一种芽孢表面稳定展示海藻糖合酶的工程菌及其构建方法与流程
本发明涉及一种芽孢表面稳定展示海藻糖合酶的工程菌及其构建方法,特别涉及一种芽孢表面稳定展示海藻糖合酶的枯草芽孢杆菌工程菌及其构建方法,属于生物工程技术领域。
背景技术:
枯草芽孢杆菌(Bacillus subtilis)是一类好氧型、内生抗逆孢子的杆状细菌,是革兰氏阳性细菌的典型代表,具有无治病性,表达系统稳定的特点;其细胞壁的组成简单,在细胞壁合成过程中没有内毒素的生成,已经被美国、日本、欧盟和中国认定为安全无毒的菌株,广泛应用于食品、饲料等行业。芽孢(内生孢子或称孢子)是芽孢杆菌营养细胞受到营养缺乏,代谢产物积累,或温度变化等外部环境因子的触发而形成的休眠体,芽孢的形成需要经过一系列复杂的生理生化反应。
近年来以芽孢衣壳蛋白为分子载体,通过芽孢表面展示技术将外源目的蛋白展示于重组枯草芽孢杆菌芽孢表面,制备具有生物活性的重组芽孢抗体和酶制剂已成为人们研究和关注的热点。构建重组枯草芽孢杆菌的芽孢表面展示体系时,利用芽孢衣壳蛋白融合展示海藻糖合酶,将底物麦芽糖转化为海藻糖,不仅能够避免因诱导剂的使用而造成限制,而且融合酶不需要穿过细胞膜,将海藻糖合酶直接应用于高端海藻糖的生产制备中,避免因细胞破碎,杂质分离造成的成本增加及热源物质对海藻糖的污染,该法是海藻糖生产的有效技术手段。但是由于芽孢易受萌发剂刺激而萌发,使展示芽孢在转化麦芽糖制备海藻糖体系中稳定性较差,海藻糖合酶异源表达后,只能利用一次,不能充分回收利用。
技术实现要素:
本发明主要针对现有技术的不足,提供一种芽孢表面稳定展示海藻糖合酶的工程菌及其构建方法。
本发明技术方案如下:
一种芽孢表面稳定展示海藻糖合酶的枯草芽孢杆菌工程菌,使枯草芽孢杆菌中编码芽孢萌发的皮层溶解酶基因sleB和cwlJ失活后获得;
所述sleB基因核苷酸序列如SEQ ID NO.1所示;cwlJ基因核苷酸序列如SEQ ID NO.2所示。
根据本发明优选的,所述编码芽孢萌发的皮层溶解酶基因sleB和cwlJ失活为通过基因敲除、基因增补、基因替换或基因突变引起编码内切纤维素酶基因celB的启动子、终止子或编码区的变化导致编码芽孢萌发的皮层溶解酶基因sleB和cwlJ无法表达。
上述芽孢表面稳定展示海藻糖合酶的枯草芽孢杆菌工程菌的构建方法,步骤如下:
(i)以枯草芽孢杆菌为模板,进行PCR扩增,分别得到sleB1,cwlJ1片段;
sleB1片段扩增的PCR引物序列如下:
sleB-up:5’-CGGGATCCCGGGGGATGATGTGGTCGAG-3’; SEQ ID NO.3
sleB-down:5’-ACTGACTCACTCAAAAATAACCCCCGCTACT-3’; SEQ ID NO.4
cwlJ1片段扩增的PCR引物序列如下:
cwlJ-up:5’-CGGGATCCCGTTCTGAAGTAATGAAATATGATG-3’; SEQ ID NO.5
cwlJ-down:5’-CTATGGTGTGTGGGAAAAGCAGTGGACTTAAATCT-3’; SEQ ID NO.6
(ii)以质粒pPIC9k为模板,进行PCR扩增,得到kmr片段;
所述的kmr片段PCR扩增引物序列如下:
kmr-up:5’-CGGGGGTTATTTTTGAGTGAGTCAGTCATAGGGAG-3’; SEQ ID NO.7
kmr-down:5’-CGGGATCCCGGGTTGAGGCCGTTGAGCA-3’; SEQ ID NO.8
(iii)以pPICZαA质粒为模板,进行PCR扩增,得到zeor片段;
所述的zeor片段PCR扩增引物序列如下:
zeor-up:5’-TAAGTCACACTGCTTTTCCCACACACCATAGCTTCA-3’; SEQ ID NO.9
zeor-down:5’-CGGGATCCCGGTTGGTCTCCAGCTTGCAAA-3’; SEQ ID NO.10
(iv)利用重叠PCR技术连接sleB1和kmr片段,制得sleB1-kmr片段,步骤如下:
初次PCR扩增,扩增体系如下:
2×Taq PCR MasterMix 12.5μl,模板sleB1 1μl,模板kmr 1μl,用ddH2O补足25μl;
初次PCR扩增,扩增程序如下:
95℃预变性5min;94℃变性30sec,57℃退火30sec,72℃延伸3.5min,5个循环;
补充PCR扩增,扩增体系如下:
向初次扩增后的体系中加入2×Taq PCR MasterMix 12.5μl,10μmol/L引物sleB-up 1μl,10μmol/L引物kmr-down 1μl,用ddH2O补足50μl;
补充PCR扩增,扩增程序如下:
95℃预变性5min;94℃变性30sec,56℃退火30sec,72℃延伸4.5min,30个循环;72℃延伸10min,-20℃保存;
(v)利用重叠PCR技术连接cwlJ1和zeor片段,制得cwlJ1-zeor片段,步骤如下:
初次PCR扩增,扩增体系如下:
2×Taq PCR MasterMix 12.5μl,模板cwlJ1 1μl,模板zeor 1μl,用ddH2O补足25μl;
初次PCR扩增,扩增程序如下:
95℃预变性5min;94℃变性30sec,57℃退火30sec,72℃延伸3min,5个循环;
补充PCR扩增,扩增体系如下:
向初次扩增后的体系中加入2×Taq PCR MasterMix 12.5μl,10μmol/L引物cwlJ-up 1μl,10μmol/L引物zeor-down 1μl,用ddH2O补足50μl;
补充PCR扩增,扩增程序如下:
95℃预变性5min;94℃变性30sec,56℃退火30sec,72℃延伸4min,30个循环;72℃延伸10min,-20℃保存;
(vi)将步骤(iv)制得的sleB1-kmr片段转化枯草芽孢杆菌获得sleB基因缺失菌后,再将步骤(v)制得的cwlJ1-zeor基因转化sleB基因缺失菌中获得sleB,cwlJ基因双缺失菌株,经筛选,制得芽孢表面稳定展示海藻糖合酶的枯草芽孢杆菌。
根据本发明优选的,所述步骤(i)中,sleB1片段扩增的PCR扩增体系为50μl:
2×Taq PCR MasterMix 25μl,10μmol/L引物sleB-up 2.5μl,10μmol/L引物sleB-down 2.5μl,模板2.5μl,用ddH2O补足50μl;
sleB1片段扩增的PCR扩增程序如下:
95℃预变性5min;95℃变性30sec,56℃退火30sec,72℃延伸1.5min,30个循环;72℃延伸10min,-20℃保存。
根据本发明优选的,所述步骤(i)中,cwlJ1片段扩增的PCR扩增体系为50μl:
2×Taq PCR MasterMix 25μl,10μmol/L引物cwlJ-up 2.5μl,10μmol/L引物cwlJ-down 2.5μl,模板2.5μl,用ddH2O补足50μl;
cwlJ1片段扩增的PCR扩增程序如下:
95℃预变性5min;95℃变性30sec,56℃退火30sec,72℃延伸1.5min,30个循环;72℃延伸10min,-20℃保存。
根据本发明优选的,所述步骤(ii)中,所述的kmr片段PCR扩增体系为50μl:
2×Taq PCR MasterMix 25μl,10μmol/L引物kmr-up 2.5μl,10μmol/L引物kmr-down 2.5μl,模板2.5μl,用ddH2O补足50μl;
所述的kmr片段PCR扩增程序如下:
95℃预变性5min;95℃变性30sec,56℃退火30sec,72℃延伸3.5min,30个循环;72℃延伸10min,-20℃保存。
根据本发明优选的,所述步骤(iii)中,所述的zeor片段PCR扩增体系为50μl:
2×Taq PCR MasterMix 25μl,10μmol/L引物zeor-up 2.5μl,10μmol/L引物zeor-down 2.5μl,模板2.5μl,用ddH2O补足50μl;
所述的zeor片段PCR扩增程序如下:
95℃预变性5min;95℃变性30sec,56℃退火30sec,72℃延伸3min,30个循环;72℃延伸10min,-20℃保存。
根据本发明优选的,所述步骤(vi)中的枯草芽孢杆菌为枯草芽孢杆菌168。枯草芽孢杆菌168来源于杭州宝赛生物有限公司。
根据本发明优选的,所述步骤(vi)中的转化为:将枯草芽孢杆菌感受态细胞在2500V、25uF的条件下电击一次,时间常数=4.5~5.0ms。
根据本发明优选的,所述步骤(vi)中的筛选,包括卡那青霉素筛选和博来霉素筛选。
根据本发明进一步优选的,所述卡那青霉素筛选步骤如下:
在含有卡那青霉素抗生素的平板上进行白斑筛选,挑取白色单一斑点,接种到含有卡那青霉素的液体LB培养基,培养至对数末期,对能在含有卡那青霉素抗生素的LB培养基上生长的菌液进行PCR验证,将能够扩增出目的条带的转化子进行提取质粒,对提取的质粒进行酶切验证,含有目的条带的,即得;
含有卡那青霉素的液体LB培养基,每升组分如下:
10g蛋白胨、10g NaCl、5g酵母浸粉、5mg卡那青霉素,定容至1L。
根据本发明进一步优选的,所述博来霉素筛选步骤如下:
在含有博来霉素抗生素的平板上进行白斑筛选,挑取白色单一斑点,接种到含有博来霉素的液体LB培养基,培养至对数末期,对能在含有博来霉素抗生素的LB培养基上生长的菌液进行PCR验证,将能够扩增出目的条带的转化子进行提取质粒,对提取的质粒进行酶切验证,含有目的条带的,即得;
含有博来霉素的液体LB培养基,每升组分如下:
10g蛋白胨、10g NaCl、5g酵母浸粉、5mg博来霉素,定容至1L。
有益效果
1、本发明首次通过失活芽孢萌发的皮层溶解酶基因sleB和cwlJ弱化芽孢萌发的方式提高芽孢表面展示海藻糖合酶的稳定性,经实际验证,可有效提高海藻糖合酶的酶活,12h内海藻糖合酶酶活可保持95%以上;
2、本发明所述的枯草芽孢杆菌工程菌表达体系具有安全、无内毒素的特点,在食品与医药产业化领域倍受认可;
3、本发明构建的芽孢表面稳定展示海藻糖合酶的枯草芽孢杆菌工程菌,既不影响菌株本身形成芽孢,同时还弱化了芽孢在麦芽糖转化生成海藻糖体系中的萌发。
附图说明
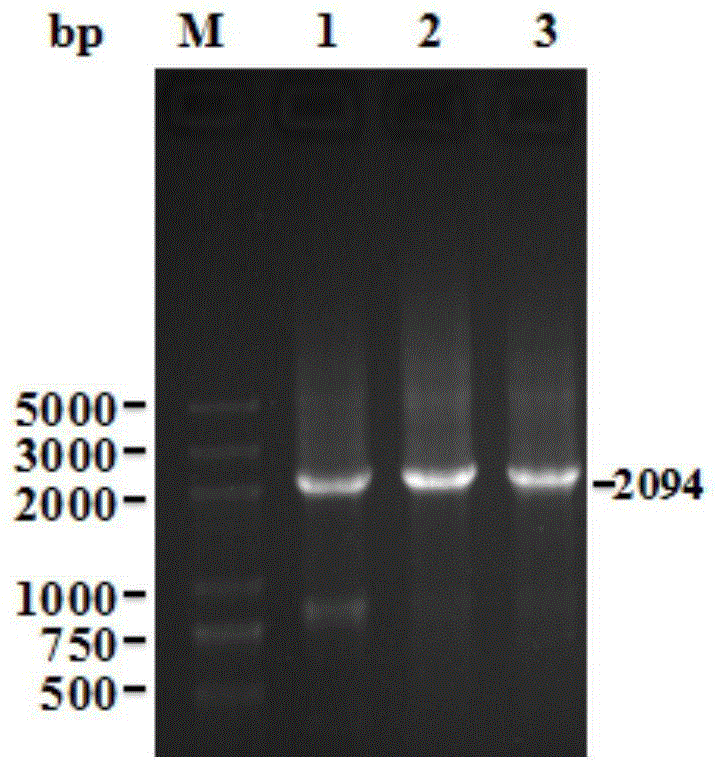
图1为本发明的sleB基因敲除转化子的琼脂糖凝胶电泳图;
图中:泳道M为DNA分子量标记(DNA marker),泳道1-3为转化子条带,大小为2094bp;
图2为本发明的cwlJ基因敲除转化子的琼脂糖凝胶电泳图;
图中:泳道M为DNA分子量标记(DNA marker),泳道1-3为转化子条带,大小为1656bp;
图3为本发明的麦芽糖转化生成海藻糖的高效液相色谱;
图中:M为麦芽糖,T为海藻糖糖,A为Bacillus Subtilis 168转化麦芽糖生成海藻糖色谱,B为Bacillus Subtilis 168ΔsleBΔcwlJ工程菌转化麦芽糖生成海藻糖色谱;
图4为本发明的芽孢表面稳定展示海藻糖合酶的枯草芽孢杆菌海藻糖酶相对酶活;
图中:芽孢表面稳定展示海藻糖合酶的枯草芽孢杆菌的海藻糖合酶的相对酶活在12h内可保持95%以上,较枯草芽孢杆菌原始菌有显著提高。
具体实施方式
下面结合实施例对本发明的技术方案做进一步阐述,但本发明所保护范围不限于此。
生物材料来源:
枯草芽孢杆菌168、穿梭质粒pPIC9k和pPICZαA均购自杭州宝赛生物有限公司;
实施例1:基因敲除片段构建
(i)提取Bacillus Subtilis 168DNA,以DNA为模板,进行PCR扩增,得到同源臂sleB1;
所述的PCR引物序列如下:
sleB-up:5’-CGGGATCCCGGGGGATGATGTGGTCGAG-3’;
sleB-down:5’-ACTGACTCACTCAAAAATAACCCCCGCTACT-3’;
所述的PCR扩增体系为:
所述的PCR扩增程序如下:
95℃预变性5min;94℃变性30sec,56℃退火30sec,72℃延伸1.5min,30个循环;72℃终延伸10min,4℃保存;
琼脂糖凝胶电泳检验PCR产物,长度为592bp,使用SanPrep柱式DNA胶回收试剂盒(上海生工)进行胶回收,回收产物-20℃保存,备用;
(ii)提取Bacillus Subtilis 168 DNA,以DNA为模板,进行PCR扩增,得到同源臂cwlJ1;
所述的PCR引物序列如下:
cwlJ-up:5’-CGGGATCCCGTTCTGAAGTAATGAAATATGATG-3’;
cwlJ-down:5’-CTATGGTGTGTGGGAAAAGCAGTGGACTTAAATCT-3’;
所述的PCR扩增体系为:
所述的PCR扩增程序如下:
95℃预变性5min;94℃变性30sec,56℃退火30sec,72℃延伸1min,30个循环;72℃终延伸10min,4℃保存;
琼脂糖凝胶电泳检验PCR产物,长度为451bp,使用SanPrep柱式DNA胶回收试剂盒(上海生工)进行胶回收,回收产物-20℃保存,备用;
(iii)提取穿梭质粒pPIC9k的DNA,以DNA为模板,进行PCR扩增,得到kmr片段;
所述的PCR引物序列如下:
kmr-up:5’-CGGGGGTTATTTTTGAGTGAGTCAGTCATAGGGAG-3’;
kmr-down:5’-CGGGATCCCGGGTTGAGGCCGTTGAGCA-3’;
所述的PCR扩增体系为:
所述的PCR扩增程序如下:
95℃预变性5min;94℃变性30sec,56℃退火30sec,72℃延伸3.5min,30个循环;72℃终延伸10min,4℃保存;
琼脂糖凝胶电泳检验PCR产物,长度为1502bp,使用SanPrep柱式DNA胶回收试剂盒(上海生工)进行胶回收,回收产物-20℃保存,备用;
(iv)提取穿梭质粒pPICZαA的DNA,以DNA为模板,进行PCR扩增,得到zeor片段;
所述的PCR引物序列如下:
zeor-up:5’-TAAGTCACACTGCTTTTCCCACACACCATAGCTTCA-3’;
zeor-down:5’-CGGGATCCCGGTTGGTCTCCAGCTTGCAAA-3’;
所述的PCR扩增体系为:
所述的PCR扩增程序如下:
95℃预变性5min;94℃变性30sec,57℃退火30sec,72℃延伸3min,30个循环;72℃终延伸10min,4℃保存;
琼脂糖凝胶电泳检验PCR产物,长度为1205bp,使用SanPrep柱式DNA胶回收试剂盒(上海生工)进行胶回收,回收产物-20℃保存,备用;
(v)将步骤(i)制得的sleB1片段与步骤(iii)制得的kmr片段进行重叠PCR,制得sleB1-kmr片段;
所述的重叠PCR的扩增体系为:
所述的重叠PCR的初次扩增程序如下:
95℃预变性5min;94℃变性30sec,57℃退火30sec,72℃延伸3.5min,5个循环;72℃终延伸10min;
所述的重叠PCR的补充扩增程序如下:
95℃预变性5min;94℃变性30sec,56℃退火30sec,72℃延伸4.5min,30个循环;72℃终延伸10min,4℃保存;
琼脂糖凝胶电泳检验PCR产物,长度为2094bp,使用SanPrep柱式DNA胶回收试剂盒(上海生工)进行胶回收,回收产物-20℃保存,备用;
(vi)将步骤(ii)制得的cwlJ1片段与步骤(iv)制得的zeor片段进行重叠PCR,制得cwlJ1-zeor片段;
所述的重叠PCR的扩增体系为:
所述的重叠PCR的初次扩增程序如下:
95℃预变性5min;94℃变性30sec,57℃退火30sec,72℃延伸3min,5个循环;72℃终延伸10min;
所述的重叠PCR的补充扩增程序如下:
95℃预变性5min;94℃变性30sec,56℃退火30sec,72℃延伸4min,30个循环;72℃终延伸10min,4℃保存;
琼脂糖凝胶电泳检验PCR产物,长度为1656bp,使用SanPrep柱式DNA胶回收试剂盒(上海生工)进行胶回收,回收产物-20℃保存,备用;
实施例2:制备Bacillus Subtilis 168感受态
(i)挑取新鲜LB固体培养基表面的Bacillus Subtilis 168单菌落,接种于10mL GM培养基中,37℃、220r/min,过夜培养;
(ii)取1mL上述菌液转接到100mLGM培养基,37℃、220r/min培养至OD600=1.0;
(iii)将菌液转移至100mL离心管,冰浴10min,使菌体停止生长;
(iv)冰浴后4℃、7000r/min、5min离心,收集菌体;
(v)离心后的菌体用预冷的电转缓冲液(ETM)洗涤2-3次;
(vi)洗涤结束后,使用1000μL电转缓冲液重悬菌体;
(vii)将制备好的感受态细胞分装100μL每管,-80℃保存,备用。
其中,GM培养基:LB培养基+0.5M山梨醇;
ETM:0.5M山梨醇+0.5M甘露醇+10%甘油,余量水;
RM培养基:LB培养基+0.5M山梨醇+0.38M甘露醇;
实施例3:sleB1-kmr片段电转化Bacillus Subtilis 168及阳性重组子的鉴定
(i)将sleB1-kmr片段用限制性内切酶BamH I酶切;
酶切体系(40μL)如下:
(ii)浓缩纯化酶切产物
(1)加入1/10体积3M醋酸钠和2.5倍体积无水乙醇,置于-20℃冰箱20min;
(2)12000r/min,离心5min得沉淀;
(3)300μL体积百分比浓度为75%的乙醇溶液重悬沉淀;
(4)12000r/min,离心5min,除去乙醇,37℃风干30min;
(5)加入15~18μL ddH2O重悬DNA,并置于-20℃保存。
(iii)电转化
首先利用核酸超微量分光光度计测定sleB1-kmr片段浓度,达到100~400ng/uL浓度后进行电转化,电转化条件为电压2000V,电击时间5ms,将得到的细胞经液体复苏培养基在37℃复苏培养4h,取200μL涂布在含浓度25μmol/mL卡那霉素的LB固体培养基上,在37℃培养1~2天,筛选具有卡那霉素抗性的转化子;
所述液体复苏培养基每升组分如下:
蛋白胨10g,酵母浸粉5g,氯化钠10g,山梨醇91g,甘露醇69g,蒸馏水1000mL。
(iv)挑取上述阳性重组菌落,接种到含卡那霉素抗性的液体LB培养基中37℃培养过夜,培养完成后,使用上海生物工程有限公司提供的试剂盒提取重组菌DNA,并以获得的基因组为模板,sleBF和kmR为引物进行PCR扩增,扩增产物利用琼脂糖凝胶电泳进行验证;
所述的PCR引物序列如下:
sleB-up:5’-CGGGATCCCGGGGGATGATGTGGTCGAG-3’
kmr-down:5’-CGGGATCCCGGGTTGAGGCCGTTGAGCA-3’
其中,下划线标识的为酶切位点
所述的PCR扩增体系为20μl:
所述的PCR扩增程序如下:
95℃预变性5min;94℃变性30sec,56℃退火30sec,72℃延伸3.5min,30个循环;72℃延伸10min,4℃保存,琼脂糖凝胶电泳检验PCR产物。
实施例4cwlJ1-zeor片段电转化Bacillus Subtilis 168ΔsleB及阳性重组子的鉴定
(i)按照实施例2中的步骤制备Bacillus Subtilis 168ΔsleB感受态
(ii)将cwlJ1-zeor片段用限制性内切酶BamH I酶切;酶切体系(40μL)如下:
(iii)按照实施例3(ii)中的步骤浓缩纯化酶切产物
(iv)电转化
首先利用核酸超微量分光光度计测定cwlJ1-zeor片段浓度,达到100~400ng/uL浓度后进行电转化,电转化条件为电压2000V,电击时间5ms,将得到的细胞经液体复苏培养基在37℃复苏培养4h,取200μL涂布在含浓度25μmol/mL卡那霉素和50μmol/mL博来霉素的LB固体培养基上,在37℃培养1~2天,筛选具有卡那霉素和博来霉素抗性的转化子;
所述液体复苏培养基每升组分如下:
蛋白胨10g,酵母浸粉5g,氯化钠10g,山梨醇91g,甘露醇69g,蒸馏水1000mL。
(v)挑取上述阳性重组菌落,分别依次接种到含卡那霉素和博来霉素抗性的液体LB培养基中37℃培养过夜,培养完成后,使用上海生物工程有限公司提供的试剂盒提取重组菌DNA,并以获得的基因组为模板,cwlJF和zeoR为引物进行PCR扩增,扩增产物利用琼脂糖凝胶电泳进行验证;
所述的PCR引物序列如下:
cwlJ-up:5’-CGGGATCCCGTTCTGAAGTAATGAAATATGATG-3’
zeor-down:5’-CGGGATCCCGGTTGGTCTCCAGCTTGCAAA-3’
其中,下划线标识的为酶切位点
所述的PCR扩增体系为20μl:
所述的PCR扩增程序如下:
95℃预变性5min;94℃变性30sec,56℃退火30sec,72℃延伸3min,30个循环;72℃终延伸10min,4℃保存,琼脂糖凝胶电泳检验PCR产物。
最终制得芽孢表面稳定展示海藻糖合酶的枯草芽孢杆菌。
实施例5:芽孢表面稳定展示海藻糖合酶的枯草芽孢杆菌在麦芽糖转化生成海藻糖体系中的应用:
(1)将芽孢表面稳定展示海藻糖合酶的枯草芽孢杆菌接种于活化培养基中于37℃下活化培养3~4h,然后按体积百分比为1%的比例接种于装液量为30~50mL三角瓶,其中抗生素浓度为25mg/mL,培养条件为35~37℃、200~220rpm、10~14h,得到Bacillus Subtilis 168ΔsleBΔcwlJ的菌液;
活化培养基配方如下,单位g/L:
蛋白胨9~11、酵母浸粉5~7、氯化钠9~11,蒸馏水定容,pH7.0~7.4;121℃灭菌20~30min;
(2)将步骤(1)中得到Bacillus Subtilis 168ΔsleBΔcwlJ的菌液按体积百分比2~4%的接种量接种于80~100mL发酵培养基中,在35~37℃、180~220rpm的条件下摇瓶发酵48h,得到发酵液;
摇瓶发酵培养基组分如下,单位g/L:
葡萄糖5~10、玉米浆10~15、磷酸二氢钾2~3、硫酸锰0.3~0.5、结晶硫酸镁2~3、氯化钠4~6,蒸馏水定容;115℃灭菌20~30min;
(3)芽孢表面稳定展示海藻糖合酶的枯草芽孢杆菌在麦芽糖转化生成海藻糖体系中海藻糖合酶酶活检测
将发酵液8000r/min离心20min,弃上清,用去离子水洗涤沉淀2次,并用去离子水悬浮,加入溶菌酶使其终浓度为2mg/mL,37℃处理30min破坏营养细胞,8000r/min离心10min,烘干,称量干菌体重量,最后用去离子水悬浮得到芽孢悬浮液。取5ml发酵48h后的重组菌芽孢悬浮液与10ml pH 7.5的磷酸盐缓冲液配制的终浓度30%的麦芽糖浆混匀后制备成海藻糖合酶的反应体系。将此反应体系置于50℃恒温水浴震荡摇床中反应12h,每隔4h取样,测定海藻糖合酶的酶活。
酶活力单位定义:在酶的最佳酶反应条件下(25℃,pH 8.0),将300g/L麦芽糖转化为海藻糖,每小时产生1μmol海藻糖所需的酶量定义一为个酶活单位(U)。
经实验证明原始菌的相对酶活在4h后酶活就下降到65%,10h后稳定在50%,而本专利中构建的芽孢表面稳定展示海藻糖合酶的枯草芽孢杆菌海藻糖合酶的相对酶活在12h内可稳定在95%以上,与原始菌相比海藻糖合酶酶活有显著提高。
SEQUENCE LISTING
<110> 齐鲁工业大学
<120> 一种芽孢表面稳定展示海藻糖合酶的工程菌及其构建方法
<160> 10
<170> PatentIn version 3.5
<210> 1
<211> 874
<212> DNA
<213> Bacillus Subtilis
<400> 1
atgaagtcca aaggatcgat tatggcatgt ctcatccttt tttctactga aacgatccat 60
tcaaagaggg gcaacagggg atgatgtggt cgagcttcag gcgcgtcttc aatacaacgg 120
atattataac ggaaaaattg acggggttta tggatggggg acgtactggg cagttcgaaa 180
ttttcaggat caattcgggt taaaagaggt tgacggcctt gtaggagcta aaacaaagca 240
aaccttaata tgtaaatcaa aatactatcg tgaatatgtc atggaacagc tcaataaagg 300
gaatacattc acgcattacg gaaaaattcc gctaaagtat cagacgaaac catcaaaagc 360
agcaacacaa aaggcaagac aacaagcaga agcacggcag aaacagcctg cggaaaaaac 420
aacgcagaag cctaaagcga atgcgaataa acagcaaaac aatacaccag caaaagcaag 480
aaaacaggat gcggtagcag cgaacatgcc tggtggattt tccaacaacg atatcaggct 540
gcttgctcaa gcggtttatg gcgaagcccg gggcgagccg tacgaggggc aggttgctat 600
tgcagcagtc attttaaacc gtttgaacag cccgttattt ccaaattcag tagcgggggt 660
tatttttgag ccgcttgcct tcacagcagt agccgacgga caaatttaca tgcagccgaa 720
tgaaacggca cgagaagcag tgctggatgc catcaatggc tgggacccat cagaggaagc 780
actttactac tttaatccgg atacggctac aagtccgtgg atttgggggc gtccgcagat 840
taaaagaatc ggtaaacaca ttttctgtga gtag 874
<210> 2
<211> 429
<212> DNA
<213> Bacillus Subtilis
<400> 2
atggcggtcg tgagagcaac gagtgcggat gtcgatttga tggcaaggct gctcagagcg 60
gaagcggaag gcgaaggcaa gcaggggatg ctgcttgtcg gcaacgttgg aattaatcgg 120
ctgcgggcga attgctcaga ttttaaaggc ctccgcacca tcaggcagat gatttatcag 180
ccacacgcgt ttgaggctgt gactcatgga tatttttatc aaagggcgcg agatagcgag 240
cgtgcccttg cacgccggtc gattaatggt gaaaggcgct ggcctgcaaa atttagttta 300
tggtacttca ggccgcaggg ggactgtcca gcccagtggt ataaccagcc gtttgtggcc 360
agatttaagt cacactgctt ttatcagccg acggcggaga cgtgtgaaaa tgtatataac 420
acattttag 429
<210> 3
<211> 28
<212> DNA
<213> 人工合成
<400> 3
cgggatcccg ggggatgatg tggtcgag 28
<210> 4
<211> 31
<212> DNA
<213> 人工合成
<400> 4
actgactcac tcaaaaataa cccccgctac t 31
<210> 5
<211> 33
<212> DNA
<213> 人工合成
<400> 5
cgggatcccg ttctgaagta atgaaatatg atg 33
<210> 6
<211> 35
<212> DNA
<213> 人工合成
<400> 6
ctatggtgtg tgggaaaagc agtggactta aatct 35
<210> 7
<211> 35
<212> DNA
<213> 人工合成
<400> 7
cgggggttat ttttgagtga gtcagtcata gggag 35
<210> 8
<211> 28
<212> DNA
<213> 人工合成
<400> 8
cgggatcccg ggttgaggcc gttgagca 28
<210> 9
<211> 36
<212> DNA
<213> 人工合成
<400> 9
taagtcacac tgcttttccc acacaccata gcttca 36
<210> 10
<211> 30
<212> DNA
<213> 人工合成
<400> 10
cgggatcccg gttggtctcc agcttgcaaa 30
- 还没有人留言评论。精彩留言会获得点赞!