一种重组枯草芽孢杆菌产透明质酸裂解酶的方法与流程
本发明涉及一种重组枯草芽孢杆菌产透明质酸裂解酶的方法,属于生物工程技术领域。
背景技术:
透明质酸酶(hyaluronidase)是对催化降解透明质酸酶的通称。透明质酸酶分布较广,在蛇毒、蜂毒、蝎毒以及蜘蛛等的毒液中都检测到了透明质酸酶。在人体组织及体液中也发现广泛存在着透明质酸酶。此外,一些细菌、真菌以及非脊椎动物如水蛭、甲壳类动物等也产生透明质酸酶。1971年,Meyer根据透明质酸酶作用机制的不同,将其分为三类:第一类是内切-β-N-乙酰氨基葡糖苷酶(EC 3.2.1.35),为水解酶,作用于β-1,4糖苷键,降解终产物主要为四糖,也可作用于软骨素或硫酸软骨素,并有转糖苷活性。哺乳动物来源和动物毒液来源的透明质酸酶属于此类。研究最多的是睾丸、蜂毒以及溶酶体透明质酸酶;第二类是水蛭、十二指肠来源的透明质酸酶,为内切-β-葡糖苷酸酶(EC 3.2.1.36),作用于β-1,3糖苷键,也为水解酶,主要降解终产物为四糖,可特异性降解透明质酸;第三类是细菌来源的透明质酸酶(EC 4.2.2.1),此类为透明质酸裂解酶(hyaluronate lyase),作用于β-1,4糖苷键,通过β-消去机制得到4,5-不饱和双糖。
近年开始用X-射线晶体衍射法研究透明质酸酶的结构和作用机制。目前见到报道的有Streptococcus pneumoniae和S.agalactiae(B族Streptococcus,GBS)透明质酸酶。完整的细菌透明质酸酶包含4个结构域:N末端的糖结合域,间隔域,催化反应域,和C末端调节域,两个末端结构域与相邻结构域间由一段连接多肽相连。
产透明质酸酶的微生物包括很多革兰氏阴性与阳性菌,革兰氏阴性菌所产酶大多存在于周质空间,不大可能与发病机制相关。这些菌有Aeromonas,Bacteroides,Fusobacterium等。而产透明质酸酶的革兰氏阳性菌有Streptococcus,Staphylococcus,Clostridium,Propionibacterium,Peptostreptococcus以及Streptomyces。其中,对于链球菌透明质酸酶的报道很多且深入。引人注目的是,这些菌大多都是致病菌,可引起人或动物的皮肤或粘膜感染,利用产生的酶降解透明质酸,破坏其防御体系,降低粘度,便于入侵。温和噬菌体如Streptococcus pyogenes和S.equi的透明质酸酶,基因序列间有很大同源性,但与细菌透明质酸酶的序列相似性很小。噬菌体透明质酸酶以多聚体存在,且能专一性降解透明质酸,并且为非进行性降解。S.pyogenes噬菌体携带透明质酸酶穿透细菌荚膜,到达受体部位,进而感染细菌。
技术实现要素:
本发明的目的在于提供一种产透明质素裂解酶的重组枯草芽孢杆菌,是在枯草芽孢杆菌中导入编码透明质酸裂解酶的基因,诱导表达透明质酸裂解酶。
本发明的另一目的在于提供上述重组枯草芽孢杆菌在生产透明质素裂解酶中的应用。
本发明的有一目的在于提供一种生产透明质素裂解酶的方法。
本发明的目的可以通过以下技术方案实现:
一种产透明质酸裂解酶的重组枯草芽孢杆菌,是在枯草芽孢杆菌中导入编码透明质酸裂解酶的基因构建得到的,所述编码透明质酸裂解酶的基因具有如SEQ ID NO.1所示核苷酸序列,该基因重组整合在枯草芽孢杆菌基因组上,并置于木糖诱导型启动子PxylA下诱导表达。
本发明中编码所述透明质酸裂解酶的基因是来源于酿脓链球菌(Streptococcus pyogenes)噬菌体H4489A的HylP;HylP的核苷酸序列具有如SEQ ID NO.1所示的核苷酸序列。编码透明质酸裂解酶的基因HylP重组整合在基因组上进行诱导表达,启动子为木糖诱导型启动子PxylA。
所述透明质酸裂解酶的N端带有信号肽amyX和组氨酸标签6×His-tag。所述信号肽amyX的基因序列如SEQ ID NO.3所示。
在枯草芽孢杆菌中导入编码透明质酸裂解酶的基因的过程是构建含有透明质酸裂解酶基因的重组质粒,将含有透明质酸裂解酶基因的重组质粒转化枯草芽孢杆菌。优选的,上述的含有透明质酸裂解酶基因的重组质粒采用以下步骤构建:
(1)设计引物HylP-F、HylP-R,以基因合成的pUC19-HylP为模板,进行PCR扩增获得HylP基因片段;设计引物AmyX-F、AmyX-R,以质粒pMA0911-amyX为模板,进行PCR扩增获得amyX片段;
HylP-F:CACCACCACCACCACCACCGTCGGAGGTGCAGGAATTG
HylP-R:TCCCCGCGGTTACACTTTATGCTTCCGGC
AmyX-F:CGCGGATCC ATGGTCAGCATCCGCCGCAG
AmyX-R:GTGGTGGTGGTGGTGGTGCATGATTTCCTTTTGTTCAG;
(2)将HylP基因片段与amyX片段通过组氨酸标签进行两步融合PCR,PCR扩增获取的amyX-His6-HylP片段,将amyX-His6-HylP片段与质粒pAX01分别采用BamHI和SacII进行双酶切,回收产物进行连接,得到重组质粒pAX01-H6HylPA1。
所述两步融合PCR的过程为:第一步体系50μl:2μl HylP片段,2μl amyX片段,25μl PrimerStar(mix),21μl ddH2O。按照如下程序:94℃,3min;94℃,30s,55℃,30s,72℃,1min,5个循环;72℃,5min;12℃,保持。第二步体系50μl:1μl第一步PCR产物,1μl AmyX-F,1μl HylP-R,25μl PrimerStar mix,22μl ddH2O。
所述枯草芽孢杆菌为Bacillus subtilis 168。
上述的重组枯草芽孢杆菌在发酵生产透明质酸裂解酶中的应用。
一种生产透明质酸裂解酶的方法,发酵培养上述的重组枯草芽孢杆菌并采用木糖诱导透明质酸裂解酶基因表达获得透明质酸裂解酶。
所述发酵培养的培养基为以蔗糖或葡萄糖作为碳源的无机盐培养基:20g/L酵母粉,50g/L蔗糖或葡萄糖;硫酸钾3.9g/L;50mM磷酸盐缓冲液,pH 7.0。
所述发酵培养的培养条件为:在37℃下,发酵40~60h;接种后1~4h向培养基中添加2g/L的木糖诱导透明质酸裂解酶基因表达。
本发明的有益效果:
本发明利用枯草芽孢杆菌产透明质酸裂解酶,与其他产透明质酸裂解酶菌相比,具有非常大的应用优势。首先,本发明过程中使用的宿主为食品级,完全满足医疗卫生和食品安全的要求,无内毒素和病原感染的风险,不会对产物的医用食品安全带来隐患;其次,枯草芽孢杆菌培养成本简单,生产强度大。基于应用分析,本发明方法在工业上用于制备透明质酸裂解酶具有潜在而广泛的价值。
附图说明
图1为PCR和凝胶核酸电泳鉴定透明质酸裂解酶基因HylP在枯草芽孢杆菌中的整合构建。
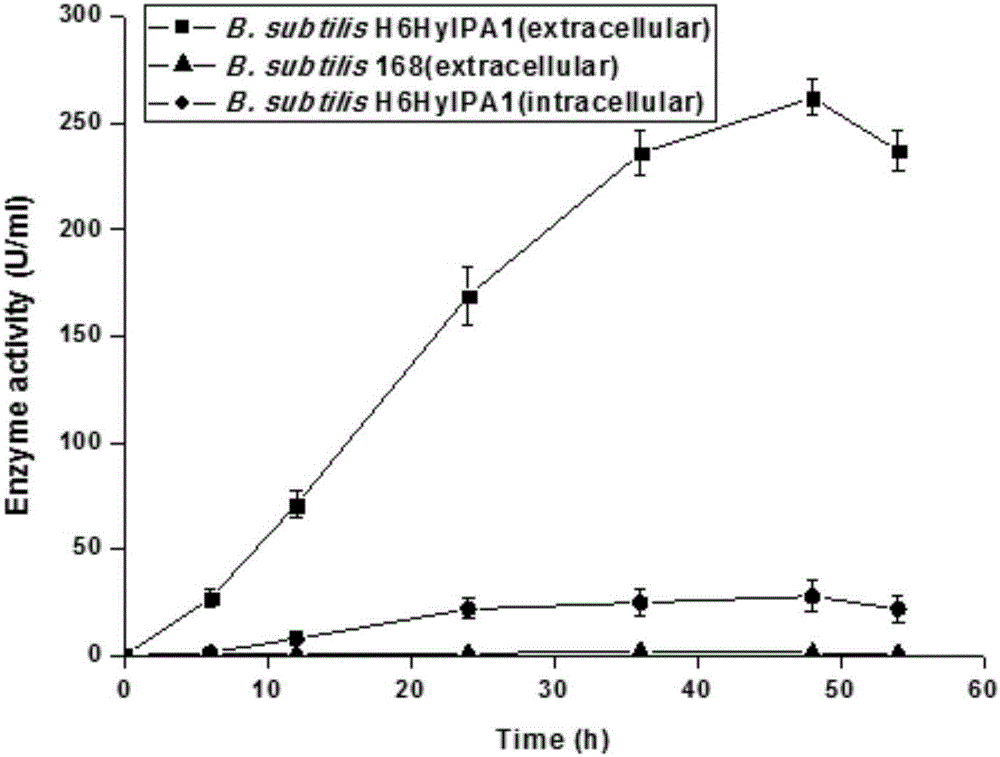
图2为枯草芽孢杆菌产透明质酸裂解酶的胞外与胞内酶活曲线(含对照菌):B.subtilis H6HylPA1胞外酶活;B.subtilis H6HylPA1胞内酶活;B.subtilis 168胞外酶活。
具体实施方式
序列表所示为本发明所述核苷酸序列信息:
(1)SEQ ID NO.1序列信息为来源于酿脓链球菌(Streptococcus pyogenes)噬菌体H4489A的透明质酸裂解酶基因HylP编码序列;
(2)SEQ ID NO.2序列信息为枯草芽孢杆菌组成型启动子PxylA的基因序列;
(3)SEQ ID NO.3序列信息为信号肽amyX的基因序列。
实施例1整合重组质粒pAX01-H6HylPA1构建
透明质酸裂解酶基因HylP来源于酿脓链球菌(Streptococcus pyogenes)噬菌体H4489A,基因合成HylP基因,根据已公布的基因信息序列,设计引物HylP-F、HylP-R,以基因合成的pUC19-HylP(将如SEQ ID NO.1所示的透明质酸裂解酶基因HylP克隆至pUC19载体得到,由南京拓达生物科技有限公司合成)为模板,采用标准的PCR扩增体系和程序,扩增获取目标基因。
引物序列信息:5’-3’方向
HylP-F:CACCACCACCACCACCACCGTCGGAGGTGCAGGAATTG
HylP-R:TCCCCGCGGTTACACTTTATGCTTCCGGC
AmyX-F:CGCGGATCCATGGTCAGCATCCGCCGCAG
AmyX-R:GTGGTGGTGGTGGTGGTGCATGATTTCCTTTTGTTCAG
在HylP基因上下游两端分别引入组氨酸标签6×His-tag和SacII限制性酶切位点,PCR扩增获取的HylP片段,采用琼脂糖凝胶核酸电泳进行切胶回收。在信号肽amyX基因上下游两端分别引入BamHI限制性酶切位点和组氨酸标签6×His-tag,以质粒pMA0911-amyX为(购自南京拓达生物科技有限公司,该质粒上含有amyX信号肽,可以此为模板进行PCR扩增,SEQ ID NO.3所示的基因序列即为该质粒上amyX信号肽的序列)模板,PCR扩增获取的amyX片段,采用琼脂糖凝胶核酸电泳进行切胶回收。HylP片段与amyX片段通过组氨酸标签进行两步融合PCR,第一步体系50μl:2μl HylP片段,2μl amyX片段,25μl PrimerStar(mix),21μl ddH2O。按照如下程序:94℃,3min;94℃,30s,55℃,30s,72℃,1min,5个循环;72℃,5min;12℃,保持。第二步体系50μl:1μl第一步PCR产物,1μl AmyX-F,1μl HylP-R,25μl PrimerStar mix(Takara),22μl ddH2O。PCR扩增获取的amyX-His6-HylP片段,采用琼脂糖凝胶核酸电泳进行切胶回收,与质粒pAX01分别采用BamHI和SacII进行双酶切,回收产物进行连接,体系10μl:1μl双切的载体,4μl双切的目的片段,5μl Solution I连接酶,16℃连接过夜,转化JM109感受态细胞,挑取单菌落PCR验证,阳性重组子进行测序,比对正确,重组质粒pAX01-H6HylPA1构建成功。重组质粒pAX01-H6HylPA1转化Bacillus subtilis 168,以20μg/ml的红霉素平板进行筛选整合重组子,并对重组菌株进行PCR验证和测序验证,对成功整合pAX01-H6HylPA1的枯草芽孢杆菌菌株命名为Bacillus subtilis H6HylPA1。
实施例2重组枯草芽孢杆菌菌株的摇瓶发酵
挑取上述构建的重组枯草芽孢杆菌B.subtilis H6HylPA1,单克隆接种于5ml LB培养基,置于200rpm 37℃过夜培养。16h后接种于250ml三角摇瓶(装液量25ml)中,发酵培养基为无机盐培养基:20g/L酵母粉,50g/L蔗糖;硫酸钾3.9g/L;50mM磷酸盐缓冲液,pH 7.0。按10%(v/v)的接种量转接于摇瓶,置于200rpm 37℃培养,在接种后第2h添加2g/L的木糖进行诱导HylP基因表达,发酵培养54h,并定期取样,分别为第6h,12h,24h,36h,48h,54h。
发酵液在8000×g下4℃离心5min。发酵液上清转移置另一离心管中,测定胞外上清中的粗酶活。另外,取2OD各取样时间的菌体样品,通过20mg/ml浓度的溶菌酶处理,37℃,20min,除去菌体细胞壁。8000×g下4℃离心5min除去溶菌酶,加入0.5ml 100mM醋酸钠缓冲液和5mM CaCl2溶液(pH 6.0)重悬细胞,使用fastprep震荡破碎裂解细胞,测定胞内酶活,与同时期胞外酶活比较。透明质酸底物浓度为0.1mg/ml,以100mM醋酸钠缓冲液和5mM CaCl2溶液(pH 6.0)溶解底物。酶活测定的反应体系为0.5ml,包括0.2%(w/v,g/ml)底物和一定量的粗酶液。在30℃反应20min后,加入2ml的5M HCl终止反应。通过分光光度计测定在235nm处的吸光值。酶活单位定义为:产生不饱和透明质酸寡糖的蛋白量等同于使用在235nm处消光系数为5500M-1cm-1的1μmol不饱和透明质酸双糖。
从附图1看出,利用菌落PCR,通过HylP基因上下游引物HylP-F/R扩增基因全长。图中泳道11为5000DL Marker,依据泳道2-10和12-19目的条带位置,可判断所获得的条带大小为3222bp,同时泳道1对照菌B.subtilis 168中未见相同目的条带,因此证明pAX01-H6HylPA1重组质粒已成功整合至B.subtilis 168基因组,表达框PxylA-amyX-His6-HylP构建成功。
从附图2看出,重组枯草芽孢杆菌B.subtilis H6HylPA1的胞外酶活随时间延长而逐渐增加,第48h时达到最高,为262U/ml,此后酶活降低。相应地,将枯草芽孢杆菌菌体破碎,测定胞内酶活,发现菌体内仍存在少量酶活,证明信号肽amyX与透明质酸裂解酶HylP相匹配,可将大部分酶转运至胞外。对照菌B.subtilis 168的胞外几乎没有酶活,证明只有当HylP基因存在时,才能产生透明质酸裂解酶。
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
序列表
<110> 颜萌
<120> 一种重组枯草芽孢杆菌产透明质酸裂解酶的方法
<160> 7
<170> PatentIn version 3.3
<210> 1
<211> 3222
<212> DNA
<213> 酿脓链球菌(Streptococcus pyogenes)噬菌体
<400> 1
cgtcggaggt gcaggaattg gcccttgaga tggcttataa agatgtggtt gacggtaata 60
atgccacgat agcagggcag tggtcagaca gcccgcaaat gattttagat ggaggtagtt 120
aatgactgaa aatataccat taagagtcca atttaagcgc atgagcgctg atgagtgggc 180
tcgtagtgat gtcatcttgc ttgagggtga gataggtttt gagactgaca ctggttttgc 240
taagtttggc gatggtcaaa acacttttag taagcttaag taccttactg gtcccaaagg 300
tcctaaagga gacactggtc tccaaggtaa aactggagga actggtcctc ggggccctgc 360
tggcaagcct ggaacgacag attatgatca actccaaaat aaaccagatc taggtgcgtt 420
tgcacaaaaa gaagaaacta atagtaaaat caccaaatta gaatcaagca aagcagataa 480
aagcgctgtt tactcaaaag cagagtcaaa aatagagcta gacaaaaaat tgagcttaac 540
aggcggcata gtgacaggac aactacagtt taaacctaat aaaagtggta ttaaaccctc 600
atcttccgta ggaggagcga ttaacattga tatgtctaaa tcggaaggtg ctgctatggt 660
gatgtataca aataaagata ctactgatgg accattgatg attttacgtt ctgacaaaga 720
tacgtttgat cagtcagctc aatttgtgga ttacagcggt aagactaatg ctgtaaatat 780
tgtaatgcgc cagccaagcg cacctaattt ttcctcggca cttaatataa ccagtgccaa 840
cgaaggcggt agtgcgatgc aaattagagg cgtcgaaaaa gcgctaggaa cgctcaaaat 900
cacacacgaa aacccaaacg ttgaggcaaa atacgatgaa aacgctgcag cgttatctat 960
tgatatcgtt aaaaaacaga aaggcggaaa aggtactgct gctcaaggaa tctacattaa 1020
ctcaacatca ggcacagctg gtaaaatgct cagaatcaga aataaaaatg aagacaaatt 1080
ttatgtaggt ccagatggcg gctttcactc aggtgcaaat tcaactgtag ctggtaatct 1140
aacagttaaa gatccaacat ctggaaaaca tgctgcgact aaagattacg tagatgaaaa 1200
aattgctgag ttaaaaaaac tcatactaaa aaaatagatt aaggaggata aatgagcaga 1260
gacccaacat atacaataaa cgagcacgac ttatcttttg cagatggtcg tttttatgtg 1320
acctttaagg cagataagtc aagtgagact gtgagactta acagtagttg ccttggcaat 1380
accataatca aaaagctaca ggtcgaggat gacaatacaa tgcacgactt tgtaaagcct 1440
aaagttacca ctcaacaagc ttttggacta gctcagcagg tcaaagagct tgatttacag 1500
ctaaaagacc ctaagtcaga tttgtggggc aaaatcaagt tcaataataa ggcaatgcta 1560
gtcgagtacg ccaacaaaga gatgtcaagt gccattgcgc aatcagctga gcagatattg 1620
ttacaagtca agtctattga tgatgaacga tattccaaat ttgagcaaac tctgaatggt 1680
atcaaacaaa ctgtcaaaag tgagtcagtt gaatccgcac gtactcagct agcatcaatg 1740
tttgatagtc gtattagtgg acttgatggc aaatacagtc gtttaagcca aacaattgat 1800
agtcttagca gtcgtcttga tgatggtgtt ggtaactact caacgctatc tcaaaaggta 1860
agtggcattg atttacgagt tagtaatgca gctaatgatg tttctcgatt gtctcagaca 1920
gcacaaggat tacagtcaca aatcacaaat gcaaaccaaa attacagcag tttgtctcag 1980
actgtacagg gactacaaac aactgtacgt gataatcaat caaatgctac aagtcggatt 2040
aatcagttaa gtgatttaat cagtactaaa gtgactaagg gcgacgtcga aacaactatt 2100
gctcaaagtt acgacaagat agccttcgca atcagggata aactcccagc aagcaagatg 2160
actggcagtg agattatctc ggcaatcaat cttgataggt ctggggttaa aatcactggc 2220
aaaaacatca cattagacgg taacagctac atcagcaacg ctgttatcaa agatgctcac 2280
attgctaaca tggatgctgg taagattaac actggttatc ttaatgctag tagaattgcg 2340
gcagaagcta tcactggcga caaaatcaag atggactatg ctttttttaa taagctcact 2400
gctaatgagg gatattttag gaccttattt gctaaaaata tctttaccac atctgtacag 2460
gctgtcacta cgtctgcgag taagattaca ggtggtgtgc tttctgccac gaatggtgcg 2520
agtaggtggg atttgaatag tgcaaatatt gattttaatc gagatgccac aattaatttt 2580
aacagcaaaa acaatgcgct tgttcgaaaa tcaggtacca acactgcttt tgttcacttt 2640
agcaatgcga caccaaaagg ctatagaggc tcagcgttgt atgcgtcaat cgggataacc 2700
tcatcaggag atggcatcga cagcgcttcg tctggacgtt tctgtggagt taggtttttc 2760
cggtacgctg aagggttaca gcatacagca aaggtcgatc aagccgaaat ttatggtgat 2820
gatattgtct ttagcgacga ttttaacatc gatcgtggct ttaagatgcg gcctagccta 2880
atgccaaaaa tggtcgactt aaacaagatg taccaggcaa ttttggctct cggccgctgc 2940
tggctgcatg ctaataacac ggcttggtcg tggaattttg atacacgcag cgcaatcatc 3000
gcagaatata acgcacacat taataactta taggagaaac aatggattta acgcttaaaa 3060
acaaagattt aaacacacta tatagtgtac tagacaaaat caaaatcacg aacatgcgag 3120
caaaccgggg aattcgtaat catggtcata gctgtttcct gtgtgaaatt gttatccgct 3180
cacaattcca cacaacatac gagccggaag cataaagtgt aa 3222
<210> 2
<211> 206
<212> DNA
<213> Bacillus subtilis
<400> 2
catatctaat attataacta aattttctaa aaaaaacatt gaaataaaca tttattttgt 60
atatgatgag ataaagttag tttattggat aaacaaacta actcaattaa gatagttgat 120
ggataaactt gttcacttaa atcaaagggg gaaatgacaa atggtccaaa ctagtgatat 180
ctaaaaatca aagggggaaa tgtaca 206
<210> 3
<211> 90
<212> DNA
<213> Bacillus subtilis
<400> 3
atggtcagca tccgccgcag cttcgaagcg tatgtcgatg acatgaatat cattactgtt 60
ctgattcctg ctgaacaaaa ggaaatcatg 90
<210> 4
<211> 38
<212> DNA
<213> 人工序列
<400> 4
caccaccacc accaccaccg tcggaggtgc aggaattg 38
<210> 5
<211> 29
<212> DNA
<213> 人工序列
<400> 5
tccccgcggt tacactttat gcttccggc 29
<210> 6
<211> 29
<212> DNA
<213> 人工序列
<400> 6
cgcggatcca tggtcagcat ccgccgcag 29
<210> 7
<211> 39
<212> DNA
<213> 人工序列
<400> 7
gtggtggtgg tggtggtgc atgatttcct tttgttcag 39
- 还没有人留言评论。精彩留言会获得点赞!