一种转基因大豆WH8055转化事件外源插入载体的右侧翼序列的制作方法
本发明涉及转基因大豆领域,具体而言,涉及一种转基因大豆wh8055转化事件外源插入载体的右侧翼序列。
背景技术:
大豆是世界上主要的油料作物。2015年全球转基因作物的种植面积高达1.797亿公顷,其中大豆种植面积为0.916亿公顷,占总面积的51%[1]。中国不仅是农业生产大国,更是农业消费大国。自1996年以来,中国已成为大豆净进口国。随着国内需求不断地增加,转基因大豆进口量也在逐年增大。仅在2015年,中国转基因大豆进口量高达8169万吨,同比增长14.4%[2]。
对转基因大豆生物安全问题进行科学研究和评价是对转基因大豆进行监督管理,保障其健康发展的重要技术基础,而转基因检测技术是生物安全评价的关键技术之一[3],对于转基因植株的身份验证具有重大意义,并有助于对外源基因的表达机制和外源基因在受体基因组中产生的影响进行研究,从而有助对于转基因植株的生物安全性做出评估[4]。
hal1基因是酿酒酵母中与耐盐相关的基因,该基因的过量表达可以提高酵母对nacl的胁迫,而敲除调该基因,则大大降低酵母的耐盐性。目前,已经有一些hal1转基因植物,但尚未研究获得hal1转基因大豆,更缺少能够有效对hal1转基因大豆进行检测的方法。
有鉴于此,特提出本发明。
技术实现要素:
本发明的第一目的在于提供一种转基因大豆wh8055转化事件外源插入载体的右侧翼序列,所述右侧翼序列能够特异性地指示第3号染色体上第43998179位核苷酸处插入pchal1的t-dna片段的转基因大豆。
本发明的第二目的在于提供一种所述右侧翼序列在检测转基因大豆中的应用,所述应用通过所述右侧翼序列可以准确判断所述样品是否来自于第3号染色体上第43998179位核苷酸处插入pchal1的t-dna片段的转基因大豆。
本发明的第三目的在于提供根据所述右侧翼序列设计的pcr引物对,所述引物对通过pcr反应能够快速、准确地检测所述样品的第3号染色体上第43998179位核苷酸处是否插入pchal1的t-dna片段,具有检测方便、特异性好以及准确率高的优点。
本发明的第四目的在于提供一种pcr检测试剂盒,所述试剂盒将上述引物对与其他辅助试剂集成在一起,具有使用方便的优点。
本发明的第五目的在于提供所述引物对或试剂盒的使用方法,通过所述方法可以快速、准确地检测样品的第3号染色体上第43998179位核苷酸处是否插入pchal1的t-dna片段。
为了实现本发明的上述目的,特采用以下技术方案:
一种转基因大豆wh8055转化事件外源插入载体的右侧翼序列,其核苷酸序列如seqidno:4所示。
本发明建立在吉林省农业科学院生物所提供的转基因大豆wh8055基础之上。转基因大豆wh8055是通过农杆菌介导大豆子叶节方法将带有hal1基因的质粒pchal1的t-dna插入到大豆威廉姆斯82的基因组上而获得的转基因大豆,其耐盐性好,农艺性状佳,具有良好的应用前景和育种价值。但在本发明之前,尚无人知晓t-dna在转基因大豆wh8055上的具体插入位点,更加无法建立能够有效鉴定该转基因大豆的方法。
本发明精心设计3’sp1、3’sp2和3’sp3序列,结合takara公司的genomewalkingkit试剂盒,通过tail-pcr和测序比对获得转基因大豆wh8055转化时间外源插入载体的右侧翼序列。
本发明所述右侧翼序列的核苷酸序列如seqidno:4所示,其包括pchal1上t-dna的hal1基因序列、nos3’utr序列以及所述t-dna右侧的大豆基因组序列。由此可见,所述右侧翼序列既包括插入载体的t-dna序列,又包括其插入位点右侧的大豆基因组序列,只有在第3号染色体第43998179位处插入pchal1t-dna的转基因大豆才具有上述右侧翼序列,pchal1以及非转基因大豆或其他转基因大豆都不具备上述右侧翼序列。
因此,本发明所述右侧翼序列能够特异性地指示在第3号染色体第43998179位处插入pchal1t-dna的转基因大豆,尤其是转基因大豆wh8055或由其转育而成的转基因大豆。基于该序列,可以特异性地检测待测样品是否源自上述转基因大豆,在转基因大豆的检测以及种质资源的鉴定方面具有重要意义和广泛的应用前景。
本发明还涉及上述右侧翼序列在检测转基因大豆中的应用,所述转基因大豆的第3号染色体第43998179位核苷酸处插入pchal1的t-dna片段;优选地,所述转基因大豆为wh8055或由其转育的转基因大豆。
本发明所述应用基于上述右侧翼序列,可以通过分子生物学方法,例如pcr、分子杂交,对前述目标转基因大豆进行特异性检测,具有操作简单、准确率高等优点。
在一些具体的实施方式中,根据上述右侧翼序列设计用于检测所述转基因大豆的pcr引物对或杂交探针。
本发明还涉及上述右侧翼序列设计的pcr引物对,所述引物对的核苷酸序列分别如seqidno:6和seqidno:7所示。
本发明所述引物对分别设计在t-dna和所述t-dna插入位点右侧的大豆基因组上,只有以目标转基因大豆为模板才能通过所述引物对扩增获得700bp左右的产物,而以pchal1、非转基因大豆或其他品种的转基因大豆为模板则无法扩增获得目标产物。因此,本发明所述pcr引物对能够用于特异性地检测第3号染色体第43998179位核苷酸处插入pchal1的t-dna片段的转基因大豆,尤其是wh8055或由其转育的转基因大豆。另外,本发明所述引物对还具有特异性好、扩增效率高等优点。因此,本发明所述引物对对提高转基因材料的鉴定效率、转基因产品的管理与检测,以及种质资源的鉴定具有重要的意义。
本发明还涉及一种pcr检测试剂盒,所述试剂盒包括上述引物对,以及其他试剂。
本发明上述试剂盒将上述引物对与pcr用到的其他辅助试剂集成在一起,具有使用方便的优点。
在一些具体的实施方式中,所述其他试剂包括dna聚合酶、dntps、pcr反应缓冲液、阳性对照品和阴性对照品中的一种或多种。
在一些具体的实施方式中,所述dna聚合酶选自taq、bst、vent、phi29、pfu、tru、tth、tl1、tac、tne、tma、tih、tf1、pwo、kod、sac、sso、poc、pab、mth、pho、es4dna聚合酶、klenow片段中的一种或多种。
优选地,所述dna聚合酶为taqdna聚合酶。
更优选地,所述taqdna聚合酶为热启动taqdna聚合酶。
在一些具体的实施方式中,所述其他试剂包括pcr反应的预混液,所述预混液为dna聚合酶、dntps和pcr反应缓冲液的混合物。
本发明还涉及上述引物对或上述试剂盒的使用方法,所述方法包括:
(1)以待测大豆样品的dna为模板,使用上述引物对或上述试剂盒中的引物对和其他试剂进行pcr扩增;
(2)根据pcr扩增产物判定所述待测大豆的第3号染色体上第43998179位核苷酸处是否插入pchal1的t-dna片段。
本发明上述使用方法通过简单的pcr方法即可检测样品的第3号染色体上第43998179位核苷酸处是否插入的hal1基因,具有检测方便、特异性好、以及准确性好的优点。
在一些具体的实施方式中,所述pcr反应的退火温度为49~51℃,循环数为28~32次;优选地,所述退火温度为50℃,所述循环数为30次。
与现有技术相比,本发明的有益效果为:
(1)本发明涉及转基因大豆wh8055,所述大豆的第3号染色体上第43998179位核苷酸处插入pchal1上的t-dna序列,从而获得hal1基因,所述转基因大豆能够耐盐碱,并且表现出良好的农艺性状,具有广阔的应用前景。本发明首次获得转基因大豆wh8055转化事件外源插入载体右侧翼序列、设计相应的pcr引物和建立特异性pcr检测方法,能够准确地检测待测样品是否来自第3号染色体上第43998179位核苷酸处插入pchal1的t-dna序列的转基因大豆,该方法灵敏,准确,简单,可靠,具有广阔的市场前景,对提高转基因材料的鉴定效率、转基因产品的管理与检测以及种质资源的鉴定具有重要的意义。
(2)本发明上述引物对经精心设计和反复筛选方最终确定,分别设计在t-dna和t-dna侧翼的大豆基因组上,只有以t-dna插入到第3号染色体的第43998179位核苷酸处的转基因大豆为模板才能扩增得到目的片段,其他模板均不能扩增获得目的片段,可容易地判断检测对象是否为t目标转基因大豆。另外,本发明上述引物对还具有特异性和准确性好、检测效率高的优点。
附图说明
为了更清楚地说明本发明具体实施方式或现有技术中的技术方案,下面将对具体实施方式或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图是本发明的一些实施方式,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
图1为pchal1质粒的t-dna图谱,其中,所述t-dna包括左边界序列(lb)、bar基因、hal1基因和右边界(rb);
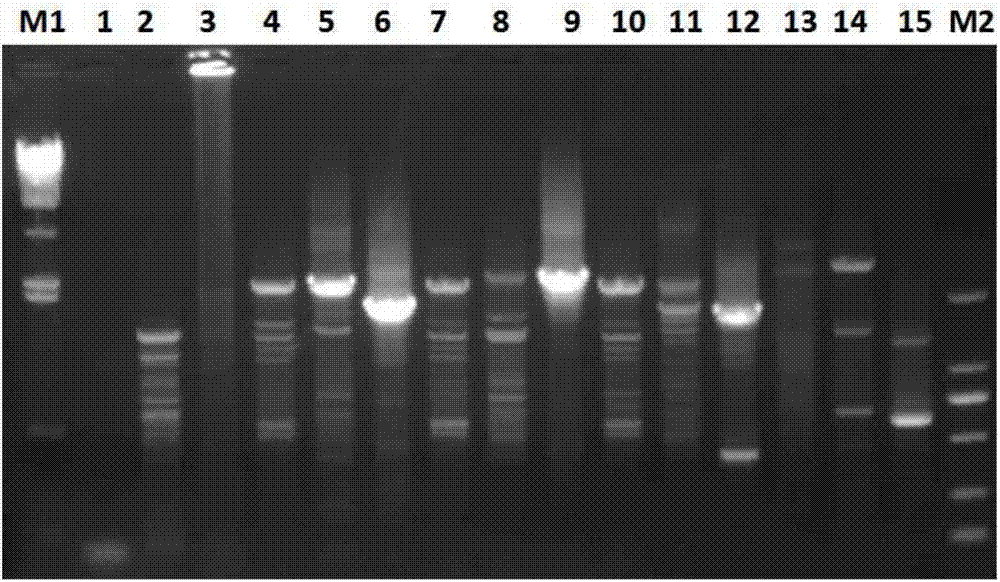
图2为右边界tail-pcr电泳结果,其中,m1:λ-hindⅲdigest;1-3:ap11st2nd3rd;4-6:ap21st2nd3rd;7-9:ap31st2nd3rd;10-12:ap41st2nd3rd;13-15:正对照1st2nd3rd;m2:dl2000;
图3为右边界tail-pcr的测序结果,其中,阴影部分为已知的hal1基因的序列,加框部分为nos3’utr序列,斜体部分为大豆基因组序列;
图4为大豆基因组chr03:43998179..43998678序列;
图5为转基因大豆wh8055右边界引物位置图,其中3’f设计在t-dna上,3’r设计在插入位点右侧的大豆基因组上;
图6为转基因大豆wh8055的特异性pcr检测的电泳结果图,其中,m:marker,1为转基因大豆wh8055的pcr反应产物。
具体实施方式
下面将结合实施例对本发明的实施方案进行详细描述,但是本领域技术人员将会理解,下列实施例仅用于说明本发明,而不应视为限制本发明的范围。实施例中未注明具体条件者,按照常规条件或制造商建议的条件进行。所用试剂或仪器未注明生产厂商者,均为可以通过市购买获得的常规产品。
实施例1获取转基因大豆wh8055插入载体的右侧翼序列
一、实验材料
转基因大豆wh8055(由吉林省农业科学院生物所大豆课题组提供):以大豆威廉姆斯82为受体材料,以质粒pchal1为转化载体,通过农杆菌介导大豆子叶节转化方法(即,转基因大豆wh8055转化事件)获得,所述质粒pchal1的图谱参见说明书附图图1;所述转基因大豆wh8055表现出耐盐碱以及良好的农艺性状,经实施例1最终确定其t-dna的插入位点为大豆第3号染色体第第43998179位核苷酸处。
二、右侧翼序列的获取
1、dna的提取
(1)样品采集:于6月中旬取大田种植的转基因大豆wh8055的叶片,在-80℃下保存。
(2)dna的提取:采用ctab方法提取所述大豆叶片的基因组dna,具体检测方法参见参考文献5。
(3)dna浓度和纯度的检测:使用nanodrop检测步骤(2)所述dna样品的浓度和纯度。
2、右侧翼序列的获取
本实施例采用takara公司genomewalkingkit试剂盒获取转基因大豆wh8055中外源t-dna整合位点处右边界的侧翼序列。
(1)根据已知hal1序列设计3条右边界特异引物3’sp1(seqidno:1)、3’sp2(seqidno:2)和3’sp3(seqidno:3)(表1)。
表1右边界特异引物
(2)以1μl(500ng)的转基因大豆基因组dna为模板,以3’sp1、3’sp2、3’sp3和takaragenomewalkingkit(codeno.6108)试剂盒中的ap1、ap2和ap3、ap4,以及ck引物,按takaragenomewalkingkit(codeno.6108)试剂盒说明书进行5次,每次3轮tail-pcr反应。
(3)对pcr产物进行电泳。图2显示引物ap1、ap2、ap3的第3轮pcr反应(3、6、9泳道)均得到比试剂盒所提供正对照清晰的特异性条带,相同引物的3’ap4-3rd的扩增产物条带(12泳道,大小约为2.4kp)切胶回收进行测序,即获得插入载体pchal1的右侧翼序列。所述右侧翼序列的具体核苷酸序列(seqidno:4)如图3所示,其中阴影部分为已知的hal1基因序列,加框部分为nos3’utr序列,斜体部分为大豆基因组序列。
3、外源t-dna在大豆基因组中整合位点的确定
在soybase.org网站(https://www.soybase.org/gb2/gbrowse/gmax2.0/)比对转基因大豆wh8055中外源t-dna整合位点处右侧序列和大豆基因组,发现所述右侧序列在大豆基因组chr03:43998179..43998678(seqidno:5)(图4)上是连续存在的,即获得t-dna在大豆基因组中的整合位点:大豆基因组chr03:43998179(即,大豆第3号染色体上第43998179位核苷酸处)。
实施例2转基因大豆wh8055的特异性pcr检测
1、pcr引物的设计
根据实施例1获得的转基因大豆wh8055的右边界侧翼序列以及t-dna序列设计能够特异性检测t-dna插入事件的引物对。
所述引物对的一条引物在t-dna上,另一条引物在大豆基因组上:如3’f上游引物在t-dna上,3’r下游引物在右边界基因组上(图5)。引物对3’f(seqidno:6)和3r’(seqidno:7)序列如表2所示。
表2转基因大豆wh8055的特异性pcr检测引物对
2、转基因大豆wh8055的特异性pcr检测
以转基因大豆wh8055的基因组dna为模板,使用上述pcr引物对进行pcr检测。其中,具体反应条件如下所示:
第1步:94℃2min;
第2步:94℃30s;
第3步:50℃30s;
第4步:72℃30s;
第5步:72℃5min;
第6步:16℃1h;
第2~4步循环30次。
3、扩增产物利用1%琼脂糖凝胶检测,在质粒和阴性材料均不能扩增到条带,只有转基因阳性材料获得了预期大小的片段(图6)。
最后应说明的是:以上各实施例仅用以说明本发明的技术方案,而非对其限制;尽管参照前述各实施例对本发明进行了详细的说明,但本领域的普通技术人员应当理解:其依然可以对前述各实施例所记载的技术方案进行修改,或者对其中部分或者全部技术特征进行等同替换;而这些修改或者替换,并不使相应技术方案的本质脱离本发明各实施例技术方案的范围。
参考文献:
[1]clivejames.2015年全球生物技术/转基因作物商业化发展态势[j].中国生物工程杂志,2016,36(4):1-11.(jamesc.globalbiotechnology/gmcropscommercializationdevelopmenttrendin2015[j].chinabiotechnology2016,36(4):1-11(inchinese));
[2]崔宁波,张正岩.转基因大豆研究及应用进展[j].西北农业学报,2016,25(8):1111-1124.(cuinb,zhangzy.researchandapplicationoftransgenicsoybean[j].actaageiculturaeboreali-occidentalissinia2016,25(8):1111-1124.);
[3]郭斌,祁洋,尉亚辉.转基因植物检测技术的研究进展[j].中国生物工程杂志,2010,30(2):120-126.(guob,qiy,wieyh.progressintheresearchoftransgenicplanttesting[j].chinabiotechnology2010,30(2):120-126.);
[4]刘蓓.外源基因插入位点旁侧序列的研究方法及进展[j].农业与技术,2012(4):97.(wangxb,jianglx,2,weil,etal.integrationandinsertionsiteofepspsgeneonthesoybeangenomeingeneticallymodifiedglyphosate-resistantsoybean[j].actaagronomicasinica2010,36(3):365-375.);
[5]fultontm,chunwongsej,tanksleysd.microprepprotocolforextractionofdnafromtomatoandotherherbaceousplants[j].plantmolecularbiologyreporter,1995,13(3):207-209。
sequencelisting
<110>吉林省农业科学院
<120>一种转基因大豆wh8055转化事件外源插入载体的右侧翼序列
<160>7
<170>patentinversion3.3
<210>1
<211>23
<212>dna
<213>人工序列
<400>1
ttggtaagctccgtcatcgaaac23
<210>2
<211>22
<212>dna
<213>人工序列
<400>2
atgcggacgggaaacatatcag22
<210>3
<211>22
<212>dna
<213>人工序列
<400>3
gagtggcctttctccaattgag22
<210>4
<211>1684
<212>dna
<213>人工序列
<400>4
atgcatttcaaagatttaggattgcatgactacactctcaaaaacttgatgtatgagaat60
aattgctgtaaattttatgatgccgtggatgaaaacaacatctcatatgttttaaaattt120
gttccctcagatgtgacttcggaaggggatactttcccattcgtggatcgctttcaagta180
aaggaaggtgtttttttggtatattcctcaaatgactttggaaaagaaggtacggactac240
tttacttatactggtagtggtggaaatgaggttcacatctcgggcacctcttcagaagca300
ggaataaaaccgcagtttattgaaacttgccatccaaaacatcttaagcggggaacaaaa360
gagcaggaagatataaatagtagtacctcaaagaaaagtgcagttatcaacaatttttcg420
ggtgaaaaaacaccaaatccaaggccacagagttccaacatttcagaaagagagacgtat480
gtcggaatattgaacgtcaaatgtaaaaataagaactcatcgaaaatacgaagtgaaaaa540
ttggtaagctccgtcatcgaaacaaagcatacgccaggattggcatctattttatcgaaa600
gaaggcactacatatccgaataatgcggacgggaaacatatcagtatcgtgaatccatcc660
tcaaaaatatatcattcatcccataaacagattgttaaaacgcctatccctaagagtggc720
ctttctccaattgagagatgccctttcaatggtcaaaatattaaatgctactcaccaaga780
ccactagatcatgaaagtccccaacgtgatttcaataataactttcagctgagaatactg840
aagagctcggtgttgcaaaggagacaatcaacacagaatagttgaggtgaccagctcgaa900
tttccccgatcgttcaaacatttggcaataaagtttcttaagattgaatcctgttgccgg960
tcttgcgatgattatcatataatttctgttgaattacgttaagcatgtaataattaacat1020
gtaatgcatgacgttatttatgagatgggtttttatgattagagtcccgcaattatacat1080
ttaatacgcgatagaaaacaaaatatagcgcgcaaactaggataaattatcgcgcgcggt1140
gtcatctatgttactagatcgggaattaaactatcagtgtttgaacaaaaaaataagaag1200
tcaatatatcataaattttgagtgtttttatttcttaaaaacaaaaaataagaagttaaa1260
tcaaatatatttttaaaattttaatcttttaaaaatgaaaacaatttttaaaaaataaag1320
aaaagaaagaaaacaaaataaaaatgctgagcaaatacattgtgacgggttttgttccaa1380
aaatgctcgaaaacgaaaaagcagtttcccgcgaatcttagtgaggatgccacgttggca1440
atccgcgtctcaccttctcgaccaatcagcgcggattatcgggccctatataattccatt1500
ccactcacaaccctttcgtctgcgcctgatttctcagttctctcacacactcattcggtg1560
ctccgaatctcagattctagggttagggttcccaatttctccaccgatcagaaaatgagt1620
tcaactggtggttctaccaagggcggaaggggcaagcccaaagcctccaaatccgtttca1680
agat1684
<210>5
<211>500
<212>dna
<213>glycinemax
<400>5
acaaaaaaataagaagtcaatatatcataaattttgagtgtttttatttcttaaaaacaa60
aaaataagaagttaaatcaaatatatttttaaaattttaatcttttaaaaatgaaaacaa120
tttttaaaaaataaagaaaagaaagaaaacaaaataaaaatgctgagcaaatacattgtg180
acgggttttgttccaaaaatgctcgaaaacgaaaaagcagtttcccgcgaatcttagtga240
ggatgccacgttggcaatccgcgtctcaccttctcgaccaatcagcgcggattatcgggc300
cctatataattccattccactcacaaccctttcgtctgcgcctgatttctcagttctctc360
acacactcattcggtgctccgaatctcagattctagggttagggttcccaatttctccac420
cgatcagaaaatgagttcaactggtggttctaccaagggcggaaggggcaagcccaaagc480
ctccaaatccgtttcaagat500
<210>6
<211>22
<212>dna
<213>人工序列
<400>6
gagtggcctttctccaattgag22
<210>7
<211>20
<212>dna
<213>人工序列
<400>7
caacgtggcatcctcactaa20
- 还没有人留言评论。精彩留言会获得点赞!