一种Clec4F纳米抗体及其应用的制作方法
本发明属于生物制药技术领域,涉及一种Clec4F纳米抗体,以及该抗体在制备靶向结合Clec4F阳性巨噬细胞和竞争抑制Clec4F和N-acetylgalactosamine(GalNAc)分子结合中的用途。
背景技术:
Clec4F蛋白全称为C-type lectin domain family 4,member F(简称Clec4F),是一个包含550个氨基酸的二型跨膜C型凝集素(C type Lectin),蛋白质的C末端结构域和其他C型凝集素的碳水化合物识别区同源。研究表明,其蛋白质集中在肝脏组织巨噬细胞中表达,因此,Clec4F也常被称为柯弗氏细胞(Kupffer cell)受体。
最初Clec4F在大鼠的肝脏中分离和发现,当时认为它是肝脏海藻糖结合凝集素(hepatic fucose-binding lectin)。随后发现Clec4F与半乳糖(galactose),N-乙酰氨基半乳糖(N-acetylgalactosamine,GalNAc)和糖脂类(Glycolipids)结合。Clec4F可能在从血液中清除半乳糖和海藻糖的糖蛋白的过程中发挥了重要作用。
Kupffer cell是位于肝脏血窦部位的最重要的一群肝脏组织巨噬细胞,Clec4F可以作为表面标记物将Kupffer cell与肝脏中其它组织巨噬细胞和单核吞噬细胞区分开。C型凝集素参与了将脂类抗原提呈至抗原递呈细胞的过程。NKT细胞是一种特殊的抗原提呈细胞,其主要特征为T细胞受体基因表达的恒定性、CD1d的限制性以及细胞因子产生的迅速、高水平性。NKT细胞既能增强免疫反应又能抑制免疫反应,从而在抗肿瘤、抗感染、抑制自身免疫性疾病及移植耐受中发挥重要的作用。这种细胞可以被一种叫做α-半乳糖神经酰胺(alpha-galactosylceramide,α-GalCer)的糖脂分子激活。NKT细胞被激活以后可以释放大量的白介素4(IL-4),干扰素(IFN-γ)和其他细胞因子,NKT细胞也被认为是免疫系统内至关重要的调节细胞。小鼠NKT细胞占血液、外周淋巴结和脾脏中总T细胞数的0.5%。而超过30%的肝脏T细胞有NKT细胞表型,研究表明肝脏中的Kupffer cell是关键的NKT细胞提呈细胞。Kupffer cell可以和NKT细胞通过CD1d形成稳定的连接,从而激活NKT细胞。同时Clec4F参与了α-GalCer抗原的提呈,α-GalCer与CD1d连接,从而激活NKT细胞。因此Clec4F可以作为Kupffer cell的表面标记物,而且在糖脂抗原的提呈方面也有重要的贡献。因此针对竞争抑制Clec4F与N-acetylgalactosamine(GalNAc)分子结合所设计的抗体,将阻断NKT细胞的激活通路,缓解免疫反应。
抗体和抗体衍生物是研究细胞表面受体最常用的方法,特异性抗体不但可以用于基础实验研究和临床标本检测,而且可以作为受体的在体示踪剂。骆驼属动物体内存在缺失轻链的天然重链抗体,这种抗体只包含一个重链可变区(VHH)和两个常规的CH2与CH3区,用噬菌体文库筛选和单克隆表达获得VHH单域抗体,也称为纳米抗体(Camelid single-domain antibody-fragment,nanobody,Nb)。纳米抗体的大小仅是普通IgG抗体的十分之一,具有高稳定性、高亲和力及容易标记等优点。通过二十几年的研究,纳米抗体作为检验、治疗等特有的工具应用于多个研究领域。
目前尚未见到Clec4F纳米抗体的研究报道,主要原因在于:Clec4F纳米抗体的筛选与免疫动物对Clec4F重组蛋白的免疫反应程度、构建噬菌体文库和构建表达载体的技术水平,以及免疫文库的筛选等诸多实验因素有关。筛选获得不但可以与Clec4F重组蛋白具有高亲和力,而且同时能与肝脏中自然表达的Clec4F受体结合的纳米抗体需要大量的实验积累。另外,Clec4F为新近发现的凝集素反应蛋白家族分子,Clec4F作为构建纳米抗体的靶标需要实验进行确定。
技术实现要素:
本发明的目的在于提供一种Clec4F纳米抗体及其应用。本发明对Kupffer cell表面受体Clec4F构建纳米抗体,作为靶向结合Kupffer cell的分子探针,可以用于研究组织巨噬细胞在急慢性肝炎和肝癌中的作用,以及作为NKT细胞激活的阻断剂。
为达到上述目的,本发明采用了以下技术方案:
本发明公开了一种Clec4F纳米抗体,包括框架区和互补决定区,该纳米抗体特异性识别Clec4F蛋白(例如能够抗鼠源Clec4F)。
所述Clec4F纳米抗体的VHH链包括四个框架区域(framework region,FR)FR1~FR4和三个抗原互补决定区域(complementarity-determining region,CDR)CDR1~CDR3;所述Clec4F纳米抗体的VHH链的氨基酸序列如SEQ.ID.NO.1所示。
其中,四个框架区域FR1、FR2、FR3和FR4的氨基酸序列分别如SEQ.ID.NO.2、SEQ.ID.NO.4、SEQ.ID.NO.6和SEQ.ID.NO.8所示;三个抗原互补决定区域CDR1、CDR2和CDR3的氨基酸序列分别如SEQ.ID.NO.3、SEQ.ID.NO.5和SEQ.ID.NO.7所示。
本发明公开了编码Clec4F纳米抗体的核苷酸序列,如SEQ.ID.NO.9所示。该DNA序列编码的蛋白质结构选自:Clec4F纳米抗体的VHH链或者Clec4F纳米抗体。
本发明还提供一种原核表达载体,它含有上述编码Clec4F纳米抗体的核苷酸序列。本发明还提供了一种原核宿主细胞,它含有上述原核表达载体。
本发明还公开了上述Clec4F纳米抗体在炎症检测中的作用,具体为Clec4F纳米抗体应用于体外或体内结合Kupffer cell表面Clec4F受体,从而监测炎症的发生和发展的过程。所述炎症包括急性肝炎,慢性肝炎,肝癌等。
本发明还公开了上述Clec4F纳米抗体在制备Clec4F受体拮抗剂中的用途。
本发明还涉及Clec4F纳米抗体在非疾病诊断治疗目的的免疫学检查分析中的应用,可以通过将Clec4F纳米抗体在制备吸附Clec4F蛋白的试剂中的使用,例如可以将本发明Clec4F纳米抗体制作成Clec4F免疫亲和柱,吸取或者富集Clec4F或富集能与Clec4F结合的受体或者细胞,以供进一步研究。
本发明所述的氨基酸序列可以作为前体,进行随机、点突变或者人源化改造,获得亲和力、特异性或者稳定性更高,免疫原性低的突变体。
与现有技术相比,本发明具有以下有益的技术效果:
本发明通过深入研究肝脏中Kupffer cell表面C型凝集素家族成员Clec4F的配体识别和受体活化的机制,采用噬菌体展示技术对Clec4F纳米抗体进行表达;通过生物淘筛技术筛选出与抗原具有较高结合力的纳米抗体;本发明表达并优化获得亲和力高且稳定均一的Clec4F纳米抗体蛋白,开辟了靶向Clec4F的纳米抗体在肝炎和肝癌的分子影像学和NKT细胞活性抑制剂的新领域,具有良好的研究价值和应用前景。
附图说明
图1为Clec4F纳米抗体噬菌体文库的DNA电泳图。
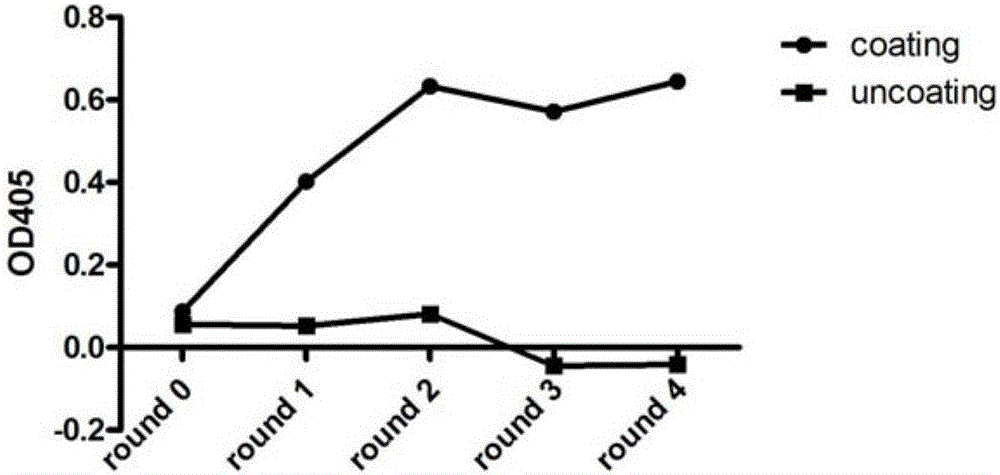
图2为Clec4F纳米抗体噬菌体展示文库的生物淘筛结果(用ELISA检查连续4次生物淘筛的Clec4F特异性噬菌体富集率)。
图3为Clec4F纳米抗体纯化鉴定图,Ladder:相对分子量标准(kDa)。
图4为用ELISA检测Clec4F纳米抗体亲和力曲线。
图5为利用纳米抗体在刀豆素A(Concanavalin A,Con A)诱导的小鼠急性肝炎中测定Clec4F表达的结果,图中按照从左到右,从上至下的顺序分别显示正常小鼠肝脏(Naive)、ConA诱导小鼠产生急性肝炎3小时、6小时、12小时、24小时、48小时小鼠肝脏的四种染色的融合结果。
具体实施方式
下面结合附图和实施例对本发明做进一步的详细说明,所述是对本发明的解释而不是限定。
根据前期的肝炎和正常肝组织的表达谱筛选出了Clec4F分子,并验证了Clec4F对肝脏巨噬细胞表达的专一性,从而进一步确定Clec4F为构建纳米抗体的靶标。在此基础上,本发明利用噬菌体展示技术,从羊驼免疫的重链抗体中筛选能与靶重组蛋白Clec4F特异性结合的纳米抗体克隆,以作为Kupffer cell的特异性探针,用于肝炎和肝癌的分子探针和NKT细胞的活性抑制剂。
本发明采用的Clec4F特异性结合的纳米抗体的筛选路线:1、通用Clec4F重组蛋白对羊驼进行免疫,分离血液中的淋巴细胞,利用噬菌体展示技术,构建噬菌体展示文库;2、经过生物淘筛法获得与Clec4F蛋白结合的噬菌体;3、经测序后和生物比对后,用酶联免疫吸附测定(enzyme-linked immuno sorbent assay,ELISA)法筛选出抗Clec4F的高亲和力纳米抗体,并用Kupper cell细胞验证纳米抗体的结合能力。
1、以Clec4F为抗原的纳米抗体文库的构建
1)羊驼淋巴细胞中VHH基因的扩增
使用小鼠淋巴瘤细胞系NS0(中科院上海生科院细胞资源中心)真核表达体系表达,并利用N端的His标签纯化得到的Clec4F重组蛋白(Entrez Gene IDs:51811,Ala65-Fly548区域)作为抗原,Clec4F蛋白的糖基增强了抗原的免疫原性,用100μg Clec4F重组蛋白免疫羊驼五次,每次间隔3周,在此过程中免疫动物体内的浆细胞分泌大量针对Clec4F的抗体。获得全血50mL后,通过淋巴分离液对不同细胞组分进行分层,分离出淋巴细胞。用Trizol法提取淋巴细胞RNA,经过反转录试剂盒(Invitrogen)获得cDNA。随后通过两次PCR反应确保分离出编码构建VHH的基因:
第一次PCR获得重链抗体基因:用淋巴细胞的cDNA作为PCR模板,加入覆盖免疫球蛋白IgG所涉及的上下游引物Primer call 01和Primer call 02,用于扩增出IgG的不同亚型,同时加入dNTP、Taq DNA聚合酶(Roche)等进行PCR反应,1%琼脂糖胶鉴定PCR结果,胶回收IgG2和IgG3的基因条带(600-800bps),并测定胶回收DNA浓度。
第二次巢式PCR获得VHH基因同时插入酶切位点:加入覆盖IgG2和IgG3重链区域VHH序列的Primer A6E和Primer 38作为引物,此上下游引物分别设计了酶切位点Not I和Nco I,同时加入dNTP、Taq DNA聚合酶(Roche)等进行PCR反应,取少量PCR产物用1%琼脂糖胶鉴定结果,获得建库用VHH大量DNA并用DNA纯化试剂盒(Qiagen)纯化PCR产物。
2)VHH基因与噬菌体载体pHEN4的链接、转染和文库的表达
在PCR反应过程中通过引物在目的基因两端加入酶切位点,便于构建噬菌体表达pHEN4质粒,具体为将pHEN4噬菌体表达载体(Addgene)和第二次PCR获得的DNA产物,用Not I和Nco I限制性内切酶进行双酶切,置于37℃水浴中过夜,1%琼脂糖胶鉴定酶切结果,再加入DNA连接酶之后过夜连接酶切DNA和pHEN4载体,并纯化重组质粒条带。
通过电转染将重组质粒转入过夜培养的TG1大肠杆菌(Addgene)感受态细胞中,之后将转染宿主菌在LB液体培养基中于37℃摇床培养1小时,再用LB培养基分别进行1/10,1/102,1/103倍稀释,分别取500μL稀释过的菌液涂布在LB固体培养皿表面,过夜于37℃培养箱中培养TG1细胞,第二天根据平板上的菌落数计算出库容量为2×107。
随机挑选30个克隆,用针对pHEN4质粒的酶切位点区域设计的RP和GIII上下游引物,同时加入dNTP、Taq DNA聚合酶(Roche)等进行PCR反应,如图1所示,经过1%琼脂糖胶鉴定确定噬菌体展示文库的插入率达到为74%以上,证明文库构建成功。
2、采用噬菌体展示技术筛选与Clec4F蛋白结合的噬菌体
1)噬菌体文库中的生物淘筛环节,是保证获得特异性结合Clec4F噬菌体的重要步骤,本发明进行了连续4次生物淘筛,淘筛中的洗脱步骤去除不能和Clec4F抗原结合的噬菌体,而能与抗原结合的噬菌体在每一轮淘筛中得到了富集。具体操作为在96孔板的两孔中分别包被(coating)或者不包被(uncoating)Clec4F重组蛋白,标记为“+”或“-”,随后将噬菌体文库分别与2个孔在室温进行孵育。第1轮生物淘筛用PBST缓冲液(Sigma)洗涤多次后,去除不能与Clec4F结合的噬菌体,剩下能够与Clec4F结合的噬菌体,使用TEA溶液(Sigma)将其溶解下来并与helper phage(Thermo fisher)同时感染TG1宿主菌,经过夜培养后,进入下一轮新的筛选,过程同上。
2)ELISA法检测生物淘筛富集率,用脱脂奶粉封闭Clec4F包被的ELISA平板,分别加入4轮生物淘筛的噬菌体培养液,随后加入辣根过氧化物酶anti-phage antibody-HRP抗体(Life technology),室温孵育1小时后,再加入酶底物ABTS和H2O2,经过分光光度计OD405nm测定ELISA显色结果,从图2可以看出,每一轮筛选,表达VHH的噬菌体与Clec4F的结合酶学反应信号增强,从而判断Clec4F特异性噬菌体在连续4轮筛选中得到了富集。
3、用酶联免疫学方法筛选和Clec4F结合的阳性单克隆
从每轮生物淘筛的TG1细菌培养皿中随机挑选克隆,置于LB培养基培养至细菌指数生长期后加入IPTG诱导剂,过夜表达蛋白。第二天去除每个克隆的培养基上清,裂解TG1细胞后将细胞裂解液,加入提前用脱脂奶粉封闭过的Clec4F包被的ELISA平板,再加入anti-HA mouse antibody单克隆抗体(Covance),室温孵育1h,最后加入碱性磷酸酶标记anti-mouse IgG Ab(Life technology)和碱性磷酸酶底物,经过分光光度计A405nm测定ELISA显色结果。对吸光度值高于阴性对照3倍的阳性克隆进行测序(见表1),根据测序结果按照IMGT法将纳米抗体进行比对和分组,从互补确定区(complementarity-determining region)CDR3相同组中选取表达量高的有代表性的纳米抗体进行后续实验。
表1.生物淘筛中随机挑选TG1细胞单克隆的PE-ELISA统计结果
表1为生物淘筛第2和3轮(Round 2and Round 3)随机挑选克隆进行PE-ELISA的统计结果,表1中斜体数字显示2轮筛选中的阳性克隆数,阳性信号(Specific signal)高于背景信号(background)3倍(>3fold)的克隆被定义为阳性克隆。
4、纳米抗体大量表达和纯化
用Nco I和Eco9II限制性内切酶对含有纳米抗体基因的pHEN4质粒进行双酶切反应,将所得目的基因片段插入pHEN6空质粒载体(Addgene)后,转染Escherichia coli WK6(Addgene)感受态大肠杆菌。将WK6在1L培养基中培养至生长指数期,用IPTG诱导表达纳米抗体,过夜培养后离心收集WK6,获得含有纳米抗体的细菌裂解液,用金属螯合亲和色谱(Immobilized metal ion affinity chromatography,IMAC)亲和层析法进行初步纯化,用0.5M咪唑洗脱带有His tag的纳米抗体,再用体积排阻层析(size exclusion chromatography)对纳米抗体进行二次纯化,用PBS进行洗脱,通过A 260测定纳米抗体浓度,蛋白质凝胶电泳方法和考马斯亮蓝法染色鉴定纳米抗体的纯度和分子量大小,并计算纳米抗体的表达量(见图3、表2)。
表2.Clec4F纳米抗体在1升TB培养基中的表达量
表2中:2.22代表第2组第22个,5.10代表第5组第10个,6.19代表第6组第19个,8.37代表第8组第37个,10.16代表第10组第16个,12.75代表第12组第75个,NbBCII10作为阴性对照。
参见图3,经过IMAC亲和层析法和体积排阻层析法纯化后,用12%SDS-PAGE凝胶电泳分析Clec4F纳米抗体和阴性对照抗体的纯度,考马斯亮蓝进行蛋白质染色,结果说明纳米抗体的大小在15kDa左右,无其它杂蛋白。
5、采用ELISA技术测定纳米抗体与Clec4F蛋白的亲和力
用Clec4F重组蛋白包被96孔平板过夜,用脱脂奶粉室温封闭平板2小时,将纳米抗体(Nb2.22纳米抗体的VHH链包括四个框架区域FR1~FR4和三个抗原互补决定区域CDR1~CDR3;纳米抗体的VHH链的氨基酸序列如SEQ.ID.NO.1所示)进行1/2梯度稀释后加入平板孵育1小时,再加入anti-mouse His Ab单克隆抗体(eBioscience)和碱性磷酸酶标记的anti-mouse IgG Ab-AP单克隆抗体(Roche),加入底物反应后,用微孔读板仪测OD 405nm的值。
进行梯度稀释的Clec4F纳米抗体与包被在ELISA平板上的Clec4F重组蛋白反应,通过将实验获得的A405值用X=log(X)方程进行对数函数转化,之后用纵坐标浓度数据取标准误,再用Sigmoidal拟合曲线法进行ELISA的曲线绘制和拟合,纳米抗体Nb2.22、5.10、6.19、8.37、10.16和12.75都和Clec4F重组蛋白有结合反应,因此酶学反应为阳性,同型对照抗体BCII10和Clec4F完全没有结合,据此证明Clec4F纳米抗体可以特异结合Clec4F重组蛋白(见图4)。
6、纳米抗体在小鼠刀豆素A(Concanavalin A,Con A)诱导的急性肝炎中测定Clec4F表达。
如采用步骤5中纳米抗体和anti-HA-AF594(Molecular Probes),联合DAPI,F4/80(巨噬细胞标记)单克隆抗体anti-F4/80-AF488(Invitrogen)或CD31(内皮细胞标记)单克隆抗体anti-CD31-AF647(Invitrogen)对小鼠肝脏冰冻切片进行染色,结果表明(图5),在正常小鼠肝脏中显示大量Kupffer cell,随着炎症的发生ConA诱导小鼠急性肝炎后3小时,6小时和12小时后Kupffer cell凋亡,48小时后Kupffer cell在肝脏中重新出现,证明纳米抗体能够通过检测Kupffer cell表面Clec4F标志物,从而观测急性肝炎中肝脏的损伤、修复和组织巨噬细胞的再生。
序列表
<110> 西安交通大学
<120> 一种Clec4F纳米抗体及其应用
<160> 9
<210> 1
<211> 121
<212> PRT
<213>Alpaca
<400> 1
Asp Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly
1 5 10 15
Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ala Thr Phe Ile
16 20 25 30
Thr Tyr Gly Met Thr Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg
31 35 40 45
Glu Phe Val Val Thr Gly Asn Gly Ala Gly Thr Thr Tyr Leu Pro
46 50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn
61 65 70 75
Thr Val Tyr Leu Gln Met Ser Ser Leu Lys Pro Glu Asp Thr Ala
76 80 85 90
Val Tyr Tyr Cys Gly Gly Arg Arg Trp Val Pro Ala Thr Ala Val
91 95 100 105
Asp Gly Val Ala Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser
106 110 115 120
Ser
121
<210> 2
<211> 25
<212> PRT
<213> Alpaca
<400> 2
Asp Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly
1 5 10 15
Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
16 20 25
<210> 3
<211> 8
<212> PRT
<213> Alpaca
<400> 3
Gly Ala Thr Phe Ile Thr Tyr Gly
1 5
<210> 4
<211> 15
<212> PRT
<213> Alpaca
<400> 4
Met Thr Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
1 5 10 15
<210> 5
<211> 8
<212> PRT
<213> Alpaca
<400> 5
Val Thr Gly Asn Gly Ala Gly Thr
1 5
<210> 6
<211> 38
<212> PRT
<213> Alpaca
<400> 6
Thr Tyr Leu Pro Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
1 5 10 15
Asn Ala Lys Asn Thr Val Tyr Leu Gln Met Ser Ser Leu Lys Pro
16 20 25 30
Glu Asp Thr Ala Val Tyr Tyr Cys
31 35
<210> 7
<211> 16
<212> PRT
<213> Alpaca
<400>7
Gly Gly Arg Arg Trp Val Pro Ala Thr Ala Val Asp Gly Val Ala Tyr
1 5 10 15
<210>8
<211>11
<212> PRT
<213> Alpaca
<400>8
Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser
1 5 10
<210>9
<211>390
<212> DNA
<213> 人工合成
<400>9
gatgtgcagc tggtggagtc tgggggaggt ttggttcagg ctgggggctc tctgagactc 60
tcctgcgcag cctctggagc cactttcatt acgtatggca tgacctggtt ccgccaggct 120
ccagggaagg agcgtgagtt tgtggcagct gtgaccggga atggtgccgg cacaacgtat 180
ctgccctccg tgaagggccg atttaccatc tccagagaca acgccaagaa cacggtgtat 240
ctgcaaatga gcagcctgaa gcctgaggac acggccgttt attactgtgg cgggcggcga 300
tgggtacccg ctactgcagt cgatcaagtc gcgtactggg gccaggggac ccaggtcacc 360
gtctcctcac accaccatca ccatcactaa 390
- 还没有人留言评论。精彩留言会获得点赞!