一株谷氨酸棒杆菌及其合成色氨酸的关键基因的制作方法
本发明属于微生物育种和发酵工程领域,具体涉及发酵生产色氨酸的谷氨酸棒杆菌高产菌株的构建及其色氨酸合成有关基因trpD、trpC和trpB的突变位点。
背景技术:
:
色氨酸是唯一有吲哚支链的芳香族氨基酸,化学名称为2-氨基-3-吲哚基丙酸。色氨酸是人体所必需的8种氨基酸之一,其来源依靠食物供给,L-色氨酸主要有两条去路,一条是合成蛋白质,另一条是进入分解代谢。L-色氨酸在其分解代谢过程中,会产生5-羟色氨酸、烟酸、松果体激素和黄尿酸等多种生理活性物质,这些生物活性物质有重要的医药用途;此外,L-色氨酸可以作为食品添加剂,调味剂和抗氧化防腐剂;近年来,在饲料中大量使用了甲硫氨酸和赖氨酸,色氨酸成为饲料中的主要限制性氨基酸,发现在饲料中加入少量的氨基酸就有很好的效果。因此L-色氨酸在医药、食品和饲料等方面有重要的工业价值。
色氨酸的生产方法有蛋白水解提取法、化学合成法、酶催化法和微生物发酵法。蛋白水解法原料来源有限。化学合成需要高温高压,合成的是D-色氨酸和L-色氨酸的混合物,需要另外的精制过程。酶催化所用底物吲哚和丝氨酸价格昂贵,且酶本身也不安全。因此,前三种方法在工业生产中受到极大限制。目前微生物发酵法是色氨酸生产的主要方法。主要生产菌种为大肠杆菌,而大肠杆菌发酵过程易积累乙酸等副产物,严重抑制菌体生长、产酸水平和糖酸转化率;另外,大肠杆菌产内毒素严重限制了产品的应用领域。
谷氨酸棒杆菌为GRAS生物安全菌,为潜在的色氨酸生产菌种。谷氨酸棒状杆菌在氨基酸发酵领域有着举足轻重的地位,至今已经被安全应用了近60年。目前,包括谷氨酸、赖氨酸、缬氨酸、亮氨酸、异亮氨酸、丙氨酸、天冬氨酸等大多数氨基酸都是利用谷氨酸棒状杆菌发酵生产。在谷氨酸棒杆菌中,由莽草酸途径的中间物分支酸合成色氨酸的4个酶由6个基因组成的trpEGDCBA色氨酸操纵子编码。trpE和trpG分别编码邻氨基苯甲酸合酶(ANS)的大小亚基,ANS催化分支酸到邻氨基苯甲酸的第一步反应。trpD编码邻氨基苯甲酸磷酸核糖转移酶(PRT),trpC编码双功能酶磷酸核糖邻氨基苯甲酸异构酶/吲哚-3-甘油磷酸合酶,trpB和trpA分别编码色氨酸合酶(TS)的β链和α链。ANS受终产物色氨酸的强烈抑制,抑制50%酶活的色氨酸浓度为0.0015mM(Sugimoto和Shiio(1983).Agricultural and Biological Chemistry.47:2295–2305)。对PRT和TS的50%酶活抑制浓度分别为0.15mM(Sugimoto和Shiio(1983).Agricultural and Biological Chemistry.47:2295–2305)和7.7mM(Sugimoto和Shiio(1982).Agricultural and Biological Chemistry.46:2711–2718)。Matsui等通过5-氟色氨酸抗性选育的谷氨酸棒杆菌AJ12036编码ANS大亚基的trpE基因上一个点突变使得丝氨酸密码子(AGC)变为精氨酸密码子(CGC),相应多肽链上氨基酸的改变(Ser38Arg)导致ANS对色氨酸的反馈抑制不敏感(Matsui等(1987).Journal of Bacteriology.109:5330–5332)。O’GARA和Dunican发现产生色氨酸的谷氨酸棒杆菌ATCC 21850编码PRT的trpD基因上两个点突变,带来多肽链上两个氨基酸的改变(Ser149Phe,Ala162Glu),导致PRT对色氨酸和5-甲基色氨酸具有更高的抗性(O’GARA和Dunican(1995).Applied and Environmental Microbiology.61(12):4477–4479)。
综上所述,谷氨酸棒杆菌色氨酸操纵子编码的酶受到终产物色氨酸的反馈抑制,成为限制利用谷氨酸棒杆菌高产色氨酸的重要因素,而目前有关解除色氨酸反馈调节的报道较少,相应菌株色氨酸生产水平也很低。只有日本研究者报道利用质粒过表达解除反馈抑制的3-脱氧-D-阿拉伯庚酮糖-7-磷酸合成酶、ANS和PRT,获得了高产色氨酸的谷氨酸棒杆菌(Katsumata和Ikeda(1993).Nature Biotechnology.12:921–925;Ikeda和Katsumata(1999).Applied and Environmental Microbiology.65(6):2497–2502)。然而,之后再没有见到该菌株的相关报道,其中关键酶的突变位点也不清楚。因此,继续选育解除色氨酸反馈抑制、得到产生色氨酸的谷氨酸棒杆菌就显得十分必要。
在前期工作中,我们通过诱变手段获得了一株谷氨酸棒杆菌生产菌株,并申请了专利。此外,在进一步的诱变工作中,我们得到了一株谷氨酸棒杆菌,该菌株的色氨酸产量进一步提高,通过对色氨酸操纵子的测序发现了色氨酸操纵子trpD、trpC和trpB都发生了基因突变,推测trpDCB的突变可能部分解除了色氨酸对其所编码酶的反馈抑制,从而提高了色氨酸的产量。为了验证色氨酸产量的提高是由trpDCB的突变造成的,本发明以C.glutamicum TP607为出发菌株,对其基因组进行编辑,将发现的trpDCB的突变位点整合到C.glutamicum TP607的基因组上,得到了生产菌株C.glutamicum TP608,摇瓶发酵测定色氨酸的产量,发酵结果表明该菌株色氨酸产量大幅提升,进一步验证了色氨酸产量的提高是由于trpDCB的突变部分解除了色氨酸对其所编码酶的反馈抑制。
技术实现要素:
:
本发明的目的在于提供一株发酵生产色氨酸的谷氨酸棒杆菌高产菌株及其色氨酸合成有关基因trpD、trpC和trpB的突变位点。
所述高产菌株为谷氨酸棒杆菌(C.glutamicum)TP608,是在谷氨酸棒杆菌(C.glutamicum)TP607(保藏编号CGMCC No.12302)的基础上通过基因工程手段,对该菌株的基因组进行编辑,将发现的色氨酸操纵子上trpDCB的突变位点整合到C.glutamicum TP 607的基因组上得到的;
trpDCB的突变位点具体如下:
(1)将邻氨基苯甲酸磷酸核糖转移酶(PRT)的编码基因trpD的第446位碱基C突变为T,使得所编码的氨基酸由丝氨酸变成苯丙氨酸(Ser149Phe);
(2)将双功能酶磷酸核糖邻氨基苯甲酸异构酶/吲哚-3-甘油磷酸合酶的编码基因trpC的第193位碱基C突变成T,使得所编码的氨基酸由苯丙氨酸变成丝氨酸(Phe 65Ser);
(3)将色氨酸合酶(TS)β链编码基因trpB的第1076-1077位间增加了三个碱基ACG,第1079位的C突变为A,第1080位的A突变为G,导致丙氨酸突变成精氨酸(Ala360Arg),同时增加了一个谷氨酸;
上述序列对应序列表中的序列编号见表1;
在本发明中采用如下定义:
1、氨基酸和DNA核酸序列的命名法
使用氨基酸残基的公认IUPAC命名法,用三字母代码形式。DNA核酸序列采用公认IUPAC命名法。
2、突变体的标识
采用“原始氨基酸位置替换的氨基酸”来表示突变体中突变的氨基酸,如Ser149Phe,表示位置149位的氨基酸由Ser替换成Phe,位置的编号对应于突变前氨基酸序列的编号;采用“原始碱基位置替换的碱基”来表示突变体中突变的碱基,如C446T,表示位置116位的碱基由C替换成T,位置的编号对应于突变前核苷酸序列的编号。
表1
所述高产菌株的构建方法如下:
(1)设计引物,通过重叠PCR获得内含上述突变位点的3个重叠片段trp1,trp2和trp3;
(2)将重叠片段trp1,trp2和trp3分别与载体pK18mobsacB连接;
(3)将所得质粒依次电转化入C.glutamicum TP607电转感受态中得高产菌株。
有益效果:
1、根据本发明,通过基因工程手段在菌株C.glutamicumTP607的基础上,对trpD、trpC和trpB基因进行定点突变,获得了新的谷氨酸棒杆菌突变株C.glutamicum TP608。推测解除了色氨酸对这些关键酶的反馈抑制。通过摇瓶发酵发现,C.glutamicumTP608色氨酸产量约为TP607的3.4倍;
2、本发明为色氨酸合成有关基因提供了新的突变位点,为色氨酸的生物合成提供了新的研究方向。
附图说明:
图1 trpD突变基因与出发基因同源比对;
图2 trpC突变基因与出发基因同源比对;
图3 trpB突变基因与出发基因同源比对;
图4发酵样品中单位菌体的产酸量
其中,菌株1为C.glutamicumTP607;菌株2为C.glutamicumTP607-trp1;
菌株3为C.glutamicum TP607-trp1-trp 2;菌株4为C.glutamicum TP608。
具体实施方式:
实施例1:C.glutamicumTP608的构建
1.C.glutamicum TP607(CGMCC No.12302)基因组DNA的提取
将单菌落接种于LB摇管中,32℃过夜培养12h后,按2%接种量接入30mL LB培养基中进行摇瓶培养过夜。提取基因组步骤如下:
(1)将菌液13000rpm离心2min,去上清液,收集到1.5mL的EP管中。
(2)加600μL pH=8.0的TE buffer悬浮菌体,13000rpm离心2min,弃上清液。
(3)加600μL TE重新悬浮菌体,加入6μlRNAase(10μg/mL),30μl溶菌酶(50μg/mL)混匀,37℃保温30min以上。
(4)加30μL 10%SDS和10μL蛋白酶K,混匀,65℃保温15min以上。
(5)加入100μL 5mol/L NaCl溶液混匀后,再加入80μL CTAB/NaCl溶液,颠倒混匀50次,65℃水浴10min。
(6)加入750μL酚:氯仿:异戊醇(25:24:1)抽提,颠倒混匀50次,13000rpm离心15min。
(7)将上清液吸入新的1.5mL EP管中,加入等体积的酚:氯仿:异戊醇(25:24:1)抽提,颠倒混匀50次,13000rpm离心15min。
(8)将上清液吸入新的1.5mL EP管中,加1倍体积异丙醇,颠倒混合,-80℃冷激15min,13000rpm离心10min,弃上清。
(9)加入70%乙醇,轻弹重悬,静置6-7min,13000rpm离心2-5min,去上清。
(10)烘箱干燥(37℃)后,加入50μL去离子水,溶解DNA,测DNA浓度,于-20℃保存。
2.谷氨酸棒杆菌电转化感受态的制备
(1)从-80℃甘油保菌管中用接种针挑取谷氨酸棒杆菌,在LB平板培养基上三区划线,放于32℃培养箱中,过夜培养。
(2)在划线平板培养基上挑取单菌落,接种到5mL的BHIS培养基试管中,30℃,200r/min培养12-14h。
(3)按1%接种量转接到装液量为100mL BHIS培养基(含150μL Tween 80和2.5g甘氨酸)的500mL圆底瓶中,在18℃低温条件下以160rpm培养至少18h开始测定OD600值。
(4)待OD600大约在0.5-0.7之间时放于冰上静置20min。
(5)在超净台中,将菌液收集到50mL离心管中,4℃,6500rpm,离心10min,弃上清。
(6)用50mL预冷的10%甘油重悬洗菌,4℃,6500rpm,离心10min,弃上清,重复洗三次。
(7)用2mL预冷的10%甘油将菌体重悬,用预冷的EP管,100μL/管分装,备注感受态名称、制作时间,-80℃保存备用。
BHIS培养基(g/L):Brain-Heart Infusion(脑心浸提物)37,Sorbital(山梨醇)91,pH 7.0~7.2,115℃高压蒸汽灭菌15min。
3.pK18mobsacB整合载体的构建
(1)目的基因的扩增
根据GenBank公布的谷氨酸棒杆菌13032中trpD基因的序列(Gene ID:1020974)、trpC基因的序列(Gene ID:1020975)和trpB基因的序列(Gene ID:1020976)以及它们的上、下游序列,设计特异性引物(5’-3’)(表1):
表1.引物序列
注:“_”标注的是酶切位点的序列,“□”标注的是构建的突变位点
trpD上、下游同源臂的扩增:以C.glutamicum TP607的基因组为模板,以trpD-up-S和trpD-up-A为引物,进行PCR扩增片段trpD的上游同源臂约500bp;以trpD-down-S和trpD-down-A为引物,进行PCR扩增片段trpD的下游同源臂约500bp。
trpC上、下游同源臂的扩增:以C.glutamicum TP607的基因组为模板,以trpC-up-S和trpC-up-A为引物,进行PCR扩增片段trpC的上游同源臂约500bp;以trpC-down-S和trpC-down-A为引物,进行PCR扩增片段trpC的下游同源臂约500bp。
trpB上、下游同源臂的扩增:以C.glutamicum TP607的基因组为模板,以trpB-up-S和trpB-up-A为引物,进行PCR扩增片段trpB的上游同源臂约500bp;以trpB-down-S和trpB-down-A为引物,进行PCR扩增片段trpB的下游同源臂约500bp。
反应条件为:95℃预变性5min,98℃变性10s,55℃退火5s,72℃延伸30s,30个循环后,72℃最后延伸10min(高保真Prime STAR HS聚合酶)。反应结束后取3μL扩增产物用1%的琼脂糖凝胶电泳检测片段大小。PCR产物回收采用液相回收纯化DNA试剂盒回收。
(2)重叠PCR引物突变位点
trpD定点突变(C446T)的构建方法:以突变位点(C446T)为分界点来划分上下游同源臂,上游同源臂为分界点以上约500bp,下游同源臂为分界点以下约500bp,突变位点设计在trpD-up-A和trpD-down-S上(表1中用□标记的碱基)。以回收的trpD上游同源臂和下游同源臂为模板(添加量为摩尔比1:1),以trpD-up-S和trpD-down-A为引物,进行重叠PCR,得到内含定点突变的重叠片段trp1。
trpC定点突变(C193T)的构建方法:以突变位点(C193T)为分界点来划分上下游同源臂,上游同源臂为分界点以上约500bp,下游同源臂为分界点以下约500bp,突变位点设计在trpC-up-A和trpC-down-S上(表1中用□标记的碱基)。以回收的trpC上游同源臂和下游同源臂为模板(添加量为摩尔比1:1),以trpC-up-S和trpC-down-A为引物,进行重叠PCR,得到内含定点突变的重叠片段trp2。
trpB定点突变的构建方法:以突变位点为分界点来划分上下游同源臂,上游同源臂为分界点以上约500bp,下游同源臂为分界点以下约500bp,突变位点设计在trpB-up-S和trpB-down-A(表1中用□标记的碱基)。以回收的trpB上游同源臂和下游同源臂为模板(添加量为摩尔比1:1),以trpB-up-S和trpB-down-A为引物,进行重叠PCR,得到内含定点突变的重叠片段trp3。
(3)PCR产物回收
PCR产物以及酶切体系采用液相回收纯化DNA试剂盒回收。
(4)酶切和solutionI连接
双酶切回收的重叠片段trp1,trp2和trp3以及载体pK18mobsacB,酶切反应体系见表2,酶切时间0.5-1h,酶切温度37℃。具体的酶切位点为:trp1酶切位点HindIII和PstI;trp2的酶切位点HindIII和PstI;trp3的酶切位点SalI和BamHI(具体酶切位点在表1中用下划线标出)。酶切完成以后,采用液相回收纯化DNA试剂盒回收。将回收的片段和载体进行连接:trp1(HindIII和PstI双酶切)和pK18mobsacB(HindIII和PstI双酶切)连接,连接体系为trp1-1;trp2(HindIII和PstI双酶切)和pK18mobsacB(HindIII和PstI双酶切)连接,连接体系为trp2-1;trp3(SalI和BamHI双酶切)和pK18mobsacB(SalI和BamHI双酶切)连接,连接体系为trp3-1。
连接反应体系10μL:目的片段与载体测定后加入体系中,摩尔比为3:1-9:1之间,Solution I连接酶5μL,其余用去离子水补齐。连接反应条件:16℃,4h。
表2双酶切体系
(5)连接体系的转化与鉴定
用CaCl2法将连接体系trp1-1、trp2-1和trp3-1转化入大肠杆菌DH5α化转感受态细胞。菌落PCR鉴定阳性转化子,引物采用pK18mobsacB的鉴定引物(T载的鉴定引物),其中trp1-1,trp2-1和trp3-1对应的阳性转化子分别为pK18-trp1、pK18-trp2和pK18-trp3。碱性裂解法提取质粒pK18-trp1、pK18-trp2和pK18-trp3DNA,进行质粒的酶切鉴定。
(6)对上述(步骤5)鉴定正确的质粒pK18-trp1、pK18-trp2和pK18-trp3,送去金唯智公司进行测序。
(7)基因序列同源对比
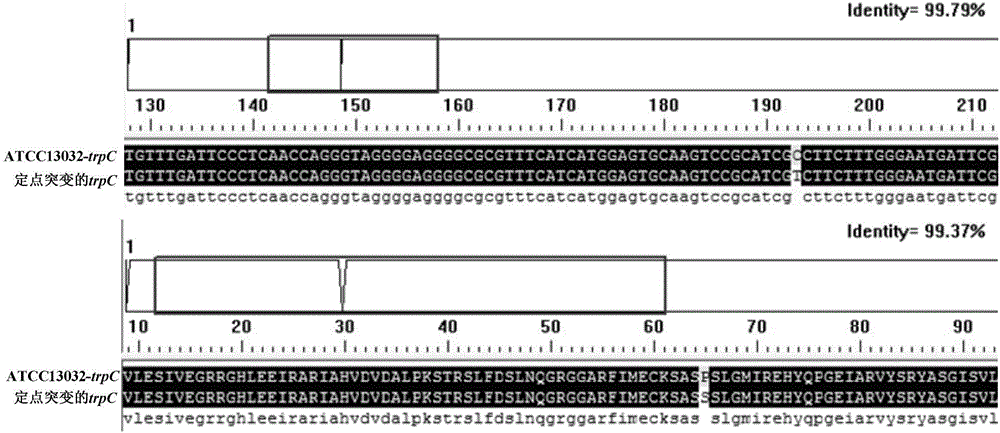
用DNAMAN软件对trpD重叠基因测序结果与GenBank中序列(Gene ID:1020974)进行比对,发现突变株第446位碱基C突变为T(图1)。对应氨基酸残基的变化:第149位丝氨酸变成苯丙氨酸(Ser149Phe)(图1)。同构建的定点突变一致。
使用DNAMAN软件对测得的trpC重叠基因测序与GenBank中序列(Gene ID:1020975)进行序列比对,发现trpC的第193位碱基C突变成T,使得所编码的氨基酸由苯丙氨酸变成丝氨酸(Phe65Ser)。同构建的定点突变一致。
用DNAMAN软件对测得的trpB重叠基因测序与GenBank中序列(Gene ID:1020976)进行序列比对,发现突变株的色氨酸合酶(TS)β链编码基因trpB的第1076-1077位增加了三个碱基ACG,第1079位的C突变为A,第1080为的A突变为G(图3)。对应氨基酸残基的变化为:丙氨酸突变成精氨酸,同时增加了一个谷氨酸残基(图3)。同构建的定点突变一致。
4.谷氨酸棒杆菌基因定点突变整合过程
(1)将整合质粒pK18-trp1电转到C.glutamicum TP607电转感受态中筛选出发生一轮交换的转化子,进一步筛选发生二轮交换的转化子,得到菌株C.glutamicum TP607-trp1(整合上trpD定点突变的菌株);
(2)在步骤(1)得到菌株的基础上,将整合质粒pK18-trp2电转到C.glutamicum TP607-trp1的电转感受态中,筛选出发生一轮交换的转化子,进一步筛选发生二轮交换的转化子,得到菌株C.glutamicum TP607-trp1-trp2(整合上trpD和trpC定点突变的菌株);
(3)在步骤(2)得到菌株的基础上,将整合质粒pK18-trp3电转到C.glutamicum TP607-trp1-trp2的电转感受态中,筛选出发生一轮交换的转化子,进一步筛选发生二轮交换的转化子,得到菌株C.glutamicum TP608(整合上trpD、trpC和trpB定点突变的菌株)
具体的电转化和一轮二轮转化子的筛选步骤如下:
①取适量重组质粒电转化C.glutamicum电转化感受态细胞中;
②电击后,立即加入1mL 46℃预热的复苏培养基BHIS,迅速46℃水浴热激6min,然后32℃、200rpm/min复苏2h;
③复苏完成以后,8000rpm/min离心2min富集菌体,涂布LB抗性平板(Kmr10μg/mL),32℃培养36~48h,将平板上长出的单菌落对点到15%蔗糖LB平板和LB卡那霉素抗性平板(Kmr10μg/mL),32℃培养12~24h;
④挑选出在LB卡那霉素抗性平板(Kmr10μg/mL)上生长,在15%蔗糖LB平板不生长表型的菌落,转接LB摇管,32℃、200rpm/min培养12~24h,提取上述菌落的基因组,以Kana-N和Kana-C为引物,具体引物序列见表3,进行Kana抗性基因PCR,验证发生第一次重组的重组子,保菌;
⑤在重组菌在含有15%蔗糖的摇管中传代两次(由于在15%蔗糖存在情况下,sacB基因表达对宿主菌具有致死效应,因此可用来筛选发生第二次同源重组的菌株);
⑥进行适当的稀释,涂布到15%蔗糖的平板上,32℃培养24~36h,将长出的单菌落对点到15%蔗糖平板和LB卡那霉素抗性平板(Kmr10μg/mL),32℃培养12~24h,筛选出在15%蔗糖平板生长同时在LB卡那霉素抗性平板(Kmr10μg/mL)不再生长表型的单菌落;
⑦将上述表型正确的单菌落转接到LB摇管中,32℃、200rpm/min过夜培养,提取基因组,以基因组DNA为模板,以设计的同源重组引物,进行PCR扩增,使用试剂盒对PCR产物进行回收,将回收的产物进行测序,筛选出发生定点突变的阳性转化子,保菌。
表3.鉴定引物序列
实施例2:摇瓶进行色氨酸发酵
1.菌种
摇瓶发酵所用菌株如下:
C.glutamicumTP607;C.glutamicumTP607-trp1;
C.glutamicumTP607-trp1-trp2;C.glutamicum TP608;
2.菌种活化
斜面培养:取-80℃保藏菌种划线接种于活化斜面,32℃培养24h,并传代一次,32℃培养12h。
活化培养基(g/L):蛋白胨10,牛肉膏10,酵母粉5,NaCl 2.5,玉米浆15mL/L,琼脂25,pH 7.0-7.2,灭菌条件:121℃、20min。
3.种子培养
种子培养:使用500mL三角瓶培养种子,装液量为30mL,菌种使用二代斜面,接种量为两环二代斜面,接种方法为用接种环刮取二代斜面种子,转移到相应的种子培养基中。培养温度32℃,pH 7.0±0.2,转速200rpm,培养时间12h,菌体生物量OD600达到10±2左右,接种到发酵培养基中。
种子培养基(g/L):葡萄糖35,酵母粉5,玉米浆60mL/L,豆粕水解液20mL/L,KH2PO41.5,MgSO40.5,FeSO4·7H2O 0.01,MnSO4·H2O 0.01,VB1 0.001,pH7.0-7.2,灭菌条件:121℃、20min。
4.摇瓶发酵培养
发酵培养:采用摇瓶培养,使用500mL挡板瓶,装液量30mL,接种量为10%,接种方法为使用5mL无菌移液管吸取3ml培养好的种子,接种到发酵培养基中。培养条件为培养温度32℃,转速200rpm,pH 7.0±0.2使用氨水调节pH,发酵周期为36小时。
发酵培养基(g/L):葡萄糖100,玉米浆25mL/L,豆粕水解液25mL/L,(NH4)2SO4 3,KH2PO4 2.2,MgSO4 0.7,FeSO4·7H2O 0.01,MnSO4·H2O 0.01,VB1 0.001,pH 7.0-7.2,灭菌条件:121℃,20min。
5.分析方法
(1)菌体生物量测定:
菌体稀释20倍或100倍,使用V1200紫外分光光度计,在600nm测定样品的吸光度。取一定量样品,烘干测干重,求得OD与细胞干重的对应关系。根据发酵液的OD值计算菌体干重。
(2)色氨酸产量的测定:
氨基酸浓度含量的测定:使用HPLC测定。检测条件为:仪器Agilent 1100高效液相色谱仪;色谱柱Phenomenex Gemini 5u C18(150*4.6mm,美国菲罗门公司);单流动相:10%乙腈;柱温33℃;检测波长278nm;流速1.0m L/min,梯度洗脱。
根据上述方法测定发酵样品中的色氨酸产量,结果见图4。
图4中中横坐标为四个菌株,纵坐标为摇瓶发酵36h样品中色氨酸的含量,单位为每克干重菌体的色氨酸生产能力。由图可以观察到,菌株C.glutamicum TP607-trp1和菌株C.glutamicum TP607相比,色氨酸的产量明显提高,说明trpD突变位点的整合能够提高色氨酸的产量,推测trpD突变位点的引入可能部分解除了色氨酸对该基因编码酶的反馈抑制,提高了该酶的活力;菌株C.glutamicum TP607-trp1-trp2和菌株C.glutamicum TP607-trp1相比,色氨酸的产量提高,说明了trpC突变位点的整合能够提高色氨酸的产量;菌株C.glutamicum TP608和菌株C.glutamicum TP607-trp1-trp2相比色氨酸的产量明显提高,说明trpB突变位点的整合能够提高色氨酸的产量,推测trpB突变位点的引入可能部分解除了色氨酸对该基因编码酶的反馈抑制,提高了该酶的活力。C.glutamicumTP608色氨酸产量约为TP607的3.4倍。
SEQUENCE LISTING
<110> 天津科技大学
莲花健康产业股份有限公司
<120> 一株谷氨酸棒杆菌及其合成色氨酸的关键基因
<130> 1
<160> 24
<170> PatentIn version 3.5
<210> 1
<211> 1047
<212> DNA
<213> CGMCC No.12302
<400> 1
atgacttctc cagcaacact gaaagttctc aacgcctact tggataaccc cactccaacc 60
ctggaggagg caattgaggt gttcaccccg ctgaccgtgg gtgaatacga tgacgtgcac 120
atcgcagcgc tgcttgccac catccgtact cgcggtgagc agttcgctga tattgccggc 180
gctgccaagg cgttcctcgc ggcggctcgt ccgttcccga ttactggcgc aggtttgcta 240
gattccgctg gtactggtgg cgacggtgcc aacaccatca acatcaccac cggcgcatcc 300
ctgatcgcag catccggtgg agtgaagctg gttaagcacg gcaaccgttc ggtgagctcc 360
aagtccggct ccgccgatgt gctggaagcg ctgaatattc ctttgggcct tgatgtggat 420
cgtgctgtga agtggttcga agcgtccaac ttcaccttcc tgttcgcacc tgcgtacaac 480
cctgcgattg cgcatgtgca gccggttcgc caggcgctga aattccccac catcttcaac 540
acgcttggac cattgctgtc cccggcgcgc ccggagcgtc agatcatggg cgtggccaat 600
gccaatcatg gacagctcat cgccgaggtc ttccgcgagt tgggccgtac acgcgcgctt 660
gttgtgcatg gcgcaggcac cgatgagatc gcagtccacg gcaccacctt ggtgtgggag 720
cttaaagaag acggcaccat cgagcattac accatcgagc ctgaggacct tggccttggc 780
cgctacaccc ttgaggatct cgtaggtggc ctcggcactg agaacgccga agctatgcgc 840
gctactttcg cgggcaccgg ccctgatgca caccgtgatg cgttggctgc gtccgcaggt 900
gcgatgttct acctcaacgg cgatgtcgac tccttgaaag atggtgcaca aaaggcgctt 960
tccttgcttg ccgacggcac cacccaggca tggttggcca agcacgaaga gatcgattac 1020
tcagaaaagg agtcttccaa tgactag 1047
<210> 2
<211> 1425
<212> DNA
<213> CGMCC No.12302
<400> 2
atgactagta ataatctgcc cacagtgttg gaaagcatcg tcgagggtcg tcgcggacac 60
ctggaggaaa ttcgcgctcg catcgctcac gtggatgtgg atgcgcttcc aaaatccacc 120
cgttctctgt ttgattccct caaccagggt aggggagggg cgcgtttcat catggagtgc 180
aagtccgcat cgccttcttt gggaatgatt cgtgagcact accagccggg tgaaatcgct 240
cgcgtgtact ctcgctacgc cagcggcatt tccgtgctgt gcgagccgga tcgttttggt 300
ggcgattacg atcacctcgc taccgttgcc gctacctctc atcttccggt gctgtgcaaa 360
gacttcatca ttgatcctgt ccaggtacac gcggcgcgtt actttggtgc tgatgccatc 420
ctgctcatgc tctctgtgct tgatgatgaa gagtacgcag cactcgctgc cgaggctgcg 480
cgttttgatc tggatatcct caccgaggtt attgatgagg aggaagtcgc ccgcgccatc 540
aagctgggtg cgaagatctt tggcgtcaac caccgcaacc tgcatgatct gtccattgat 600
ttggatcgtt cacgtcgcct gtccaagctc attccagcag atgccgtgct cgtgtctgag 660
tctggcgtgc gcgataccga aaccgtccgc cagctaggtg ggcactccaa tgcattcctc 720
gttggctccc agctgaccag ccaggaaaac gtcgatctgg cagcccgcga attagtctac 780
ggccccaaca aagtctgcgg actcacctca ccaagtgcag cacaaaccgc tcgcgcagcg 840
ggtgcggtct acggcgggct catcttcgaa gaggcatcgc cacgcaatgt ttcacgtgaa 900
acattgcaaa aaatcatcgc cgcagagccc aacctgcgct acgtcgcggt cagccgtcgc 960
acctccgggt acaaggattt gcttgtcgac ggcatcttcg ccgtacaaat ccacgcccca 1020
ctgcaggaca gcgtcgaagc agaaaaggca ttgatcgccg ccgttcgtga agaggttgga 1080
ccgcaggtcc aggtctggcg cgcgatctcg atgtccagcc ccttgggggc tgaagtggca 1140
gctgcggtgg agggtgacgt cgataagcta attcttgatg cccatgaagg tggcagcggg 1200
gaagtattcg actgggctac ggtgccggcc gctgtgaagg caaagtcttt gctcgcggga 1260
ggcatctctc cggacaacgc tgcgcaggca ctcgctgtgg gctgcgcagg tttggacatc 1320
aactctggcg tggaataccc cgccggtgca ggcacgtggg ctggggcgaa agacgccggc 1380
gcgctgctga aaattttcgc gaccatctcc acattccatt actaa 1425
<210> 3
<211> 1123
<212> DNA
<213> cgmcc no.12302
<400> 3
atgactgaaa aagaaaactt gggcggctcc acgctgctgc ctgcatactt cggtgaattc 60
ggcggccagt tcgtcgcgga atccctcctg cctgctctcg accagctgga gaaggccttc 120
gttgacgcga ccaacagccc agagttccgc gaagaactcg gcggctacct ccgcgattac 180
ctcggccgcc caaccccgct gaccgaatgc tccaacctgc cactcgcagg cgaaggcaaa 240
ggctttgcgc ggatcttcct caagcgcgaa gacctcgtcc acggcggtgc acacaaaact 300
aaccaggtga tcggccaggt gctgcttgcc aagcgcatgg gcaaaacccg catcatcgca 360
gagaccggcg caggccagca cggcaccgcc accgctctcg catgtgcgct catgggcctc 420
gagtgcgttg tctacatggg cgccaaggac gttgcccgcc agcagcccaa cgtctaccgc 480
atgcagctgc acggcgcgaa ggtcatcccc gtggaatctg gttccggcac cctgaaggac 540
gccgtgaatg aagcgctgcg cgattggacc gcaaccttcc acgagtccca ctaccttctc 600
ggcaccgccg ccggcccgca cccattccca accatcgtgc gtgaattcca caaggtgatc 660
tctgaggaag ccaaggcaca gatgctagag cgcaccggca agcttcccga cgttgtggtc 720
gcctgtgtcg gtggtggctc caacgccatc ggcatgttcg cagacttcat tgacgatgaa 780
ggtgtagagc tcgtcggcgc tgagccagcc ggtgaaggcc tcgactccgg caagcacggc 840
gcaaccatca ccaacggtca gatcggcatc ctgcacggca cccgttccta cctgatgcgc 900
aactccgacg gccaagtgga agagtcctac tccatctccg ccggacttga ttacccaggc 960
gtcggcccac agcacgcaca cctgcacgcc accggccgcg ccacctacgt tggtatcacc 1020
gacgccgaag ccctccaagc attccagtac ctcgcccgct acgaaggcat catccccgca 1080
ctggaatcct cacacgcgtt cgcctacgca ctcaagcgcg cca 1123
<210> 4
<211> 1047
<212> DNA
<213> 人工序列
<400> 4
atgacttctc cagcaacact gaaagttctc aacgcctact tggataaccc cactccaacc 60
ctggaggagg caattgaggt gttcaccccg ctgaccgtgg gtgaatacga tgacgtgcac 120
atcgcagcgc tgcttgccac catccgtact cgcggtgagc agttcgctga tattgccggc 180
gctgccaagg cgttcctcgc ggcggctcgt ccgttcccga ttactggcgc aggtttgcta 240
gattccgctg gtactggtgg cgacggtgcc aacaccatca acatcaccac cggcgcatcc 300
ctgatcgcag catccggtgg agtgaagctg gttaagcacg gcaaccgttc ggtgagctcc 360
aagtccggct ccgccgatgt gctggaagcg ctgaatattc ctttgggcct tgatgtggat 420
cgtgctgtga agtggttcga agcgttcaac ttcaccttcc tgttcgcacc tgcgtacaac 480
cctgcgattg cgcatgtgca gccggttcgc caggcgctga aattccccac catcttcaac 540
acgcttggac cattgctgtc cccggcgcgc ccggagcgtc agatcatggg cgtggccaat 600
gccaatcatg gacagctcat cgccgaggtc ttccgcgagt tgggccgtac acgcgcgctt 660
gttgtgcatg gcgcaggcac cgatgagatc gcagtccacg gcaccacctt ggtgtgggag 720
cttaaagaag acggcaccat cgagcattac accatcgagc ctgaggacct tggccttggc 780
cgctacaccc ttgaggatct cgtaggtggc ctcggcactg agaacgccga agctatgcgc 840
gctactttcg cgggcaccgg ccctgatgca caccgtgatg cgttggctgc gtccgcaggt 900
gcgatgttct acctcaacgg cgatgtcgac tccttgaaag atggtgcaca aaaggcgctt 960
tccttgcttg ccgacggcac cacccaggca tggttggcca agcacgaaga gatcgattac 1020
tcagaaaagg agtcttccaa tgactag 1047
<210> 5
<211> 1425
<212> DNA
<213> 人工序列
<400> 5
atgactagta ataatctgcc cacagtgttg gaaagcatcg tcgagggtcg tcgcggacac 60
ctggaggaaa ttcgcgctcg catcgctcac gtggatgtgg atgcgcttcc aaaatccacc 120
cgttctctgt ttgattccct caaccagggt aggggagggg cgcgtttcat catggagtgc 180
aagtccgcat cgtcttcttt gggaatgatt cgtgagcact accagccggg tgaaatcgct 240
cgcgtgtact ctcgctacgc cagcggcatt tccgtgctgt gcgagccgga tcgttttggt 300
ggcgattacg atcacctcgc taccgttgcc gctacctctc atcttccggt gctgtgcaaa 360
gacttcatca ttgatcctgt ccaggtacac gcggcgcgtt actttggtgc tgatgccatc 420
ctgctcatgc tctctgtgct tgatgatgaa gagtacgcag cactcgctgc cgaggctgcg 480
cgttttgatc tggatatcct caccgaggtt attgatgagg aggaagtcgc ccgcgccatc 540
aagctgggtg cgaagatctt tggcgtcaac caccgcaacc tgcatgatct gtccattgat 600
ttggatcgtt cacgtcgcct gtccaagctc attccagcag atgccgtgct cgtgtctgag 660
tctggcgtgc gcgataccga aaccgtccgc cagctaggtg ggcactccaa tgcattcctc 720
gttggctccc agctgaccag ccaggaaaac gtcgatctgg cagcccgcga attagtctac 780
ggccccaaca aagtctgcgg actcacctca ccaagtgcag cacaaaccgc tcgcgcagcg 840
ggtgcggtct acggcgggct catcttcgaa gaggcatcgc cacgcaatgt ttcacgtgaa 900
acattgcaaa aaatcatcgc cgcagagccc aacctgcgct acgtcgcggt cagccgtcgc 960
acctccgggt acaaggattt gcttgtcgac ggcatcttcg ccgtacaaat ccacgcccca 1020
ctgcaggaca gcgtcgaagc agaaaaggca ttgatcgccg ccgttcgtga agaggttgga 1080
ccgcaggtcc aggtctggcg cgcgatctcg atgtccagcc ccttgggggc tgaagtggca 1140
gctgcggtgg agggtgacgt cgataagcta attcttgatg cccatgaagg tggcagcggg 1200
gaagtattcg actgggctac ggtgccggcc gctgtgaagg caaagtcttt gctcgcggga 1260
ggcatctctc cggacaacgc tgcgcaggca ctcgctgtgg gctgcgcagg tttggacatc 1320
aactctggcg tggaataccc cgccggtgca ggcacgtggg ctggggcgaa agacgccggc 1380
gcgctgctga aaattttcgc gaccatctcc acattccatt actaa 1425
<210> 6
<211> 1126
<212> DNA
<213> 人工序列
<400> 6
atgactgaaa aagaaaactt gggcggctcc acgctgctgc ctgcatactt cggtgaattc 60
ggcggccagt tcgtcgcgga atccctcctg cctgctctcg accagctgga gaaggccttc 120
gttgacgcga ccaacagccc agagttccgc gaagaactcg gcggctacct ccgcgattac 180
ctcggccgcc caaccccgct gaccgaatgc tccaacctgc cactcgcagg cgaaggcaaa 240
ggctttgcgc ggatcttcct caagcgcgaa gacctcgtcc acggcggtgc acacaaaact 300
aaccaggtga tcggccaggt gctgcttgcc aagcgcatgg gcaaaacccg catcatcgca 360
gagaccggcg caggccagca cggcaccgcc accgctctcg catgtgcgct catgggcctc 420
gagtgcgttg tctacatggg cgccaaggac gttgcccgcc agcagcccaa cgtctaccgc 480
atgcagctgc acggcgcgaa ggtcatcccc gtggaatctg gttccggcac cctgaaggac 540
gccgtgaatg aagcgctgcg cgattggacc gcaaccttcc acgagtccca ctaccttctc 600
ggcaccgccg ccggcccgca cccattccca accatcgtgc gtgaattcca caaggtgatc 660
tctgaggaag ccaaggcaca gatgctagag cgcaccggca agcttcccga cgttgtggtc 720
gcctgtgtcg gtggtggctc caacgccatc ggcatgttcg cagacttcat tgacgatgaa 780
ggtgtagagc tcgtcggcgc tgagccagcc ggtgaaggcc tcgactccgg caagcacggc 840
gcaaccatca ccaacggtca gatcggcatc ctgcacggca cccgttccta cctgatgcgc 900
aactccgacg gccaagtgga agagtcctac tccatctccg ccggacttga ttacccaggc 960
gtcggcccac agcacgcaca cctgcacgcc accggccgcg ccacctacgt tggtatcacc 1020
gacgccgaag ccctccaagc attccagtac ctcgcccgct acgaaggcat catcccacgc 1080
gagctggaat cctcacacgc gttcgcctac gcactcaagc gcgcca 1126
<210> 7
<211> 348
<212> PRT
<213> CGMCC No.12302
<400> 7
Met Thr Ser Pro Ala Thr Leu Lys Val Leu Asn Ala Tyr Leu Asp Asn
1 5 10 15
Pro Thr Pro Thr Leu Glu Glu Ala Ile Glu Val Phe Thr Pro Leu Thr
20 25 30
Val Gly Glu Tyr Asp Asp Val His Ile Ala Ala Leu Leu Ala Thr Ile
35 40 45
Arg Thr Arg Gly Glu Gln Phe Ala Asp Ile Ala Gly Ala Ala Lys Ala
50 55 60
Phe Leu Ala Ala Ala Arg Pro Phe Pro Ile Thr Gly Ala Gly Leu Leu
65 70 75 80
Asp Ser Ala Gly Thr Gly Gly Asp Gly Ala Asn Thr Ile Asn Ile Thr
85 90 95
Thr Gly Ala Ser Leu Ile Ala Ala Ser Gly Gly Val Lys Leu Val Lys
100 105 110
His Gly Asn Arg Ser Val Ser Ser Lys Ser Gly Ser Ala Asp Val Leu
115 120 125
Glu Ala Leu Asn Ile Pro Leu Gly Leu Asp Val Asp Arg Ala Val Lys
130 135 140
Trp Phe Glu Ala Ser Asn Phe Thr Phe Leu Phe Ala Pro Ala Tyr Asn
145 150 155 160
Pro Ala Ile Ala His Val Gln Pro Val Arg Gln Ala Leu Lys Phe Pro
165 170 175
Thr Ile Phe Asn Thr Leu Gly Pro Leu Leu Ser Pro Ala Arg Pro Glu
180 185 190
Arg Gln Ile Met Gly Val Ala Asn Ala Asn His Gly Gln Leu Ile Ala
195 200 205
Glu Val Phe Arg Glu Leu Gly Arg Thr Arg Ala Leu Val Val His Gly
210 215 220
Ala Gly Thr Asp Glu Ile Ala Val His Gly Thr Thr Leu Val Trp Glu
225 230 235 240
Leu Lys Glu Asp Gly Thr Ile Glu His Tyr Thr Ile Glu Pro Glu Asp
245 250 255
Leu Gly Leu Gly Arg Tyr Thr Leu Glu Asp Leu Val Gly Gly Leu Gly
260 265 270
Thr Glu Asn Ala Glu Ala Met Arg Ala Thr Phe Ala Gly Thr Gly Pro
275 280 285
Asp Ala His Arg Asp Ala Leu Ala Ala Ser Ala Gly Ala Met Phe Tyr
290 295 300
Leu Asn Gly Asp Val Asp Ser Leu Lys Asp Gly Ala Gln Lys Ala Leu
305 310 315 320
Ser Leu Leu Ala Asp Gly Thr Thr Gln Ala Trp Leu Ala Lys His Glu
325 330 335
Glu Ile Asp Tyr Ser Glu Lys Glu Ser Ser Asn Asp
340 345
<210> 8
<211> 474
<212> PRT
<213> CGMCC No.12302
<400> 8
Met Thr Ser Asn Asn Leu Pro Thr Val Leu Glu Ser Ile Val Glu Gly
1 5 10 15
Arg Arg Gly His Leu Glu Glu Ile Arg Ala Arg Ile Ala His Val Asp
20 25 30
Val Asp Ala Leu Pro Lys Ser Thr Arg Ser Leu Phe Asp Ser Leu Asn
35 40 45
Gln Gly Arg Gly Gly Ala Arg Phe Ile Met Glu Cys Lys Ser Ala Ser
50 55 60
Pro Ser Leu Gly Met Ile Arg Glu His Tyr Gln Pro Gly Glu Ile Ala
65 70 75 80
Arg Val Tyr Ser Arg Tyr Ala Ser Gly Ile Ser Val Leu Cys Glu Pro
85 90 95
Asp Arg Phe Gly Gly Asp Tyr Asp His Leu Ala Thr Val Ala Ala Thr
100 105 110
Ser His Leu Pro Val Leu Cys Lys Asp Phe Ile Ile Asp Pro Val Gln
115 120 125
Val His Ala Ala Arg Tyr Phe Gly Ala Asp Ala Ile Leu Leu Met Leu
130 135 140
Ser Val Leu Asp Asp Glu Glu Tyr Ala Ala Leu Ala Ala Glu Ala Ala
145 150 155 160
Arg Phe Asp Leu Asp Ile Leu Thr Glu Val Ile Asp Glu Glu Glu Val
165 170 175
Ala Arg Ala Ile Lys Leu Gly Ala Lys Ile Phe Gly Val Asn His Arg
180 185 190
Asn Leu His Asp Leu Ser Ile Asp Leu Asp Arg Ser Arg Arg Leu Ser
195 200 205
Lys Leu Ile Pro Ala Asp Ala Val Leu Val Ser Glu Ser Gly Val Arg
210 215 220
Asp Thr Glu Thr Val Arg Gln Leu Gly Gly His Ser Asn Ala Phe Leu
225 230 235 240
Val Gly Ser Gln Leu Thr Ser Gln Glu Asn Val Asp Leu Ala Ala Arg
245 250 255
Glu Leu Val Tyr Gly Pro Asn Lys Val Cys Gly Leu Thr Ser Pro Ser
260 265 270
Ala Ala Gln Thr Ala Arg Ala Ala Gly Ala Val Tyr Gly Gly Leu Ile
275 280 285
Phe Glu Glu Ala Ser Pro Arg Asn Val Ser Arg Glu Thr Leu Gln Lys
290 295 300
Ile Ile Ala Ala Glu Pro Asn Leu Arg Tyr Val Ala Val Ser Arg Arg
305 310 315 320
Thr Ser Gly Tyr Lys Asp Leu Leu Val Asp Gly Ile Phe Ala Val Gln
325 330 335
Ile His Ala Pro Leu Gln Asp Ser Val Glu Ala Glu Lys Ala Leu Ile
340 345 350
Ala Ala Val Arg Glu Glu Val Gly Pro Gln Val Gln Val Trp Arg Ala
355 360 365
Ile Ser Met Ser Ser Pro Leu Gly Ala Glu Val Ala Ala Ala Val Glu
370 375 380
Gly Asp Val Asp Lys Leu Ile Leu Asp Ala His Glu Gly Gly Ser Gly
385 390 395 400
Glu Val Phe Asp Trp Ala Thr Val Pro Ala Ala Val Lys Ala Lys Ser
405 410 415
Leu Leu Ala Gly Gly Ile Ser Pro Asp Asn Ala Ala Gln Ala Leu Ala
420 425 430
Val Gly Cys Ala Gly Leu Asp Ile Asn Ser Gly Val Glu Tyr Pro Ala
435 440 445
Gly Ala Gly Thr Trp Ala Gly Ala Lys Asp Ala Gly Ala Leu Leu Lys
450 455 460
Ile Phe Ala Thr Ile Ser Thr Phe His Tyr
465 470
<210> 9
<211> 374
<212> PRT
<213> CGMCC No.12302
<400> 9
Met Thr Glu Lys Glu Asn Leu Gly Gly Ser Thr Leu Leu Pro Ala Tyr
1 5 10 15
Phe Gly Glu Phe Gly Gly Gln Phe Val Ala Glu Ser Leu Leu Pro Ala
20 25 30
Leu Asp Gln Leu Glu Lys Ala Phe Val Asp Ala Thr Asn Ser Pro Glu
35 40 45
Phe Arg Glu Glu Leu Gly Gly Tyr Leu Arg Asp Tyr Leu Gly Arg Pro
50 55 60
Thr Pro Leu Thr Glu Cys Ser Asn Leu Pro Leu Ala Gly Glu Gly Lys
65 70 75 80
Gly Phe Ala Arg Ile Phe Leu Lys Arg Glu Asp Leu Val His Gly Gly
85 90 95
Ala His Lys Thr Asn Gln Val Ile Gly Gln Val Leu Leu Ala Lys Arg
100 105 110
Met Gly Lys Thr Arg Ile Ile Ala Glu Thr Gly Ala Gly Gln His Gly
115 120 125
Thr Ala Thr Ala Leu Ala Cys Ala Leu Met Gly Leu Glu Cys Val Val
130 135 140
Tyr Met Gly Ala Lys Asp Val Ala Arg Gln Gln Pro Asn Val Tyr Arg
145 150 155 160
Met Gln Leu His Gly Ala Lys Val Ile Pro Val Glu Ser Gly Ser Gly
165 170 175
Thr Leu Lys Asp Ala Val Asn Glu Ala Leu Arg Asp Trp Thr Ala Thr
180 185 190
Phe His Glu Ser His Tyr Leu Leu Gly Thr Ala Ala Gly Pro His Pro
195 200 205
Phe Pro Thr Ile Val Arg Glu Phe His Lys Val Ile Ser Glu Glu Ala
210 215 220
Lys Ala Gln Met Leu Glu Arg Thr Gly Lys Leu Pro Asp Val Val Val
225 230 235 240
Ala Cys Val Gly Gly Gly Ser Asn Ala Ile Gly Met Phe Ala Asp Phe
245 250 255
Ile Asp Asp Glu Gly Val Glu Leu Val Gly Ala Glu Pro Ala Gly Glu
260 265 270
Gly Leu Asp Ser Gly Lys His Gly Ala Thr Ile Thr Asn Gly Gln Ile
275 280 285
Gly Ile Leu His Gly Thr Arg Ser Tyr Leu Met Arg Asn Ser Asp Gly
290 295 300
Gln Val Glu Glu Ser Tyr Ser Ile Ser Ala Gly Leu Asp Tyr Pro Gly
305 310 315 320
Val Gly Pro Gln His Ala His Leu His Ala Thr Gly Arg Ala Thr Tyr
325 330 335
Val Gly Ile Thr Asp Ala Glu Ala Leu Gln Ala Phe Gln Tyr Leu Ala
340 345 350
Arg Tyr Glu Gly Ile Ile Pro Ala Leu Glu Ser Ser His Ala Phe Ala
355 360 365
Tyr Ala Leu Lys Arg Ala
370
<210> 10
<211> 348
<212> PRT
<213> 人工序列
<400> 10
Met Thr Ser Pro Ala Thr Leu Lys Val Leu Asn Ala Tyr Leu Asp Asn
1 5 10 15
Pro Thr Pro Thr Leu Glu Glu Ala Ile Glu Val Phe Thr Pro Leu Thr
20 25 30
Val Gly Glu Tyr Asp Asp Val His Ile Ala Ala Leu Leu Ala Thr Ile
35 40 45
Arg Thr Arg Gly Glu Gln Phe Ala Asp Ile Ala Gly Ala Ala Lys Ala
50 55 60
Phe Leu Ala Ala Ala Arg Pro Phe Pro Ile Thr Gly Ala Gly Leu Leu
65 70 75 80
Asp Ser Ala Gly Thr Gly Gly Asp Gly Ala Asn Thr Ile Asn Ile Thr
85 90 95
Thr Gly Ala Ser Leu Ile Ala Ala Ser Gly Gly Val Lys Leu Val Lys
100 105 110
His Gly Asn Arg Ser Val Ser Ser Lys Ser Gly Ser Ala Asp Val Leu
115 120 125
Glu Ala Leu Asn Ile Pro Leu Gly Leu Asp Val Asp Arg Ala Val Lys
130 135 140
Trp Phe Glu Ala Phe Asn Phe Thr Phe Leu Phe Ala Pro Ala Tyr Asn
145 150 155 160
Pro Ala Ile Ala His Val Gln Pro Val Arg Gln Ala Leu Lys Phe Pro
165 170 175
Thr Ile Phe Asn Thr Leu Gly Pro Leu Leu Ser Pro Ala Arg Pro Glu
180 185 190
Arg Gln Ile Met Gly Val Ala Asn Ala Asn His Gly Gln Leu Ile Ala
195 200 205
Glu Val Phe Arg Glu Leu Gly Arg Thr Arg Ala Leu Val Val His Gly
210 215 220
Ala Gly Thr Asp Glu Ile Ala Val His Gly Thr Thr Leu Val Trp Glu
225 230 235 240
Leu Lys Glu Asp Gly Thr Ile Glu His Tyr Thr Ile Glu Pro Glu Asp
245 250 255
Leu Gly Leu Gly Arg Tyr Thr Leu Glu Asp Leu Val Gly Gly Leu Gly
260 265 270
Thr Glu Asn Ala Glu Ala Met Arg Ala Thr Phe Ala Gly Thr Gly Pro
275 280 285
Asp Ala His Arg Asp Ala Leu Ala Ala Ser Ala Gly Ala Met Phe Tyr
290 295 300
Leu Asn Gly Asp Val Asp Ser Leu Lys Asp Gly Ala Gln Lys Ala Leu
305 310 315 320
Ser Leu Leu Ala Asp Gly Thr Thr Gln Ala Trp Leu Ala Lys His Glu
325 330 335
Glu Ile Asp Tyr Ser Glu Lys Glu Ser Ser Asn Asp
340 345
<210> 11
<211> 474
<212> PRT
<213> 人工序列
<400> 11
Met Thr Ser Asn Asn Leu Pro Thr Val Leu Glu Ser Ile Val Glu Gly
1 5 10 15
Arg Arg Gly His Leu Glu Glu Ile Arg Ala Arg Ile Ala His Val Asp
20 25 30
Val Asp Ala Leu Pro Lys Ser Thr Arg Ser Leu Phe Asp Ser Leu Asn
35 40 45
Gln Gly Arg Gly Gly Ala Arg Phe Ile Met Glu Cys Lys Ser Ala Ser
50 55 60
Ser Ser Leu Gly Met Ile Arg Glu His Tyr Gln Pro Gly Glu Ile Ala
65 70 75 80
Arg Val Tyr Ser Arg Tyr Ala Ser Gly Ile Ser Val Leu Cys Glu Pro
85 90 95
Asp Arg Phe Gly Gly Asp Tyr Asp His Leu Ala Thr Val Ala Ala Thr
100 105 110
Ser His Leu Pro Val Leu Cys Lys Asp Phe Ile Ile Asp Pro Val Gln
115 120 125
Val His Ala Ala Arg Tyr Phe Gly Ala Asp Ala Ile Leu Leu Met Leu
130 135 140
Ser Val Leu Asp Asp Glu Glu Tyr Ala Ala Leu Ala Ala Glu Ala Ala
145 150 155 160
Arg Phe Asp Leu Asp Ile Leu Thr Glu Val Ile Asp Glu Glu Glu Val
165 170 175
Ala Arg Ala Ile Lys Leu Gly Ala Lys Ile Phe Gly Val Asn His Arg
180 185 190
Asn Leu His Asp Leu Ser Ile Asp Leu Asp Arg Ser Arg Arg Leu Ser
195 200 205
Lys Leu Ile Pro Ala Asp Ala Val Leu Val Ser Glu Ser Gly Val Arg
210 215 220
Asp Thr Glu Thr Val Arg Gln Leu Gly Gly His Ser Asn Ala Phe Leu
225 230 235 240
Val Gly Ser Gln Leu Thr Ser Gln Glu Asn Val Asp Leu Ala Ala Arg
245 250 255
Glu Leu Val Tyr Gly Pro Asn Lys Val Cys Gly Leu Thr Ser Pro Ser
260 265 270
Ala Ala Gln Thr Ala Arg Ala Ala Gly Ala Val Tyr Gly Gly Leu Ile
275 280 285
Phe Glu Glu Ala Ser Pro Arg Asn Val Ser Arg Glu Thr Leu Gln Lys
290 295 300
Ile Ile Ala Ala Glu Pro Asn Leu Arg Tyr Val Ala Val Ser Arg Arg
305 310 315 320
Thr Ser Gly Tyr Lys Asp Leu Leu Val Asp Gly Ile Phe Ala Val Gln
325 330 335
Ile His Ala Pro Leu Gln Asp Ser Val Glu Ala Glu Lys Ala Leu Ile
340 345 350
Ala Ala Val Arg Glu Glu Val Gly Pro Gln Val Gln Val Trp Arg Ala
355 360 365
Ile Ser Met Ser Ser Pro Leu Gly Ala Glu Val Ala Ala Ala Val Glu
370 375 380
Gly Asp Val Asp Lys Leu Ile Leu Asp Ala His Glu Gly Gly Ser Gly
385 390 395 400
Glu Val Phe Asp Trp Ala Thr Val Pro Ala Ala Val Lys Ala Lys Ser
405 410 415
Leu Leu Ala Gly Gly Ile Ser Pro Asp Asn Ala Ala Gln Ala Leu Ala
420 425 430
Val Gly Cys Ala Gly Leu Asp Ile Asn Ser Gly Val Glu Tyr Pro Ala
435 440 445
Gly Ala Gly Thr Trp Ala Gly Ala Lys Asp Ala Gly Ala Leu Leu Lys
450 455 460
Ile Phe Ala Thr Ile Ser Thr Phe His Tyr
465 470
<210> 12
<211> 375
<212> PRT
<213> 人工序列
<400> 12
Met Thr Glu Lys Glu Asn Leu Gly Gly Ser Thr Leu Leu Pro Ala Tyr
1 5 10 15
Phe Gly Glu Phe Gly Gly Gln Phe Val Ala Glu Ser Leu Leu Pro Ala
20 25 30
Leu Asp Gln Leu Glu Lys Ala Phe Val Asp Ala Thr Asn Ser Pro Glu
35 40 45
Phe Arg Glu Glu Leu Gly Gly Tyr Leu Arg Asp Tyr Leu Gly Arg Pro
50 55 60
Thr Pro Leu Thr Glu Cys Ser Asn Leu Pro Leu Ala Gly Glu Gly Lys
65 70 75 80
Gly Phe Ala Arg Ile Phe Leu Lys Arg Glu Asp Leu Val His Gly Gly
85 90 95
Ala His Lys Thr Asn Gln Val Ile Gly Gln Val Leu Leu Ala Lys Arg
100 105 110
Met Gly Lys Thr Arg Ile Ile Ala Glu Thr Gly Ala Gly Gln His Gly
115 120 125
Thr Ala Thr Ala Leu Ala Cys Ala Leu Met Gly Leu Glu Cys Val Val
130 135 140
Tyr Met Gly Ala Lys Asp Val Ala Arg Gln Gln Pro Asn Val Tyr Arg
145 150 155 160
Met Gln Leu His Gly Ala Lys Val Ile Pro Val Glu Ser Gly Ser Gly
165 170 175
Thr Leu Lys Asp Ala Val Asn Glu Ala Leu Arg Asp Trp Thr Ala Thr
180 185 190
Phe His Glu Ser His Tyr Leu Leu Gly Thr Ala Ala Gly Pro His Pro
195 200 205
Phe Pro Thr Ile Val Arg Glu Phe His Lys Val Ile Ser Glu Glu Ala
210 215 220
Lys Ala Gln Met Leu Glu Arg Thr Gly Lys Leu Pro Asp Val Val Val
225 230 235 240
Ala Cys Val Gly Gly Gly Ser Asn Ala Ile Gly Met Phe Ala Asp Phe
245 250 255
Ile Asp Asp Glu Gly Val Glu Leu Val Gly Ala Glu Pro Ala Gly Glu
260 265 270
Gly Leu Asp Ser Gly Lys His Gly Ala Thr Ile Thr Asn Gly Gln Ile
275 280 285
Gly Ile Leu His Gly Thr Arg Ser Tyr Leu Met Arg Asn Ser Asp Gly
290 295 300
Gln Val Glu Glu Ser Tyr Ser Ile Ser Ala Gly Leu Asp Tyr Pro Gly
305 310 315 320
Val Gly Pro Gln His Ala His Leu His Ala Thr Gly Arg Ala Thr Tyr
325 330 335
Val Gly Ile Thr Asp Ala Glu Ala Leu Gln Ala Phe Gln Tyr Leu Ala
340 345 350
Arg Tyr Glu Gly Ile Ile Pro Arg Glu Leu Glu Ser Ser His Ala Phe
355 360 365
Ala Tyr Ala Leu Lys Arg Ala
370 375
<210> 13
<211> 34
<212> DNA
<213> 人工序列
<400> 13
cccaagctta tgacttctcc agcaacactg aaag 34
<210> 14
<211> 42
<212> DNA
<213> 人工序列
<400> 14
cgaacaggaa ggtgaagttg aacgcttcga accacttcac ag 42
<210> 15
<211> 42
<212> DNA
<213> 人工序列
<400> 15
ctgtgaagtg gttcgaagcg ttcaacttca ccttcctgtt cg 42
<210> 16
<211> 28
<212> DNA
<213> 人工序列
<400> 16
aaactgcagc aagcaaggaa agcgcctt 28
<210> 17
<211> 31
<212> DNA
<213> 人工序列
<400> 17
cccaagcttc gctacaccct tgaggatctc g 31
<210> 18
<211> 43
<212> DNA
<213> 人工序列
<400> 18
ctcacgaatc attcccaaag aagacgatgc ggacttgcac tcc 43
<210> 19
<211> 43
<212> DNA
<213> 人工序列
<400> 19
ggagtgcaag tccgcatcgt cttctttggg aatgattcgt gag 43
<210> 20
<211> 28
<212> DNA
<213> 人工序列
<400> 20
aaactgcaga cctagctggc ggacggtt 28
<210> 21
<211> 27
<212> DNA
<213> 人工序列
<400> 21
cgcgtcgaca tgaagcgctg cgcgatt 27
<210> 22
<211> 40
<212> DNA
<213> 人工序列
<400> 22
gtgaggattc cagctcgcgt gggatgatgc cttcgtagcg 40
<210> 23
<211> 40
<212> DNA
<213> 人工序列
<400> 23
cgctacgaag gcatcatccc acgcgagctg gaatcctcac 40
<210> 24
<211> 28
<212> DNA
<213> 人工序列
<400> 24
cgcggatcct ccaagccacg ggtgaaag 28
- 还没有人留言评论。精彩留言会获得点赞!