3,7‑二羟基卓酚酮生物合成基因簇及其应用的制作方法
本发明属于微生物基因资源和基因工程领域,具体涉及一种抗生素-3,7二羟基卓酚酮的生物合成基因簇及其应用。
背景技术:
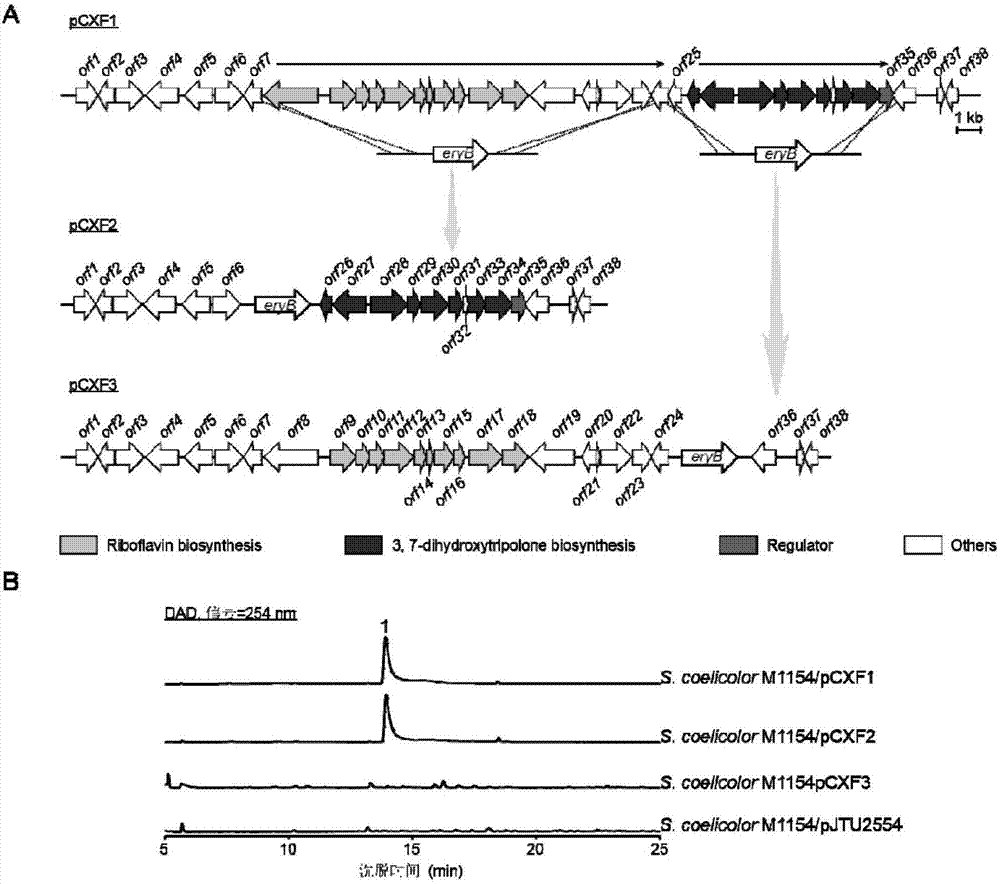
:3,7二羟基卓酚酮是一类具有稀有七元芳香环结构的天然产物,最初于1988年在streptomycestropolofaciensno.k611-97(atcc53548)中发现并报道。3,7二羟基卓酚酮具有非常好的抗革兰氏阳性细菌、革兰氏阴性菌、分枝杆菌及真菌等广谱抗菌活性,同时还具有非常好的抗肿瘤尤其是b16黑色素瘤的活性以及抗病毒如艾滋病毒(hiv)、乙型肝炎病毒(hbv)及疱疹病毒(hsv)等活性。3,7二羟基卓酚酮因其特殊的α-羟基酮的结构,可以螯合金属离子,因而其可以发挥金属离子螯合剂的功能,抑制金属酶的活性。基于卓酚酮类化合物的稀有结构骨架与优异的生物活性,卓酚酮类化合物已经成为了一个非常重要的药物研发的先导化合物(leadcompound),近期对于卓酚酮类化合物尤其是β-thujaplicin的结构改造的工作也取得了很大进展。然而3,7-二羟基卓酚酮由于产量的限制,导致其在发现后的近30年中取得的研究进展十分有限。相对于真菌以及植物中研究的比较透彻的卓酚酮类化合物的生物合成途径,细菌中的卓酚酮类化合物的生物合成途径还没有得到很好的揭示。尤其是3,7-二羟基卓酚酮七元芳香环上的羟基化修饰目前为止还没有报道,然而3,7-二羟基卓酚酮上的羟基化修饰是其螯合金属离子发挥抗菌、抗癌以及抗病毒作用所必需的。因而3,7-二羟基卓酚酮生物合成途径的揭示,对于理解细菌卓酚酮类化合物的合成机理,并进一步利用其进行结构改造具有重要意义。在本发明中,通过高通量异源表达筛选的方法,获得了3,7-二羟基卓酚酮的生物合成基因簇。结合后续的dna测序以及分子生物学与生物化学等技术手段,阐明了3,7-二羟基卓酚酮的生物合成机制,尤其是3,7-二羟基卓酚酮七元芳香环上羟基化修饰。基于此研究,本发明可以利用3,7-二羟基卓酚酮生物合成基因簇中的羟基化酶对其他卓酚酮结构类似物进行修饰,提高相应化合物的生物活性;亦或是在羟基化修饰的基础上进一步的结合化学衍生修饰等手段合成更多活性优良的化合物。技术实现要素:本发明的目的在于提供一种3,7-二羟基卓酚酮的生物合成基因簇及其所包含的蛋白的功能的研究应用。具体地,本发明提供了由基因工程菌株streptomycescoelicolorm1154/pcxf1cgmccno.13916产生的具有广谱抗菌、抗癌以及抗病毒活性的抗生素-3,7-二羟基卓酚酮(3,7-dihydroxytropolone)的生物合成基因簇的克隆、异源表达、测序分析、生物合成途径研究、相关酶的功能研究及其应用。所述天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf1已于2017年3月23日递交中国微生物菌种保藏管理委员会普通微生物中心保藏,保藏地址为北京市朝阳区北辰西路1号院3号,中国科学院微生物研究所,菌种保藏编号为cgmccno.13916。中断3,7-二羟基卓酚酮生物合成基因簇的天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf3已于2017年4月7日递交中国微生物菌种保藏管理委员会普通微生物中心保藏,保藏地址为北京市朝阳区北辰西路1号院3号,中国科学院微生物研究所,菌种保藏编号为cgmccno.14005。中断单加氧酶合成基因trlc的天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf6已于2017年3月23日递交中国微生物菌种保藏管理委员会普通微生物中心保藏,保藏地址为北京市朝阳区北辰西路1号院3号,中国科学院微生物研究所,菌种保藏编号为cgmccno.13918。中断黄素还原酶合成基因trld的天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf7已于2017年3月23日递交中国微生物菌种保藏管理委员会普通微生物中心保藏,保藏地址为北京市朝阳区北辰西路1号院3号,中国科学院微生物研究所,菌种保藏编号为cgmccno.13919。中断单加氧酶合成基因trle的天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf8已于2017年3月23日递交中国微生物菌种保藏管理委员会普通微生物中心保藏,保藏地址为北京市朝阳区北辰西路1号院3号,中国科学院微生物研究所,菌种保藏编号为cgmccno.13920。中断氧化酶合成基因trlf的天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf9已于2017年3月23日递交中国微生物菌种保藏管理委员会普通微生物中心保藏,保藏地址为北京市朝阳区北辰西路1号院3号,中国科学院微生物研究所,菌种保藏编号为cgmccno.13921。本发明的目的是通过以下技术方案来实现的:第一方面,本发明涉及一种3,7-二羟基卓酚酮的生物合成基因簇,所述基因簇包括编码3,7-二羟基卓酚酮生物合成所涉及的10个基因:s1、负责3,7-二羟基卓酚酮生物合成的结构基因,包括trla、trlb、trlc、trld、trle、trlf、trlh以及trli;其中,trla位于如seqidno.1所示基因簇核苷酸序列的第1-480位,编码酰基辅酶a脱水酶,大小为159个氨基酸;trlb位于如seqidno.1所示基因簇核苷酸序列的第530-1984位,编码3-脱氧-d-阿糖基庚酮酸-7-磷酸合成酶,大小为484个氨基酸;trlc位于如seqidno.1所示基因簇核苷酸序列的第2248-3840位,编码单加氧酶,大小为530个氨基酸;trld位于如seqidno.1所示基因簇核苷酸序列的第3837-4394位,编码黄素还原酶,大小为185个氨基酸;trle位于如seqidno.1所示基因簇核苷酸序列的第4439-5641位,编码单加氧酶,大小为400个氨基酸;trlf位于如seqidno.1所示基因簇核苷酸序列的第5697-6308位,编码氧化酶,大小为203个氨基酸;trlh位于如seqidno.1所示基因簇核苷酸序列的第6536-7297位,编码硫酯酶,大小为253个氨基酸;trli位于如seqidno.1所示基因簇核苷酸序列的第7299-8508位,编码预苯酸脱水酶-分支酸变位酶双功能酶,大小为402个氨基酸;s2、负责3,7-二羟基卓酚酮生物合成的调控基因,包括trlj;trlj位于如seqidno.1所示基因簇核苷酸序列的第8483-9112位,编码tetr家族转录调控蛋白,大小为209个氨基酸;s3、其他功能蛋白基因,包括trlg;trlg位于如seqidno.1所示基因簇核苷酸序列的第6327-6539位,编码其他功能蛋白,大小为70个氨基酸。优选的,所述基因簇的碱基序列如seqidno.1中第1-9112位所示。第二方面,本发明还涉及一种上述3,7-二羟基卓酚酮的生物合成基因簇在催化合成3,7-二羟基卓酚酮及其类似物中的用途。第三方面,本发明还涉及一种上述3,7-二羟基卓酚酮生物合成基因簇编码的蛋白。优选的,编码蛋白的氨基酸序列如seqidno.2-11所示。其中,如seqidno.4所示的蛋白为单加氧酶trlc。如seqidno.5所示的蛋白为黄素还原酶trld。如seqidno.6所示的蛋白为单加氧酶trle。如seqidno.7所示的蛋白为氧化酶trlf。第四方面,本发明还涉及一种上述3,7-二羟基卓酚酮的生物合成基因簇编码的蛋白在催化合成3,7-二羟基卓酚酮及其类似物中的用途。第五方面,本发明还涉及一种含有上述3,7-二羟基卓酚酮的生物合成基因簇的表达载体。优选的,所述表达载体的大小为46,192bp,携带39,142bp的外源片段。第六方面,本发明还涉及一种携带上述的表达载体,即染色体上整合有外源的上述3,7-二羟基卓酚酮生物合成基因簇的重组表达菌株。优选的,所述的重组表达菌株是天蓝色链霉菌。第七方面,本发明还涉及一种基因工程菌株,所述基因工程菌株携带上述3,7-二羟基卓酚酮生物合成基因簇。优选的,所述基因工程菌株为天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf1cgmccno.13916。第八方面,本发明还涉及一种基因工程菌株,所述菌株携带上述3,7-二羟基卓酚酮的生物合成基因簇,且其中的某一种或几种基因被失活。优选的,3,7-二羟基卓酚酮生物合成基因簇被基因失活的基因工程菌株为天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf3cgmccno.14005。优选的,3,7-二羟基卓酚酮生物合成基因簇缺失的菌株不能产生3,7-二羟基卓酚酮。优选的,trlc基因失活的基因工程菌株为天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf6cgmccno.13918。优选的,trlc的失活菌株不能产生3,7-二羟基卓酚酮,但能合成3,7-二羟基卓酚酮的前体卓酚酮(tropolone)。优选的,trld基因失活的基因工程菌株为天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf7cgmccno.13919。优选的,trld的失活菌株不能产生3,7-二羟基卓酚酮,但能合成3,7-二羟基卓酚酮的前体7-羟基卓酚酮(tropolone)。优选的,trle基因失活的基因工程菌株为天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf8cgmccno.13920。优选的,trle的失活菌株不能产生3,7-二羟基卓酚酮,但能合成3,7-二羟基卓酚酮的前体2,4,6-环庚三烯酮(tropone)。优选的,trlf基因失活的基因工程菌株为天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf9cgmccno.13921。优选的,trlf的失活菌株不能产生3,7-二羟基卓酚酮,但能合成3,7-二羟基卓酚酮的前体1,4,6-环庚三烯-1-羧酸(cyclohepta-1,4,6-triene-1-formicacid)。第九方面,本发明还涉及一种中断3,7-二羟基卓酚酮生物合成基因簇的天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf3cgmccno.14005。第十方面,本发明还涉及一种中断单加氧酶合成基因trlc的天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf6cgmccno.13918。第十一方面,本发明还涉及一种中断黄素还原酶合成基因trld的天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf7cgmccno.13919。第十二方面,本发明还涉及一种中断氧化酶合成基因trle的天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf8cgmccno.13920。第十三方面,本发明还涉及一种中断氧化酶合成基因trlf的天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf9cgmccno.13921。第十四方面,本发明还涉及一种上述的天蓝色链霉菌在催化合成卓酚酮、7-羟基卓酚酮、环庚三烯酮或1,4,6-环庚三烯-1-羧酸中的用途。其中,天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf6cgmccno.13918用于催化合成卓酚酮。天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf7cgmccno.13919用于催化合成7-羟基卓酚酮和少量卓酚酮。天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf8cgmccno.13920用于催化合成2,4,6-环庚三烯酮。天蓝色链霉菌(streptomycescoelicolor)m1154/pcxf9cgmccno.13921用于催化合成1,4,6-环庚三烯-1-羧酸。本发明的原理在于:卓酚酮类化合物是一类具有稀有七元芳香环结构的次级代谢产物,具有非常好的抗菌、抗癌以及抗病毒的活性。相对于真菌以及植物中卓酚酮类化合物的生物合成研究报道,微生物中的卓酚酮类化合物的生物合成机制仍然不是很清楚。由于原核生物中的卓酚酮类化合物的生物合成机制未知,因而从dna序列的角度去钓取3,7-二羟基卓酚酮的生物合成基因簇就显得无从下手。然而微生物次级代谢产物往往是聚集成簇的排列,根据这一特性,本发明利用高通量异源表达的方法,结合生物活性筛选,确定了3,7-二羟基卓酚酮的生物合成基因簇,并在异源表达菌株天蓝色链霉菌中实现了表达。进一步的利用dna测序、遗传学、分子生物学以及生物化学等分析手段与方法,解析了3,7-二羟基卓酚酮的生物合成途径。通过体内的敲除实验和积累代谢产物的分离纯化鉴定,通过体外生化实验,确证了多步氧化酶的功能和生物活性,为后续的改造和利用提供了依据。与现有技术相比,本发明具有如下有益成果:1)本发明首次确定了3,7-二羟基卓酚酮的生物合成基因簇,碱基序列如seqidno.1所示,全长9112个碱基。所述基因簇来源于赭石菌核链霉菌s.ochraceiscleroticusb222;通过异源表达,测序分析及功能研究,确定了3,7-二羟基卓酚酮的生物合成基因簇共包含10个基因;其中trla、trlb、trlc、trld、trle、trlf、trlh以及trli为结构基因;trlg为其他功能基因;trlj为调控基因;本发明所提供的基因及其蛋白可以用来寻找和发现用于医药、工业或农业的化合物、基因或蛋白。2)本发明在异源表达宿主天蓝色链霉菌(streptomycescoelicolor)m1154中实现了3,7-二羟基卓酚酮生物合成基因簇的高效表达;3)本发明通过测序、分子生物学以及生物化学等手段解析了3,7-二羟基卓酚酮的生物合成途径;4)本发明通过单基因敲除获得了一系列的基因缺失菌株以及3,7-二羟基卓酚酮生物合成的前体;为发酵生产卓酚酮、7-羟基卓酚酮、环庚三烯酮以及1,4,6-环庚三烯-1-羧酸提供了菌种来源;5)本发明报道了首例催化七元芳香环上羟化的蛋白,并确定了trlcd以及trle负责了3,7-二羟基卓酚酮七元芳香环上2,3,7位的顺序羟化反应。附图说明通过阅读参照以下附图对非限制性实施例所做的详细描述,本发明的其他特征、目的和优点将会变得更为突出:图1为异源表达筛选3,7-二羟基卓酚酮生物合成基因簇的生物活性检测图(a)、s.coelicolorm1154/pcxf1表型图(b)以及3,7-二羟基卓酚酮异源表达液相检测图谱(c);图2为3,7-二羟基卓酚酮生物合成基因簇的确定示意图;其中,a,pcxf1质粒的大片段缺失示意图;b,pcxf1大片段敲除质粒异源表达产物液相检测图谱;图3为3,7-二羟基卓酚酮生物合成基因簇及其基因功能示意图;图4为3,7-二羟基卓酚酮生物合成基因簇中trlc单基因敲除示意图、凝胶检测图及hplc检测图谱;其中,a,pcr-targeting敲除trlc基因示意图;b,敲除突变质粒的凝胶检测图;c,trlc敲除质粒异源表达菌株液相检测图谱;图5为3,7-二羟基卓酚酮生物合成基因簇中trld单基因敲除示意图、凝胶检测图及hplc检测图谱;其中,a,pcr-targeting敲除trld基因示意图;b,敲除突变质粒的凝胶检测图;c,trld敲除质粒异源表达菌株液相检测图谱;图6为3,7-二羟基卓酚酮生物合成基因簇中trle单基因敲除示意图、凝胶检测图及hplc检测图谱;其中,a,pcr-targeting敲除trle基因示意图;b,敲除突变质粒的凝胶检测图;c,trle敲除质粒异源表达菌株液相检测图谱;图7为3,7-二羟基卓酚酮生物合成基因簇中trlf单基因敲除示意图、凝胶检测图及hplc检测图谱;其中,a,pcr-targeting敲除trlf基因示意图;b,敲除突变质粒的凝胶检测图;c,trlf敲除质粒异源表达菌株液相检测图谱;图8为化合物2的核磁及化学结构图;其中,a为氢谱图,b为碳谱图;图9为化合物5的核磁及化学结构图;其中,a为氢谱图,b为碳谱图;图10为3,7-二羟基卓酚酮生物合成途径示意图。具体实施方式下面结合附图和具体实施例对本发明进行详细说明,以下实例将有助于本领域从业人员更深入地理解本发明。以下实施例中未特殊说明的实验方法均为常规方法,未特殊说明的材料、试剂均可从商业途径获取。lb培养基:用于大肠杆菌的培养;胰蛋白胨10g,酵母提取物5g,氯化钠5g,去离子水定容至1,000ml,ph7.0。2×yt培养基:大肠杆菌-链霉菌属间接合转移;胰蛋白胨16g,酵母提取物10g,氯化钠5g,去离子水定容至1,000ml,ph7.0。sfm培养基:链霉菌培养产孢;黄豆饼粉20g,甘露醇20g,自来水定容至1,000ml,ph7.2~7.4。黄豆饼粉20g先用1,000ml自来水浸泡,121℃灭菌20min,然后过滤收集黄豆饼粉浸提液配制培养基。r3培养基:链霉菌发酵;葡萄糖10g,酵母提取物5g,酪蛋白氨基酸0.1g,l-脯氨酸3g,mgcl2·6h2o10g,cacl2·2h2o4g,k2so40.25g,kh2po40.05g,tes5.6g,微量元素溶液2ml,去离子水定容至1,000ml,ph7.2。微量元素溶液(/l)包括:相应固体培养基需要添加15g琼脂粉,sfm固体培养基添加20g琼脂粉。本发明所涉及的赭石菌核链霉菌s.ochraceiscleroticusb222菌株,分离自神农架山区,在文献“石春芝,吴后波,岳莹玉,陶天申.神农架林区和自然保护区链霉菌资源考察[j].氨基酸和生物资源,2002,(02):1-4”中公开过。异源表达宿主天蓝色链霉菌(streptomycescoelicolor)m1154在文献“gomez-escribanojp,bibbmj.engineeringstreptomycescoelicolorforheterologousexpressionofsecondarymetabolitegeneclusters.microbialbiotechnology,2011,4(2):207-215.”中公开过,公众可以从johninnescentre的mervynjbibb教授处获取。构建cosmid文库所用载体pjtu2554在文献“lil,xuz,xux,etal.themildiomycinbiosynthesis:initialstepsforsequentialgenerationof5-hydroxymethylcytidine5′-monophosphateand5-hydroxymethylcytosineinstreptoverticilliumrimofacienszju5119.chembiochem,2008,9(8):1286-1294.”中公开过,公众可以从上海交通大学邓子新教授处获取。cosmid文库宿主e.colixl-1bluemr/puz8002菌株在文献“chenl,wangy,guoh,etal.high-throughputscreeningforstreptomycesantibioticbiosynthesisactivators.appliedandenvironmentalmicrobiology,2012,78(12):4526-4528.”中公开过,公众可以从上海交通大学陶美凤教授处获得。实施例1、streptomycessp.b222cosmid文库的构建(一)取streptomycessp.b222新鲜孢子接种至30mltsby培养基(添加0.6%甘氨酸)中,30℃,220rpm培养16-24h。(二)离心收集菌体,称取0.5g湿菌体至50ml离心管中,加入5mlset(75mmnacl,25mmedta,20mmtris,ph7.5)缓冲液,振荡打散。加入终浓度为1mg/ml的溶菌酶,37℃温浴10min。加入1/10体积的10%sds溶液后,再加入终浓度为0.5mg/ml的蛋白酶k,55℃温浴2h。加入1/3体积的5mnacl溶液,并加入一倍体积的中性酚仿,室温颠倒混匀30min;4,000rpm离心30min后吸取上清至另一新的50ml离心管中,加入等体积氯仿再次颠倒混匀30min除去残留的蛋白。4,000rpm离心30min后吸取上清至另一新的50ml离心管中,加入两倍体积的无水乙醇沉淀基因组dna。挑取白色絮状的基因组dna至30ml75%无水乙醇中,室温静置10min洗去盐离子,重复75%无水乙醇洗涤步骤。倒去75%无水乙醇,室温晾干dna后,加入3mlte(ph8.0)溶解。(三)利用限制性内切酶sau3ai对streptomycessp.b222基因组dna进行部分酶切。设置梯度酶切体系(12个梯度),每个梯度酶切体系为200μl。第一管中加入200μl基因组dna,40μl10×h缓冲液,159μl无菌水以及1μlsau3ai;第2至12管中加入20μl10×h缓冲液以及180μl基因组dna。从第一管溶液中吸取200μl溶液至第2管中混合均匀得到sau3ai稀释两倍的酶切体系,进一步再从第2管中吸取200μl溶液至第3管中混合均匀得到sau3ai稀释四倍的酶切体系,依此类推稀释至第12管,获得相应稀释度的酶切体系。37℃温浴30min后,65℃温浴20min失活。每管取10μl酶切样品进行pfge检测,检测条件为:initialswitchtime:1s;finalswitchtime:6s;voltage:6v/cm;includedangle:120°;runtime:14-16h;temperature:14℃。选择酶切大小为45-60kb左右的梯度体系,混合进行pfge凝胶电泳回收dna,方法同上。切下45-60kb左右胶条用低熔点琼脂糖凝胶制备一块新的凝胶,再次进行pfge检测除去携带的小片段dna,检测条件为:initialswitchtime:4s;finalswitchtime:4s;voltage:6v/cm;includedangle:120°;runtime:14-16h;temperature:14℃。切下45-60kb左右胶条回收基因组dna,溶于10μlte(ph8.0)中。(四)对总dna进行去磷酸化处理,去磷酸化体系为:8μl基因组dna,1μlfastap缓冲液,1μlfastap。37℃温浴15min后,于75℃温浴5min失活。将去磷酸化的基因组dna与经hpai与bamhi双酶切处理的pjtu2554载体进行酶连,酶连体系为:10μl基因组dna去磷酸化溶液,1μlpjtu2554(经hpai酶切去磷酸化,然后经bamhi酶切回收载体),1μl10×fast-linkligasebuffer,1μl10mmratp,1μlfast-linkligase。(五)利用maxplaxtmlamdapackagingextracts对酶连产物进行包装并转染e.colixl-1bluemr/puz8002感受态细胞。涂布lb平板(添加0.1%阿伯拉霉素与0.1%卡那霉素),筛选转染子,获得一个包含2,000个克隆的cosmid文库。将克隆挑至96孔板中置于-80℃进行保存。实施例2、3,7-二羟基卓酚酮生物合成基因簇的克隆链霉菌基因组文库异源表达活性筛选方法可参考文献“chenl,wangy,guoh,etal.high-throughputscreeningforstreptomycesantibioticbiosynthesisactivators.appliedandenvironmentalmicrobiology,2012,78(12):4526-4528.”与文献“xum,wangy,zhaoz,etal.functionalgenomeminingformetabolitesencodedbylargegeneclustersthroughheterologousexpressionofawhole-genomebacterialartificialchromosomelibraryinstreptomycesspp.appliedandenvironmentalmicrobiology,2016,82(19):5795-5805.”(一)从-80℃冰箱中取出streptomycesochraceiscleroticusb222(即,s.ochraceiscleroticusb222)的cosmid文库(12块96孔板),37℃培养箱放置2h至融化完全。(二)在96孔板中加入130μl新鲜lb培养基,利用复制器将streptomycessp.b222的cosmid文库接种至96孔板中,37℃,220rpm培养4~6h至od6000.4~0.6。(三)利用复制器将大肠杆菌菌液影印到均匀涂布了一层热激过的变铅青链霉菌(streptomyceslividans)sbt5孢子的sfm平板,在生物安全柜中吹干,30℃培养12~16h。(四)在sfm平板上覆盖三甲氧苄啶和阿泊拉霉素至终浓度为50μg/ml,在生物安全柜中吹干,30℃培养4~6天至接合转移子长出。(五)利用复制器将长出的接合转移子影印至含有萘啶酮酸(25μg/ml)和阿泊拉霉素(50μg/ml)的sfm平板上除去残留的大肠杆菌和受体菌变铅青链霉菌(streptomyceslividans)sbt5,30℃培养4~6天至产孢完全。(六)利用复制器将产孢的接合转移子转移至发酵培养基r3上,30℃发酵培养4~6天至产素完全。(七)在产素完全的平板上覆盖一层含有1%枯草芽孢杆菌的软琼脂(0.5%琼脂),37℃培养过夜,观察抑菌圈的生成。观察到的抑菌圈如图1a所示,11a11(表达3,7-二羟基卓酚酮的接合转移子)克隆发酵产生的化合物能够显著抑制生测指示菌枯草芽孢杆菌的生长。实施例3、3,7-二羟基卓酚酮的异源表达(一),从streptomycesochraceiscleroticusb222的cosmid文库文库中挑取11a11克隆至3ml新鲜lb培养基(添加0.1%阿伯拉霉素)中,37℃,220rpm培养过夜。(二),将提取11a11的cosmiddna转化大肠杆菌e.coliet12567电转感受态细胞,得到e.coliet12567/11a11(e.coliet12567/pcxf1)菌株,为描述方便,后续11a11cosmid质粒均以pcxf1表示。(三),利用大肠杆菌-链霉菌三亲本接合转移的方法,以e.coliet12567/pcxf1为供体菌,s.coelicolorm1154为受体菌,在e.coliet12567/pub307的辅助下,将pcxf质粒转入s.coelicolorm1154中,得到3,7-二羟基卓酚酮的表达菌株s.coelicolorm1154/pcxf1cgmccno.,如图1b所示;由图1b可知,3,7-二羟基卓酚酮的表达对宿主s.coelicolorm1154有明显地抑制作用。s.coelicolorm1154/pcxf1较之于野生型s.coelicolorm1154菌株不能产孢。(四),挑取三个s.coelicolorm1154/pcxf1菌株的接合转移子接种至r3固体培养基上,30℃发酵4~6天。(五),发酵结束后,收集r3固体发酵培养物,利用等体积乙酸乙酯(0.1%乙酸)萃取3遍。收集乙酸乙酯萃提液,真空浓缩旋干,再用1ml甲醇溶解粗样。(六),12,000rpm离心10min或用0.22μm滤膜过滤甲醇样品后,进行高效液相色谱分析,进样量为10μl。液相分析色谱柱型号为:agilentzorbaxsb-c18(5μm,4.6×250mm)。流动相为:a相为水相(添加1‰三氟乙酸),b相为乙腈。分析条件为:0min,5%b;0-15min,b相由5%浓度升至40%;15-25min,b相由40%浓度升至100%;25-30min,b相100%浓度维持5min:30-31min,b相由100%浓度降至5%;31-38min,b相5%浓度平衡7min。流速为0.6ml/min,室温,流动相比例按体积比计算。如图1c所示,3,7-二羟基卓酚酮在s.coelicolorm1154菌株中能够正常表达,出峰时间位于14min。实施例4、3,7-二羟基卓酚酮的生物合成基因簇的确定pcxf1大小为46,192bp,携带有39,142bp的外源片段,对这39,142bp的外源片段序列进行基因功能分析发现其上包含一簇核黄素合成相关基因(orf9-orf18)与一簇芳香类化合物代谢相关基因(orf26-orf35)。为了确定pcxf1上与3,7-二羟基卓酚酮生物合成相关的基因,本发明对pcxf1质粒进行了大片段的敲除。利用pcr-targeting技术分别敲除了orf7-orf25与orf25-orf35这两段大片段dna,获得pcxf2与pcxf3质粒。pcr-targeting技术可参考文献“bertoltgust,gregl.challis,kayfowler,tobiaskieser,andkeithf.chater;pcr-targetedstreptomycesgenereplacementidentifiesaproteindomainneededforbiosynthesisofthesesquiterpenesoilodorgeosmin,pnas2003,vol.100,no.4,1541-1546”。orf7-orf25大片段敲除引物序列如下:seqidno.12(δrib-f,5′-3′):gccaccgcaccgccgtacgggccgggccgcggccgtccgatattccggggatccgtcgacseqidno.13(δrib-r,5′-3′):gcggccgacgatctgctggtggacgtcggcttccacgccgtgtaggctggagctgcttcorf7-orf25大片段敲除验证引物序列如下:seqidno.14(ver-δrib-f,5′-3′):atgtcaccgctgcgcaccgaseqidno.15(ver-δrib-r,5′-3′):acctccggtacggaccagga1)利用δrib-f/r引物从pjtu6722上扩增红霉素抗性基因eryb,利用pcr-targeting技术将红霉素抗性基因eryb基因插入pcxf1质粒中替换orf7-orf25大片段dna,得到orf7-orf25的敲除质粒pcxf2,如图2a所示。2)将pcxf2质粒大肠杆菌-链霉菌属间三亲本接合转移转入s.coelicolorm1154中即可获得orf7-orf25大片段敲除的表达菌株s.coelicolorm1154/pcxf2。3)将敲除菌株s.coelicolorm1154/pcxf2接种至r3发酵培养基上,30℃发酵培养4-6天后提取发酵产物进行液相检测。液相检测图谱如图2b所示,14min的3,7-二羟基卓酚酮的洗脱峰仍然存在,表明这段基因不参与3,7-二羟基卓酚酮的生物合成,s.coelicolorm1154/pcxf2仍然能够合成3,7-二羟基卓酚酮。orf25-orf35大片段敲除引物序列如下:seqidno.16(δaro-f,5′-3′):gcgacgggtgcgacgggtgcggaacatgggagcggccctatattccggggatccgtcgacseqidno.17(δaro-r,5′-3′):gtgtggctccgccacgccgtccgctccgcgctgacccgggtgtaggctggagctgcttcorf25-orf35大片段敲除验证引物序列如下:seqidno.18(ver-δaro-f,5′-3′):tcacgaacagcttcacccactseqidno.19(ver-δaro-r,5′-3′):ggcatcgaactgttcgacctt1)利用δaro-f/r引物从pjtu6722上扩增红霉素抗性基因eryb,利用pcr-targeting技术将eryb基因插入pcxf1中替换orf25-orf35大片段dna,得到orf25-orf35的敲除质粒pcxf3,如图2a所示。2)将pcxf3质粒大肠杆菌-链霉菌属间三亲本接合转移转入s.coelicolorm1154中即可获得orf25-orf35大片段敲除的表达菌株s.coelicolorm1154/pcxf3。3)将敲除菌株s.coelicolorm1154/pcxf3接种至r3发酵培养基上,30℃发酵培养4-6天后提取发酵产物进行液相检测。液相检测图谱如图2b所示,14min的3,7-二羟基卓酚酮的洗脱峰消失,表明这段基因参与3,7-二羟基卓酚酮的生物合成。因而3,7-二羟基卓酚酮的生物合成基因簇应当包含orf26-orf35这段区域内的10个基因,共9112bp。各基因编码的蛋白质及其功能分析见表1,基因组织排布如图3所示。核苷酸序列见seqidno.1,蛋白质序列见seqidno.2-11。表1,3,7-二羟基卓酚酮生物合成基因簇蛋白功能注释基因基因位置蛋白序列蛋白功能trla1-480seqidno.2酰基辅酶a水合酶trlb530-1984seqidno.33-脱氧-d-阿糖基庚酮酸-7-磷酸合成酶trlc2248-3840seqidno.4单加氧酶trld3837-4394seqidno.5黄素还原酶trle4439-5641seqidno.6单加氧酶trlf5697-6308seqidno.7氧化酶trlg6327-6539seqidno.8未知功能trlh6536-7297seqidno.9硫酯酶trli7299-8507seqidno.10预苯酸脱水酶-分支酸变位酶trlj8483-9112seqidno.11tetr转录调控蛋白实施例5、3,7-二羟基卓酚酮生物合成基因trlc的敲除细菌来源的卓酚酮类化合物的七元芳香环的合成已经由robintuefel等在文献“teufelr,friedricht,fuchsg.anoxygenasethatformsanddeoxygenatestoxicepoxide.nature,2012,483(7389):359-362.”中进行了报道。环庚三烯酚酮七元芳香环是由分支酸途径合成的苯乙酸经苯乙酸代谢途径后合成。3,7-二羟基卓酚酮生物合成基因簇中的trlb及trli基因编码的蛋白是苯乙酸合成所必需的蛋白,表明3,7-二羟基卓酚酮七元芳香环骨架的合成可能采用了类似的苯乙酸代谢途径。然而3,7-二羟基卓酚酮结构上的羟基化的修饰及其催化顺序仍然未知,本发明将目标锁定在trlc、trld、trle及trlf四个氧化还原反应相关的蛋白上。利用pcr-targeting技术对trlc基因进行单基因敲除:trlc基因的敲除引物序列如下:seqidno.20(δtrlc-f,5′-3′):acgagtacctggagagcctccgggacggccgggaagtcaggatctatattccggggatccgtcgacseqidno.21(δtrlc-r,5′-3′):cgtacttgtccaggtagggccggacctcggggttggaccagtcgatgtgtaggctggagctgcttctrlc基因敲除验证引物序列如下:seqidno.22(ver-δtrlc-f,5′-3′):caccggctcgaaaggtgatcseqidno.23(ver-δtrlc-r,5′-3′):gtagttcatctcgtacagctc1)利用δtrlc-f/r引物从pjtu6722上扩增红霉素抗性基因eryb,利用pcr-targeting技术将eryb基因插入pcxf1中替换trlc,得到trlc的敲除质粒pcxf1(δtrlc::eryb)。将pcxf1(δtrlc::eryb)质粒转入bt340菌株中,在flp重组酶的作用下,将eryb基因抹除,留下81bp的scar片段,得到trlc基因敲除质粒pcxf6,如图4a所示。pcxf6敲除质粒的验证胶图如图4b所示。野生型的pcr产物大小为1401bp,敲除质粒的pcr产物大小为358bp。2)将pcxf6质粒大肠杆菌-链霉菌属间三亲本接合转移转入s.coelicolorm1154中即可获得trlc基因敲除的表达菌株s.coelicolorm1154/pcxf6。3)将敲除菌株s.coelicolorm1154/pcxf6接种至r3发酵培养基上,30℃发酵培养4-6天后提取发酵产物进行液相检测。液相检测图谱如图4c所示,14min的3,7-二羟基卓酚酮的洗脱峰消失,在16.5min出现了一个新的洗脱峰3。实施例6、3,7-二羟基卓酚酮生物合成基因trld的敲除同上利用pcr-targeting技术对trld基因进行单基因敲除:trld基因的敲除引物序列如下:seqidno.24(δtrld-f,5′-3′):gagcccccgtcctgccggaccaggcccaactcaggcacgtactcgatattccggggatccgtcgacseqidno.25(δtrld-r,5′-3′):gacagccggtgtgcgggccgacggcccagtcggcggcctgctccatgtgtaggctggagctgcttctrld基因敲除验证引物序列如下:seqidno.26(ver-δtrld-f,5′-3′):gagtacgacgagcacggctgseqidno.27(ver-δtrld-r,5′-3′):cctgttcgctgccgaaggc1)利用δtrld-f/r引物从pjtu6722上扩增红霉素抗性基因eryb,利用pcr-targeting技术将eryb基因插入pcxf1中替换trld,得到trld的敲除质粒pcxf1(δtrld::eryb)。将pcxf1(δtrld::eryb)质粒转入bt340菌株中,在flp重组酶的作用下,将eryb基因抹除,留下81bp的scar片段,得到trld基因敲除质粒pcxf7,如图5a所示。pcxf7敲除质粒的验证胶图如图5b所示。野生型的pcr产物大小为703bp,敲除质粒的pcr产物大小为530bp。2)将pcxf7质粒大肠杆菌-链霉菌属间三亲本接合转移转入s.coelicolorm1154中即可获得trld基因敲除的表达菌株s.coelicolorm1154/pcxf7。3)将敲除菌株s.coelicolorm1154/pcxf7接种至r3发酵培养基上,30℃发酵培养4-6天后提取发酵产物进行液相检测。液相检测图谱如图5c所示,14min的3,7-二羟基卓酚酮的洗脱峰消失,在16.5min及17.5min出现了两个新的洗脱峰2与3。实施例7、3,7-二羟基卓酚酮生物合成基因trle的敲除同上利用pcr-targeting技术对trle基因进行单基因敲除:trle基因的敲除引物序列如下:seqidno.28(δtrle-f,5′-3′):gactgcgcaggcagggcgtcgaggcggtcgtccacgagcaggcgcatattccggggatccgtcgacseqidno.29(δtrle-r,5′-3′):gtgccaggaagtgggcgaggaccaccgcgtcctccagggcctggttgtgtaggctggagctgcttctrle基因敲除验证引物序列如下:seqidno.30(ver-δtrle-f,5′-3′):ccttcaccgaagtcgcaccseqidno.31(ver-δtrle-r,5′-3′):cgacgcagacgctcgtagg1)利用δtrle-f/r引物从pjtu6722上扩增红霉素抗性基因eryb,利用pcr-targeting技术将eryb基因插入pcxf1中替换trle,得到trle的敲除质粒pcxf1(δtrle::eryb)。将pcxf1(δtrle::eryb)质粒转入bt340菌株中,在flp重组酶的作用下,将eryb基因抹除,留下81bp的scar片段,得到trle基因敲除质粒pcxf8,如图6a所示。pcxf7敲除质粒的验证胶图如图6b所示。野生型的pcr产物大小为703bp,敲除质粒的pcr产物大小为530bp。2)将pcxf8质粒大肠杆菌-链霉菌属间三亲本接合转移转入s.coelicolorm1154中即可获得trle基因敲除的表达菌株s.coelicolorm1154/pcxf8。3)将敲除菌株s.coelicolorm1154/pcxf8接种至r3发酵培养基上,30℃发酵培养4-6天后提取发酵产物进行液相检测。液相检测图谱如图6c所示,14min的3,7-二羟基卓酚酮的洗脱峰消失,在14.1min出现了一个新的洗脱峰4。实施例8、3,7-二羟基卓酚酮生物合成基因trlf的敲除同上利用pcr-targeting技术对trlf基因进行单基因敲除:trlf基因的敲除引物序列如下:seqidno.32(δtrlf-f,5′-3′):atggcaaggacctactccttcgaaggcaacgtacccgtcgtgcatatattccggggatccgtcgacseqidno.33(δtrlf-r,5′-3′):ctcggtggcggcctccgccgcgtgcagaccggtgagacagcgctgtgtgtaggctggagctgcttctrlf基因敲除验证引物序列如下:seqidno.34(ver-δtrlf-f,5′-3′):gatccacgggcacgacatcseqidno.35(ver-δtrlf-r,5′-3′):cgtcctgacggtcttgtcc1)利用δtrlf-f/r引物从pjtu6722上扩增红霉素抗性基因eryb,利用pcr-targeting技术将eryb基因插入pcxf1中替换trlf,得到trlf的敲除质粒pcxf1(δtrlf::eryb)。将pcxf1(δtrlf::eryb)质粒转入bt340菌株中,在flp重组酶的作用下,将eryb基因抹除,留下81bp的scar片段,得到trlf基因敲除质粒pcxf9,如图7a所示。pcxf8敲除质粒的验证胶图如图7b所示。野生型的pcr产物大小为782bp,敲除质粒的pcr产物大小为417bp。2)将pcxf9质粒大肠杆菌-链霉菌属间三亲本接合转移转入s.coelicolorm1154中即可获得trlf基因敲除的表达菌株s.coelicolorm1154/pcxf9。3)将敲除菌株s.coelicolorm1154/pcxf9接种至r3发酵培养基上,30℃发酵培养4-6天后提取发酵产物进行液相检测。液相检测图谱如图7c所示,14min的3,7-二羟基卓酚酮的洗脱峰消失,在22min出现了一个新的洗脱峰5。实施例9、中间体化合物2的结构鉴定将s.coelicolorm1154/pcxf7接种至3lr3固体培养基上,30℃发酵培养4-6天。收集固体发酵培养物,利用等体积乙酸乙酯(添加0.5%乙酸)萃取3遍。35℃旋干后,利用甲醇溶解样品,进一步利用干法拌样将样品上载至chp20p大孔吸附柱(φ3.5cm×30cm)上。利用甲醇与水进行梯度洗脱,从纯水起始至纯甲醇,10%的梯度递增,每个梯度洗脱500ml,洗脱液收集至250ml三角瓶中,每瓶100ml。每瓶取出20μl样品进行hplc检测,方法同实施例3种的步骤(六)所述,收集含有化合物2的洗脱组分。将各组分室温静置过夜,直至出现棕色晶体,挑出晶体用甲醇洗涤后进行x-ray分析,同时挑取晶体洗涤后用氘代氯仿溶解后进行nmr检测,确定化合物2为7-羟基卓酚酮。化合物2的核磁谱图见附图8。实施例10、中间体化合物3的结构鉴定将s.coelicolorm1154/pcxf6接种至3lr3固体培养基上,30℃发酵培养4-6天。收集固体发酵培养物,利用等体积乙酸乙酯(添加0.5%乙酸)萃取3遍。35℃旋干后,利用甲醇溶解样品,进一步利用干法拌样将样品上载至chp20p大孔吸附柱(φ3.5cm×30cm)上。利用甲醇与水进行梯度洗脱,从纯水起始至纯甲醇,10%的梯度递增,每个梯度洗脱500ml,洗脱液收集至250ml三角瓶中,每瓶100ml。每瓶取出20μl样品进行hplc检测,方法同前文所述,收集含有化合物3的洗脱组分。将各组分室温静置过夜,直至出现棕色晶体,挑出晶体用甲醇洗涤后进行x-ray分析,同时与卓酚酮标准品进行hplc和高分辨质谱比对,确定化合物3为卓酚酮。实施例11、中间体化合物4的结构鉴定将s.coelicolorm1154/pcxf8接种至3lr3固体培养基上,30℃发酵培养4-6天。收集固体发酵培养物,利用等体积乙酸乙酯(添加0.5%乙酸)萃取3遍。35℃旋干后,利用甲醇溶解样品,进一步利用干法拌样将样品上载至chp20p大孔吸附柱(φ3.5cm×30cm)上。利用甲醇与水进行梯度洗脱,从纯水起始至纯甲醇,10%的梯度递增,每个梯度洗脱500ml,洗脱液收集至250ml三角瓶中,每瓶100ml。每瓶取出20μl样品进行hplc检测,方法同前文所述,收集含有化合物4的洗脱组分。合并含有化合物4的组分,35℃浓缩旋干后用甲醇溶解,进一步利用干法拌样将样品上载至ymc反相c18柱(φ2cm×20cm)上。利用甲醇与水进行梯度洗脱,起始洗脱体系为20%甲醇,5%的梯度递增洗脱至50%甲醇,每个梯度洗脱200ml,洗脱液收集至15ml玻璃试管中,每管约10ml。每个试管中取出20μl样品进行hplc检测,方法同前文所述,将仅含有化合物4的组分合并浓缩旋干,获得5mg化合物4纯品。通过高分辨质谱检测,气相质谱和hplc检测,与2,4,6-环庚三烯酮进行比对,确定化合物4为2,4,6-环庚三烯酮。实施例12、中间体化合物5的结构鉴定将s.coelicolorm1154/pcxf9接种至5lr3固体培养基上,30℃发酵培养4-6天。收集固体发酵培养物,利用等体积乙酸乙酯(添加0.5%乙酸)萃取3遍。35℃旋干后,利用甲醇溶解样品,进一步利用干法拌样将样品上载至chp20p大孔吸附柱(φ3.5cm×30cm)上。利用甲醇与水进行梯度洗脱,从纯水起始至纯甲醇,10%的梯度递增,每个梯度洗脱500ml,洗脱液收集至250ml三角瓶中,每瓶100ml。每瓶取出20μl样品进行hplc检测,方法同前文所述,收集含有化合物4的洗脱组分。合并含有化合物5的组分,35℃浓缩旋干后用甲醇溶解,进一步利用干法拌样将样品上载至ymc反相c18柱(φ2cm×20cm)上。利用甲醇与水进行梯度洗脱,起始洗脱体系为20%甲醇,5%的梯度递增洗脱至50%甲醇,每个梯度洗脱200ml,洗脱液收集至15ml玻璃试管中,每管约10ml。每个试管中取出20μl样品进行hplc检测,方法同前文所述,将仅含有化合物5的组分合并浓缩旋干,获得5mg化合物5纯品。利用氘代氯仿溶解样品后进行nmr检测,确定化合物5为1,4,6-环庚三烯-1-羧酸。化合物5的结构与核磁图谱见附图9。实施例13、3,7-二羟基卓酚酮的生物合成途径结合生物信息学分析3,7-二羟基卓酚酮生物合成基因簇中各蛋白的功能,本发明实施例中的基因敲除与中间体的结构等结果以及之前关于细菌来源环庚三烯酮七元芳香环的合成报道,本发明对细菌中3,7-二羟基卓酚酮的生物合成途径进行了推测。细菌中的3,7-二羟基卓酚酮的生物合成途径可能包含以下几个步骤(图10):首先经过分支酸代谢途径,在七磷酸景天庚酮糖合酶trlb的作用下催化4-磷酸-d-赤藓糖(化合物18)和磷酸烯醇式丙酮酸(化合物19)合成3-脱氧-d-阿糖基庚酮酸-7-磷酸(化合物17),起始分支酸途径,以合成大量的分支酸(化合物16)。再由双功能酶trlh的脱水酶结构域将分支酸脱水形成预苯酸(化合物15),后又在另一预苯酸变位酶的结构域作用下变构形成苯丙酮酸(化合物14)。苯丙酮酸降解形成苯乙酸(化合物13),进入苯乙酸降解途径,首先由苯乙酸辅酶a转移酶将苯乙酸活化形成苯乙酰-coa(化合物12),再由环氧化酶paaabce在苯乙酰-coa上形成环氧,合成1,2-环氧苯乙酰-coa(化合物11),再经由异构酶paag开环生成氧杂环庚三烯-coa(化合物10),进一步在烯酰-coa水合酶trla的催化下发生开环生成3-酮-5,6-脱氢庚酰-coa-半缩醛(化合物9)。3-酮-5,6-脱氢庚酰-coa-半缩醛中间体极其不稳定,经一步分子内自发的knoevenagel缩合反应生成2-羟基-1,4,6-环庚三烯-1-甲酰-coa(化合物8);2-羟基-1,4,6-环庚三烯-1-甲酰-coa经一步还原生成2-羟基-4,6-环庚二烯-1-甲酰-coa(化合物7),进一步自发脱水反应生成1,4,6-环庚三烯-1-甲酰-coa(化合物6),而后在硫酯酶trlg的催化下水解释放coa生成1,4,6-环庚三烯-1-羧酸(化合物5);1,4,6-环庚三烯-1-羧酸在氧化酶trlf的催化下发生脱羧氧化生成2,4,6-环庚三烯酮(化合物4);经单加氧酶trle催化后在2,4,6-环庚三烯酮c2位发生羟化生成卓酚酮(化合物3);trlc与trld分别为单加氧酶的氧化酶亚基与还原酶亚基,经trlc与trld催化后在卓酚酮的c7与c3位先后发生羟化生成7-羟基卓酚酮(化合物2)以及终产物3,7-二羟基卓酚酮(化合物1)。以上对本发明的具体实施例进行了描述。需要理解的是,本发明并不局限于上述特定实施方式,本领域技术人员可以在权利要求的范围内做出各种变形或修改,这并不影响本发明的实质内容。sequencelisting<110>上海交通大学<120>3,7-二羟基卓酚酮生物合成基因簇及其应用<130>dag29671<160>35<170>patentinversion3.5<210>1<211>9112<212>dna<213>streptomycesochraceiscleroticussoc7<400>1tcagcggcgtacggccacttcctcgcgctgggcgccgccggcctccgcgcggccgaacag60cagccgcgtacgcatccggtacaacgcctcgccccgctggttgtccgcctcgacgacgaa120ccggaccgtgcccgtgtccgggcgccggggatggggtgcggcctcctccacggtggcccg180tccgcgcacgtcgtcgccggggcgcaccgggcgcaggaaccggacctcgtccatgccgta240ggagccctggcaggcggagccggcgagcagggcgcggacgaactgccccatgaccaccgc300gccggtgtgccagccgctggccaccaggccgccgaagggggagcgggccgccgcctcggg360atccaggtggaagggcatcgggtcccacacccgcgcgtagccgatgatgtcctcgctcgt420gagccgggtggtccccagctcgtaggtgctgccgacgcggaagtgctcgaagtgccgcac480gggggtgctccttcgtctcgggggtctcgcgggggccggggcccggcactcagccggggg540acgccgcgtgggccgcgtacaccggcgcgcccaccgggcggatcagggcgggctccgggc600gggggcggcccaggcggcgcctctcggcgctgaaccactcggcgacccggaaggccaggt660ccatcgactgcccgcggttcaggcgggggtcgcacgccgtctcgtaccgcagaggcagat720cgtcggagaggacctcgtcgctcccgcccacgcactcggtgacgtcctccccggtgagct780ccacgtgcaccccgccggggtgcgtgcccagcgcccggtggacctcgaagaagcccgtga840tctcgtccaggacgtcctcgaagcgccggctcttgtggccgctcggcgcggtgaaggtgt900tgccgtgcatcgggtcgcacgcccacaccacccgggcgccctccgcgcgcaccgcctcca960ccagggcgggcagccggtcgcgtaccaggccccgccccatccgcgcgatcaacgtgagcc1020ggcccggggtgcgcagcggatccagccggtccaccagggcccgtacgtcgtcgacggcgg1080cgtcgggacccagcttgacgccgagcgggttgctgaccccggcggcgaacgcgacgtgcg1140cgccgtccagccggcgggtgcgctcgccgatccagagcaggtgcccgctcagggcgtacc1200gccggcccgtgtcggggtccgttcgcaccagggcggactcgtagtcgaggagcagcgcct1260cgtggctggcgaacacctccgtggtgcgcagcgccgaggtgctcataccgcaggcccgga1320tgaacgacagcgccgcgtcgatctcgtccgccagccgctcgtagcggtgtcccgccgcgg1380acagcgccacgaagtcacggttccactcgtgcacccggcgcagatccgcgtgcccgccgg1440cggtgagggcgcgcagcaggttgagcgtcgctgcggacgcctggtagacacgctccagcc1500gggccgggtcaggggtgcgggcctcggcggtgaactccagcccgttgacggcatcgcccc1560ggtacgagggcagcgtcacgccgccccgggtctcggtgggctgtgagcggggcttggcgt1620actggccggcgatacggccgaccttgaccaccgggacgccggccgcgtacgtcagcacgg1680cggccatctggaggaggacctccagcttgttgcggaccgactccgcgcccaccccgtcca1740gggtctccgcgcagtcgccgccctggagcaggaagccccgccccagggctacttcgccga1800gccggtcgagcagggtgtcgcactcctggccgaacaccagcggcggtgcggcggacaggg1860ccgacaaagtggtccgcagcgccgtgggatcgggccagtcgggctgctgcgccacgggca1920gcgcccgccacgcttcgacagagggatcggcaagtgagttctcgtccatctccagggatt1980ccacgctgaaaacctcaacgccttctgttccgagggcagggcacggggttgcggtgcgcg2040cagcctgtcacgcgccggccgggccccggcagtgggacgttgtggctagaagggcggatc2100tactacccgggtactagcgccagcggccgactgtcggggccggccgcactcctggaccat2160ttcggcaggcgccctcgtcgtcggcacccgacggcgggcgcggttcgccgcatgtctcca2220cgccaccggctcgaaaggtgatcccgcatgagcagcaccgtcaacccgtccgggtccgtg2280tcccgcccgcagaccggggacgagtacctggagagcctccgggacggccgggaagtcagg2340atctacggcgagaaggtgcaggacgtcacggctcacccggccttccgcaacaactcccgc2400atggtggcccggctcttcgacgcgctccacgaccccgagcgcaacagcaagctggtcgtc2460cccaccgacaccggcaacggcggtttcacccacccgtacttcaaggccccgtacacctcc2520ggcgacctgcggaccgccgcgcgggccatggaggagtgggcgcgcatgacgtacggctgg2580ctgggccgcacccccgacttcaaggccgccttcctgtccaccctgggctcgaacaaggag2640cactacgcgccgttcgaggacaacgcgtccgcgtggtaccgcaaggcacaggagaacgtg2700ctcttcatcggccacggcatcgtcaacccgccgatcgaccgcaaccgggggctgaccgag2760ctcaaggacgtgatgctgacggtggaccgggagaccgacgccggcctcgtggtctccggc2820gccaaggtcgtcggcaccggggccgcgctcacccagcacatcttcatcggcagctacggg2880gtcataccgaagggcgcgaaggagttctccgcgtacttcatcgtgccgaccgacagcccc2940ggcctgaagatcatctcccggccgtcgtacgagttcgcctcggccaccgtcagcagcccc3000ttcgaccagccgctcagcagccgcctggacgagaacgacggcattctcgtcttcgaccag3060gtgctgatcccctgggagaacgtcttctgctacgacgtggacaaggcgaacgacttcttc3120tacgggtcaggcttcttctaccgggccatgttccacgcctgcgtccgcttcgccgtgaag3180atggacttcctgaccggcctgctcgccaagtgcctggagaccacgggcacctcggagttc3240cgcggggtgcagagccggctcggcgaggtcctcgcctaccgcaacatgttctgggccctg3300gtcgactccatggccgagaaccccaccaagtggcccgacggcaccgtcacccccaacggc3360gagaccgcgctggccttccgcgtcctgtcctcgtcgatcttccccaaggtgcgtgagatc3420ttcctgcgggatctcggcagcgcgctgatctacaacaactcccacgccctcgactggtcc3480aaccccgaggtccggccctacctggacaagtacgtccgcggcctgggcgtggaggccatc3540gaccgggtgaagctgatgaagctggcatgggacgcggtcggcaccgagttcggggcccgc3600tgggagctgtacgagatgaactacgcgggcaaccacgagcagatccggttcgaggcgctg3660cacgcccagcaggtctccgggaagttcgacgactacaagcagctcgtcgaccggtgcctg3720tcggagtacgacgagcacggctggaaggtccccgacctcgtcgaccccaaggacaccacc3780gtcatcggcgccggcgcaccggacaccggcacgaccgccccccgcccggccgcacggtga3840gcagccacacccccgccccgggcggacccaccgccccgcccggagcccccgtcctgccgg3900accaggcccaactcaggcacgtactcggccagttcgtgaccggggtgtgcgcggtgtcga3960cccgcttcgacacccgctccggcgtacgccacgacgccatcgcggtgaactcgttcacct4020cggtctccctcgacccgccgctcgtgtcgatgtacctgcgcgacgactccaccttcctgc4080ggcgggtccgcacgtccggccagtgggccgtgtcgttcctcggcaaccgggcgcgctccc4140tggtcggcgtcctcaccgcgcccgcggcccggcgccctccggtggagcaggccgccgact4200gggccgtcggcccgcacaccggctgtctggtcctgccggacggccccggaacgctcgaat4260gcgctctgcacctcacgcaggcactcggcgaccatgtcctcgtggtcggacgggtgctgg4320gcgcggtgggccgcgaccacgaacccctggtcttccaccgcggcgccttcaccgaagtcg4380caccggcgccgtgaccacgctccaccggccttcggcagcgaacaggaaaggagaccccat4440ggcgaggaaacgcccgtacgtggcggtggtgggcggcggcatcggcggcctggccgtcgc4500gctgggactgcgcaggcagggcgtcgaggcggtcgtccacgagcaggcgcacgcactgtc4560ccaccagggggcggggatcgcgatcggcgcgaacggacaccgggccctgcgggaactggg4620cgtcgccaagcggctgaccgcctctgccgcccggccgtcgcgcgccgacttccggcactg4680gcgcaccggccggtccatggtgtcgcaccgcctgaccggcctgtacgaggaacgcttcgg4740cgcccccttctggaccgtggaacgcgcggccgtccagcaggctctgctcgccgaactggg4800cccgcgccacgtgcgcctgggcgcgcgctgcaccggggtggacaggacggccgacggcgc4860cgtgatccgcttcgaggacggcggcgaggcggaggccgacgcggtcgtcggggcggacgg4920tatccactccgcggtgcgccacagcctcttcggcccgcaggaggccgtgttctccggcac4980cagcggataccgcgccctggtcccgatggaccggctgcggcacgttcccgaactcgccga5040accggtgctgtggctgtggctcggaccgggccggcacttcatcgcctacccggtggccga5100cggcagcgcgctgaacttcctggccgtcgtgcccgaccgcacgtggacggtggagtcctg5160gtccacggagggcgacgctgccgagctgcgggccgccttcgacggctggcacccgttcgt5220cacggaggtcctcggcgcctgcgaacgtccgggccgctgggcgctgtacgaccgggagcc5280gcagcgcgtgtggagttccggcgcggtgacgctgctgggagatgccgcgcacgcgatgct5340gccgcaccacggacagggcgccaaccaggccctggaggacgcggtggtcctcgcccactt5400cctggcacggacggacaccggcggtgtgccgtcggccctgcgcgcctacgagcgtctgcg5460tcggccgcgcacccggctgctccaggcgggctcccgtaagaacgccggctgcttccagct5520ccccgacgggccgcaggccgaagcccgcaacgcaaggctggccaccctgcccgacgacgt5580cgcctggatccacgggcacgacatcctcggctccctgcctgtggccacgtcaccggcctg5640aaccgcgggcgggggccgccgaccgcgacgaccatcgacgaacggtgaggaacaccatgg5700caaggacctactccttcgaaggcaacgtacccgtcgtgcatcccacggcgttcgtgcacc5760ccgacgccgtgctgatcgggtcggtcgacatcggtccggggtgctatgtcggccccctcg5820cgagcctgcggggcgacttcgggcacatcgaactccgggcgggctccaacgtccaggacg5880gctgcgtcctgcactgcttcccgggggccgacacggtcgtggaggaggacggacacgtcg5940gccacggcagcgtgctgcacggctgccgggtgggccgggacagcctcatcggcatgaagt6000ccgtcctcatggacggtgtcgtggtgggaacgcgggcgttcgtcggggccggcagcttcg6060tcaagtcccgcttccaggtaccggagcggcatctggtggcgggcagcccggccaaggtgg6120tgcgtgaactcaccgccgacgagatcgcctggaagggcaacgggaccgcccagtaccaga6180agctggcccagcgctgtctcaccggtctgcacgcggcggaggccgccaccgagcgcaccg6240ccccggcggcccccgccgaacccggcgagcacgaacacgtcaccctccacgcctaccggt6300cccgctgacgaagcacccgaggcaccttgtcgcggccgatccgcgcaccggacaagaccg6360tcaggacgggccccgcgacggcgccgggctcgcgcgaacgccaggcccattcaccggacg6420tcacgttcagggacgtcacgttcacggacgtcatgttcaaggacttcacgctcaaggacc6480ttccgcggctccccagggccgcgacccttccccaccccctccaggaggcatgttcatgaa6540cagctcagccacctcccgactcgtcgtcggcgaccggttccgcgacttcgccgaggccgt6600cggcgccgagacgttctcgcggctcgcccgctcgctcgacgcgggcgacctggacgcggt6660cgagggaccgctgcgcgtggtcaccggccagggcgtcggcgagttcgagatctcctacct6720cgaggacgccgtccgccggcggcagctggacgaccggatcgagctggtccactcggccgg6780gcagcccgcccgtcgccaggaactgcacaaggcccgcgaggacaacgtgctgatcgccgg6840gctcacacaggtcgacgacaccgcctacgaggccgccctgaggctgcacgaccacaacga6900actgctcgtcgacgtccaggaccgggtgcacgtgcagggcatggtggccgtcgaggcggc6960ccgtcagatgttcgtcgcggtggtggagcgctacttcacggcccgctggccgcagcagcg7020ctactacatcgtcctcaactccatcaacaccacgttctccaacttcctcttccccgtggg7080tgccacggtgcggctgacggtggacgagtcggaggtgtcggagcccagccggctcacgtt7140ccgtacgacggtggaggtggagcaggccggacggcccgccgctgccgtcaccatcgacgc7200ggccgccttcgcccccgggctgctggaggagaaggagctgcgccgggccaagagcgcggt7260ggcgagcacgatggacgccgcactggccatcgcgtgacatgaggacgccgacaccggaca7320cccggacgcgccccgcaccgggacgactcccgtacccggagtggcggttcgcctacctgg7380gcccggaaggaaccttcaccgagctggccctgcggtccctcccggaggccacgggcgccc7440ggctgtcaccggctgccacggccaccgaggccctgacgctggtgtgccggggcgccgcgg7500acgccgccatgctgcccgtccacaacacggtggccggggtggtcgcggacacggtgcgcg7560cgctcgccgagtcacccgcgctgaccgtcctcagggaagtcgtgctccccgtggaattcg7620cccttctggtgcgccccggcacacgacgcgcgcggatccgcacggtgagcggccatccgc7680acgcgggagcgcaggtcggcggatggctcgacagccgggcgccgcaggcgcgttggacac7740cggcgccgtccaacgccgaggccgcgcgcagggtacgggaccgggagtgcgacgccgcgg7800tcgccggggagttcaccgccgcgcactacggccttcaggtcctggacgccgggatacagg7860acaccgccggtgcggtcacccggttcctgctgtgcgcgcgtcccggcaggtggagcggcc7920cgcgtcccgccgccgaccgcacctcgctggtcgggcgtctcgcggcccggcccgaggagg7980ccggcgggctgctcgacggcctgcggagccaccccttcctgcggtcggtgtccctgcgga8040ccgtgtccgacggccgcagcggctcggtcctgttcgccgactgcccgggaggcgccggac8100aggccgcgacagcgcgcgcggtcggcctgctgcgcctgcggctgcccggactgagagtcc8160tggggccgtacccctcggcgacccatgtcccgtcgtacgaacggagcgagaccatgcaca8220ccgagaccgccgccgaagccggcctctcagcggagaccgggtcccgggacatcgcccggc8280tgcgcgaccggatcgactccctggaccacctgctcatcggcacgctcagggagcggctgg8340aggcgtcccgcgagatccagcggatccgtacccgaagcggcggctcccaggtcgaccccg8400ggcgcgaggccgccgtccggggccggtacgccgaggaactgggggagggcggaacgggcg8460tcgccgaggcgctgctggacatgtgccgggggcggtcaccacggtgaacccgcccccgac8520gacgacggcgacgtcgtcaccccgcccccgtcgcggccgtgaggacgtcctcacggccgc8580gacggccttgttccacgagcggggttacgacgcgacgagcatgagcgacatcgccgcacg8640cctcggcttcaccaaggcggcgctgtaccggcacgtgacgggcaaggccgaactgctgcg8700ggcgatcacgcggccggtccgcgcggacgtgcgggccctgctggccgcgagcaccgccgg8760agacgaccggcccgtcgaccaactctcggagctgttacgagggttggccggcgccgcggc8820ggccgacccgggccggtacgtcctcttctgggggccggacggcgagcggagcccgcacgg8880gcccgacgccgtgtgccgtgcggcggtggtccagcggctgacggaactgctggagcgcgc8940cgccgaccgcggcgagatacgcgacggcatcgttccccacctggccgcccggctgctcgt9000cggcgcggtcaccggtccgggccggccggtgtcaccgtcggccgtggacgtcctgctcga9060cgggccggtgcggcgtctgaacgacggggcaggccgggggcgggggcgctga9112<210>2<211>159<212>prt<213>streptomycesochraceiscleroticussoc7<400>2valarghisphegluhispheargvalglyserthrtyrgluleugly151015thrthrargleuthrsergluaspileileglytyralaargvaltrp202530aspprometprophehisleuaspproglualaalaalaargserpro354045pheglyglyleuvalalaserglytrphisthrglyalavalvalmet505560glyglnphevalargalaleuleualaglyseralacysglnglyser65707580tyrglymetaspgluvalargpheleuargprovalargproglyasp859095aspvalargglyargalathrvalgluglualaalaprohisproarg100105110argproaspthrglythrvalargphevalvalglualaaspasngln115120125argglyglualaleutyrargmetargthrargleuleupheglyarg130135140alaglualaglyglyalaglnargglugluvalalavalargarg145150155<210>3<211>484<212>prt<213>streptomycesochraceiscleroticussoc7<400>3valgluserleuglumetaspgluasnserleualaaspproserval151015glualatrpargalaleuprovalalaglnglnproasptrpproasp202530prothralaleuargthrthrleuseralaleuseralaalapropro354045leuvalpheglyglnglucysaspthrleuleuaspargleuglyglu505560valalaleuglyargglypheleuleuglnglyglyaspcysalaglu65707580thrleuaspglyvalglyalagluservalargasnlysleugluval859095leuleuglnmetalaalavalleuthrtyralaalaglyvalproval100105110vallysvalglyargilealaglyglntyralalysproargsergln115120125prothrgluthrargglyglyvalthrleuprosertyrargglyasp130135140alavalasnglyleugluphethralaglualaargthrproasppro145150155160alaargleugluargvaltyrglnalaseralaalathrleuasnleu165170175leuargalaleuthralaglyglyhisalaaspleuargargvalhis180185190glutrpasnargaspphevalalaleuseralaalaglyhisargtyr195200205gluargleualaaspgluileaspalaalaleuserpheileargala210215220cysglymetserthrseralaleuargthrthrgluvalphealaser225230235240hisglualaleuleuleuasptyrgluseralaleuvalargthrasp245250255proaspthrglyargargtyralaleuserglyhisleuleutrpile260265270glygluargthrargargleuaspglyalahisvalalaphealaala275280285glyvalserasnproleuglyvallysleuglyproaspalaalaval290295300aspaspvalargalaleuvalaspargleuaspproleuargthrpro305310315320glyargleuthrleuilealaargmetglyargglyleuvalargasp325330335argleuproalaleuvalglualavalargalagluglyalaargval340345350valtrpalacysasppromethisglyasnthrphethralaproser355360365glyhislysserargargphegluaspvalleuaspgluilethrgly370375380phephegluvalhisargalaleuglythrhisproglyglyvalhis385390395400valgluleuthrglygluaspvalthrglucysvalglyglyserasp405410415gluvalleuseraspaspleuproleuargtyrgluthralacysasp420425430proargleuasnargglyglnsermetaspleualapheargvalala435440445glutrppheseralagluargargargleuglyargproargproglu450455460proalaleuileargprovalglyalaprovaltyralaalahisala465470475480alaserprogly<210>4<211>530<212>prt<213>streptomycesochraceiscleroticussoc7<400>4metserserthrvalasnproserglyservalserargproglnthr151015glyaspglutyrleugluserleuargaspglyarggluvalargile202530tyrglyglulysvalglnaspvalthralahisproalapheargasn354045asnserargmetvalalaargleupheaspalaleuhisaspproglu505560argasnserlysleuvalvalprothraspthrglyasnglyglyphe65707580thrhisprotyrphelysalaprotyrthrserglyaspleuargthr859095alaalaargalametgluglutrpalaargmetthrtyrglytrpleu100105110glyargthrproaspphelysalaalapheleuserthrleuglyser115120125asnlysgluhistyralaprophegluaspasnalaseralatrptyr130135140arglysalaglngluasnvalleupheileglyhisglyilevalasn145150155160proproileaspargasnargglyleuthrgluleulysaspvalmet165170175leuthrvalasparggluthraspalaglyleuvalvalserglyala180185190lysvalvalglythrglyalaalaleuthrglnhisilepheilegly195200205sertyrglyvalileprolysglyalalysglupheseralatyrphe210215220ilevalprothraspserproglyleulysileileserargproser225230235240tyrgluphealaseralathrvalserserpropheaspglnproleu245250255serserargleuaspgluasnaspglyileleuvalpheaspglnval260265270leuileprotrpgluasnvalphecystyraspvalasplysalaasn275280285aspphephetyrglyserglyphephetyrargalametphehisala290295300cysvalargphealavallysmetasppheleuthrglyleuleuala305310315320lyscysleugluthrthrglythrserglupheargglyvalglnser325330335argleuglygluvalleualatyrargasnmetphetrpalaleuval340345350aspsermetalagluasnprothrlystrpproaspglythrvalthr355360365proasnglygluthralaleualapheargvalleuserserserile370375380pheprolysvalarggluilepheleuargaspleuglyseralaleu385390395400iletyrasnasnserhisalaleuasptrpserasnprogluvalarg405410415protyrleuasplystyrvalargglyleuglyvalglualaileasp420425430argvallysleumetlysleualatrpaspalavalglythrgluphe435440445glyalaargtrpgluleutyrglumetasntyralaglyasnhisglu450455460glnileargpheglualaleuhisalaglnglnvalserglylysphe465470475480aspasptyrlysglnleuvalaspargcysleuserglutyraspglu485490495hisglytrplysvalproaspleuvalaspprolysaspthrthrval500505510ileglyalaglyalaproaspthrglythrthralaproargproala515520525alaarg530<210>5<211>185<212>prt<213>streptomycesochraceiscleroticussoc7<400>5valserserhisthrproalaproglyglyprothralaproprogly151015alaprovalleuproaspglnalaglnleuarghisvalleuglygln202530phevalthrglyvalcysalavalserthrargpheaspthrargser354045glyvalarghisaspalailealavalasnserphethrservalser505560leuaspproproleuvalsermettyrleuargaspaspserthrphe65707580leuargargvalargthrserglyglntrpalavalserpheleugly859095asnargalaargserleuvalglyvalleuthralaproalaalaarg100105110argproprovalgluglnalaalaasptrpalavalglyprohisthr115120125glycysleuvalleuproaspglyproglythrleuglucysalaleu130135140hisleuthrglnalaleuglyasphisvalleuvalvalglyargval145150155160leuglyalavalglyargasphisgluproleuvalphehisarggly165170175alaphethrgluvalalaproalapro180185<210>6<211>400<212>prt<213>streptomycesochraceiscleroticussoc7<400>6metalaarglysargprotyrvalalavalvalglyglyglyilegly151015glyleualavalalaleuglyleuargargglnglyvalglualaval202530valhisgluglnalahisalaleuserhisglnglyalaglyileala354045ileglyalaasnglyhisargalaleuarggluleuglyvalalalys505560argleuthralaseralaalaargproserargalaaspphearghis65707580trpargthrglyargsermetvalserhisargleuthrglyleutyr859095glugluargpheglyalaprophetrpthrvalgluargalaalaval100105110glnglnalaleuleualagluleuglyproarghisvalargleugly115120125alaargcysthrglyvalaspargthralaaspglyalavalilearg130135140phegluaspglyglyglualaglualaaspalavalvalglyalaasp145150155160glyilehisseralavalarghisserleupheglyproglngluala165170175valpheserglythrserglytyrargalaleuvalprometasparg180185190leuarghisvalprogluleualagluprovalleutrpleutrpleu195200205glyproglyarghispheilealatyrprovalalaaspglyserala210215220leuasnpheleualavalvalproaspargthrtrpthrvalgluser225230235240trpserthrgluglyaspalaalagluleuargalaalapheaspgly245250255trphisprophevalthrgluvalleuglyalacysgluargprogly260265270argtrpalaleutyrasparggluproglnargvaltrpsersergly275280285alavalthrleuleuglyaspalaalahisalametleuprohishis290295300glyglnglyalaasnglnalaleugluaspalavalvalleualahis305310315320pheleualaargthraspthrglyglyvalproseralaleuargala325330335tyrgluargleuargargproargthrargleuleuglnalaglyser340345350arglysasnalaglycyspheglnleuproaspglyproglnalaglu355360365alaargasnalaargleualathrleuproaspaspvalalatrpile370375380hisglyhisaspileleuglyserleuprovalalathrserproala385390395400<210>7<211>203<212>prt<213>streptomycesochraceiscleroticussoc7<400>7metalaargthrtyrserphegluglyasnvalprovalvalhispro151015thralaphevalhisproaspalavalleuileglyservalaspile202530glyproglycystyrvalglyproleualaserleuargglyaspphe354045glyhisilegluleuargalaglyserasnvalglnaspglycysval505560leuhiscyspheproglyalaaspthrvalvalglugluaspglyhis65707580valglyhisglyservalleuhisglycysargvalglyargaspser859095leuileglymetlysservalleumetaspglyvalvalvalglythr100105110argalaphevalglyalaglyserphevallysserargpheglnval115120125progluarghisleuvalalaglyserproalalysvalvalargglu130135140leuthralaaspgluilealatrplysglyasnglythralaglntyr145150155160glnlysleualaglnargcysleuthrglyleuhisalaalagluala165170175alathrgluargthralaproalaalaproalagluproglygluhis180185190gluhisvalthrleuhisalatyrargserarg195200<210>8<211>70<212>prt<213>streptomycesochraceiscleroticussoc7<400>8leuserargproileargalaproasplysthrvalargthrglypro151015alathralaproglyserarggluargglnalahisserproaspval202530thrpheargaspvalthrphethraspvalmetphelysaspphethr354045leulysaspleuproargleuproargalaalathrleuprohispro505560leuglnglualacysser6570<210>9<211>253<212>prt<213>streptomycesochraceiscleroticussoc7<400>9metasnserseralathrserargleuvalvalglyaspargphearg151015aspphealaglualavalglyalagluthrpheserargleualaarg202530serleuaspalaglyaspleuaspalavalgluglyproleuargval354045valthrglyglnglyvalglygluphegluilesertyrleugluasp505560alavalargargargglnleuaspaspargilegluleuvalhisser65707580alaglyglnproalaargargglngluleuhislysalaarggluasp859095asnvalleuilealaglyleuthrglnvalaspaspthralatyrglu100105110alaalaleuargleuhisasphisasngluleuleuvalaspvalgln115120125aspargvalhisvalglnglymetvalalavalglualaalaarggln130135140metphevalalavalvalgluargtyrphethralaargtrpprogln145150155160glnargtyrtyrilevalleuasnserileasnthrthrpheserasn165170175pheleupheprovalglyalathrvalargleuthrvalaspgluser180185190gluvalsergluproserargleuthrpheargthrthrvalgluval195200205gluglnalaglyargproalaalaalavalthrileaspalaalaala210215220phealaproglyleuleugluglulysgluleuargargalalysser225230235240alavalalaserthrmetaspalaalaleualaileala245250<210>10<211>402<212>prt<213>streptomycesochraceiscleroticussoc7<400>10metargthrprothrproaspthrargthrargproalaproglyarg151015leuprotyrproglutrpargphealatyrleuglyprogluglythr202530phethrgluleualaleuargserleuproglualathrglyalaarg354045leuserproalaalathralathrglualaleuthrleuvalcysarg505560glyalaalaaspalaalametleuprovalhisasnthrvalalagly65707580valvalalaaspthrvalargalaleualagluserproalaleuthr859095valleuarggluvalvalleuprovalgluphealaleuleuvalarg100105110proglythrargargalaargileargthrvalserglyhisprohis115120125alaglyalaglnvalglyglytrpleuaspserargalaproglnala130135140argtrpthrproalaproserasnalaglualaalaargargvalarg145150155160aspargglucysaspalaalavalalaglygluphethralaalahis165170175tyrglyleuglnvalleuaspalaglyileglnaspthralaglyala180185190valthrargpheleuleucysalaargproglyargtrpserglypro195200205argproalaalaaspargthrserleuvalglyargleualaalaarg210215220progluglualaglyglyleuleuaspglyleuargserhisprophe225230235240leuargservalserleuargthrvalseraspglyargserglyser245250255valleuphealaaspcysproglyglyalaglyglnalaalathrala260265270argalavalglyleuleuargleuargleuproglyleuargvalleu275280285glyprotyrproseralathrhisvalprosertyrgluargserglu290295300thrmethisthrgluthralaalaglualaglyleuseralagluthr305310315320glyserargaspilealaargleuargaspargileaspserleuasp325330335hisleuleuileglythrleuarggluargleuglualaserargglu340345350ileglnargileargthrargserglyglyserglnvalaspprogly355360365argglualaalavalargglyargtyralaglugluleuglyglugly370375380glythrglyvalalaglualaleuleuaspmetcysargglyargser385390395400proarg<210>11<211>209<212>prt<213>streptomycesochraceiscleroticussoc7<400>11valproglyalavalthrthrvalasnproproprothrthrthrala151015thrserserproargproargargglyarggluaspvalleuthrala202530alathralaleuphehisgluargglytyraspalathrsermetser354045aspilealaalaargleuglyphethrlysalaalaleutyrarghis505560valthrglylysalagluleuleuargalailethrargprovalarg65707580alaaspvalargalaleuleualaalaserthralaglyaspasparg859095provalaspglnleusergluleuleuargglyleualaglyalaala100105110alaalaaspproglyargtyrvalleuphetrpglyproaspglyglu115120125argserprohisglyproaspalavalcysargalaalavalvalgln130135140argleuthrgluleuleugluargalaalaaspargglygluilearg145150155160aspglyilevalprohisleualaalaargleuleuvalglyalaval165170175thrglyproglyargprovalserproseralavalaspvalleuleu180185190aspglyprovalargargleuasnaspglyalaglyargglyarggly195200205arg<210>12<211>60<212>dna<213>artificialsequence<220><223>正向<400>12gccaccgcaccgccgtacgggccgggccgcggccgtccgatattccggggatccgtcgac60<210>13<211>59<212>dna<213>artificialsequence<220><223>反向<400>13gcggccgacgatctgctggtggacgtcggcttccacgccgtgtaggctggagctgcttc59<210>14<211>20<212>dna<213>artificialsequence<220><223>正向<400>14atgtcaccgctgcgcaccga20<210>15<211>20<212>dna<213>artificialsequence<220><223>反向<400>15acctccggtacggaccagga20<210>16<211>60<212>dna<213>artificialsequence<220><223>正向<400>16gcgacgggtgcgacgggtgcggaacatgggagcggccctatattccggggatccgtcgac60<210>17<211>59<212>dna<213>artificialsequence<220><223>反向<400>17gtgtggctccgccacgccgtccgctccgcgctgacccgggtgtaggctggagctgcttc59<210>18<211>21<212>dna<213>artificialsequence<220><223>正向<400>18tcacgaacagcttcacccact21<210>19<211>21<212>dna<213>artificialsequence<220><223>反向<400>19ggcatcgaactgttcgacctt21<210>20<211>66<212>dna<213>artificialsequence<220><223>正向<400>20acgagtacctggagagcctccgggacggccgggaagtcaggatctatattccggggatcc60gtcgac66<210>21<211>66<212>dna<213>artificialsequence<220><223>反向<400>21cgtacttgtccaggtagggccggacctcggggttggaccagtcgatgtgtaggctggagc60tgcttc66<210>22<211>20<212>dna<213>artificialsequence<220><223>正向<400>22caccggctcgaaaggtgatc20<210>23<211>21<212>dna<213>artificialsequence<220><223>反向<400>23gtagttcatctcgtacagctc21<210>24<211>66<212>dna<213>artificialsequence<220><223>正向<400>24gagcccccgtcctgccggaccaggcccaactcaggcacgtactcgatattccggggatcc60gtcgac66<210>25<211>66<212>dna<213>artificialsequence<220><223>反向<400>25gacagccggtgtgcgggccgacggcccagtcggcggcctgctccatgtgtaggctggagc60tgcttc66<210>26<211>20<212>dna<213>artificialsequence<220><223>正向<400>26gagtacgacgagcacggctg20<210>27<211>19<212>dna<213>artificialsequence<220><223>反向<400>27cctgttcgctgccgaaggc19<210>28<211>66<212>dna<213>artificialsequence<220><223>正向<400>28gactgcgcaggcagggcgtcgaggcggtcgtccacgagcaggcgcatattccggggatcc60gtcgac66<210>29<211>66<212>dna<213>artificialsequence<220><223>反向<400>29gtgccaggaagtgggcgaggaccaccgcgtcctccagggcctggttgtgtaggctggagc60tgcttc66<210>30<211>19<212>dna<213>artificialsequence<220><223>正向<400>30ccttcaccgaagtcgcacc19<210>31<211>19<212>dna<213>artificialsequence<220><223>反向<400>31cgacgcagacgctcgtagg19<210>32<211>66<212>dna<213>artificialsequence<220><223>正向<400>32atggcaaggacctactccttcgaaggcaacgtacccgtcgtgcatatattccggggatcc60gtcgac66<210>33<211>66<212>dna<213>artificialsequence<220><223>反向<400>33ctcggtggcggcctccgccgcgtgcagaccggtgagacagcgctgtgtgtaggctggagc60tgcttc66<210>34<211>19<212>dna<213>artificialsequence<220><223>正向<400>34gatccacgggcacgacatc19<210>35<211>19<212>dna<213>artificialsequence<220><223>反向<400>35cgtcctgacggtcttgtcc19当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1