异位表达的不同基因组合及其在提高植物营养组织油含量的应用的制作方法
本发明属于基因工程技术领域,具体涉及异位表达的不同基因组合及其在提高植物营养组织油含量的应用。
背景技术:
植物油用途广泛,不仅是食用油的主要来源,而且可用于动物饲料、肥皂、表面活性剂、化妆品、涂料、润滑油,以及生物柴油的生产。据报道,食用油的消耗量在2030年之前将翻倍;化学工业界还寄望在20年后,植物油能取代40%的原油,因而对植物油的需求正急剧增加。然而,由于人口的增长和可耕地面积的减少,中国将面临食用油和化工原油短缺的严峻局面。人们寄希望通过提高和改变油料植物的农艺和品质性状(其中包括种子产量、油脂的含量及其脂肪酸组分),使植物不仅是食用油的主要来源,而且还成为可持续地生产生物能源和化工原料的潜在工厂。
通常情况下,油料植物生产植物油的主要场所在种子和果实中,营养器官中有的含量则很低,不足种子中油含量的百分之一,然而在某些逆境条件下或植物衰老过程中,叶片或茎秆中的油含量呈显著增加的趋势,这表明,营养器官中也存在“产油机器”,且在某些特定条件下,其运转效率可以得到增加。普遍认为,植物种子内油脂的合成和积累取决于以下四个关键因素:(1)将光合产物转化为脂肪酸的调控因子;(2)将脂肪酸链连接在甘油骨架上的脂肪酸酰基转移酶;(3)参与油脂降解的脂酶活性;(4)参与油滴形成和稳定的油脂蛋白。上述关键酶基因和调控因子在不同的亚细胞上被精确地调控,具有高度的时空协同性,而在营养组织中,尽管存有部分上述基因和调控因子,但表达量极低,且相互之间缺乏协同性(chapmanetal.2013)。因此,若通过基因工程手段,在不同的亚细胞器内协同、有序地表达相关基因,同时降低脂肪酶的活性,能否将油料种子中的高效“产油机器”整合到植物营养器官中从而提高其产油能力成为本发明的研究课题。
在经典的kennedy途径中,acyl-coa:二酰甘油酰基转移酶(dgat)被认为是参与种子油脂合成的关键限速酶,且发现衰老叶片中,此酶参与了tag的合成,因此,为了剖析营养组织中脂肪酸组装到甘油骨架上的机制,一些研究致力于调查dgat基因在组成型表达的启动子(如35s)调控下对植物营养器官中油合成的影响,结果显示,atdgat1基因在烟草幼苗中的过表达可使其tag含量增至野生型的5.9倍,而转基因叶片中,tag含量高达野生型的7倍。
亦有研究表明,在营养组织中过表达脂肪酸合成途径中的关键酶基因或转录因子,能加速光合产物向脂肪酸的转化以及油脂的合成。丙二酰辅酶a是脂肪酸合成的重要前体,klaus等发现,丙二酰辅酶a合成途径中的限速酶—乙酰辅酶a羧化酶(accase)的过表达可使马铃薯块茎积累中性的三酰甘油(tag)。随后,mendoza等在拟南芥中过表达参与种子成熟和油脂积累调控过程的转录因子leafycotyledon2(lec2),发现在营养组织中积累了种子特异的mrna,并且储存性tag含量有所增加,但与此同时,幼苗出现体细胞胚胎发生现象,且组织扭曲变形,影响转基因植株的正常生长。这说明,关键基因与合适启动子的有机结合对操控植物营养组织中油脂的合成和积累至关重要。但是,基于载体承受性和表达量的限制,选择何种基因组合并怎样合理地构建转录单元从而更好地实现基因均衡表达仍然是需要首先考虑和解决的技术问题。
技术实现要素:
针对上述问题,本发明的目的在于公开异位表达的不同基因组合及其在提高植物营养组织油含量的应用,构建表达载体,使其在植物营养器官中异位或异源表达参与种子油生产的关键基因,增强油脂合成与积累能力,有效地提高营养器官的含油量。
在植物营养组织中异位表达的不同基因组合,含有所述的基因组合的表达载体引入以下模块:
a、调控油脂合成的四种目的基因lec2、dgat1、oleo2或gat1中的至少一种;
b、控制目的基因均衡表达的转录单元;
c、多克隆位点。
进一步的,所述表达载体为含有lec2、dgat1、oleo2和gat1基因组合的载体。
进一步的,所述表达载体中启动lec2基因表达的启动子为35s,终止该基因表达的终止子为nos;启动dgat1基因表达的启动子为35s,终止该基因表达的终止子为at4g25710基因的3’utr;启动oleo2基因表达的启动子为at4g25700基因的启动子,终止该基因表达的终止子为nos;启动gat1基因表达的启动子为35s,终止该基因表达的终止子为nos。
更进一步的,所述表达载体是以pbi121双元载体为骨架构建的。
进一步的,目的基因还包括与lec2、dgat1、oleo2或gat1的核苷酸序列同源性达到90%以上的基因;或包括编码的氨基酸序列与拟南芥中lec2、dgat1或oleo2编码的氨基酸序列,或与酿酒酵母中gat1编码的氨基酸序列同源性达到95%以上的基因。
进一步的,构建上述表达载体的方法,包括以下步骤:
a、选择pbi121双元载体为构建骨架;在35s的5’端引入包括bamhi,xmai,smai,xhoi和saci的多克隆位点后将其插入pbi121中获得pbi121-35s;
b、扩增35s-nosdna片段;然后在35s-nosdna片段的5’端加入kpni-clai-stui多酶切位点,3’端加入hindiii-clai-ecori多酶切位点,再通过kpni和ecori将35s-nosdna片段克隆到载体pyes2中,获得中间载体pyes2-35s-nos;
c、以拟南芥基因组dna为模板,扩增含有at4g25710基因的3’utr和at4g25700基因的启动子(bch1)及5’utr的dna片段,再在该dna片段的5’端加入bamhi酶切位点,3’端加入saci酶切位点,然后通过bamhi和saci将该dna片段插入到载体pyes2-35s-nos中,获得中间载体pyes2-35s-3’utr-bch1-5’utr-nos;
d、以拟南芥的cdna为模板,扩增oleo2目的基因,然后在oleo2目的基因的5’末端和3’末端均加上saci单酶切位点,再通过单酶切位点saci将oleo2目的基因克隆到载体pyes2-35s-3’utr-bch1-5’utr-nos,获得中间载体pyes2-35s-3’utr-bch1-5’utr-oleo2-nos;
e、以拟南芥的cdna为模板,扩增dgat1目的基因,然后在dgat1目的基因的5’末端和3’末端均加上bamhi单酶切位点,再通过单酶切位点bamhi将dgat1目的基因克隆到载体pyes2-35s-3’utr-bch1-5’utr-oleo2-nos,获得中间载体pyes2-35s-3’utr-dgat1-bch1-5’utr-oleo2-nos,即pyes2-dgat1-oleo2;
f、以酵母基因组为模板,扩增gat1目的基因,在gat1目的基因的5’端引入酶切位点bamhi,3’端引入酶切位点xhoi,通过bamhi和xhoi将gat1目的基因克隆到载体pbi121-35s,构成单基因表达载体pbi121-35s-gat1;然后将中间载体pyes2-dgat1-oleo2中的基因片段dgat1-oleo2通过clai位点克隆到载体pbi121-35s-gat1,获得三基因植物表达载体pbi121-dgat1-oleo2-gat1;
g、以拟南芥的cdna为模板,扩增lec2目的基因,再在其3’端引入xbai、5’端引入saci,然后将lec2目的基因克隆到pbi121-35s,构成单基因表达载体pbi121-lec2;在扩增的lec2目的基因的5’末端和3’末端均加上stui单酶切位点,再通过单酶切位点stui将lec2目的基因克隆到三基因植物表达载体pbi121-dgat1-oleo2-gat1,获得四基因表达载体pbi121-lec2-dgat1-oleo2-gat1。
本发明还保护所述的不同基因组合在提高植物营养组织油含量上的应用。
进一步的,所述植物为烟草、大豆、油菜、向日葵、花生、玉米、高粱或苜蓿。
本发明另外还保护所述的不同基因组合在提高与植物营养组织中油合成与积累相关联的物质生产中的应用。
进一步的,所述与植物营养组织中油合成与积累相关联的物质生产为脂溶性物质生产。
本发明的有益效果:
1、本发明利用基因工程手段,通过合理设计并成功构建了含有lec2、dgat1、oleo2和gat1基因组合的植物表达载体,其中的基因与驱使其有效表达的强度不同的启动子连接,再利用农杆菌转染将其导入植物的营养组织中,使目的基因在植物营养器官中异位或异源表达,并得到稳定遗传,最后筛选得到营养组织内高油的转基因植株,为创建在植物营养组织中生产植物油的体系提供分子手段。
2、本发明构建的基因表达载体,转化到拟南芥中,转基因植株中lec2与其它基因发生了互作,使得油脂含量相较lec2基因单独存在时得到显著增加,pl2、pdog1、pldo和pldog1的转基因植株中的三酰甘油的含量与野生型拟南芥相比,增加的倍数分别为2.08,2.47,8.99,5.10;这种互作也使得14:0和16:0的脂肪酸含量明显下降,20:0以上的超长链脂肪酸含量增加。将构建的基因表达载体转化到烟草中,阳性转基因植株与野生相比,叶片中tag的含量增加可达18.3倍,这也进一步说明,本发明所述基因组合和基因表达载体形成了提高植物营养器官含油量较优的表达体系,为探究提高植物营养器官中油脂含量或与油脂和合成与积累相关的物质的生产体系提供了条件。
附图说明
图1是重组载体pbi121-35smcs的构建流程图;
图2是重组载体pyes2-35s-nos的构建流程图;
图3是重组载体pyes2-35s-3’utr-bch1-5’utr-nos的构建流程图;
图4是重组载体pyes2-35s-3’utr-bch1-5’utr-oleo2-nos的构建流程图;
图5是重组载体pyes2-dgat1-oleo2的构建流程图;
图6是重组载体pbi12135s-gat1的构建流程图;
图7是重组载体pbi121-lec2的构建流程图;
图8是重组载体pbi121-dgat1-oleo2-gat1的构建流程图;
图9是重组载体pbi121-lec2-dgat1-oleo2-gat1的构建流程图;
图10是pyes2-dgat1-oleo2载体的酶切鉴定图;其中:a,泳道1、2、3和4:用bamhi酶切;m:1kbdnaladder;
图11是pyes2-dgat1-oleo2载体的pcr鉴定图;泳道1、2、3和4:片段35s-dgat1;
图12是pbi121-dgat1-oleo2-gat1双元载体的酶切鉴定图;其中:a,泳道1和2:stui酶切;泳道1’和2’:stui和noti酶切;m:1kbdnaladder;
图13是pbi121-dgat1-oleo2-gat1双元载体的酶切鉴定图b,泳道1和2:clai酶切;
图14是pbi121-dgat1-oleo2-gat1双元载体的pcr鉴定图c,泳道1和2:目的基因gat1;m:1kbdnaladder;
图15是双元载体pbi121-lec2-dgat1-oleo2-gat1的酶切鉴定图;其中:a,泳道1和2:用stui酶切pbi121-lec2-dgat1-oleo2-gat1;m:1kbdnaladder;
图16是双元载体pbi121-lec2-dgat1-oleo2-gat1的pcr鉴定图;b,泳道1和2:目的片段lec2-dgat1;m:dl2000;
图17是薄层色谱法分离三酰甘油的结果图;其中:a,泳道1:橄榄油;泳道2和3:野生拟南芥wt;泳道4、5和6:转基因植株pbi121-lec211号;泳道7、8和9:转基因植株pbi121-lec214号。b,泳道1和2:转基因植株pbi121-dgat1-oleo2-gat117号;泳道5、6和7:转基因植株pbi121-dgat1-oleo2-lec26号;泳道8和9:转基因植株pbi121-dgat1-oleo2-lec2-gat1。
图18是野生型拟南芥wt的气相色谱分析图;
图19是转基因拟南芥pbi121-dgat1-oleo2-gat1的气相色谱分析图;
图20是转基因拟南芥pbi121-lec2的气相色谱分析图;
图21是转基因拟南芥pbi121-lec2-dgat1-oleo2的气相色谱分析图;
图22是转基因拟南芥pbi121-lec2-dgat1-oleo2-gat1的气相色谱分析图;
图23是不同转基因拟南芥中脂肪酸组分对比分析图;
图24是不同转基因拟南芥中tag含量分析图;
其中,在图23和图24中:wt:野生拟南芥;lec2:转pbi121-lec2拟南芥;3-gat1:转pbi121-dgat1-oleo2-gat1拟南芥;l-d-o:转pbi121-lec2-dgat1-oleo2拟南芥;4-gat1:转pbi121-lec2-dgat1-oleo2-gat1拟南芥。
图25是烟草中三酰甘油含量分析图。
具体实施方式
下面通过具体的实施例对本发明做进一步的解释说明。需要说明的是,以下实施例应理解为说明性的,而非限制本发明的保护范围,对本领域技术人员而言,在不背离本发明实质和范围的前提下,作出的一些非本质的改进和调整仍属于本发明的保护范围。
实施例:
1、转录单元的构建和目的基因的筛选
为了构建不同的植物转录单元,本申请选择可以在营养器官中组成性表达的35s和bch1作为启动子。此外,还选择参与油脂合成的目的基因:(i)酵母中编码3-磷酸甘油酰基转移酶的基因gat1;(ii)编码二酰甘油酰基转移酶的一个基因dgat1,前者和后者分别参与油脂合成的第一步和第三步酰化反应;(iii)oleosin2(oleo2)编码油体嵌合蛋白,可维持油滴的完整性和稳定性;(iv)将营养细胞转变为胚性细胞的调控因子lec2,其在叶片中异位表达能增加叶片油的合成和积累。
以上四种目的基因中gat1来源于酿酒酵母(saccharomycescerevisiae),dgat1、oleo2和lec2来源于拟南芥;除自身外,四种目的基因还包括与各自的核苷酸序列同源性达90%以上的基因或编码的氨基酸序列与各自的氨基酸序列同源性达95%以上的基因。
2、中间表达载体的构建
为了在一个植物双元载体上克隆多个基因,在载体pbi121-gus上引入一个多克隆位点,构建载体pbi121-35smcs。此外,还构建四个中间载体,分别为pyes2-35s-nos,pyes2-35s-3’utr-bch1-5’utr-nos,pyes2-35s-3’utr-bch1-5’utr-oleo2-nos和pyes2-dgat1-oleo2,它们可作为在双元表达载体中插入多基因的辅助载体。
2.1载体pbi121-35smcs的构建
在pbi121-gus中插入启动子35s和多克隆位点,构建重组载体pbi121-35smcs。具体步骤为:以质粒pbi121-gus为模板,用引物zzf47和zzf48进行pcr扩增,获得35spromoter。然后以35spromoter为模板,分别用3对引物zzf47和zzf49,zzf47和zzf50,zzf47和zzf51依次对其纯化回收产物进行pcr扩增,最终获得dna片段35s-mcs。其中多克隆位点包括bamhi,xmai,smai,xhoi和saci。通过酶切位点hindiii和saci将dna片段35smcs克隆到载体pbi121-gus,从而获得新的双元载体pbi121-35smcs,如图1所示。
分别用clai和ecori,hindiii和saci对pbi121-35smcs载体进行双酶切,将酶切产物进行凝胶电泳,酶切的片段分别为1595bp和11313bp,915bp和11993bp,正确。且经测序验证,序列正确。
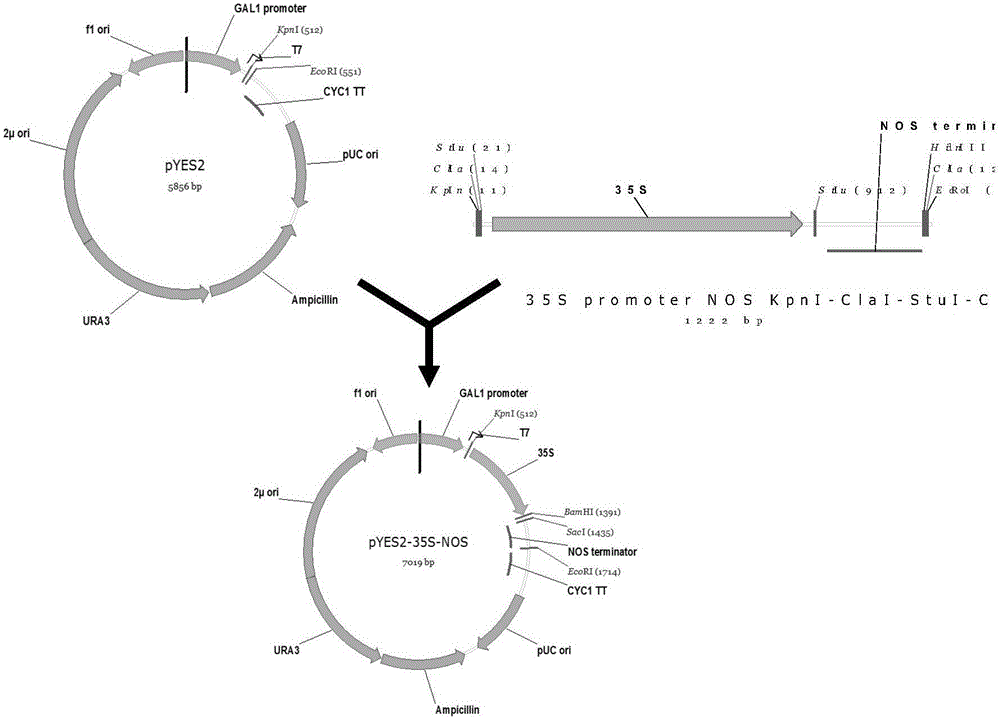
2.2中间载体pyes2-35s-nos的构建
以pbi121-35smcs为模板,用引物zzf52和zzf53进行pcr扩增,获得35s-nos。然后以纯化回收的35s-nos-t为模板,用引物zzf54和zzf55进行pcr扩增,在5’端和3’端加入多酶切位点。通过酶切位点kpni和ecori将35s-nos克隆到载体pyes2,从而获得中间载体pyes2-35s-nos,如图2所示。
分别用kpni和ecori,bamhi和saci对pyes2-35s-nos载体进行双酶切,将酶切产物进行凝胶电泳,酶切的片段分别为5817bp和1202bp,44bp和6975bp,正确。且经测序验证,序列正确。
2.3中间载体pyes2-35s-3’utr-bch1-5’utr-nos的改造
以拟南芥基因组dna为模板,用引物zzf56和zzf57进行pcr扩增,获得3’utr-bch1-5’utr。其中,3’utr为at4g25710基因的终止子,bch1为at4g25700基因的启动子。然后以纯化回收的3’utr-bch1-5’utr为模板,用引物zzf58和zzf59进行pcr扩增,在5’端和3’端加入bamhi、saci酶切位点。通过酶切位点bamhi和saci将目的基因3’utr-bch1-5’utr插入到载体pyes2-35s-nos,获得中间载体pyes2-35s-3’utr-bch1-5’utr-nos,如图3所示。
分别用kpni和ecori,bamhi和saci对pyes2-35s-3’utr-bch1-5’utr-nos载体进行双酶切,将酶切产物进行凝胶电泳,酶切的片段分别为3662bp和5817bp,2504bp和6975bp,正确。且经测序验证,序列正确。
2.4中间载体pyes2-35s-3’utr-bch1-5’utr-oleo2-nos的构建
以拟南芥的cdna为模板,用引物zzf60和zzf61进行pcr扩增获得目的基因oleo2(基因序列如seqidno:3所示,编码的氨基酸序列如seqidno:7所示),然后以纯化回收产物为模板,分别用引物zzf62和zzf63,进行pcr扩增,分别在5’末端和3’末端加上单酶切位点,最终获得带有酶切位点的目的基因oleo2(at5g40420)-saci-saci。通过酶切位点saci将目的基因oleo2-saci-saci克隆到载体pyes2-35s-3’utr-bch1-5’utr-nos,获得中间载体pyes2-35s-3’utr-bch1-5’utr-oleo2-nos,如图4所示。
分别用saci和ecori,saci对pyes2-35s-3’utr-bch1-5’utr-oleo2-nos载体进行酶切,将酶切产物进行凝胶电泳,酶切的片段分别为880bp和9229bp,630bp和9479bp,酶切验证正确。同时,以pyes2-35s-3’utr-bch1-5’utr-oleo2-nos为模板,zzf56(bch1fp)和zzf61(oleo2rp)为引物,pcr扩增bch1-oleo2基因片段并验证其方向性,结果扩增出一条3105bp的亮条带,条带大小正确且方向性正确。且经测序验证,序列正确。
2.5中间载体pyes2-dgat1-oleo2的构建
以拟南芥的cdna为模板,用引物zzf64和zzf65进行pcr扩增获得目的基因dgat1(基因序列如seqidno:2所示,编码的氨基酸序列如seqidno:6所示),然后以纯化回收产物为模板,用引物zzf66和zzf67进行pcr扩增,分别在5’末端和3’末端加上单酶切位点,最终获得带有酶切位点的目的基因dgat1(at2g19450)-bamhi-bamhi,通过酶切位点bamhi将目的基因dgat1-bamhi-bamhi克隆到载体pyes2-35s-3’utr-bch1-5’utr-oleo2-nos,最终获得中间载体pyes2-35s-3’utr-dgat1-bch1-5’utr-oleo2-nos,即pyes2-dgat1-oleo2,如图5所示。
用bamhi对pyes2-dgat1-oleo2载体进行单酶切,将酶切产物进行凝胶电泳,酶切的片段为1575bp和10109bp,如图10所示。图11所示,以pyes2-dgat1-oleo2为模板,zzf52(35sfp)和zzf65(dgat1rp)为引物,pcr扩增出一条2408bp的亮条带,来验证其方向性。且经测序验证,序列正确。
3植物表达载体的构建
运用传统的基因克隆方法,先构建单基因表达载体pbi121-35sgat1,然后将中间载体pyes2-dgat1-oleo2中的基因片段dgat1-oleo2通过clai位点克隆到载体pbi121-35sgat1,最终获得三基因植物表达载体pbi121-dgat1-oleo2-gat1。用引物zzf72和zzf73,zzf74和zzf75扩增单基因表达载体pbi121-lec2获得lec2ptu-stui-stui,通过酶切位点stui将目的片段lec2ptu-stui-stui插入三基因表达载体pbi121-dgat1-oleo2-gat1,最终获得四基因表达载体pbi121-lec2-dgat1-oleo2-gat1。
3.1单基因表达载体pbi121-35sgat1的构建
以酵母基因组为模板,用引物gat1fpbamhi和gat1rpxhoi进行pcr扩增,于gat1的5’端引入酶切位点bamhi,3’端引入酶切位点xhoi,最终获得目的基因gat1(ykr076w)-bamhi-xhoi。其中,gat1的基因序列如seqidno:1所示,编码的氨基酸序列如seqidno:5所示。通过双酶切位点bamhi和xhoi将目的基gat1-bamhi-xhoi克隆到载体pbi121-35smcs,构成单基因表达载体pbi12135s-gat1,如图6所示。
分别用clai和xhoi,bamhi和xhoi对pbi12135s-gat1载体进行双酶切,将酶切产物进行凝胶电泳,酶切的片段分别为3525bp和11598bp,2241bp和12882bp,正确。且经测序验证,序列正确。
3.2.单基因表达载体pbi121-lec2的构建
通过双酶切位点xbai和saci将目的基因lec2-xbai-saci插入双元载体pbi121-35smcs,构成单基因表达载体pbi121-lec2,如图7所示。其中lec2的基因序列如seqidno:4所示,编码的氨基酸序列如seqidno:8所示。
3.3三基因表达载体pbi121-dgat1-oleo2-gat1的构建
通过酶切位点clai将目的片段dgat1-oleo2从载体pyes2-dgat1-oleo2上切下,克隆到单基因表达载体pbi12135s-gat1,构成三基因表达载体pbi121-dgat1-oleo2-gat1,如图8所示。
图12所示,分别用stui,stui和noti对pbi121-dgat1-oleo2-gat1载体进行酶切,将酶切产物进行凝胶电泳,酶切的片段分别为20982bp,4640bp和16342bp。且经测序验证,序列正确。图13所示,用clai对pbi121-dgat1-oleo2-gat1载体进行酶切,将酶切产物进行凝胶电泳,酶切的片段为5859bp和15123bp。图14所示,以pbi121-dgat1-oleo2-gat1为模板,gat1fpbamhi和gat1rpxhoi为引物,pcr扩增出一条2247bp的亮条带。
3.4四基因表达载体pbi121-lec2-dgat1-oleo2-gat1的构建
以单基因表达载体pbi121-lec2为模板,用引物zzf72和zzf73,zzf74和zzf75进行pcr扩增,获得lec2ptu-stui-stui。通过酶切位点stui将目的片段lec2ptu-stui-stui插入三基因表达载体pbi121-dgat1-oleo2-gat1,最终获得四基因表达载体pbi121-lec2-dgat1-oleo2-gat1,如图9所示。
图15所示,用stui对pbi121-lec2-dgat1-oleo2-gat1载体进行单酶切,将酶切产物进行凝胶电泳,酶切的片段分别为2234bp和21842bp。且经测序验证,序列正确。图16所示,以pbi121-lec2-dgat1-oleo2-gat1为模板,zzf68和zzf65为引物,pcr扩增出一条3808bp的亮条带。
4根癌农杆菌gv3101的转化与鉴定
制备感受态根癌农杆菌gv3101,将构建的5种双元表达载体pbi121-35smcs,pbi121-gat1,pbi121-lec2,pbi121-dgat1-oleo2-gat1和pbi121-lec2-dgat1-oleo2-gat1分别进行根癌农杆菌的转化,然后再提取根癌农杆菌中的质粒,并对其进行进一步酶切或pcr验证。
5拟南芥的遗传转化
采用拟南芥花序侵染法,即用分别含有5种表达载体pbi121-35smcs,pbi121-gat1,pbi121-lec2,pbi121-dgat1-oleo2-gat1和pbi121-lec2-dgat1-oleo2-gat1的根癌农杆菌侵染有大量花苞的哥伦比亚型拟南芥,并获得转基因拟南芥植株。
6转基因拟南芥的筛选鉴定
6.1转基因拟南芥的抗性筛选
用含50μg/mlkan的1/2ms的固体培养基分别对t1代转基因拟南芥种子进行抗性筛选。观察拟南芥幼苗的生长状况,如果叶片仍为绿色,且根长较长,则为t1代转基因植株。而非转基因植株的性状则是叶片变黄,根长较短。将转基因植株移栽至基质(泥炭土:蛭石:珍珠岩=6:3:1,v/v/v)中,移栽时要将根部粘连的培养基洗掉,移栽的过程中要防止根部的损伤。待叶片长大后,提取叶片基因组dna,pcr验证。待其成熟后,收获t2代种子。
同理对t2代单拷贝转基因拟南芥的筛选,t2代中若抗性性状分离比为3:1,证明是单拷贝。将单拷贝转基因拟南芥移栽至基质中,待叶片长大后,需再进行pcr验证。待其成熟后,收获t3代种子。
同理对t3代纯合转基因拟南芥的筛选,t3代中如果不出现抗性性状分离,则证明是纯合株系。选着全抗的纯合株系用于后续试验。将纯合转基因拟南芥移栽至基质中,待叶片长大后,再进行pcr验证。待其成熟后,收获t4代种子。
对转基因拟南芥的抗性筛选结果为:待分别侵染过5种根癌农杆菌pbi121-35smcs,pbi121-gat1,pbi121-lec2,pbi121-dgat1-oleo2-gat1和pbi121-lec2-dgat1-oleo2-gat1的野生型拟南芥成熟后,分别收获种子,干燥处理后置于4℃冰箱保存。将种子灭菌后均匀撒播于含50μg/ml卡那霉素的1/2ms培养基上,成功侵染的拟南芥种子能够正常生长,而未成功侵染的拟南芥种子在卡那霉素的作用下逐渐黄化死亡。将初步筛选到的幼苗移栽到基质中,置于温室中培养。单基因表达载体的转化率为10%,三基因表达载体的转化率为5%,而四基因表达载体的转化率很低,甚至达不到1%。
6.2转基因拟南芥的pcr鉴定
取生长2-3周的5种转基因拟南芥pbi121-35smcs,pbi121-gat1,pbi121-lec2,pbi121-dgat1-oleo2-gat1和pbi121-lec2-dgat1-oleo2-gat1的新鲜叶片,提取转基因拟南芥叶片中的基因组dna。以转基因拟南芥基因组dna为模板,分别以zzf47和zzf48,gat1fp和gat1rp,zzf52和zzf65,zzf56和zzf61,zzf68和zzf65引物,pcr扩增插入片段35s,gat1,35s-dgat1,bch1-oleo2和lec2-nos,片段大小分别为911bp,2206bp,2431bp,3125bp和3808bp。结果证明拟南芥中成功导入目的基因。
6.3转基因拟南芥的脂质分析
分别提取5种转基因拟南芥pbi121-35smcs,pbi121-gat1,pbi121-lec2,pbi121-dgat1-oleo2-gat1和pbi121-lec2-dgat1-oleo2-gat1叶片或茎中的脂质,用薄层色谱法分离三酰甘油,然后用气相色谱法分析三酰甘油中的脂肪酸组分及相对含量。
6.3.1转基因拟南芥中三酰甘油(tag)的提取和分离
取4μl100μg/μl的橄榄油做对照,取50μl转基因拟南芥的油脂点于薄层色谱板,然后将薄层色谱板置于含有饱和展开剂正己烷:乙醚:乙酸(70:30:1,v/v/v)的单槽层析缸中层析。待其吹干后,置于碘蒸汽中显色,直至斑点完全显现,如图17所示。从薄层色谱板上可以看出转基因植株中三酰甘油的含量有明显增加。
6.3.2三酰甘油含量的测定
将薄层色谱板上的tag刮下,去甲酯化,用气相色谱仪对其分析,结果如图18-22所示。
从气相色谱图18-22看出,转基因拟南芥营养器官中tag的组分发生明显变化。图23得出,目的基因lec2促进了长链脂肪酸合成与积累,这一现象与lec2可将营养细胞转变为胚性细胞这一功能是一致的。当lec2与dgat1、oleo2、gat1共同存在时,与野生拟南芥相比较,14:0和16:0的脂肪酸含量下降20%,20:0以上的超长链脂肪酸含量增加15%-20%。当只含有转基因lec2时,与野生拟南芥相比较,14:0和16:0的脂肪酸含量无明显下降,20:0以上的超长链脂肪酸含量增加5%-10%。说明lec2与其它基因发生了互作。
图24表明,当lec2存在时,转基因拟南芥中三酰甘油含量有所增加,这与之前的报道是一致的。当lec2与dgat1、oleo2、gat1共同存在时,三酰甘油含量增加幅度增加,进一步说明lec2与其它基因发生了互作。不存在lec2基因时,dgat1、oleo2和gat1可增加转基因拟南芥三酰甘油的含量。pl2、pdog1、pldo和pldog1的转基因植株中的三酰甘油的含量与野生型拟南芥相比,增加的倍数分别为2.08,2.47,8.99,5.10。
7烟草的遗传转化
以大田栽培品种k326的叶片作为外植体,通过根瘤农杆菌介导法,将植物表达载体pbi121-lec2-dgat1-oleo2-gat1导入烟草中,用含50μg/ml卡那霉素(kan)的1/2ms的固体培养基经过(kan)筛选,获得转基因烟草植株,具体步骤为:取温室中生长约2个月的k326幼嫩叶片,进行表面消毒,放于1/2ms培养基上,暗处,25±1℃预培养2-3天。农杆菌(已转入植物表达载体pbi121-lec2-dgat1-oleo2-gat1)侵染后黑暗条件下共培养2天,然后将叶片转移到筛选培养基上,25±1℃,16h光照,8h黑暗,培养40天,将芽切下转入生根培养基中,14天后获得转基因烟草植株。
8.转基因烟草的筛选鉴定
以提取的转基因烟草pbi121-35smcs,pbi121-gat1,pbi121-dgat1-oleo2-gat1和pbi121-lec2-dgat1-oleo2-gat1的基因组dna为模板,用引物zzf47和zzf48,gat1fp和gat1rp,zzf52和zzf65,zzf56和zzf61,zzf68和zzf65pcr扩增插入片段35s,gat1,35s-dgat1,bch1-oleo2和lec2-nos进行pcr验证,58株转基因植株中有22株能同时扩增出5个外源基因片段,阳性率为37.93%。
提取t0代转基因烟草植株(pbi121-lec2-dgat1-oleo2-gat1)叶片中的总脂,然后进行tlc分离(薄层层析法),通过气相色谱分析叶片中三酰甘油(tag)含量与脂肪酸组分。结果可知,从22株阳性转化植株中筛选出2株高油植株5-13、5-22。如图25所示,与野生相比,tag的含量分别增加18.3、10.9倍。
本发明所述lec2、dgat1、oleo2和gat1的基因组合,在启动子作用下协同表达和作用,能够显著提高拟南芥或烟草营养组织中的油脂含量,可将此方法进一步应用于大豆、油菜、花生或玉米等其它植物中,有望促进生物燃料的生产;或将其应用于苜蓿、高粱等饲料作物中提高饲料的营养。
以上实施例中所用引物及其序列表如下表1所示。
表1:引物序列表
sequencelisting
<110>浙江农林大学
<120>异位表达的不同基因组合及其在提高植物营养组织油含量的应用
<130>1
<160>8
<170>patentinversion3.3
<210>1
<211>2232
<212>dna
<213>酿酒酵母
<400>1
atgtctgctcccgctgccgatcataacgctgccaaacctattcctcatgtacctcaagcg60
tcccgacggtacaaaaattcatacaatggattcgtatacaatatacatacatggctgtat120
gatgtgtctgtatttctgtttaatattttgttcactattttcttcagagaaattaaggta180
cgtggtgcatataacgttcccgaagttggggtgccaaccatccttgtgtgtgcccctcat240
gcaaatcagttcatcgacccggctttggtaatgtcgcaaacccgtttgctgaagacatca300
gcgggaaagtcccgatccagaatgccttgttttgttactgctgagtcgagttttaagaaa360
agatttatctctttctttggtcacgcaatgggcggtattcccgtgcctagaattcaggac420
aacttgaagccagtggatgagaatcttgagatttacgctccggacttgaagaaccacccg480
gaaatcatcaagggccgctccaagaacccacagactacaccagtgaactttacgaaaagg540
ttttctgccaagtccttgcttggattgcccgactacttaagtaatgctcaaatcaaggaa600
atcccggatgatgaaacgataatcttgtcctctccattcagaacatcgaaatcaaaagtg660
gtggagctcttgactaatggtactaattttaaatatgcagagaaaatcgacaatacggaa720
actttccagagtgtttttgatcacttgcatacgaagggctgtgtaggtattttccccgag780
ggtggttctcatgaccgtccttcgttactacccatcaaggcaggtgttgccattatggct840
ctgggcgcagtagccgctgatcctaccatgaaagttgctgttgtaccctgtggtttgcat900
tatttccacagaaataaattcagatctagagctgttttagaatacggcgaacctatagtg960
gtggatgggaaatatggcgaaatgtataaggactccccacgtgagaccgtttccaaacta1020
ctaaaaaagatcaccaattctttgttttctgttaccgaaaatgctccagattacgatact1080
ttgatggtcattcaggctgccagaagactatatcaaccggtaaaagtcaggctacctttg1140
cctgccattgtagaaatcaacagaaggttacttttcggttattccaagtttaaagatgat1200
ccaagaattattcacttaaaaaaactggtatatgactacaacaggaaattagattcagtg1260
ggtttaaaagaccatcaggtgatgcaattaaaaactaccaaattagaagcattgaggtgc1320
tttgtaactttgatcgttcgattgattaaattttctgtctttgctatactatcgttaccg1380
ggttctattctcttcactccaattttcattatttgtcgcgtatactcagaaaagaaggcc1440
aaagagggtttaaagaaatcattggttaaaattaagggtaccgatttgttggccacatgg1500
aaacttatcgtggcgttaatattggcaccaattttatacgttacttactcgatcttgttg1560
attattttggcaagaaaacaacactattgtcgcatctgggttccttccaataacgcattc1620
atacaatttgtctatttttatgcgttattggttttcaccacgtattcctctttaaagacc1680
ggtgaaatcggtgttgaccttttcaaatctttaagaccactttttgtttctattgtttac1740
cccggtaagaagatcgaagaaatccaaacaacaagaaagaatttaagtctagagttgact1800
gctgtttgtaacgatttaggacctttggttttccctgattacgataaattagcgactgag1860
atattctctaagagagacggttatgatgtctcttctgatgcagagtcttctataagtcgt1920
atgagtgtacaatctagaagccgctcttcttctatacattctattggctcgctagcttct1980
aacgccctatcaagagtgaattcaagaggctcgttgaccgatattccaattttttctgat2040
gcaaagcaaggtcaatggaaaagtgaaggtgaaactagtgaggatgaggatgaatttgat2100
gagaaaaatcctgccatagtacaaaccgcacgaagttctgatctaaataaggaaaacagt2160
cgcaacacaaatatatcttcgaagattgcttcgctggtaagacagaaaagagaacacgaa2220
aagaaagaatga2232
<210>2
<211>1591
<212>dna
<213>拟南芥
<400>2
cttctggatccttcgaaatggcgattttggattctgctggcgttactacggtgacggaga60
acggtggcggagagttcgtcgatcttgataggcttcgtcgacggaaatcgagatcggatt120
cttctaacggacttcttctctctggttccgataataattctccttcggatgatgttggag180
ctcccgccgacgttagggatcggattgattccgttgttaacgatgacgctcagggaacag240
ccaatttggccggagataataacggtggtggcgataataacggtggtggaagaggcggcg300
gagaaggaagaggaaacgccgatgctacgtttacgtatcgaccgtcggttccagctcatc360
ggagggcgagagagagtccacttagctccgacgcaatcttcaaacagagccatgccggat420
tattcaacctctgtgtagtagttcttattgctgtaaacagtagactcatcatcgaaaatc480
ttatgaagtatggttggttgatcagaacggatttctggtttagttcaagatcgctgcgag540
attggccgcttttcatgtgttgtatatccctttcgatctttcctttggctgcctttacgg600
ttgagaaattggtacttcagaaatacatatcagaacctgttgtcatctttcttcatatta660
ttatcaccatgacagaggttttgtatccagtttacgtcaccctaaggtgtgattctgctt720
ttttatcaggtgtcactttgatgctcctcacttgcattgtgtggctaaagttggtttctt780
atgctcatactagctatgacataagatccctagccaatgcagctgataaggccaatcctg840
aagtctcctactacgttagcttgaagagcttggcatatttcatggtcgctcccacattgt900
gttatcagccaagttatccacgttctgcatgtatacggaagggttgggtggctcgtcaat960
ttgcaaaactggtcatattcaccggattcatgggatttataatagaacaatatataaatc1020
ctattgtcaggaactcaaagcatcctttgaaaggcgatcttctatatgctattgaaagag1080
tgttgaagctttcagttccaaatttatatgtgtggctctgcatgttctactgcttcttcc1140
acctttggttaaacatattggcagagcttctctgcttcggggatcgtgaattctacaaag1200
attggtggaatgcaaaaagtgtgggagattactggagaatgtggaatatgcctgttcata1260
aatggatggttcgacatatatacttcccgtgcttgcgcagcaagataccaaagacactcg1320
ccattatcattgctttcctagtctctgcagtctttcatgagctatgcatcgcagttcctt1380
gtcgtctcttcaagctatgggcttttcttgggattatgtttcaggtgcctttggtcttca1440
tcacaaactatctacaggaaaggtttggctcaacggtggggaacatgatcttctggttca1500
tcttctgcattttcggacaaccgatgtgtgtgcttctttattaccacgacctgatgaacc1560
gaaaaggatctatgtcatgaggatccactaa1591
<210>3
<211>872
<212>dna
<213>拟南芥
<400>3
attacaaagaaaataggtaaaaacaatttctcattagcttacaatggcggatacacaccg60
tgtcgaccgtactgatagacactttcaatttcagtcgccctatgaaggcggccgaggtca120
aggtcagtatgaaggtgaccgtggttacggtggtggcggttacaagagcatgatgcctga180
aagtggcccatctagtacccaagtattgtccctgttgattggagtccctgtcgtcggttc240
gctacttgccttggctggattacttctagctggttcggtgatcggcttaatggttgcttt300
accactatttctcctcttcagcccggttatagtcccagcggctctaactatcgggcttgc360
aatgacaggctttttagcctcggggatgttcggtctaaccgggcttagctcaatctcatg420
ggtcatgaactatcttcgtgggacaaggagaactgtgcctgagcaattggagtatgctaa480
gaggagaatggctgatgcggttggctacgcaggacaaaagggcaaagaaatgggccagca540
tgtgcagaacaaggcccaagatgttaaacaatatgatatttctaagccacatgacactac600
cactaagggtcatgagactcaggggaggacgacggctgcatgatgagttttcagtatgaa660
cggtagatatgtgttttcactattatgtcgttttttctgcattttcaatatgatgttatg720
tgttttttttgtttggctttttgttgaaccgtgtatgtgttttatgtttttgtaagcatg780
aaagatcgcaagtgttgtggtaatatttgaatgtaataatatgataagttgataaatcat840
gggaacatttaaattaggtggacatgtttagc872
<210>4
<211>2249
<212>dna
<213>拟南芥
<400>4
attgaaggcctgcaggtccccagattagccttttcaatttcagaaagaatgctaacccac60
agatggttagagaggcttacgcagcaggtctcatcaagacgatctacccgagcaataatc120
tccaggaaatcaaataccttcccaagaaggttaaagatgcagtcaaaagattcaggacta180
actgcatcaagaacacagagaaagatatatttctcaagatcagaagtactattccagtat240
ggacgattcaaggcttgcttcacaaaccaaggcaagtaatagagattggagtctctaaaa300
aggtagttcccactgaatcaaaggccatggagtcaaagattcaaatagaggacctaacag360
aactcgccgtaaagactggcgaacagttcatacagagtctcttacgactcaatgacaaga420
agaaaatcttcgtcaacatggtggagcacgacacacttgtctactccaaaaatatcaaag480
atacagtctcagaagaccaaagggcaattgagacttttcaacaaagggtaatatccggaa540
acctcctcggattccattgcccagctatctgtcactttattgtgaagatagtggaaaagg600
aaggtggctcctacaaatgccatcattgcgataaaggaaaggccatcgttgaagatgcct660
ctgccgacagtggtcccaaagatggacccccacccacgaggagcatcgtggaaaaagaag720
acgttccaaccacgtcttcaaagcaagtggattgatgtgatatctccactgacgtaaggg780
atgacgcacaatcccactatccttcgcaagacccttcctctatataaggaagttcatttc840
atttggagagaacacgggggactctagaaaatggataacttcttaccctttccctcttct900
aacgcaaactctgtccaagaactctctatggatcctaacaacaatcgctcgcacttcaca960
acagtccctacttatgatcatcatcaggctcagcctcatcacttcttgcctccgttttca1020
tacccggtggagcagatggcggcggtgatgaatcctcagccggtttacttatcggagtgt1080
tatcctcagatcccggttacgcaaaccggaagtgaattcggttctctggttggtaatcct1140
tgtttgtggcaagagagaggtggttttcttgatccgcgtatgacgaagatggcaaggatc1200
aacaggaaaaacgccatgatgagatcaagaaacaactctagccctaattctagtccaagt1260
gagttggttgattcaaagagacagctgatgatgcttaacttgaaaaataacgtgcagatc1320
tccgacaagaaagatagctaccaacagtccacatttgataacaagaagcttagggttttg1380
tgtgagaaggaattgaagaacagcgatgttgggtcactcgggaggatagttctaccaaag1440
agagatgcagaagcaaatcttccgaagctatctgataaagaaggaatcgttgtacagatg1500
agagatgttttctctatgcagtcttggtctttcaaatacaagttttggtccaataacaag1560
agcagaatgtatgtcctcgagaacacaggagaatttgtgaagcaaaatggagctgagata1620
ggagactttttaacaatatacgaggacgaaagcaagaatctctacttcgccatgaatgga1680
aattcgggaaaacaaaatgaaggaagagaaaatgagtcgagggaaaggaaccactacgaa1740
gaggcaatgcttgattacataccaagagacgaagaggaagcttccattgcaatgctcatc1800
ggaaatctaaacgatcactatcccatccctaacgatctcatggacctcaccactgacctt1860
cagcaccatcaagccacgtcctcatcaatgccacctgaggatcacgcgtacgtgggttca1920
tccgatgatcaggtgagctttaacgactttgagtggtggtgagagctcgaatttccccga1980
tcgttcaaacatttggcaataaagtttcttaagattgaatcctgttgccggtcttgcgat2040
gattatcatataatttctgttgaattacgttaagcatgtaataattaacatgtaatgcat2100
gacgttatttatgagatgggtttttatgattagagtcccgcaattatacatttaatacgc2160
gatagaaaacaaaatatagcgcgcaaactaggataaattatcgcgcgcggtgtcatctat2220
gttactagatcgggaattcaggcctaatg2249
<210>5
<211>743
<212>prt
<213>酿酒酵母
<400>5
metseralaproalaalaasphisasnalaalalysproileprohis
151015
valproglnalaserargargtyrlysasnsertyrasnglypheval
202530
tyrasnilehisthrtrpleutyraspvalservalpheleupheasn
354045
ileleuphethrilephephearggluilelysvalargglyalatyr
505560
asnvalprogluvalglyvalprothrileleuvalcysalaprohis
65707580
alaasnglnpheileaspproalaleuvalmetserglnthrargleu
859095
leulysthrseralaglylysserargserargmetprocyspheval
100105110
thralagluserserphelyslysargpheileserphepheglyhis
115120125
alametglyglyileprovalproargileglnaspasnleulyspro
130135140
valaspgluasnleugluiletyralaproaspleulysasnhispro
145150155160
gluileilelysglyargserlysasnproglnthrthrprovalasn
165170175
phethrlysargpheseralalysserleuleuglyleuproasptyr
180185190
leuserasnalaglnilelysgluileproaspaspgluthrileile
195200205
leuserserpropheargthrserlysserlysvalvalgluleuleu
210215220
thrasnglythrasnphelystyralaglulysileaspasnthrglu
225230235240
thrpheglnservalpheasphisleuhisthrlysglycysvalgly
245250255
ilepheprogluglyglyserhisaspargproserleuleuproile
260265270
lysalaglyvalalailemetalaleuglyalavalalaalaasppro
275280285
thrmetlysvalalavalvalprocysglyleuhistyrphehisarg
290295300
asnlyspheargserargalavalleuglutyrglygluproileval
305310315320
valaspglylystyrglyglumettyrlysaspserproarggluthr
325330335
valserlysleuleulyslysilethrasnserleupheservalthr
340345350
gluasnalaproasptyraspthrleumetvalileglnalaalaarg
355360365
argleutyrglnprovallysvalargleuproleuproalaileval
370375380
gluileasnargargleuleupheglytyrserlysphelysaspasp
385390395400
proargileilehisleulyslysleuvaltyrasptyrasnarglys
405410415
leuaspservalglyleulysasphisglnvalmetglnleulysthr
420425430
thrlysleuglualaleuargcysphevalthrleuilevalargleu
435440445
ilelyspheservalphealaileleuserleuproglyserileleu
450455460
phethrproilepheileilecysargvaltyrserglulyslysala
465470475480
lysgluglyleulyslysserleuvallysilelysglythraspleu
485490495
leualathrtrplysleuilevalalaleuileleualaproileleu
500505510
tyrvalthrtyrserileleuleuileileleualaarglysglnhis
515520525
tyrcysargiletrpvalproserasnasnalapheileglnpheval
530535540
tyrphetyralaleuleuvalphethrthrtyrserserleulysthr
545550555560
glygluileglyvalaspleuphelysserleuargproleupheval
565570575
serilevaltyrproglylyslysileglugluileglnthrthrarg
580585590
lysasnleuserleugluleuthralavalcysasnaspleuglypro
595600605
leuvalpheproasptyrasplysleualathrgluilepheserlys
610615620
argaspglytyraspvalserseraspalagluserserileserarg
625630635640
metservalglnserargserargserserserilehisserilegly
645650655
serleualaserasnalaleuserargvalasnserargglyserleu
660665670
thraspileproilepheseraspalalysglnglyglntrplysser
675680685
gluglygluthrsergluaspgluaspglupheaspglulysasnpro
690695700
alailevalglnthralaargserseraspleuasnlysgluasnser
705710715720
argasnthrasnileserserlysilealaserleuvalargglnlys
725730735
arggluhisglulyslysglu
740
<210>6
<211>520
<212>prt
<213>拟南芥
<400>6
metalaileleuaspseralaglyvalthrthrvalthrgluasngly
151015
glyglygluphevalaspleuaspargleuargargarglysserarg
202530
seraspserserasnglyleuleuleuserglyseraspasnasnser
354045
proseraspaspvalglyalaproalaaspvalargaspargileasp
505560
servalvalasnaspaspalaglnglythralaasnleualaglyasp
65707580
asnasnglyglyglyaspasnasnglyglyglyargglyglyglyglu
859095
glyargglyasnalaaspalathrphethrtyrargproservalpro
100105110
alahisargargalaarggluserproleuserseraspalailephe
115120125
lysglnserhisalaglyleupheasnleucysvalvalvalleuile
130135140
alavalasnserargleuileilegluasnleumetlystyrglytrp
145150155160
leuileargthraspphetrppheserserargserleuargasptrp
165170175
proleuphemetcyscysileserleuserilepheproleualaala
180185190
phethrvalglulysleuvalleuglnlystyrilesergluproval
195200205
valilepheleuhisileileilethrmetthrgluvalleutyrpro
210215220
valtyrvalthrleuargcysaspseralapheleuserglyvalthr
225230235240
leumetleuleuthrcysilevaltrpleulysleuvalsertyrala
245250255
histhrsertyraspileargserleualaasnalaalaasplysala
260265270
asnprogluvalsertyrtyrvalserleulysserleualatyrphe
275280285
metvalalaprothrleucystyrglnprosertyrproargserala
290295300
cysilearglysglytrpvalalaargglnphealalysleuvalile
305310315320
phethrglyphemetglypheileilegluglntyrileasnproile
325330335
valargasnserlyshisproleulysglyaspleuleutyralaile
340345350
gluargvalleulysleuservalproasnleutyrvaltrpleucys
355360365
metphetyrcysphephehisleutrpleuasnileleualagluleu
370375380
leucyspheglyasparggluphetyrlysasptrptrpasnalalys
385390395400
servalglyasptyrtrpargmettrpasnmetprovalhislystrp
405410415
metvalarghisiletyrpheprocysleuargserlysileprolys
420425430
thrleualaileileilealapheleuvalseralavalphehisglu
435440445
leucysilealavalprocysargleuphelysleutrpalapheleu
450455460
glyilemetpheglnvalproleuvalpheilethrasntyrleugln
465470475480
gluargpheglyserthrvalglyasnmetilephetrppheilephe
485490495
cysilepheglyglnprometcysvalleuleutyrtyrhisaspleu
500505510
metasnarglysglysermetser
515520
<210>7
<211>199
<212>prt
<213>拟南芥
<400>7
metalaaspthrhisargvalaspargthrasparghispheglnphe
151015
glnserprotyrgluglyglyargglyglnglyglntyrgluglyasp
202530
argglytyrglyglyglyglytyrlyssermetmetproglusergly
354045
proserserthrglnvalleuserleuleuileglyvalprovalval
505560
glyserleuleualaleualaglyleuleuleualaglyservalile
65707580
glyleumetvalalaleuproleupheleuleupheserprovalile
859095
valproalaalaleuthrileglyleualametthrglypheleuala
100105110
serglymetpheglyleuthrglyleuserserilesertrpvalmet
115120125
asntyrleuargglythrargargthrvalprogluglnleuglutyr
130135140
alalysargargmetalaaspalavalglytyralaglyglnlysgly
145150155160
lysglumetglyglnhisvalglnasnlysalaglnaspvallysgln
165170175
tyraspileserlysprohisaspthrthrthrlysglyhisgluthr
180185190
glnglyargthrthralaala
195
<210>8
<211>362
<212>prt
<213>拟南芥
<400>8
metaspasnpheleupropheproserserasnalaasnservalgln
151015
gluleusermetaspproasnasnasnargserhisphethrthrval
202530
prothrtyrasphishisglnalaglnprohishispheleupropro
354045
phesertyrprovalgluglnmetalaalavalmetasnproglnpro
505560
valtyrleuserglucystyrproglnileprovalthrglnthrgly
65707580
serglupheglyserleuvalglyasnprocysleutrpglngluarg
859095
glyglypheleuaspproargmetthrlysmetalaargileasnarg
100105110
lysasnalametmetargserargasnasnserserproasnserser
115120125
prosergluleuvalaspserlysargglnleumetmetleuasnleu
130135140
lysasnasnvalglnileserasplyslysaspsertyrglnglnser
145150155160
thrpheaspasnlyslysleuargvalleucysglulysgluleulys
165170175
asnseraspvalglyserleuglyargilevalleuprolysargasp
180185190
alaglualaasnleuprolysleuserasplysgluglyilevalval
195200205
glnmetargaspvalphesermetglnsertrpserphelystyrlys
210215220
phetrpserasnasnlysserargmettyrvalleugluasnthrgly
225230235240
gluphevallysglnasnglyalagluileglyasppheleuthrile
245250255
tyrgluaspgluserlysasnleutyrphealametasnglyasnser
260265270
glylysglnasngluglyarggluasngluserarggluargasnhis
275280285
tyrgluglualametleuasptyrileproargaspgluglugluala
290295300
serilealametleuileglyasnleuasnasphistyrproilepro
305310315320
asnaspleumetaspleuthrthraspleuglnhishisglnalathr
325330335
sersermetthrprogluasphisalatyrvalglyserseraspasp
340345350
glnvalserpheasnasppheglutrptrp
355360
- 还没有人留言评论。精彩留言会获得点赞!