水稻吲哚乙酸氨基化合成酶基因OsGH3.6的编码序列及其应用的制作方法
本发明属于基因工程技术领域,具体涉及一种在水稻中表达的吲哚乙酸氨基化合成酶基因osgh3.6的编码序列及其应用。具体包括:吲哚乙酸氨基化合成酶基因osgh3.6基因的核苷酸编码序列的克隆,对水稻此基因内源的不同器官、组织的空间表达模式,干旱、高盐胁迫以及植物激素处理后的表达模式变化进行分析鉴定,以及osgh3.6tilling突变体的检测以及根长和产量分析。
背景技术:
生长素在植物体内分布很广,如根、茎、叶、花、果实、种子及胚芽鞘等。但主要集中在生长旺盛的部位(胚芽鞘、幼嫩的茎尖、根尖、叶片、受精后的子房和幼嫩的种子等),而在趋向衰老的组织和器官中,生长素的含量相对较少(ljungetal.,2001;friml,2003)。在植物的生长发育过程中,体内合成的生长素是可以从其合成部位向作用部位运输的。
生长素早期响应基因分为三大类:aux/iaa、gh3家族和saur(smallauxin-uprna)基因家族。其中,aux/iaa蛋白在生长素信号通路中起着转录抑制的作用,它是生长素早期响应因子中研究最为清楚和主要的一类基因家族。研究发现,aux/iaa是核蛋白,含有i、ii、iii、iv四个结构域(abeletal.,1994);其中i结构域可以决定它是转录抑制子(tiwarietal.,2004),ii结构域起着快速降解aux/iaa蛋白的作用,iii和iv结构域能与arf的iii和iv结构域相互作用(ouelletetal.,2001)。水稻中有31个aux/iaa成员。其中,osiaa11功能获得突变可抑制水稻侧根原基的发生(zhuetal.,2012)。osiaa13通过参与生长素信号途径,从而调控与水稻侧根起始的相关基因的表达(kitomietal.,2012)。osiaa23可介导的生长素信号通路,对水稻的根尖静止中心细胞的胚后维持起一定的作用(nietal.,2011)。
第一个gh3家族基因最早是在大豆中发现的,以后相继在拟南芥、水稻和葡萄等中也有发现。至今水稻中有13个gh3基因家族成员,各成员在水稻染色体上的分布也各有不同。通过构建同源进化树分析发现,可将水稻中的gh3家族基因分为两大类(mukeshetal.,2006)。近年来,通过突变体筛选及功能基因组学研究,水稻中的有些gh3家族蛋白的功能已被确定。发现gh3家族的基因属于吲哚乙酸氨基化合成酶基因,可以催化iaa与氨基酸结合,使游离的iaa含量降低,从而行使生理生化的功能。有研究报道,发现osgh3.1和osgh3-8可通过降低水稻中生长素的含量,减少细胞壁的松弛,从而增强防御反应和对病原菌的抗性(domingoetal.,2009;dingetal.,2008)。osgh3-2过表达株系中,根系呈细丝状,侧根的数目减少,表明osgh3-2可能参与调控水稻侧根的发育(duetal.,2012)。tld1/osgh3.13还可通过下调吲哚乙酸的含量,来改变水稻株型和提高水稻抗旱性(zhangetal.,2009)。
水稻根系的发育受自身内部和外界环境因素的影响,五大植物激素中的生长素和细胞分裂素对水稻根系的发育发挥着重要的调控作用。其中,生长素的代谢、转运和信号转导紧密地影响着根系的生长发育。研究发现,有些基因与生长素共同在水稻根系发育中起作用,也有一些与生长素相关的水稻突变体,它们可以表现出冠根畸形的表型。如crownlessroot1(crl1)/adventitiousrootless1(arl1)编码了一个含有as2/lob结构域的转录因子,并作为生长素应答因子介导生长素信号通路,从而正向调控水稻冠根的形成;其突变体的冠根较少(inukaietal.,2005)。另外,还发现crl4/osgnom1蛋白负责协助生长素外流载体pin1的极性定位,通过介导生长素参与水稻冠根的形成;其突变体crl4的不定根显著减少,侧根减少和根的向重力性也受到影响(kitomietal.,2008)。oscand1与拟南芥中的cand1同源,可以参与生长素信号通路中的g2/m细胞周期在冠根原基的转化,从而影响冠状根的伸出(wangetal.,2011)。最近发现过表达水稻中的mir393a或mir393b,会导致生长素的受体基因ostir1和osafb2下调,生长素信号通路受阻,从而影响主根的生长和冠根的起始(xiaetal.,2012)。
根系是一株植物的全部根的总称,它在植物生长发育过程中起着极其重要的作用。根系有吸收水分及养分、固定和支撑植株等功能,是植物激素、有机酸和氨基酸等物质合成与转化的重要场所。水稻属于禾本科单子叶作物,其根系由胚根(种子根)、冠根(不定根)和侧根组成(coudertetal.,2010)。胚根是在发芽时期形成的第一条根,它是由根顶端分生组织分化形成的。冠根是由茎节处的中柱鞘细胞依次经过垂周分裂和平周分裂而成,从下位节向上位节发根。侧根是冠根分生出的次生根,可以分为大侧根和小侧根。通过对水稻根的径向切面观察,发现包括中柱(韧皮部、木质部和中柱鞘)、内皮层和皮质(通气组织、厚壁组织、外皮层和表皮)组成(rebouillatetal.,2009)。它的径向结构反映了水稻根系能在有氧和缺氧条件下的生存的能力。
技术实现要素:
本发明的目的在于提出一种新的水稻基因,还提供该水稻基因的蛋白编码序列,并提供该水稻基因的应用。
本发明提供的水稻中表达的吲哚乙酸氨基化合成酶基因osgh3.6基因编码序列及其应用,具体包括:吲哚乙酸氨基化合成酶基因osgh3.6基因的核苷酸编码序列的克隆,序列的同源比对,对水稻此基因内源的不同器官、组织的空间表达模式,干旱、高盐胁迫以及植物激素处理后的表达模式变化进行分析鉴定,以及osgh3.6tilling突变体的检测以及根长和产量分析。
本发明通过克隆水稻生长素螯合基因osgh3.6,对其时空表达模式及胁迫响应方式进行确定,结果显示osgh3.6基因在各个器官中组成型表达,但在不同部位叶片以及根中的表达都无显著差异,在茎中的表达量显著升高。盐胁迫下,osgh3.6的表达显著升高,而干旱处理后,osgh3.6的表达显著降低。在kt处理下,osgh3.6的表达量基本无变化,而6-ba和iaa处理后,osgh3.6的表达显著降低;在2,4-d和naa处理后,osgh3.6的表达都出现了而不同程度的升高。osgh3.6tilling突变体在幼苗期与野生型相比根长明显变长,而在成熟期,osgh3.6tilling突变体在株高、千粒重与结实率与野生型相比都无明显变化。
本发明首先提供一种从水稻中克隆出的新的吲哚乙酸氨基化合成酶基因,记为osgh3.6,为具有特定序列的dna分子,其中开放阅读框为1809bp,其核苷酸序列为seqidno.1所示。
本发明还提供编码所述基因osgh3.6的蛋白质分子,该序列编码602个氨基酸残基,分子量65.25kda,等电点为7.24,氨基酸序列为seqidno.2所示。
本发明还提供用于调取获得水稻样品中基因osgh3.6的一对核苷酸引物。该引物根据基因osgh3.6设计,使用此对引物对水稻样品cdna进行pcr扩增可获得长1809bp的基因片段。具体的引物序列为:
forwardprimer:5'atggcgaccgccgccgccgacgagc3'(seqidno.3)
reverseprimer:5'ctagtgagcagctggttcagtgat3'(seqidno.4)。
本发明提供检测水稻基因osgh3.6在不同器官表达模式的方法,即利用所述基因osgh3.6的核苷酸序列作为设计探针引物的保守区段,调取其序列的引物序列:
forwardprimer:5'cactagcatctgtctcattgtgtca3'(seqidno.5)
reverseprimer:5'accttgtcagtgccggaatt3'(seqidno.6)。
对水稻cdna样品进行real-timepcr,然后检测该基因在茎、叶、根中的表达;样品为水稻的rna经过逆转录后所得cdna;其步骤如下:
(1)提取水稻器官的总rna(trizol,市售);
(2)利用反转录试剂盒(市售)将总rna反转录成cdna,根据seqidno.5和seqidno.6设计引物,根据seqidno.1,跨越orf区和3`utr的78bp作为pcr产物,进行实时定量pcr检测。
本发明还提供检测水稻基因osgh3.6在高盐、干旱胁迫以及激素处理下的表达模式变化的方法,即将水稻进行高盐、干旱胁迫以及激素处理后,提取水稻叶片中的rna;利用反转录试剂盒将rna反转录成cdna,利用引物seqidno.5与seqidno.6,进行定量pcr检测。其步骤如下:
(1)将两周大的水稻苗置于150mm氯化钠溶液中,28℃培养高盐胁迫处理0、2、4、8、12以及24h;干旱处理0、2、4、8、12以及24h;置于10μmiaa、10μmkt、10μm2,4-d、10μmnaa、10μm6-ba中,28℃分别培养0、2、4、8、12以及24h进行激素处理;
(2)提取前述个处理的水稻苗的叶和根中的总rna(trizol,市售);
(3)利用反转录试剂盒(市售)将总rna反转录成cdna,利用引物seqidno.5和seqidno.6,根据seqidno.1,跨越orf区和3`utr的77bp作为pcr产物,进行实时定量pcr检测。
本发明还提供检测水稻osgh3.6tilling突变体与野生型水稻在根长及产量改变的方法,其步骤如下:
(1)突变体株系与野生型的种子浸泡在水中24h,让种子充分吸涨。随后将种子玻璃培养皿内,放在水稻恒温培养箱内37℃催芽至露白,每天换水防止长霉。选取萌发一致的种子,将其转移至种子架上,置于基础营养液中,28℃培养箱培养;
(2)分别提取突变体以及野生型对照组中的总dna,利用引物进行实时荧光定量pcr检测,确定突变位置以及筛选纯合株系。对应的pcr引物序列为:
forwardprimer:5'attctgtctggggaagctatgtgaga3'(seqidno.7)
reverseprimer:5'ccagtcatccctctcgcagatt3'(seqidno.8);
(3)观察统计每天实验组与对照组的生长情况,并在一周时,观察并测量突变体株系与野生型株系的根长。
本发明还提供osgh3.6tilling突变体水稻与野生型水稻在成熟期株高及产量改变的方法,其步骤如下:
(1)突变体株系与野生型的种子浸泡在水中24h,让种子充分吸涨。随后将种子玻璃培养皿内,放在水稻恒温培养箱内37℃催芽至露白,每天换水防止长霉;
(2)将催芽后的种子均匀地散在苗床上,表面不能积水,种子播撒后,用手抹平,让种子陷入泥中,不应太深;
(3)育苗3-4周后,三叶期,根系发达方可移苗。过早会造成根部损伤,后期发育不良。移苗后1-2周可按每颗苗0.5g尿素少量施肥。浇水一般提前将水放在温室内1天温热后再浇;
(4)成熟期测量突变体与野生型水稻的株高,并统计千粒重与结实率。
本发明中,可选用本领域已经知道的各种载体,如市售的载体以及质粒。
可见,本发明提供的水稻基因osgh3.6可用于植物品种改良,如用于改善水稻抗高盐、干旱胁迫、激素胁迫的等性能,从而提高水稻产量。
附图说明
图1水稻osgh3.6的不同器官表达模式分析。其中,a为水稻根、茎、二叶、三叶、四叶、五叶、六叶的结构图;b为水稻二叶、三叶、四叶、五叶、六叶的代表图;c为各个器官中osgh3.6的表达量。
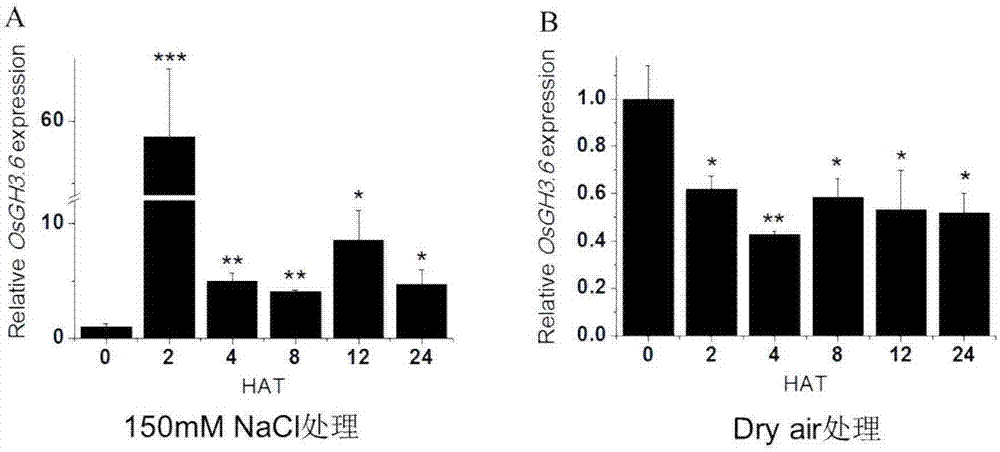
图2为水稻osgh3.6基因在高盐和干旱处理下的表达分析。其中,a是用150mmnacl处理0-24h后水稻的osgh3.6基因表达量变化;b是在干燥空气中处理0-24h后水稻的osgh3.6基因表达量变化。
图3为水稻osgh3.6基因在6-ba和kt处理下的表达分析。其中,a是10μmkt处理0-24h后水稻的osgh3.6基因表达量变化;b是10μm6-ba处理0-24h后水稻的osgh3.6基因表达量变化。
图4为水稻osgh3.6基因在iaa、2,4-d和naa处理下的表达分析。其中,a是10μmiaa处理0-24h后水稻的osgh3.6基因表达量变化;b是10μm2,4-d处理0-24h后水稻的osgh3.6基因表达量变化;c是10μmnaa处理0-24h后水稻的osgh3.6基因表达量变化。
图5为osgh3.6的两个突变体g19和g20与wt的osgh3.6dna序列(a,b)与蛋白质序列(c,d)的比对。
图6为生长一周osgh3.6tilling突变体与野生型水稻wt幼苗的根长分析。
图7为成熟期osgh3.6tilling突变体与野生型水稻wt的株高、千粒重与结实率分析。其中,a为osgh3.6tilling突变体与野生型表型;b为株高对比照相;c为千粒重统计;d为结实率对比。
具体实施方式
下面结合具体实施实例进一步阐释本发明。应理解,这些实例仅以用于说明本发明而不用于限制本发明的范围。下列实例中未注明具体的实验方法,均可按照常规方法进行。如sambrook等分子克隆:实验手册(newyork:coldspringharborlaboratorypress,1989)中所述条件,或按照制造生产厂商的使用说明。
实施例1,水稻基因osgh3.6的克隆
1.水稻品种nipponbare(oryzasativajaponica)在培养箱(spx-250-gb,shanghai,china)中培养:生长条件为光周期16h/8h(l/d),28℃;
2.rna提取。取100mg左右新鲜的水稻植物组织材料,液氮充分研磨。加1mltrizol试剂,涡旋15s后室温放置5min。加0.2ml氯仿,去蛋白,12000rpm离心10min后上清转移至新的离心管,加等体积异丙醇,充分混匀,室温放置10min,12krpm离心10min,弃上清,用depc处理过的水配制的75%乙醇1ml洗涤沉淀,重复一次。室温干燥5-10min,溶于20μldepc水中,测od值,电泳检测;
3.基因的克隆。以逆转录的水稻cdna第一链为模板,利用正向引物和反向引物进行pcr,获得基因全长,具体序列信息参见seqidno.1。
实施例2,水稻osgh3.6基因器官表达模式分析
分别提取水稻茎、幼叶、老叶、根等中的总rna,利用反转录试剂盒将总rna反转录成cdna,利用引物seqidno.5和seqidno.6,进行实时荧光定量pcr检测(图1)。结果显示,osgh3.6基因在各个器官中组成型表达,但在不同部位叶片以及根中的表达都无显著差异,在茎中的表达量显著升高。
实施例,3水稻基因osgh3.6在干旱、高盐胁迫以及植物激素处理下的表达模式分析
对两周大的水稻幼苗分别进行10μmiaa、10μmkt、10μm2,4-d、10μmnaa、10μm6-ba、150mmnacl以及干旱(dryair)处理0、2、4、8、12以及24h,分别提取叶中的总rna,利用反转录试剂盒将总rna反转录成cdna,利用引物seqidno.5和seqidno.6,进行实时荧光定量pcr检测。结果显示,盐胁迫下(图2a),osgh3.6的表达显著升高,而干旱处理(图2b)后,osgh3.6的表达显著降低。在kt处理下,osgh3.6的表达量基本无变化(图3a),而6-ba处理后,osgh3.6的表达显著降低(图3b);iaa处理后,osgh3.6的表达显著降低,在2,4-d和naa处理后,osgh3.6的表达都出现了不同程度的升高(图4a,b,c)。
实施例4,osgh3.6tilling突变体的分子鉴定
分别提取突变体以及野生型对照组中的总dna,利用引物seqidno.7和seqidno.8,进行实时荧光定量pcr检测与测序(图5)。根据osgh3-6的突变体g19和g20分别与wt的gh3.6dna序列比对,结果发现:g19在452bp处,g突变为a;g20在452bp处,g突变为a(图5a,b)。以上均在osgh3-6的编码区发生了突变,为有义突变。osgh3-6的两个突变体g19和g20与wt的gh4蛋白(图5c,d)比对发现,均在第87处的组氨酸(h)突变为精氨酸(r)。
实施例5,osgh3.6tilling突变体水稻根长的改变
28℃培养一周大的osgh3.6tilling突变体与野生型wt对照组进行根长测量(图6)。结果显示,osgh3.6tilling突变体与野生型wt相比,其根长显著变长。
实施例6,osgh3.6tilling突变体水稻株高、千粒重与结实率的改变
对成熟期osgh3.6tilling突变体与野生型wt对照组进行株高测量(图7a)。结果显示,osgh3.6tilling突变体与野生型wt相比,其株高表型无显著变化(图7b)。对成熟期osgh3.6tilling突变体与野生型wt对照组进行千粒重(图7c)与结实率的统计(图7d)。结果显示,osgh3.6tilling突变体与野生型wt相比也都无显著变化。
参考文献
abels,oellerpw,theologisa.earlyauxin-inducedgenesencodeshort-livednuclearproteins[j].procnatlacadsci,1994,91:326-330.
couderty,perinc,courtoisb,etal.geneticcontrolofrootdevelopmentinrice,themodelcereal[j].trendsplantsci,2010,15:219-226.
dingxh,caoyl,huangll,etal.activationoftheindole-3-aceticacid-amidosynthetasegh3-8suppressesexpansinexpressionandpromotessalicylate-andjasmonate-independentbasalimmunityinrice[j].plantcell,2008,20(1):228-240.
domingoc,andrésf,tharreaud,etal.constitutiveexpressionofosgh3.1reducesauxincontentandenhancesdefenseresponseandresistancetoafungalpathogeninrice[j].molplantmicrobeinteract,2009,22(2):201-210.
duh,wun,fuj,wangs,lix,xiaoj,xiongl.agh3familymember,osgh3-2,modulatesauxinandabscisicacidlevelsanddifferentiallyaffectsdroughtandcoldtoleranceinrice[j].jexpbot,2012,63(18):6467-6480.
frimlj.auxintransport-shapingtheplant[j].plantbiology,2003,6:7-12.
inukaiy,sakamotot,ueguchi-tanakam,etal.crownrootless1,whichisessentialforcrownrootformationinrice,isatargetofanauxinresponsefactorinauxinsignaling[j].plantcell,2005,17(5):1387-1396.
kitomiy,inahashih,takehisah,etal.osiaa13-mediatedauxinsignalingisinvolvedinlateralrootinitiationinrice[j].plantsci,2012,190:116-122.
kitomiy,ogawaa,kitanoh,etal.crl4regulatescrownrootformationthroughauxintransportinrice[j].plantroot,2008,2:19-28.
ljungk,ostina,lioussannel,sandbeigg.developmentalregulationofindole-3-aceticacidturnoverinscotspineseedlings[j].plantphysiol,2001,125:464-475.
mukeshj,navneetk,akhileshk,etal.theauxin-responsivegh3genefamilyinrice(oryzasativa)[j].functintegrgenomics,2006,6:36-46.
nij,wanggh,zhuzx,etal.osiaa23-mediatedauxinsignalingdefinespostembryonicmaintenanceofqcinrice[j].plantj,2011,68(3):433-442.
ouelletf,overvoordepj,theologisa.iaa17/axr3:biochemicalinsightintoanauxinmutantphenotype[j].plantcell,2001,13:829-841.
rebouillata,dievarta,verdeiljl,etal.geneticofricerootdevelopment[j].rice,2009,2:15-34.
tiwarisb,hageng,guilfoyletj.aux/iaaproteinscontainapotenttranscriptionalrepressiondomain[j].plantcell,2004,16:533543.
wangxf,heff,maxx,etal.oscand1isrequiredforcrownrootemergenceinrice[j].molplant,2011,4(2):289-299.
xiak,wangr,oux,fangz,tianc,duanj,wangy,zhangm.ostir1andosafb2downregulationviaosmir393overexpressionleadstomoretillers,earlyfloweringandlesstolerancetosaltanddroughtinrice[j].plosone,2012,7(1):e30039.
zhuzx,liuy,liusj,etal.again-of-functionmutationinosiaa11affectslateralrootdevelopmentinrice[j].molplant,2012,5(1):154-161.
zhangsw,lich,caoj,zhangyc,zhangsq,xiayf,sundy,suny.alteredarchitectureandenhanceddroughttoleranceinriceviathedown-regulationofindole-3-aceticacidbytld1/osgh3.13activation[j].plantphysiol,2009,151(4):1889-1901.。
序列表
<110>复旦大学
<120>水稻吲哚乙酸氨基化合成酶基因osgh3.6的编码序列及其应用
<130>001
<160>8
<170>siposequencelisting1.0
<210>1
<211>1809
<212>dna
<213>oryzasativa
<400>1
atggcgaccgccgccgccgacgagctcgcgctgcggcggctggacgacgccacccgcgac60
gcccggaggcttcagctcgagaccctccgcgccatcctcgccgagaatgccggcgccgcc120
tacctccgccggtacatcccctccgacggcggcgcccaccacctcctccgctccaccgac180
ctcgccgcggcggccgacgaattccgcctcctcgtccccgtcacctcctacgacgactac240
gccgagtccatccgccgcgtcgcggatggcgacgctgcccccgacgagctctcgccgcgc300
cccctcctctgcttcttcctcagctctgggacgagctcgctgcggccgaagctgataccc360
tacttggattcgccgggggcgagggccgccaccgccgccgtgatgcaggccaactccgcc420
ctcgtcagaaggctgttccctccacggccggcggtgagcaaggcgctgtggttcttgtac480
gccggcgaggtcaggaagacgaagggtggatacgaggccatggcggcgacggcgtggggc540
atccggagcagcggcatccggggagcctcgccggtgatgtccgcctgcgtcagccccgcc600
gaggtcatcctcggcgccgaccaccagcagcagatgtactgccacctcctctgcggcctc660
cgccgatgggacgccgtcgactgcatccgcgcgccgtacgccgccgccctggcgcgcgcc720
ctccgcctcctccagtccaagtggcgtcaactctgcgatgacctcgagtgcgggacagtc780
tgcgccgacgtcgtcaccgacgcggcgatgagaggcgccgtccaggacggagtgctcgcc840
gggccatgccccgagctcgccggccgcgtccgccgaatctgcgagagggatgactggcgc900
ggcgtgctgcgccaactgtggccggacgcgaggtacatctcctgcgtcacgacggggacc960
atggagcagtacttcccggcgatcaagcacttcgccggcgaggcgttgccggtgctcgga1020
acggactacctcgcctccgagtgcgccatcggcatcaacctggagcgcacgtcgccgcct1080
gaggaaaccacctacgtgctcctccccagggcggcatacttcgagttcatcccgttcgac1140
atggacgccgccggccgcggcgccgccgcggcagagccggtggacatcgccggcgtggag1200
gccggcaagacgtacgagttggtggccaccacgttccgtgggctgtaccggtacaaagtc1260
ggcgacgtcgtgaagatcgccggcttccaccactcctcccctcggcttcagttcgtgacc1320
cgcgcgccgccgccgcaagaacacggcgaggtgctcaccgagagggacgtcatggccgcc1380
atggacaccttccagctcatgctaaaggacggcggcgaggttatcgaatttgcggcgttc1440
atcattgacggcgacggaggccagcggcggcggcgatgcgcgacgatcgccgtcgaggtg1500
agcaatggaagcaagctgctcgaccatgagaggtcagccgccttcctcaggcgatgcacc1560
gcgccgctcgagggctgcctcggcggagcgtacaggctgagcagagccaccggcgacgtc1620
gcgccgctggaggtcgccgtggtgaggcccggcacgttcgacaggctcgccgaggccgcg1680
atccgcggcggcgctccggcgaaccagtacaagccgcccaagatcgtcaggcaccggcac1740
ctcgtcgatgtgctgcaatcctccgtggtgtgcagcagcagctcaatcactgaaccagct1800
gctcactag1809
<210>2
<211>602
<212>prt
<213>oryzasativa
<400>2
metalathralaalaalaaspgluleualaleuargargleuaspasp
151015
alathrargaspalaargargleuglnleugluthrleuargalaile
202530
leualagluasnalaglyalaalatyrleuargargtyrileproser
354045
aspglyglyalahishisleuleuargserthraspleualaalaala
505560
alaaspglupheargleuleuvalprovalthrsertyraspasptyr
65707580
alagluserileargargvalalaaspglyaspalaalaproaspglu
859095
leuserproargproleuleucysphepheleuserserglythrser
100105110
serleuargprolysleuileprotyrleuaspserproglyalaarg
115120125
alaalathralaalavalmetglnalaasnseralaleuvalargarg
130135140
leupheproproargproalavalserlysalaleutrppheleutyr
145150155160
alaglygluvalarglysthrlysglyglytyrglualametalaala
165170175
thralatrpglyileargserserglyileargglyalaserproval
180185190
metseralacysvalserproalagluvalileleuglyalaasphis
195200205
glnglnglnmettyrcyshisleuleucysglyleuargargtrpasp
210215220
alavalaspcysileargalaprotyralaalaalaleualaargala
225230235240
leuargleuleuglnserlystrpargglnleucysaspaspleuglu
245250255
cysglythrvalcysalaaspvalvalthraspalaalametarggly
260265270
alavalglnaspglyvalleualaglyprocysprogluleualagly
275280285
argvalargargilecysgluargaspasptrpargglyvalleuarg
290295300
glnleutrpproaspalaargtyrilesercysvalthrthrglythr
305310315320
metgluglntyrpheproalailelyshisphealaglyglualaleu
325330335
provalleuglythrasptyrleualaserglucysalaileglyile
340345350
asnleugluargthrserproproglugluthrthrtyrvalleuleu
355360365
proargalaalatyrpheglupheilepropheaspmetaspalaala
370375380
glyargglyalaalaalaalagluprovalaspilealaglyvalglu
385390395400
alaglylysthrtyrgluleuvalalathrthrpheargglyleutyr
405410415
argtyrlysvalglyaspvalvallysilealaglyphehishisser
420425430
serproargleuglnphevalthrargalaproproproglngluhis
435440445
glygluvalleuthrgluargaspvalmetalaalametaspthrphe
450455460
glnleumetleulysaspglyglygluvalilegluphealaalaphe
465470475480
ileileaspglyaspglyglyglnargargargargcysalathrile
485490495
alavalgluvalserasnglyserlysleuleuasphisgluargser
500505510
alaalapheleuargargcysthralaproleugluglycysleugly
515520525
glyalatyrargleuserargalathrglyaspvalalaproleuglu
530535540
valalavalvalargproglythrpheaspargleualaglualaala
545550555560
ileargglyglyalaproalaasnglntyrlysproprolysileval
565570575
arghisarghisleuvalaspvalleuglnserservalvalcysser
580585590
serserserilethrgluproalaalahis
595600
<210>3
<211>25
<212>dna
<213>oryzasativa
<400>3
atggcgaccgccgccgccgacgagc25
<210>4
<211>24
<212>dna
<213>oryzasativa
<400>4
ctagtgagcagctggttcagtgat24
<210>5
<211>25
<212>dna
<213>oryzasativa
<400>5
cactagcatctgtctcattgtgtca25
<210>6
<211>20
<212>dna
<213>oryzasativa
<400>6
accttgtcagtgccggaatt20
<210>7
<211>26
<212>dna
<213>oryzasativa
<400>7
attctgtctggggaagctatgtgaga26
<210>8
<211>22
<212>dna
<213>oryzasativa
<400>8
ccagtcatccctctcgcagatt22
- 还没有人留言评论。精彩留言会获得点赞!