一种长链非编码RNA的高通量芯片处理及分析流程控制方法与流程
本发明涉及医学基因组学和计算生物学领域,具体涉及一种长链非编码RNA的高通量芯片数据处理及分析流程控制方法。
背景技术:
长链非编码RNA(long non-coding RNA,lncRNA)是一类转录本长度超过200nt、不编码蛋白的RNA。长链非编码RNA起初被认为是基因组转录的“噪音”,不具有生物学功能。然而,近年来的研究表明长链非编码RNA能在表观遗传、转录及转录后水平上调控基因表达,参与了X染色体沉默、基因组印记以及染色质修饰、转录激活、转录干扰、核内运输等多种重要的调控过程,与人类疾病的发生、发展和防治都有着密切联系。长链非编码RNA通常较长,具有mRNA样结构,有些具有poly(A)尾巴,有些没有poly(A)尾巴,分化过程中有动态的表达与不同的剪接方式,与编码基因相比,长链非编码RNA表达量更低。且具有组织特异性即不同组织之间的长链非编码RNA表达量不同和时空特异性即同一组织或器官的不同生长阶段,其中的长链非编码RNA表达量也会变化。长链非编码RNA可从染色质重塑、转录调控及转录后加工等多种层面实现对基因表达的调控,然而长链非编码RNA的功能太过多种多样,其作用机制又了解的太少,给科研工作者带来了挑战和困难,尤其是面对高通量大数据的时候。如何分析长链非编码RNA大数据,研究其潜在功能成为该领域目前急需解决的问题。
技术实现要素:
本发明的目的是提供一种长链非编码RNA高通量芯片数据处理及分析流程控制方法,以解决现有的技术对长链非编码RNA高通量芯片数据处理中的不准确性、以及不懂如何分析长链非编码RNA等问题。
为实现上述目的,本发明采用的技术方案为:
一种长链非编码RNA的高通量芯片处理及分析流程控制方法,包括如下步骤:
步骤1,自定义参数配置文件的生成:导入长链非编码RNA高通量原始芯片数据,经过信号值筛选和标准化得到理论上有效的长链非编码RNA,在此基础上进行生物信息学参数分析;
步骤2,输入步骤:用户根据需要,输入设定的各参数配置文件;
步骤3,分析步骤:根据上述步骤输入设定的参数配置文件,通过长链非编码RNA高通量数据处理流程模块生成对应的自动化分析流程;
步骤4,执行及输出步骤:执行上述步骤所生成的自动化分析流程,获得并输出长链非编码RNA分析结果报告。
优选的,所述的步骤1具体包括如下步骤:
步骤1.1,导入长链非编码RNA高通量芯片原始信号值文件;
步骤1.2,对上述步骤导入的长链非编码RNA高通量芯片原始信号文件进行质量分析并剔除低质量信号数据,获得经过筛选的信号数据;
步骤1.3,将上述步骤获得的经过筛选的数据进行前景值和背景值校正,得到消除噪音污染的长链非编码RNA信号数据;
步骤1.4,将上述步骤得到的信号数据进行标准化,并去除极值,得到理论上有效的长链非编码RNA表达值。
优选的,所述的步骤1.2中,所述低质量信号数据是指扫描微阵列芯片荧光强度作为RNA表达信号值且荧光强度小于30的数据,同一探针的重复信号数据采用中位数计算法取中位值作为该探针的表达值。
优选的,所述的步骤1.3中,使用针对Affymetrix芯片原理设计的Affy软件包中的MAS5或者RMA方法根据不同的芯片类型进行芯片数据预处理,不同的芯片类型是指单、双色通道;MAS5得到的数据是原始信号强度,RMA得到的是经过对数变换的信号值。
优选的,所述的步骤1.4中,使用limma软件包进行芯片间归一化,得到标准化的长链非编码RNA表达谱数据。
优选的,所述的步骤1中,生物信息学参数分析包括差异表达长链非编码RNA的筛选,长链非编码RNA的功能性分析和对长链非编码RNA的调控机制分析。
优选的,所述的差异表达长链非编码RNA的筛选包括输入指令选取1.5倍或者2倍的差异倍数,选用三个标准Benjamini–Hochberg方法、FDR方法或者Bonforroni方法校正P-value得到差异表达的长链非编码RNA。
优选的,所述的对长链非编码RNA的功能性分析包括长链非编码RNA和基因数据的共表达分析,基因本体分析,代谢通路分析,化学反应分析和调控网络的构建;
其中,所述的长链非编码RNA和基因数据的共表达分析采用Pearson相关系数法或Spearman相关系数法,相关系数>=0.9,Benjamini–Hochberg方法、FDR方法或者Bonforroni方法校正P-value;
所述的基因本体分析采用g:Profiler法从生物过程、分子功能和细胞组分三个成分进行注释和富集分析;
所述的代谢通路分析和化学反应分析采用g:Profiler法通过KEGG和Reactive数据库信息进行分析;
优选的,对长链非编码RNA的调控机制分析包括intergenic类型的长链非编码RNA的筛选,长链非编码RNA的microRNA结合位点预测,microRNA靶基因预测和竞争性内源RNA调控网络的构建;
其中,所述的intergenic类型的长链非编码RNA的筛选整合国际权威数据库RefSeq、UCSC、GENCODE、RNAdb、NRED和UCR数据库信息;
所述的长链非编码RNA的microRNA结合位点预测采用miRWalk和DIANA-lncbase数据库信息;
所述的microRNA靶基因预测采用miRWalk和TargetScan数据库信息。
有益效果:利用本发明,将长链非编码RNA各分析步骤模块分和流程分,能够单独运行一个模块或流程中的局部分析模块,并进行模块内规定数据分析流程的快速执行。从而通过不同模块的选取,帮助科研人员迅速完成一套高通量数据的前期数据质控、功能分析和结果报告。该工具能够优化生物信息分析人员和科研人员的工作时间,显著提高工作效率,降低科研成本。本发明的分析流程思路清晰,其实现方法简单,可广泛应用于生物学研究工作中,也可用于临床相关应用。
本发明的方法首先由系统生成自定义参数配置文件,再根据用户设定参数后的自定义参数文件和高通量数据处理流程模块生成与数据流程对应的批处理可执行文件;由系统执行批处理可执行文件,实现数据流程自动化,最终生成结果报告文件。从而能高效的帮助生物信息分析人员完成一套标准化的高通量数据分析流程,甚至可以让非生物信息专业的科研人员独立完成高通量数据分析。达到优化科研人员的工作效率,降低科研成本的目的。本发明提出了一种新颖且可靠的长链非编码RNA高通量数据分析方法,在任何物种中通用,其实现方法简单,应用范围广泛。
附图说明
图1是长链非编码RNA自动化分析流程;
图2是长链非编码RNA生物信息学分析步骤;
图3是长链非编码RNA-共表达基因网络示意图;
图4是长链非编码RNA生物通路富集调控示意图;
图5是长链非编码RNA作为竞争性内源RNA的调控示意图。
具体实施方式
下面结合附图对本发明作更进一步的说明。
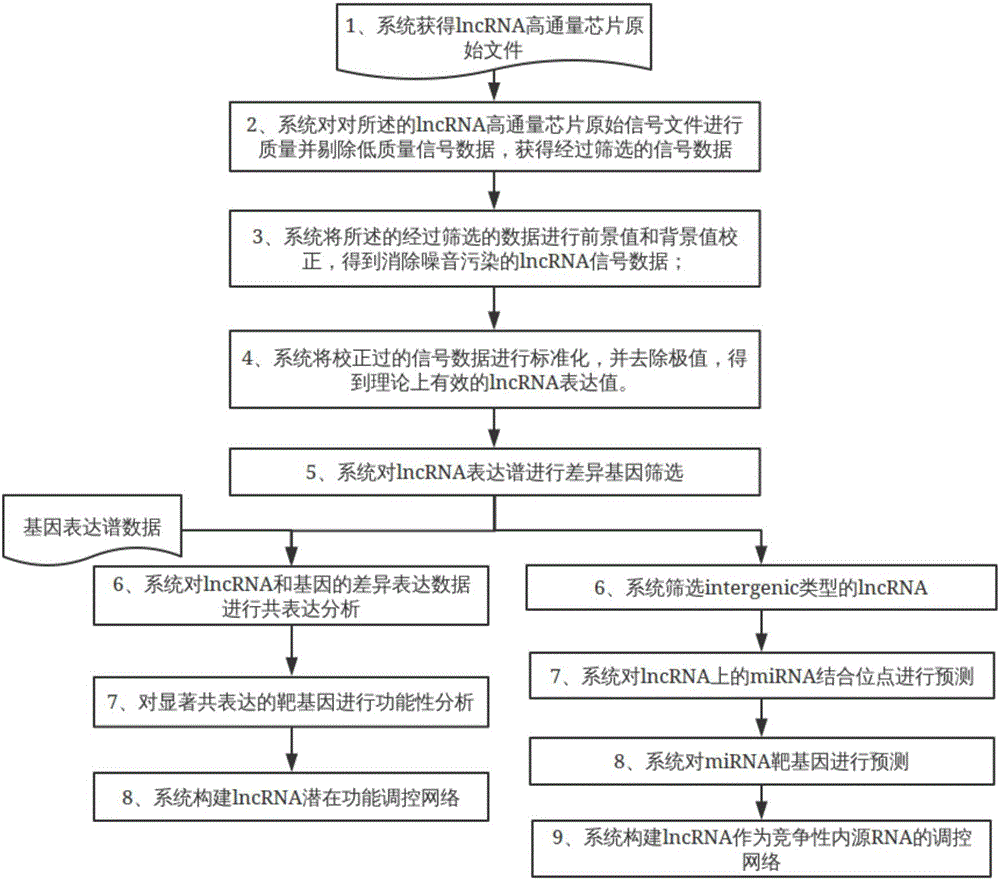
如图1所示,本发明的一种长链非编码RNA的高通量芯片处理及分析流程控制方法,包括如下步骤:
步骤1,自定义参数配置文件的生成:导入长链非编码RNA高通量原始芯片数据,经过信号值筛选和标准化得到理论上有效的长链非编码RNA,在此基础上进行生物信息学参数分析;
步骤2,输入步骤:用户根据需要,输入设定的各参数配置文件;
步骤3,分析步骤:根据上述步骤输入设定的参数配置文件,通过长链非编码RNA高通量数据处理流程模块生成对应的自动化分析流程;
步骤4,执行及输出步骤:执行上述步骤所生成的自动化分析流程,获得并输出长链非编码RNA分析结果报告。
其中,步骤1具体包括如下步骤:
步骤1.1,导入长链非编码RNA高通量芯片原始信号值文件;
步骤1.2,对上述步骤导入的长链非编码RNA高通量芯片原始信号文件进行质量分析并剔除低质量信号数据,获得经过筛选的信号数据;其中,低质量信号数据是指扫描微阵列芯片荧光强度作为RNA表达信号值且荧光强度小于30的数据,同一探针的重复信号数据采用中位数计算法取中位值作为该探针的表达值;
步骤1.3,将上述步骤获得的经过筛选的数据进行前景值和背景值校正,得到消除噪音污染的长链非编码RNA信号数据;其中,使用针对全球销量第一的Affymetrix芯片原理设计的Affy软件包中的MAS5或者RMA方法根据不同的芯片类型进行芯片数据预处理,不同的芯片类型是指单、双色通道;MAS5得到的数据是原始信号强度,RMA得到的是经过对数变换的信号值;
步骤1.4,将上述步骤得到的信号数据进行标准化,并去除极值,得到理论上有效的长链非编码RNA表达值;其中,使用目前芯片处理最通用的limma软件包进行芯片间归一化,得到标准化的长链非编码RNA表达谱数据。
如图2所示,步骤1中,生物信息学参数分析包括差异表达长链非编码RNA的筛选,长链非编码RNA的功能性分析和对长链非编码RNA的调控机制分析。
其中,差异表达长链非编码RNA的筛选包括输入指令选取1.5倍或者2倍的差异倍数(Fold change),选用国际最通用的三个标准Benjamini–Hochberg方法、FDR方法或者Bonforroni方法校正P-value得到差异表达的长链非编码RNA。
长链非编码RNA的功能性分析包括长链非编码RNA和基因数据的共表达分析,基因本体分析,代谢通路分析,化学反应分析和调控网络的构建。
对长链非编码RNA的调控机制分析包括intergenic类型的长链非编码RNA的筛选,长链非编码RNA的microRNA结合位点预测,microRNA靶基因预测和竞争性内源RNA调控网络的构建。
在本发明的一个实施方案中,在R平台,使用limma软件包的linear model线性拟合数据,通过经验Bayes t test得到差异表达的长链非编码RNA结果。
linear model是limma软件的线性模型算法,用来分析实验以及评估差异表达。
E[yj]=Xαj
上式中,Yj表示gene J的表达值;X是实验设计矩阵;Αj是系数向量。
经验Bayes t test检验是检验样本平均数与总体平均数的离差统计量。
上式中,为样本平均数;μ为总体平均数;N为样本容量;σx为样本标准差。
在本发明的一个实施方案中,在R平台,对差异长链非编码RNA的结果进行错误发现率矫正。可以采用Benjamini–Hochberg,FDR和Bonferroni方法。
Benjamini–Hochberg方法
上式中,α是给定的显著性阀值;K代表样本容量;M代表从小到大的排列顺序。
FDR方法
上式中,M0代表零假设是真的时候的样本总数;M代表样本容量;Q为显著性阀值。
Bonferroni方法
P=α/k
上式中,α是给定的显著性阀值;K是样本容量。
在本发明的一个实施方案中,在R平台,对长链非编码RNA的表达谱数据和基因表达谱数据进行共表达分析,可以使用Pearson和Spearman两种算法进行计算。相关系数>=0.9,选用Benjamini–Hochberg方法、FDR或者Bonforroni方法校正P-value。
Pearson相关系数是用来衡量两个数据集合是否在一条线上面,它用来衡量定距变量间的线性关系。
上式中,Z:代表正态分布中,数据偏离中心点的距离;等于变量减掉平均数再除以标准差;N为样本容量。
Spearman相关系数对原始变量分布不作要求,属于非参数统计方法。
rs=1-6∑(Xi-Yi)2/n(n2-1)
上式中,Xi和Yi分别为两个变量按大小排位的等级;n为样本容量。
在本发明的一个实施方案中,在R平台,与长链非编码RNA显著共表达的基因采用g:Profiler法从生物过程、分子功能和细胞组分三个成分进行基因本体注释和富集分析,差异显著可以用Benjamini–Hochberg和Bonferroni。
在本发明的一个实施方案中,在R平台,采用g:Profiler法整合KEGG和Reactive数据库信息对与长链非编码RNA显著共表达的基因进行代谢通路和化学反应分析,差异显著可以用Benjamini–Hochberg和Bonferroni方法。
在本发明的一个实施方案中,因为长链非编码RNA和共表达的基因具有相近的功能,在得到基因的基因本体、代谢或者反应信息确定其功能后,将两者结合,生成含有这些信息的网络文件。可以用Cytoscape软件打开,图形化展示长链非编码RNA潜在功能调控网络。
在本发明的一个实施方案中,对intergenic类型的长链非编码RNA的筛选整合了国际权威数据库NCBI的RefSeq、UCSC、GENCODE和NRED等数据库信息。
RefSeq数据库
RefSeq数据库,即RefSeq参考序列数据库,美国国家生物信息技术中心(NCBI)提供的具有生物意义上的非冗余的基因和蛋白质序列。
UCSC数据库
UCSC Genome Browser是由University of California Santa Cruz(UCSC)创立和维护的,该站点包含有人类、小鼠和大鼠等多个物种的基因组草图,并提供一系列的网页分析工具。站点用户可以通过它可靠和迅速地浏览基因组的任何一部分,并且同时可以得到与该部分有关的基因组注释信息,如已知基因,预测基因,表达序列标签,信使RNA,CpG岛,克隆组装间隙和重叠,染色体带型,小鼠同源性等。用户也可以因为教育或科研目的加上他们自己的注释信息。UCSC Genome Browser目前应用相当广泛,比如Ensembl就是使用它的人类基因组序列草图为基础的。
GENCODE数据库
国家人类基因组研究所(NHGRI)发起了一项公众研究ENCODE——the Encyclopedia Of DNA Elements,DNA元素百科全书。自2003九月起,开展识别所有在人类基因组序列的功能元素的项目。Wellcome Trust Sanger研究所进行基因注释功能集成放大的GENCODE项目。
NRED数据库
NRED数据库提供人和小鼠的长链非编码RNA在芯片数据的表达信息。
在本发明的一个实施方案中,对长链非编码RNA序列的miRNA结合位点预测采用miRWalk和DIANA-LncBase数据库信息进行预测,并取其交集。
miRWalk数据库
miRWalk是一个综合性数据库,不仅提供来自人类、小鼠和大鼠的长链非编码RNA的预测信息和经过验证的位于其靶基因上的结位点,也提供mRNA的预测信息和验证信息,共整合了13个公共数据库资源。
DIANA-LncBase数据库
DIANA-LncBase保存了全转录组实验验证的和计算预测的人类和小鼠lncRNAs上的miRNA识别元件(miRNA recognition elements,MREs)。其分析包括了大量的lncRNA资源的整合,相关高通量HITS-CLIP和PAR-CLIP实验数据,以及最新的计算靶预测。其中有效的实验支持条目超过了5000对相互作用,计算预测的相互作用超过1000万对。DIANA-LncBase保存了每个miRNA-lncRNA对的详细信息,例如外部链接,转录本基因组位置的图形绘制,结合位点的表征,lncRNA组织表达以及MREs的保守性得分和预测得分。
在本发明的一个实施方案中,对microRNA靶基因预测采用miRWalk和TargetScan数据库信息进行预测,并取其交集。
TargetScan数据库
TargetScan是由长链非编码RNA领域大牛Bartel实验室开发的数据库。基于靶mRNA序列的进化保守等特征搜寻动物的长链非编码RNA靶基因。是预测长链非编码RNA靶标假阳性率较低的数据库。
在本发明的一个实施方案中,得到的intergenic类型长链非编码RNA、长链非编码RNA通过结合位点吸附的microRNA以及microRNA调控的靶基因数据,构建长链非编码RNA作为竞争性内源RNA的调控网络,生成含有所有信息的文件。可以用Cytoscape软件打开,图形化展示长链非编码RNA的竞争性内源RNA调控网络。
以下结合具体实施例对上述方案做进一步说明。应理解,这些实施例是用于说明本发明而不是限制本发明的范围。实施例中采用的实施条件可以根据具体应用要求的条件做进一步调整,未注明的实施条件通常为常规实验中的条件。
实施例
首先对原始数据进行过滤处理,然后去除低质量信号和噪音污染的数据,经过标准化后得到有效的长链非编码RNA表达值。基于长链非编码RNA分析结果,可以基于其序列特征,进行靶基因预测;也可以结合共表达的基因表达谱对其进行功能预测。长链非编码RNA-共表达基因网络如图3所示。在上述分析的基础上,可进行一系列的统计学和可视化分析。
1.长链非编码RNA原始信号文件如表1所示
分析平台:R平台
分析软件:Affy,limma
表1
列名解释:
2.长链非编码RNA芯片表达结果如表2所示
分析平台:R平台
分析软件:limma,sva
表2
列名解释:
3.差异表达的长链非编码RNA结果如表3所示
分析平台:R平台
分析软件:limma,openxlsx
表3
列名解释:
4.长链非编码RNA和基因的相关系数如表4所示
分析平台:R平台
分析方法:Pearson,Spearman
表4
列名解释:
5.基因功能分析
为了得到与长链非编码RNA显著共表达的基因的功能,通过g:Profiler对其从生物过程、分子功能和细胞组成进行基因本体分析,代谢通路分析和化学反应分析。
分析平台:R平台
分析软件:g:Profiler
结果如表5-9所示,生物通路富集调控示意图如图4所示。
表5生物通路富集分析
列名解释
表6分子功能富集分析
列名解释:
表7细胞组分富集分析
列名解释:
表8 KEGG代谢通路富集分析
列名解释:
表9 Reactive化学反应富集分析
列名解释:
6.长链非编码RNA潜在功能调控网络的构建
分析平台:R平台
图形化软件:Cytoscape
7.长链非编码RNA类型筛选
通过整合多个国际权威数据库NCBI的RefSeq、UCSC、GENCODE和NRE等对长链非编码RNA的注释信息,筛选出intergenic类型的长链非编码RNA。
结果如表10所示:
表10多数据库长链非编码RNA注释信息
列名解释
8.长链非编码RNA结合的microRNA靶基因预测
对长链非编码RNA结合的miRNA靶基因预测采用miRWalk和TargetScan数据库信息进行预测,并取其交集。
结果如表11-13所示:
表11 miRWalk预测结果
列名解释:
表12 miRWalk验证结果
列名解释:
表13 TargetScan结果
列名解释:
9.长链非编码RNA作为竞争性内源RNA的调控网络构建
将得到的intergenic类型长链非编码RNA、长链非编码RNA通过结合位点吸附的microRNA以及microRNA调控的靶基因数据,构建长链非编码RNA作为竞争性内源RNA的调控网络,生成含有所有信息的文件。
图形化软件:Cytoscape,长链非编码RNA作为竞争性内源RNA的调控示意图如图5所示。
以上显示和描述了本发明的基本原理、主要特征和本发明的优点。本行业的技术人员应该了解,本发明不受上述实例的限制,上述实例和说明书中描述的只是说明本发明的原理,在不脱离本发明精神和范围的前提下本发明还会有各种变化和改进,这些变化和改进都落入要求保护的本发明范围内。本发明要求保护范围由所附的权利要求书及其等同物界定。
- 还没有人留言评论。精彩留言会获得点赞!
- 一种构建Hi‑C高通量测序文库的方法与制造工艺
- 桑葚病原菌高通量鉴定及种属分类方法及其应用与制造工艺
- 基于高通量测序技术检测乳腺癌/卵巢癌易感基因的多重PCR特异性引物、试剂盒及方法与制造工艺
- 基于高通量测序检测人EGFR基因变异的质控方法及试剂盒与制造工艺
- 基于高通量测序技术的小麦病原微生物检测方法及其应用与制造工艺
- 一种基于二代高通量测序技术的地中海贫血症的基因检测试剂盒的制造方法与工艺
- 一种基于集群的高通量数据分析方法与制造工艺
- 高通量双折射干涉成像光谱仪装置及其成像方法与制造工艺
- 高通量测序检测人循环肿瘤DNA EGFR基因的捕获探针及试剂盒的制造方法与工艺
- 一种高通量测序检测无创亲子鉴定的方法与制造工艺