一种ZNF667蛋白的RNA干扰质粒及其应用方法与流程
本发明属于肿瘤分子生物学的
技术领域:
,具体涉及ZNF667蛋白的短发夹RNA干扰质粒及其应用方法。
背景技术:
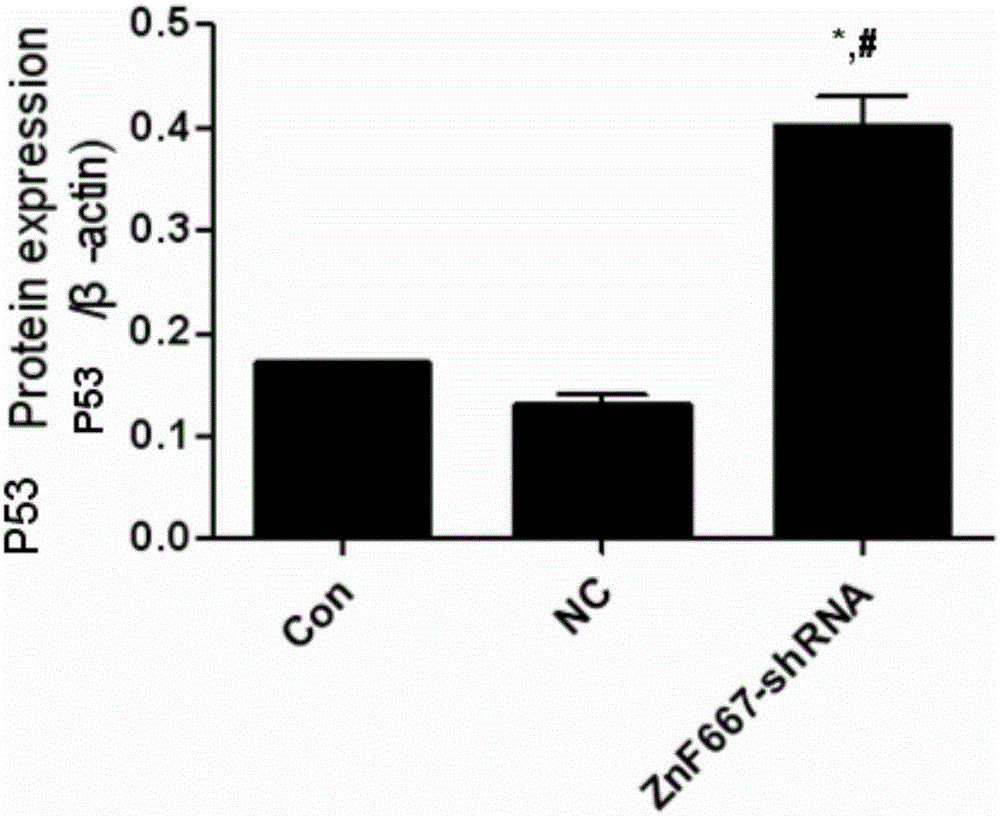
:ZNF667是在大鼠心肌缺血预适应中发现的一种表达上调的基因,通过与基因库的比对发现没有与之对应的序列,它是一个全新的基因,将其命名为ZNF667,Genbank登录号为AY221750。通过生物信息学分析,发现与之对应的蛋白是一种KRAB型锌指蛋白,含有14个锌指结构,具有转录调控的作用。ZNF667的ORF为1827bp,编码608个氨基酸。目前对于ZNF667相关功能研究结果如下:(1)ZNF667是一种新发现的KRAB型锌指蛋白,能抑制SRE和AP-1的转录活性,具有转录抑制作用;(2)ZNF667定位在细胞核内,主要在心脏和脑组织中表达,在肝、肾、骨骼肌、睾丸中表达次之,在其他组织表达则较少;(3)ZNF667基因在缺血、缺氧、炎症、氧化应激条件下表达增多,具有典型的应激反应性;(4)ZNF667在应激状态下促进细胞生长;(5)ZNF667具有抗心肌细胞凋亡作用,H2O2处理H9C2心肌细胞再瞬时转染ZNF667后能明显的抑制Fas等蛋白的表达,且这种抗凋亡作用与直接和Fas启动子结合而抑制其表达有关;ZNF667能抑制TNF-ɑ所致的细胞凋亡,且这种抗凋亡作用也与直接和Bid启动子结合而抑制其表达有关。(6)ZNF667过表达后可以通过抑制Bax和P53的表达。有关ZNF667的功能学研究表明:脑星形细胞瘤病理级别的升高,ZNF667mRNA及蛋白的含量呈上升趋势,尤其是在恶性程度高的Ⅲ、Ⅳ级脑星形细胞瘤中,含量明显升高。但是ZNF667在其他癌中的表达,运用ZNF667短发夹RNA干扰质粒对癌症进行治疗,尚没有任何研究。本发明通过使用ZNF667发夹干扰RNA(shRNA)质粒,转染HepG2肝癌细胞系,建立ZNF667稳定削减的肝癌细胞系,通过研究发现,HepG2-ZNF667-shRNA细胞系表现为细胞增殖减慢,克隆形成率减低,浸润与侵袭能力下降。裸鼠成瘤实验证实,抑制HepG2细胞中ZNF667的表达,可以从在体水平抑制HepG2细胞成瘤。本发明从细胞功能学水平和在体动物实验水平证实了削减HepG2肝癌细胞中的ZNF667蛋白的表达,可以抑制HepG2细胞的恶性度,起到治疗作用。技术实现要素:本发明的目的是通过构建一种ZNF667蛋白的发夹短发夹RNA干扰质粒,探讨削减ZNF667蛋白对HepG2细胞的生物学特性的影响,进而将这种质粒用于抑制肝癌的治疗。一种ZNF667蛋白的短发夹RNA干扰质粒,根据ZNF667的序列设计能够产生干扰ZNF667蛋白表达的siRNA的DNA片段,插入到短发夹RNA干扰载体中构建而成。上述质粒以人ZNF667的mRNA序列为模板,以及载体启动子启动表达siRNA对序列的要求,设计合成能够产生干扰ZNF667蛋白表达的siRNA的DNA片段。siRNA干扰的目标基因ZNF667序列如下:siRNA-1:CGAATATCTCTCACACGACAT;siRNA-2:CGCCAATCATTTCTTATTGAA;siRNA-3:CAACCCTTATTCTGCATCTAA。优选为siRNA-2:CGCCAATCATTTCTTATTGAA上述质粒选用的原始质粒表达载体优选为pRNAT-U6.1/Neo。DNA片段插入到载体时的酶切位点为BamHI和HindIII。根据ZNF667的序列设计能够产生干扰ZNF667蛋白表达的siRNA的DNA片段,具体序列如下:ZNF667-shRNA-1a链序列:5’-GATCCCCGAATATCTCTCACACGACATCTCGAGATGTCGTGTGAGAGATATTCGTTTTTGGAT-3’ZNF667-shRNA-1b链序列:5’-AGCTATCCAAAAACGAATATCTCTCACACGACATCTCGAGATGTCGTGTGAGAGATATTCGGG-3’ZNF667-shRNA-2a链序列:5’-GATCCCCGCCAATCATTTCTTATTGAACTCGAGTTCAATAAGAAATGATTGGCGTTTTTGGAT-3’ZNF667-shRNA-2b链序列:5’-AGCTATCCAAAAAGCCAATCATTTCTTATTGAACTCGAGTTCAATAAGAAATGATTGGCGGG-3’ZNF667-shRNA-3a链序列:5’-GATCCCCAACCCTTATTCTGCATCTAACTCGAGTTAGATGCAGAATAAGGGTTGTTTTTGGAT-3’ZNF667-shRNA-3b链序列:5’-AGCTATCCAAAAACAACCCTTATTCTGCATCTAACTCGAGTTAGATGCAGAATAAGGGTTGGG-3’。对照shRNAa链序列:5’-GATCCCTTCTCCGAACGTGTCACGTCTCGAGACGTGACACGTTCGGAGAATTTTTGGAT-3’对照shRNAb链序列:5’-AGCTATCCAAAAATTCTCCGAACGTGTCACGTCTCGAGACGTGACACGTTCGGAGAAGG-3’。构建的三个ZNF667短发夹干扰质粒载体的序列见SEQNO:17-19。优选见SEQNO:18。所述的ZNF667蛋白的短发夹RNA干扰质粒的应用方法,用于制备抑制肝癌的制剂。本发明通过在线生物信息学软件选择设计ZNF667的干扰序列,按照RNA干扰的原则构建ZNF667的短发夹RNA干扰质粒。质粒的构建及筛选含有ZNF667的siRNA序列的短发夹RNA干扰质粒采用氨苄青霉素(ampicilin,Amp)抗性基因做为筛选标志。采用新霉素(G418)筛选方法构建稳定转染ZNF667-shRNA和其空白对照质粒的HEPG2细胞株。MTT法检测转染干扰质粒ZNF667-shRNA,转染空白对照质粒,空白细胞的三组HepG2细胞株的增殖,克隆形成实验监测三组细胞株的克隆形成能力。划痕试验检测三组细胞的迁移能力,Transwell实验检测其浸润能力。裸鼠成瘤实验从在体层次检验三组细胞的恶性程度,免疫印迹检测靶点分子P53,Bcl-2的表达。结果是本发明成功的构建了ZNF667发夹干扰质粒(ZNF667-shRNA),并将其转染HepG2细胞,建立了稳转干扰质粒ZNF667-shRNA的细胞株。MTT实验数据表明,转染了ZNF667-shRNA的细胞株生长较HepG2,HepG2-ZNF667E(转染空白对照质粒)细胞显著减慢;细胞克隆形成能力较空白对照HepG2组和空白对照质粒组HepG2-ZNF667E组减弱;划痕实验表明:和HepG2空白细胞,HepG2-ZNF667E(即转空白对照质粒细胞株NC组)相比,转染了ZNF667-shRNA的细胞株的迁移距离在24小时,48小时明显降低;Transwell侵袭实验结果表明:转染了ZNF667-shRNA的细胞株细胞侵袭能力较HepG2组和HepG2-ZNF667E组减弱。免疫印迹结果显示:转染了ZNF667-shRNA的细胞中的P53的表达较HepG2,HepG2-ZNF667E组细胞株明显升高,Bcl-2的表达较HepG2,HepG2-ZNF667E细胞株明显降低。这为肝癌的治疗提供了新的途径和思路。附图说明图1为ZNF667-shRNA-1a链测序结果;图2为ZNF667-shRNA-2a链测序结果;图3为ZNF667-shRNA-3a链测序结果;图4为HepG2细胞转染空白对照质粒(NC)、ZNF667-shRNA1(Sh1)、ZNF667-shRNA2(Sh2)、ZNF667-shRNA3(Sh3)质粒后,以及空白细胞蛋白表达检测条带;图5为HepG2细胞转染空白对照质粒(NC)、ZNF667-shRNA1(Sh1)、ZNF667-shRNA2(Sh2)、ZNF667-shRNA3(Sh3)质粒后,以及空白细胞蛋白表达检测削减效率;*和空白细胞相比,&和空白对照质粒组相比;图6为稳定转染ZNF667-shRNA,NC(空白对照质粒)和Con(空白细胞)的HepG2细胞中ZNF667蛋白进行Westernblot检测结果;图7为稳定转染ZNF667-shRNA,NC(空白对照质粒)和Con(空白细胞)的HepG2细胞中ZNF667蛋白表达的统计学分析;*和空白细胞相比,&和空白对照质粒组相比;图8为HepG2细胞转染ZNF667-shRNA质粒、空白对照质粒(NC)后和Con(空白细胞)生长曲线图;*和Con(空白细胞)相比,#和NC(空白对照质粒)组相比;图9为HepG2细胞转染ZNF667-shRNA质粒、空白对照质粒(NC)后和Con(空白细胞)细胞克隆形成图;图10为HepG2细胞转染ZNF667-shRNA质粒、空白对照质粒(NC)后和Con(空白细胞)细胞克隆的统计学分析;图11为HepG2细胞转染ZNF667-shRNA质粒、空白对照质粒(NC)后和Con(空白细胞)的TransWell实验结果;图12为HepG2细胞转染ZNF667-shRNA质粒、空白对照质粒(NC)后和Con(空白细胞)TransWell实验的统计学分析结果;*和Con(空白细胞)相比,&和NC(空白对照质粒)组相比;图13为HepG2细胞转染ZNF667-shRNA质粒、空白对照质粒(NC)后和Con(空白细胞)的细胞划痕实验结果;图14为HepG2细胞转染ZNF667-shRNA质粒、空白对照质粒(NC)后和Con(空白细胞)的细胞划痕实验的统计学分析结果;*和Con(空白细胞)相比,#和NC(空白对照质粒)组相比;图15为Westernblot检测HepG2细胞转染ZNF667-shRNA质粒、空白对照质粒(NC)后和Con(空白细胞)中P53和Bcl2、ZNF667蛋白表达;图16为Westernblot检测HepG2细胞转染ZNF667-shRNA质粒、空白对照质粒(NC)后和Con(空白细胞)中Bcl2蛋白表达分析结果;*和Con(空白细胞)相比,#和NC(空白对照质粒)组相比;图17为Westernblot检测HepG2细胞转染ZNF667-shRNA质粒、空白对照质粒(NC)后和Con(空白细胞)中P53蛋白表达分析结果;*和Con(空白细胞)相比,#和NC(空白对照质粒)组相比;图18为HepG2细胞转染ZNF667-shRNA质粒、空白对照质粒(NC)后和HepG2(空白细胞)三组细胞裸鼠成瘤实物体积和生长曲线图;图19为HepG2细胞转染ZNF667-shRNA质粒、空白对照质粒(NC)后和HepG2(空白细胞)三组细胞裸鼠成瘤瘤体体积及重量统计学分析;图20为HepG2细胞转染ZNF667-shRNA质粒、空白对照质粒(NC)后和HepG2(空白细胞)三组细胞裸鼠成瘤,瘤体中ZNF667蛋白的含量检测及统计学分析。具体实施方式以下结合具体实施方式旨在进一步说明本发明,而非限制本发明。材料和方法:材料:人肝癌细胞株HepG2购自中南大学细胞分子中心,正常生长培基为1640+10%FBS,细胞呈长梭形,贴壁生长。质粒表达载体pRNAT-U6.1/Neo,全长6380bp(见序列表中SEQNO.21),包含绿色荧光蛋白(Geenfluorescentprotein,GFP)做为检识标记,氨苄青霉素(ampicilin,Amp)抗性基因做为原核筛选标志,用来筛选已经成功把shRNA片段克隆入pRNAT-U6.1/Neo质粒,新霉素(neomycin,又被称为G418)抗性基因做为真核筛选标志,用于筛选稳定转染了ZNF667-shRNA质粒的细胞。克隆酶切位点BamHI和HindIII。保证插入片段的方向正确,防止载体自体连接。此载体购于上海吉凯基因化学技术有限公司。方法:1.ZNF667干扰位点的选择及寡脱氧核苷酸的设计在NCBI数据库中查找人ZNF667的mRNA完整序列,以此为模板,同时参考siRNA设计原则。根据干扰载体pRNAT-U6.1/Neo启动子启动表达siRNA对序列的要求,利用在线siRNATatgetfinder软件(http://genomics.jp/sidirect/)设计合成3条针对人ZNF667序列的siRNA的DNA片段。在线输入ZNF667mRNA1序列(NM_022103.3)分析获得的序列,选择GC含量在40%~55%之间的靶基因序列作为潜在优选,并在GenBank表达序列标签(EsT)数据库中用BLAsT检索,将选定的序列和相应的基因组数据库进行比较,排除和其他编码序列同源的序列,以确定其为特异性序列。为了提高序列正确退火成为双链DNA分子的效率,将包含能够产生干扰目的基因表达的siRNA序列的DNA按照以下原则设计为a链和b链寡核苷酸序列,在a链和b链序列加入间隔序列CTCGAG,以避免形成终止信号,使其能形成发夹结构,a链模板的5'端添加了GATCCC,b链模板的3’端添加了GG,与BamHI切后形成的粘性末端互补,a链3’端添加了TTTTTGGAT,b链模板的5'端添加了AGCTATCCAAAAA,与HindIII酶切后形成的粘性末端互补。针对ZNF667的3个siRNA的a链和b链由上海吉凯基因有限公司合成。作为ZNF667的短发夹RNA干扰质粒的空白对照质粒的shRNA序列(shRNA-NC)的a链和b链由上海吉凯基因化学技术有限公司合成。2.ZNF667siRNA干扰片段合成将合成的八条Oligo片段先稀释成100μM,两两配对各吸5μL混在一管中,混匀后置于PCR仪中95℃,5min,室温静置20min,形成双链Oligo片段。然后再将双链OligoDNA片段稀释成10μM,用于插入到pRNAT-U6.1/Neo双酶切产生的载体酶切片段中。表1构建的干扰质粒序列3.shRNA载体的构建使用BamHI和HindIII对pRNAT-U6.1/Neo载体进行酶切消化,将消化后的产物置于37℃,4h,经0.8%琼脂糖凝胶电泳分离,胶回收试剂盒回收纯化大片段备用。按Takara公司说明书,将载体与插入子按照一定的比例匹配,设置连接反应,16℃反应过夜。将连接产物转化至感受态的大肠杆菌DH5α菌种中,氨苄青霉素筛选,挑取阳性克隆,摇菌、小提质粒,构建的3个ZNF667shRNA真核载体分别命名为ZNF667-shRNA1,ZNF667-shRNA2,ZNF667-shRNA3,无意对照shRNA真核载体被命名NC。构建的载体还需后继进行测序,进行验证。4.shRNA载体的鉴定抽提质粒后送上海申工公司测序。5.干扰质粒的削减效率的鉴定将1×105个HepG2细胞接种至已用0.1%明胶包被过的6孔板中,铺板48小时后转染。细胞转染共分成5组:空白细胞组,转染三个shRNA组和空白对照shRNA组,细胞转染按照LipofectaminTM2000说明书操作,所有细胞均放在37℃,5%CO2恒温培养箱中进行培养,48小时后收取总蛋白。通过检测ZNF667蛋白的表达来确定3个发夹短发夹RNA干扰质粒的削减效率。最终定下2号质粒ZNF667-shRNA2为最佳备选质粒。6.稳定转染ZNF667-shRNA2载体的HepG2细胞株的建立①细胞准备培养HepG2细胞至80~90%满瓶,消化收集细胞,以1×105个/mL的细胞密度接种2个6孔细胞培养板,每孔接种2mL细胞悬液,每株细胞接种1孔,37℃、5%CO2细胞培养箱培养24小时至细胞80%满孔时开始转染。②质粒转染种板后次日,各取4μg质粒(将空白载体编号为ZNF667-E、上述的2号质粒编号为ZNF667-AS2)分别用0.25mL无血清培养基稀释,充分混匀5min。取20μL(10μL×2)lipo2000转染试剂,用0.5mL(0.25mL×2)无血清培养基稀释,充分混匀5min。6孔板细胞弃完全培养基,用无血清培养基洗涤2次,同时补加0.5mL无血清培养基。分别取0.25mL两种质粒悬液加入转染试剂的2个离心管中,混匀。室温静置20min,并在30min内加入六孔板中。③细胞筛选HepG2细胞G418预筛选:培养HepG2细胞至80~90%满瓶,消化收集细胞,以1×104个/ml的细胞密度接种24孔板中,调整G418浓度为0、100、200、300、400、500、600、700、800、900、1000g/ml加入24孔中,观察细胞。最终挑选600μg/mL,为G418筛选浓度。稳株筛选:将上述已经转染ZNF667-E、ZNF667-AS2的HepG2细胞标记为HepG2-ZNF667-E、HepG2-ZNF667-AS2。转染48h后,加入600μg/mL的G418到六孔板中,观察细胞,每两天换液,7天后HepG2组细胞基本死亡,调整G418浓度为200μg/mL为维持浓度,继续培养,扩增细胞:10天培养后HepG2组细胞全部死亡,其他组(即转染了ZNF667-E和ZNF667-AS2的HepG2细胞组)降低G418浓度至200μg/mL,维持200μg/mL的G418浓度扩增细胞至满瓶。7.细胞增殖能力检测①MTT法分别收集各组对数期HepG2稳定株细胞,调整细胞悬液浓度;取3块96孔板,每孔加入100μL细胞悬液,铺板使待测细胞为2×103cell/孔(边缘孔用无菌PBS填充以消除边缘效应),每组设置5个复孔;5%CO2,37℃培养箱培养,分别于24h,48h,72h后取出一块96孔板检测:检测时每孔加入10μLMTT溶液(5mg/ml,即0.5%MTT)溶液,细胞培养箱继续培养4h,终止培养;每孔加入150ul二甲基亚砜,置摇床上低速振荡10min,使结晶物充分溶解。用酶标仪检测各孔波长490nm的吸光值。②克隆形成实验取对数生长期的各组HepG2细胞(共3组),用胰酶消化液消化并用1640完全培养基制成单细胞悬液,使单个细胞百分率在95%以上。细胞计数,并用含10%FBS的1640培养基调整细胞浓度至1×104个/ml备用;接种35mm细胞培养皿,200cells/孔,每组细胞重复种3个孔,以确保实验数据的可靠性。补足3ml完全培养基,充分混匀后置于5%CO2,37℃培养箱培养。经常观察,当培养皿中出现肉眼可见的克隆时,终止培养。固定与染色:弃去上清液,用PBS小心浸洗2次。每孔加2mL4%多聚甲醛固定细胞15min。去固定液,加1mlGIMSA染色液染色20min,流水缓慢冲洗去除多余染色液,空气干燥。计数克隆:将平板倒置并叠加一张带网格的透明胶片,用肉眼直接计数克隆数量。结果计算采用如下公式:克隆形成率=(克隆数/接种细胞数)×100%。8.浸润功能检测(Transwell侵袭实验)分别消化收集各组HepG2细胞,用无血清培养基重悬细胞,调整细胞密度为1×106cells/ml。吸掉已充分浸润基底层膜后的侵袭小室内的培养基;加500μL的含有10%FBS的培养基至下室(侵袭小室外,24孔板孔内);加200μL已调节好细胞密度的细胞悬液至侵袭小室内;37℃、5%CO2细胞培养箱孵育24h;用棉签轻柔擦拭以去除侵袭小室内未侵过基底层膜的细胞,以及基底层(ECMatrixgel),小心操作以免刺穿底层聚碳酸酯膜;在新的24孔板孔中加500μL染色液,分别将侵袭小室浸入染色20min;将侵袭小室在有水的烧杯中浸没几次冲洗掉多余的染料,然后在空气中干燥;通过显微镜随机取不同视野拍照采集;然后用10%的乙酸溶解膜上的染料,500μL/孔,再转至96孔板中(150μL/孔),检测570nm波长下各孔OD值。9.迁移能力检测(划痕实验)分别消化收集各组HepG2细胞,用含10%FBS的培养基将各组细胞配成单个细胞悬液,以每孔3×105个细胞接种到6孔板;待细胞铺满孔底,用20μL的tip头垂直板面划痕,用无血清培养基洗涤2次,将脱落下来的细胞洗去;用无血清培养基培养细胞,24h后更换成含3%FBS培养基继续培养;以划痕时间为起点,每隔24h拍摄各组细胞图片。10.裸鼠移植瘤动物模型制备①9只裸鼠随机数字表法分为3组:HepG2细胞,ZNF667-NC组,ZNF667-shRNA组;②将处于对数生长期癌细胞培养液丢弃,用无菌中性PBS洗涤细胞2次,用0.25%的胰蛋白酶消化,从培养瓶洗脱下,吹打成单细胞悬液,1000转/min离心smin,收集细胞,用0.9%的生理盐水悬浮细胞,定容,并将细胞密度调至注射的细胞量为2×106个细胞/ml.③对超净台紫外灯照射30分钟,75%酒精擦拭台面;用75%酒精擦培养笼,其它用具均高压灭菌处理。注射细胞悬液前用75%酒精局部皮肤消毒裸鼠接种处皮肤,每只裸鼠接种0.2ml细胞悬液,用无菌一次性注射器分别接种于裸鼠颈背部或腋下皮下。④接种后每3天密切观察成瘤情况及肿瘤的生长情况。裸鼠致瘤成功后每5天测量肿瘤体积并记录,绘制各组种植瘤生长曲线。第42日实验结束,以脱颈法处死所有裸鼠,解剖取出肿瘤组织,测量肿瘤最长径和横径,称取肿瘤质量。用公式分别计算肿瘤体积。计算公式如下:肿瘤体积=肿瘤长径×肿瘤短径2×0.5。结果:1.ZNF667-shRNA质粒的鉴定挑选阳性菌落,抽提质粒,以T7为测序引物,对构建载体进行序列通测,通过测序峰图进行核对分析,证实插入片段为FoxA1shRNADNA片段,且无碱基突变(图1-图3),说明shRNA载体构建成功,符合预期设计,能够表达siRNA,预期能对ZNF667蛋白的表达予以消减。2.ZNF667-shRNA对HepG2细胞中ZNF667蛋白表达的抑制作用Westernblotting检测转染sh短发夹RNA干扰质粒后HepG2细胞ZNF667蛋白表达量的变化,结果发现:在HepG2细胞中,收集空白质粒对照组、ZNF667-con组(空白细胞组)和ZNF667-shRNA1(转染1号质粒)、ZNF667-shRNA2(转染2号质粒)、ZNF667-shRNA3(转染3号质粒)总共5组的标化细胞总蛋白进行检测,结果显示ZNF667-shRNA1、ZNF667-shRNA2、ZNF667-shRNA3组蛋白表达条带比空白细胞组(Con)和空白质粒(NC)组条带明显减弱(图4),统计学分析证实这三组质粒在HepG2细胞的干扰效果都具有显著性差异(图5),ZNF667-shRNA2的干扰效率是最佳的,被确定为可用于后继实验的干扰质粒。3.稳定转染ZNF667-shRNA2质粒及对照质粒的HepG2细胞株的建立与鉴定按照前述稳转细胞株建立的方法,筛选HepG2稳转细胞株的最佳G418浓度为600ug/ml,获得稳转细胞株后,维持培养浓度为200ug/ml。最终建立的稳转细胞株被命名为ZNF667-shRNA(HepG2细胞转染ZNF667-shRNA2质粒组),NC(空白对照质粒组)。收集转染了ZNF667-shRNA,NC和空白细胞蛋白,进行Westernblot检测,将胶片进行扫描,用图像分析软件IPP6.0对图像进行灰度分析(图6)。统计学分析结果表明(图7),ZNF667-shRNA2质粒在稳定转入HepG2细胞中后,能够削减90%的蛋白表达。转染了ZNF667-shRNA,NC和空白细胞中ZNF667的表达差异具有统计学意义。4.ZNF667-shRNA细胞增殖能力减弱采用基于3-(4,5)-dimethylthiahiazo(-z-y1)-3,5-di-phenytetrazoliumromide(化学名:3-(4,5-二甲基噻唑-2)-2,5-二苯基四氮唑溴盐)的细胞计数试剂盒检测三株细胞的增殖能力。使用酶标仪在490mM波长处测定OD值,活细胞线粒体中的琥珀酸脱氢酶能使外源性MTT还原为水不溶性的蓝紫色结晶甲臢并沉积在细胞中,而死细胞无此功能。用酶标仪在490nm波长处测定其光吸收值,在一定细胞数范围内,MTT结晶形成的量与细胞数成正比。从而间接地反应细胞的增殖。0小时,24小时,48小时与72小时490mM波长处的OD值见表1,曲线分析表明:HepG2细胞转染空白对照质粒ZNF667-NC后,稳定株细胞生长较HepG2细胞稍有减慢;HepG2细胞转染ZNF667-shRNA2质粒后,细胞株生长较HepG2-NC和空白细胞显著减慢(图8),差异具有统计学意义。表1HepG2各组稳定株细胞MTT不同时间点数据5.稳转ZNF667-shRNA(2号质粒)的HepG2细胞株克隆形成能力降低克隆形成试验是测定细胞增殖能力的有效方法之一,克隆形成率反映细胞群体依赖性和增殖能力两个重要性状。HepG2细胞转染质粒ZNF667-shRNA后,ZNF667-shRNA组细胞克隆形成能力较HepG2组减弱,较空白对照质粒组NC减弱(图9,表2)。克隆形成的差异具有统计学意义(图10)。表2.HepG2各组克隆形成率数据6.稳转ZNF667-shRNA(2号质粒)的HepG2细胞株浸润与迁移能力降低采用划痕试验与transwell侵袭试验检测ZNF667的表达改变是否能够影响HepG2细胞的侵袭能力。Transwell侵袭试验实验结果采用醋酸洗脱液在酶标仪上570nm的OD值来间接代表穿过Transwell小室的细胞数,结果表明:转染了ZNF667-shRNA的细胞株侵袭能力下降,具体表现为570nm波长时吸光值下降了25%(图11,表3)。实验结果表明:ZNF667-shRNA组细胞侵袭能力较HepG2组减弱,较空白对照质粒组NC组减弱,且差异具有统计学意义(图12)。表3HepG2三组细胞Transwell侵袭实验结果ConNCZNF667-shRNAOD1.233±0.0261.228±0.0280.814±0.029划痕实验(Scratchingtest)是一种用于检测细胞运动的实验方法,该实验可用于检测贴壁生长肿瘤细胞的侵袭转移能力。在全部三组细胞细胞铺满孔底后,使用10ul枪头垂直板面划痕,以划痕时间为起点,每隔24h拍摄各组细胞图片,和HepG2细胞,NC组稳转细胞株相比,HepG2-ZNF667-shRNA细胞株的迁移距离在24小时,48小时明显降低(表4,图13),且差异具有统计学意义(图14)。表4.HepG2三组细胞划痕实验迁移距离(mm)7HepG2三株细胞中的P53,Bcl-2蛋白的表达为了进一步探讨削减ZNF667蛋白表达是如何引起HepG2细胞生长,增殖,浸润及侵袭能力的改变的,选择了Bcl2,P53两个蛋白为靶点分子进行检测,结果表明,和HepG2细胞及NC细胞株相比,HepG2-ZNF667-shRNA细胞株中的P53和Bcl2两个蛋白表达有明显改变。Bcl2蛋白的表达明显降低,p53蛋白的表达明显升高(图15),图16和17为Bcl2蛋白和P53蛋白表达分析结果;结果表明,削减HepG2三个细胞株中的ZNF667蛋白极有可能是通过降低Bcl2,升高p53的表达来达到改变HepG2细胞的生物学特性,从而从体外细胞水平表现出削减其恶性程度的。8.裸鼠成瘤实验将HepG2细胞及HepG2-NC细胞株,HepG2-ZNF667-shRNA细胞株进行裸鼠成瘤实验。第42天处死裸鼠,老鼠的成瘤体积以及生长曲线见图18.瘤体体积及重量统计学分析见图19,三组细胞裸鼠成瘤,瘤体中ZNF667蛋白的含量检测及统计学分析结果见图20。SEQUENCELISTING<110>中南大学湘雅二医院<120>一种ZNF667蛋白的RNA干扰质粒及其应用方法<130>无<160>21<170>PatentInversion3.3<210>1<211>21<212>DNA<213>siRNA-1<400>1cgaatatctctcacacgacat21<210>2<211>22<212>DNA<213>siRNA-2<400>2cgccaatcatttcttattgaac22<210>3<211>21<212>DNA<213>siRNA-3<400>3caacccttattctgcatctaa21<210>4<211>63<212>DNA<213>ZNF667-shRNA-1a链序列<400>4gatccccgaatatctctcacacgacatctcgagatgtcgtgtgagagatattcgtttttg60gat63<210>5<211>63<212>DNA<213>ZNF667-shRNA-1b链序列<400>5agctatccaaaaacgaatatctctcacacgacatctcgagatgtcgtgtgagagatattc60ggg63<210>6<211>64<212>DNA<213>ZNF667-shRNA-2a链序列<400>6gatccccgccaatcatttcttattgaactcgagttcaataagaaatgattggcgtttttg60gat63<210>7<211>62<212>DNA<213>ZNF667-shRNA-2b链序列<400>7agctatccaaaaagccaatcatttcttattgaactcgagttcaataagaaatgattggcg60gg62<210>8<211>63<212>DNA<213>ZNF667-shRNA-3a链序列<400>8gatccccaacccttattctgcatctaactcgagttagatgcagaataagggttgtttttg60gat63<210>9<211>63<212>DNA<213>ZNF667-shRNA-3b链序列<400>9agctatccaaaaacaacccttattctgcatctaactcgagttagatgcagaataagggtt60ggg63<210>10<211>59<212>DNA<213>对照shRNAa链序列<400>10gatcccttctccgaacgtgtcacgtctcgagacgtgacacgttcggagaatttttggat59<210>11<211>59<212>DNA<213>对照shRNAb链序列<400>11agctatccaaaaattctccgaacgtgtcacgtctcgagacgtgacacgttcggagaagg59<210>12<211>6<212>DNA<213>a链和b链序列加入间隔序列<400>12ctcgag6<210>13<211>6<212>DNA<213>a链模板的5'端添加的序列<400>13gatccc6<210>14<211>2<212>DNA<213>b链模板的3'端添加的序列<400>14gg2<210>15<211>9<212>DNA<213>a链3'端添加的序列<400>15tttttggat9<210>16<211>13<212>DNA<213>b链模板的5'端添加的序列<400>16agctatccaaaaa13<210>17<211>938<212>DNA<213>质粒shRNA1<400>17tccggatctcctaagggaggagagcatgaattctcgagcggccgcccccttcaccgaggg60cctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataat120tggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagta180ataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgct240taccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaa300caccggcgaatatctctcacacgacatctcgagatgtcgtgtgagagatattcgttttta360attctcgacctcgagacaaatgcagtattcatccacaagcttaagtttaaaccgctgatc420agcctcgactgtgccttctaaatagtaatcaattacggggtcattagttcatagcccata480tatggagttccgcgttacataacttacggtaaatggcccgcctggctgaccgcccaacga540cccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggacttt600ccattgacgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatcaagt660gtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggcccgcctggca720ttatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtattagt780catcgctattaccatggtgatgcggttttggcagtacatcaatgggcgtggatagcggtt840tgactcacggggatttccaagtctccaccccattgacgtcaatgggagtttgttttggca900ccaaaatcaacgggactttccaaaatgtcgtaacaact938<210>18<211>1008<212>DNA<213>质粒shRNA2<400>18tttccctcacttaaggggaggagaagcatgaattctcgagcggccgcccccttcaccgag60ggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagata120attggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaag180taataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatg240cttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacga300aacaccggcgccaatcatttcttattgaactcgagttcaataagaaatgattggcgtttt360taattctcgacctcgagacaaatggcagtattcatccacaagcttaagtttaaaccgctg420atcagcctcgactgtgccttctaaatagtaatcaattacggggtcattagttcatagccc480atatatggagttccgcgttacataacttacggtaaatggcccgcctggctgaccgcccaa540cgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggac600tttccattgacgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatca660agtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggcccgcctg720gcattatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtatt780agtcatcgctattaccatggtgatgcggttttggcagtacatcatgggcgtggatagcgg840tttgactcacggggatttccaagtctccaccccattgacgtcaatggagtttgttttggc900accaaatcaccggactttccaaaatgtcgtacactccgccccattgacgcaaatgggcgg960tagccgtgtacggtgggagttctatataagtccgagctggttagtgac1008<210>19<211>1017<212>DNA<213>质粒shRNA3<400>19tcccgtcactaagggaggagagcatgaattctcgagcggccgcccccttcaccgagggcc60tatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattg120gaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaat180aatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgctta240ccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaaca300ccggcaacccttattctgcatctaactcgagttagatgcagaataagggttgtttttaat360tctcgacctcgagacaaatggcagtattcatccacaagcttaagtttaaaccgctgatca420gcctcgactgtgccttctaaatagtaatcaattacggggtcattagttcatagcccatat480atggagttccgcgttacataacttacggtaaatggcccgcctggctgaccgcccaacgac540ccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggactttc600cattgacgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatcaagtg660tatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcat720tatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtattagtc780atcgctattaccatggtgatgcggttttggcagtacatcaatgggcgtggatagcggttt840gactcacggggatttccaagtctccaccccattgacgtcaatgggagtttgttttggcac900caaaatcaacgggactttccaaaatgtcgtacaactccgccccattgacgcaatggggcg960gtaggcggggtacggtgggaggtctaatataaggcagaagctggtttagtgaaccgt1017<210>20<211>1010<212>DNA<213>con-shRNA<400>20tacctcactaagggaggagaagcatgaattccccagtggaaagacgcgcaggcaaaacgc60accacgtgacggagcgtgaccgcgcgccgagcgcgcgccaaggtcgggcaggaagagggc120ctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataatt180agaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaa240taatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgctt300accgtaacttgaaagtatttcgatttcttgggtttatatatcttgtggaaaggacgcggg360atcccttctccgaacgtgtcacgtctcgagacgtgacacgttcggagaatttttggatag420cttaagtttaaaccgctgatcagcctcgactgtgccttctaaatagtaatcaattacggg480gtcattagttcatagcccatatatggagttccgcgttacataacttacggtaaatggccc540gcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgtatgttcccat600agtaacgccaatagggactttccattgacgtcaatgggtggagtatttacggtaaactgc660ccacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatga720cggtaaatggcccgcctggcattatgcccagtacatgaccttatgggactttcctacttg780gcagtacatctacgtattagtcatcgctattaccatggtgatgcgggttttggcagtaca840tcaatgggcgtggatagcggtttgactcacggggatttccaagtctccaccccattgacg900tcaatgggagtttgttttggcaccaaaatcaacgggactttccaaaatgtcgtacaactc960cgccccattgacgcaaatggggcggtaggcgtgtacggtgggaggtctat1010<210>21<211>6380<212>DNA<213>pRNAT-U6.1/neo载体<400>21aatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttgggtttatatat60cttgtggaaaggacgcgggatccgagctcggtaccaagcttaagtttaaaccgctgatca120gcctcgactgtgccttctaaatagtaatcaattacggggtcattagttcatagcccatat180atggagttccgcgttacataacttacggtaaatggcccgcctggctgaccgcccaacgac240ccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggactttc300cattgacgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatcaagtg360tatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcat420tatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtattagtc480atcgctattaccatggtgatgcggttttggcagtacatcaatgggcgtggatagcggttt540gactcacggggatttccaagtctccaccccattgacgtcaatgggagtttgttttggcac600caaaatcaacgggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggc660ggtaggcgtgtacggtgggaggtctatataagcagagctggtttagtgaaccgtcagatc720cgctagcgctaccggtcgccaccatggccagcaagggcgaggagctgttcaccggcgtgg780tgcccatcctggtggagctggacggcgatgtgaatggccacaagttcagcgtgagcggcg840agggcgagggcgatgccacctacggcaagctgaccctgaagttcatctgcaccaccggca900agctgcctgtgccctggcccaccctggtgaccaccctgagctacggcgtgcagtgcttct960cacgctaccccgatcacatgaagcagcacgacttcttcaagagcgccatgcctgagggct1020acatccaggagcgcaccatcttcttcgaggatgacggcaactacaagtcgcgcgccgagg1080tgaagttcgagggcgataccctggtgaatcgcatcgagctgaccggcaccgatttcaagg1140aggatggcaacatcctgggcaataagatggagtacaactacaacgcccacaatgtgtaca1200tcatgaccgacaaggccaagaatggcatcaaggtgaacttcaagatccgccacaacatcg1260aggatggcagcgtgcagctggccgaccactaccagcagaatacccccatcggcgatggcc1320ctgtgctgctgcccgataaccactacctgtccacccagagcgccctgtccaaggacccca1380acgagaagcgcgatcacatgatcctgctggagttcgtgaccgccgccggcatcacccacg1440gcatggacgagctgtacaagtgaggactagataactgaacttgtttattgcagcttataa1500tggttacaaataaagcaatagcatcacaaatttcacaaataaagcatttttttcactgca1560ttctagttgtggtttgtccaaactcatcaatgtatcttatcatgtctgtgggaagacaat1620agcaggcatgctggggatgcggtgggctctatggcttctgaggcggaaagaaccagctgg1680ggctctagggggtatccccacgcgccctgtagcggcgcattaagcgcggcgggtgtggtg1740gttacgcgcagcgtgaccgctacacttgccagcgccctagcgcccgctcctttcgctttc1800ttcccttcctttctcgccacgttcgccggctttccccgtcaagctctaaatcgggggctc1860cctttagggttccgatttagtgctttacggcacctcgaccccaaaaaacttgattagggt1920gatggttcacgtagtgggccatcgccctgatagacggtttttcgccctttgacgttggag1980tccacgttctttaatagtggactcttgttccaaactggaacaacactcaaccctatctcg2040gtctattcttttgatttataagggattttgccgatttcggcctattggttaaaaaatgag2100ctgatttaacaaaaatttaacgcgaattaattctgtggaatgtgtgtcagttagggtgtg2160gaaagtccccaggctccccagcaggcagaagtatgcaaagcatgcatctcaattagtcag2220caaccaggtgtggaaagtccccaggctccccagcaggcagaagtatgcaaagcatgcatc2280tcaattagtcagcaaccatagtcccgcccctaactccgcccatcccgcccctaactccgc2340ccagttccgcccattctccgccccatggctgactaattttttttatttatgcagaggccg2400aggccgcctctgcctctgagctattccagaagtagtgaggaggcttttttggaggcctag2460gcttttgcaaaaagctcccgggagcttgtatatccattttcggatctgatcaagagacag2520gatgaggatcgtttcgcatgattgaacaagatggattgcacgcaggttctccggccgctt2580gggtggagaggctattcggctatgactgggcacaacagacaatcggctgctctgatgccg2640ccgtgttccggctgtcagcgcaggggcgcccggttctttttgtcaagaccgacctgtccg2700gtgccctgaatgaactgcaggacgaggcagcgcggctatcgtggctggccacgacgggcg2760ttccttgcgcagctgtgctcgacgttgtcactgaagcgggaagggactggctgctattgg2820gcgaagtgccggggcaggatctcctgtcatctcaccttgctcctgccgagaaagtatcca2880tcatggctgatgcaatgcggcggctgcatacgcttgatccggctacctgcccattcgacc2940accaagcgaaacatcgcatcgagcgagcacgtactcggatggaagccggtcttgtcgatc3000aggatgatctggacgaagagcatcaggggctcgcgccagccgaactgttcgccaggctca3060aggcgcgcatgcccgacggcgaggatctcgtcgtgacccatggcgatgcctgcttgccga3120atatcatggtggaaaatggccgcttttctggattcatcgactgtggccggctgggtgtgg3180cggaccgctatcaggacatagcgttggctacccgtgatattgctgaagagcttggcggcg3240aatgggctgaccgcttcctcgtgctttacggtatcgccgctcccgattcgcagcgcatcg3300ccttctatcgccttcttgacgagttcttctgagcgggactctggggttcgaaatgaccga3360ccaagcgacgcccaacctgccatcacgagatttcgattccaccgccgccttctatgaaag3420gttgggcttcggaatcgttttccgggacgccggctggatgatcctccagcgcggggatct3480catgctggagttcttcgcccaccccaacttgtttattgcagcttataatggttacaaata3540aagcaatagcatcacaaatttcacaaataaagcatttttttcactgcattctagttgtgg3600tttgtccaaactcatcaatgtatcttatcatgtctgtataccgtcgacctctagctagag3660cttggcgtaatcatggtcatagctgtttcctgtgtgaaattgttatccgctcacaattcc3720acacaacatacgagccggaagcataaagtgtaaagcctggggtgcctaatgagtgagcta3780actcacattaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgcca3840gctgcattaatgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgctcttc3900cgcttcctcgctcactgactcgctgcgctcggtcgttcggctgcggcgagcggtatcagc3960tcactcaaaggcggtaatacggttatccacagaatcaggggataacgcaggaaagaacat4020gtgagcaaaaggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgttttt4080ccataggctccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcg4140aaacccgacaggactataaagataccaggcgtttccccctggaagctccctcgtgcgctc4200tcctgttccgaccctgccgcttaccggatacctgtccgcctttctcccttcgggaagcgt4260ggcgctttctcatagctcacgctgtaggtatctcagttcggtgtaggtcgttcgctccaa4320gctgggctgtgtgcacgaaccccccgttcagcccgaccgctgcgccttatccggtaacta4380tcgtcttgagtccaacccggtaagacacgacttatcgccactggcagcagccactggtaa4440caggattagcagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcctaa4500ctacggctacactagaagaacagtatttggtatctgcgctctgctgaagccagttacctt4560cggaaaaagagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtttttt4620tgtttgcaagcagcagattacgcgcagaaaaaaaggatctcaagaagatcctttgatctt4680ttctacggggtctgacgctcagtggaacgaaaactcacgttaagggattttggtcatgag4740attatcaaaaaggatcttcacctagatccttttaaattaaaaatgaagttttaaatcaat4800ctaaagtatatatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacc4860tatctcagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagat4920aactacgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagaccc4980acgctcaccggctccagatttatcagcaataaaccagccagccggaagggccgagcgcag5040aagtggtcctgcaactttatccgcctccatccagtctattaattgttgccgggaagctag5100agtaagtagttcgccagttaatagtttgcgcaacgttgttgccattgctacaggcatcgt5160ggtgtcacgctcgtcgtttggtatggcttcattcagctccggttcccaacgatcaaggcg5220agttacatgatcccccatgttgtgcaaaaaagcggttagctccttcggtcctccgatcgt5280tgtcagaagtaagttggccgcagtgttatcactcatggttatggcagcactgcataattc5340tcttactgtcatgccatccgtaagatgcttttctgtgactggtgagtactcaaccaagtc5400attctgagaatagtgtatgcggcgaccgagttgctcttgcccggcgtcaatacgggataa5460taccgcgccacatagcagaactttaaaagtgctcatcattggaaaacgttcttcggggcg5520aaaactctcaaggatcttaccgctgttgagatccagttcgatgtaacccactcgtgcacc5580caactgatcttcagcatcttttactttcaccagcgtttctgggtgagcaaaaacaggaag5640gcaaaatgccgcaaaaaagggaataagggcgacacggaaatgttgaatactcatactctt5700cctttttcaatattattgaagcatttatcagggttattgtctcatgagcggatacatatt5760tgaatgtatttagaaaaataaacaaataggggttccgcgcacatttccccgaaaagtgcc5820acctgacgtcgacggatcgggagatctcccgatcccctatggtgcactctcagtacaatc5880tgctctgatgccgcatagttaagccagtatctgctccctgcttgtgtgttggaggtcgct5940gagtagtgcgcgagcaaaatttaagctacaacaaggcaaggcttgaccgacaattgcatg6000aagaatctgcttagggttaggcgttttgcgctgcttcgcgatgtacgggccagatatacg6060cgtttaatacgactcactatagggagagagagagaattaccctcactaaagggaggagaa6120gcatgaattccccagtggaaagacgcgcaggcaaaacgcaccacgtgacggagcgtgacc6180gcgcgccgagcgcgcgccaaggtcgggcaggaagagggcctatttcccatgattccttca6240tatttgcatatacgatacaaggctgttagagagataattagaattaatttgactgtaaac6300acaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgca6360gttttaaaattatgttttaa6380当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1