柠檬酸产量提高的重组霉菌的构建方法及应用与流程
本发明涉及生物工程技术领域,尤其涉及一种柠檬酸产量提高的重组霉菌的构建方法及应用。
背景技术:
黑曲霉是重要的工业生产菌,目前对黑曲霉进行代谢改造的研究集中于中心代谢通路和呼吸链的改造,包括糖酵解途径、TCA循环、rTCA循环的关键酶的单基因表达和基因协同表达,以及交替氧化酶的过量表达和敲除,但这些改造对柠檬酸产量的影响甚微,上述方法中只有增强rTCA循环途径促进了柠檬酸产量的提高。
在柠檬酸工业生产中,原料为淀粉,淀粉经过淀粉酶液化后过滤,得到清液和混液,通过清液和混液进行勾兑得到种子和发酵培养基,此时培养基中约一半的碳源为葡萄糖,另一半碳源以多聚葡萄糖的形式存在。而工业生产菌在发酵初期合成大量糖化酶,将多聚葡萄糖分解为葡萄糖,因此,在发酵前期培养基中碳源基本以葡萄糖的形式存在,所以柠檬酸发酵对碳源的吸收实际上是指对葡萄糖的吸收。
Torres测定了黑曲霉吸收葡萄糖存在2个Km值,分别为260μM和3.67mM,说明存在高亲和力和低亲和力两套转运系统,而且由低亲和力的转运系统提供柠檬酸发酵所需的代谢流,但该转运系统仅在葡萄糖浓度>50g·L-1时起作用。因葡萄糖的转运是柠檬酸发酵的第一个步骤,该转运系统对柠檬酸发酵有直接的影响,因此调整柠檬酸发酵生产过程中的葡萄糖转运系统,有可能增强柠檬酸的产量。
技术实现要素:
为解决上述技术问题,本发明的目的是提供一种柠檬酸产量提高的重组霉菌的构建方法及应用,通过启动高亲和力葡萄糖转运蛋白的表达,以增强葡萄糖在发酵后期的吸收,进而提高了霉菌中柠檬酸的发酵产量。
为实现上述目的,本发明采用了以下技术方案:
在一方面,本发明提供了一种柠檬酸产量提高的重组霉菌的构建方法,包括以下步骤:
(1)构建HGT1蛋白表达框,HGT1蛋白表达框包含组成型启动子、与组成型启动子连接的HGT1蛋白编码基因以及与HGT1蛋白编码基因连接的终止子;
(2)构建抗性基因表达框,抗性基因表达框包含组成型启动子、与组成型启动子连接的hph基因以及与hph基因连接的终止子;
(3)将步骤(1)HGT1蛋白表达框和步骤(2)得到的抗性基因表达框转化到霉菌中,得到重组霉菌。
进一步地,在步骤(1)中,HGT1蛋白编码基因来源于黑曲霉。
进一步地,在步骤(1)中,HGT1蛋白编码基因的核苷酸序列如SEQ ID NO.1所示。
进一步地,HGT1蛋白编码基因具有转运葡萄糖的功能,其编码的氨基酸序列可发生1个或多个氨基酸的取代、缺失或插入。
优选地,HGT1蛋白编码基因编码的氨基酸序列如SEQ ID NO.2所示。
进一步地,在步骤(1)中和步骤(2)中,组成型启动子为PgpdA启动子、gpdA启动子、Pmbf启动子或Pgla启动子。
进一步地,PgpdA启动子的序列如SEQ ID NO.3所示。
进一步地,在步骤(1)和步骤(2)中,终止子为trp终止子。
进一步地,trp终止子的序列如SEQ ID NO.4所示。
进一步地,在步骤(2)中,hph基因的序列如SEQ ID NO.5所示。
进一步地,在步骤(2)中,构建潮霉素抗性基因表达框,便于阳性克隆子的筛选。抗性基因表达框以pAN7-1为模板克隆得到。
进一步地,在步骤(3)中,霉菌为黑曲霉、米曲霉或构巢曲霉。
优选地,在步骤(3)中,霉菌为黑曲霉Aspergillus niger H915-1,米曲霉100-8或构巢曲霉FGSC A4。
进一步地,HGT1蛋白表达框的构建方法,包括以下步骤:
(1)将终止子序列酶切后,连接到pUC19质粒上,得到pUC-trp载体;
(2)将组成型启动子序列PgpdA酶切后,连接到pUC-trp载体上,得到pUC-PgpdA-trp载体;组成型启动子序列PgpdA还可以替换为gpdA启动子、Pmbf启动子或Pgla启动子;
(3)将HGT1序列双酶切后,片段连接到pUC-PgpdA-trp载体上,得到pUC-PgpdA-HGT1-trp表达框。
进一步地,转化方法包括以下步骤:
(1)将HGT1蛋白表达框和抗性基因表达框经PEG介导法转入霉菌原生质体中;
(2)在潮霉素抗性的高渗软琼脂PDA平板上培养得到阳性克隆,再将经培养4-7天的阳性克隆转移至含150-180mg/L潮霉素的平板上进一步培养得到阳性克隆,验证正确的即为重组霉菌。
由于霉菌在发酵后期通过表达高亲和力葡萄糖转运蛋白来吸收葡萄糖,基于这一研究基础,本发明实现了高亲和力葡萄糖转运蛋白HGT1在不同霉菌中的表达,以增强葡萄糖在发酵后期的吸收。
在另一方面,本发明提供了一种HGT1蛋白表达框,包括组成型启动子、与组成型启动子连接的HGT1蛋白编码基因以及与HGT1蛋白编码基因连接的终止子。
进一步地,组成型启动子为PgpdA启动子、gpdA启动子、Pmbf启动子或Pgla启动子。
进一步地,HGT1蛋白编码基因来源于黑曲霉,HGT1蛋白编码基因的核苷酸序列如SEQ ID NO.1所示。
进一步地,终止子为trp终止子。
在又一方面,本发明还提供了一种由上述方法构建的重组霉菌株。
本发明还提供了上述方法所构建的重组霉菌在发酵生产柠檬酸中的应用。
进一步地,本发明的重组霉菌发酵生产柠檬酸的条件为:在30-35℃的温度下发酵72-120h,优选转速为250r/min左右。
借由上述方案,本发明至少具有以下优点:
本发明利用组成型启动子启动HGT1蛋白在霉菌中表达,通过增强发酵后期葡萄糖的摄取,从而提高柠檬酸的产量;本发明方法对产柠檬酸的黑曲霉菌株具有普遍适用性,可使黑曲霉发酵生产柠檬酸产量提高近10%,利用Aspergillus niger H915-1作为宿主,柠檬酸产量提高了14.7%,发酵周期缩短了6h。
附图说明
图1是黑曲霉菌株中柠檬酸和葡萄糖的含量测试结果;
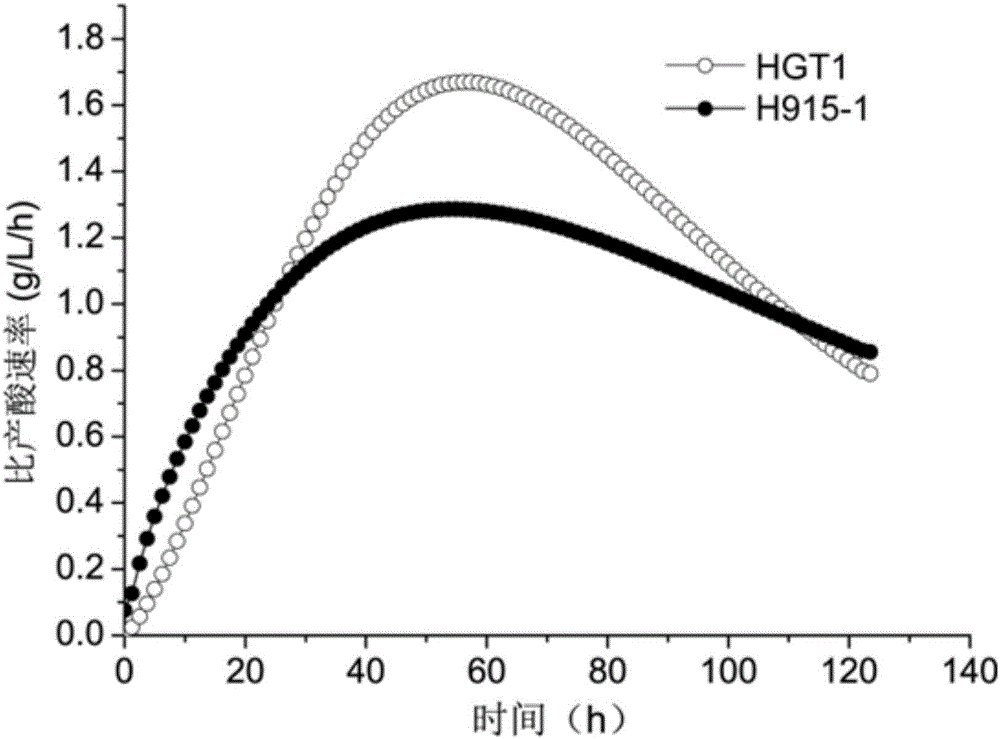
图2是发酵生产时,柠檬酸的比产酸速率测试结果。
具体实施方式
下面结合附图和实施例,对本发明的具体实施方式作进一步详细描述。以下实施例用于说明本发明,但不用来限制本发明的范围。
实施例1 黑曲霉RNA的提取
将黑曲霉孢子接种到柠檬酸发酵培养基中,35℃下250r/min培养48h,用mirocloth滤布收集菌球,再用无菌超纯水洗涤3遍,滤干水分后迅速在液氮中冷冻。用液氮研磨的方法将组织充分磨碎,采用QIAGEN公司RNeasy Plant Mini Kit试剂盒提取黑曲霉总RNA。用TAKARA公司PrimeScript RT reagent Kit with gDNA Eraser将RNA反转录成cDNA。
实施例2 黑曲霉基因组DNA的提取
将黑曲霉孢子接种到ME液体培养基(3%麦芽提取物,0.5%胰蛋白胨)中,35℃下250r/min培养48h,用mirocloth滤布收集菌球,再用无菌超纯水洗涤3遍,滤干水分后迅速在液氮中冷冻。用液氮研磨的方法将组织充分磨碎,采用QIAGEN公司DNeasy Plant Mini Kit提取丝状真菌基因组。
实施例3 HGT1蛋白表达框和潮霉素抗性表达框的构建
利用引物trp-F(序列如SEQ ID NO.6所示)和trp-R(序列如SEQ ID NO.7所示)以pAN7-1为模板扩增trp终止子,序列上下游含Pst I和Hin dIII酶切位点,连接到pMD19上测序,然后用这两个限制性内切酶酶切,将该序列连接到同样酶切的pUC19上,得到pUC19-trp。
利用引物Pgpd-F(序列如SEQ ID NO.8所示)和Pgpd-R(序列如SEQ ID NO.9所示)从pAN7-1中扩增PgpdA启动子,序列两头含Eco RI和Kpn I酶切位点,酶切后,将该序列连接到同样酶切的pUC19-trp,得到pUC-PgpdA-trp。
利用引物HGT1-F(序列如SEQ ID NO.10所示)和HGT1-R(序列如SEQ ID NO.11所示)从实施例1中得到的黑曲霉cDNA中扩增HGT1基因,基因两头含Kpn I和Pst I酶切位点,连接到同样酶切的pUC-PgpdA-trp上,形成PgpdA-HGT1-trp表达框。
潮霉素抗性表达框通过引物gpd-F(序列如SEQ ID NO.12所示)和Ttrp-R-2(序列如SEQ ID NO.13所示)从质粒pAN7-1中扩增得到。
本实施例中,扩增条件如下:94℃预变性3min,随后进行30次循环94℃20s、55℃20s和72℃3min,最后72℃反应10min。
本实施例中,酶切条件如下:37℃酶切2小时。
实施例4 黑曲霉原生质体的制备和转化
按3×105/ml的浓度将黑曲霉孢子接种至PDA液体培养基,在30℃下200r/min培养过夜。用mirocloth滤布收集菌球,并用无菌水清洗菌球。称取一定量的裂解酶,并用渗透压稳定剂KMC溶解,用无菌滤膜过滤除菌。
称取一定量菌球,加入到酶解液中,37℃、100r/min震荡培养约3h直至菌丝完全消化为原生质体,4℃、1000rpm离心10min,弃上清,加入相同体积预冷的STC,4℃、1000rpm离心10min,弃上清,洗涤2次,加入100μL STC,混匀。
向100μL黑曲霉原生质体中加入10μL线性化核酸片段(HGT1蛋白表达框和潮霉素抗性表达框)和330μL PEG缓冲液,冰上放置20min,加入2mL PEG,室温放置10min,依次加入4mL STC和4mL于48℃预热的上层培养基,铺板于含有180mg/L潮霉素的下层培养基上。平板在35℃倒置培养4-7天,直至出现菌落,挑取单菌落继代。每个菌落进行3次单孢子继代。
实施例5 黑曲霉转化子产量的验证
将黑曲霉接种于PDA培养基上,35℃下生孢培养5-7天,刮取孢子,以106/mL的接种量接种于种子培养基,37℃、250r/min培养24h,以1/10接种量转接发酵培养基,35℃、250r/min发酵72h。发酵液离心去除菌体,稀释10倍,用滤膜过滤后用HPLC法检测柠檬酸含量,HPLC法检测条件如下:
仪器:Agilent 1200高效液相色谱仪(配紫外可见检测器、示差检测器和工作站);色谱条件:HPX87H色谱柱(4.6×250mm,5μm),流动相为5mM硫酸溶液,流量为0.6mL/min,进样量为10μL,柱温为30℃,210nm波长紫外光检测。
图1反映了在不同菌株中,柠檬酸和葡萄糖的含量结果,其中H915-1代表野生黑曲霉菌株,HGT1代表本发明的重组菌株,从图中可以看出,本发明的重组黑曲霉菌株的柠檬酸产量得到提高,其产量比野生型菌株提高了14.6%左右,且发酵后期,葡萄糖的含量更低,说明HGT1蛋白促进了葡萄糖的吸收,且葡萄糖吸收的增强可以增加柠檬酸产量。
图2是上述两种菌株的柠檬酸比产酸速率测试结果,可以看出,本发明的重组菌株的柠檬酸产出速率更高,原因是HGT1蛋白的过量表达增强了葡萄糖的转运,可以提高产酸速率,说明葡萄糖吸收是提高柠檬酸产量的限制性步骤。与野生型黑曲霉菌株相比,本发明的重组黑曲霉菌株的发酵时间缩短了6h。
以上所述仅是本发明的优选实施方式,并不用于限制本发明,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明技术原理的前提下,还可以做出若干改进和变型,这些改进和变型也应视为本发明的保护范围。
序列表
<110> 江南大学
<120> 柠檬酸产量提高的重组霉菌的构建方法及应用
<160> 13
<170> PatentIn version 3.3
<210> 1
<211> 1644
<212> DNA
<213> HGT1基因
<400> 1
1 ATGTTGATTG GCAACATCTA CGTGATTGCG AGCGTCGCCG TCGTCGGCGG CGGCTTGTTT
61 GGCTTTGATA TTTCCTCCAT GTCCGCTCAG CTGAGCGAGA ATTCCTATCT ATGCTATTTC
121 AACCAGGGCC CTAAGGGTCC TCCCTTCACT GACGATGAGG ACTGTTCTGG TCCCACATCC
181 CTCAACCAGG GTGGCATCAC GGCAGCCATG GCAGCTGGAT CTTGGCTGGG TGCTTTGATT
241 TCAGGTCCGC TTTCCGATCG CATCGGACGT AAGACGTCCA TCATGGTCGG CTGTGTCGTT
301 TGGCTGATTG GTTCCACCAT TATGTGCGCT TCTCAGAACA TCGGTATGCT CGTTGTTGGG
361 CGGGTCATCA ACGGTCTCGC GGTGGGCATT GAGTCTGCTC AGGTCCCCGT CTACATCAGT
421 GAACTTTCGC CACCCTCCAA GCGCGGTCGG TTCGTAGGTA TGCAGCAATG GGCCATTACA
481 TGGGGTATCC TCATTATGTT CTATATCTCG TATGGCTGCT CCTTCATCGG GGGACAGAAG
541 TCCTACAACT ACAGCACTGC TTCTTGGCGA GTCCCCTGGG GCTTGCAGAT GCTGCCGGCT
601 GTTTTCCTTT TCCTTGGAAT GCTGGTTCTG CCCGAATCTC CTCGTTGGCT GGCGCGCAAG
661 GATCGTTGGG AGGACTGCCA TCGGGTCTTG GCTCTCGTCC ACGCCAAGGG GGATCTCAAC
721 CATCCTTTCG TTGCGCTAGA GTTGCAGGAC ATTCGGGATA TGTGTGAGCT TGAACGTCAG
781 TTCAAAGATG TCACTTACCT TGACCTGTTC AAACCGAGGA TGATCAACCG CACGTTGATC
841 GGTCTGTTCA TGCAGATCTG GTCTCAGCTG ACCGGCATGA ATGTGATGAT GTACTACATC
901 ACGTATCTCT TTTCTATGGC TGGATACACC GGCGATTCCA CCCTCCTTGC CTCCTCCATT
961 CAGTATATTA TCAATGTGTT TATGACCCTC CCGGCACTGA TCTGGATGGA CAAGTGGGGT
1021 CGTCGCATGC CATTGCTGGT TGGCGCAGCT CTGATGGCCA TCTTGATGTA TGCCAATGGT
1081 GCGATCATGG CGGTTCATGG TGTTGTGGTG CCCGGGGGCA TCAATGGGGT TGCAGCTGAG
1141 TCCATGCGTC TTCACGGCGC TCCTGCCAAA GGTTTGATTG CTTGCACATA CCTCTTCGTC
1201 GCGTCGTATG CGCCTACCTG GGGTCCTGTA TCCTGGACGT ACCCCCCTGA GCTGTATCCG
1261 CTGCGGCTGC GTGGTAAGGG AGTGGCTTTG TCGACCTCAG GTAACTGGGC GTTTAACACT
1321 GCATTGGGGC TGTTTACACC CACTGCATTT GAGAACATCC GCTGGAAGAC CTACATCATG
1381 TTTGGGGTGT TCAACACTGC CATGTTCTTG CACGTGCTGT TCCTGTTCCC GGAGACTGCA
1441 GGAAAGACGC TCGAAGAGAC CGAAGCCATG TTCGAGGATC CCAACGGCAT CAAGTACATT
1501 GGCACACCGG CCTGGAAGAC CAAGATGAAG ACCCGGCAGG TGGAGCAGCT GGAGCACGGT
1561 CAAGTGGATA TAGAGTCCAA GATTGAAACC CGCCACGCAG AGACCACTGA GACCGCTCCC
1621 AAATCCCAGG AGGCCACAGC ATAA
<210> 2
<211> 547
<212> 氨基酸
<213> HGT1蛋白编码基因编码的氨基酸
<400> 2
Met Leu Ile Gly Asn Ile Tyr Val Ile Ala Ser Val Ala Val Val
1 5 10 15
Gly Gly Gly Leu Phe Gly Phe Asp Ile Ser Ser Met Ser Ala Gln
20 25 30
Leu Ser Glu Asn Ser Tyr Leu Cys Tyr Phe Asn Gln Gly Pro Lys
35 40 45
Gly Pro Pro Phe Thr Asp Asp Glu Asp Cys Ser Gly Pro Thr Ser
50 55 60
Leu Asn Gln Gly Gly Ile Thr Ala Ala Met Ala Ala Gly Ser Trp
65 70 75
Leu Gly Ala Leu Ile Ser Gly Pro Leu Ser Asp Arg Ile Gly Arg
80 85 90
Lys Thr Ser Ile Met Val Gly Cys Val Val Trp Leu Ile Gly Ser
95 100 105
Thr Ile Met Cys Ala Ser Gln Asn Ile Gly Met Leu Val Val Gly
110 115 120
Arg Val Ile Asn Gly Leu Ala Val Gly Ile Glu Ser Ala Gln Val
125 130 135
Pro Val Tyr Ile Ser Glu Leu Ser Pro Pro Ser Lys Arg Gly Arg
140 145 150
Phe Val Gly Met Gln Gln Trp Ala Ile Thr Trp Gly Ile Leu Ile
155 160 165
Met Phe Tyr Ile Ser Tyr Gly Cys Ser Phe Ile Gly Gly Gln Lys
170 175 180
Ser Tyr Asn Tyr Ser Thr Ala Ser Trp Arg Val Pro Trp Gly Leu
185 190 195
Gln Met Leu Pro Ala Val Phe Leu Phe Leu Gly Met Leu Val Leu
200 205 210
Pro Glu Ser Pro Arg Trp Leu Ala Arg Lys Asp Arg Trp Glu Asp
215 220 225
Cys His Arg Val Leu Ala Leu Val His Ala Lys Gly Asp Leu Asn
230 235 240
His Pro Phe Val Ala Leu Glu Leu Gln Asp Ile Arg Asp Met Cys
245 250 255
Glu Leu Glu Arg Gln Phe Lys Asp Val Thr Tyr Leu Asp Leu Phe
260 265 270
Lys Pro Arg Met Ile Asn Arg Thr Leu Ile Gly Leu Phe Met Gln
275 280 285
Ile Trp Ser Gln Leu Thr Gly Met Asn Val Met Met Tyr Tyr Ile
290 295 300
Thr Tyr Leu Phe Ser Met Ala Gly Tyr Thr Gly Asp Ser Thr Leu
305 310 315
Leu Ala Ser Ser Ile Gln Tyr Ile Ile Asn Val Phe Met Thr Leu
320 325 330
Pro Ala Leu Ile Trp Met Asp Lys Trp Gly Arg Arg Met Pro Leu
335 340 345
Leu Val Gly Ala Ala Leu Met Ala Ile Leu Met Tyr Ala Asn Gly
350 355 360
Ala Ile Met Ala Val His Gly Val Val Val Pro Gly Gly Ile Asn
365 370 375
Gly Val Ala Ala Glu Ser Met Arg Leu His Gly Ala Pro Ala Lys
380 385 390
Gly Leu Ile Ala Cys Thr Tyr Leu Phe Val Ala Ser Tyr Ala Pro
395 400 405
Thr Trp Gly Pro Val Ser Trp Thr Tyr Pro Pro Glu Leu Tyr Pro
410 415 420
Leu Arg Leu Arg Gly Lys Gly Val Ala Leu Ser Thr Ser Gly Asn
425 430 435
Trp Ala Phe Asn Thr Ala Leu Gly Leu Phe Thr Pro Thr Ala Phe
450 440 445 450
Glu Asn Ile Arg Trp Lys Thr Tyr Ile Met Phe Gly Val Phe Asn
455 460 465
Thr Ala Met Phe Leu His Val Leu Phe Leu Phe Pro Glu Thr Ala
470 475 480
Gly Lys Thr Leu Glu Glu Thr Glu Ala Met Phe Glu Asp Pro Asn
485 490 495
Gly Ile Lys Tyr Ile Gly Thr Pro Ala Trp Lys Thr Lys Met Lys
500 505 510
Thr Arg Gln Val Glu Gln Leu Glu His Gly Gln Val Asp Ile Glu
515 520 525
Ser Lys Ile Glu Thr Arg His Ala Glu Thr Thr Glu Thr Ala Pro
530 535 540
Lys Ser Gln Glu Ala Thr Ala
545
<210> 3
<211> 2310
<212> DNA
<213> PgpdA启动子
<400> 3
1 CAATTCCCTT GTATCTCTAC ACACAGGCTC AAATCAATAA GAAGAACGGT TCGTCTTTTT
61 CGTTTATATC TTGCATCGTC CCAAAGCTAT TGGCGGGATA TTCTGTTTGC AGTTGGCTGA
121 CTTGAAGTAA TCTCTGCAGA TCTTTCGACA CTGAAATACG TCGAGCCTGC TCCGCTTGGA
181 AGCGGCGAGG AGCCTCGTCC TGTCACAACT ACCAACATGG AGTACGATAA GGGCCAGTTC
241 CGCCAGCTCA TTAAGAGCCA GTTCATGGGC GTTGGCATGA TGGCCGTCAT GCATCTGTAC
301 TTCAAGTACA CCAACCCTCT TCTGATCCAG TCGATCATCC CGCTGAAGGG CGCTTTCGAA
361 TCGAATCTGG TTAAGATCCA CGTCTTCGGG AAGCCAGCGA CTGGTGACCT CCAGCGTCCC
421 TTTAAGGCTG CCAACAGCTT TCTCAGCCAG GGCCAGCCCA AGACCGACAA GGCCTCCCTC
481 CAGAACGCCG AGAAGAACTG GAGGGGTGGT GTCAAGGAGG AGTAAGCTCC TTATTGAAGT
541 CGGAGGACGG AGCGGTGTCA AGAGGATATT CTTCGCTCTG TATTATAGAT AAGATGATGA
601 GGAATTGGAG GTAGCATAGC TTCATTTGGA TTTGCTTTCC AGGCTGAGAC TCTAGCTTGG
661 AGCATAGAGG GTCCCTTTGG CTTTCAATAT TCTCAAGTAT CTCGAGTTTG AACTTATTCC
721 CGTGAACCTT TTATTCACCA ATGAGCATTG GAATGAACAT GAATCTGAGG ACTGCAATCG
781 CCATGAGGTT TTCGAAATAC ATCCGGATGT CGAAGGCTTG GGGCACCTGC GTTGGTTGAA
841 TTTAGAACGT GGCACTATTG ATCATCCGAT AGCTCTGCAA AGGGCGTTGC ACAATGCAAG
901 TCAAACGTTG CTAGCAGTTC CAGGTGGAAT GTTATGATGA GCATTGTATT AAATCAGGAG
961 ATATAGCATG ATCTCTAGTT AGCTCACCAC AAAAGTCAGA CGGCGTAACC AAAAGTCACA
1021 CAACACAAGC TGTAAGGATT TCGGCACGGC TACGGAAGAC GGAGAAGCCC ACCTTCAGTG
1081 GACTCGAGTA CCATTTAATT CTATTTGTGT TTGATCGAGA CCTAATACAG CCCCTACAAC
1141 GACCATCAAA GTCGTATAGC TACCAGTGAG GAAGTGGACT CAAATCGACT TCAGCAACAT
1201 CTCCTGGATA AACTTTAAGC CTAAACTATA CAGAATAAGA TGGTGGAGAG CTTATACCGA
1261 GCTCCCAAAT CTGTCCAGAT CATGGTTGAC CGGTGCCTGG ATCTTCCTAT AGAATCATCC
1321 TTATTCGTTG ACCTAGCTGA TTCTGGAGTG ACCCAGAGGG TCATGACTTG AGCCTAAAAT
1381 CCGCCGCCTC CACCATTTGT AGAAAAATGT GACGAACTCG TGAGCTCTGT ACAGTGACCG
1441 GTGACTCTTT CTGGCATGCG GAGAGACGGA CGGACGCAGA GAGAAGGGCT GAGTAATAAG
1501 CGCCACTGCG CCAGACAGCT CTGGCGGCTC TGAGGTGCAG TGGATGATTA TTAATCCGGG
1561 ACCGGCCGCC CCTCCGCCCC GAAGTGGAAA GGCTGGTGTG CCCCTCGTTG ACCAAGAATC
1621 TATTGCATCA TCGGAGAATA TGGAGCTTCA TCGAATCACC GGCAGTAAGC GAAGGAGAAT
1681 GTGAAGCCAG GGGTGTATAG CCGTCGGCGA AATAGCATGC CATTAACCTA GGTACAGAAG
1741 TCCAATTGCT TCCGATCTGG TAAAAGATTC ACGAGATAGT ACCTTCTCCG AAGTAGGTAG
1801 AGCGAGTACC CGGCGCGTAA GCTCCCTAAT TGGCCCATCC GGCATCTGTA GGGCGTCCAA
1861 ATATCGTGCC TCTCCTGCTT TGCCCGGTGT ATGAAACCGG AAAGGCCGCT CAGGAGCTGG
1921 CCAGCGGCGC AGACCGGGAA CACAAGCTGG CAGTCGACCC ATCCGGTGCT CTGCACTCGA
1981 CCTGCTGAGG TCCCTCAGTC CCTGGTAGGC AGCTTTGCCC CGTCTGTCCG CCCGGTGTGT
2041 CGGCGGGGTT GACAAGGTCG TTGCGTCAGT CCAACATTTG TTGCCATATT TTCCTGCTCT
2101 CCCCACCAGC TGCTCTTTTC TTTTCTCTTT CTTTTCCCAT CTTCAGTATA TTCATCTTCC
2161 CATCCAAGAA CCTTTATTTC CCCTAAGTAA GTACTTTGCT ACATCCATAC TCCATCCTTC
2221 CCATCCCTTA TTCCTTTGAA CCTTTCAGTT CGAGCTTTCC CACTTCATCG CAGCTTGACT
2281 AACAGCTACC CCGCTTGAGC AGACATCACC
<210> 4
<211> 771
<212> DNA
<213> trp终止子
<400> 4
1 GATCCACTTA ACGTTACTGA AATCATCAAA CAGCTTGACG AATCTGGATA TAAGATCGTT
61 GGTGTCGATG TCAGCTCCGG AGTTGAGACA AATGGTGTTC AGGATCTCGA TAAGATACGT
121 TCATTTGTCC AAGCAGCAAA GAGTGCCTTC TAGTGATTTA ATAGCTCCAT GTCAACAAGA
181 ATAAAACGCG TTTCGGGTTT ACCTCTTCCA GATACAGCTC ATCTGCAATG CATTAATGCA
241 TTGGACCTCG CAACCCTAGT ACGCCCTTCA GGCTCCGGCG AAGCAGAAGA ATAGCTTAGC
301 AGAGTCTATT TTCATTTTCG GGAGACGAGA TCAAGCAGAT CAACGGTCGT CAAGAGACCT
361 ACGAGACTGA GGAATCCGCT CTTGGCTCCA CGCGACTATA TATTTGTCTC TAATTGTACT
421 TTGACATGCT CCTCTTCTTT ACTCTGATAG CTTGACTATG AAAATTCCGT CACCAGCCCC
481 TGGGTTCGCA AAGATAATTG CACTGTTTCT TCCTTGAACT CTCAAGCCTA CAGGACACAC
541 ATTCATCGTA GGTATAAACC TCGAAAATCA TTCCTACTAA GATGGGTATA CAATAGTAAC
601 CATGGTTGCC TAGTGAATGC TCCGTAACAC CCAATACGCC GGCCGAAACT TTTTTACAAC
661 TCTCCTATGA GTCGTTTACC CAGAATGCAC AGGTACACTT GTTTAGAGGT AATCCTTCTT
721 TCTAGAAGTC CTCGTGTACT GTGTAAGCGC CCACTCCACA TCTCCACTCG A
<210> 5
<211> 1020
<212> DNA
<213> hph终止子
<400> 5
1 ATGCCTGAAC TCACCGCGAC GTCTGTCGAG AAGTTTCTGA TCGAAAAGTT CGACAGCGTC
61 TCCGACCTGA TGCAGCTCTC GGAGGGCGAA GAATCTCGTG CTTTCAGCTT CGATGTAGGA
121 GGGCGTGGAT ATGTCCTGCG GGTAAATAGC TGCGCCGATG GTTTCTACAA AGATCGTTAT
181 GTTTATCGGC ACTTTGCATC GGCCGCGCTC CCGATTCCGG AAGTGCTTGA CATTGGGGAA
241 TTCAGCGAGA GCCTGACCTA TTGCATCTCC CGCCGTGCAC AGGGTGTCAC GTTGCAAGAC
301 CTGCCTGAAA CCGAACTGCC CGCTGTTCTG CAGCCGGTCG CGGAGGCCAT GGATGCGATC
361 GCTGCGGCCG ATCTTAGCCA GACGAGCGGG TTCGGCCCAT TCGGACCGCA AGGAATCGGT
421 CAATACACTA CATGGCGTGA TTTCATATGC GCGATTGCTG ATCCCCATGT GTATCACTGG
481 CAAACTGTGA TGGACGACAC CGTCAGTGCG TCCGTCGCGC AGGCTCTCGA TGAGCTGATG
541 CTTTGGGCCG AGGACTGCCC CGAAGTCCGG CACCTCGTGC ACGCGGATTT CGGCTCCAAC
601 AATGTCCTGA CGGACAATGG CCGCATAACA GCGGTCATTG ACTGGAGCGA GGCGATGTTC
661 GGGGATTCCC AATACGAGGT CGCCAACATC TTCTTCTGGA GGCCGTGGTT GGCTTGTATG
721 GAGCAGCAGA CGCGCTACTT CGAGCGGAGG CATCCGGAGC TTGCAGGATC GCCGCGGCTC
781 CGGGCGTATA TGCTCCGCAT TGGTCTTGAC CAACTCTATC AGAGCTTGGT TGACGGCAAT
841 TTCGATGATG CAGCTTGGGC GCAGGGTCGA TGCGACGCAA TCGTCCGATC CGGAGCCGGG
901 ACTGTCGGGC GTACACAAAT CGCCCGCAGA AGCGCGGCCG TCTGGACCGA TGGCTGTGTA
961 GAAGTACTCG CCGATAGTGG AAACCGACGC CCCAGCACTC GTCCGAGGGC AAAGGAATAG
<210> 6
<211> 31
<212> DNA
<213> 引物trp-F
<400> 6
1 ctgcaggatc cacttaaacg ttactgaaat c
<210> 7
<211> 28
<212> DNA
<213> 引物trp-R
<400> 7
1 AAGCTTCTCG AGTGGAGATG TGGAGTGG
<210> 8
<211> 40
<212> DNA
<213> 引物Pgpd-F
<400> 8
1 GAATTCGCGG CCGCCAATTC CCTTGTATCT CTACACACAG
<210> 9
<211> 26
<212> DNA
<213> 引物Pgpd-R
<400> 9
1 GGTACCGGTG ATGTCTGCTC AAGCGG
<210> 10
<211> 45
<212> DNA
<213> 引物HGT1-F
<400> 10
1 CTTGAGCAGA CATCACCGGT ACCATGTTGA TTGGCAACAT CTACG
<210> 11
<211> 44
<212> DNA
<213> 引物HGT1-R
<400> 11
1 TAACGTTTAA GTGGATCGGA TCCTTATGCT GTGGCCTCCT GGGA
<210> 12
<211> 26
<212> DNA
<213> 引物gpd-F
<400> 12
1 CAATTCCCTT GTATCTCTAC ACACAG
<210> 13
<211> 22
<212> DNA
<213> 引物Ttrp-R-2
<400> 13
1 CTCGAGTGGA GATGTGGAGT GG
- 还没有人留言评论。精彩留言会获得点赞!