一种基于对称环状哑铃模板的核酸恒温指数扩增技术及其在microRNA检测中的应用的制作方法
本发明属于分子检测技术领域,具体涉及一种基于对称环状哑铃模板的核酸恒温指数扩增技术及其在microRNA检测中的应用。
背景技术:
MicroRNA(miRNA)是一类对基因表达具有调控作用的非编码小分子RNA(18~24碱基)。它在动植物生长、发育、分化和生殖等过程中发挥着重要作用。人类约30%的基因受miRNA调控,且miRNA的表达水平与人类重大疾病密切相关。对miRNA的定量检测有助于深入理解其作用机制,对疾病的诊断与治疗以及相关基因药物的开发等具有重要意义。由于miRNA的序列短,在细胞或者体液内的表达水平非常低、易降解且同源miRNA之间仅相差1~2个碱基,要求检测方法必须具有高选择性和高灵敏度。Northern印迹技术是当前分析miRNA的标准方法,但该方法操作繁琐、耗时长、灵敏度低,检测需要的样品量大,对RNase污染敏感,分析结果易受实验条件影响。Microarray方法可实现miRNA的高通量、多组分同时检测,但该方法特异性和灵敏度相对较低,且芯片制作和检测成本高。反转录荧光定量PCR(RT-qPCR)方法具有很高的检测灵敏度,但由于miRNA序列很短,需要多种技术将其转录成cDNA后才能进行PCR;PCR反应需要精确的温度循环装置,因此,该方法操作复杂,耗时长,成本高。
核酸恒温扩增技术由于能在恒温的条件下产生较高的扩增效率,成为miRNA检测方法研究与应用热点。其中,恒温指数扩增反应(Exponential Amplification Reaction,EXPAR)技术备受关注。该技术是由D.J.Galas等在2003年建立起来的。基于3′端与5′端序列完全相同,且均与引物序列互补的线性模板,在DNA聚合酶和特殊的切口酶(nicking enzyme)共同作用下,可以在恒温条件下对短链核酸(10~25碱基)进行高效、快速的扩增,一般在几分钟内可对目标核酸放大106~109倍。与PCR扩增技术相比,EXPAR方法反应迅速,无需精确的热循环装置,扩增倍数媲美于PCR技术。因此,该技术已被广泛应用于短核酸的检测,特别是对miRNA的定量分析。但由于传统EXPAR技术的扩增模板为线性对称模板,引物在5′端和3′端杂交的几率和速率也相同。杂交在3′端的引物能够正常扩增,而杂交在5′端的引物无法扩增,因此,扩增反应损失了50%的扩增效率;在3'端的非特异性扩增也会极大降低扩增特异性。
技术实现要素:
本发明的目的在于通过设计一个基于toehold exchange原理的对称环状哑铃扩增模板,解决扩增反应中扩增效率损失难题,在获得100%的二次方指数扩增同时,保证扩增反应的特异性,实现更为快速、高效、高选择性的核酸恒温指数扩增并应用于短链核酸的灵敏检测。
本发明所采取的技术方案是:
一种环状哑铃形扩增模板,包括两个环部和一个颈部,两个环部上均设有长度可调节的toehold序列,所述的toehold序列及相连颈部的部分序列与靶序列互补,环部的另一侧含有切口酶识别位点。
作为优选的,所述toehold序列的长度≤14bp。
进一步优选的,所述toehold序列的长度为9-14bp。
作为优选的,与toehold序列相连的颈部与靶序列互补部分的长度为9-14bp。
一种microRNA检测试剂盒,其包括以上所述的环状哑铃形扩增模板,所述靶序列即为目标microRNA。
作为优选的,所述试剂盒还包括A液和B液,其中A液包括:切口酶缓冲液、dNTP、RNase抑制剂;B液包括:DNA聚合酶缓冲溶液、DNA聚合酶、切口酶、以及能与双链DNA结合发光的荧光染料。
一种高效率的microRNA检测方法,包括如下步骤:
(1)从样品中提取总microRNA;
(2)加入权利要求1-4任一项所述的环状哑铃形扩增模板进行恒温指数扩增;
(3)根据检测到的荧光强度判断目标microRNA的浓度。
所述恒温指数扩增体系中还含有dNTP、RNase抑制剂、DNA聚合酶缓冲溶液、DNA聚合酶、切口酶缓冲液、切口酶、以及能与双链DNA结合发光的荧光染料。
作为优选的,所述恒温指数扩增体系中,环状哑铃形扩增模板的用量为0.05-0.25μM。
作为优选的,所述DNA聚合酶为Vent(exo-)聚合酶,其用量为0.05-0.3U/μL;所述切口酶为Nt.BstNBI切口酶,其用量为0.05-0.25U/μL。
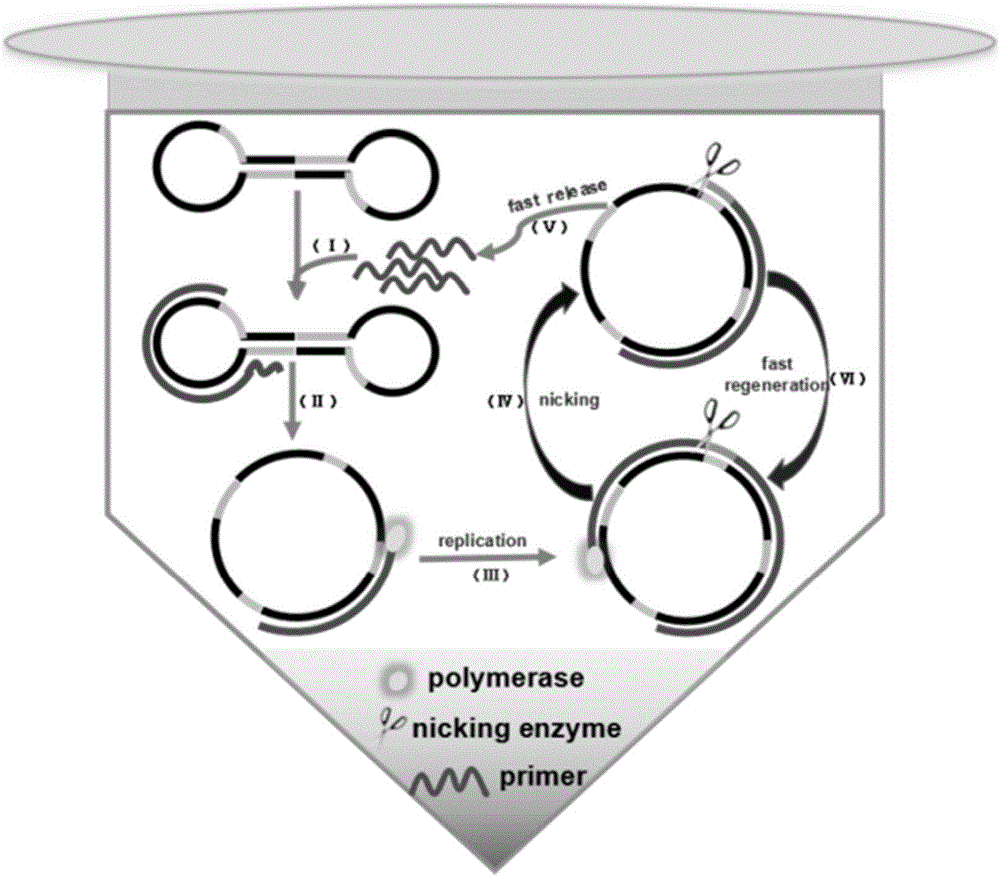
本发明设计的环状哑铃形扩增模板(结构见图1),模板的两个环部各包含一个与靶序列互补的序列。在扩增反应中,靶序列可与任一互补序列杂交,确保扩增反应为100%的二次方指数扩增效率;另外,只有与哑铃环模板上完全互补的引物才能通过toehold exchange链置换反应将环状哑铃形模板的颈部杂交序列打开从而发生扩增反应。通过调节toehold的长度,链置换反应的动力学常数可以高达106,实现高特异性。扩增原理如图2所示。首先target引物在环状哑铃形模板任一端环部上根据碱基互补配对原则与其互补的序列杂交,然后通过toehold exchange链置换反应将环状哑铃形模板颈部杂交序列打开,target序列完全与环状模板杂交,在DNA聚合酶的作用下进行延伸合成双链DNA,切口酶(限制性内切酶)辨认特异性DNA序列(黄色),在延伸链上切开一个缺口,暴露其3'端;DNA聚合酶以切口为起点,以未被切断的环状链为模板,合成新链,并将“旧链”剥离置换下来,被置换的单链DNA又可作为target与另一新环状哑铃形模板杂交,指导新一轮的合成,新合成的双链DNA又恢复了切口酶位点,切口酶在此位点重新切开一个缺口,延长、剥离等步骤又重新开始;上述各反应步骤在恒温条件下反复、连续地进行,target引物分子呈指数扩增,直到原料耗尽或酶活性丧失。
本发明的有益效果是:
本发明通过设计一个基于toehold链置换的对称环形哑铃形扩增模板,对广泛应用的指数扩增方法进行改进。该方法较现有的指数扩增方法,具有以下优点:
1、反应速度更快。扩增所需的时间更少,缩短了检测的时间。
2、特异性更好。可以很好的区别单碱基错配引起的非特异性扩增。
3、线性范围更宽。检测下限低至单拷贝数,可用于超微量目标核酸的检测。
4、具有更好的精确性,重复性和稳定性,可发展成为试剂盒并向市场推广。
附图说明
图1:环状哑铃形扩增模板结构示意图;
图2:本发明环状哑铃形模板指数扩增原理图;
图3:标准EXPAR模板结构示意图;
图4:biotin标记的toehold扩增模板结构示意图;
图5:环状哑铃形模板的电泳表征(1、T4连接酶处理前;2、T4连接酶处理后;3、核酸外切酶处理后);
图6:不同toehold长度模板反应曲线;
图7:不同引物对不同toehold长度模板敏感度;
图8:不同扩增模板实时扩增曲线(circle-EXPAR:环状哑铃形模板扩增;toehold-EXPAR:单toehold模板扩增;EXPAR:标准指数扩增模板扩增);
图9:聚合酶用量优化图;
图10:切口酶用量优化图;
图11:模板用量优化图;
图12:不同let-7a浓度下的扩增曲线图;
图13:Let-7a检测的标准曲线;
图14:不同引物扩增曲线;
图15:方法特异性实验结果图。
具体实施方式
下面结合实施例对本发明作进一步的说明,但并不局限于此。
实施例1基于对称环状哑铃形模板的核酸恒温指数扩增技术方案
1、环状哑铃形扩增模板的制备与表征
(1)环状哑铃形扩增模板的合成
根据待测miRNA序列设计一系列具有不同toehold长度的线型探针,根据toehold的碱基个数分别命名为TN。在探针的5′端修饰一磷酸基团,通过T4DNA连接酶连接探针的3′和5′端,形成环形探针,再利用核酸外切酶I和核酸外切酶III水解为反应的线型探针,获得环状哑铃形扩增模板。
(2)环状哑铃形扩增模板的表征
将(1)中制备的环状哑铃形扩增模板进行电泳表征,验证模板的制备情况。
2、测定miRNA的含量
(1)扩增模板优化
a)反应热力学计算
将待测miRNA及其同源miRNAs与具有不同toehold长度的哑铃形扩增模板杂交的反应分解成若干步骤,采用热力学软件计算每一步的反应商,找出最佳指数扩增模板。
b)扩增动力学
等量待测miRNA与具有不同toehold长度的哑铃形扩增模板反应,实时荧光定量PCR记录扩增曲线,考察扩增效率与扩增模板toehold长度的规律,验证理论计算结果。
c)扩增敏感度
等量待测miRNA或同源miRNAs分别与具有不同toehold长度的哑铃形扩增模板反应,实时荧光定量PCR记录扩增曲线。根据扩增曲线的POI值计算不同扩增模板对待测miRNA和同源miRNAs的扩增敏感度,验证理论计算结果,选择最佳哑铃形扩增模板。
(d)不同方法模板扩增动力学曲线
固定待测miRNA,聚合酶,切口酶的用量,扩增模板用量,分别采用标准EXPAR模板(结构见图3),biotin标记的toehold扩增模板(结构见图4),环状哑铃形模板进行扩增实验,用实时荧光定量PCR记录扩增曲线,考察不同模板扩增曲线的POI值差异,POI值越小的扩增效率越高。
(2)miRNA测定条件优化
选择一个环状哑铃扩增模板,通过单因素轮换法优化miRNA测定条件。
a)聚合酶用量
固定待测miRNA,哑铃形扩增模板,切口酶的用量,梯度改变聚合酶用量,实时荧光定量PCR记录扩增曲线,考察扩增曲线及空白实验的POI差值,差值最大时所对应的聚合酶用量为实验最佳用量。
b)切口酶用量
固定待测miRNA,哑铃形扩增模板,聚合酶的用量,梯度改变切口酶用量,实时荧光定量PCR记录扩增曲线,考察扩增曲线及空白实验的POI差值,差值最大时所对应的切口酶用量为实验最佳用量。
c)扩增模板用量
固定待测miRNA,聚合酶,切口酶的用量,梯度改变哑铃形扩增模板用量,实时荧光定量PCR记录扩增曲线,考察扩增曲线及空白实验的POI差值,差值最大时所对应的扩增模板用量为实验最佳用量。
(3)标准曲线获得
在冰上配制A和B两部分溶液,A溶液包含切口酶缓冲液、dNTP、RNase抑制剂、最佳哑铃形扩增模板、以及一系列不同浓度的待测miRNA;B溶液包含聚合酶缓冲溶液、最佳量聚合酶、切口酶、SYBR Green I荧光染料。将A和B溶液混合,立即利用实时荧光定量PCR记录扩增曲线,求得POI值,绘制待测miRNA测定标准曲线。
(4)特异性评估
在冰上配制A和B两部分溶液,A溶液包含切口酶缓冲液、dNTP、RNase抑制剂、最佳哑铃形扩增模板、以及不同的同源miRNAs;B溶液包含聚合酶缓冲溶液、最佳量聚合酶、切口酶、SYBR Green I荧光染料。将A和B溶液混合,立即利用实时荧光定量PCR记录扩增曲线,求得POI值。根据此POI值以及标准曲线计算该方法的特异性。
(5)样品中待测miRNA含量测定
从样品中提取总miRNA。在冰上配制A和B两部分溶液,A溶液包含切口酶缓冲液、dNTP、RNase抑制剂、最佳哑铃形扩增模板、以及一系列不同浓度的待测miRNA;B溶液包含聚合酶缓冲溶液、最佳量聚合酶、切口酶、SYBR Green I荧光染料。将A和B溶液混合,立即利用实时荧光定量PCR记录扩增曲线,求得POI值。根据此POI值,在标准曲线上查出相应的的浓度。
实施例2
下面以检测miRNA let-7家族中let-7a为例,进一步说明本发明的技术方案。
1、环状哑铃形扩增模板的合成
let-7a序列:5'-UGA GGU AGU AGG UUG UAU AGUU-3'(SEQ ID NO.7)。
针对let-7a序列设计一系列toehold长度不同的直链型探针,根据toehold碱基个数分别命名为T9~T14。探针碱基序列如下:(下划线为toehold区域,加粗部分为切口酶识别序列)T14:5′-(PO4)TTA ACT ATA CAA CCT ACT ACC TCACCC CGT ATA GTT AAC TAT ACA ACC TAC TAC CTC ACC CCG TAT AG-3′(SEQ ID NO.1);
T13:5′-(PO4)TTA ACT ATA CAA CCT ACT ACC TCACCC TGT ATA GTT AAC TAT ACA ACC TAC TAC CTC ACC CTG TAT AG-3′(SEQ ID NO.2);
T12:5′-(PO4)TTA ACT ATA CAA CCT ACT ACC TCACCT TGT ATA GTT AAC TAT ACA ACC TAC TAC CTC ACC TTG TAT AG-3′(SEQ ID NO.3);
T11:5′-(PO4)TTA ACT ATA CAA CCT ACT ACC TCACGT TGT ATA GTT AAC TAT ACA ACC TAC TAC CTC ACG TTG TAT AG-3’(SEQ ID NO.4)
T10:5′-(PO4)TTA ACT ATA CAA CCT ACT ACC TCAGGT TGT ATA GTT AAC TAT ACA ACC TAC TAC CTC AGG TTG TAT AG-3′(SEQ ID NO.5);
T9:5′-(PO4)TTA ACT ATA CAA CCT ACT ACC TCAAGG TTG TAT AGT TAA CTA TAC AAC CTA CTA CCT CAA GGT TGT ATA G-3′(SEQ ID NO.6)。
分别将上述探针在95℃变性5min,自然冷却到室温,自杂交生成开口的哑铃型探针。分别取30μL该探针溶液,在16℃下加入5μL 10×T4DNA连接酶缓冲液、2μL T4DNA连接酶(100U/μL),反应2h后在65℃变性10min终止连接反应。然后向溶液加入核酸外切酶I(20U/μL)、核酸外切酶III(100U/μL),在37℃反应14h,彻底水解溶液中未闭合环的核酸探针。之后,在85℃变性20min,冷却至室温,用nanodrop 2000测定溶液浓度,稀释至1μM,保存在-20℃备用。
2、环状哑铃形扩增模板的电泳表征
选取上述1中合成的哑铃形扩增模板T11各步骤产物进行电泳实验,验证探针的合成效果。配制15%的非变性凝胶电泳,在180V电压下进行电泳,电泳图如图5,图中第3个条带只有1个明显的条带,证明成功合成了环状哑铃形模板。
3、测定let-7a含量
以let-7a为目标miRNA,let-7a家族其他miRNA为错配引物,序列如下:
let-7a:5'-UGA GGU AGU AGG UUG UAU AGU U-3'(SEQ ID NO.7);
let-7b:5'-UGA GGU AGU AGG UUG UGU GGU U-3'(SEQ ID NO.8);
let-7c:5'-UGA GGU AGU AGG UUG UAU GGU U-3'(SEQ ID NO.9);
let-7d:5'-AGA GGU AGU AGG UUG CAU AGU-3'(SEQ ID NO.10);
let-7e:5'-UGA GGU AGG AGG UUG UAU AGU-3'(SEQ ID NO.11);
let-7f:5'-UGA GGU AGU AGA UUG UAU AGU U-3'(SEQ ID NO.12);
let-7g:5'-UGA GGU AGU AGU UUG UAC AGU-3'(SEQ ID NO.13);
let-7i:5'-UGA GGU AGU AGU UUG UGC UGU U-3'(SEQ ID NO.14)。
(1)环状哑铃探针优化
a)热力学计算
将引物及其错配与环杂交的反应分解成若干步骤,采用热力学软件计算每一步的反应商。通过热力学计算发现:在55℃,toehold长度变短,引物和模板杂交速率变慢。考虑反应速率及特异性,当toehold长度为11时为最优模板。
b)扩增动力学
在冰上配制A和B两部分溶液,A溶液包含Nt.BstNBI缓冲液、具有不同toehold长度的哑铃型扩增模板(T9~T14)、dNTP、RNase抑制剂、let-7a;B溶液包含1×ThermoPol聚合酶缓冲溶液、Vent(exo-)聚合酶、Nt.BstNBI切口酶、SYBR Green I荧光染料。在55℃将A和B溶液混合后定容至20μL,立即利用伯乐CFX-connect记录实时荧光定量PCR扩增曲线,考察扩增效率与扩增模板toehold长度的规律。反应溶液各组分含量为:0.5×Nt.BstNBI缓冲液(25mM tris-HCl pH 7.90,50mM NaCl,5mM MgCl,0.5mM EDTA)、扩增模板(0.15μM)、dNTP(250μM)、RNase抑制剂(0.8U/μL)、let-7a(10pM)、1×ThermoPol聚合酶缓冲溶液(10mM KCl,10mM(NH4)2SO4,20mM Tris-HCl pH 8.8,2mM MgSO4,0.1%TritonX-100)、Vent(exo-)聚合酶(0.1U/μL)、Nt.BstNBI切口酶(0.15U/μL)、0.4×SYBR Green I荧光染料、DEPC水。如图6所示,随着toehold长度的增加,扩增速率减慢,符合理论计算结果;当Toehold长度>12,反应速率明显降低,toehold长度<11,反应速率相差不大。
c)扩增敏感度
同上述方法,等量let-7a或同源miRNAs分别与T9~T14的哑铃模板反应,实时荧光定量PCR记录扩增曲线。根据POI值计算不同引物对不同环的敏感度,如图7所示,T11模板对错配引物的敏感度最强。综合实验现象及理论计算,选择T11模板为最优反应模板。
(d)不同方法模板扩增动力学曲线
同上述方法,固定待测miRNA,聚合酶,切口酶的用量,扩增模板用量,分别采用标准EXPAR模板(结构见图3),biotin标记的toehold扩增模板(结构见图4),最优环状哑铃形模板进行扩增实验,用实时荧光定量PCR记录扩增曲线,考察不同模板扩增曲线的POI值差异,POI值越小的扩增效率越高,由图8可知环状哑铃型模板扩增曲线的POI值最小,说明本方法扩增效率明显提高。
(2)测定条件优化
通过单因素轮换法对let-7a测定条件进行优化,以T11环状哑铃形探针为扩增模板。
a)聚合酶量
在冰上配制A和B两部分溶液,A溶液包含切Nt.BstNBI缓冲液、模板T11、dNTP、RNase抑制剂、let-7a;B溶液包含聚合酶缓冲溶液、0.05、0.1、0.2、0.3U/μL Vent(exo-)聚合酶、切口酶、SYBR Green I荧光染料。将A和B溶液混合,立即利用伯乐CFX-connect记录实时荧光定量PCR扩增曲线,求得POI值。如图9所示,当聚合酶含量为0.1U/μL时,let-7a与空白的POI差值最大,背景值最小,故选择聚合酶为0.1U/μL作为此指数扩增反应的最佳条件。
b)切口酶量
同上述方法,固定let-7a,T11,Vent(exo-)用量,改变Nt.BstNBI用量为0.0.05、0.1、0.15、0.2、0.25U/uL,实时荧光定量PCR记录扩增曲线,考察扩增曲线及空白实验的POI差值。如图10所示,当Nt.BstNBI含量为0.15U/μL时,let-7a与空白的POI差值最大,作为指数扩增切口酶的最佳用量。
c)模板量
同上述方法,固定let-7a,Vent(exo-),Nt.BstNBI用量,改变T11用量为0.05、0.1、0.15、0.2、0.25μM,实时荧光定量PCR记录扩增曲线,考察扩增曲线及空白实验的POI差值。如图11所示,当T11含量为为0.15μM时,let-7a与空白的POI差值最大,作为指数扩增模板最佳用量。
4、标准曲线获得
根据最佳反应条件,在冰上配制一系列不同let-7a浓度的待测溶液。在55℃将A和B溶液混合后定容至20μL,立即利用伯乐CFX-connect记录实时荧光定量PCR扩增曲线(图12),获得POI值。进行线性回归(图13),let-7a在1aM-10nM之间与POI呈线性关系,线性方程为:POI=–2.01logAlet-7a–18.94(R=0.9974)。
5、特异性评估
根据最佳反应条件,在冰上配制一系列含10pM let-7a或同源miRNAs(let-7b、let-7c、let-7d、let-7e、let-7f、let-7g、let-7i)的待测溶液。在55℃将A和B溶液混合后定容至20μL,立即利用伯乐CFX-connect记录实时荧光定量PCR扩增曲线(图14),获得POI值,根据标准曲线及POI值求出各错配序列对let-7a检测的影响(图15)。
6、目标样品含量的测定
在一定量样品中提取出目标物后,根据最佳反应条件(同5),在冰上配制A和B两部分溶液,A部分包含Nt.BstNBI缓冲液、模板T11、dNTP、RNase抑制剂、样品目标物;B部分包含聚合酶缓冲溶液、Vent(exo-)聚合酶、内切酶、SYBR Green I荧光染料。将A和B两部分溶液混合,立即在定量PCR仪上监控扩增曲线,求得POI值,根据此POI值,在标准曲线上查出相应的的浓度。
从样品中提取总miRNA。在冰上配制A和B两部分溶液,A溶液包含切口酶缓冲液、dNTP、RNase抑制剂、最佳哑铃型扩增模板、以及一系列不同浓度的待测miRNA;B溶液包含聚合酶缓冲溶液、最佳量聚合酶、切口酶、SYBR Green I荧光染料。将A和B溶液混合,立即利用实时荧光定量PCR记录扩增曲线,求得POI值。根据此POI值,在标准曲线上查出相应的的浓度。将本方法测定的实际样品数据与测定miRNA的标准方法RT-qPCR测定的数据进行对比。如表一,本方法对测定miRNA取得了较好的结果。
表一:实际样品测试情况
以上实施例表明,本发明的技术方案在获得100%的二次方指数扩增同时,保证扩增反应的特异性,实现更为快速、高效、高选择性的核酸恒温指数扩增并应用于短链核酸的灵敏检测。
以上实施例仅为介绍本发明的优选案例,对于本领域技术人员来说,在不背离本发明精神的范围内所进行的任何显而易见的变化和改进,都应被视为本发明的一部分。
SEQUENCE LISTING
<110> 中山大学
<120> 一种基于对称环状哑铃模板的核酸恒温指数扩增技术及其在microRNA检测中
的应用
<130>
<160> 14
<170> PatentIn version 3.5
<210> 1
<211> 86
<212> DNA
<213> 人工序列
<400> 1
ttaactatac aacctactac ctcaaaaaga ctcccccgta tagttaacta tacaacctac 60
tacctcaaaa agactccccc gtatag 86
<210> 2
<211> 86
<212> DNA
<213> 人工序列
<400> 2
ttaactatac aacctactac ctcaaaaaga ctcccctgta tagttaacta tacaacctac 60
tacctcaaaa agactcccct gtatag 86
<210> 3
<211> 86
<212> DNA
<213> 人工序列
<400> 3
ttaactatac aacctactac ctcaaaaaga ctcccttgta tagttaacta tacaacctac 60
tacctcaaaa agactccctt gtatag 86
<210> 4
<211> 86
<212> DNA
<213> 人工序列
<400> 4
ttaactatac aacctactac ctcaaaaaga ctccgttgta tagttaacta tacaacctac 60
tacctcaaaa agactccgtt gtatag 86
<210> 5
<211> 86
<212> DNA
<213> 人工序列
<400> 5
ttaactatac aacctactac ctcaaaaaga ctcggttgta tagttaacta tacaacctac 60
tacctcaaaa agactcggtt gtatag 86
<210> 6
<211> 88
<212> DNA
<213> 人工序列
<400> 6
ttaactatac aacctactac ctcaaaaaga ctcaggttgt atagttaact atacaaccta 60
ctacctcaaa aagactcagg ttgtatag 88
<210> 7
<211> 22
<212> RNA
<213> human
<400> 7
ugagguagua gguuguauag uu 22
<210> 8
<211> 22
<212> RNA
<213> human
<400> 8
ugagguagua gguugugugg uu 22
<210> 9
<211> 22
<212> RNA
<213> human
<400> 9
ugagguagua gguuguaugg uu 22
<210> 10
<211> 21
<212> RNA
<213> human
<400> 10
agagguagua gguugcauag u 21
<210> 11
<211> 21
<212> RNA
<213> human
<400> 11
ugagguagga gguuguauag u 21
<210> 12
<211> 22
<212> RNA
<213> human
<400> 12
ugagguagua gauuguauag uu 22
<210> 13
<211> 21
<212> RNA
<213> human
<400> 13
ugagguagua guuuguacag u 21
<210> 14
<211> 22
<212> RNA
<213> human
<400> 14
ugagguagua guuugugcug uu 22
- 还没有人留言评论。精彩留言会获得点赞!