生产蛋白质的方法与流程
本申请是申请日为2010年6月10日、申请号为201080026008.5、发明名称为“生产蛋白质的方法”的发明专利申请的分案申请。
本发明涉及生产目的蛋白质的方法,其包括将含有基因片段和位于该基因片段两端的转座子序列的蛋白质表达载体导入悬浮性哺乳动物细胞,其中所述基因片段含有编码目的蛋白质的dna和选择性标记基因,将插入在一对转座子序列之间的基因片段整合入该哺乳动物细胞的染色体中以获得能够表达目的蛋白质的哺乳动物细胞;和悬浮培养该哺乳动物细胞;和能够表达目的蛋白质的悬浮性哺乳动物细胞。
背景技术:
利用重组dna技术生产外源蛋白质被用于各种产业,如制药业和食品业。在多数情况下,通过以下方法进行重组蛋白质的生产:将含有编码目的蛋白质的核苷酸序列的表达载体导入宿主,如大肠杆菌(escherichiacoli)、酵母、昆虫细胞、植物细胞、和动物细胞,选择表达载体被整合入染色体的转化体,并进一步在适当培养条件下培养所述细胞株。
然而,为有效开发能够生产外源蛋白质的宿主,必需选择对每种目标蛋白质具有良好生产性的宿主细胞,因此在个体宿主的外源蛋白质生产技术方面要求进一步的技术创新。
与动物细胞不同,在细菌系统、如大肠杆菌、和酵母系统中,很多情况下难以实现翻译后修饰,如糖链修饰,并因此在生产具有活性的蛋白质方面产生了问题。
由于在昆虫系统中所生产的蛋白质受到翻译后修饰,如磷酸化和加入糖链,该系统具有可以表达有原始生理活性的蛋白质的优点。但是,由于分泌蛋白的糖链结构与哺乳动物源细胞的不同,当将该蛋白质应用于制药用途时,抗原性等成为问题。
此外,由于导入外源基因时在昆虫细胞系统中使用重组病毒,存在从安全角度出发要求病毒灭活和隔离的问题。
在动物细胞系统中,对于源自高等动物包括人类的蛋白质,可以用与活体内产生的蛋白质更相似的方式进行翻译后修饰,如磷酸化、加入糖链、和折叠。这样的精确翻译后修饰对于蛋白质在其重组蛋白质中再现其所具有的原始生理活性是必需的,并且用哺乳动物细胞作为宿主的蛋白质生产系统通常应用于要求这样的生理活性的药品等。
但是用哺乳动物细胞作为宿主的蛋白质表达系统通常生产性低,且在很多情况下导致导入的基因的稳定性问题。改进用哺乳动物培养细胞作为宿主的蛋白质生产性不仅在生产治疗药物、诊断剂等中很重要,而且极大地促进了它们的研究和开发。因此,迫切需要开发一种基因表达系统,其易于使利用哺乳动物培养细胞、特别是中华仓鼠卵巢细胞(cho细胞)作为宿主获得高生产性细胞株成为可能。
转座子是可以从染色体上的一个基因座转移到其它基因座的可转座基因元件。转座子是用于分子生物和遗传学研究的有力工具,并在昆虫或线虫(例如,黑腹果蝇(drosophilamelanogaster)或秀丽隐杆线虫(caenorhabditiselegans))和植物中用于诸如诱变、基因捕获和制备转基因个体的目的。但是,对于脊椎动物包括哺乳动物细胞,这样的技术的开发已延迟。
然而,近年已报道了在脊椎动物中具有活性的转座子,并且其中一些已显示出在哺乳动物细胞、如源自小鼠和人类的细胞中具有活性。典型实例包括从青鳉(鳉鱼)克隆的转座子tol1(专利文献1)和tol2(非专利文献1)、从存在于鲑(onchorhynchus)鱼科基因组中的非自主转座子再构建的sleepingbeauty(非专利文献2)、源自蛙的人工转座子frogprince(非专利文献3)和源自昆虫的转座子piggybac(非专利文献4)。
这些dna转座子已经作为基因转移工具用于诱变、基因捕获、转基因个体的制备、抗药蛋白质的表达等,以向哺乳动物细胞的基因组中引入新的表现型(非专利文献5至12)。
在昆虫的情况下,研究了用源自鳞翅目(lepidoptera)昆虫的转座子piggybac向蚕染色体中导入外源基因以表达由所述外源基因编码的蛋白质的方法,并已公开了运用上述技术的蛋白质生产方法(专利文献2)。
但是,由于所表达的目的蛋白质的表达水平不充足并且在蚕的整个身体中产生,导致了经济问题,因为需要先进的纯化技术从含有大量污染蛋白质的体液中以高纯度形式回收所表达的外源蛋白质。
此外,用源自青鳉的转座子tol2在哺乳动物细胞中表达与g418抗性有关的蛋白质的实例是已知的(非专利文献12)。
引用列表
专利文献
专利文献1wo2008/072540
专利文献2日本国特表2001-532188号公报
非专利文献
非专利文献1nature383,30(1996)
非专利文献2cell91,501-510(1997)
非专利文献3nucleicacidsres,31,6873-6881(2003)
非专利文献4insectmol.biol.5,141-151(1996)
非专利文献5genetics.166,895-899(2004)
非专利文献6plosgenet,2,e169(2006)
非专利文献7proc.natl.acad.sci.usa95,10769-10773(1998)
非专利文献8proc.natl.acad.sci.usa98:6759-6764(2001)
非专利文献9nature436,221-226(2005)
非专利文献10nucleicacidsres,31,6873-6881(2003)
非专利文献11nucleicacidsres.35,e87(2007)
非专利文献12procnatlacadsciusa,103,15008-15013(2006)
发明概要
技术问题
为了生产和分析目的蛋白质,需要用哺乳动物源培养细胞挑选稳定并高水平表达目的蛋白质的细胞株,但制备和培养生产目的蛋白质的细胞要求相当多的劳动和时间。
此外,虽然已经了解目的蛋白质是利用转座子序列在哺乳动物细胞中表达的,但是不了解用转座子序列制备可以高水平表达目的蛋白质并因此可以用作蛋白质生产系统的细胞;用转座子序列制备可以高水平生产目的蛋白质的哺乳动物细胞的方法;和用该细胞生产蛋白质的方法。
如上所述,需要通过建立可以利用哺乳动物培养细胞有效并在短时间内高水平生产目的蛋白质的蛋白质生产系统,来大量表达目的蛋白质。因此,本发明的目的是提供可以有效建立的能够高水平表达目的蛋白质的细胞,和利用该细胞生产目的蛋白质的方法。
问题的解决方案
为解决上述问题,本发明人已经进行了深入研究并由此发现可以通过以下方法有效制备能够高水平表达目的蛋白质的哺乳动物细胞:将含有基因片段和位于该基因片段两端的转座子序列的蛋白质表达载体导入悬浮性哺乳动物细胞,其中所述基因片段含有编码目的蛋白质的dna和选择性标记基因;和将插入在一对(两个)转座子序列之间的基因片段整合入哺乳动物细胞的染色体。此外,发现可以用该细胞有效生产目的蛋白质,并从而完成了本发明。
发明详述
具体地,本发明如下:
1.生产目的蛋白质的方法,其包括将含有基因片段和位于该基因片段两端的转座子序列的蛋白质表达载体导入悬浮性哺乳动物细胞,其中所述基因片段含有编码目的蛋白质的dna和选择性标记基因;将插入在一对转座子序列之间的基因片段整合入该哺乳动物细胞的染色体以获得能够表达目的蛋白质的哺乳动物细胞;和悬浮培养该哺乳动物细胞。
2.生产目的蛋白质的方法,其包括下列步骤(a)至(c):
(a)将下列表达载体(a)和(b)同时导入悬浮性哺乳动物细胞的步骤:
(a)含有包含编码目的蛋白质的dna的基因片段和位于该基因片段两端的转座子序列的表达载体,
(b)含有编码转座酶的dna的表达载体,其中所述转座酶识别转座子序列并具有将插入在一对转座子序列之间的基因片段转移入染色体中的活性,
(b)从步骤(a)中所导入的表达载体瞬间表达转座酶、以将插入在一对转座子序列之间的基因片段整合入哺乳动物细胞的染色体而获得能够表达目的蛋白质的悬浮性哺乳动物细胞的步骤,和
(c)悬浮培养步骤(b)中获得的能够表达目的蛋白质的悬浮性哺乳动物细胞以生产目的蛋白质的步骤;
3.获得能够表达目的蛋白质的悬浮性哺乳动物细胞的方法,其包括将含有基因片段和位于该基因片段两端的转座子序列的蛋白质表达载体导入悬浮性哺乳动物细胞,其中所述基因片段含有编码目的蛋白质的dna和选择性标记基因;和将插入在一对转座子序列之间的基因片段整合入该哺乳动物细胞的染色体;
4.上述第1至3项中任一项所述的方法,其中悬浮性哺乳动物细胞是能够在无血清培养基中存活和增殖的细胞;
5.上述第1至4项中任一项所述的方法,其中悬浮性哺乳动物细胞是选自悬浮性cho细胞、per.c6细胞、大鼠骨髓瘤细胞yb2/3hl.p2.g11.16ag.20(或也称为yb2/0)和适应于悬浮培养的悬浮性小鼠骨髓瘤细胞ns0的至少一种细胞,其中所述悬浮性cho细胞中的cho细胞适应于悬浮培养;
6.上述第5项中所述的方法,其中cho细胞是选自cho-k1、cho-k1sv、dukxb11、cho/dg44、pro-3和cho-s的至少一种细胞;
7.上述第1至6项中任一项所述的方法,其中选择性标记基因是环己酰亚胺抗性基因;
8.上述第7项所述的方法,其中环己酰亚胺抗性基因是编码人核糖体蛋白质l36a的突变体的基因;
9.上述第8项所述的方法,其中突变体是人核糖体蛋白质l36a的第54位脯氨酸被其它氨基酸取代的突变体;
10.上述第9项所述的方法,其中其它氨基酸是谷氨酰胺;
11.上述第1至10项中任一项所述的方法,其中一对转座子序列是源自在哺乳动物细胞中发挥功能的一对dna型转座子的核苷酸序列;
12.上述第11项所述的方法,其中源自一对dna型转座子的核苷酸序列是源自一对tol1转座子的核苷酸序列或源自一对tol2转座子的核苷酸序列;
13.上述第12项所述的方法,其中源自一对tol2转座子的核苷酸序列是含有seqidno:2所示核苷酸序列和seqidno:3所示核苷酸序列的核苷酸序列;
14.上述第12项所述的方法,其中源自一对tol1转座子的核苷酸序列是seqidno:14所示核苷酸序列和seqidno:15所示核苷酸序列;
15.能够生产目的蛋白质的悬浮性哺乳动物细胞,其中导入含有基因片段和位于该基因片段两端的转座子序列的蛋白质表达载体,以将插入在一对转座子序列之间的基因片段整合入染色体,所述基因片段含有编码目的蛋白质的dna和选择性标记基因;
16.能够生产目的蛋白质的悬浮性哺乳动物细胞,其中同时导入含有基因片段及位于该基因片段两端的转座子序列的表达载体(a)和含有编码转座酶(转移酶)的dna的表达载体(b),以将插入在一对转座序列之间的基因片段整合入染色体,所述基因片段含有编码目的蛋白质的dna和选择性标记基因,而所述转座酶识别转座子序列并具有将插入在一对转座子序列之间的基因片段转移入染色体的活性;
17.上述第15或16项所述的细胞,其中细胞是能够在无血清培养基中存活和增殖的细胞;
18.上述第15至17项中任一项所述的细胞,其中细胞是选自悬浮性cho细胞、per.c6细胞、大鼠骨髓瘤细胞yb2/3hl.p2.g11.16ag.20(或也称为yb2/0)和适应于悬浮培养的悬浮性小鼠骨髓瘤细胞ns0的至少一种悬浮性哺乳动物细胞,其中所述悬浮性cho细胞中的cho细胞适应于悬浮培养;
19.上述第18项所述的细胞,其中cho细胞是选自cho-k1、cho-k1sv、dukxb11、cho/dg44、pro-3和cho-s的至少一种细胞;
20.上述第15至19项中任一项所述的细胞,其中选择性标记基因是环己酰亚胺抗性基因;
21.上述第20项所述的细胞,其中环己酰亚胺抗性基因是编码人核糖体蛋白质l36a的突变体的基因;
22.上述第21项所述的细胞,其中突变体是人核糖体蛋白质l36a的第54位脯氨酸被其它氨基酸取代的突变体;
23.上述第22项所述的细胞,其中其它氨基酸是谷氨酰胺;
24.上述第15至23项中任一项所述的细胞,其中一对转座子序列是源自在哺乳动物细胞中发挥功能的一对dna型转座子的核苷酸序列;
25.上述第24项所述的细胞,其中源自一对dna型转座子的核苷酸序列是源自一对tol1转座子的核苷酸序列或源自一对tol2转座子的核苷酸序列;
26.上述第25项所述的细胞,其中源自一对tol2转座子的核苷酸序列是seqidno:2所示核苷酸序列和seqidno:3所示核苷酸序列;
27.上述第25项所述的细胞,其中源自一对tol1转座子的核苷酸序列是seqidno:14所示核苷酸序列和seqidno:15所示核苷酸序列;
28.蛋白质表达载体,其含有基因片段和位于该基因片段两端的一对转座子序列,其中所述基因片段含有编码目的蛋白质的dna和选择性标记基因;
29.上述第28项所述的蛋白质表达载体,其中一对转座子序列是源自一对tol1转座子的核苷酸序列或源自一对tol2转座子的核苷酸序列;
30.上述第29项所述的蛋白质表达载体,其中源自一对tol2转座子的核苷酸序列是seqidno:2所示核苷酸序列和seqidno:3所示核苷酸序列;
31.上述第29项所述的蛋白质表达载体,其中源自一对tol1转座子的核苷酸序列是seqidno:14所示核苷酸序列和seqidno:15所示核苷酸序列。
发明的有益效果
根据本发明的蛋白质生产方法,可以利用哺乳动物细胞有效生产目标蛋白质。此外,本发明的细胞可以作为蛋白质生产细胞用于高效生产重组蛋白质。
附图说明
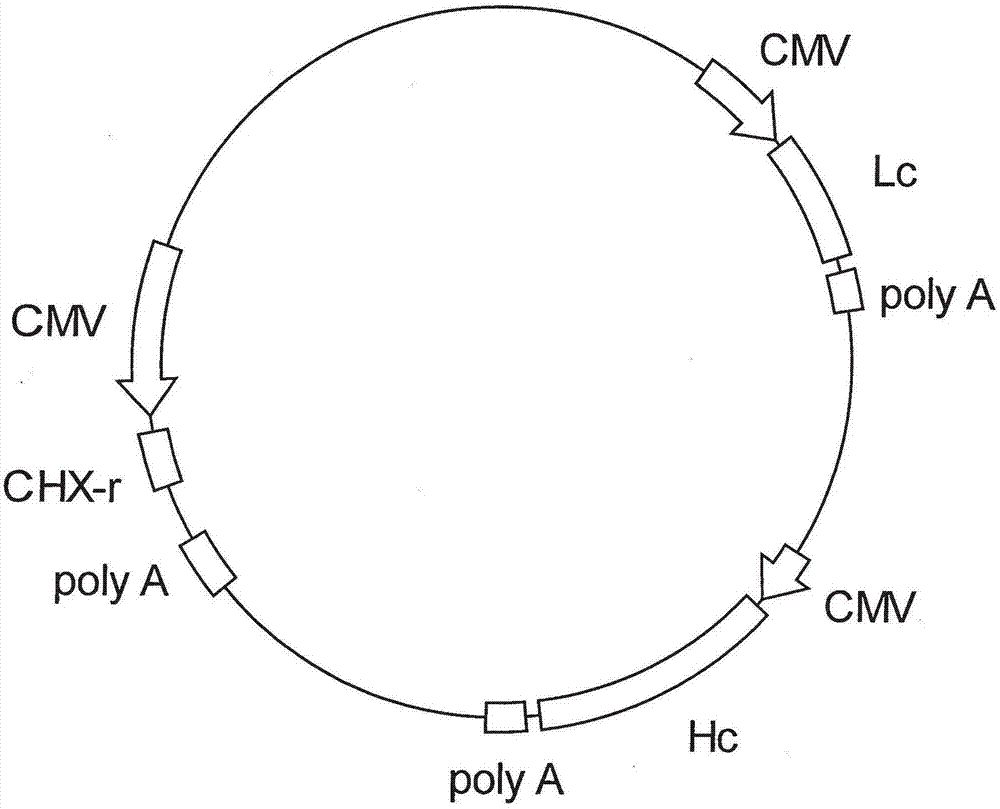
图1显示用于表达抗人流感m2抗体的转座子载体的示意图。tol2-l表示tol2转座子的左端(seqidno:2),tol2-r表示tol2转座子的右端(seqidno:3),cmv表示cmv启动子,polya表示多聚腺苷酸化位点,hc表示人类抗体h链cdna,lc表示人类抗体l链cdna,和chx-r表示环己酰亚胺抗性基因。
图2显示抗人流感m2抗体表达载体的示意图。cmv表示cmv启动子,polya表示多聚腺苷酸化位点,hc表示人类抗体h链cdna,lc表示人类抗体l链cdna和chx-r表示环己酰亚胺抗性基因。
图3显示tol2转座酶表达载体的示意图。caggs表示caggs启动子,polya表示多聚腺苷酸化位点,和tpasecdna表示tol2转座酶cdna。
图4a显示当使用表达抗人流感m2抗体的tol2转座子载体时,检测悬浮性cho-k1细胞中抗人流感m2抗体表达水平的结果。纵坐标表示抗体生产量(μg/ml),和横坐标表示悬浮性cho-k1细胞的转基因克隆数量。
图4b显示当使用表达抗人流感m2抗体的tol2转座子载体时,检测粘附性cho-k1细胞中抗人流感m2抗体表达水平的结果。纵坐标表示抗体生产量(μg/ml),和横坐标表示粘附性cho-k1细胞的转基因克隆数量。
图5显示表达抗人流感m2抗体的tol1转座子载体的示意图。tol1-l表示tol1转座子的左端(seqidno:14),tol1-r表示tol1转座子的右端(seqidno:15),cmv表示cmv启动子,polya表示多聚腺苷酸化位点,hc表示人类抗体h链cdna,lc表示人类抗体l链cdna,和chx-r表示环己酰亚胺抗性基因。
图6显示tol1转座酶表达载体的示意图。caggs表示caggs启动子,polya表示多聚腺苷酸化位点,和tpasecdna表示tol1转座酶cdna。
图7显示当使用表达抗人流感m2抗体的tol1转座子载体时,检测悬浮性cho-k1细胞中抗人流感m2抗体表达水平的结果。纵坐标表示抗体生产量(μg/ml),和横坐标表示悬浮性cho-k1细胞的转基因克隆数量。
具体实施方式
本发明涉及生产目的蛋白质的方法,其包括将含有基因片段和位于该基因片段两端的转座子序列的蛋白质表达载体导入悬浮性哺乳动物细胞,其中所述基因片段含有编码目的蛋白质的dna和选择性标记基因;将插入在一对(两个)转座子序列之间的基因片段整合入该哺乳动物细胞的染色体以获得能够表达目的蛋白质的哺乳动物细胞;和悬浮培养该哺乳动物细胞。
本发明生产目的蛋白质的方法的实例包括包含下列步骤(a)至(c)的方法:
(a)将下列表达载体(a)和(b)同时导入悬浮性哺乳动物细胞的步骤:
(a)含有基因片段和位于该基因片段两端的转座子序列的表达载体,其中所述基因片段含有编码目的蛋白质的dna,
(b)含有编码转座酶的dna的表达载体,其中所述转座酶识别转座子序列并具有将插入在一对转座子序列之间的基因片段转移入染色体的活性,
(b)从步骤(a)中所导入的表达载体瞬间表达转座酶、以将插入在一对转座子序列之间的基因片段整合入该哺乳动物细胞的染色体而获得能够表达目的蛋白质的悬浮性哺乳动物细胞的步骤,和
(c)悬浮培养步骤(b)中获得的能够表达目的蛋白质的悬浮性哺乳动物细胞以生产目的蛋白质的步骤;
此外,本发明涉及能够生产目的蛋白质的悬浮性哺乳动物细胞,其中导入含有基因片段及位于该基因片段两端的转座子序列的蛋白质表达载体,以将插入在一对转座子序列之间的基因片段整合入染色体,所述基因片段含有编码目的蛋白质的dna和选择性标记基因。
此外,本发明涉及能够生产目的蛋白质的悬浮性哺乳动物细胞,所述细胞中同时导入含有基因片段和位于该基因片段两端的转座子序列的表达载体(a)以及含有编码转座酶(转移酶)的dna的表达载体(b),以将插入一对转座序列之间的基因片段整合入染色体,所述基因片段含有编码目的蛋白质的dna和选择性标记基因,而所述转座酶识别转座子序列并具有将插入在一对转座子序列之间的基因片段转移入染色体的活性。
本说明书中的术语“转座子”是可转座的基因元件,是指在染色体上迁移或从一个染色体迁移到其它染色体(转座)同时保持一定结构的基因单元。
转座子含有重复转座子序列(也称为反向重复序列(ir序列)或末端反向重复序列(tir序列))的基因单元和编码转座酶的核苷酸序列,其中所述重复转座子序列以相同方向或相反方向位于基因单元的两端,其中所述转座酶识别转座子位点以将存在于转座子序列之间的基因转移。
从转座子翻译的转座酶可以通过识别转座子两端的转座子序列、切掉插入在一对转座子序列之间的dna片段并将该片段插入待转移的位点,从而转移dna。
本说明书中的术语“转座子序列”指被转座酶识别的转座子的核苷酸序列,其与ir序列或tir序列的意思相同。含有该核苷酸序列的dna可以含有不完全重复的部分,只要其可以通过转座酶的活性被转移(插入基因组中的其它位置)并含有对转座酶特异的转座子序列即可。
如本发明所用的转座子序列,优选为源自dna型转座子的核苷酸序列,且更优选为源自一对天然或人工dna型转座子的核苷酸序列,其可以被转座酶识别并在哺乳动物细胞中转座。
源自dna型转座子的核苷酸序列的实例包括源自青鳉鱼源tol1转座子和tol2转座子的核苷酸序列、从鲑鱼科基因组中存在的非自主转座子再构建的sleepingbeauty、蛙源人工转座子frogprince和昆虫源转座子piggybac。
其中,特别优选源自含有seqidno:6所示核苷酸序列的青鳉鱼源tol2转座子和含有seqidno:13所示核苷酸序列的青鳉鱼源tol1转座子的核苷酸序列。
源自一对tol2转座子的核苷酸序列的实例包括序列表seqidno:6所示tol2转座子核苷酸序列中第1至2299位核苷酸序列和第4148至4682位核苷酸序列。
作为源自一对tol2转座子的核苷酸序列,更优选序列表seqidno:1所示tol2转座子核苷酸序列中第1至200位核苷酸序列(seqidno:2)(以下称作“tol2-l序列”)和第2285至2788位核苷酸序列(seqidno:3)(以下称作“tol2-r序列”)。
源自一对tol1转座子的核苷酸序列的实例包括含有序列表seqidno:13所示tol1转座子核苷酸序列中第1至157位核苷酸序列和第1748至1855位核苷酸序列的核苷酸序列。
作为源自一对tol1转座子的核苷酸序列,更优选序列表seqidno:13所示tol1转座子核苷酸序列中第1至200位核苷酸序列(seqidno:14)(以下称作“tol1-l序列”)和第1351至1855位核苷酸序列(seqidno:15)(以下称作“tol1-r序列”)。
本发明所用的转座子序列的实例包括可以利用源自上述转座子的转座子序列的部分序列,通过调整该核苷酸序列的长度和通过由添加、缺失或取代修饰该核苷酸序列,从而控制其转移反应。
至于转座子的转移反应的控制,可以分别通过促进或抑制转座酶对转座子序列的识别而促进或抑制转移反应。
本说明书中的术语“转座酶”指识别具有转座子序列的核苷酸序列并将存在于核苷酸序列间的dna转移入染色体或从该染色体转移到其它染色体的酶。
转座酶的实例包括源自青鳉鱼的tol1和tol2、从鲑鱼科基因组中存在的非自主转座子再构建的sleepingbeauty、源自蛙的人工转座子frogprince和源自昆虫的转座子piggybac。
作为转座酶,可以用天然酶,并可以用部分氨基酸被取代、缺失、插入和/或添加的任何转座酶,只要其保持与转座酶相同的转移活性即可。通过控制转座酶的酶活性,可以控制存在于转座子序列间的dna的转移反应。
为了分析它是否具有与转座酶相同的转移活性,可以用日本国特开2003-235575号公报中公开的2-成分分析系统测定。
具体地,可以分别利用含有tol2转座酶缺失的tol2转座子(tol2源非自主转座子)的质粒和含有tol2转座酶的质粒,分析非自主tol2元件是否可以通过转座酶的活性被转移并插入到哺乳动物细胞的染色体。
本说明书中的术语“非自主转座子”指缺失了存在于转座子内的转座酶并因此不能进行其自主转移的转座子。通过允许转座酶蛋白质、编码转座酶蛋白质的mrna或编码转座酶蛋白质的dna同时存在于宿主细胞内,非自主转座子可以将插入在非自主转座子的转座子序列间的dna转移到宿主细胞的染色体中。
转座酶基因指编码转座酶的基因。为了改进其在哺乳动物细胞中的表达效率,可以将调整kozak共有序列(kozakm.,nucleicacidsres.,12,857-872(1984))或核糖体结合序列、shine-dalgarno序列与起始密码子之间的间隔调整到适当距离(如6至18个碱基)的序列连接到该基因的翻译起始密码子atg的上游位点。
根据本发明的方法,为了将表达载体中含有编码目的蛋白质的dna和选择性标记基因的基因片段整合入宿主细胞的染色体,将含有基因片段及位于该基因片段两端的转座子序列的表达载体导入宿主细胞,所述基因片段含有编码目的蛋白质的dna和选择性标记基因,并允许转座酶作用于导入细胞的表达载体中所包含的转座子序列。
为了允许转座酶作用于导入细胞的表达载体中包含的转座子序列,可以将转座酶注射入细胞,或将含有编码转座酶的dna的表达载体与含有编码目的蛋白质的dna和选择性标记基因的表达载体一起导入宿主细胞。此外,通过将编码转座酶基因的rna导入宿主细胞,可以在该细胞中表达转座酶。
不具体限定表达载体。可以根据导入含有转座酶基因的表达载体的宿主细胞、用途等,通过从本领域技术人员已知的表达载体中任意选择而使用任何表达载体。
为了用本发明方法生产由两个以上多肽构建的蛋白质,通过将编码该两个以上多肽的dna整合入相同或不同表达载体,然后将表达载体导入宿主细胞,可以将dna整合入细胞的染色体中。
可以将转座酶插入表达载体以与目的蛋白质一起表达,或可以将转座酶插入与表达载体不同的载体。可以允许转座酶瞬时起作用或持续起作用,但为了制备稳定生产的细胞,优选允许转座酶瞬时起作用。
作为允许转座酶瞬时起作用的方法,实例包括以下方法,其包括:制备含有编码转座酶的dna的表达载体和含有编码目的蛋白质的dna的表达载体,然后将两个表达质粒同时导入宿主细胞。
本说明书中的术语“表达载体”指用于导入哺乳动物细胞以表达目的蛋白质的表达载体。本发明所用的表达载体具有在表达盒两侧存在至少一对转座子序列的结构。
本说明书中术语“表达盒”指具有表达目的蛋白质所必须的基因表达控制区域和编码目的蛋白质的序列的核苷酸序列。基因表达控制区域的实例包括增强子、启动子和终止子。表达盒可以包含选择性标记基因。
可以使用任何启动子,只要其在动物细胞中发挥功能即可。实例包括巨细胞病毒(cmv)的ie(立即早期)基因的启动子、sv40早期启动子、逆转录病毒启动子、金属硫蛋白启动子、热休克启动子、srα启动子、莫洛尼鼠白血病病毒、增强子等。并且,人cmv的ie基因的增强子可以与该启动子一起使用。
“选择性标记基因”指可以用于区分导入质粒载体的细胞和缺乏该载体的细胞的任意其它基因。
选择性标记基因的实例包括抗药基因(新霉素抗性基因、dhfr基因、嘌呤霉素抗性基因、杀稻瘟菌素抗性基因、潮霉素抗性基因、和环己酰亚胺抗性基因(日本国特开2002-2682879号公报))、荧光和生物发光标记基因(如绿色荧光蛋白gfp)等。
本发明中,优选的选择性标记是抗药基因,且特别优选的选择性标记为环己酰亚胺抗性基因。此外,通过对选择性标记基因进行基因修饰,也可以改变选择性标记蛋白质的抗药性能和发光性能。
环己酰亚胺(以下有时称为chx)是蛋白质合成抑制剂,作为用chx抗性基因作为选择性标记基因的实例,酵母(kondok.j.bacteriol.,177,247171-7177(1995))和动物细胞(日本国特开2002-262879号公报)的例子是公知的。
在动物细胞的例子中,已发现对环己酰亚胺的抗性是由转化体提供的,其中所述转化体表达由序列表的seqidno:7所示核苷酸序列编码的蛋白质,在该蛋白质中由序列表的seqidno:5所示核苷酸序列编码的人核糖体蛋白质亚基l36a中的第54位脯氨酸被谷氨酰胺取代。
没有具体限定导入上述含有转座子序列的蛋白质表达载体、转座酶表达质粒载体和rna的方法。实例包括磷酸钙转染、电穿孔、脂质体方法、基因枪方法、脂质体转染等。
直接以蛋白质形式导入转座酶的方法的实例包括通过显微注射或内吞作用供入细胞。可以使用《新基因工程手册》中所述方法进行基因转移,该手册由村松正实和山本雅所著,由羊土社出版,isbn9784897063737。
宿主细胞可以是任何哺乳动物细胞,只要其可以传代培养并稳定表达目的蛋白质即可。宿主细胞的实例包括per.c6细胞、人白血病细胞namalwa细胞、猴细胞cos细胞、大鼠骨髓瘤细胞yb2/3hl.p2.g11.16ag.20(也称为yb2/0)、小鼠骨髓瘤细胞ns0、小鼠骨髓瘤细胞sp2/0-ag14、叙利亚仓鼠细胞bhk、hbt5637(日本国特开昭63-000299号公报)、中华仓鼠卵巢细胞cho细胞(journalofexperimentalmedicine,108,945(1958);proc.natl.acad.sci.usa,601275(1968);genetics,55,513(1968);chromosoma,41,129(1973);methodsincellscience,18,115(1996);radiationresearch,148,260(1997);proc.natl.acad.sci.usa,77,4216(1980);proc.natl.acad.sci.,60,1275(1968);cell,6,121(1975);molecularcellgenetics,appendixi,ii(pp.883-900))、cho/dg44、cho-k1(atccccl-61)、dukxb11(atccccl-9096)、pro-5(atccccl-1781)、cho-s(lifetechnologies,目录号11619)、pro-3和cho細胞亚株。
此外,通过改变上述宿主细胞以适合蛋白质生产、修饰染色体dna、导入外源基因等,上述宿主细胞也可以用于本发明的蛋白质生产方法。
此外,为了控制与所生产的目的蛋白质结合的糖链的结构,也可以使用获得凝集素抗性的lec13[somaticcellandmoleculargenetics,12,55(1986)]和缺失α1,6-岩藻糖基转移酶基因的cho细胞(wo2005/35586,wo2002/31140)作为宿主细胞。
目的蛋白质可以是任何蛋白质,只要其可以用本发明方法表达即可。具体的,实例包括人血清蛋白、肽激素、生长因子、细胞因子、凝血因子、纤溶系统蛋白质、抗体、和各种蛋白质的部分片段、等等。
目的蛋白质的优选实例包括单克隆抗体,如嵌合抗体、人源化抗体和人抗体,fc融合蛋白,和白蛋白结合蛋白,及其片段。
可以用各种方法控制用本发明方法获得的单克隆抗体的效应子活性。例如,已知方法是用于控制岩藻糖(以下也称为核心岩藻糖)量的方法,,其中所述岩藻糖通过与抗体fc区域第297位天冬酰胺(asn)结合的复合型n-连接糖链的还原端的α-1,6键与n-乙酰葡萄糖胺(glcnac)相连(wo2005/035586,wo2002/31140,和wo00/61739);通过改变抗体fc区域的氨基酸基团来控制单克隆抗体的效应子活性的方法,等等。可以用上述任何方法控制用本发明方法生产的单克隆抗体的效应子活性。
“效应子活性”指由抗体fc区域引起的抗体依赖活性。作为效应子活性,已知的有抗体依赖性细胞毒(adcc活性)、补体依赖性细胞毒(cdc活性)、吞噬细胞如巨噬细胞或树突状细胞的抗体依赖性胞吞作用(adp活性)、等等。
此外,通过控制单克隆抗体fc区域的复合型n-连接糖链的核心岩藻糖含量,可以提高或降低抗体的效应子活性。
作为降低与抗体的fc相连的复合型n-连接糖链所连接的岩藻糖含量的方法,可以通过用缺乏编码α1,6-岩藻糖基转移酶基因的cho细胞表达抗体而获得无岩藻糖连接的抗体,该无岩藻糖连接的抗体具有高度adcc活性。
另一方面,作为提高与抗体fc相连的复合型n-连接糖链所连接的岩藻糖含量的方法,可以通过用导入编码α1,6-岩藻糖基转移酶的基因的宿主细胞表达抗体而获得岩藻糖连接的抗体,该岩藻糖连接的抗体具有的adcc活性低于无岩藻糖连接的抗体。
另外,通过改变抗体fc区域的氨基酸残基,可以提高或降低adcc活性或cdc活性。例如,可以用us2007/0148165所述的fc区域的氨基酸序列提高抗体的cdc活性。
另外,通过如6737056或7297775或7317091号美国专利所述改变氨基酸,可以提高或降低抗体的adcc活性或cdc活性。
本发明中的术语“悬浮性哺乳动物细胞”指不与用于促进培养细胞粘附的涂覆型细胞培养支持体如微珠、用于组织培养的培养容器(也称为组织培养或粘附培养容器等)等粘附、并可以通过悬浮于培养液中而存活和生长的细胞。
当细胞不与细胞培养支持体粘附时,其可以在培养液中以单细胞状态存活和生长或以两个以上细胞凝集形成的细胞团状态存活和生长。
此外,作为本发明所用的悬浮性哺乳动物细胞,优选在不含胎牛血清(以下称为fcs)等的无血清培养基可以存活和生长中并同时悬浮于培养液中不与细胞培养支持体粘附的细胞,并更优选悬浮于不含蛋白质的无蛋白质培养基中时可以存活和生长的哺乳动物细胞。
作为组织培养的培养容器,其可以是任何培养容器,如培养瓶、培养皿等,只要其施加了用于粘附培养的涂层即可。具体地,例如,可以用市售组织培养瓶(greiner制造)、粘附培养瓶(sumitomobakelite制造)等证实是否是悬浮性哺乳动物细胞。
作为本发明所用的悬浮性哺乳动物细胞,其可以是通过使原本具有悬浮特性的细胞进一步适应于悬浮培养而制备的细胞,或者通过使粘附性哺乳动物细胞适应于悬浮培养条件制备的悬浮性哺乳动物细胞。
原本具有悬浮特性的细胞的实例包括per.c6细胞、大鼠骨髓瘤细胞yb2/3hl.p2.g11.16ag.20(或也称为yb2/0)、cho-s细胞(invitrogen制造)等。
上述“通过使粘附性哺乳动物细胞适应于悬浮培养条件制备的悬浮性哺乳动物细胞”可以用mol.biotechnol.,2000,15(3),249-57中所述的方法或以下所示方法制备,并可以通过建立表现出增殖特性和存活特性与悬浮培养适应前相似或优于适应于悬浮培养前的细胞而制备(j.biotechnol.2007,130(3),282-90)。
术语“与悬浮培养适应前相似”指适应于悬浮培养的细胞的存活率、增殖速率(倍增时间)等与适应悬浮培养前的细胞基本相同。
根据本发明使粘附性哺乳动物细胞适应于悬浮培养条件的方法的实例包括下列方法。将含血清培养基的血清含量降低到1/10,并以较高细胞浓度反复传代培养。当哺乳动物细胞开始能够存活和增殖时,进一步降低血清含量并反复传代培养。通过该方法可以制备在无血清状态下能够存活和增殖的悬浮性哺乳动物细胞。
此外,也可以通过以下方法制备悬浮性哺乳动物细胞,所述方法包括在培养液中添加适当的非离子表面活性剂如pluronic-f68等而培养。
通过适应悬浮培养条件获得悬浮特性的粘附性哺乳动物细胞的实例包括小鼠骨髓瘤细胞ns0、cho细胞等。
本发明中,作为悬浮性哺乳动物细胞所具有的特性,当悬浮培养2×105细胞/ml的细胞时,培养3或4天后的细胞浓度优选为5×105细胞/ml以上,更优选为8×105细胞/ml以上,特别优选为1×106细胞/ml以上,最优选为1.5×106细胞/ml以上。
此外,本发明的悬浮性哺乳动物细胞的倍增时间优选为48小时以下,更优选为24小时以下,特别优选为18小时以下,最优选为11小时以下。
用于悬浮培养的培养基的实例包括市售培养基,如cd-cho培养基(invitrogen制造),ex-cell325-pf培养基(safcbiosciences制造)、sfm4cho培养基(hyclone制造)等。此外,也可以通过混合培养哺乳动物细胞所必需的糖、氨基酸等而获得。
可以使用在能够悬浮培养的培养条件下可以用于悬浮培养的培养容器来培养悬浮性哺乳动物细胞。培养容器的实例包括用于细胞培养的96孔板(corning制造)、t-培养瓶(bectondickinson制造)、erlenmeyer培养瓶(corning制造)等。
至于培养条件,例如,其可以在5%co2的气氛中和37℃培养温度下静置培养。也可以使用振荡培养设备,如专用于悬浮培养的培养设备wavebioreactor(gehealthcarebioscience制造)。
至于使用wavebioreactor设备时悬浮性哺乳动物细胞的悬浮培养条件,可以通过gehealthcarebioscience主页http://www.gelifesciences.co.jp/tech_support/manual/pdf/cellcult/wave_03_16.pdf中所述的方法培养细胞。
除振荡培养外,也可以用旋转搅拌设备如生物反应器培养。可以通过cytotechnology(2006)52:199-207中所述方法等用生物反应器进行培养。
本发明中,当用悬浮性哺乳动物细胞以外的细胞株时,可以用任何细胞株,只要其是通过上述方法适应于悬浮培养的哺乳动物细胞并是可以用于本发明的蛋白质生产方法的细胞株即可。
通过在含有目的蛋白质的培养液或细胞匀浆中将目的蛋白质与目的蛋白质以外的杂质分离,来执行纯化悬浮性哺乳动物细胞生产的目的蛋白质。分离方法的实例包括离心、渗析、硫酸铵沉淀、柱层析、过滤等。可以基于目的蛋白质与杂质的理化性质差异和它们对柱载体的亲和性差异进行分离。
纯化目的蛋白质的方法可以,例如,通过《蛋白质实验概述》(第一卷)-重组蛋白质的提取、分离和表达(日文版书籍的翻译)(冈田雅人和宫崎香编辑,羊土社出版,isbn9784897069180)记载的方法实施。
本文所引用的文献、如科学论文、专利、专利申请的全部内容分别通过在与所具体描述内容同等的程度上进行参考纳入本文。
为易于理解,前文通过说明优选实施方案介绍了本发明。以下,基于实施例进一步具体介绍本发明,但提供上述阐述和以下实施例仅用于例示目的而不是出于限定本发明的目的。相应地,本发明的范围不限于本文具体介绍的实施方案和实施例,而仅由权利要求限定。
下文所述与基因重组有关的各种实验技术,如克隆等,根据下列文献记载的基因工程技术实施:j.sambrook,e.f.frisch和t.maniatis所著的molecularcloning第二版,和currentprotocols出版的由frederickm.ausubel等所著的currentprotocolsinmolecularbiology等。
实施例
[实施例1]
表达抗人流感m2抗体的转座子载体的制备
用含有哺乳动物细胞基因表达盒的质粒作为蛋白质表达的质粒载体,该质粒含有插入在一对tol2转座子序列之间的任意人类抗体基因和抗药标记基因。
所用基因的各dna是基于已知核苷酸序列人工化学合成,或通过制备其两端序列的引物并然后用合适的dna源作为模板进行pcr而获得。为了进行后期基因操作,向引物末端加入限制性酶的限制性位点。
在日本国特开2003-235575/号公报中所公开的非自主tol2转座子的核苷酸序列(seqidno:1)中,用第1-200位核苷酸序列(tol2-l序列)(seqidno:2)和第2285至2788位核苷酸序列(tol2-r序列)(seqidno:3)作为转座子序列。
用下列方法制备含有一对转座子序列(takarabioinc.制造)的各合成dna片段。制备含有在tol2-r序列的5’端和3’端都连接有限制性酶nrui识别序列的核苷酸序列的dna片段。然后制备含有在tol2-l序列5’端连接有fsei限制性酶识别序列并在其3’端连接有asci限制性酶识别序列的核苷酸序列的dna片段。
接着,将由此制备的含有tol2-r序列和tol2-l序列的dna片段插入表达载体n5lg1-m2-z3载体(wo2006/061723),所述n5lg1-m2-z3载体含有编码抗人流感m2抗体z3g1的氨基酸序列的核苷酸序列。
用n5lg1-m2-z3载体(wo2006/061723)作为抗体基因表达盒,所述n5lg1-m2-z3载体中在cmv增强子/启动子的控制下被插入了编码抗人流感m2抗体z3g1(atcc保藏号pta-5968:2004年3月13日保藏,美国典型培养物保藏中心(americantypeculturecollection),manassas,va,美国)的h链的核苷酸序列(seqidno:8)和编码其l链(seqidno:9)的核苷酸序列(seqidno:10和seqidno:11)。
将含有tol2-r序列的dna片段插入在n5lg1-m2-z3载体中含有抗体基因表达盒和抗性标记基因的基因片段5’端侧的限制性酶nrui位点中。然后,将含有tol2-l序列的dna片段插入到在3’端侧的限制性酶fsei和asci位点中。
此外,在cmv增强子/启动子的控制下,通过将环己酰亚胺抗性基因表达盒插入n5lg1-m2-z3载体的fsei识别位点而构建表达抗人流感m2抗体的转座子载体(图1),其中所述环己酰亚胺抗性基因表达盒与编码环己酰亚胺抗性基因(人核糖体蛋白质l36a第54位脯氨酸被谷氨酰胺取代的基因)的核苷酸序列(seqidno:5)相连,其中所述fsei识别位点与tol2转座子序列相连。
另一方面,将不包含转座子序列的载体命名为抗人流感m2抗体表达载体并用作对照载体(图2)。
[实施例2]
转座酶表达载体的制备
用与目的抗体表达载体无关的表达载体来表达转座酶。即,将编码青鳉鱼来源的tol2转座酶(seqidno:4)的基因插入pcaggs载体(gene,108,193-200,1991)的caggs启动子下游,并用作表达载体(图3)。
[实施例3]
悬浮性cho细胞的制备
将已用含10%血清(fcs)的α-mem培养基(invitrogen制造)培养的粘附性cho细胞剥离并通过胰蛋白酶处理而回收,并用含10%fcs的新鲜α-mem培养基在5%co2培养器中37℃振荡培养。若干天后,确认这些细胞的生长,然后以2×105细胞/ml的浓度将它们接种到含有5%fcs的α-mem培养基中进行振荡培养。
再若干天后,用含有5%fcs的α-mem培养基同样进行接种。最后,用下述方法制备适应于悬浮培养的细胞:用无血清的α-mem培养基反复传代培养和振荡培养,并确认细胞与血清存在下它们的培养情况具有相同的生长能力。
(2)产抗体的cho细胞的制备
用实施例1和实施例2中所制备的表达抗人流感m2抗体的转座子载体(以下称为转座子载体)和tol2转座酶表达载体pcaggs-t2tp(图3,kawakamik.&nodat.,genetics,166,895-899(2004))作为表达载体。此外,用无转座子序列的抗人流感m2抗体表达载体作为对照。
通过将上述表达载体导入适应悬浮培养的cho-k1细胞(美国典型培养物保藏中心,目录号ccl-61)或hek293细胞(freestyle293f细胞,invitrogen)比较获得环己酰亚胺抗性克隆的频次。
将各细胞(4×106细胞)悬浮于400μlpbs,用电穿孔法直接以环状dna形式共转染表达抗人流感m2抗体的转座子载体(10μg)和tol2转座酶表达载体(25μg)。在这一点上,为瞬时表达tol2转座酶,将tol2转座酶表达载体直接以环状dna形式导入,以防止整合入宿主染色体。
此外,作为对照,按照电穿孔法的标准基因转移方法,用限制性酶使抗人流感m2抗体表达载体(10μg)线性化,然后导入各细胞。
在电压为300v、静电容量为500μf和室温的条件下,使用电穿孔仪(genepulserxcell系统(bio-rad制造)),用间隙宽度为4mm的电击杯(bio-rad制造)进行电穿孔。
用电穿孔法转染后,将各细胞接种到三个96孔板,并在co2培养器中培养3天,其中对cho细胞使用safcbiosciences制造的ex-cell325-pf培养基,对hek293细胞使用freestyle-293培养基(invitrogen制造)。
接着,从转染第4天培养基更换之日起,向培养基中加入3μg/ml环己酰亚胺,使得在环己酰亚胺存在下培养细胞,随后培养3周并每周进行培养基更换。
培养3周后,计数发现环己酰亚胺抗性克隆的孔数,结果显示在表1和表2中。
[表1]
表1环己酰亚胺抗性细胞(cho细胞)的数量比较
[表2]
表2环己酰亚胺抗性细胞(hek293细胞)的数量比较
如表1所示,将各抗人流感m2抗体表达转座子载体或抗人流感m2抗体表达载体导入悬浮性cho-k1细胞。结果,与其它细胞株的情况相似,没有从导入抗人流感m2抗体表达载体的细胞中获得环己酰亚胺抗性转化体,但是从导入表达抗人流感m2抗体的转座子载体的细胞中高频获得了环己酰亚胺抗性转化体。
另一方面,如表2所示,将表达抗人流感m2抗体的转座子载体或抗人流感m2抗体表达载体导入hek293细胞时,没有获得环己酰亚胺抗性转化体。
基于这些结果,发现插入在一对转座子序列之间的目的蛋白质编码基因和环己酰亚胺抗性基因被有效导入宿主细胞、即悬浮性哺乳动物细胞的染色体中。
(3)悬浮性cho细胞和粘附性cho细胞的抗体生产的检测
为检测悬浮性cho细胞或粘附性cho细胞的抗体生产效率,检测了各细胞株所生产的抗体量。使用适应于悬浮培养的悬浮性cho-k1细胞作为悬浮性cho细胞,此外,使用适应悬浮培养前的粘附性cho-k1细胞作为粘附性cho细胞。
用电穿孔法分别将抗人流感m2抗体表达转座子载体(10μg)和tol2转座酶表达载体(25μg)导入悬浮性cho-k1细胞和粘附性cho-k1细胞。之后,将悬浮性cho-k1细胞和粘附性cho-k1细胞各接种至96孔板。
对悬浮性cho-k1细胞使用悬浮细胞培养基(ex-cell325pf,safcbiosciences制造),对粘附性cho-k1细胞使用含10%血清的α-mem培养基。在co2培养器中将各细胞培养3天。转染第4天培养基更换之日起,向培养基中加入3μg/ml环己酰亚胺,以在环己酰亚胺存在下培养细胞,并进一步将细胞培养3周。这种情况下,每周进行培养基更换。
对于悬浮性cho-k1细胞,将1×106个细胞接种到6孔板并在co2培养器中振荡培养3天,用hplc测定培养上清液中抗人流感m2抗体蛋白质的量。
对于粘附性cho-k1细胞,当细胞在6孔板上达到汇合时(2×106个)进行培养基更换,静置培养3天后,用hplc测定培养上清液中抗体蛋白质的量。
根据yeastres.,(2007),1307-1316中所述的方法测定培养上清液中的抗体浓度。结果显示在图4a和图4b中。
如图4a所示,当使用适应于悬浮培养的cho-k1细胞时,获得大量表现出相当高抗体表达水平的细胞。另一方面,如图4b所示,当使用粘附性cho-k1细胞时,仅获得表现出hplc检测限度(5μg/ml)以下表达水平的细胞。
基于这些结果发现,对于用转座子载体表达目的蛋白质,用悬浮性哺乳动物细胞时可以高水平表达目的蛋白质。
另外,从实施例1至3的结果发现,通过用适应于悬浮培养的悬浮性哺乳动物细胞有效制备可以高度表达外源基因的生产细胞,本发明方法可以用作生产目的蛋白质的新方法。
[实施例4]
用于表达抗人流感m2抗体的tol转座子载体的制备
与实施例1的方式相同,用含有哺乳动物细胞基因表达盒的质粒作为蛋白质表达质粒载体,该质粒含有插入一对tol1转座子序列之间的任意人类抗体基因和抗药标记基因。
所用基因的各dna是基于已知序列信息人工化学合成的,或通过制备其两端序列的引物并使用合适dna源作为模板进行pcr而获得。为了后期进行的基因操作,向引物末端加入限制性酶切位点。
在序列表的seqidno:13所示的非自主tol1转座子核苷酸序列(wo2008/072540)中,用第1-200位核苷酸序列(tol1-l序列)(seqidno:14)和第1351至1855位核苷酸序列(tol1-r序列)(seqidno:15)作为转座子序列。
用下列方法制备各含有一对转座子序列的各合成dna片段。制备含有在tol1-r序列的5’端和3’端都连接有限制性酶nrui识别序列的核苷酸序列的dna片段,然后,制备含有在tol1-l序列5’端连接有fsei限制性酶识别序列并在其3’端连接有asci限制性酶识别序列的核苷酸序列的dna片段。
接着,将由此制备的含有tol1-r序列和tol1-l序列的dna片段插入表达载体n5lg1-m2-z3载体。将含有tol1-r序列的dna片段插入n5lg1-m2-z3载体的限制性酶nrui位点,该位点存在于含有抗体基因表达盒和抗性标记基因的基因片段的5’端一侧,并将含有tol1-l序列的dna片段插入存在于其3’端一侧的限制性酶fsei和asci位点中。
此外,在cmv增强子/启动子的控制下,通过将连有环己酰亚胺抗性基因(人核糖体蛋白质l36a第54位脯氨酸突变为谷氨酰胺的基因)的环己酰亚胺抗性基因表达盒插入n5lg1-m2-z3载体中与tol1转座子序列相连的fsei识别位点来构建表达抗人流感m2抗体的tol1转座子载体(图5)。
[实施例5]
tol1转座酶表达载体的制备
用与目的抗体表达载体无关的表达载体来表达转座酶。即,在cmv增强子/启动子的控制下将tol1转座酶基因表达盒插入pbluescriptiisk(+)(stratagene制造),并用作ptol1ase表达载体(图6),其中所述tol1转座酶基因表达盒与编码青鳉鱼来源的tol1转座酶的dna片段相连,含有序列表的seqidno:16所示核苷酸序列。
[实施例6]
(1)生产抗体的cho细胞的制备
用实施例4和实施例5的表达抗人流感m2抗体的tol1转座子载体(以下称为tol1转座子载体)和tol2转座酶表达载体ptol1ase作为表达载体。此外,以与实施例3(1)相同的方式通过适应悬浮培养制备的cho-k1细胞被用作细胞。
将上述表达载体导入适应于悬浮培养的cho-k1细胞,并测定获得环己酰亚胺抗性克隆的频次。将适应于悬浮培养的cho-k1细胞(4×106个细胞)悬浮于400μlpbs,用电穿孔法直接以环状dna形式共转染表达抗人流感m2抗体的tol1转座子载体(10μg)和tol1转座酶表达载体(50μg)。为了实现瞬时表达tol1转座酶,直接以环状dna形式导入tol1转座酶表达载体,以防止整合入宿主染色体。
在电压为300v、静电容量为500μf和室温的条件下,使用电穿孔仪(genepulserxcell系统(bio-rad制造)),用间隙宽度为4mm的电击杯(bio-rad制造)进行电穿孔。
用电穿孔法转染后,将各细胞接种到两个96孔板,并在co2培养器中对cho细胞使用ex-cell325-pf培养基(safcbiosciences制造)培养3天。接着,从转染第4天培养基更换之日起,向培养基中加入3μg/ml环己酰亚胺,以在环己酰亚胺存在下培养细胞,随后培养3周并每周进行培养基更换。
培养3周后,计数发现环己酰亚胺抗性克隆的孔数,结果显示在表3中。表3中实验1至实验3各显示了进行三次基因转移的结果。
[表3]
如表3所示,当将表达抗人流感m2抗体的tol1转座子载体导入悬浮性cho-k1细胞时,以高频率获得了环己酰亚胺抗性转化体,所述频率与导入表达抗人流感m2抗体的tol2转座子载体的实施例3相近。
基于这些结果发现,在使用tol1转座子的情况下,也将插入在一对转座子序列之间的抗体基因和环己酰亚胺抗性基因有效转导入宿主细胞即悬浮性哺乳动物细胞的染色体。
(2)悬浮性cho-k1细胞的抗体生产的检测
用悬浮性cho-k1细胞检测悬浮性cho-k1细胞的抗体生产效率。用电穿孔法将表达抗人流感m2抗体的转座子载体(10μg)和tol1转座酶表达载体(50μg)导入适应于悬浮培养的悬浮性cho-k1细胞。
之后,分别将细胞接种到两个96孔板,并用悬浮培养基ex-cee325pf在co2培养器中培养3天。从电穿孔后第4天更换培养基起,在3μg/ml环己酰亚胺存在下培养细胞3周。在此情况下,每周进行培养基更换。
对于悬浮性cho-k1细胞,将1×106个细胞接种到6孔板并在co2培养器中振荡培养3天,用hplc测定培养上清液中抗人流感m2抗体蛋白质的量。
根据yeastres.,(2007),1307-1316中所述的方法测定培养上清液中的抗体浓度。结果显示在图7中。
如图7所示,在使用tol1转座子的情况下,也获得大量表现出相当高的抗体表达水平的细胞。由该结果发现,与使用tol2转座子来源的核苷酸序列的情况相似,当用tol1转座子来源的核苷酸序列作为转座子序列时,也可以获得能够高度表达目的蛋白质的悬浮性哺乳动物细胞。
虽然已详细并参考具体实施方案介绍了本发明,但其中可以进行各种变化和修改而不脱离其精神和范围,这对于本领域技术人员来说是显而易见的。该申请是基于2009年6月11日提交的日本专利申请特愿2009-140626号和2009年6月11日提交的美国临时专利申请no.61/186138,通过引用的方式将这两件专利申请的全部内容纳入本申请,并且将本文引用的全部文献整体纳入。
工业实用性
通过本发明生产蛋白质的方法,可以用悬浮性哺乳动物细胞有效制造目的蛋白质。可以用本发明的细胞作为生产重组蛋白质的蛋白质生产细胞。
序列表自由文本
seqidno:1-人工序列的说明:非自主tol2转座子的核苷酸序列
seqidno:2-人工序列的说明:tol2-l序列
seqidno:3-人工序列的说明:tol2-r序列
seqidno:7-人工序列的说明:环己酰亚胺抗性基因的核苷酸序列
seqidno:8-人工序列的说明:环己酰亚胺抗性基因所编码的蛋白质的氨基酸序列
seqidno:9-人工序列的说明:编码m2z3抗体h链的核苷酸序列
seqidno:10-人工序列的说明:编码m2z3抗体h链的氨基酸序列
seqidno:11-人工序列的说明:编码m2z3抗体l链的核苷酸序列
seqidno:12-人工序列的说明:编码m2z3抗体l链的氨基酸序列
seqidno:13-人工序列的说明:非自主tol1转座子的核苷酸序列
seqidno:14-人工序列的说明:tol1-l序列
seqidno:15-人工序列的说明:tol1-r序列
序列表
<110>大学共同利用机关法人情报·系统研究机构
协和发酵麒麟株式会社
<120>生产蛋白质的方法
<130>spi170911-61
<150>jp2009-140626
<151>2009-06-11
<150>us61/186138
<151>2009-06-11
<160>17
<170>patentinversion3.3
<210>1
<211>2788
<212>dna
<213>artificial
<220>
<223>descriptionofartificialsequence;nonautologustol2transposon
<400>1
cagaggtgtaaagtacttgagtaattttacttgattactgtacttaagtattatttttgg60
ggatttttactttacttgagtacaattaaaaatcaatacttttacttttacttaattaca120
tttttttagaaaaaaaagtactttttactccttacaattttatttacagtcaaaaagtac180
ttattttttggagatcacttcattctattttcccttgctattaccaaaccaattgaattg240
cgctgatgcccagtttaatttaaatgttatttattctgcctatgaaaatcgttttcacat300
tatatgaaattggtcagacatgttcattggtcctttggaagtgacgtcatgtcacatcta360
ttaccacaatgcacagcaccttgacctggaaattagggaaattataacagtcaatcagtg420
gaagaaaatggaggaagtatgtgattcatcagcagctgcgagcagcacagtccaaaatca480
gccacaggatcaagagcacccgtggccgtatcttcgcgaattcttttctttaagtggtgt540
aaataaagattcattcaagatgaaatgtgtcctctgtctcccgcttaataaagaaatatc600
ggccttcaaaagttcgccatcaaacctaaggaagcatattgaggtaagtacattaagtat660
tttgttttactgatagtttttttttttttttttttttttttttttgggtgtgcatgtttt720
gacgttgatggcgcgccttttatatgtgtagtaggcctattttcactaatgcatgcgatt780
gacaatataaggctcacgtaataaaatgctaaaatgcatttgtaattggtaacgttaggt840
ccacgggaaatttggcgcctattgcagctttgaataatcattatcattccgtgctctcat900
tgtgtttgaattcatgcaaaacacaagaaaaccaagcgagaaatttttttccaaacatgt960
tgtattgtcaaaacggtaacactttacaatgaggttgattagttcatgtattaactaaca1020
ttaaataaccatgagcaatacatttgttactgtatctgttaatctttgttaacgttagtt1080
aatagaaatacagatgttcattgtttgttcatgttagttcacagtgcattaactaatgtt1140
aacaagatataaagtattagtaaatgttgaaattaacatgtatacgtgcagttcattatt1200
agttcatgttaactaatgtagttaactaacgaaccttattgtaaaagtgttaccatcaaa1260
actaatgtaatgaaatcaattcaccctgtcatgtcagccttacagtcctgtgtttttgtc1320
aatataatcagaaataaaattaatgtttgattgtcactaaatgctactgtatttctaaaa1380
tcaacaagtatttaacattataaagtgtgcaattggctgcaaatgtcagttttattaaag1440
ggttagttcacccaaaaatgaaaataatgtcattaatgactcgccctcatgtcgttccaa1500
gcccgtaagacctccgttcatcttcagaacacagtttaagatattttagatttagtccga1560
gagctttctgtgcctccattgagaatgtatgtacggtatactgtccatgtccagaaaggt1620
aataaaaacatcaaagtagtccatgtgacatcagtgggttagttagaattttttgaagca1680
tcgaatacattttggtccaaaaataacaaaacctacgactttattcggcattgtattctc1740
ttccgggtctgttgtcaatccgcgttcacgacttcgcagtgacgctacaatgctgaataa1800
agtcgtaggttttgttatttttggaccaaaatgtattttcgatgcttcaaataattctac1860
ctaacccactgatgtcacatggactactttgatgtttttattacctttctggacatggac1920
agtataccgtacatacattttcagtggagggacagaaagctctcggactaaatctaaaat1980
atcttaaactgtgttccgaagatgaacggaggtgttacgggcttggaacgacatgagggt2040
gagtcattaatgacatcttttcatttttgggtgaactaaccctttaatgctgtaatcaga2100
gagtgtatgtgtaattgttacatttattgcatacaatataaatatttatttgttgttttt2160
acagagaatgcacccaaattacctcaaaaactactctaaattgacagcacagaagagaaa2220
gatcgggacagatctcatatgctcgagggcccatctggcctgtgtttcagacaccaggga2280
gtctctgctcacgtttcctgctatttgcagcctctctatcaagactaatacacctcttcc2340
cgcatcggctgcctgtgagaggcttttcagcactgcaggattgcttttcagccccaaaag2400
agctaggcttgacactaacaattttgagaatcagcttctactgaagttaaatctgaggtt2460
ttacaactttgagtagcgtgtactggcattagattgtctgtcttatagtttgataattaa2520
atacaaacagttctaaagcaggataaaaccttgtatgcatttcatttaatgttttttgag2580
attaaaagcttaaacaagaatctctagttttctttcttgcttttacttttacttccttaa2640
tactcaagtacaattttaatggagtacttttttacttttactcaagtaagattctagcca2700
gatacttttacttttaattgagtaaaattttccctaagtacttgtactttcacttgagta2760
aaatttttgagtactttttacacctctg2788
<210>2
<211>200
<212>dna
<213>artificial
<220>
<223>descriptionofartificialsequence;tol2-ltransposonsequence
<400>2
cagaggtgtaaagtacttgagtaattttacttgattactgtacttaagtattatttttgg60
ggatttttactttacttgagtacaattaaaaatcaatacttttacttttacttaattaca120
tttttttagaaaaaaaagtactttttactccttacaattttatttacagtcaaaaagtac180
ttattttttggagatcactt200
<210>3
<211>504
<212>dna
<213>artificial
<220>
<223>descriptionofartificialsequence;tol2-rtransposonsequence
<400>3
ctgctcacgtttcctgctatttgcagcctctctatcaagactaatacacctcttcccgca60
tcggctgcctgtgagaggcttttcagcactgcaggattgcttttcagccccaaaagagct120
aggcttgacactaacaattttgagaatcagcttctactgaagttaaatctgaggttttac180
aactttgagtagcgtgtactggcattagattgtctgtcttatagtttgataattaaatac240
aaacagttctaaagcaggataaaaccttgtatgcatttcatttaatgttttttgagatta300
aaagcttaaacaagaatctctagttttctttcttgcttttacttttacttccttaatact360
caagtacaattttaatggagtacttttttacttttactcaagtaagattctagccagata420
cttttacttttaattgagtaaaattttccctaagtacttgtactttcacttgagtaaaat480
ttttgagtactttttacacctctg504
<210>4
<211>2156
<212>dna
<213>oryziaslatipes
<220>
<221>cds
<222>(85)..(2034)
<400>4
acgtcatgtcacatctattaccacaatgcacagcaccttgacctggaaattagggaaatt60
ataacagtcaatcagtggaagaaaatggaggaagtatgtgattcatcagca111
metglugluvalcysaspserserala
15
gctgcgagcagcacagtccaaaatcagccacaggatcaagagcacccg159
alaalaserserthrvalglnasnglnproglnaspglngluhispro
10152025
tggccgtatcttcgcgaattcttttctttaagtggtgtaaataaagat207
trpprotyrleuarggluphepheserleuserglyvalasnlysasp
303540
tcattcaagatgaaatgtgtcctctgtctcccgcttaataaagaaata255
serphelysmetlyscysvalleucysleuproleuasnlysgluile
455055
tcggccttcaaaagttcgccatcaaacctaaggaagcatattgagaga303
seralaphelysserserproserasnleuarglyshisilegluarg
606570
atgcacccaaattacctcaaaaactactctaaattgacagcacagaag351
methisproasntyrleulysasntyrserlysleuthralaglnlys
758085
agaaagatcgggacctccacccatgcttccagcagtaagcaactgaaa399
arglysileglythrserthrhisalaserserserlysglnleulys
9095100105
gttgactcagttttcccagtcaaacatgtgtctccagtcactgtgaac447
valaspservalpheprovallyshisvalserprovalthrvalasn
110115120
aaagctatattaaggtacatcattcaaggacttcatcctttcagcact495
lysalaileleuargtyrileileglnglyleuhispropheserthr
125130135
gttgatctgccatcatttaaagagctgattagtacactgcagcctggc543
valaspleuproserphelysgluleuileserthrleuglnprogly
140145150
atttctgtcattacaaggcctactttacgctccaagatagctgaagct591
ileservalilethrargprothrleuargserlysilealagluala
155160165
gctctgatcatgaaacagaaagtgactgctgccatgagtgaagttgaa639
alaleuilemetlysglnlysvalthralaalametsergluvalglu
170175180185
tggattgcaaccacaacggattgttggactgcacgtagaaagtcattc687
trpilealathrthrthraspcystrpthralaargarglysserphe
190195200
attggtgtaactgctcactggatcaaccctggaagtcttgaaagacat735
ileglyvalthralahistrpileasnproglyserleugluarghis
205210215
tccgctgcacttgcctgcaaaagattaatgggctctcatacttttgag783
seralaalaleualacyslysargleumetglyserhisthrpheglu
220225230
gtactggccagtgccatgaatgatatccactcagagtatgaaatacgt831
valleualaseralametasnaspilehisserglutyrgluilearg
235240245
gacaaggttgtttgcacaaccacagacagtggttccaactttatgaag879
asplysvalvalcysthrthrthraspserglyserasnphemetlys
250255260265
gctttcagagtttttggtgtggaaaacaatgatatcgagactgaggca927
alapheargvalpheglyvalgluasnasnaspilegluthrgluala
270275280
agaaggtgtgaaagtgatgacactgattctgaaggctgtggtgaggga975
argargcysgluseraspaspthraspsergluglycysglyglugly
285290295
agtgatggtgtggaattccaagatgcctcacgagtcctggaccaagac1023
seraspglyvalglupheglnaspalaserargvalleuaspglnasp
300305310
gatggcttcgaattccagctaccaaaacatcaaaagtgtgcctgtcac1071
aspglypheglupheglnleuprolyshisglnlyscysalacyshis
315320325
ttacttaacctagtctcaagcgttgatgcccaaaaagctctctcaaat1119
leuleuasnleuvalserservalaspalaglnlysalaleuserasn
330335340345
gaacactacaagaaactctacagatctgtctttggcaaatgccaagct1167
gluhistyrlyslysleutyrargservalpheglylyscysglnala
350355360
ttatggaataaaagcagccgatcggctctagcagctgaagctgttgaa1215
leutrpasnlysserserargseralaleualaalaglualavalglu
365370375
tcagaaagccggcttcagcttttaaggccaaaccaaacgcggtggaat1263
sergluserargleuglnleuleuargproasnglnthrargtrpasn
380385390
tcaacttttatggctgttgacagaattcttcaaatttgcaaagaagca1311
serthrphemetalavalaspargileleuglnilecyslysgluala
395400405
ggagaaggcgcacttcggaatatatgcacctctcttgaggttccaatg1359
glygluglyalaleuargasnilecysthrserleugluvalpromet
410415420425
tttaatccagcagaaatgctgttcttgacagagtgggccaacacaatg1407
pheasnproalaglumetleupheleuthrglutrpalaasnthrmet
430435440
cgtccagttgcaaaagtactcgacatcttgcaagcggaaacgaataca1455
argprovalalalysvalleuaspileleuglnalagluthrasnthr
445450455
cagctggggtggctgctgcctagtgtccatcagttaagcttgaaactt1503
glnleuglytrpleuleuproservalhisglnleuserleulysleu
460465470
cagcgactccaccattctctcaggtactgtgacccacttgtggatgcc1551
glnargleuhishisserleuargtyrcysaspproleuvalaspala
475480485
ctacaacaaggaatccaaacacgattcaagcatatgtttgaagatcct1599
leuglnglnglyileglnthrargphelyshismetphegluasppro
490495500505
gagatcatagcagctgccatccttctccctaaatttcggacctcttgg1647
gluileilealaalaalaileleuleuprolyspheargthrsertrp
510515520
acaaatgatgaaaccatcataaaacgaggcatggactacatcagagtg1695
thrasnaspgluthrileilelysargglymetasptyrileargval
525530535
catctggagcctttggaccacaagaaggaattggccaacagttcatct1743
hisleugluproleuasphislyslysgluleualaasnserserser
540545550
gatgatgaagattttttcgcttctttgaaaccgacaacacatgaagcc1791
aspaspgluaspphephealaserleulysprothrthrhisgluala
555560565
agcaaagagttggatggatatctggcctgtgtttcagacaccagggag1839
serlysgluleuaspglytyrleualacysvalseraspthrargglu
570575580585
tctctgctcacgtttcctgctatttgcagcctctctatcaagactaat1887
serleuleuthrpheproalailecysserleuserilelysthrasn
590595600
acacctcttcccgcatcggctgcctgtgagaggcttttcagcactgca1935
thrproleuproalaseralaalacysgluargleupheserthrala
605610615
ggattgcttttcagccccaaaagagctaggcttgacactaacaatttt1983
glyleuleupheserprolysargalaargleuaspthrasnasnphe
620625630
gagaatcagcttctactgaagttaaatctgaggttttacaactttgag2031
gluasnglnleuleuleulysleuasnleuargphetyrasnpheglu
635640645
tagcgtgtactggcattagattgtctgtcttatagtttgataattaaatacaa2084
acagttctaaagcaggataaaaccttgtatgcatttcatttaatgttttttgagattaaa2144
agcttaaacaag2156
<210>5
<211>649
<212>prt
<213>oryziaslatipes
<400>5
metglugluvalcysaspserseralaalaalaserserthrvalgln
151015
asnglnproglnaspglngluhisprotrpprotyrleuarggluphe
202530
pheserleuserglyvalasnlysaspserphelysmetlyscysval
354045
leucysleuproleuasnlysgluileseralaphelysserserpro
505560
serasnleuarglyshisilegluargmethisproasntyrleulys
65707580
asntyrserlysleuthralaglnlysarglysileglythrserthr
859095
hisalaserserserlysglnleulysvalaspservalpheproval
100105110
lyshisvalserprovalthrvalasnlysalaileleuargtyrile
115120125
ileglnglyleuhispropheserthrvalaspleuproserphelys
130135140
gluleuileserthrleuglnproglyileservalilethrargpro
145150155160
thrleuargserlysilealaglualaalaleuilemetlysglnlys
165170175
valthralaalametsergluvalglutrpilealathrthrthrasp
180185190
cystrpthralaargarglysserpheileglyvalthralahistrp
195200205
ileasnproglyserleugluarghisseralaalaleualacyslys
210215220
argleumetglyserhisthrphegluvalleualaseralametasn
225230235240
aspilehisserglutyrgluileargasplysvalvalcysthrthr
245250255
thraspserglyserasnphemetlysalapheargvalpheglyval
260265270
gluasnasnaspilegluthrglualaargargcysgluseraspasp
275280285
thraspsergluglycysglygluglyseraspglyvalgluphegln
290295300
aspalaserargvalleuaspglnaspaspglypheglupheglnleu
305310315320
prolyshisglnlyscysalacyshisleuleuasnleuvalserser
325330335
valaspalaglnlysalaleuserasngluhistyrlyslysleutyr
340345350
argservalpheglylyscysglnalaleutrpasnlysserserarg
355360365
seralaleualaalaglualavalglusergluserargleuglnleu
370375380
leuargproasnglnthrargtrpasnserthrphemetalavalasp
385390395400
argileleuglnilecyslysglualaglygluglyalaleuargasn
405410415
ilecysthrserleugluvalprometpheasnproalaglumetleu
420425430
pheleuthrglutrpalaasnthrmetargprovalalalysvalleu
435440445
aspileleuglnalagluthrasnthrglnleuglytrpleuleupro
450455460
servalhisglnleuserleulysleuglnargleuhishisserleu
465470475480
argtyrcysaspproleuvalaspalaleuglnglnglyileglnthr
485490495
argphelyshismetphegluaspprogluileilealaalaalaile
500505510
leuleuprolyspheargthrsertrpthrasnaspgluthrileile
515520525
lysargglymetasptyrileargvalhisleugluproleuasphis
530535540
lyslysgluleualaasnserserseraspaspgluaspphepheala
545550555560
serleulysprothrthrhisglualaserlysgluleuaspglytyr
565570575
leualacysvalseraspthrarggluserleuleuthrpheproala
580585590
ilecysserleuserilelysthrasnthrproleuproalaserala
595600605
alacysgluargleupheserthralaglyleuleupheserprolys
610615620
argalaargleuaspthrasnasnphegluasnglnleuleuleulys
625630635640
leuasnleuargphetyrasnpheglu
645
<210>6
<211>4682
<212>dna
<213>oryziaslatipes
<400>6
cagaggtgtaaagtacttgagtaattttacttgattactgtacttaagtattatttttgg60
ggatttttactttacttgagtacaattaaaaatcaatacttttacttttacttaattaca120
tttttttagaaaaaaaagtactttttactccttacaattttatttacagtcaaaaagtac180
ttattttttggagatcacttcattctattttcccttgctattaccaaaccaattgaattg240
cgctgatgcccagtttaatttaaatgttatttattctgcctatgaaaatcgttttcacat300
tatatgaaattggtcagacatgttcattggtcctttggaagtgacgtcatgtcacatcta360
ttaccacaatgcacagcaccttgacctggaaattagggaaattataacagtcaatcagtg420
gaagaaaatggaggaagtatgtgattcatcagcagctgcgagcagcacagtccaaaatca480
gccacaggatcaagagcacccgtggccgtatcttcgcgaattcttttctttaagtggtgt540
aaataaagattcattcaagatgaaatgtgtcctctgtctcccgcttaataaagaaatatc600
ggccttcaaaagttcgccatcaaacctaaggaagcatattgaggtaagtacattaagtat660
tttgttttactgatagtttttttttttttttttttttttttttttgggtgtgcatgtttt720
gacgttgatggcgcgccttttatatgtgtagtaggcctattttcactaatgcatgcgatt780
gacaatataaggctcacgtaataaaatgctaaaatgcatttgtaattggtaacgttaggt840
ccacgggaaatttggcgcctattgcagctttgaataatcattatcattccgtgctctcat900
tgtgtttgaattcatgcaaaacacaagaaaaccaagcgagaaatttttttccaaacatgt960
tgtattgtcaaaacggtaacactttacaatgaggttgattagttcatgtattaactaaca1020
ttaaataaccatgagcaatacatttgttactgtatctgttaatctttgttaacgttagtt1080
aatagaaatacagatgttcattgtttgttcatgttagttcacagtgcattaactaatgtt1140
aacaagatataaagtattagtaaatgttgaaattaacatgtatacgtgcagttcattatt1200
agttcatgttaactaatgtagttaactaacgaaccttattgtaaaagtgttaccatcaaa1260
actaatgtaatgaaatcaattcaccctgtcatgtcagccttacagtcctgtgtttttgtc1320
aatataatcagaaataaaattaatgtttgattgtcactaaatgctactgtatttctaaaa1380
tcaacaagtatttaacattataaagtgtgcaattggctgcaaatgtcagttttattaaag1440
ggttagttcacccaaaaatgaaaataatgtcattaatgactcgccctcatgtcgttccaa1500
gcccgtaagacctccgttcatcttcagaacacagtttaagatattttagatttagtccga1560
gagctttctgtgcctccattgagaatgtatgtacggtatactgtccatgtccagaaaggt1620
aataaaaacatcaaagtagtccatgtgacatcagtgggttagttagaattttttgaagca1680
tcgaatacattttggtccaaaaataacaaaacctacgactttattcggcattgtattctc1740
ttccgggtctgttgtcaatccgcgttcacgacttcgcagtgacgctacaatgctgaataa1800
agtcgtaggttttgttatttttggaccaaaatgtattttcgatgcttcaaataattctac1860
ctaacccactgatgtcacatggactactttgatgtttttattacctttctggacatggac1920
agtataccgtacatacattttcagtggagggacagaaagctctcggactaaatctaaaat1980
atcttaaactgtgttccgaagatgaacggaggtgttacgggcttggaacgacatgagggt2040
gagtcattaatgacatcttttcatttttgggtgaactaaccctttaatgctgtaatcaga2100
gagtgtatgtgtaattgttacatttattgcatacaatataaatatttatttgttgttttt2160
acagagaatgcacccaaattacctcaaaaactactctaaattgacagcacagaagagaaa2220
gatcgggacctccacccatgcttccagcagtaagcaactgaaagttgactcagttttccc2280
agtcaaacatgtgtctccagtcactgtgaacaaagctatattaaggtacatcattcaagg2340
acttcatcctttcagcactgttgatctgccatcatttaaagagctgattagtacactgca2400
gcctggcatttctgtcattacaaggcctactttacgctccaagatagctgaagctgctct2460
gatcatgaaacagaaagtgactgctgccatgagtgaagttgaatggattgcaaccacaac2520
ggattgttggactgcacgtagaaagtcattcattggtgtaactgctcactggatcaaccc2580
tggaagtcttgaaagacattccgctgcacttgcctgcaaaagattaatgggctctcatac2640
ttttgaggtactggccagtgccatgaatgatatccactcagagtatgaaatacgtgacaa2700
ggttgtttgcacaaccacagacagtggttccaactttatgaaggctttcagagtttttgg2760
tgtggaaaacaatgatatcgagactgaggcaagaaggtgtgaaagtgatgacactgattc2820
tgaaggctgtggtgagggaagtgatggtgtggaattccaagatgcctcacgagtcctgga2880
ccaagacgatggcttcgaattccagctaccaaaacatcaaaagtgtgcctgtcacttact2940
taacctagtctcaagcgttgatgcccaaaaagctctctcaaatgaacactacaagaaact3000
ctacagatctgtctttggcaaatgccaagctttatggaataaaagcagccgatcggctct3060
agcagctgaagctgttgaatcagaaagccggcttcagcttttaaggccaaaccaaacgcg3120
gtggaattcaacttttatggctgttgacagaattcttcaaatttgcaaagaagcaggaga3180
aggcgcacttcggaatatatgcacctctcttgaggttccaatgtaagtgtttttcccctc3240
tatcgatgtaaacaaatgtgggttgtttttgtttaatactctttgattatgctgatttct3300
cctgtaggtttaatccagcagaaatgctgttcttgacagagtgggccaacacaatgcgtc3360
cagttgcaaaagtactcgacatcttgcaagcggaaacgaatacacagctggggtggctgc3420
tgcctagtgtccatcagttaagcttgaaacttcagcgactccaccattctctcaggtact3480
gtgacccacttgtggatgccctacaacaaggaatccaaacacgattcaagcatatgtttg3540
aagatcctgagatcatagcagctgccatccttctccctaaatttcggacctcttggacaa3600
atgatgaaaccatcataaaacgaggtaaatgaatgcaagcaacatacacttgacgaattc3660
taatctgggcaacctttgagccataccaaaattattcttttatttatttatttttgcact3720
ttttaggaatgttatatcccatctttggctgtgatctcaatatgaatattgatgtaaagt3780
attcttgcagcaggttgtagttatccctcagtgtttcttgaaaccaaactcatatgtatc3840
atatgtggtttggaaatgcagttagattttatgctaaaataagggatttgcatgatttta3900
gatgtagatgactgcacgtaaatgtagttaatgacaaaatccataaaatttgttcccagt3960
cagaagcccctcaaccaaacttttctttgtgtctgctcactgtgcttgtaggcatggact4020
acatcagagtgcatctggagcctttggaccacaagaaggaattggccaacagttcatctg4080
atgatgaagattttttcgcttctttgaaaccgacaacacatgaagccagcaaagagttgg4140
atggatatctggcctgtgtttcagacaccagggagtctctgctcacgtttcctgctattt4200
gcagcctctctatcaagactaatacacctcttcccgcatcggctgcctgtgagaggcttt4260
tcagcactgcaggattgcttttcagccccaaaagagctaggcttgacactaacaattttg4320
agaatcagcttctactgaagttaaatctgaggttttacaactttgagtagcgtgtactgg4380
cattagattgtctgtcttatagtttgataattaaatacaaacagttctaaagcaggataa4440
aaccttgtatgcatttcatttaatgttttttgagattaaaagcttaaacaagaatctcta4500
gttttctttcttgcttttacttttacttccttaatactcaagtacaattttaatggagta4560
cttttttacttttactcaagtaagattctagccagatacttttacttttaattgagtaaa4620
attttccctaagtacttgtactttcacttgagtaaaatttttgagtactttttacacctc4680
tg4682
<210>7
<211>321
<212>dna
<213>artificial
<220>
<223>descriptionofartificialsequence;cycloheximideresistantgene
<220>
<221>cds
<222>(1)..(321)
<400>7
atggtcaacgtacctaaaacccgaagaaccttctgtaagaagtgtggc48
metvalasnvalprolysthrargargthrphecyslyslyscysgly
151015
aagcatcagcctcacaaagtgacacagtataagaagggcaaggattct96
lyshisglnprohislysvalthrglntyrlyslysglylysaspser
202530
ttgtatgcccagggaaggaggcgctatgatcggaagcagagtggctat144
leutyralaglnglyargargargtyrasparglysglnserglytyr
354045
ggtgggcagacaaagcaaattttccggaagaaggctaagaccacaaag192
glyglyglnthrlysglnilephearglyslysalalysthrthrlys
505560
aagattgtgctaaggctggaatgtgttgagcctaactgcagatccaag240
lysilevalleuargleuglucysvalgluproasncysargserlys
65707580
aggatgctggccattaagagatgcaagcattttgaactgggaggagat288
argmetleualailelysargcyslyshisphegluleuglyglyasp
859095
aagaagagaaagggccaagtgatccagttctaa321
lyslysarglysglyglnvalileglnphe
100105
<210>8
<211>106
<212>prt
<213>artificial
<220>
<223>syntheticconstruct
<400>8
metvalasnvalprolysthrargargthrphecyslyslyscysgly
151015
lyshisglnprohislysvalthrglntyrlyslysglylysaspser
202530
leutyralaglnglyargargargtyrasparglysglnserglytyr
354045
glyglyglnthrlysglnilephearglyslysalalysthrthrlys
505560
lysilevalleuargleuglucysvalgluproasncysargserlys
65707580
argmetleualailelysargcyslyshisphegluleuglyglyasp
859095
lyslysarglysglyglnvalileglnphe
100105
<210>9
<211>1404
<212>dna
<213>artificial
<220>
<223>m2z3heavychain
<220>
<221>cds
<222>(1)..(1404)
<400>9
atggactggacctggagcatccttttcttggtggcagcagcaacaggt48
metasptrpthrtrpserileleupheleuvalalaalaalathrgly
151015
gcccactcccaggttcagctggtgcagtctggagctgaggtgaagaag96
alahisserglnvalglnleuvalglnserglyalagluvallyslys
202530
cctggggcctcagtgaaggtctcctgcaaggcttctggttacaccttt144
proglyalaservallysvalsercyslysalaserglytyrthrphe
354045
accagctatggtatcagctgggtgcgacaggcccctggacaagggctt192
thrsertyrglyilesertrpvalargglnalaproglyglnglyleu
505560
gagtggatgggatggatcagcgcttacaatggtaacacaaactatgca240
glutrpmetglytrpileseralatyrasnglyasnthrasntyrala
65707580
cagaagctccagggcagagtcaccatgaccacagacacatccacgagc288
glnlysleuglnglyargvalthrmetthrthraspthrserthrser
859095
acagcctacatggagctgaggagcctgagatctgacgacacggccgtg336
thralatyrmetgluleuargserleuargseraspaspthralaval
100105110
tattactgtgcgagggcagcagctggcggatacttccagcactggggc384
tyrtyrcysalaargalaalaalaglyglytyrpheglnhistrpgly
115120125
cagggcaccctggtcaccgtctcctcagctagcaccaagggcccatcg432
glnglythrleuvalthrvalserseralaserthrlysglyproser
130135140
gtcttccccctggcaccctcctccaagagcacctctgggggcacagcg480
valpheproleualaproserserlysserthrserglyglythrala
145150155160
gccctgggctgcctggtcaaggactacttccccgaaccggtgacggtg528
alaleuglycysleuvallysasptyrpheprogluprovalthrval
165170175
tcgtggaactcaggcgccctgaccagcggcgtgcacaccttcccggct576
sertrpasnserglyalaleuthrserglyvalhisthrpheproala
180185190
gtcctacagtcctcaggactctactccctcagcagcgtggtgaccgtg624
valleuglnserserglyleutyrserleuserservalvalthrval
195200205
ccctccagcagcttgggcacccagacctacatctgcaacgtgaatcac672
proserserserleuglythrglnthrtyrilecysasnvalasnhis
210215220
aagcccagcaacaccaaggtggacaagaaagttgagcccaaatcttgt720
lysproserasnthrlysvalasplyslysvalgluprolyssercys
225230235240
gacaaaactcacacatgcccaccgtgcccagcacctgaactcctgggg768
asplysthrhisthrcysproprocysproalaprogluleuleugly
245250255
ggaccgtcagtcttcctcttccccccaaaacccaaggacaccctcatg816
glyproservalpheleupheproprolysprolysaspthrleumet
260265270
atctcccggacccctgaggtcacatgcgtggtggtggacgtgagccac864
ileserargthrprogluvalthrcysvalvalvalaspvalserhis
275280285
gaagaccctgaggtcaagttcaactggtacgtggacggcgtggaggtg912
gluaspprogluvallyspheasntrptyrvalaspglyvalgluval
290295300
cataatgccaagacaaagccgcgggaggagcagtacaacagcacgtac960
hisasnalalysthrlysproargglugluglntyrasnserthrtyr
305310315320
cgtgtggtcagcgtcctcaccgtcctgcaccaggactggctgaatggc1008
argvalvalservalleuthrvalleuhisglnasptrpleuasngly
325330335
aaggagtacaagtgcaaggtctccaacaaagccctcccagcccccatc1056
lysglutyrlyscyslysvalserasnlysalaleuproalaproile
340345350
gagaaaaccatctccaaagccaaagggcagccccgagaaccacaggtg1104
glulysthrileserlysalalysglyglnproarggluproglnval
355360365
tacaccctgcccccatcccgggatgagctgaccaagaaccaggtcagc1152
tyrthrleuproproserargaspgluleuthrlysasnglnvalser
370375380
ctgacctgcctggtcaaaggcttctatcccagcgacatcgccgtggag1200
leuthrcysleuvallysglyphetyrproseraspilealavalglu
385390395400
tgggagagcaatgggcagccggagaacaactacaagaccacgcctccc1248
trpgluserasnglyglnprogluasnasntyrlysthrthrpropro
405410415
gtgctggactccgacggctccttcttcctctacagcaagctcaccgtg1296
valleuaspseraspglyserphepheleutyrserlysleuthrval
420425430
gacaagagcaggtggcagcaggggaacgtcttctcatgctccgtgatg1344
asplysserargtrpglnglnglyasnvalphesercysservalmet
435440445
catgaggctctgcacaaccactacacgcagaagagcctctccctgtct1392
hisglualaleuhisasnhistyrthrglnlysserleuserleuser
450455460
ccgggtaaatga1404
proglylys
465
<210>10
<211>467
<212>prt
<213>artificial
<220>
<223>syntheticconstruct
<400>10
metasptrpthrtrpserileleupheleuvalalaalaalathrgly
151015
alahisserglnvalglnleuvalglnserglyalagluvallyslys
202530
proglyalaservallysvalsercyslysalaserglytyrthrphe
354045
thrsertyrglyilesertrpvalargglnalaproglyglnglyleu
505560
glutrpmetglytrpileseralatyrasnglyasnthrasntyrala
65707580
glnlysleuglnglyargvalthrmetthrthraspthrserthrser
859095
thralatyrmetgluleuargserleuargseraspaspthralaval
100105110
tyrtyrcysalaargalaalaalaglyglytyrpheglnhistrpgly
115120125
glnglythrleuvalthrvalserseralaserthrlysglyproser
130135140
valpheproleualaproserserlysserthrserglyglythrala
145150155160
alaleuglycysleuvallysasptyrpheprogluprovalthrval
165170175
sertrpasnserglyalaleuthrserglyvalhisthrpheproala
180185190
valleuglnserserglyleutyrserleuserservalvalthrval
195200205
proserserserleuglythrglnthrtyrilecysasnvalasnhis
210215220
lysproserasnthrlysvalasplyslysvalgluprolyssercys
225230235240
asplysthrhisthrcysproprocysproalaprogluleuleugly
245250255
glyproservalpheleupheproprolysprolysaspthrleumet
260265270
ileserargthrprogluvalthrcysvalvalvalaspvalserhis
275280285
gluaspprogluvallyspheasntrptyrvalaspglyvalgluval
290295300
hisasnalalysthrlysproargglugluglntyrasnserthrtyr
305310315320
argvalvalservalleuthrvalleuhisglnasptrpleuasngly
325330335
lysglutyrlyscyslysvalserasnlysalaleuproalaproile
340345350
glulysthrileserlysalalysglyglnproarggluproglnval
355360365
tyrthrleuproproserargaspgluleuthrlysasnglnvalser
370375380
leuthrcysleuvallysglyphetyrproseraspilealavalglu
385390395400
trpgluserasnglyglnprogluasnasntyrlysthrthrpropro
405410415
valleuaspseraspglyserphepheleutyrserlysleuthrval
420425430
asplysserargtrpglnglnglyasnvalphesercysservalmet
435440445
hisglualaleuhisasnhistyrthrglnlysserleuserleuser
450455460
proglylys
465
<210>11
<211>708
<212>dna
<213>artificial
<220>
<223>m2z3lightchian
<220>
<221>cds
<222>(1)..(708)
<400>11
atggccagcttccctctcctcctcaccctcctcactcactgtgcaggg48
metalaserpheproleuleuleuthrleuleuthrhiscysalagly
151015
tcctgggcccagtctgtgctgactcagccaccctcagcgtctgggacc96
sertrpalaglnservalleuthrglnproproseralaserglythr
202530
cccgggcagagggtcaccatctcttgttctggaagcaactccaacatc144
proglyglnargvalthrilesercysserglyserasnserasnile
354045
ggaagtaaaactgtaaactggtaccagcagctcccaggaacggccccc192
glyserlysthrvalasntrptyrglnglnleuproglythralapro
505560
aaactcctcatctctagtaataatcagcggccctcaggggtccctgac240
lysleuleuileserserasnasnglnargproserglyvalproasp
65707580
cgattctctggctccaagtctggcacctcagcctccctggccatcagt288
argpheserglyserlysserglythrseralaserleualaileser
859095
gggctccagtctgaggatgaggctgattattactgtgcagcatgggat336
glyleuglnsergluaspglualaasptyrtyrcysalaalatrpasp
100105110
gacagcctgaatggtgtggtattcggcggagggaccaagctgaccgtc384
aspserleuasnglyvalvalpheglyglyglythrlysleuthrval
115120125
ctaggtcagcccaaggctgccccctcggtcactctgttcccaccctcc432
leuglyglnprolysalaalaproservalthrleupheproproser
130135140
tctgaggagcttcaagccaacaaggccacactggtgtgtctcataagt480
serglugluleuglnalaasnlysalathrleuvalcysleuileser
145150155160
gacttctacccgggagccgtgacagtggcctggaaggcagatagcagc528
aspphetyrproglyalavalthrvalalatrplysalaaspserser
165170175
cccgtcaaggcgggagtggagaccaccacaccctccaaacaaagcaac576
provallysalaglyvalgluthrthrthrproserlysglnserasn
180185190
aacaagtacgcggccagcagctacctgagcctgacgcctgagcagtgg624
asnlystyralaalasersertyrleuserleuthrprogluglntrp
195200205
aagtcccacaaaagctacagctgccaggtcacgcatgaagggagcacc672
lysserhislyssertyrsercysglnvalthrhisgluglyserthr
210215220
gtggagaagacagtggcccctacagaatgttcatag708
valglulysthrvalalaprothrglucysser
225230235
<210>12
<211>235
<212>prt
<213>artificial
<220>
<223>syntheticconstruct
<400>12
metalaserpheproleuleuleuthrleuleuthrhiscysalagly
151015
sertrpalaglnservalleuthrglnproproseralaserglythr
202530
proglyglnargvalthrilesercysserglyserasnserasnile
354045
glyserlysthrvalasntrptyrglnglnleuproglythralapro
505560
lysleuleuileserserasnasnglnargproserglyvalproasp
65707580
argpheserglyserlysserglythrseralaserleualaileser
859095
glyleuglnsergluaspglualaasptyrtyrcysalaalatrpasp
100105110
aspserleuasnglyvalvalpheglyglyglythrlysleuthrval
115120125
leuglyglnprolysalaalaproservalthrleupheproproser
130135140
serglugluleuglnalaasnlysalathrleuvalcysleuileser
145150155160
aspphetyrproglyalavalthrvalalatrplysalaaspserser
165170175
provallysalaglyvalgluthrthrthrproserlysglnserasn
180185190
asnlystyralaalasersertyrleuserleuthrprogluglntrp
195200205
lysserhislyssertyrsercysglnvalthrhisgluglyserthr
210215220
valglulysthrvalalaprothrglucysser
225230235
<210>13
<211>1855
<212>dna
<213>artificial
<220>
<223>nonautologustol1transposon
<400>13
cagtagcggttctaggcacgggccgtccgggcggtggcctggggcggaaaactgaagggg60
ggcggcaccggcggctcagccctttgtaatatattaatatgcaccactattggtttactt120
atgtcacagtttgtaagtttgtaacagcctgaacctggccgcgccgccgccctcgccccg180
cagctgcgctctcctgtctttgagaagtagacacaaatgtgtgtgaagaaggagaaggga240
gggggcgcggggtgagcacggagcgtcgccgcgtttgcgcatgcgcaaaacctggctggc300
tcatctttcaggggaggcgacggtcgcgggcttgatgaaaaaaataaaagtaaaaactgc360
gactgcgccgtcatgtagcgaatcagcgcccctggctgtagctgcacgcgctcctgctgg420
aaatgtgtgaagaggggggggggggggggggctgcggggaatcagttcaattgtgggacg480
cttccaaattaagtggctaggtggggacaagggcgggggtttgaatctacttcataaaac540
ctttttatattataagtcagtcataaggtgacattctataacctacattttaataaaggt600
ataaaaaatatattctgctttttttgggttaattttgtgtgaaatgtccaaataaaaaaa660
atggcaacacaaaacaatgctgtcactaaggtgacagttggttcagtcgacggacttgat720
gccttcttcgtgacgtgaggacatttatgccaaacaaacgccaataaacatctaaaatat780
ggaaaagaaaaggtcaaagccatctggtgcccaatttagaaagaaaagaaaagaagaaga840
ggagaaaagagataaagaaaagggtaagtcctcacagcttgatgcatgttttttctaaat900
tctaatgctacctgccctacaacaacgttgccgatgaaaactttattttggtcgatgacc960
aacactgaattaggcccaaatgttgcaaatagcgtcatttttttttttttttttagattt1020
tattcttaaaaatttgctctgccttaacttgtaacattagttatgattcatgtgtctgtc1080
tgctctgctgtaacacaaaggttttgttgggttttgctgttgtatactagctcataatgt1140
taaaaaagctgtgatggttacacagcatgctggtgctgccataagatgctaatggggcaa1200
ataatttgagattggtcattaatttaataatcatttgtggcagcctaaacgttttcacaa1260
tgtttttttgacatttaactggggatttaggggttaattttgagcctgcatatgaagttt1320
attttttatttgttttacaaatgtgggattatatttttagccaatagaatttccataaat1380
ctgtaggtagttttaaaaatgaatatttaccatttactgcaactctatggggacaaaaca1440
taatgtaacaggtcataactaaaaatgtgccaatcaaaggattgaagacggaaaacatga1500
gttaatttttcttctctgaagtagagatcgatatagaacatgacaatttaaatttccaat1560
tcataaatgtttttaaaatatttattttatattatttatttaacattgagtttgattcaa1620
tattttcttagctaactgtatttttgccatgcttatggtcttttattttttgtgttctga1680
taacttttataatgcttttcagaattttgacatcttttgtatccacttcttaatttcaat1740
gacaataaaacatttcagttgacgaagacaaacaaagttctgttgtgactatgggggggg1800
ggggcgcctggggatggtctcgcccggggagtaattcagggtagaaccgccactg1855
<210>14
<211>200
<212>dna
<213>artificial
<220>
<223>tol1-ltransposonsequence
<400>14
cagtagcggttctaggcacgggccgtccgggcggtggcctggggcggaaaactgaagggg60
ggcggcaccggcggctcagccctttgtaatatattaatatgcaccactattggtttactt120
atgtcacagtttgtaagtttgtaacagcctgaacctggccgcgccgccgccctcgccccg180
cagctgcgctctcctgtctt200
<210>15
<211>505
<212>dna
<213>artificial
<220>
<223>tol1-rtransoposonsequence
<400>15
atatttttagccaatagaatttccataaatctgtaggtagttttaaaaatgaatatttac60
catttactgcaactctatggggacaaaacataatgtaacaggtcataactaaaaatgtgc120
caatcaaaggattgaagacggaaaacatgagttaatttttcttctctgaagtagagatcg180
atatagaacatgacaatttaaatttccaattcataaatgtttttaaaatatttattttat240
attatttatttaacattgagtttgattcaatattttcttagctaactgtatttttgccat300
gcttatggtcttttattttttgtgttctgataacttttataatgcttttcagaattttga360
catcttttgtatccacttcttaatttcaatgacaataaaacatttcagttgacgaagaca420
aacaaagttctgttgtgactatggggggggggggcgcctggggatggtctcgcccgggga480
gtaattcagggtagaaccgccactg505
<210>16
<211>2745
<212>dna
<213>oryziaslatipes
<220>
<221>cds
<222>(30)..(2585)
<400>16
gccaaacaaacgccaaaaacatctaaaatatggagaaaaaaaggtcaaagcca53
metglulyslysargserlyspro
15
tctggtgcccaatttagaaagaaaagaaaagaagaagaggagaaaaga101
serglyalaglnphearglyslysarglysgluglugluglulysarg
101520
gataaagaaaagggggcacttctaagatattttggatcgtctaccact149
asplysglulysglyalaleuleuargtyrpheglyserserthrthr
25303540
gctcaagatgagacatctacctccctgccagctatctcatcagccaca197
alaglnaspgluthrserthrserleuproalaileserseralathr
455055
gtcacagtctcaccccctcaggatgagctaccatctacatcctctgct245
valthrvalserproproglnaspgluleuproserthrserserala
606570
actcatgtagttccacagttgttacctgagcaaagttttgatagtgag293
thrhisvalvalproglnleuleuprogluglnserpheaspserglu
758085
gctgaagacgttgttccatctacgtctacccagcttgagacttcagaa341
alagluaspvalvalproserthrserthrglnleugluthrserglu
9095100
atgcctggtgatgaaaccccactgaccccgactgctgaggaccagcct389
metproglyaspgluthrproleuthrprothralagluaspglnpro
105110115120
ctaccaactgaccctgcaaagtggccctcacctctgactgacaggata437
leuprothraspproalalystrpproserproleuthraspargile
125130135
cggatggagctggttcgaagaggaccaagtagcataccacctgacttt485
argmetgluleuvalargargglyproserserileproproaspphe
140145150
gttttcccaagaaatgacagtgatgggagaagttgtcatcaccactat533
valpheproargasnaspseraspglyargsercyshishishistyr
155160165
ttcaggaagacactagtaagtggtgaaaaaatagcaagaacttggttg581
phearglysthrleuvalserglyglulysilealaargthrtrpleu
170175180
atgtattcaaaagtgaagaacagcctcttttgcttttgttgcaaattg629
mettyrserlysvallysasnserleuphecysphecyscyslysleu
185190195200
ttttccaacaaaaacattaatttaacaacttctggtacagcaaactgg677
pheserasnlysasnileasnleuthrthrserglythralaasntrp
205210215
aaacatgcaagcacatacctcacagcacacgaaaaaagcccagaacac725
lyshisalaserthrtyrleuthralahisglulysserprogluhis
220225230
ctcaattgtatgaaagcatggaaggaactgtcagggaggatcagaagt773
leuasncysmetlysalatrplysgluleuserglyargileargser
235240245
gggaaaacaattgataagcaggagatggcacttctggaagaggagcgg821
glylysthrileasplysglnglumetalaleuleugluglugluarg
250255260
gtgagatggagagcagtgctaacccgtctcattgctattgtgcagtca869
valargtrpargalavalleuthrargleuilealailevalglnser
265270275280
ctggcagttcggaatttggctctaaggggacacacagaaacactgttc917
leualavalargasnleualaleuargglyhisthrgluthrleuphe
285290295
acatcatcaaatgggaattttttgaaagaggttgaactgatggccagg965
thrserserasnglyasnpheleulysgluvalgluleumetalaarg
300305310
tttgatcccataatgaaagatcatcttaaccgtgtattaagaggaaca1013
pheaspproilemetlysasphisleuasnargvalleuargglythr
315320325
gcaagtcacaacagctacataggccatcatgtgcagaatgaacttatt1061
alaserhisasnsertyrileglyhishisvalglnasngluleuile
330335340
gatttgttgagcagcaaaatcctatccgctatagtggatgacatcaaa1109
aspleuleuserserlysileleuseralailevalaspaspilelys
345350355360
aaggcaaaatatttttcaataattctggactgcactctggatataagc1157
lysalalystyrpheserileileleuaspcysthrleuaspileser
365370375
cacacagaacagttgtcagttataattagagtggtgtcactgatggag1205
histhrgluglnleuservalileileargvalvalserleumetglu
380385390
aagcctcagatcagggaacattttatggggtttttggaggcagaggag1253
lysproglnilearggluhisphemetglypheleuglualagluglu
395400405
tccacaggccagcacttggcatccatgatcttaaacagacttgaggag1301
serthrglyglnhisleualasermetileleuasnargleugluglu
410415420
ttaggaatttcttttgaagactgcagaggacaatcatatgataatggg1349
leuglyileserphegluaspcysargglyglnsertyraspasngly
425430435440
gcaaatatgaaaggcaaaaataagggagtacaagccaggctcttagaa1397
alaasnmetlysglylysasnlysglyvalglnalaargleuleuglu
445450455
aagaatccccgtgctctgtttttgccatgcggtgcacacacattgaat1445
lysasnproargalaleupheleuprocysglyalahisthrleuasn
460465470
ttagttgtgtgtgatgctgctaagagatctgttgatgctatgagctac1493
leuvalvalcysaspalaalalysargservalaspalametsertyr
475480485
tttggtgtcctgcaaaagctttacactttattttcagcctctgcccaa1541
pheglyvalleuglnlysleutyrthrleupheseralaseralagln
490495500
cgatgggccatactgaagagtcaggtgagcatcactctaaagtcgtgg1589
argtrpalaileleulysserglnvalserilethrleulyssertrp
505510515520
acagaaacaaggtgggagagcaaaatcaaaagcatcgagcccatgagg1637
thrgluthrargtrpgluserlysilelysserilegluprometarg
525530535
taccagggagctgcagtgagagaggctttaatagaagtgagagacaag1685
tyrglnglyalaalavalargglualaleuilegluvalargasplys
540545550
accaaagacccagttataaaggctgaggcccagtctttgtctgaagag1733
thrlysaspprovalilelysalaglualaglnserleusergluglu
555560565
gtagggtcgtaccgcttcaacatctgcacagtcgtatggcatgacatt1781
valglysertyrargpheasnilecysthrvalvaltrphisaspile
570575580
ctatctacaataaagcatgtcagcaaactcatgcagtctccaaatatg1829
leuserthrilelyshisvalserlysleumetglnserproasnmet
585590595600
catgtggacctagctgtgagtcttttgaagaagactgaacaaagtctc1877
hisvalaspleualavalserleuleulyslysthrgluglnserleu
605610615
cagagctacagggcaaatggctttgtgaatgcacagatggcagccaaa1925
glnsertyrargalaasnglyphevalasnalaglnmetalaalalys
620625630
gaaatgtgcaaggaaatgaatgtcgaggctattttgaaacaaaaaaga1973
glumetcyslysglumetasnvalglualaileleulysglnlysarg
635640645
ataagatccacaaagtgccaattctcgtatgaatcacacgatgagcct2021
ileargserthrlyscysglnphesertyrgluserhisaspglupro
650655660
ttcagtgacgcacttaaaaagttggaggttgaatttttcaatgttgtt2069
pheseraspalaleulyslysleugluvalgluphepheasnvalval
665670675680
gttgatgaagccttgtcagccatcgcggagaggttttccacattggaa2117
valaspglualaleuseralailealagluargpheserthrleuglu
685690695
gttgtacaaaacagatttggggttttgaccaatttcccaagccttgga2165
valvalglnasnargpheglyvalleuthrasnpheproserleugly
700705710
gacgaggagctgacggagcaatgcgaggcactaggcaacatactccat2213
aspglugluleuthrgluglncysglualaleuglyasnileleuhis
715720725
tttgagaagaactgggatttggacagtagagagcttgttcaggaaatc2261
pheglulysasntrpaspleuaspserarggluleuvalglngluile
730735740
aagaacttgcctaacttaccatcaacgactccaagtctccttgagctc2309
lysasnleuproasnleuproserthrthrproserleuleugluleu
745750755760
atctctttcatgtctgataaggatctatcagaaatctatccgaacttt2357
ileserphemetserasplysaspleusergluiletyrproasnphe
765770775
tggactgctctcaggattgcactcaccttgccagtcactgtggctcaa2405
trpthralaleuargilealaleuthrleuprovalthrvalalagln
780785790
gcagagaggagcttttcaaaactaaaattgatcaagtcgtacctgagg2453
alagluargserpheserlysleulysleuilelyssertyrleuarg
795800805
tcaacaatgtcacaggagcgactcactaaccttgccgttgttagcatc2501
serthrmetserglngluargleuthrasnleualavalvalserile
810815820
aatcactcagtaggggagcagatatcatatgatgatgttattgacgag2549
asnhisservalglygluglnilesertyraspaspvalileaspglu
825830835840
tttgcatcaagaaaggctaggaaggttaggttttagttggtgtttt2595
phealaserarglysalaarglysvalargphe
845850
ctgttattgtattggtgctgcagttatatttattttagcgtgtcatttgtgtgataaaag2655
gtttgtgctttataatatttattttatattatttattcaatattgagtttgattcaatat2715
tttcttagctaactgtatttttgccatgct2745
<210>17
<211>851
<212>prt
<213>oryziaslatipes
<400>17
metglulyslysargserlysproserglyalaglnphearglyslys
151015
arglysgluglugluglulysargasplysglulysglyalaleuleu
202530
argtyrpheglyserserthrthralaglnaspgluthrserthrser
354045
leuproalaileserseralathrvalthrvalserproproglnasp
505560
gluleuproserthrserseralathrhisvalvalproglnleuleu
65707580
progluglnserpheaspserglualagluaspvalvalproserthr
859095
serthrglnleugluthrserglumetproglyaspgluthrproleu
100105110
thrprothralagluaspglnproleuprothraspproalalystrp
115120125
proserproleuthraspargileargmetgluleuvalargarggly
130135140
proserserileproproaspphevalpheproargasnaspserasp
145150155160
glyargsercyshishishistyrphearglysthrleuvalsergly
165170175
glulysilealaargthrtrpleumettyrserlysvallysasnser
180185190
leuphecysphecyscyslysleupheserasnlysasnileasnleu
195200205
thrthrserglythralaasntrplyshisalaserthrtyrleuthr
210215220
alahisglulysserprogluhisleuasncysmetlysalatrplys
225230235240
gluleuserglyargileargserglylysthrileasplysglnglu
245250255
metalaleuleugluglugluargvalargtrpargalavalleuthr
260265270
argleuilealailevalglnserleualavalargasnleualaleu
275280285
argglyhisthrgluthrleuphethrserserasnglyasnpheleu
290295300
lysgluvalgluleumetalaargpheaspproilemetlysasphis
305310315320
leuasnargvalleuargglythralaserhisasnsertyrilegly
325330335
hishisvalglnasngluleuileaspleuleuserserlysileleu
340345350
seralailevalaspaspilelyslysalalystyrpheserileile
355360365
leuaspcysthrleuaspileserhisthrgluglnleuservalile
370375380
ileargvalvalserleumetglulysproglnilearggluhisphe
385390395400
metglypheleuglualaglugluserthrglyglnhisleualaser
405410415
metileleuasnargleuglugluleuglyileserphegluaspcys
420425430
argglyglnsertyraspasnglyalaasnmetlysglylysasnlys
435440445
glyvalglnalaargleuleuglulysasnproargalaleupheleu
450455460
procysglyalahisthrleuasnleuvalvalcysaspalaalalys
465470475480
argservalaspalametsertyrpheglyvalleuglnlysleutyr
485490495
thrleupheseralaseralaglnargtrpalaileleulyssergln
500505510
valserilethrleulyssertrpthrgluthrargtrpgluserlys
515520525
ilelysserilegluprometargtyrglnglyalaalavalargglu
530535540
alaleuilegluvalargasplysthrlysaspprovalilelysala
545550555560
glualaglnserleuserglugluvalglysertyrargpheasnile
565570575
cysthrvalvaltrphisaspileleuserthrilelyshisvalser
580585590
lysleumetglnserproasnmethisvalaspleualavalserleu
595600605
leulyslysthrgluglnserleuglnsertyrargalaasnglyphe
610615620
valasnalaglnmetalaalalysglumetcyslysglumetasnval
625630635640
glualaileleulysglnlysargileargserthrlyscysglnphe
645650655
sertyrgluserhisaspglupropheseraspalaleulyslysleu
660665670
gluvalgluphepheasnvalvalvalaspglualaleuseralaile
675680685
alagluargpheserthrleugluvalvalglnasnargpheglyval
690695700
leuthrasnpheproserleuglyaspglugluleuthrgluglncys
705710715720
glualaleuglyasnileleuhispheglulysasntrpaspleuasp
725730735
serarggluleuvalglngluilelysasnleuproasnleuproser
740745750
thrthrproserleuleugluleuileserphemetserasplysasp
755760765
leusergluiletyrproasnphetrpthralaleuargilealaleu
770775780
thrleuprovalthrvalalaglnalagluargserpheserlysleu
785790795800
lysleuilelyssertyrleuargserthrmetserglngluargleu
805810815
thrasnleualavalvalserileasnhisservalglygluglnile
820825830
sertyraspaspvalileaspgluphealaserarglysalaarglys
835840845
valargphe
850
- 还没有人留言评论。精彩留言会获得点赞!