高产MonacolinK的红曲霉基因工程菌的构建方法及其应用与流程
本发明属于微生物基因工程技术领域,涉及高产monacolink的红曲霉基因工程菌的构建方法及其应用。
背景技术:
红曲霉(monascuspurpureuswent.)中文别名红曲、红糟、红大米,存在于树木、土壤和堆积物等。是我国重要的微生物资源,能产生广泛的具有生物活性的天然产物,具有很高的商业价值。红曲霉生长适应的温度范围很广,一般温度15~42℃,最适温度为25~30℃,生长最适ph值为3.5~5,可耐浓度高达10%的乙醇。在麦芽汁琼脂培养基上生长良好,菌落初为白色,老熟后变成淡粉色、紫色或灰黑色,多形成红色。
红曲的应用已有上千年的历史,其医疗功效在中国古籍中早有记载,可用于酿酒、制醋、做豆腐乳的着色剂和调味剂,也可做中药。现有研究表明,红曲具有降血脂、降血压、抗氧化、抗癌、抗菌、抗疲劳、预防老年痴呆症等多重功效。
monacolins是红曲霉的主要代谢产物之一。研究表明monacolink(又称lovastain、mevinollin和mewacor)是一种抑制胆固醇合成的药物,最初由日本学者远藤章于1979年从红曲霉的次级代谢产物中发现。monacolink具有抑制胆固醇合成系统中的限制酶3-羟基-3-甲基戊二酰coa还原酶(hmg-coa)活性,从而降低人体和动物血浆中的胆固醇水平,是良好的血脂调节物质。此外,monacolink还具有抑制肿瘤、预防和治疗动脉硬化、冠心病、高血压、肥胖症等的作用。
目前,monacolink主要是以红曲霉通过固态发酵或液态发酵的方式进行生产。菌株产目标物质的能力是影响微生物发酵生产功能性物质的产量的首要因素。因此,需要不断的研究筛选出高产目的产物的菌株。基因工程菌是获得高产目标产物菌株的一个重要手段。目前,还没有采用基因工程的方法获得高产monacolink的红曲霉的相关报道。
技术实现要素:
本发明针对现有技术的不足,提供一种高产monacolink的红曲霉基因工程菌的构建方法及其应用,利用本发明方法,可以增强红曲霉中monacolink的代谢途径,进而从根本上解决红曲霉次级代谢产物中monacolink产量低的问题。不仅如此,本发明还意外发现,本发明工程菌可以降低桔霉素的含量,达到改善红曲产品质量、保证红曲产品的安全性的目的。
为实现上述目标,因此,本发明第一方面,提供一种高产monacolink的红曲霉基因工程菌的构建方法,通过在红曲霉菌中过表达monacolink编码基因mok基因,获得高产monacolink的红曲霉基因工程菌。所述mok基因为mokc基因、mokd基因、moke基因、moki基因中的一种。
所述mokc、mokd、moke、moki基因的碱基序列分别如seqidno.1至seqidno.4所示。
进一步,所述红曲霉菌为低产桔霉素红曲霉菌。
具体的,本发明高产monacolink的红曲霉基因工程菌的构建方法,包括如下步骤:
(1)以红曲霉cdna为模板,用特异引物扩增monacolink编码基因mok基因片段;
(2)构建红曲霉m1的monacolink基因的克隆质粒载体pbc-hygro-mok(mokc/mokd/moke/moki);
(3)将构建好的质粒电击转化进入红曲霉感受态细胞中,潮霉素b抗性筛选转基因菌株。
所述步骤(1)特异引物序列上游引物和下游引物分别带有酶切位点,引物序列如表1所示的seqidno.5-seqidno.12:
表1.特异性引物表
另一方面,本发明还提供了所构建的高产monacolink的红曲霉基因工程菌在发酵获得高产量、低桔霉素的monacolink的用途。monacolink的产量较出发菌红曲霉最高提高2.3倍,而桔霉素含量低于0.25μg/l。
本发明具有如下有益效果:
(1)本发明成功扩增到mok的mokc、mokd、moke、moki四个基因片段。我们在研究中发现以cdna为模板,设计能够完整扩增目的条带、并且两端包含合适的酶切位点的特异性引物十分困难。发明人经过反复引物设计-合成-pcr测序结果验证,才获得了本发明表1所用的4对特异性引物。
(2)本发明成功构建红曲霉m1的monacolink基因的克隆载体pbc-hygro-mok。本发明通过特异性引物的设计,可成功酶切pbc-hygro和mokc/mokd/moke/moki片段,然后连接后获得重组质粒,本发明方法方便、高效。
(3)本发明采用基于原生质体的电击转化介导红曲霉m1的monacolink基因过表达方法,可一次转入大量质粒,不会影响到红曲霉基因组序列长度和碱基序列。实践中发现,当前热门的remi转化方法并不适用于本发明,因为remi转化不能够将质粒切开后连接于红曲霉基因的dna上。经过大量研究和筛查,最终确定采用本发明提供的电击转化的方法和条件。
(4)本发明所得基因工程菌产monacolink能力强,产monacolink的能力比其出发菌红曲霉最高提高2.3倍。
(5)本发明所得基因工程菌在高产monacolink的同时,将桔霉素含量降低至0.25μg/l以下,远低于国标中相关规定(液态样品中桔霉素的定量限为50μg/l,对固态样品中桔霉素的定量限为1mg/kg)。进而保证红曲产品的安全性,达到改善红曲产品质量的目的。
附图说明
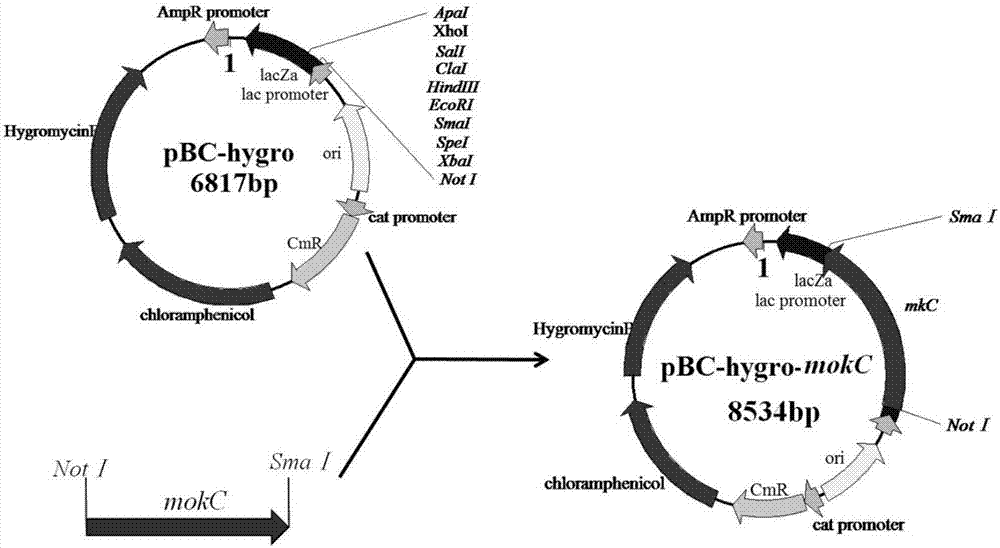
图1为过表达质粒pbc-hygro-mok的构建技术路线示意图(以pbc-hygro-mokc为例)。
图2为紫色红曲霉m1转录组rna的琼脂糖凝胶电泳图;m:dl2000dnamarker;泳道1-3:紫色红曲霉m1转录组rna。
图3为目标基因pcr产物的琼脂糖凝胶电泳图;m:dl2000dnamarker;泳道1-3:mokc基因的pcr产物。
图4为目标基因pcr产物的琼脂糖凝胶电泳图;m:dl2000dnamarker;泳道1-5:mokd基因的pcr产物。
图5为目标基因pcr产物的琼脂糖凝胶电泳图;m:dl2000dnamarker;泳道1-5:moke基因的pcr产物。
图6为目标基因pcr产物的琼脂糖凝胶电泳图;m:dl2000dnamarker;泳道1-5:moki基因的pcr产物。
图7为重组质粒pbc-hygro-mokc酶切验证的琼脂糖凝胶电泳图;m:dl10000dnamarker;泳道1:pbc-hygro-mokc/quickcutsmai+quickcutnoti;泳道2:pbc-hygro/quickcutsmai+quickcutnoti。
图8为重组质粒pbc-hygro-mokd酶切验证的琼脂糖凝胶电泳图;m:dl10000dnamarker;泳道1:pbc-hygro-mokc/quickcutsmai+quickcutnoti;泳道2:pbc-hygro/quickcutsmai+quickcutnoti。
图9为重组质粒pbc-hygro-moke酶切验证的琼脂糖凝胶电泳图;m:dl10000dnamarker;泳道1:pbc-hygro-mokc/quickcutsmai+quickcutnoti;泳道2:pbc-hygro/quickcutsmai+quickcutnoti。
图10为重组质粒pbc-hygro-moki酶切验证的琼脂糖凝胶电泳图;m:dl10000dnamarker;泳道1:pbc-hygro-mokc/quickcutsmai+quickcutnoti;泳道2:pbc-hygro/quickcutsmai+quickcutnoti。
图11为重组质粒pcr验证;m:dl2000dnamarker;泳道1:pbc-hygro-mokc;泳道2:pbc-hygro。
图12为重组质粒pcr验证;m:dl2000dnamarker;泳道1:pbc-hygro-mokd;泳道2:pbc-hygro。
图13为重组质粒pcr验证;m:dl2000dnamarker;泳道1:pbc-hygro-moke;泳道2:pbc-hygro。
图14为重组质粒pcr验证;m:dl2000dnamarker;泳道1:pbc-hygro-moki;泳道2:pbc-hygro。
图15为紫色红曲霉m1对潮霉素b的耐受浓度以及电击条件筛选。
图16为紫色红曲霉m1电击转入重组质粒后在不同浓度潮霉素平板上的生长情况。
图17为导入pbc-hygro质粒的红曲霉转化子monacolink产量分析
图18为导入pbc-hygro-mokc质粒的红曲霉转化子monacolink产量分析。
图19为导入pbc-hygro-mokd质粒的红曲霉转化子monacolink产量分析。
图20为导入pbc-hygro-moke质粒的红曲霉转化子monacolink产量分析。
图21为导入pbc-hygro-moki质粒的红曲霉转化子monacolink产量分析。
图22为导入mok基因的红曲霉转化子及空白质粒的转化子与原始出发菌株的桔霉素产量比较。
图23为红曲霉转化菌株潮霉素转录组的pcr电泳结果。
m:dl2000dnamarker;泳道1-2:pbc5、pbc6菌株cdna的pcr;泳道3-4:c2、c8菌株cdna的pcr;泳道5-6:d7、d8菌株cdna的pcr。
图24为潮霉素基因测序结果比对(mokc)。
具体实施方式
下面结合具体试验方法和附图对本发明的技术方案及其所产生的技术效果做进一步的阐述,下述说明仅是为了解释本发明,但不以任何方式对本发明加以限制,基于本发明教导所作的任何变换或替换,均属于本发明的保护范围。本发明所述方法如无特殊说明,均为本领域常规方法。所用试剂如无特殊说明,均可以从商业途径获得。
实施例一、高产monacolink的红曲霉基因工程菌的构建
1.过表达质粒构建技术路线
本发明高产monacolink的红曲霉基因工程菌,以pbc-hygro质粒为过表达质粒,过表达mokc或mokd或moke或moki基因获得。路线是:提取红曲霉rna,反转录为cdna,以cdna为模板扩增目的片段。将目的片段分别双酶切连接后,构建重组质粒,以pbc-hygro-mokc为例,技术路线如图1所示。
2.红曲霉rna的提取
将发酵培养至第8d的紫色红曲霉m1菌丝体纯化后进行转录组rna的提取,提取方法为本领域常规方法。取7μlrna样品进行1%琼脂糖凝胶电泳检测,电泳结果如图2所示,得到清晰的rna条带,且28s条带的亮度是18s的二倍,说明rna提取成功。
3.cdna的反转录
按照试剂盒(常规市售反转录试剂盒即可,如fermentask1622revertaidtmfirststrandcdnasynthesiskit反转录试剂盒)步骤,将rna反转录成cdna。
4.目的基因的扩增
以紫色红曲霉m1的cdna为模版,用上下游引物来扩增mokc/mokd/moke/moki基因。用来扩增mokc/mokd/moke/moki基因的特异性引物序列如表1所示。
pcr反应体系和pcr反应条件如下所示:
表2.扩增mok基因的pcr反应体系
pcr反应条件:
取7μlpcr产物进行电泳检测,电泳结果如图3至图6所示,pcr扩增后均得到单一条带,图3所示扩增条带长度约1500bp;图4所示扩增条带长度约0.8kb;图5所示扩增条带长度约1.0kb;图6所示扩增条带长度约1.8kb;条带长度分别与ncbi上公布的mokc/mokd/moke/moki基因片段大小一致,并且测序验证相似度高达100%,表明目标基因扩增成功。切胶回收获得目的片段。
表1.特异性引物表
5.pbc-hygro-mok重组质粒的构建与验证
分别用内切酶酶切pbc-hygro和mokc/mokd/moke/moki片段,酶切产物纯化后进行连接,连接产物转化大肠杆菌感受态dh5α,在34μg/ml氯霉素平板上筛选阳性转化子,提取质粒,取7μl重组质粒与原始质粒进行电泳检测,重组质粒长度与预期大小相符。
将大小正确的重组质粒用同一组内切酶进行双酶切验证,取7μl酶切产物进行电泳检测,如图7至图10所示,经过双酶切后分别得到大小在1500bp、0.8kb、1.0kb、1.8kb左右的目的片段带,与预期大小相符,初步预测质粒构建成功。
将大小正确的重组质粒和原始质粒分别用表1的特异性引物进行pcr验证,pcr条件同上。结果如图11至图14所示,重组质粒中有一条大小正确的扩增条带,原始质粒中没有扩增条带,进一步表明本发明重组质粒构建成功。
将上述重组质粒的pcr产物酶切回收后进行测序验证,测序结果与ncbi公布的mokc/mokd/moke/moki基因进行序列比对,同源性为100%,充分证明本发明pbc-hygro-mok重组质粒构建成功。
其中,酶切体系如下:
37℃酶切90min。
连接体系如下:
16℃条件下孵育过夜。
6.红曲霉原生质体的制备
(1)红曲霉接种至斜面培养基,30℃培养7d~10d。
(2)用0.9%无菌生理盐水洗涤斜面并收集孢子,制备成均一浓度的孢子悬液(1×108-1×109个/ml)。
(3)取200μl均一孢子悬液涂布在已铺有玻璃纸的pda固体培养基上,30℃培养40-45h。
(4)用涂布棒刮取玻璃纸上的菌丝体并收集到500目尼龙布上,用0.6mmgso4溶液洗1-2次。
(5)取约0.3g菌丝体于100ml三角瓶中,并加入30ml裂解酶液,于30℃,60r/min酶解2.5h,然后用四层擦镜试纸过滤,滤液于4℃,7000r/min离心10min,弃上清。收集原生质体。
7.红曲霉潮霉素耐受性检测及电压条件筛选
将上述步骤6获得的紫色红曲霉m1的原生质体涂布于含不同浓度梯度的潮霉素b抗性平板上,30℃培养4-5d,抑菌情况如图15所示。从图中可以看出,随着潮霉素b浓度的增长,紫色红曲霉m1的菌落数逐渐减少,当潮霉素b浓度为10μg/ml时,紫色红曲霉m1基本不生长,当潮霉素b浓度为10μg/ml时,加入质粒与不加入质粒的紫色红曲霉m1生长情况出现差异。因此,将浓度为10μg/ml潮霉素b确定为m1的最佳抑制浓度。
在现有电击转化条件的基础上,对红曲霉m1的电击转化条件进行筛选,如图15所示,当电压在3.0kv/cm时,10μg/ml的潮霉素b平板上的单菌落数量相对较多,因此,将电压在3.0kv/cm、潮霉素b浓度为10μg/ml作为实验电击转化条件。
8.基于原生质体的电击转化
将上述步骤5构建的重组质粒电击转化进入红曲霉感受态细胞中,生长结果如图16所示。将10μg/ml的潮霉素b平板上的单菌落在潮霉素平板上传代培养5代,获得基因工程菌。
电击转化步骤如下:
(1)红曲霉原生质体准备
将收集的原生质体沉淀用1.0mol/l预冷的山梨醇溶液洗涤2次,然后再将其平均分成2份。一份用预冷的1.0mol/l山梨醇溶液重悬后,于-80℃保存。另一份则用1.0mol/l山梨醇溶液重悬后置于冰上备用。
(2)基于原生质体的电击转化
①将重悬于预冷山梨醇溶液中的原生质体离心,弃上清,重悬于电击转化液i中,调整原生质体浓度至5x107个/ml;
②取70μl电机转化细胞,加入1μg质粒;
③将电击转化杯预冷,在电压强度为2.5kv/cm时进行电击;
④点击后立即加入900μl预冷的复苏培养基,转入1.5ml无菌离心管中,冰浴10min;
⑤30℃、100r/min培养2h;
⑥取200μl转化液涂布于含0、5、8、10、20μg/ml潮霉素b的再生培养基上,30℃培养4-5天。
9.基因工程菌发酵培养
将上述步骤8的基因工程菌进行发酵培养,条件如下:菌株在pda斜面30℃培养7d~10d,收集孢子并制备成均一的孢子悬液浓度为1×107~1×108个/ml。10%接种量接种装有30ml马铃薯葡萄糖液体培养基的250ml摇瓶,200r/min,30℃培养2天。以15%的接种量转接装有70mlyes液体培养基(4%蔗糖,2%酵母浸提物)的500ml摇瓶,28℃静止培养14天。
10.含不同转化子的基因工程菌的monacolink产量检测
monacolink的检测:采用hplc法,色谱柱:inertsilods-3c18(150mm×4.6mm×5μm),流动相:0.1%磷酸:甲醇=1:3,流速1ml/min,检测器为紫外检测器(pda),检测波长237nm,检测温度30℃,进样量10μl。
空载质粒(pbc-hygro)转化子monacolink产量如图17。取产量处于平均值的pbc5或pbc6作为对照菌株进行后续产量比较研究。
导入pbc-hygro-mokc质粒的转化子monacolink产量如图18,与对照菌株(pbc5或pbc6)相比,c2,c8转化子的monacolink产量增加最为显著,即c2、c8转化子的monacolink产量分别上调了2.0、2.3倍。
导入pbc-hygro-mokd质粒的转化子monacolink产量如图19,与对照菌株(pbc5或pbc6)相比,d7、d8转化子的monacolink产量增加最为显著,即d7、d8转化子的monacolink产量分别上调了1.5、2.2倍。
导入pbc-hygro-moke质粒的转化子monacolink产量如图20,与对照菌株(pbc5或pbc6)相比,e1、e3转化子的monacolink产量增加最为显著,即e1、e3转化子的monacolink产量分别上调了0.7、0.9倍。
导入pbc-hygro-moki质粒的转化子monacolink产量如图21,与对照菌株(pbc5或pbc6)相比,i1转化子的monacolink产量增加最为显著,即i1转化子的monacolink产量分别上调了0.1倍。
因此,选取monacolink产量处于平均值的pbc5、pbc6作为候选对照菌株,选取monacolink产量最高的c2、、c8作为候选mokc基因的过表达菌株,选取monacolink产量最高的d7、d8作为候选mokd基因的过表达菌株,选取monacolink产量最高的e1、e3作为候选mokd基因的过表达菌株,选取monacolink产量最高的i1作为候选moki基因的过表达菌株,进行潮霉素验证。
11.含不同转化子的基因工程菌的桔霉素产量检测
桔霉素检测——hplc法:将发酵液与菌丝体混合研磨,取10ml研碎液,加乙醇定容至30ml,混匀,60℃水浴1h。3000r/min离心15min。用孔径为0.45μm偏氟微孔滤膜微滤后直接用于hplc分析。检测条件:荧光检测波长λex=331nm,λem=500nm;流动相:v乙氟:v水=35:65(ph调2.5);出峰时间:12.324min,流速:1.0ml/min,柱温:28℃。
将导入pbc-hygro-mokc/mokd/moke/moki质粒的转化子c8、d8、e3、i1的桔霉素产量与原始出发菌株m1(来自中国普通微生物菌种保藏管理中心cgmcc3.0568)的桔霉素产量进行对比,以导入pbc-hygro空白质粒的pbc5为空白对照。结果如表3及图22所示。
表3.各菌株桔霉素产量
由以上结果可以看出,导入mok基因的转化子c8、d8、e3、i1中没有检测到桔霉素,说明过表达mokc/mokd/moke/moki基因后,对桔霉素的编码基因的表达起到了一定的抑制作用。
12.转化子的潮霉素pcr验证
提取红曲霉转化子pbc5,pbc6,c2,c8,d7,d8,e1,e3,i1,i5的rna,反转录cdna,以cdna为模板,以hph-f和hph-r为引物(序列如表4),pcr潮霉素基因,pcr结果如图23。从图中看出,可以准确扩增出1000bp左右条带,初步证明过表达菌株构建成功。将潮霉素片段切胶回收后,进行测序分析,与原质粒中潮霉素片段进行对比分析,相似度高达99%(图24,以mokc为例),证明过表达菌株构建成功。
表4.潮霉素引物
sequencelisting
<110>北京工商大学
<120>高产monacolink的红曲霉基因工程菌的构建方法及其应用
<130>
<160>14
<170>patentinversion3.3
<210>1
<211>1575
<212>dna
<213>monascuspurpureuswent.
<400>1
atgacagttccgacagatacggtctcgcgtcgcctccagtctctcgcttggagcgacata60
aagcagcatgccccatggctgccgtcttctcgcactcttgtctcgggatttctctgtttg120
atccttcttcagattctgtactctcgtggacgcaaatccgacctgcgggtgtataacccg180
aagaaatggtgggaactcaccaccatgagggccaagcgggagttcgatgcgaatgcgccc240
gcatggattgaggcctggttctccaagaacgaccaaccgctccgtttcattgtcgactct300
ggctattgtaccattttgccctcatcgatggccgacgaattccggaagatgaaggagctt360
tgcatgtacaagtttttgggcacggacttccactctcatcttcccggattcgacggcttc420
aaagaagtcaccagagacgcacatctcatcaccaaggtggtcatgaatcagttccaaacc480
caagctgcgaaatacaccaagcctcttgccgatgaggccagcgcgacaattgcagatatc540
tttggggacaacaaggaatggcacacggctcctgtctataacgagtgtttagatttggtg600
acacgcactgtcactttcatcatggtcggggataaactggcgcacaacgaggaatggttg660
gatatcgccaagcaccacgcggtgactatggcgattcaggctcgccaactccgcctctgg720
cccgtcattctgcgccctatagtccactggctcgagccccagggagccaaactgcgggcg780
caagtccgacgagcccggcagcttctcgagcccatcatccaggagcgacgcgccgagaag840
gccaaatgcctcgcccaagggatcgagccgccgcgctacgtcgattccattcagtggttt900
gaagacaccgccaagggccaatggtacgatgcggcgggggcgcagttggccatggacttt960
gccggcatctacggcacctctgacctgatgatcggcgggctggtcgacatcgtccgccac1020
ccgcatctcatcgagccccttcgcaacgagatccggaccgtcatcggcgaggaaggctgg1080
acgccggcctcgttgtacaaactcaagcttctggacagctgtctcaaggaatcgcagcgc1140
gtcaagcccgtagaatgcgccacgatgcgcagctacgcgctccagaacgtgaccttctcc1200
aatggaacctttgtcccgaaaggcgagctggtagctgtggcggccgaccgcatgagcaac1260
cctgaagtctggcctgagccgaagaagtacgacccgtaccgatacatgcgcctgcgagag1320
gatcccgacaaggcgttcagtgcccagctggagaacaccaacggtaaccatatcggcttt1380
gggtggcatcctcgggcgtgtcccggacggttcttcgcctccaaggagatcaagatcatg1440
ttggcgtttctgctgattcggtacgactggaagctggtgccaaacgagccgttgcagtat1500
taccgtcattccttcagcgtgcgcatccatcctgccaccaagcttatgatgcgccgtcgg1560
gacgaagatctctga1575
<210>2
<211>792
<212>dna
<213>monascuspurpureuswent.
<400>2
atgcgtatccaacgcacccccgcgccccccaaagcgccccgggccctcctctgcgtgcac60
ggcgccggctgctcccccgccatcttccgcgtgcagctctccaaactccgcgccgcttta120
cgggaagactttgaattcgtctacgcaacggcacccttcccttccgcccctggacccggg180
atcctgcccacgttcgaaggcctcgggccctattacacctggttcgagggttctccgtct240
ggtgctgctgccaaaggggataacagcaacagcaacgacagcaactcttctcccaccgta300
cacgaccgcctcgccgccgtccacgaacccgtccgccgcgccatcgccgaatggcagacc360
cagaaccccagcatccccatcgtgggaaccgtcagtttctccgagggcgctcttgtgacc420
gccctgctgctctggcagcagcagatgggtaggataccctggctcccggtcatgcaggtt480
gcgatgttcatctgctgctggtatcagcatgaggcgactcagtatatgcgggaggaggta540
ggctgcggcggtgatggtggcattgacggtgagaagttggtgatccggggggtgctgtcg600
ctgcacctgcagggtcgtgatgactttgccctggcgggttcgaagatggtggtggcgcag660
cattttgtgcccggggaggcgcaggtgttggagtttgcgggccggcatcatttcccgaat720
cggccgcgcgatgtgttggagacggttaagcggtttaggcagctgtgtgtgagggctaga780
gtcattgggtga792
<210>3
<211>1083
<212>dna
<213>monascuspurpureuswent.
<400>3
ttaccccaacttcaccacaaccttctctcccgacaactgcccagtcctaaccaactccat60
cccctgcttaatggcctcgaacccgccaggaatcacccgcagaggatggtgcacgatctt120
cccatcctccacgagccttgccgcaacgcgccacaactcctcgccgaatgcgcggtcctt180
ctcgctccccggccgcccatacggcgccggccaagtcgacccttccccgaaaatggtcgg240
ccccagggtccagtcggcggtgaccattttccgggtcgccgcgtgttccgggaacgggtt300
cagggagacatagcggccgccggcgcggccgatggcggcgaagcagagggtggtggactc360
gacgttggtgatgcaatcgagggcgtagcggaggttgtttttggtgtatgtgcgaatcgt420
ctgcgcgagaccggcatcacgatagtcaaagacatcttctgcgccgtgctttttggcgag480
gtcgaaattgtgcggggagcatgtggctatggggatgtatccggacaagcgtagcatctg540
catcacaatcgtcgcagtagcggtgctacccccatacaccaaaacatacacgggcttcgg600
aagggccggtgccgtcgtagcattgggatccggtaaaggcagcccgagcagcttcatcgc660
caggccggccgtgctgatgcccgcgggcaacgacgcggcctgctcgaaggaccagccctc720
agggaccgtggcccagatgcggccgcgggtgaccgtgtactgcgagaaggcgccctggtc780
gggggtgcgcggacacatctcgttctgggcgccgtagacccggtccccgacttggatgtg840
cgtgacctgcgatcccacggcgacgacggtgccggcgtagtcggtgcccaggaaggcgta900
gggggtggcgaattggccgcgcatcttcgtgtcgctggggttgagggctacggcctcgac960
gcgcacgcagacctgatcgggggggaggcttggacagggggcgacgtcccagatggtgac1020
tttgtcatgctcgtccactgtcagcgcagtctggtgaggtggtagggtgaaggtgatggt1080
cat1083
<210>4
<211>1632
<212>dna
<213>monascuspurpureuswent.
<400>4
ctagactcgttcatcgcggccgaaccccgtcctttcctccccccaccaaatcaaaccgac60
ctgacccattccccacgcaaacaaaaaagtggctcccgatgccccgaccgcaaggtagaa120
tgtatggtctatccctttacagtatgccaggagaacctgagaaaggagatttgcggggac180
gacatcgcggaagcccgtggcgcccgcggccgtcaccgcctctgcgctgacggcgggcgc240
atactcgcgtaagccggcgtcaaggccggcgctgaagacgagcttggccaaggtgaggaa300
gagggagccgccgaaggtctggccgaacatggccagcgagataccgagcgcactcatttg360
tggtggcagggcatgttgaatggcaatgatgggggtctgcattccacatcctcgaccaaa420
gccccccatgaattgatacatcacccacgctgcgattgaggtatgcggctggaaggtcga480
taccaggcctgcggagatagatacgatgatcccactggcaagcgcccagggaatgtaata540
ccccgttttgccgatgattgcgccggagacgatcgccatcacaatttgtccccctatgcc600
tggaagcatatacaccccactcatggtgggagagacgtccttcacggcctggaagtagat660
aggaaggtagtaagagaaggtgagcaaggccccggagaaaaagcccaagaaaagacacga720
gcaccagacttgtcgacgactggccaccgagaaaggaatcatcgccatcgagttgcctac780
gtagcgttcccagaacccaaacaccaccagcgagatgccggcaccacagaacaagccgat840
gattacggagctatcccacatataatcggacccgccccattctaacgcaagtgagatcat900
cacggcaaaggcggcgaagagcacaaaccccaggagatcgagctgactcaggatgacccg960
gaccccaacagctctgggagcaggcattgtgcaatccgatgttggcacactcttaggaat1020
gtggatggcgagaaggaggatggcggccagcgctccgacaggaagattgatatagaagca1080
ccaccgccaacttgcgtgttgggtaaaagcacctcccagcaatggtccacagacaatggc1140
aatttggctcagacccatcatgatcccgattaacagtggttgctgttgttttggagctgc1200
agaggcgaggatggtgatggctccattggtgagccccgatcctcccatgccagccactgc1260
gcgccccacgatcaacatagttgaacaacgcgcggcgccacagagagctgatccgacttc1320
aaacaccccgaggaatgctaggaaggtatacttcaatgtcaagagagtgtagagcttccc1380
tgccaagggctgtagagcacagctcgacaaaagatacgcacttccataccatccaacatc1440
cccaagggaatgaaactgagcagtgatatgggggatagctgtcacaatgatagacatgtc1500
caacaacatcaaaaacaccaccagggtcaccgaggttactaccagtcctagctttaggcc1560
ggtgacatggctgacctggacttcagtggtgcagctctgtggcttctctttctcagactg1620
gtgggaagccat1632
<210>5
<211>39
<212>dna
<213>人工序列
<400>5
aaggaaaaaagcggccgcatgacagttccgacagatacg39
<210>6
<211>29
<212>dna
<213>人工序列
<400>6
tcccccgggtcagagatcttcgtcccgac29
<210>7
<211>27
<212>dna
<213>人工序列
<400>7
gctctagaatgcgtatccaacgcaccc27
<210>8
<211>29
<212>dna
<213>人工序列
<400>8
cccaagctttcacccaatgactctagccc29
<210>9
<211>30
<212>dna
<213>人工序列
<400>9
gctctagaatgaccatcaccttcaccctac30
<210>10
<211>30
<212>dna
<213>人工序列
<400>10
cccaagcttttaccccaacttcaccacaac30
<210>11
<211>27
<212>dna
<213>人工序列
<400>11
gctctagaatggcttcccaccagtctg27
<210>12
<211>28
<212>dna
<213>人工序列
<400>12
cccaagcttctagactcgttcatcgcgg28
<210>13
<211>21
<212>dna
<213>人工序列
<400>13
atgcctgaactcaccgcgacg21
<210>14
<211>21
<212>dna
<213>人工序列
<400>14
gataaggaaacgggagcctgc21
- 还没有人留言评论。精彩留言会获得点赞!