多囊卵巢综合征基因表达分析的内参基因组合及应用的制作方法
本发明涉及分子生物学和疾病基因研究和诊断领域。具体的,本发明涉及在多囊卵巢综合征(pcos)的基因表达分析的研究中采用的内参基因组合和检测多囊卵巢综合征的内参基因组合。
背景技术:
多囊卵巢综合征(pcos),症状包括排卵功能障碍、多囊卵巢、雄激素过多,是常见的妇科内分泌疾病,并且是女性生殖失败的主要原因(kovacsandwood,2001;wild,etal.,2000)。根据不同的诊断标准,其患病率的估计是6%~18%(azziz,etal.,2004;christian,etal.,2003;march,etal.,2010)。多囊卵巢综合征患者易发生多种代谢紊乱包括胰岛素抵抗、2型糖尿病(t2dm)、肥胖、血脂异常、慢性低水平炎症和心血管疾病(cvd)以及心理障碍(azziz,etal.,2004;ehrmann,2005;jayasenaandfranks,2014;kovacsandwood,2001;orio,etal.,2004;ozcandag,etal.,2015;spritzer,etal.,2015;stepto,etal.,2013;wild,etal.,2000)。
尽管pcos的确切发病机制仍不清楚,但经推断其机制可能涉及到卵泡发育过程中卵巢颗粒细胞(gcs)介导的异常的激素反应。已有的研究表明,pcos的发生与pcos患者的颗粒细胞中促黄体生成激素(lh)相对于lh的升高以及高表达和过度激活的促黄体激素/绒毛膜促性腺激素受体相关,阻碍了卵泡的成熟和排卵过程(ehrmann,etal.,1995;jakimiuk,etal.,2001;kanamarlapudi,etal.,2016;willis,etal.,1998;yong,etal.,1992)。此外,颗粒细胞功能受损还会导致卵巢内雄激素和体
正常和疾病样本中基因表达谱的比较为pcos发病机制的研究提供了宝贵的资料。实时定量pcr(qrt-pcr)现已被大量、广泛的用于检测器官和组织甚至单细胞中的基因表达差异,常被用来去寻找pcos的调控基因、相关的信号通路以及非侵入性的生物标志物(adams,etal.,2016;chazenbalk,etal.,2012;deresende,etal.,2012;lemieux,etal.,1999;long,etal.,2014;salilew-wondim,etal.,2015;schmidt,etal.,2014;wissing,etal.,2014)。
在基因表达研究中的各种方法,包括测序法、qrt-pcr和microarray(基因芯片)中,均需要使用内参基因。内参基因通常在所有样本和实验条件下都能稳定地表达,而且具有与研究对象相似的属性,例如rna稳定性和大小等,由此表达量可以与研究对象进行比较。内参基因在很大程度上能够影响测序法、qrt-pcr和microarray(基因芯片)的准确性。现在的多数研究只是选择一个内参基因,如β-肌动蛋白(actb)、18s核糖体rna、肽基脯氨酸异构酶b(peptidylprolylisomeraseb,ppib)或甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphatedehydrogenase)等。越来越多的证据发现这些原来被认为是能够稳定表达的内参基因实际上在不同生理或病理条件下的表达是有变化的(meller,etal.,2005;patel,etal.,2002)。采用不恰当的看家基因(housekeepinggene)可能会导致数据曲解引起基因表达的低估或高估(dheda,etal.,2005;murphyandbustin,2009;nestorov,etal.,2013)。
因此本领域还需要在患者或正常人的样品中对多囊卵巢综合征(pcos)通过基因表达分析进行研究和检测,特别是寻找在颗粒细胞样品中进行基因表达分析时,对基因表达进行标准化(normalization)的更可靠的内参基因或基因组合。
技术实现要素:
本发明提供了对健康人群和多囊卵巢综合征(pcos)患者的样本进行有关的研究,特别是在颗粒细胞样本进行基因表达分析时作为内参基因的基因组合。本发明也提供了对多囊卵巢综合征(pcos)患者的样本进行研究或检测,特别是在颗粒细胞样本中通过分析基因表达来进行研究或检测多囊卵巢综合征时作为内参基因的基因组合。
具体的,本发明提供了次黄嘌呤磷酸核糖转移酶1(hprt1)、核糖体磷酸化蛋白大亚基p0(ribosomalproteinlateralstalksubunitp0,rplp0)和羟甲基胆素合成酶(hydroxymethylbilanesynthase,hmbs)的基因的组合在研究多囊卵巢综合征中作为内参基因的用途,特别是在人的颗粒细胞样品中研究多囊卵巢综合征时作为内参基因。该研究通常是通过细胞中的基因表达谱分析来进行。
颗粒细胞是卵泡(follicle)中卵母细胞四周一层扁平或立方形细胞。在卵泡开始发育、卵细胞成长的同时,这些颗粒细胞由扁平变为立方形,并由单层增生成复层。
内参基因是用于检测基因表达水平变化时用于参照物的基因。内参基因需要选择在研究对象(组织和细胞等)中表达相对恒定的基因。内参基因可以在例如实时荧光定量pcr、northernblot等方法中用作校正例如上样量误差或上样过程中的操作误差等差异,保证实验结果的准确性。通常采用看家基因(house-keepinggenes)作为内参基因。在测试中,可以采用一个或多个基因作为内参基因。
次黄嘌呤磷酸核糖转移酶1(hypoxanthine-guaninephosphoribosyltransferase),即hprt1,是一种人类蛋白,其作用是将5-磷酸核糖-1-焦磷酸盐中的5-磷酸核苷基团转移至嘌呤上,可催化次黄嘌呤转化为肌苷酸也可催化鸟嘌呤转化为环磷酸鸟苷。hprt1在核苷酸再利用合成途径中扮演重要角色。编码hprt的基因为hprt1(ensemb1:ensg00000165704)。
核糖体磷酸化蛋白大亚基p0(ribosomalproteinlateralstalksubunitp0),即rplp0,是一种人类蛋白,是组成核糖体60s亚基的一个核糖体蛋白,属于l10p核糖体蛋白家族。它是一种带有一个羧基末端的中性磷蛋白,与同样带有羧基末端的酸性核糖体磷酸蛋白p1和p2非常相似。p0蛋白可以与2个p1及p2形成的二聚体共同组成一个五聚体。p0蛋白位于细胞质内。作为一个典型的基因编码的核糖体蛋白,它有许多个已加工假基因分散在基因组中。编码rplp0的基因为rplp0(ensemb1:ensg00000089157)。
羟甲基胆素合成酶(hydroxymethylbilanesynthase),即hmbs,是一种人类蛋白,是羟甲基胆素合成酶超家族中的一员。这个蛋白是胆素从头到尾合成途径的第三个酶,可催化四个胆色素原分子合成线性的羟甲基胆素。编码hmbs的基因为hmbs(ensemb1:ensg00000256269)。
本发明提供了采用hprt1,rplp0和hmbs这三个基因的组合在用于制备研究或检测多囊卵巢综合征的试剂盒或装置中作为内参基因的用途。优选的,所述试剂盒或装置用于在受试者的颗粒细胞样品中进行研究或检测多囊卵巢综合征。对多囊卵巢综合征的检测也包括对多囊卵巢综合征的诊断。在本发明的其中一个方面,所述试剂盒为用于通过实时荧光定量pcr方法研究或检测多囊卵巢综合征的试剂盒。在本发明的其中又一个方面,所述装置为用于研究或检测多囊卵巢综合征的基因芯片。
本发明还提供了用于研究或检测多囊卵巢综合征的试剂盒或装置,其中采用hprt1,rplp0和hmbs的组合作为内参基因。优选的,所述试剂盒或装置用于在颗粒细胞样品中研究或检测多囊卵巢综合征。在本发明的其中一个方面,所述试剂盒为用于通过实时荧光定量pcr方法研究或检测多囊卵巢综合征的试剂盒。在本发明的其中又一个方面,所述有时装置为用于研究或检测多囊卵巢综合征的基因芯片。
在本文中,蛋白符号不用斜体,并全部大写;基因符号使用斜体。例如蛋白核糖体磷酸化蛋白大亚基p0写为rplp0,编码该蛋白的基因写为rplp0。但有时在本文中基因符号也不使用斜体。例如有时本文中“rplp0”或“rplp0基因”表示编码蛋白rplp0的基因rplp0。
附图说明
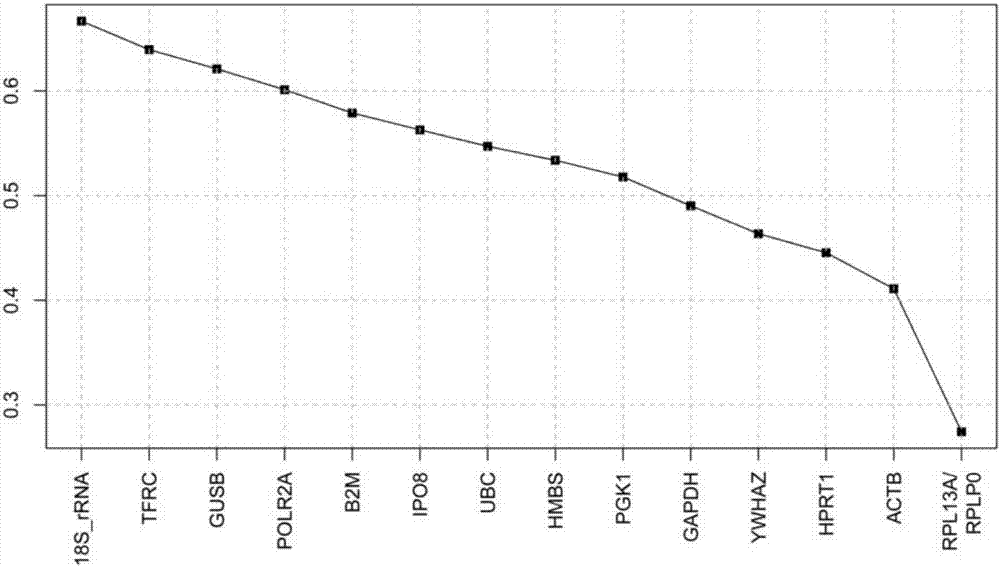
图1为测试人员的颗粒细胞样品的qrt-pcr的循环阈值(ct)。
图2为测试人员的颗粒细胞样品的基因表达稳定性的genorm算法测试结果。
图3为测试人员的颗粒细胞样品的内参基因组合中基因个数的genorm算法。
图4为测试人员的颗粒细胞样品的基因表达稳定性的normfinder算法测试结果。
图5为测试人员的颗粒细胞样品的基因表达稳定性的bestkeeper算法测试结果。
具体实施方式
下面将结合实施例进一步说明本发明的实质内容和有益效果,该实施例仅用于说明本发明而非对本发明的限制。
实施例1
研究人员征集
本研究从2015年1月到2016年6月,涉及89个中国籍汉族妇女,年龄为29.12±3.01岁,来自山东大学附属医院。其中包括44个多囊卵巢综合征(pcos)女性患者和正在接受输卵管和/或男性不育治疗的45个具有正常排卵功能的妇女。患者或家属全部提供了书面同意通知书。本研究得到了道德委员会的同意。
实施例2
临床和生化检测
多囊卵巢综合征(pcos)根据2003鹿特丹标准诊断(chang,etal.,2004;fauser,etal.,2004),包括以下三个临床特征中的任何两个:1.少/无排卵;2.临床和/或生化高雄激素血症;3.超声检查多囊卵巢,以及排除其它高雄性激素血症相关的病理生理状态,包括先天性肾上腺增生,cushing’s综合征,和分泌雄激素的肿瘤。
所有相关被研究者到门诊初诊时测量人体参数,如腰围、臀围、身高和体重等。身体质量指数(bmi)的计算为重量(公斤)除以高度的平方(m2),或bmi=kg/m2。静脉血标本在经过12小时的禁食后于上午8点到10点之间采集。多囊卵巢综合征妇女的所有血液样本在卵泡期早期即月经周期第3天-5获得。血清卵泡刺激素(fsh)、黄体生成素(lh)、睾酮(t)、雌激素(e2)、孕酮(p)和催乳素(prl)使用自动生化分析仪(奥林巴斯600生化分析仪,olympusdiagnosticagmbh,co.,爱尔兰)测量。
下面的表1给出44个pcos患者及45例正常妇女的临床和生化指标。
表1多囊卵巢综合征与正常女性测试者人体临床数据
bmi:身体质量指数;e2:雌激素;p:孕酮;prl:催乳素;t:睾酮;fsh:促卵泡激素;lh:促黄体生成素。
p值<0.05为有统计学意义
本研究中的多囊卵巢综合征患者及对照组妇女的临床研究血清中雌激素(e2)、孕酮(p)、催乳素(prl)、卵泡刺激素(fsh)都没有显著性差异。正如预期的那样,pcos组bmi显著高于对照组7.8%(多囊卵巢综合征24.20±4.73公斤/平方米,对照22.45±2.99公斤/平方米,95%ci:-1.771to9.411,t=1.358,df=87,p<0.05)。除此之外,与对照组相比,pcos组表现出高水平的睾酮(t)33.9%(多囊卵巢综合征:36.12±17.6ng/dl,对照:23.87±7.77,95%ci:6.540to17.96,t=4.264df=87,p<0.0001)。尽管fsh水平在两组没有区别,但pcos患者的促黄体生成激素(lh)和lh/fsh明显升高,分别为36.4%和39.8%(pcoslh:8.10±3.87iu/l,lh:对照5.1±1.84iu/l,95%ci:1.688to4.232,t=4.624df=87,p<0.0001;pcoslh/fsh:1.33±0.74,对照0.80±lh/fsh比值:0.29,95%ci:0.2942to0.7658,t=4.467df=87,p<0.0001)。
实施例3
颗粒细胞分离
从44个多囊卵巢综合征(pcos)女性患者和对照组的正在接受输卵管和/或男性不育治疗的45个具有正常排卵功能的妇女分离得到颗粒细胞。排除有其它妇科或内科疾病史的志愿者。所有的妇女在黄体中期开始注射促性腺激素释放激素(gnrh)激动剂,通过超声波扫描和血清雌二醇测定监测卵泡大小。当检测到三个或更多的卵泡平均直径达到r1.8cm时,超声引导下取卵手术36小时前,iu给药8000-10000人绒毛膜促性腺激素(profasi,serono)。麻醉后,通过17号双腔穿刺针(k-ops-wood-1235,cook澳大利亚)取卵。在卵母细胞周围收集颗粒细胞(gcs),用pasteurpipette抽吸去除卵母细胞后,用dulbecco′smodifiedeaglemedium(dmem)培养基洗涤两次,以作后续分析。用裂解液去除红细胞。
实施例4
rna提取和cdna合成
采用trizolplusrnapurificationkit(lifetechnologies)从分离得到的颗粒细胞中提取总rna。采用nanodrop2000(thermoscientific)检测rna纯度,以a260:a280比值为1.9-2.1为合格标准。总rna用reverse-transcribekit(takara)试剂盒转录为cdna,然后用无核酸酶水稀释到20μl。得到的cdna经1∶20稀释后作为qrt-pcr的模板。
实施例5
实时荧光定量pcr(qrt-pcr)
本发明采用次黄嘌呤磷酸核糖转移酶1(hprt1)、核糖体磷酸化蛋白大亚基p0(ribosomalproteinlateralstalksubunitp0,rplp0)和羟甲基胆素合成酶(hydroxymethylbilanesynthase,hmbs)的基因的组合作为在待测者的颗粒细胞中进行多囊卵巢综合征(pcos)检测时作为qpcr分析的内参基因。为此,对实施例1-4所述的健康对照组和多囊卵巢综合征(pcos)患者的颗粒细胞中的hprt1,rplp0和hmbs的表达进行测量。
另外,作为比较,对其它以下12个基因在上述测试样品中的表达也进行了测试:
18s核糖体rna(18srrna),β-肌动蛋白(actb)、甘油醛-3-磷酸脱氢酶(gapdh)、泛素c(ubc),β-2-微球蛋白(b2m),rna聚合酶ii亚基(polr2a)、酪氨酸3-单加氧酶/色氨酸5-加单氧酶激活蛋白z(ζ-tyrosine3-monooxygenase/tryptophan5-monooxygenaseactivationprotein,ywhaz),β-葡萄糖醛酸酶(gusb),输入蛋白8(importin8,ipo8)、磷酸甘油酸激酶1(phosphoglyceratekinase1,pgk1),转铁蛋白受体(tfrc)和核糖体蛋白l13a(rpl13a)。
qrt-pcr采用自动工作站epmotion5075lh(eppendorf,germany)来对pcr试剂进行精确加样。扩增反应采用quantstudiotm7flexreal-timepcrsystems(appliedbiosystem,usa)进行,以及使用poweruptm
qpcr中针对本发明的hprt1、rplp0和hmbs基因使用的引物序列为:
hprt1-fwd:tgacactggcaaaacaatgca(5′→3′)(seqidno.1)
hprt1-rev:ggtccttttcaccagcaagct(5′→3′)(seqidno.2)
rplp0-fwd:agcccagaacactggtctc(5′→3′)(seqidno.3)
rplp0-rev:actcaggatttcaatggtgcc(5′→3′)(seqidno.4)
hmbs-fwd:ggcaatgcggctgcaa(5′→3′)(seqidno.5)
hmbs-rev:gggtacccacgcgaatcac(5′→3′)(seqidno.6)
qpcr中检测前述待测看家基因使用的引物序列如表2所示:
表2引物序列
采用qrt-pcr对44例pcos患者和45例对照妇女的上述15个候选内参基因的表达水平测定其循环阈值(ct),结果如图1所示,其ct值表现出动态的表达,范围从最低的18srna的13.81±0.54到最高的hmbs的28.25±1.13,平均ct值为21.35±1.12。18srna和β肌动蛋白是最丰富的基因,其次是gapdh,而表达最少的基因是hmbs。对这些候选内参基因的表达的测量发现它们的表达具有明显的个体间差异,标准偏差范围从18srna的0.54到ubc的1.26。可见,还需要选择更可靠的内参基因或内参基因组合才能够在对多囊卵巢综合征(pcos),特别是在颗粒细胞样品中进行比较性基因表达研究作为基因表达标准化的内部控制的内参基因。
实施例6内参基因表达稳定性分析
(1)内参基因表达稳定性评估方法
本领域采用的评估待测基因组合作为内参基因的使用的稳定性的评估方法(chen,etal.,2011;zhang,etal.,2012)采取以下公式进行综合分析:
n=几何平均数(n1,n2,n3)(1)
即n=(n1xn2xn3)1/3
其中n为进行分析的基因组群中各基因的表达的稳定性数值,数值越小,表示稳定性越好;n1为根据genorm算法得到的排名;n2为根据normfinder算法得到的排名;n3为根据bestkeeper算法得到的排名。即先采用genorm算法、normfinder算法和bestkeeper算法等三种算法测试得到各基因表达的稳定性,然后计算这三种算法的排名的几何平均数。通过公式(1)对genorm算法、normfinder算法和bestkeeper算法的排名进行综合排名,可以消除只用单一方法可能造成的误差。
genorm算法(vandesompele,etal.,2002)可选择一个理想的内参基因组合,其中的内参基因的表达比率受不同的实验条件和样品等外部因素影响最小。稳定性m值是基于一个待测内参基因和所有其他待测内参基因的成对变异分析(pair-wisevariationanalysis),其值表示该内参基因表达的稳定性。在算法中,最高m值的基因被逐一淘汰,重复这个选择和排除过程,最后选择两个最稳定表达的基因。最低m值的基因被认为是最稳定的内参基因。
normfinder算法(andersen,etal.,2004)的原理和genorm算法类似,产生基因表达稳定值,然后根据稳定值排序,以表达稳定值最小的基因作为最稳定的基因。
bestkeeper算法(pfaffl,etal.,2004)是根据与bestkeeper指数相关的系数,即被测基因组合的ct值的几何平均数,来测定基因表达的稳定性。其中测算出变异系数(cv)和标准偏差(sd)。具有最低cv和sd值的内参基因则为表达最稳定的基因。具有sd>1的基因被排除。此算法考虑了bestkeeper指数和内参基因间的pearson相关系数、关联系数(coefficientofdetermination,r2)和p值。
被测多囊卵巢综合征患者和对照组的临床特点的数值变量被表示为平均值±sd。使用spssv.19.0(spss公司,芝加哥,il,美国)进行独立样本t检验。p值小于0.05被认为有统计学意义。
(2)genorm算法测试结果
如图2所示,本发明测试的3个基因(hprt1、rplp0和hmbs)以及其它12个内参基因(18srrna、actb、gapdh、ubc、b2m、polr2a、ywhaz、gusb、ipo8、pgk1、tfrc和rpl13a),在颗粒细胞中,其表达值m的平均数都低于genorm算法设定的阈值1.5。rpl13a和rlpp0具有最低的m值0.27,属于此算法算出的表达最稳定的基因,接着是β-actin(0.41),hprt1(0.45),ywhaz(0.46),gapdh(0.49),pgk1(0.52),hmbs(0.53),ubc(0.55),ipo8(0.56),b2m(0.58),polr2a(0.60),gusb(0.62),trfc(0.64),而18srna是表达最不稳定的,m值为0.67。
(3)normfinder算法测试结果
如图4所示,根据normfinder算法,在颗粒细胞样品中,hprt1具有最低的m值0.07,属于此算法算出的表达最稳定的基因,接着是ywhaz(0.09),actb(0.42),hmbs(0.11),pgk1(0.11),ubc(0.12),gapdh(0.19),gusb(0.2),ipo8(0.21),polr2a(0.22),rplp0(0.24),18srna(0.24),tfrc(0.25),rpl13a(0.25),而b2m是表达最不稳定的,m值为0.28。
(4)bestkeeper算法测试结果
如图5所示,根据bestkeeper算法,在颗粒细胞样品中,gusb的sd(±ct)和cv(%ct)的值分别为0.70和2.67,属于此算法算出的表达最稳定的基因,接着是18srna(sd:0.40,cv:2.87),hmbs(sd:0.90,cv:3.19),ipo8(sd:0.88,cv:3.38),tfrc(sd:0.85,cv:3.45),polr2a(sd:0.88,cv:3.58),hprt1(sd:0.88,cv:3.59),rplp0(sd:0.75,cv:3.77),rpl13a(sd:0.75,cv:3.97),ywhaz(sd:0.88,cv:4.12),pgk1(sd:0.95,cv:4.30),actb(sd:0.76,cv:4.36),ubc(sd:1.00,cv:4.72),b2m(sd:0.93,cv:4.73),而gapdh(sd:0.94,cv:5.02)是表达最不稳定的。
(5)内参基因表达稳定性评估
根据(2)-(4)中根据genorm算法、normfinder算法和bestkeeper算法得到的待测基因的表达稳定性的排名,根据(1)中的公式,可得到表3:
表3内参基因稳定性排名
可见,本发明采用的内参基因组合中的三个基因的稳定性在所测的15个基因中是最稳定的。
实施例7内参基因组合中采用基因个数的优劣比较
genorm算法可通过对标准化因子进行成对差异分析(pair-wisevariationanalysis)来评估用于基因表达标准化的内参基因的合适的个数(vandesompele,eta1.,2002)。在成对差异分析中,对于任何具有(vn/n+1)的值低于阈值0.15的n个内参基因的组合,多一个(即n+1个)的内参基因不会显著提高归一化的效果,从而是不需要再多一个内参基因的。
根据实施例5的44例pcos患者和45例对照妇女的上述15个候选内参基因的表达水平的循环阈值(ct)的检测结果,通过genorm算法进行分析。
结果如图3所示,发现v2/3值(r2of0.978)超过了阈值0.15,而v3/4值(r2of0.986)正好低于阈值0.15,这表明在颗粒细胞样本中进行多囊卵巢综合征(pcos)的基因表达谱研究时用3个看家基因作为内参基因是最合适的。
越来越多的证据证明,原来被认为是在不同样品中稳定表达,差异最小的看家基因,在不同的生理或病理情况下在不同样本(组织或细胞)中的表达也存在很大差异(meller,etal.,2005;patel,etal.,2002)。本发明在多囊卵巢综合征(pcos)女性患者和具有正常排卵功能的妇女中,在通过颗粒细胞中的基因表达谱分析来研究多囊卵巢综合征时,出乎意料地发现次黄嘌呤磷酸核糖转移酶1(hprt1)、核糖体磷酸化蛋白大亚基p0(ribosomalproteinlateralstalksubunitp0,rplp0)和羟甲基胆素合成酶(hydroxymethylbilanesynthase,hmbs)的基因在综合评估(chen,etal.,2011;zhang,etal.,2012)中是表达最稳定的看家基因。并且,在基因表达谱分析中,采用这三个基因的组合比其它采用一个、两个或四个看家基因作为内参基因更加适合。
由此,本发明提供了在通过细胞中的基因表达谱分析来研究多囊卵巢综合征中,特别是在人的颗粒细胞样品中,采用次黄嘌呤磷酸核糖转移酶1(hprt1)、核糖体磷酸化蛋白大亚基p0(ribosomalproteinlateralstalksubunitp0,rplp0)和羟甲基胆素合成酶(hydroxymethylbilanesynthase,hmbs)的基因的组合作为内参基因的方法。这几个内参基因的组合能够在基因表达谱分析,例如在实时荧光定量pcr、northernblot和基因芯片等应用中,用作校正上样量误差或上样过程中的操作误差等差异,进一步保证实验结果的准确性,帮助本领域的技术人员对多囊卵巢综合征进行研究或检测(诊断)。
以下论文的全文引入到本申请,作为本发明的说明的一部分。
1.auerspergn,wongas,choikc,kangsk,leungpc:ovariansurfaceepithelium:biology,endocrinology,andpathology.endocrrev2001,22(2):255-288.
2.redjimin,gaudinf,touboulc,emilied,pallardym,biola-vidammenta,fernandezh,prevots,balabaniank,machelonv:identificationofglucocorticoid-inducedleucinezipperasakeyregulatoroftumorcellproliferationinepithelialovariancancer.molcancer2009,8:83.
3.hennessybt,colemanrl,markmanm:ovariancancer.lancet2009,374(9698):1371-1382.
4.partridgeee,bamesmn:epithelialovariancancer:prevention,diagnosis,andtreatment.cacancerjclin1999,49(5):297-320.
5.rosenthalan,fraserl,manchandar,badmanp,philpotts,mozerskyj,hadwinr,caffertyfh,benjamine,singhnetal:resultsofannualscreeninginphaseioftheunitedkingdomfamilialovariancancerscreeningstudyhighlighttheneedforstrictadherencetoscreeningschedule.jclinoncol2013,31(1):49-57.
6.zhaoh,shenj,wangd,guoy,gregorys,medicol,huq,yanl,odunsik,lelesetal:associationsbetweengeneexpressionvariationsandovariancancerriskallelesidentifiedfromgenomewideassociationstudies.plosone2012,7(11):e47962.
7.shenj,wangd,gregorysr,medicol,huq,yanl,odunsik,lelesb,ambrosonecb,liusetal:evaluationofmicrornaexpressionprofilesandtheirassociationswithriskallelesinlymphoblastoidcelllinesoffamilialovariancancer.carcinogenesis2012,33(3):604-612.
8.centherv,massartdl,denoordoe,dejongs,vandeginstebm,stemac:eliminationofuninformativevariablesformultivariatecalibration.analyticalchemistry1996,68(21):3851-3858.
9.richardr.picardrdc:cross-validationofregressionmodels.journaloftheamericanstatisticalassociation1984,79:575-583.
10.caiw,liy,shaox:avariableselectionmethodbasedonuninformativevariableeliminationformultivariatecalibrationofnear-infraredspectra.chemometricsandintelligentlaboratorysystems2008,90(2):188-194.
11.chakrabortys,dattas,dattas:surrogatevariableanalysisusingpartialleastsquares(sva-pls)ingeneexpressionstudies.bioinformatics2012,28(6):799-806.
12.xuq-s,liangy-z,duy-p:montecarlocross-validationforselectingamodelandestimatingthepredictionerrorinmultivariatecalibration.journalofchemometrics2004,18(2):112-120.
13.gourvénecs,femándezpiernaja,massartdl,rutledgedn:anevaluationofthepolishsmoothedregressionandthemontecarlocross-validationforthedeterminationofthecomplexityofaplsmodel.chemometricsandintelligentlaboratorysystems2003,68(1-2):41-51.
14.irizartyra,hobbsb,collinf,beazer-barclayyd,antonelliskj,scherfu,speedtp:exploration,normalization,andsummariesofhighdensityoligonucleotidearrayprobeleveldata.biostatistics2003,4(2):249-264.
15.ish:onthestructureofpartialleast-squaresregression.communstat-simulationcomput1988.17:581-607.
16.ish:partialleast-squaresregressionandstatisticalmodel.scandjstat1990,17:97-144.
17.barkerm.rayensw:partialleastsquaresfordiscrimination.journalofchemometrics2010,17(3):166-173.
18.martinsjpa,teófilorf,ferreirammc:computationalperformanceandcross-validationerrorprecisionoffiveplsalgorithmsusingdesignedandrealdatasets
19.dings:partialleastsquaresbasedgeneexpressionanalysisinrenalfailure.diagnosticpathology2014,9(1):137-137.
20.gosselinr,rodrigued,duchesnec:abootstrap-vipapproachforselectingwavelengthintervalsinspectralimagingapplications.chemometricsandintelligentlaboratorysystems2010,100(1):12-21.
21.wangx,e1naqaim:predictionofbothconservedandnanconservedmicrornatargetsinanimals.bioinformatics2008,24(3):325-332.
22.shannonp,markiela,oziero,baligans,wangjt,ramaged,aminn,schwikowskib,idekert:cytoseape:asoftwareenvironmentforintegratedmodelsofbiomolecularinteractionnetworks.genomeresearch2003,13(11):2498-2504.
23.hanq-j,wuh-l,caic-b,xul,yur-q:anensembleofmontecarlouninformativevariableeliminationforwavelengthselection.analyticachimicaacta2008,612(2):121-125.
24.agostinim,knightra:mir-34:frombenchtobedside.oncotarget2014,5(4):872-881.
25.kumamotok,spillareea,fujitak,horikawai,yamashitat,appellae,nagashimam,takenoshitas,yokotaj,harriscc:nutlin-3aactivatesp53tobothdown-regulateinhibitorofgrowth2andup-regulatemir-34a,mir-34b,andmir-34cexpression,andinducesenescence.cancerresearch2008,68(9):3193-3203.
26.asmarf,hotherc,kulosmang,treppendahlmb,nielsenhm,ralfkiaeru,pedersena,mollermb,ralfkiaere,denullybrownpetal:diffuselargeb-celllymphomawithcombinedtp53mutationandmir34amethylation:another″doublehit″lymphomawithverypooroutcame?oncotarget2014,5(7):1912-1925.
27.niskakoskia,kaurs,staffs,renkonen-sinisalol,lassush,jarvinenhj,mecklinjp,butzowr,peltomakip:epigeneticanalysisofsporadicandlynch-associatedovariancancersrevealshistology-specificpatternsofdnamethylation.epigenetics:officialjournalofthednamethylationsociety2014,9(12):1577-1587.
28.shrestharl,tamuran,friesa,levinn,clarkj,draviamvm:tao1kinasemaintainschromosomalstabilitybyfacilitatingpropercongressionofchromosomes.openbiol2014,4(6):130108.
29.ramahm,earnests,zhangk,zhaoy,cobbmh:taokinasesmediateactivationofp38inresponsetodnadamage.theembojournal2007,26(8):2005-2014.
30.draviamvm,stegmeierf,nalepag,sowame,chenj,lianga,hannongj,sorgerpk,harperjw,elledgesj:afunctionalgenomicscreenidentifiesarolefortao1kinaseinspindle-checkpointsignalling.naturecellbiology2007,9(5):556-564.
31.hubnernc,wanglh,kaulichm,descombesp,poseri,niggea:re-examinationofsirnaspecificityquestionsroleofpichandtao1inthespindlecheckpointandidentifiesmad2asasensitivetargetforsmallrnas.chromosoma2010,119(2):149-165.
32.westhorpefg,diezma,gurdenmd,tighea,taylorss:re-evaluatingtheroleoftao1inthespindlecheckpoint.chromosoma2010,119(4):371-379.
33.guowj,zhangym,zhangl,huangb,taoff,chenw,guozj,xuq,suny:novelmonofunctionalplatinum(ii)complexmono-ptinducesapoptosis-independentautophagiccelldeathinhumanovariancarcinomacells,distinctfromcisplatin.autophagy2013,9(7):996-1008.
34.smithdm,patels,raffoulf,hallere,millsgb,nanjundanm:arsenictrioxideinducesabeclin-1-independentautophagicpathwayviamodulationofsnon/skilexpressioninovariancarcinomacells.celldeathanddifierentiation2010,17(12):1867-1881.
35.caoj,caij,huangd,hanq,yangq,lit,dingh,wangz:mir-335representsaninvasionsuppressorgeneinovariancancerbytargetingbcl-w.oncologyreports2013,30(2):701-706.
36.caoj,caij,huangd,hanq,cheny,yangq,yangc,kuangy,lid,wangz:mir-335representsanindependentprognosticmarkerinepithelialovariancancer.americanjournalofclinicalpathology2014,141(3):437-442.
37.taylorsa,marrinanch,liug,nalel,bishopwr,kirschmeierp,lium,longbj:combiningthefarnesyltransferaseinhibitorlonafarnibwithpaclitaxelresultsinenhancedgrowthinhibitoryeffectsonhumanovariancancermodelsinvitroandinvivo.gynecologiconcology2008,109(1):97-106.
38.yangy,liul,caij,wuj,guanh,zhux,yuanj,chens,lim:targetingsmad2andsmad3bymir-136suppressesmetastasis-associatedtraitsoflungadenocarcinomacells.oncologyresearch2013,21(6):345-352.
39.shens,yueh,liy,qinj,lik,liuy,wangj:upregulationofmir-136inhumannon-smallcelllungcancercellspromoteserk1/2activationbytargetingppp2r2a.tumourbiology:thejournaloftheinternationalsocietyforoncodevelopmentalbiologyandmedicine2014,35(1):631-640.
40.leedy,jeyapalanz,fangl,yangj,zhangy,yeeay,lim,duww,shatsevat,yangbb:expressionofversican3′-untranslatedregionmodulatesendogenousmicrornafunctions.plosone2010,5(10):e13599.
41.qiuj,yel,dingj,fengw,zhangy,lvt,wangj,huak:effectsofoestrogenonlongnoncodingrnaexpressioninoestrogenreceptoralpha-positiveovariancancercells.thejournalofsteroidbiochemistryandmolecularbiology2014,141:60-70.
42.januchowskir,zawieruchap,rucinskim,nowickim,zabelm:extracellularmatrixproteinsexpressionprofilinginchemoresistantvariantsofthea2780ovariancancercellline.biomedresearchinternational2014.2014:365867.
43.yewkh,crowj,hirstj,pessettoz,godwinak:epimorphin-inducedmetsensitizesovariancancercellstoplatinum.plosone2013,8(9):e72637.
上面是对本发明进行的说明,不能将其看成是对本发明进行的限制。除非另外指出,本发明的实践将使用有机化学、聚合物化学、生物技术等的常规技术,显然除在上述说明和实施例中所特别描述之外,还可以别的方式实现本发明。其它在本发明范围内的方面与改进将对本发明所属领域的技术人员显而易见。根据本发明的教导,许多改变和变化是可行的,因此其在本发明的范围之内。
如无特别表示,本文中出现的温度的单位“度”是指摄氏度,即℃。
- 还没有人留言评论。精彩留言会获得点赞!