一种腺病毒载体及其构建方法与流程
本发明属于生物技术领域,涉及一种腺病毒载体及其制备方法。
背景技术
腺病毒是一种无包膜的线性双链dna病毒,广泛存在于大自然中,其基因组全长约36kb。腺病毒对大多数细胞有几乎100%的感染效率,包括分裂和不分裂细胞及原代细胞,除了卵细胞以外几乎在所有已知细胞中都不整合到染色体中,因此不会干扰其它的宿主基因。由于腺病毒基因组不整合到宿主的基因组,所以腺病毒在宿主体内是瞬时表达。腺病毒可以直接感染动物个体,通常用于疫苗和基因治疗。由于以上种种优点,腺病毒载体是一种应用较为广泛的病毒载体系统,其中使用最广泛的是5型血清型腺病毒载体。为了提高腺病毒载体的生物安全性,人们对腺病毒基因组进行改造,构建出缺失了腺病毒的早期表达基因序列e1和e3区的重组腺病毒载体。e1是腺病毒复制所必须的,e1的缺失使其不能自身复制,只能依靠包装细胞如293a细胞提供的反式互补进行复制扩增,而不能进行自主复制,e3基因表达的蛋白能对抗宿主的抗病毒防御系统,e3区的去除能减少宿主的体液免疫反应。
gateway技术是基于λ噬菌体位点专一重组系统的一种通用的克隆技术,能够快速地高特异性地将异源dna片断克隆到不同的载体中。其特点是通过bp反应和lr反应最终把目的基因克隆到表达载体上。在gateway技术中起到重组作用的是重组位点:attb,attp,attl和attr。在bpclonasetmiienzymemix催化的重组反应中,attb位点通常与attp位点发生重组。bp重组反应(bprecombination)是指含有attp位点的供体载体(donorvector)与含有attb位点的pcr产物或者其它克隆之间的att位点序列交换反应。供体载体携带卡那霉素抗性基因、氯霉素抗性基因和一个ccdb自杀基因。氯霉素抗性基因和ccdb自杀基因位于两个attp位点之间。由于ccdb自杀基因只能存在于ccdbt1surviralcells、ccdbt2surviralcells或者db3.1中,而对top10、stbl3、dh5a这些细胞具有致死作用。若要复制供体载体,必须把供体载体转化到ccdbt1surviralcells、ccdbt2surviralcells或者db3.1里才能随着宿主分裂而复制。bp重组反应的结果是attb-pcr产物中的目的序列交换到含有attp位点的供体载体内,而供体载体内的ccdb自杀基因和氯霉素抗性基因交换出去。bp重组反应产物被转化到非ccdbsurviralcells内,如top10,再用含有卡那霉素的lb平板筛选阳性克隆。基于卡那霉素和自杀基因的双重筛选,90%的克隆都是正确的克隆。attb和attp位点重组后,序列发生交换,导致重组后的含有目的序列的供体载体上的attp位点序列产生了变化,attp位点变成成attl位点。一般将重组后的供体载体称为入门克隆(entryclone)。
lr重组反应(lrrecombination)是指含有attl位点的入门克隆和含有attr位点的目的载体(destinationvector)之间的att位点序列交换反应。目的载体携带氨苄西林抗性基因、氯霉素抗性基因和ccdb基因。氯霉素抗性基因和ccdb自杀基因位于attr位点之间。由于ccdb自杀基因只能存在于ccdbt1surviralcells、ccdbt2surviralcells或者db3.1中,而对top10、stbl3、dh5a这些细胞具有致死作用。若要复制目的载体,必须把目的载体转化到ccdbt1surviralcells或者ccdbt2surviralcells里才能随着宿主分裂而复制。lr重组反应的结果是入门克隆attl位点间的目的序列交换到含有attr位点的目的载体内,而ccdb自杀基因和氯霉素抗性基因被交换出去。lr反应产物被转化到非ccdbsurviralcells内,如stbl3,再用含有氨苄西林的lb平板筛选阳性克隆。基于氨苄西林和自杀基因的双重筛选,90%的克隆都是正确的克隆。attl和attr位点重组后,序列发生交换,导致重组后的含有目的序列的目的载体上的attr位点序列产生了变化,重组成attb位点序列。一般将这样的目的载体称为表达克隆或者表达载体(expressionvector)。gateway系统种类有以下三种:
1)单片段重组系统(afragmentrecombinationsystem)
在单片段重组系统中,一段末端含有attb1和attb2的pcr产物和含有attp1和attp2的供体载体pdonr221发生bp反应,产生含有目的序列的入门克隆pdown-xxx。这个入门克隆接着与含有attr1和attr2的目的载体发生lr反应,产生含有一段目的序列的表达载体。
2)双片段重组系统(twofragmentsrecombinationsystem)
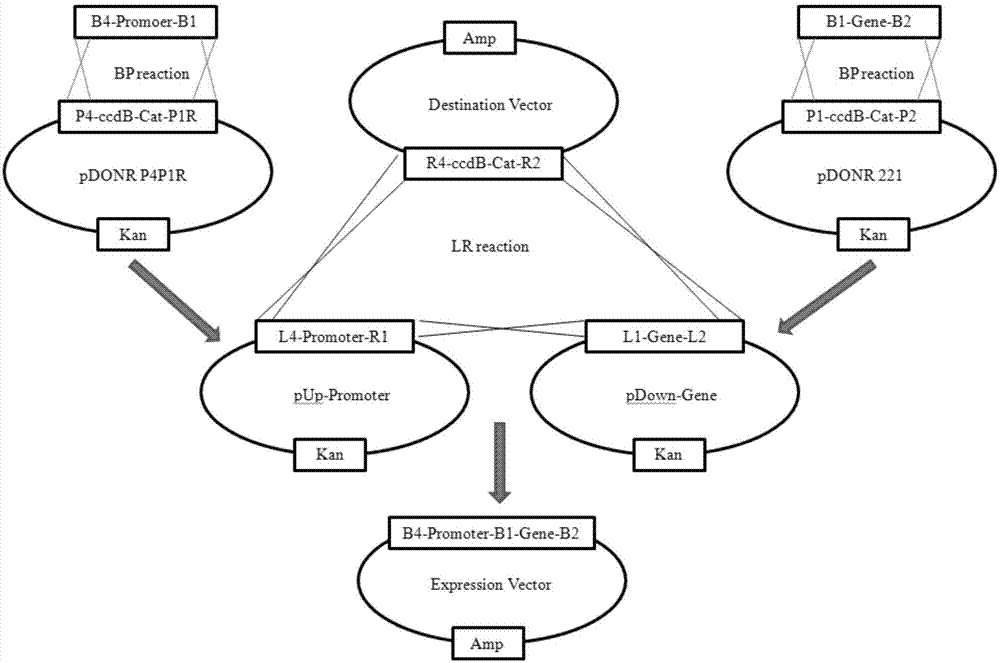
在双片段重组系统(如图1)中,一段末端含有attb4和attb1r的pcr产物和含有attp4和attp1r的供体载体pdonrp4p1r发生bp反应,产生含有目的序列的入门克隆pup-xxx。另一段末端含有attb1和attb2的pcr产物和含有attp1和attp2的供体载体pdonr221发生bp反应产生pdown-xxx。这两个入门克隆接着与含有attr4和attr2的目的载体发生lr反应,产生含有两段目的序列的表达载体。
3)三片段重组系统
在三片段重组系统中,一段末端含有attb4和attb1r的pcr产物和含有attp4和attp1r的供体载体pdonrp4p1r发生bp反应产生pup-xxx,另一段末端含有attb1和attb2的pcr产物和含有attp1和attp2的供体载体pdonr221发生bp反应pdown-xxx,第三段末端含有attb2r和attb3的pcr产物和含有attp2r和attp3的供体载体pdonrp2rp3发生bp反应产生ptail-xxx。这三个入门克隆接着与含有attr4和attr3的目的载体发生lr反应,产生含有三段目的序列的表达载体。
red/et同源重组技术是一种基于rac噬菌体rece/rect或λ噬菌体redα/redβ/redγ的大肠杆菌体内dna同源重组系统,通过同源臂,在同源重组酶的作用下,可以将的目的片段同源重组到选定区域,从而产生研究者想要的特定位点缺失、插入、点突变等。pred/et是一种温度敏感型的同源重组酶表达载体,包含一个l-阿拉伯糖诱导的pbad启动子,整个同源重组过程主要由三种蛋白来协同完成。其中来源于λ噬菌体的redα蛋白具有5’-3’核酸外切酶活性;redβ是一种退火蛋白,这两种蛋白协同作用催化整个同源重组;另外来源于λ噬菌体的redγ蛋白会与大肠杆菌内源性的recbcd核酸外切酶结合。
重组腺病毒载体作为一种比较大(~36kb)的常规载体,利用常规的酶切连接方法在操作层面上显得很吃力,面临一个问题是大片段和小片段切开以后,大片段很难回收,市面上的胶回收试剂盒回收片段都是10kb以下,因此很难对骨架载体进行有效的回收。大部分文献涉及对腺病毒骨架的改造都是基于同源重组的方法,如其中一篇文献利用大肠杆菌bj5183recbcsbcbc作为重组的宿主菌。
现有的重组腺病毒载体是基于gateway克隆技术的一种灵活高效的克隆载体pav.des1d(invitrogen公司商品),其载体图谱如图2所示。该载体保留了野生型腺病毒载体两端的itr区域及绝大部分基因组,缺失了e1和e3区,还有一个gateway克隆位点,其结构为attr1-cm-ccdb-attr2,如图2所示,由于ccdb自杀基因只能存在于ccdbt1surviralcells、ccdbt2surviralcells或者db3.1中,所以该载体需要转化到其中的一种宿主菌中进行复制。根据需求,可先将目的序列通过bp反应克隆到pdonr载体中形成入门克隆,然后再进行lr反应克隆到腺病毒载体pav.des1d上。但是该载体属于gateway载体系统中的单片段重组系统,如果是多片段克隆的话,则需要把多片段融合起来克隆到pdonr载体中才能进行后续的工作。多片段融合pcr往往容易出现融合不成功的情况,不仅增加了耗材成本和时间成本,结果也不能保证,这是不利于高效的生产的。
简而言之,现有腺病毒载体及其构建方法存在以下缺点:第一,现有腺病毒载体包含了attr1-attr2重组位点,所以一次lr反应只能克隆一个来自于入门克隆的外源目的片段,如果需要同时克隆多个外源片段,则需要用融合pcr的方法将这些片段融合成一个片段,再进行后续的构建,这不但提高了构建的难度,也增加了时间和人力成本,不利于提高生产效率。第二,骨架酶切后,很难用市面上现有的胶回收试剂盒进行切胶回收,即使回收,也不能保证是否会对骨架片段造成机械损伤,导致片段丢失。第三,传统的red/et同源重组技术出现的非预期的分子内重排、异位的概率很高,出现假阳性的概率高,真正的阳性率低。
技术实现要素:
为了解决上述存在的技术问题,本申请提供了一种重组腺病毒载体及其构建方法。使得所得腺病毒载体只要进行一步lr反应,即可将多个片段同时快速灵活高效地克隆到腺病毒载体中,大大缩短了服务周期和节约了时间耗材成本,提高了综合竞争力。并且可将阳性率从原来的1%提高到了约50%。
本发明的目的在于提供了一种重组腺病毒载体及其构建方法。
本发明所采取的技术方案是:
一种腺病毒载体的构建方法,包括以下步骤:
1)将pred/et载体中的redα基因删除,得pred/et(δα)载体;
2)将pred/et(δα)载体转入含pav.des1d质粒的db3.1感受态细胞,复苏培养;
3)将复苏培养后的产物涂布到同时含有氨苄抗生素amp、氯霉素cm和四环素tc的培养基平板上,避光培养,挑取阳性克隆菌a;
4)将上步所得阳性克隆菌a加入含l-阿拉伯糖的培养基中诱导表达同源重组酶并制备成感受态细胞,将带同源臂的kan抗性表达盒pcr产物转入其中进行重组反应,复苏培养;
5)将步骤4)的复苏培养产物涂布到含amp、kan和tc的培养基平板上,避光培养,挑取重组阳性克隆菌b;
6)将步骤5)中得到的阳性克隆菌b分别在液体培养基扩大培养,其中在含amp、kan和tc的抗性培养基中生长,且在含cm抗性培养基中不能生长的克隆菌即为目的阳性克隆菌c;
7)将步骤6)中得到的目的阳性克隆菌c加入l-阿拉伯糖诱导表达同源重组酶并制备成感受态细胞,将带同源臂的attr4-cm-ccdb-attr2或带同源臂的attr4-cm-ccdb-attr3片断转入其中进行重组反应;复苏培养;
8)将步骤7)中复苏培养产物涂布到含amp、cm和tc的培养基平板上,36.5~37.5℃培养,挑取重组阳性克隆菌d;
9)将步骤8)中得到的阳性克隆菌d分别在液体培养基扩大培养,其中在含amp和cm的抗性培养基中生长,且在含kan和tc抗性培养基中不能生长的克隆菌即为目的阳性克隆菌e;该步骤中所有培养的培养温度为36.5~37.5℃;
提取阳性克隆菌e中的质粒即得腺病毒载体。
进一步的,步骤1)中,pred/et(δα)载体中还对redγ和redβ基因序列进行了优化,优化后的redγ和redβ基因序列分别如seqidno:6、seqidno:7所示。
进一步的,步骤1)~7)中所有培养的培养温度为29.5~30.5℃。
进一步的,所述带同源臂的kan抗性片段的碱基序列如seqidno:1所示。
进一步的,所述带同源臂的attr4-cm-ccdb-attr2的碱基序列如seqidno:2所示。
进一步的,所述带同源臂的attr4-cm-ccdb-attr3的碱基序列如seqidno:3所示。
进一步的,步骤2)中,复苏培养所用的培养基为soc培养基。
上述任一所述方法构建的腺病毒载体。
含有上述所述腺病毒载体的菌体。
一种腺病毒载体pav.des2d,其与现有pav.des1d载体相比,区别点在于,pav.des2d用attr4取代了pav.des1d中的attr1序列,同时pav.des2d在attr2下游连有终止子序列。
一种腺病毒载体pav.des3d,其与现有pav.des1d载体相比,区别点在于,pav.des3d用attr4取代了pav.des1d中的attr1序列,同时用attr3取代了pav.des1d中的attr2序列,并且pav.des3d在attr3下游连有终止子序列。
本发明的有益效果是:
(1)本发明方法不需要经过重新合成,可将多片段gateway重组位点与腺病毒载体组合,构建获得pav.des2d和pav.des3d,阻止pav.des1d的商业垄断。且本发明中在db3.1感受态(抗ccdb自杀基因)中进行同源重组反应。
(2)本发明获得的pav.des2d和pav.des3d腺病毒载体,实现了在腺病毒载体上同时克隆多片段。
(3)本发明用新的pred/et(δα)介导的重组反应,虽然平板上的菌落数量减少了,但是其阳性率从原来的1%提高到了约50%,阳性克隆的正确率接近100%,大大降低了假阳性克隆的产生。这也解决了我们一直以来饱受第二步重组阳性率(实施例1步骤7中的重组反应)极低的困扰,提高了工作效率。在实际工作中,我们将比较受欢迎的启动子、目的基因及示踪基因等分别构建到相应的pdonr载体中形成对应的入门克隆,然后根据客户的不同需要进行灵活自由搭配,只要进行一步lr反应,即可将多个片段同时快速灵活高效地克隆到腺病毒载体中,大大缩短了服务周期和节约了时间耗材成本,提高了本公司的综合竞争力。
(4)本申请人已有的重组腺病毒载体是基于gateway重组系统的一种高效克隆载体,但是由于该载体包含了attr1-attr2重组位点,所以一次lr反应只能克隆一个来自于入门克隆的外源目的片段,如果客户需要同时克隆多个外源片段,则需要用融合pcr的方法将这些片段融合成一个片段,再进行后续的构建,这不但提高了构建的难度,也增加了时间和人力成本,不利于我们提高生产效率。为了提高该载体的克隆适用性,灵活性,在此基础上,我们通过特定的方法对现有的重组腺病毒载体进行改造,用attr4-attr2和attr4-attr3分别取代attr1-attr2,构建出可同时克隆2个和3个目的片段的重组腺病毒克隆载体pav.des2d和pav.des3d,从而使得重组腺病毒表达载体的构建变得高效灵活。
附图说明
图1双片段重组系统;
图2为pav.des1d载体图谱;
图3为pred/et(δα)载体图谱;
图4为pav.des2d载体图谱;
图5为pav.des3d载体图谱;
图6为本发明腺病毒载体构建方法的流程示意图;
图7为将阳性表达载体pav[exp]-cmv>egfp转染293a细胞,7天后发现明显病变斑,成功包装腺病毒;左图:白光视野;右图:绿色荧光视野;
图8为阳性表达载体pav[exp]-cmv>egfp:ires:mcherry转染293a细胞,7天后发现明显病变斑,成功包装腺病毒;左图:白光视野;中图:绿色荧光视野;右图:红色荧光视野。
具体实施方式
下面结合具体实施例对本发明作进一步的说明。
实施例1一种腺病毒载体的构建方法
1.将pred/et载体(购于生物公司)中的redα基因删除,优化redβ和redγ基因序列,获得优化改造的pred/et(δα)载体,使重组过程中产生的非特异性大片段分子内重组率显著降低,从而达到提高特异性重组效率的目的。优化改造的pred/et(δα)的结构示意图如图3所示,pred/et(δα)载体中的核心序列如seqidno:4所示,其中包括pbad启动子序列(seqidno:5)、优化后的redγ基因序列(seqidno:6)、优化后的redβ基因序列(seqidno:7)。将优化的pred/et(δα)电转到包含pav.des1d的db3.1感受态细胞中,加入soc培养基于30℃复苏培养1小时。
2.复苏培养产物涂布到同时含有氨苄抗生素(amp)、氯霉素(cm)和四环素(tc)的lb板上,30℃避光(四环素见光分解)培养两天,挑克隆pcr鉴定出含有两种质粒的阳性克隆。
3.挑取阳性克隆分别接种到含amp+cm+tc抗性的lb液体培养基中30℃过夜培养。
4.将步骤3中得到的阳性克隆菌加入l-阿拉伯糖诱导表达同源重组酶并制备成感受态细胞,将带同源臂的kan抗性片段(碱基序列如seqidno:1所示)pcr产物电转入其中进行重组反应。
5.复苏培养产物涂布到amp+kan+tc的lb板上,30℃避光(四环素见光分解)培养两天,挑克隆pcr鉴定出重组阳性克隆。
6.将步骤5中得到的阳性克隆分别接种到液体lb培养基中于30℃过夜培养,其中在amp+kan+tc抗性培养基中生长,而在cm抗性培养基中不能生长的克隆即为目的阳性克隆
7.将步骤6中得到的阳性克隆菌加入l-阿拉伯糖诱导表达同源重组酶并制备成感受态细胞,将带同源臂的attr4-cm-ccdb-attr2(seqidno:2)或attr4-cm-ccdb-attr3(seqidno:3)片段电转入其中进行重组反应。
8.复苏培养产物涂布到amp+cm+tc的lb板上,37℃培养一天,挑克隆pcr鉴定出重组阳性克隆。
9.将步骤8中得到的阳性克隆分别接种到液体lb培养基中于37℃过夜培养,其中在amp+cm抗性培养基中生长,而在kan和tc抗性培养基中不能生长的克隆即为纯的阳性克隆菌;提取阳性克隆菌的质粒即得腺病毒载体。当步骤7中转化用片段为带同源臂的attr4-cm-ccdb-attr2片段时,最终所得的腺病毒载体记为pav.des2d,其载体示意图如图4所示,图中各元件符号名称对应的中文名称如表1所示;当步骤7中转化用片段为带同源臂的attr4-cm-ccdb-attr3片段时,最终所得的腺病毒载体记为pav.des3d,其载体示意图如图5所示,图中各元件符号名称对应的中文名称如表1所示。
进一步对本实施例获得pav.des2d和pav.des3d的阳性率进行检测,结果显示,pav.des2d和pav.des3d的阳性率分别为54%、50%,验证后阳性克隆的正确率为100%(阳性率是指在一个板上的菌有的是错的,有的是对的,通过菌落pcr来扩增看是否有目的带,有目的带则认为是阳性,从而计算出阳性率;而阳性克隆正确率是指阳性克隆经过了进一步测序,序列是完全正确的数量占总的阳性克隆数的比率)。而运用现有未改造的pred/et载体(含redα基因,redβ和redγ基因序列未优化)进行本发明pav.des2d和pav.des3d的构建时,最终获得pav.des2d和pav.des3d的阳性率仅分别为1%、1.2%。可见本发明方法显著提升了特异性的重组率,重组阳性克隆正确率显著提高。
表1图4的pav.des2d和图5的pav.des3d中相应元件符号名称和中文名称
本实施例构建方法的操作步骤示意图如图6所示。
下面对本发明构建的pav.des2d和pav.des3d腺病毒载体作进一步的效果检测。
一、对pav.des2d腺病毒载体的效果检测
方法:将cmv启动子基因序列和egfp绿色荧光蛋白基因序列分别进行bp反应,得入门克隆pup-cmv和pdown-egfp。再将pup-cmv、pdown-egfp与本发明制备骨架载体pav.des2d进行一步lr反应,即可得到表达载体pav[exp]-cmv>egfp,将该表达载体转化感受态细胞,检测阳性率、阳性克隆的正确率、病毒滴度等指标。
结果:利用本发明pav.des2d仅通过一步lr反应,即可获得含2个目的片段的阳性表达载体pav[exp]-cmv>egfp,且经pcr鉴定后,所得阳性率正确率为100%。将阳性表达载体pav[exp]-cmv>egfp转染293a细胞,7天后发现明显病变斑,成功包装腺病毒,可看到非常强的绿色荧光表达(图7),且qpcr检测病毒滴度最高可达到1012pfu/ml。
二、对pav.des3d腺病毒载体的效果检测
方法:将ires-mcherry红色荧光蛋白基因序列进行bp反应,得入门克隆ptail-ires-mcherry。将上述“一”中pup-cmv、pdown-egfp和ptail-ires-mcherry与本发明制备骨架载体pav.des3d进行一步lr反应,即可得到表达载体pav[exp]-cmv>egfp:ires:mcherry,将该表达载体转化感受态细胞,检测阳性率、阳性克隆的正确率、病毒滴度等指标。
结果:利用本发明pav.des3d仅通过一步lr反应,即可获得含3个目的片段的阳性表达载体pav[exp]-cmv>egfp:ires:mcherry,且经pcr鉴定后,所得阳性率正确率为100%。将阳性表达载体pav[exp]-cmv>egfp:ires:mcherry转染293a细胞,7天后发现明显病变斑,成功包装腺病毒,可看到非常强的绿色和红色荧光表达(图8),且qpcr检测病毒滴度最高可达到1012pfu/ml。
上述结果说明,本发明成功实现了对pav.des1d载体的改造,构建出的pav.des2d和pav.des3d腺病毒载体可以分别同时克隆2个或3个目片段,将常用的一些片段进行bp反应构建成对应的入门克隆,然后根据需求进行自由搭配,只需要进行一步lr反应即可得到所需要的腺病毒表达载体,且具有很好的准确性、高效性,成功率高。大大降低了人力时间成本,提高了用户体验。本发明构建的重组腺病毒载体可根据需要任意引入目的双片段或三片段,更加灵活快速高效地克隆目的片段,提高生产效率。
另外,在本研究中发现,传统的red/et同源重组技术,会大大增加第二步重组(上述实施例1步骤7中的重组反应)中出现的非预期的分子内重排、异位的概率,出现假阳性背景,使阳性率降低。我们综合实验中出现的异常情况,删除了red/et载体上的redα基因,优化了redβ和redγ基因序列,构建成新的载体pred/et(δα)。在对比重组实验中发现,与原来利用pred/et载体相比,利用新的pred/et(δα)介导的重组反应,虽然平板上的菌落数量减少了,但是其阳性率从原来的1%提高到了约50%,阳性克隆的正确率接近100%,大大降低了假阳性克隆的产生。这也解决了我们一直以来饱受第二步重组阳性率(上述实施例1步骤7中的重组反应)极低的困扰,提高了工作效率。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
sequencelisting
<110>云舟生物科技(广州)有限公司
<120>一种腺病毒载体及其构建方法
<130>
<160>7
<170>patentinversion3.5
<210>1
<211>1395
<212>dna
<213>人工序列
<400>1
cttggatccggtacctctagaattctcgagcggccgctagcgacatcgatcaagcttgct60
acgtacgtcgacagcttgatatcgaattccgaagttcctattctctagaaagtataggaa120
cttcaggtctgaagaggagtttacgtccagccaagctagcctggcccgtgtctcaaaatc180
tctgatgttacattgcacaagataaaataatatcatcatgaacaataaaactgtctgctt240
acataaacagtaatacaaggggtgttatgagccatattcaacgggaaacgtcgaggccgc300
gattaaattccaacatggatgctgatttatatgggtataaatgggctcgcgataatgtcg360
ggcaatcaggtgcgacaatctatcgcttgtatgggaagcccgatgcgccagagttgtttc420
tgaaacatggcaaaggtagcgttgccaatgatgttacagatgagatggtcagactaaact480
ggctgacggaatttatgcctcttccgaccatcaagcattttatccgtactcctgatgatg540
catggttactcaccactgcgatccccggaaaaacagcattccaggtattagaagaatatc600
ctgattcaggtgaaaatattgttgatgcgctggcagtgttcctgcgccggttgcattcga660
ttcctgtttgtaattgtccttttaacagcgatcgcgtatttcgtctcgctcaggcgcaat720
cacgaatgaataacggtttggttgatgcgagtgattttgatgacgagcgtaatggctggc780
ctgttgaacaagtctggaaagaaatgcataaacttttgccattctcaccggattcagtcg840
tcactcatggtgatttctcacttgataaccttatttttgacgaggggaaattaataggtt900
gtattgatgttggacgagtcggaatcgcagaccgataccaggatcttgccatcctatgga960
actgcctcggtgagttttctccttcattacagaaacggctttttcaaaaatatggtattg1020
ataatcctgatatgaataaattgcagtttcatttgatgctcgatgagtttttctaatcag1080
aattggttaattggttgtaacactggcagagcattacgctgacttgacgggacggcgcaa1140
gctcatgaccaaaatcccttaacgtgagttacgcgtcgttccactgagcgtcagaccccg1200
tagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgctgcttgc1260
gctagctagagcttgcggaacccttcgaagttcctattctctagaaagtataggaacttc1320
atcagtcaggtacataacctcgagggatcgattcgacagatcactgaaatgtgtgggcgt1380
ggcttaagggtggga1395
<210>2
<211>1817
<212>dna
<213>人工序列
<400>2
cttggatccggtacctctagaattctcgagcggccgctagcgacatcgatcaactttgta60
tagaaaagttgaacgagaaacgtaaaatgatataaatatcaatatattaaattagatttt120
gcataaaaaacagactacataatactgtaaaacacaacatatccagtcactatggcggcc180
gcattaggcaccccaggctttacactttatgcttccggctcgtataatgtgtggattttg240
agttaggatccgtcgagattttcaggagctaaggaagctaaaatggagaaaaaaatcact300
ggatataccaccgttgatatatcccaatggcatcgtaaagaacattttgaggcatttcag360
tcagttgctcaatgtacctataaccagaccgttcagctggatattacggcctttttaaag420
accgtaaagaaaaataagcacaagttttatccggcctttattcacattcttgcccgcctg480
atgaatgctcatccggaactccgtatggcaatgaaagacggtgagctggtgatatgggat540
agtgttcacccttgttacaccgttttccatgagcaaactgaaacgttttcatcgctctgg600
agtgaataccacgacgatttccggcagtttctacacatatattcgcaagatgtggcgtgt660
tacggtgaaaacctggcctatttccctaaagggtttattgagaatatgtttttcgtctca720
gccaatccctgggtgagtttcaccagttttgatttaaacgtggccaatatggacaacttc780
ttcgcccccgttttcaccatgggcaaatattatacgcaaggcgacaaggtgctgatgccg840
ctggcgattcaggttcatcatgccgtttgtgatggcttccatgtcggcagaatgcttaat900
gaattacaacagtactgcgatgagtggcagggcggggcgtaaacgcgtggatccggctta960
ctaaaagccagataacagtatgcgtatttgcgcgctgatttttgcggtataagaatatat1020
actgatatgtatacccgaagtatgtcaaaaagaggtatgctatgaagcagcgtattacag1080
tgacagttgacagcgacagctatcagttgctcaaggcatatatgatgtcaatatctccgg1140
tctggtaagcacaaccatgcagaatgaagcccgtcgtctgcgtgccgaacgctggaaagc1200
ggaaaatcaggaagggatggctgaggtcgcccggtttattgaaatgaacggctcttttgc1260
tgacgagaacaggggctggtgaaatgcagtttaaggtttacacctataaaagagagagcc1320
gttatcgtctgtttgtggatgtacagagtgatattattgacacgcccgggcgacggatgg1380
tgatccccctggccagtgcacgtctgctgtcagataaagtctcccgtgaactttacccgg1440
tggtgcatatcggggatgaaagctggcgcatgatgaccaccgatatggccagtgtgccgg1500
tctccgttatcggggaagaagtggctgatctcagccaccgcgaaaatgacatcaaaaacg1560
ccattaacctgatgttctggggaatataaatgtcaggctcccttatacacagccagtctg1620
caggtcgagcagatctgctcgaccatagtgactggatatgttgtgttttacagtattatg1680
tagtctgttttttatgcaaaatctaatttaatatattgatatttatatcattttacgttt1740
ctcgttcagctttcttgtacaaagtgggatcgattcgacagatcactgaaatgtgtgggc1800
gtggcttaagggtggga1817
<210>3
<211>1802
<212>dna
<213>人工序列
<400>3
cttggatccggtacctctagaattctcgagcggccgctagcgacatcgatcaactttgta60
tagaaaagttgaacgagaaacgtaaaatgatataaatatcaatatattaaattagatttt120
gcataaaaaacagactacataatactgtaaaacacaacatatccagtcactatggtcgac180
ctgcagactggctgtgtataagggagcctgacatttatattccccagaacatcaggttaa240
tggcgtttttgatgtcattttcgcggtggctgagatcagccacttcttccccgataacgg300
agaccggcacactggccatatcggtggtcatcatgcgccagctttcatccccgatatgca360
ccaccgggtaaagttcacgggggactttatctgacagcagacgtgcactggccaggggga420
tcaccatccgtcgcccgggcgtgtcaataatatcactctgtacatccacaaacagacgat480
aacggctctctcttttataggtgtaaaccttaaactgcatttcaccagcccctgttctcg540
tcggcaaaagagccgttcatttcaataaaccgggcgacctcagctatcccttcctgattt600
tccgctttccagcgttcggcacgcagacgacgggcttcattctgcatggttgtgcttacc660
gaaccggagatattgacatcatatatgccttgagcaactgatagctgtcgctgtcaactg720
tcactgtaatacgctgcttcatagcatacctctttttgacatacttcgggtatacatatc780
agtatatattcttataccgcaaaaatcagcgcgcaaatacgcatactgttatctggcttt840
tagtaagccggatcctctagattacgccccgcctgccactcatcgcagtactgttgtaat900
tcattaagcattctgccgacatggaagccatcacaaacggcatgatgaacctgaatcgcc960
agcggcatcagcaccttgtcgccttgcgtataatatttgcccatggtgaaaacgggggcg1020
aagaagttgtccatattggccacgtttaaatcaaaactggtgaaactcacccagggattg1080
gctgagacgaaaaacatattctcaataaaccctttagggaaataggccaggttttcaccg1140
taacacgccacatcttgcgaatatatgtgtagaaactgccggaaatcgtcgtggtattca1200
ctccagagcgatgaaaacgtttcagtttgctcatggaaaacggtgtaacaagggtgaaca1260
ctatcccatatcaccagcccaccgtctttcattgccatacggaattccggatgagcattc1320
atcaggcgggcaagaatgtgaataaaggccggataaaacttgtgcttatttttctttacg1380
gtctttaaaaaggccgtaatatccagctgaacggtctggttataggtacattgagcaact1440
gactgaaatgcctcaaaatgttctttacgatgccattgggatatatcaacggtggtatat1500
ccagtgatttttttctccattttagcttccttagctcctgaaaatctcgacggatcctaa1560
ctcaaaatccacacattatacgagccggaagcataaagtgtaaagcctggggtgcctaat1620
gcggccgccatagtgactggatatgttgtgttttacagtattatgtagtctgttttttat1680
gcaaaatctaatttaatatattgatatttatatcattttacgtttctcgttcaactttat1740
tatacatagttggatcgattcgacagatcactgaaatgtgtgggcgtggcttaagggtgg1800
ga1802
<210>4
<211>1524
<212>dna
<213>人工序列
<400>4
aaaccaattgtccatattgcatcagacattgccgtcactgcgtcttttactggctcttct60
cgctaaccaaaccggtaaccccgcttattaaaagcattctgtaacaaagcgggaccaaag120
ccatgacaaaaacgcgtaacaaaagtgtctataatcacggcagaaaagtccacattgatt180
atttgcacggcgtcacactttgctatgccatagcatttttatccataagattagcggatc240
ctacctgacgctttttatcgcaactctctactgtttctccatacccgtttttttgggcta300
gcaggaggaattcaccatggatattaacaccgaaaccgaaattaaacagaaacatagcct360
gaccccgtttccggtgtttctgattagcccggcgtttcgcggccgctattttcatagcta420
ttttcgcagcagcgcgatgaacgcgtattatattcaggatcgcctggaagcgcagagctg480
ggcgcgccattatcagcagctggcgcgcgaagaaaaagaagcggaactggcggatgatat540
ggaaaaaggcctgccgcagcatctgtttgaaagcctgtgcattgatcatctgcagcgcca600
tggcgcgagcaaaaaaagcattacccgcgcgtttgatgatgatgtggaatttcaggaacg660
catggcggaacatattcgctatatggtggaaaccattgcgcatcatcaggtggatattga720
tagcgaagtgtaaaacgaatgagcaccgcgctggcgaccctggcgggcaaactggcggaa780
cgcgtgggcatggatagcgtggatccgcaggaactgattaccaccctgcgccagaccgcg840
tttaaaggcgatgcgagcgatgcgcagtttattgcgctgctgattgtggcgaaccagtat900
ggcctgaacccgtggaccaaagaaatttatgcgtttccggataaacagaacggcattgtg960
ccggtggtgggcgtggatggctggagccgcattattaacgaaaaccagcagtttgatggc1020
atggattttgaacaggataacgaaagctgcacctgccgcatttatcgcaaagatcgcaac1080
catccgatttgcgtgaccgaatggatggatgaatgccgccgcgaaccgtttaaaacccgc1140
gaaggccgcgaaattaccggcccgtggcagagccatccgaaacgcatgctgcgccataaa1200
gcgatgattcagtgcgcgcgcctggcgtttggctttgcgggcatttatgataaagatgaa1260
gcggaacgcattgtggaaaacaccgcgtataccgcggaacgccagccggaacgcgatatt1320
accccggtgaacgatgaaaccatgcaggaaattaacaccctgctgattgcgctggataaa1380
acctgggatgatgatctgctgccgctgtgcagccagatttttcgccgcgatattcgcgcg1440
agcagcgaactgacccaggcggaagcggtgaaagcgctgggctttctgaaacagaaagcg1500
gcggaacagaaagtggcggcgtaa1524
<210>5
<211>273
<212>dna
<213>人工序列
<400>5
aaaccaattgtccatattgcatcagacattgccgtcactgcgtcttttactggctcttct60
cgctaaccaaaccggtaaccccgcttattaaaagcattctgtaacaaagcgggaccaaag120
ccatgacaaaaacgcgtaacaaaagtgtctataatcacggcagaaaagtccacattgatt180
atttgcacggcgtcacactttgctatgccatagcatttttatccataagattagcggatc240
ctacctgacgctttttatcgcaactctctactg273
<210>6
<211>417
<212>dna
<213>人工序列
<400>6
atggatattaacaccgaaaccgaaattaaacagaaacatagcctgaccccgtttccggtg60
tttctgattagcccggcgtttcgcggccgctattttcatagctattttcgcagcagcgcg120
atgaacgcgtattatattcaggatcgcctggaagcgcagagctgggcgcgccattatcag180
cagctggcgcgcgaagaaaaagaagcggaactggcggatgatatggaaaaaggcctgccg240
cagcatctgtttgaaagcctgtgcattgatcatctgcagcgccatggcgcgagcaaaaaa300
agcattacccgcgcgtttgatgatgatgtggaatttcaggaacgcatggcggaacatatt360
cgctatatggtggaaaccattgcgcatcatcaggtggatattgatagcgaagtgtaa417
<210>7
<211>786
<212>dna
<213>人工序列
<400>7
atgagcaccgcgctggcgaccctggcgggcaaactggcggaacgcgtgggcatggatagc60
gtggatccgcaggaactgattaccaccctgcgccagaccgcgtttaaaggcgatgcgagc120
gatgcgcagtttattgcgctgctgattgtggcgaaccagtatggcctgaacccgtggacc180
aaagaaatttatgcgtttccggataaacagaacggcattgtgccggtggtgggcgtggat240
ggctggagccgcattattaacgaaaaccagcagtttgatggcatggattttgaacaggat300
aacgaaagctgcacctgccgcatttatcgcaaagatcgcaaccatccgatttgcgtgacc360
gaatggatggatgaatgccgccgcgaaccgtttaaaacccgcgaaggccgcgaaattacc420
ggcccgtggcagagccatccgaaacgcatgctgcgccataaagcgatgattcagtgcgcg480
cgcctggcgtttggctttgcgggcatttatgataaagatgaagcggaacgcattgtggaa540
aacaccgcgtataccgcggaacgccagccggaacgcgatattaccccggtgaacgatgaa600
accatgcaggaaattaacaccctgctgattgcgctggataaaacctgggatgatgatctg660
ctgccgctgtgcagccagatttttcgccgcgatattcgcgcgagcagcgaactgacccag720
gcggaagcggtgaaagcgctgggctttctgaaacagaaagcggcggaacagaaagtggcg780
gcgtaa786
- 还没有人留言评论。精彩留言会获得点赞!