GsHA12蛋白及其编码基因在调控植物耐逆性中的应用的制作方法
本发明属于生物技术领域,具体涉及gsha12蛋白及其编码基因在调控植物耐逆性中的应用。
背景技术:
土地盐碱化严重威胁我国乃至世界农业系统的正常发展,全世界约20%的可耕种地和50%的灌溉地受盐碱化影响。中国东北地域的松嫩平原是世界三大盐碱地之一,约有盐碱地373.33万hm2,这严重制约我国东北地区农业系统的正常发展,限制粮食产量。然而,随着我国人口的持续增长和可耕种土壤的退化,合理开发利用盐碱地是增加我国粮食产量的重要举措之一,也是实现农业可持续发展的关键因素之一。随着功能基因组学、分子生物学和基因工程技术日新月异的发展,通过转基因分子育种改良作物耐盐碱性,已成为改良和合理开发利用盐碱地的手段之一。然而,其关键在于挖掘功能显著的耐盐碱关键调控基因。
在生命有机体的生长发育过程中,一系列的生理生化反应都需要在特定ph条件下进行,如细胞质ph维持在7-8。质膜h+-atpase在植物和真菌中通过偶联atp的水解对质子进行转运,产生的ph梯度为次级转运器提供驱动力。植物质膜h+-atpase是一类质子泵,在植物营养吸收、细胞内ph调节、气孔开闭和细胞生长等生理过程中是不可或缺的。除此之外,在植物应对外界不利因素也具有重要作用,如盐碱、低温、干旱、重金属毒害、光胁迫、化学胁迫和激素胁迫等非生物胁迫。
技术实现要素:
本发明所要解决的技术问题是如何调控植物耐逆性。
为解决上述技术问题,本发明首先提供了与植物耐逆性相关蛋白质gsha12的新用途。
本发明提供了蛋白质gsha12在调控植物耐逆性中的应用。
本发明所涉及的蛋白质gsha12是如下a)或b)或c)或d)的蛋白质:
a)氨基酸序列是序列2所示的蛋白质;
b)在序列2所示的蛋白质的n端和/或c端连接标签得到的融合蛋白质;
c)将序列2所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加得到的具有相同功能的蛋白质;
d)与序列2所示的氨基酸序列具有75%或75%以上的同源性且具有相同功能的蛋白质。
其中,序列2由888个氨基酸残基组成。
为了解决上述技术问题,本发明还提供了与蛋白质gsha12相关的生物材料的新用途。
本发明提供了与蛋白质gsha12相关的生物材料在调控植物耐逆性中的应用。
所述与蛋白质gsha12相关的生物材料为下述a1)至a12)中的任一种:
a1)编码蛋白质gsha12的核酸分子;
a2)含有a1)所述核酸分子的表达盒;
a3)含有a1)所述核酸分子的重组载体;
a4)含有a2)所述表达盒的重组载体;
a5)含有a1)所述核酸分子的重组微生物;
a6)含有a2)所述表达盒的重组微生物;
a7)含有a3)所述重组载体的重组微生物;
a8)含有a4)所述重组载体的重组微生物;
a9)含有a1)所述核酸分子的转基因植物细胞系;
a10)含有a2)所述表达盒的转基因植物细胞系;
a11)含有a3)所述重组载体的转基因植物细胞系;
a12)含有a4)所述重组载体的转基因植物细胞系。
上述应用中,a1)所述核酸分子为如下1)或2)或3)所示的基因:
1)其编码序列是序列1所示的cdna分子或基因组dna分子;
2)与1)限定的核苷酸序列具有75%或75%以上同一性,且编码蛋白质gsha12的cdna分子或基因组dna分子;
3)在严格条件下与1)或2)限定的核苷酸序列杂交,且编码蛋白质gsha12的cdna分子或基因组dna分子。
其中,所述核酸分子可以是dna,如cdna、基因组dna或重组dna;所述核酸分子也可以是rna,如mrna或hnrna等。
其中,序列1由2667个核苷酸组成,编码序列2所示的氨基酸序列。
本领域普通技术人员可以很容易地采用已知的方法,例如定向进化和点突变的方法,对本发明的编码蛋白质gsha12的核苷酸序列进行突变。那些经过人工修饰的,具有编码蛋白质gsha12的核苷酸序列75%或者更高同一性的核苷酸,只要编码蛋白质gsha12且具有相同功能,均是衍生于本发明的核苷酸序列并且等同于本发明的序列。
这里使用的术语“同一性”指与天然核酸序列的序列相似性。“同一性”包括与本发明的编码序列2所示的氨基酸序列组成的蛋白质的核苷酸序列具有75%或更高,或85%或更高,或90%或更高,或95%或更高同一性的核苷酸序列。同一性可以用肉眼或计算机软件进行评价。使用计算机软件,两个或多个序列之间的同一性可以用百分比(%)表示,其可以用来评价相关序列之间的同一性。
上述75%或75%以上同一性,可为80%、85%、90%或95%以上的同一性。
上述应用中,所述严格条件是在2×ssc,0.1%sds的溶液中,在68℃下杂交并洗膜2次,每次5min,又于0.5×ssc,0.1%sds的溶液中,在68℃下杂交并洗膜2次,每次15min;或,0.1×sspe(或0.1×ssc)、0.1%sds的溶液中,65℃条件下杂交并洗膜。
上述应用中,a2)所述的含有编码蛋白质gsha12的核酸分子的表达盒(gsha12基因表达盒)是指能够在宿主细胞中表达蛋白质gsha12的dna,该dna不但可包括启动gsha12转录的启动子,还可包括终止gsha12转录的终止子。进一步,所述表达盒还可包括增强子序列。可用于本发明的启动子包括但不限于:组成型启动子;组织、器官和发育特异的启动子及诱导型启动子。合适的转录终止子包括但不限于:农杆菌胭脂碱合成酶终止子(nos终止子)、花椰菜花叶病毒camv35s终止子、tml终止子、豌豆rbcse9终止子和胭脂氨酸和章鱼氨酸合酶终止子。
可用现有的表达载体构建含有所述gsha12基因表达盒的重组载体。所述植物表达载体包括双元农杆菌载体和可用于植物微弹轰击的载体等。如pahc25、pbin438、pcambia1302、pcambia2301、pcambia1301、pcambia1300、pbi121、pcambia1391-xa或pcambia1391-xb(cambia公司)等。所述植物表达载体还可包含外源基因的3′端非翻译区域,即包含聚腺苷酸信号和任何其它参与mrna加工或基因表达的dna片段。所述聚腺苷酸信号可引导聚腺苷酸加入到mrna前体的3′端,如农杆菌冠瘿瘤诱导(ti)质粒基因(如胭脂碱合成酶基因nos)、植物基因(如大豆贮存蛋白基因)3′端转录的非翻译区均具有类似功能。使用本发明的基因构建植物表达载体时,还可使用增强子,包括翻译增强子或转录增强子,这些增强子区域可以是atg起始密码子或邻接区域起始密码子等,但必需与编码序列的阅读框相同,以保证整个序列的正确翻译。所述翻译控制信号和起始密码子的来源是广泛的,可以是天然的,也可以是合成的。翻译起始区域可以来自转录起始区域或结构基因。为了便于对转基因植物细胞或植物进行鉴定及筛选,可对所用植物表达载体进行加工,如加入可在植物中表达的编码可产生颜色变化的酶或发光化合物的基因(gus基因、萤光素酶基因等)、抗生素的标记基因(如赋予对卡那霉素和相关抗生素抗性的nptii基因,赋予对除草剂膦丝菌素抗性的bar基因,赋予对抗生素潮霉素抗性的hph基因,和赋予对氨甲喋呤抗性的dhfr基因,赋予对草甘磷抗性的epsps基因)或是抗化学试剂标记基因等(如抗除莠剂基因)、提供代谢甘露糖能力的甘露糖-6-磷酸异构酶基因。从转基因植物的安全性考虑,可不加任何选择性标记基因,直接以逆境筛选转化植株。
上述应用中,所述载体可为质粒、黏粒、噬菌体或病毒载体。
上述应用中,所述微生物可为酵母、细菌、藻或真菌,如农杆菌。
上述应用中,所述转基因植物细胞系、转基因植物组织和转基因植物器官均不包括繁殖材料。
为了解决上述技术问题,本发明还提供了蛋白质gsha12或上述相关生物材料的新用途。
本发明提供了蛋白质gsha12或上述相关生物材料在培育耐逆性提高的转基因植物中的应用。
本发明还提供了蛋白质gsha12或上述相关生物材料在植物育种中的应用。
上述应用中,所述耐逆性为耐碱性;所述耐碱性具体为耐碳酸盐胁迫;所述耐碳酸盐胁迫具体为耐nahco3胁迫。
上述应用中,所述调控为提高。
上述应用中,所述植物为单子叶植物或双子叶植物,所述双子叶植物具体可为豆科植物和/或十字花科植物和/或菊科植物;所述豆科植物可为大豆、百脉根、苜蓿或水黄皮;所述十字花科植物可为拟南芥或油菜;所述菊科植物可为向日葵;所述拟南芥可为拟南芥(哥伦比亚生态型col-0)。
为了解决上述技术问题,本发明最后提供了一种培育耐逆性提高的转基因植物的方法。
本发明提供的培育耐逆性提高的转基因植物的方法包括提高受体植物中蛋白质gsha12的表达量和/或活性,得到转基因植物的步骤;所述转基因植物的耐逆性高于所述受体植物。
上述方法中,所述耐逆性为耐碱性;所述耐碱性具体为耐碳酸盐胁迫。
上述方法中,所述耐碳酸盐胁迫为耐nahco3胁迫,具体体现为在nahco3胁迫的条件下:转基因植物的种子萌发速率高于受体植物和/或转基因植物的地上部分的鲜重高于受体植物和/或转基因植物的长势优于野生型。所述nahco3的浓度具体可为6mmnahco3或8mmnahco3或150mmnahco3。
上述方法中,所述提高受体植物中蛋白质gsha12的表达量和/或活性的方法为在受体植物中过表达蛋白质gsha12。所述过表达的方法为将蛋白质gsha12的编码基因导入受体植物。
上述方法中,所述蛋白质gsha12的编码基因的核苷酸序列是序列1所示的dna分子。在本发明的实施例中,所述蛋白质gsha12的编码基因(即序列表中序列1所示的dna分子)通过重组载体pcambia330035su-gsha12导入所述受体植物中,所述重组载体pcambia330035su-gsha12为在pcambia330035su载体中插入如序列表中序列1所示的gsha12基因。gsha12基因编码的蛋白的氨基酸序列如序列表中序列2所示。
上述方法中,所述受体植物为单子叶植物或双子叶植物,所述双子叶植物具体可为豆科植物和/或十字花科植物和/或菊科植物;所述豆科植物可为大豆、百脉根、苜蓿或水黄皮;所述十字花科植物可为拟南芥或油菜;所述菊科植物可为向日葵;所述拟南芥可为拟南芥(哥伦比亚生态型col-0)。
上述方法中,所述转基因植物理解为不仅包含将所述gsha12基因转化受体植物得到的第一代转基因植物,也包括其子代。对于转基因植物,可以在该物种中繁殖该基因,也可用常规育种技术将该基因转移进入相同物种的其它品种,特别包括商业品种中。所述转基因植物包括种子、愈伤组织、完整植株和细胞。
扩增编码上述蛋白质gsha12的核酸分子全长或其片段的引物对也属于本发明的保护范围。
本发明将gsha12基因超表达于拟南芥中,得到转gsha12拟南芥。通过实验证明:在碳酸盐胁迫下,转gsha12拟南芥的种子萌发速率和地上部分的鲜重均高于受体植物。说明gsha12基因可增强拟南芥对碳酸盐胁迫的耐性,gsha12蛋白可以为培育具有碳酸盐胁迫耐性的转基因植物的研究奠定基础。
附图说明
图1为gsha12基因在碳酸盐胁迫下的表达模式分析。
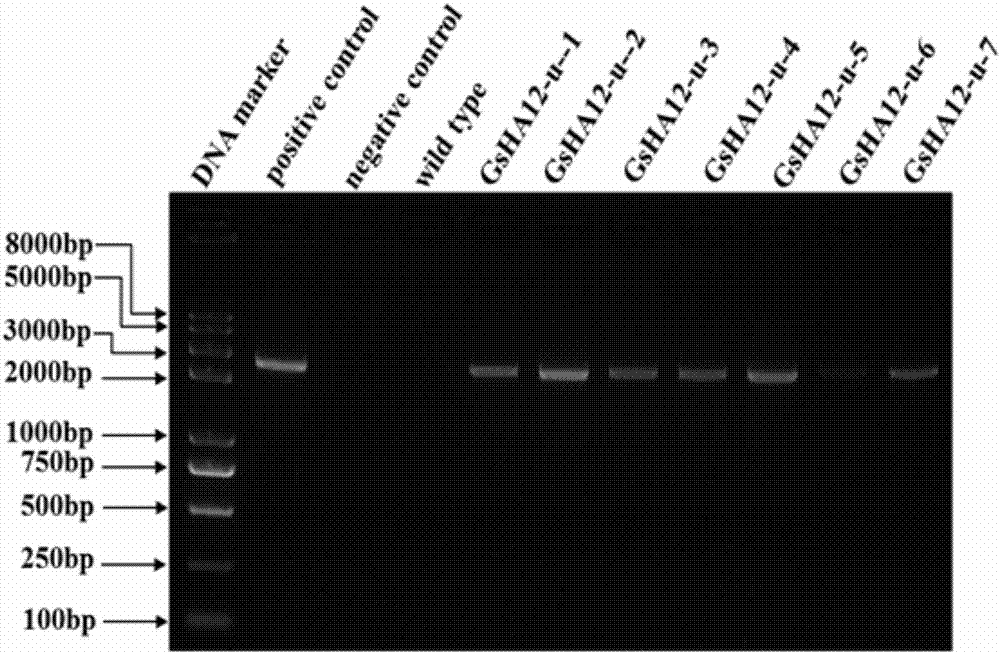
图2为转gsha12拟南芥植株的pcr检测。
图3为转gsha12拟南芥植株的rt-pcr检测。
图4为转gsha12拟南芥植株萌发期的耐碳酸盐胁迫分析。
图5为转gsha12拟南芥植株幼苗期的耐碳酸盐胁迫分析。
图6为转gsha12拟南芥植株成苗期的耐碳酸盐胁迫分析。
具体实施方式
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
下述实施例中的定量试验,均设置三次重复实验,结果取平均值。
下述实施例中的野生型大豆g07256记载于如下文献:mingzhesun,xiaolisun,yangzhao,huacai,chaoyuezhao,weiji,huiziduanmu,yangyu,yanmingzhu.ectopicexpressionofgsppck3andscmrpinmedicagosativaenhancesplantalkalinestresstoleranceandmethioninecontent.plosone2014,9(2):e89578,公众可从申请人(黑龙江八一农垦大学)处获得,该生物材料只为重复本发明的相关实验所用,不可作为其它用途使用。
下述实施例中的pcambia330035su记载于如下文献:xiaolisun,weiji,xiaodongding,xibai,huacai,shanshanyang,xueqian,mingzhesun,yanmingzhu.gsvamp72,anovelglycinesojar-snareprotein,isinvolvedinregulatingplantsalttoleranceandabasensitivity.plantcelltissorgancult2013,113:199–215,公众可从申请人(黑龙江八一农垦大学)处获得,该生物材料只为重复本发明的相关实验所用,不可作为其它用途使用。
下述实施例中的根癌农杆菌gv3101记载于如下文献:leecw,etal.agrobacteriumtumefacienspromotestumorinductionbymodulatingpathogendefenseinarabidopsisthaliana,plantcell,2009,21(9),2948-62,公众可从申请人(黑龙江八一农垦大学)处获得,该生物材料只为重复本发明的相关实验所用,不可作为其它用途使用。
下述实施例中的野生型拟南芥(哥伦比亚生态型col-0)记载于如下文献:luox,sunx,liub,etal.ectopicexpressionofawrkyhomologfromglycinesojaaltersfloweringtimeinarabidopsis[j].plosone,2013,8(8):e73295,公众可从申请人(黑龙江八一农垦大学)处获得,该生物材料只为重复本发明的相关实验所用,不可作为其它用途使用。
实施例1、野生大豆gsha12全长基因的克隆
1、总rna的提取
选取饱满的野生大豆g07256种子,用浓h2so4处理10min,无菌水冲洗3-4次,25℃暗培养2-3d催芽。待芽长到1-2cm时,将其转移至1/4hogland营养液中,置于人工气候箱中培养。取3周龄野生大豆g07256幼苗的根,采用rnapreppure试剂盒(tiangen)提取总rna。
2、cdna的获得
以oligodt为引物进行cdna第一条链的合成,方法参见invitrogen公司superscripttmiiireversetranscriptase说明书,得到野生大豆总cdna。
3、pcr扩增
以野生大豆总cdna为模板,采用引物gsha12-s和gsha12-as进行pcr扩增,得到pcr扩增产物。引物序列如下:
gsha12-s:5'-atgtggaatcctctttcatgggtca-3';
gsha12-as:5'-ctagacagtgtatgcttgctgtattgtgtc-3';
4、gsha12基因的克隆载体构建及测序
将pcr扩增产物与peasy-t载体连接,构建peasy-gsha12克隆载体。并对其进行测序。
测序结果表明:pcr扩增得到的大小为2667bp的dna片段,其核苷酸序列如序列表中序列1所示,将序列1所示的基因命名为gsha12基因,自5’端第1-2667位为orf,gsha12基因编码序列表中序列2所示的蛋白质,将序列2所示的氨基酸序列命名为gsha12蛋白。
实施例2、gsha12基因在碳酸盐胁迫条件下的表达模式分析
1、对3周龄的野生大豆g07256幼苗进行50mmnahco3碳酸盐胁迫处理,分别在处理0h、1h、3h、6h、12h、24h时取根尖组织。
2、采用rna提取试剂盒rneasyplantminikit(qiagen,hilden,germany)提取总rna,采用反转录试剂盒superscripttmiiireversetranscriptasekit(invitrogen,carlsbad,ca,usa)反转录获得cdna。
3、采用sybr定量试剂盒sybrpremixextaqtmiimix(takara,shiga,japan)于荧光定量pcr仪abi7500(appliedbiosystems,usa)上进行real-timepcr检测基因表达量。定量分析采用比较ct法(△△ct),以gapdh基因为内参,未经处理的样品作为对照。经gapdh基因均一化处理后,通过2-△△ct法计算gsha12基因表达量变化差异,以处理样本相对于未处理样本的倍数表示。gsha12基因特异引物为5’-aggcaagggctggtattcaagag-3’和5’-gagaccgtaatcctcgctctgca-3’,gapdh基因特异引物为5’-gactggtatggcattccgtgt-3’和5’-gccctctgattcctccttga-3’。
定量real-timepcr结果显示碳酸盐胁迫处理后,gsha12基因表达量呈上升趋势,并在碳酸盐胁迫处理12h后达到顶峰(图1),表明gsha12基因的表达受碳酸盐胁迫诱导。
实施例3、转gsha12拟南芥的获得及耐碳酸盐分析
一、转gsha12拟南芥的获得
1、以peasy-gsha12克隆载体质粒为模板,采用基因特异引物gsha12-u-f和gsha12-u-r进行pcr扩增,得到gsha12基因全长cds区。引物序列如下(下划线代表载体构建时所需的接头序列,其中u为user酶切位点):
gsha12-u-f:5’-ggcttaauatgtggaatcctctttcat-3’;
gsha12-u-r:5’-ggtttaauctagacagtgtatgcttgctg-3’。
2、用限制性内切酶paci和nt.bbvci对pcambia330035su载体进行双酶切,得到载体酶切产物。将获得的载体酶切产物、user酶(neb,m5505s)和步骤1获得的gsha12基因在37℃下孵育20min,利用user酶对gsha12基因片段的尿嘧啶处进行切割,形成可与pcambia330035su载体互补的粘性末端,接着25℃下孵育20min,得到重组表达载体pcambia330035su-gsha12,并送交测序。
测序结果表明:重组表达载体pcambia330035su-gsha12为在pcambia330035su载体中插入如序列表中序列1所示的gsha12基因,其保持pcambia330035su载体的其他序列不变后得到的载体。重组表达载体pcambia330035su-gsha12表达的gsha12蛋白。
3、采用冻融法,将pcambia330035su-gsha12载体转化至根癌农杆菌gv3101,获得重组农杆菌,并经pcr鉴定得到阳性转化子(含有序列表中序列1所示的gsha12转化子),用于侵染拟南芥植株。
4、转gsha12拟南芥的获得及鉴定
将上述重组农杆菌通过floral-dip法侵染野生型拟南芥(哥伦比亚生态型)。并将t0代种子表面消毒后,播种于含25mg/l固杀草的1/2ms培养基上进行筛选。将t1代抗性苗移栽至营养钵中培养,提取基因组dna,进行pcr鉴定和rt-pcr鉴定。
(1)pcr鉴定
采用easypureplantgenomicdnakit基因组提取试剂盒(全式金,ee111-01),提取野生型拟南芥植株和固杀草抗性植株的基因组dna,以获得的基因组dna为模板,并采用5'-atgtggaatcctctttcatgggtca-3'和5'-ctagacagtgtatgcttgctgtattgtgtc-3'引物进行pcr鉴定。结果如图2所示。从图中可以看出:抗性植株gsha12-u-1、gsha12-u-2、gsha12-u-3、gsha12-u-4、gsha12-u-5、gsha12-u-6和gsha12-u-7均扩增得到目的条带,而野生型拟南芥植株没有扩增得到的目的条带。
(2)rt-pcr鉴定
对pcr鉴定阳性的植株,提取总rna,利用实施例1中的real-timepcr引物(5’-aggcaagggctggtattcaagag-3’和5’-gagaccgtaatcctcgctctgca-3’)进行半定量rt-pcr,以拟南芥actin2基因为内参,检测gsha12基因在转基因植株中的表达量。actin2基因特异引物序列如下:actin2-rt-f:5’-ttacccgatgggcaagtc-3’;actin2-rt-r:5’-gctcatacggtcagcgatac-3’。
结果如图3所示。从图中可以看出:野生型拟南芥植株的rt-pcr无扩增产物,而转gsha12拟南芥均可以扩增出目的条带,表明外源基因gsha12不但已顺利整合到拟南芥的基因组上,而且能够在转基因拟南芥中正常转录表达。
将rt-pcr阳性的t1代转gsha12拟南芥单株收种子,并分别播种于含25mg/l固杀草的1/2ms培养基上进行筛选,观察t2代分离情况。如此重复,直至得到t3代转gsha12拟南芥纯合体株系。选取t3代转gsha12拟南芥纯合体株系(#1)和(#2)用于下述耐碳酸盐分析。
二、转gsha12拟南芥的耐碳酸盐分析
1、转gsha12拟南芥萌发期的耐碳酸盐分析
选取饱满的野生型拟南芥、t3代转gsha12拟南芥纯合体株系(#1)和(#2)种子,用5%naclo处理6-8min,灭菌ddh2o冲洗5-7次,4℃春化3d,一部分播种于正常的1/2ms培养基,一部分播种于含6mmnahco3的1/2ms培养基,22℃培养3d,统计种子萌发率。实验重复三次,每种处理各株系采用30株植株。
结果如图4所示。从图中可以看出:碳酸盐胁迫严重抑制了野生型与转gsha12拟南芥种子的萌发,但转gsha12拟南芥纯合体株系(#1)和(#2)的种子萌发率高于野生型。
2、转gsha12拟南芥幼苗期的耐碳酸盐分析
选取饱满的野生型拟南芥、t3代转gsha12拟南芥纯合体株系(#1)和(#2)种子,春化后播种于正常的1/2ms培养基,22℃培养11d。待拟南芥长至六叶期,将幼苗移至正常、或含8mmnahco3的1/2ms培养基竖直培养7d。实验重复三次,每种处理各株系采用15株植株。
结果如图5所示。从图中可以看出:碳酸盐胁迫抑制了野生型和转gsha12拟南芥根的伸长和地上部分的生长,并且t3代转gsha12拟南芥纯合体株系(#1)和(#2)地上部分的鲜重也显著高于野生型。
3、转gsha12拟南芥成苗期的耐碳酸盐分析
选取饱满的野生型拟南芥、t3代转gsha12拟南芥纯合体株系(#1)和(#2)种子,春化后播种于营养钵中(营养土:君子兰土:蛭石1:1:1),置于人工气候培养箱中培养。选取长势一致的4周龄拟南芥植株,每3d浇灌1次150mmnahco3(ph9.0)溶液进行碳酸盐胁迫处理,观察胁迫处理后植株长势。实验重复三次,每种处理各株系采用30株植株。
结果如图6所示。从图中可以看出:碳酸盐胁迫处理后野生型和转gsha12拟南芥都逐渐失绿发黄,变紫甚至死亡,但是t3代转gsha12拟南芥纯合体株系(#1)和(#2)的长势明显优于野生型。
上述结果表明gsha12基因超量表达显著提高了植物的耐逆性,尤其是碳酸盐胁迫耐性。
序列表
<110>黑龙江八一农垦大学
<120>gsha12蛋白及其编码基因在调控植物耐逆性中的应用
<160>2
<210>1
<211>2667bp
<212>dna
<213>野生大豆(glycinesoja)
<400>1
atgtggaatcctctttcatgggtcatggaagctgcagcaatcatggccattgctttggcc60
aatggaggaggaaaacctcctgattggcaagactttgttgggattatcacactccttatt120
atcaattcaacaataagtttcattgaggagaacaatgctggtaatgctgcggcagctctg180
atggctcgtttagcacctaaagctaagttccttcgagatgggaaatggattgaggaggat240
gctagcattcttgttcctggtgatataattagtgttaagctaggggatattatccctgcg300
gatgctcgtctacttgaaggtgatccactgaagattgatcagtctgcacttacaggcgag360
tctcttcctgtcacaaaaggccctggtgatagtgtttattcaggctccacatgcaagcag420
ggagagatcaatgcagttgttattgccacaggagttcataccttctttggcaaagctgct480
catcttgtggactccacaaatcaagttggtcatttccagaaggtcctgactgcaattggg540
aacttctgcatatgttccattgctgtgggaatgatagtagagataattgtcatgtaccca600
attcaacaccgggaatatcgtcctgggattgacaatctgcttgtgcttcttattggagga660
attcctattgccatgcctactgttttgtcagtgacaatggcaattggatcccatcgctta720
gctcagcagggtgctattactaaaagaatgacagcaatagaagagatggcaggaatggat780
gtattatgtagtgacaaaactggaactttgactttgaataaactgacagttgacaagaat840
cttattgagatttttgctaaaggagttgacgtagatactgttgttctcatggccgctcgg900
gctgcacgattggaaaaccaagatgctatagatgccgctattgtagggatgttgggtgat960
ccaaaagaggcaagggctggtattcaagaggttcacttcctacccttcaatccaactgac1020
aagcgaactgcaatcacttatatagacggtgaaagtaaaatgcatcgtgtcagcaaagga1080
gcaccagagcagattttgaatcttgcacgcaataaatcagagatagaacgcagagttcat1140
tctgtcattgataagtttgcagagcgaggattacggtctcttgcagtagcttaccaggaa1200
gttcctgatggaaagaaagaaagccaaggagggccttggcaatttattggactgttgcct1260
ttatttgacccacctagacatgatagtgctgagacaatacgaagggcattaaatcttgga1320
gtaaatgttaaaatgataacaggtgatcaactagcaataggaaaagaaacaggacgccgt1380
ctggggatgggaaccaacatgtacccttcatcggctttattgggccaaaacaaggatgaa1440
gcaattgctaccttgccagttgatgagttgattgaaaaagcagatggatttgctggtgtt1500
tttcctgaacacaaatatgagatcgtgaaacgtttacaagctaggaaacacatatgtgga1560
atgactggtgatggggttaacgatgctcctgctcttaaaaaggcagatattggaatagct1620
gtcgccgatgctactgatgcagctcgtagtgcttctgatattgttctgactgaacctggt1680
ctcagtgttatcatcagtgctgtactgaccagtcgagcaatattccaaaggatgaagaat1740
tacacaatctatgcagtttccatcacaatccgtattgtgcttggtttcatgttactggcc1800
ctcatatggcattttgattttccaccattcatggtgctgattattgctattcttaatgac1860
ggtaccattatgacgatatcaaaggacagggtgaaaccatctccatatccagatagctgg1920
aagttggccgagatctttaccactggaataattcttggtggttatttggctatgatgaca1980
gttattttcttttgggcagcatataaaacagattttttccctcaaacatttggagtctca2040
agtcttcagaaaaaggatagggatgactttagaaagcttgcctcagcaatatacctacaa2100
gttagcacaattagtcaggccctcatattcattacacgggctcggagttggtcttatgtt2160
gaacgtccgggtttgttacttgttgcagcttttgttatcgcccagctgatagctacctta2220
attgcagtttatgcaaattggagtttcgctgctattgaagggattggatggggttgggct2280
ggtgttgtttggctttacaacctcatcttttatatcccacttgactttatcaagttcata2340
attcgatatgccttgagtggaagggcttgggatcttgttattgaacaaaggattgctttt2400
acaaggaaaaaagattttggaaaggaagaacgtgaacttaaatgggcacatgcacagagg2460
acgcttcacggccttcacccaccagagactaagatgttcaatgaacgtacaagttacaca2520
gaacttaatcagatggctgaagaggctagaagacgagcagaaattgcaaggctgagagaa2580
ctgcatacacttaagggtcgtgttgagtctgtggttagactgaagggtcttaacattgac2640
acaatacagcaagcatacactgtctag2667
<210>2
<211>888
<212>prt
<213>野生大豆(glycinesoja)
<400>2
mettrpasnproleusertrpvalmetglualaalaalailemetala
151015
ilealaleualaasnglyglyglylysproproasptrpglnaspphe
202530
valglyileilethrleuleuileileasnserthrileserpheile
354045
glugluasnasnalaglyasnalaalaalaalaleumetalaargleu
505560
alaprolysalalyspheleuargaspglylystrpileglugluasp
65707580
alaserileleuvalproglyaspileileservallysleuglyasp
859095
ileileproalaaspalaargleuleugluglyaspproleulysile
100105110
aspglnseralaleuthrglygluserleuprovalthrlysglypro
115120125
glyaspservaltyrserglyserthrcyslysglnglygluileasn
130135140
alavalvalilealathrglyvalhisthrphepheglylysalaala
145150155160
hisleuvalaspserthrasnglnvalglyhispheglnlysvalleu
165170175
thralaileglyasnphecysilecysserilealavalglymetile
180185190
valgluileilevalmettyrproileglnhisargglutyrargpro
195200205
glyileaspasnleuleuvalleuleuileglyglyileproileala
210215220
metprothrvalleuservalthrmetalaileglyserhisargleu
225230235240
alaglnglnglyalailethrlysargmetthralailegluglumet
245250255
alaglymetaspvalleucysserasplysthrglythrleuthrleu
260265270
asnlysleuthrvalasplysasnleuilegluilephealalysgly
275280285
valaspvalaspthrvalvalleumetalaalaargalaalaargleu
290295300
gluasnglnaspalaileaspalaalailevalglymetleuglyasp
305310315320
prolysglualaargalaglyileglngluvalhispheleuprophe
325330335
asnprothrasplysargthralailethrtyrileaspglygluser
340345350
lysmethisargvalserlysglyalaprogluglnileleuasnleu
355360365
alaargasnlyssergluilegluargargvalhisservalileasp
370375380
lysphealagluargglyleuargserleualavalalatyrglnglu
385390395400
valproaspglylyslysgluserglnglyglyprotrpglnpheile
405410415
glyleuleuproleupheaspproproarghisaspseralagluthr
420425430
ileargargalaleuasnleuglyvalasnvallysmetilethrgly
435440445
aspglnleualaileglylysgluthrglyargargleuglymetgly
450455460
thrasnmettyrproserseralaleuleuglyglnasnlysaspglu
465470475480
alailealathrleuprovalaspgluleuileglulysalaaspgly
485490495
phealaglyvalpheprogluhislystyrgluilevallysargleu
500505510
glnalaarglyshisilecysglymetthrglyaspglyvalasnasp
515520525
alaproalaleulyslysalaaspileglyilealavalalaaspala
530535540
thraspalaalaargseralaseraspilevalleuthrgluprogly
545550555560
leuservalileileseralavalleuthrserargalailephegln
565570575
argmetlysasntyrthriletyralavalserilethrileargile
580585590
valleuglyphemetleuleualaleuiletrphispheaspphepro
595600605
prophemetvalleuileilealaileleuasnaspglythrilemet
610615620
thrileserlysaspargvallysproserprotyrproaspsertrp
625630635640
lysleualagluilephethrthrglyileileleuglyglytyrleu
645650655
alametmetthrvalilephephetrpalaalatyrlysthraspphe
660665670
pheproglnthrpheglyvalserserleuglnlyslysaspargasp
675680685
aspphearglysleualaseralailetyrleuglnvalserthrile
690695700
serglnalaleuilepheilethrargalaargsertrpsertyrval
705710715720
gluargproglyleuleuleuvalalaalaphevalilealaglnleu
725730735
ilealathrleuilealavaltyralaasntrpserphealaalaile
740745750
gluglyileglytrpglytrpalaglyvalvaltrpleutyrasnleu
755760765
ilephetyrileproleuasppheilelyspheileileargtyrala
770775780
leuserglyargalatrpaspleuvalilegluglnargilealaphe
785790795800
thrarglyslysasppheglylysglugluarggluleulystrpala
805810815
hisalaglnargthrleuhisglyleuhisproprogluthrlysmet
820825830
pheasngluargthrsertyrthrgluleuasnglnmetalagluglu
835840845
alaargargargalagluilealaargleuarggluleuhisthrleu
850855860
lysglyargvalgluservalvalargleulysglyleuasnileasp
865870875880
thrileglnglnalatyrthrval
885
- 还没有人留言评论。精彩留言会获得点赞!
- 蝎神经毒素AaIT及其编码基因与应用的制造方法与工艺
- 腈水解酶突变体及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白AsTMK1及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种花狭口蛙的基因及其编码的多肽和该多肽的用途的制造方法与工艺
- 一种泽陆蛙基因及其编码的多肽和该多肽的用途的制造方法与工艺
- 降低种子重量的白桦AP2基因及其编码蛋白的制造方法与工艺
- 可编码ArtRD蛋白基因及其蛋白和应用的制造方法与工艺
- 蝎神经毒素AaIT及其编码基因与应用的制造方法与工艺
- 腈水解酶突变体及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白AsTMK1及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 腈水解酶突变体及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白AsTMK1及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种花狭口蛙的基因及其编码的多肽和该多肽的用途的制造方法与工艺
- 一种泽陆蛙基因及其编码的多肽和该多肽的用途的制造方法与工艺
- 一种水稻MIT1基因、其编码蛋白及应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种水稻MIT1基因、其编码蛋白及应用的制造方法与工艺
- 蛋白StTrxF、编码基因及其在提高植物淀粉含量中的应用的制造方法与工艺
- 蛋白质及其编码基因在调控植物抗黄萎病性中的应用的制造方法与工艺
- 甘薯耐逆相关蛋白IbFBA2、编码基因、重组载体与应用的制造方法与工艺
- 植物抗旱相关蛋白ZmNAC111及其编码基因与应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种水稻MIT1基因、其编码蛋白及应用的制造方法与工艺
- 蛋白StTrxF、编码基因及其在提高植物淀粉含量中的应用的制造方法与工艺
- 植物抗旱相关蛋白ZmNAC111及其编码基因与应用的制造方法与工艺
- 核盘菌凝集素SSL‑6蛋白及其编码基因与应用的制造方法与工艺
- 植物抗病毒相关蛋白GmSN1及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种水稻MIT1基因、其编码蛋白及应用的制造方法与工艺
- 蛋白StTrxF、编码基因及其在提高植物淀粉含量中的应用的制造方法与工艺
- 蛋白质及其编码基因在调控植物抗黄萎病性中的应用的制造方法与工艺
- 植物抗旱相关蛋白ZmNAC111及其编码基因与应用的制造方法与工艺
- 核盘菌凝集素SSL‑6蛋白及其编码基因与应用的制造方法与工艺