用于稳定反式剪接的内含肽修饰的蛋白酶的方法和组合物与流程
相关申请的交叉引用
本申请要求申请号为62/069,653的美国临时申请的优先权,该美国临时申请于2014年10月28日提交并且通过引用并入本文,如同完整阐述。
与本申请一起以电子方式提交的标题为“sequencelisting”的序列表创建于2015年10月28日,具有59,091字节大小,通过引用并入本文,如同完整阐述。
本发明涉及能够在液体洗涤剂和家庭护理产品中紧密调节反式剪接的内含肽修饰的蛋白酶活性的方法和组合物,以便在制剂中稳定储存并快速有效地恢复蛋白酶活性,同时降低清洁效能的损失。
背景技术:
蛋白酶是各种液体洗涤剂、洗衣洗涤剂、碗碟洗涤液、油漆、涂料和工业清洁剂中的常见组分。众所周知,蛋白酶在制剂中难以处理,因为它们不仅降解其他蛋白质而且还可以自我消化。因此,蛋白酶稳定仍然是液体洗涤剂行业面临的最大挑战之一。目前的稳定技术通常基于使用高浓度的化学蛋白酶抑制剂,这会增加化学负载和成本。希望有更环保的、生物基的和低成本的解决方案以稳定蛋白酶,而不影响产品性能。之前已经证明,可以使用反式剪接的内含肽技术来控制蛋白酶活性。
技术实现要素:
本发明的一个方面涉及一种内含肽修饰的蛋白酶。所述内含肽修饰的蛋白酶包括:第一前体和第二前体。所述第一前体包括靶蛋白酶的n-外显肽和反式剪接的内含肽的可溶性增强的n-内含肽。所述n-外显肽的羧基末端与所述可溶性增强的n-内含肽的氨基末端融合。所述第二前体包括反式剪接的内含肽的可溶性增强的c-内含肽和靶蛋白酶的c-外显肽。所述可溶性增强的c-内含肽的羧基末端与c-外显肽的氨基末端融合。在剪接之前,所述第一前体与所述第二前体是分离的。所述内含肽修饰的蛋白酶与所述靶蛋白酶相比具有增强的可溶性和降低的活性。经内含肽修饰的蛋白酶的反式剪接以及n-外显肽与c-外显肽的融合,获得靶蛋白酶活性。
本发明的一个方面涉及一种表达盒。所述表达盒包括编码内含肽修饰的蛋白酶的第一前体或第二前体的一种或多种多核苷酸。所述第一前体包括靶蛋白酶的n-外显肽和反式剪接的内含肽的可溶性增强的n-内含肽。所述n-外显肽的羧基末端与所述可溶性增强的n-内含肽的氨基末端融合。所述第二前体包括反式剪接的内含肽的可溶性增强的c-内含肽和靶蛋白酶的c-外显肽。所述可溶性增强的c-内含肽的羧基末端与c-外显肽的氨基末端融合。经所述表达盒的表达,在反式剪接之前所述第一前体与所述第二前体将是分离的。所述内含肽修饰的蛋白酶与所述靶蛋白酶相比将具有增强的可溶性和降低的或抑制的活性。经内含肽修饰的蛋白酶的反式剪接以及n-外显肽与c-外显肽的融合,所述靶蛋白酶的活性将恢复。
本发明的一个方面涉及一种组合物,该组合物含有:内含肽修饰的蛋白酶的第一前体和内含肽修饰的蛋白酶的第二前体。所述第一前体包括与反式剪接的内含肽的可溶性增强的n-内含肽融合的靶蛋白酶的n-外显肽。所述n-外显肽的羧基末端与所述可溶性增强的n-内含肽的氨基末端融合。所述第二前体包括与靶蛋白酶的c-外显肽融合的反式剪接的内含肽的可溶性增强的c-内含肽。所述可溶性增强的c-内含肽的羧基末端与所述c-外显肽的氨基末端融合。在反式剪接之前所述第一前体与所述第二前体是分离的。所述内含肽修饰的蛋白酶与所述靶蛋白酶相比具有增强的可溶性和降低的活性,且经内含肽修饰的蛋白酶的反式剪接以及n-外显肽与c-外显肽的融合,获得靶蛋白酶活性。
本发明的一个方面涉及一种家庭护理产品,该家庭护理产品包括本文所公开的任意一种所述内含肽修饰的蛋白酶或所述组合物,以及一种或多种效应物(effector)。
本发明的一个方面涉及一种调节蛋白酶活性的方法。所述方法包括形成本文所公开的任意一种所述家庭护理产品。
本发明的一个方面涉及一种在混合物中储存蛋白酶的方法。所述方法包括制备本文所公开的任意一种所述内含肽修饰的蛋白酶或所述组合物。所述方法还包括将所述内含肽修饰的蛋白酶或所述组合物与具有一种或多种效应物的选自由:洗涤剂、肥皂、工业清洁剂和碗碟洗涤液组成的组中的至少一种试剂结合以形成混合物。
附图说明
当结合附图阅读时,在下面进行详细描述的本发明的实施方式将更好地被理解。为了说明本发明的目的,示出优选的实施方式的附图。然而,应当理解的是,本发明并不限于所示的确切设置和手段。在附图中:
图1示出了可溶性优化的反式剪接的ni和ic的示意图。
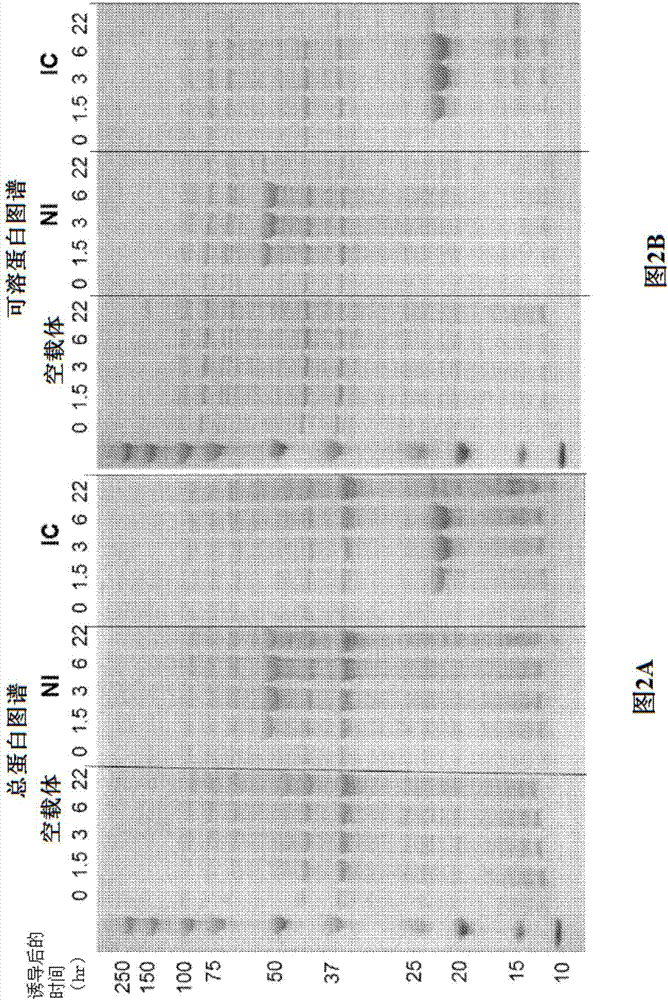
图2a-2b示出了大肠杆菌中可溶性优化的ni和ic的表达。图2a示出了总蛋白图谱。图2b示出了可溶蛋白图谱。
图3a-3e示出了用于稀释诱导型iprotease活性的洗涤剂补充剂筛选的结果,对钠盐补充剂(图3a)、钾盐补充剂(图3b)、铵盐和锂盐补充剂(图3c)、带电聚合盐补充剂(图3d)和多元醇补充剂(图3e)进行了稀释诱导型iprotease活性的测定。
图4a-4b示出了盐补充剂在不同的洗涤剂环境(洗涤剂-2(图4a)和洗涤剂-1(图4b))中表现相似。
图5a-5b示出了kcl在含水缓冲液中可逆地抑制iprotease活性。图5a示出了稀释诱导。图5b示出了盐抑制。
图6a-6b示出了kcl促进剪接前体的缔合。图6a示出了br缓冲液中连续稀释的高盐相关(ni+ic)预混物中的iprotease活性。图6b示出了br缓冲液中单独的连续稀释的ni和ic(ni-ic)的混合中的iprotease活性。
图7a-7b示出了在剪接的前体混合之后显示剪接的考马斯凝胶。图7a示出了在含水缓冲液中稀释未盐化的预混物之后的蛋白图谱。图7b示出了在含水缓冲液中稀释盐化的预混物之后的蛋白图谱。
图8a-8b示出了稀释诱导型iprotease活性基准为1%savinase。图8a示出了iprotease的稀释活性图谱。图8b示出了savinase的稀释活性图谱。
图9a-9b示出了kcl稳定的(ni+ic)预混物对于iprotease(图9a)和savinase(图9b)没有酶活性。
图10a-10b示出了在含水缓冲液中kcl稳定的(ni+ic)预混物的储存稳定性。图10a示出了未储存的预混物的iprotease活性。图10b示出了储存8周的预混物的iprotease活性。
图11a-11b示出了可以将kcl稳定的(ni+ic)预混物配制成洗涤剂-1,而不损失稀释诱导活性。图11a示出了1mkcl的稀释活性。图11b示出了缺乏1mkcl的50%洗涤剂-1的稀释活性。
图12示出了kcl在洗涤剂配方中具有双重作用:稳定用于配制的(ni+ic)预混物并保持洗涤剂中的稳定性。
图13示出了在加速老化条件下,洗涤剂-1中的iprotease储存稳定性与savinase相当。
图14示出了在洗涤剂稀释试验中,kcl稳定的(ni+ic)预混物与四种液体洗衣洗涤剂相容。
图15示出了在四种液体洗衣洗涤剂中,洗涤剂配方的iprotease的去污效果与savinase相当。
图16示出了在加速老化条件下,三种液体洗衣洗涤剂中的稀释诱导型iprotease活性的稳定性。
图17示出了在三种液体洗衣洗涤剂中在加速老化条件下的去污稳定性。
图18示出了用于表达ni和ic的双顺反子表达盒,并且包括seqidno:48的移码的rbs的核酸序列。
图19a和19b示出了iptg诱导后体外反式剪接3小时的蛋白酶活性。图19a示出了内含肽12、12、44、46、47、50、79、81和103剪接后的蛋白酶活性。图19b示出了内含肽110、116、123、128、135、143和150剪接后的蛋白酶活性。
具体实施方式
用于以下说明中的某些术语仅为方便起见,并且不是限制性的。除非特别说明,在权利要求书和说明书中相应部分使用的词语“一个(a)”和“一种(one)”被定义为包括一个或多个所引用的项。这个术语包括上面特别提到的词、它们的衍生词、以及类似含义的词语。短语“至少一种”随后有两个或两个以上的项,如“a、b或c”是指a、b或c中的任何一个以及它们的任意组合。
一种实施方式包括具有增强的可溶性的内含肽修饰的蛋白酶。内含肽修饰的蛋白酶可以包括在控制靶蛋白酶活性的位置中共同含有靶蛋白酶和与靶蛋白酶融合的内含肽的组分。靶蛋白酶和内含肽均被分割成两部分,靶蛋白的一部分与内含肽的一部分融合,而靶蛋白的另一部分与内含肽的另一部分融合。尽管两个融合序列是独立的实体,但它们在本文中统称为“内含肽修饰的蛋白酶”。单独的融合体可以被称为“第一前体”和“第二前体”。总的来说,第一前体和第二前体可以被称为内含肽修饰的蛋白酶的组分。如本文所用,n-外显肽和n-内含肽分别指靶蛋白酶和内含肽的氨基末端部分。同样,c-外显肽和c-内含肽分别指靶蛋白酶和内含肽的羧基末端部分。
内含肽修饰的蛋白酶中的内含肽是能够影响内含肽修饰的蛋白酶的反式剪接的反式剪接的内含肽。第一前体包括靶蛋白酶的n-外显肽和反式剪接的内含肽的可溶性增强的n-内含肽。n-外显肽的羧基末端可以与可溶性增强的n-内含肽的氨基末端融合。可溶性增强的n-内含肽可以包括第一可溶性增强子。n-内含肽的羧基末端可以通过第一连接体与第一可溶性增强子融合。第二前体包括反式剪接的内含肽的可溶性增强的c-内含肽和靶蛋白酶的c-外显肽。可溶性增强的c-内含肽的羧基末端可以与c-外显肽的氨基末端融合。可溶性增强的c-内含肽可以包括第二可溶性增强子。第二可溶性增强子可以通过第二连接体与c-内含肽的氨基末端融合。在内含肽修饰的蛋白酶剪接之前,第一前体与第二前体是分离的。
在一种实施方式中,第一可溶性增强子或第二可溶性增强子的至少一种可以含有、基本上由、或由硫氧还蛋白结构域(trx)、小泛素相关蛋白(sumo)、谷胱甘肽-s转移酶(gst)、麦芽糖结合蛋白(mbp)、n利用物质(anusa)或十七千道尔顿蛋白(skp)组成。第一可溶性增强子可以是与第二可溶性增强子相同或不同的可溶性增强子。第一可溶性增强子或第二可溶性增强子的至少一种可以是trx。第一种可溶性增强子可以含有、基本上由、或由与seqidno:51具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。第二可溶性增强子可以含有、基本上由、或由与seqidno:53具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。可溶性增强子可以是能够增强第一前体或第二前体的可溶性的任何其他分子。可溶性增强子可以与内含肽共价连接或融合。
在一种实施方式中,第一连接体或第二连接体可以是适于将可溶性增强子与n-内含肽或c-内含肽连接的任何连接体。连接体可以具有可变的长度、组成和灵活性。连接体可以是短而灵活的。连接体可以含有一段甘氨酸(g)和丝氨酸(s)残基。连接体可以包括ggggs(seqidno:55)、gggggs(seqidno:56)或ggggg(seqidno:57)的氨基酸序列。连接体可以被配置为限定小的结构转弯,以保持第一前体或第二前体的可溶性增强子结构域远离分割的内含肽对的接口。结构建模可用于预测连接体的最佳拟合长度和组成。
在一种实施方式中,第一连接体或第二连接体的至少一种可以是dpng连接体。dpng连接体可以含有seqidno:5的氨基酸序列。第一连接体或第二连接体可以是任何其他合适的连接体。
靶蛋白酶可以是任何蛋白酶。如前所述,当内含肽被修饰时,靶蛋白酶被分割成两部分,并且内含肽修饰的部分将包括第一前体和第二前体。如本文所用,“蛋白酶”是指催化肽键水解的酶或其部分。酶可以为但不限于本文的具有催化肽键水解的活性的氨基酸序列或蛋白。酶可以为本文的氨基酸序列或蛋白的变体,并且具有催化肽键水解的活性,其中,变体是氨基酸序列或蛋白的突变体和/或部分。变体可以具有氨基酸序列或蛋白的至少40%的活性,该氨基酸序列或蛋白具有催化肽键水解的活性。关于savinase活性,可以通过实施例2中概述的酶试验来分析活性。
靶蛋白酶可以是作为肽水解酶的分类为ec3.4的酶。靶蛋白酶是分类为ec3.4.99、ec3.4.21.62、丝氨酸蛋白酶、碱性蛋白酶、角蛋白酶和其他中的一种。靶蛋白酶可以为但不限于:金属蛋白酶、半胱氨酸蛋白酶、天冬氨酸蛋白酶或atp依赖性蛋白酶。
在一种实施方式中,靶蛋白酶可以是碱性蛋白酶。碱性蛋白酶可以是枯草杆菌蛋白酶(subtilisin)家族的蛋白酶。靶蛋白酶可以是来自迟缓芽孢杆菌(b.lentus)的枯草杆菌蛋白酶(bl,p29599,);来自短小芽孢杆菌(b.pumilus)的枯草杆菌蛋白酶(po7518);来自枯草芽孢杆菌(b.subtilis)的枯草杆菌蛋白酶(e,p04189);来自地衣芽孢杆菌(b.licheniformis)的枯草杆菌蛋白酶(dy,p00781);来自解淀粉芽孢杆菌(b.amyloliquefaciens)的枯草杆菌蛋白酶(bpn,p00782);来自芽孢杆菌ta39菌株(bacillussp.strainta39)的枯草杆菌蛋白酶(p28842);来自嗜热脂肪土芽孢杆菌(geobacillusstearothermophilus)的枯草杆菌蛋白酶(j,p29142);来自枯草芽孢杆菌纳豆亚种(b.subtilissubsp.natto)的枯草杆菌蛋白酶(nat,p35835);来自地衣芽孢杆菌(b.licheniformis)的枯草杆菌蛋白酶(carlsberg,p00780);来自枯草芽孢杆菌淀粉糖化亚种(b.subtilissubsp.amylosacchariticus)的枯草杆菌蛋白酶(amylosacchariticus,p00780);枯草杆菌蛋白酶carlsberg;枯草杆菌蛋白酶bpn;或来自短小芽孢杆菌(bacilluspumilus)的枯草杆菌蛋白酶sapb(cbs)。
枯草杆菌蛋白酶家族的蛋白酶可以是savinase。savinase是来自迟缓芽孢杆菌(bacilluslentus)的细胞外碱性蛋白酶(uniprot登录号为:p29600(http://www.uniprot.org/uniprot/p29600)和液体洗衣洗涤剂中的常见蛋白酶。靶蛋白酶可以含有、基本上由、或由与seqidno:6的序列(其是savinase,p29600的序列)具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成。
靶蛋白酶可以是来自地衣芽孢杆菌(bacilluslicheniformis)的蛋白酶(uniprot登录号为:q6pnn5)。靶蛋白酶可以含有、基本上由、或由与seqidno:8的序列(其是地衣芽孢杆菌蛋白酶,q6pnn5的序列)具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成。
靶蛋白酶可以是酸性蛋白酶。酸性蛋白酶广泛用于奶酪制作、食品和饲料制备以及皮革工业。酸性蛋白酶可以是细菌蛋白酶、真菌蛋白酶或哺乳动物蛋白酶。酸性蛋白酶可以是来自黄曲霉(aspergillusflavus)的天冬氨酸蛋白酶曲霉素a(pepa)(uniprot登录号为:b8nly9(pepa_aspfn))。靶蛋白酶可以含有、基本上由、或由与seqidno:9的序列(其是来自黄曲霉的天冬氨酸蛋白酶曲霉素a(pepa),b8nly9(pepa_aspfn)的序列)具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成。
内含肽可以是任何内含肽。内含肽可以是天然能够反式剪接的内含肽。内含肽可以是经基因工程改造的用于反式剪接的顺式剪接的内含肽。在本文的实施例17和18中描述了将顺式剪接的内含肽经基因工程改造为人工反式剪接的内含肽并人工测试分割的内含肽以恢复蛋白酶活性的方法。内含肽为多肽,其具有在翻译后从蛋白切割自身的能力,并且可以介导剩余的蛋白片段(外显子)的连接,并且可以具有在特定位点切割dna用于其繁殖的能力。内含肽可以被修饰。修饰的内含肽可以具有切割自身的能力,但可能丧失切割dna的能力。内含肽可以为但不限于mtth、pho_rada、tko_rada、sce_vma、mvma和pab_lon。内含肽可以是在inbase,内含肽数据库(http://www.neb.com/neb/inteins.html;perler等人1992procnatlacadsciusa89:5577)中发现的,其通过引用并入本文,如同完整阐述。
内含肽可以为apmvpol(多食棘阿米巴拟病毒(acanthomoebapolyphagamimivirus))、abrprp8(短柄曲霉frr2439(aspergillusbrevipesfrr2439))、aca-jer2004prp8(荚膜阿耶罗菌(ajellomycescapsulatus))、aca-h143prp8(荚膜阿耶罗菌h143)、ade-er3prp8(皮炎组织胞浆菌er-3(ajellomycesdermatitidiser-3))、aca-nam1prp8(荚膜阿耶罗菌nam1)、afu-af293prp8(烟曲霉菌椭圆变种af293菌株(aspergillusfumigatusvar.ellipticusstrainaf293))、ade-slh14081prp8(皮炎组织胞浆菌slh14081)、afu-frr0163prp8(烟曲霉菌frr0163菌株(aspergillusfumigatusstrainfrr0163))、afu-nrrl5109prp8(烟曲霉菌椭圆变种nrrl5109菌株)、ani-fgsca4prp8(构巢曲霉fgsca(aspergillusnidulansfgsca))、agi-nrrl6136prp8(巨大曲霉nrrl6136菌株(aspergillusgiganteusstrainnrrl6136))、aviprp8(绿垂曲霉frr0577菌株(aspergillusviridinutansstrainfrr0577))、bciprp8(葡萄孢菌(botrytiscinerea))、bde-jel423prp8-1(蛙壶菌jel423(batrachochytriumdendrobatidisjel423))、bde-jel197rpb2(蛙壶菌jel197)、bde-jel423eif-5b(蛙壶菌jel423)、bde-jel423prp8-2(蛙壶菌jel423)、bfu-b05prp8(灰葡萄孢霉菌b05.10(botryotiniafuckelianab05.10))、bde-jel423rpc2(蛙壶菌jel423)、civrir1(齐洛虹彩病毒(chiloiridescentvirus))、cv-ny2aorf212392(小球藻病毒ny2a(chlorellavirusny2a))、cv-ny2arir1(小球藻病毒ny2a)、czivrir1(褐新西兰肋翅鳃角金龟虹彩病毒(costelytrazealandicairidescentvirus))、cba-wm02.98prp8(杆孢隐球菌wm02.98菌株(cryptococcusbacillisporusstrainwm02.98))、cba-wm728prp8(杆孢隐球菌wm728菌株)、ceuclpp(衣滴虫(chlamydomonaseugametos))、cgaprp8(格特隐球菌(cryptococcusgattii))、claprp8(罗伦隐球菌cbs139菌株(cryptococcuslaurentiistraincbs139))、cmoclpp(绿藻utex97菌株(chlamydomonasmoewusiistrainutex97))、cmorpb2(绿藻utex97菌株)、cglvma(光滑念珠菌(candidaglabrata))、cpathrrs(近平滑念珠菌clib214菌株(candidaparapsilosisstrainclib214))、fne-aprp8(新型线黑粉菌a血清型(filobasidiellaneoformansserotypea))、cne-jec21prp8(新型隐球菌jec21(cryptococcusneoformansjec21))、fne-adprp8(新型隐球菌ad血清型)、crerpb2(莱茵衣藻(chlamydomonasreinhardtii))、crovrpb2(伦比约餐厅虫病毒bv-pw1(cafeteriaroenbergensisvirusbv-pw1))、crovrir1(伦比约餐厅虫病毒bv-pw1)、crovpol(伦比约餐厅虫病毒bv-pw1)、crovtop2(伦比约餐厅虫病毒bv-pw1)、ctrthrrs(热带念珠菌atcc750(candidatropicalisatcc750))、cstrpb2(复蚊雕蚀菌(coelomomycesstegomyiae))、ctrvma(热带念珠菌)、ddirpc2(盘基网柄菌ax4菌株(dictyosteliumdiscoideumstrainax4))、dhanvma(汉逊德巴利酵母cbs767(debaryomyceshanseniicbs767))、ctr-mya3404vma(热带念珠菌mya-3404)、dhanglt1(汉逊德巴利酵母cbs767)、fterpb2(特瑞特斯弗洛伊德拉菌utex1709菌株(floydiellaterrestrisstrainutex1709))、gthdnab(蓝隐藻(guillardiatheta))、eniprp8(构巢裸壳孢菌r20(emericellanidulansr20))、eni-fcsga4prp8(构巢裸壳孢菌fgsca4)、hav01pol(赤潮异弯藻病毒01(heterosigmaakashiwovirus01))、hcaprp8(荚膜组织胞浆菌(histoplasmacapsulatum))、iiv6rir1(无脊椎动物虹彩病毒6(invertebrateiridescentvirus6))、kex-cbs379vma(夜蛾酿酒酵母cbs379菌株(kazachstaniaexiguastraincbs379))、kla-cbs683vma(乳酸克鲁维酵母cbs683菌株(kluyveromyceslactisstraincbs683))、kla-ifo1267vma(乳酸克鲁维酵母ifo1267)、kla-nrrly1140vma(乳酸克鲁维酵母nrrly-1140)、lelvma(洛德酵母(lodderomyceselongisporus))、nauprp8(鲜橙新萨托菌nrrl4378(neosartoryaauratanrrl4378))、mca-cbs113480prp8(犬小孢子菌cbs113480(microsporumcaniscbs113480))、nfiprp8(费氏新萨托菌(neosartoryafischeri))、nfe-nrrl5534prp8(芬尼新萨托菌nrrl5534(neosartoryafennelliaenrrl5534))、ngl-frr1833prp8(光滑新萨托菌frr1833(neosartoryaglabrafrr1833))、ngl-fr2163prp8(光滑新萨托菌frr2163)、nquprp8(四绕新萨托菌nrrl4175菌株(neosartoryaquadricinctastrainnrrl4175))、nspiprp8(刺孢新萨托菌frr4595(neosartoryaspinosafrr4595))、pabr-pb01prp8(巴西副球孢子菌pb01(paracoccidioidesbrasiliensispb01))、pabr-pb03prp8(巴西副球孢子菌pb03)、panglt1(柄孢霉(podosporaanserina))、panchs2(柄孢霉)、pchprp8(产黄青霉(penicilliumchrysogenum))、pblprp8-a(布拉克须霉(phycomycesblakesleeanus))、pbr-pb18prp8(巴西副球孢子菌pb18)、pblprp8-b(布拉克须霉)、pexprp8(扩展青霉(penicilliumexpansum))、pguglt1(季也蒙毕赤酵母(pichiaguilliermondii))、pnoglt1(颖枯壳针孢sn15(phaeosphaerianodorumsn15))、pgu-altglt1(季也蒙毕赤酵母)、pstvma(树干毕赤酵母cbs6054(pichiastipitiscbs6054))、pnorpa2(颖枯壳针孢sn15)、ppudnab(紫红紫菜(porphyrapurpurea))、ptrprp8(小麦黄斑病真菌pt-1c-bf(pyrenophoratritici-repentispt-1c-bf))、pvuprp8(狐粪青霉(penicilliumvulpinum))、pyednab(条斑紫菜(porphyrayezoensis))、sca-cbs4309vma(芽殖酵母cbs4309菌株(saccharomycescastelliistraincbs4309))、sasrpb2(旋毛接合菌nrrl22631(spiromycesaspiralisnrrl22631))、scevma、vma(酿酒酵母(saccharomycescerevisiae))、sca-ifo1992vma(芽殖酵母ifo1992菌株)、sce-dh1-1avma(酿酒酵母dh1-1a菌株)、scarvma(卡里奥卡利斯酵母ufrj50791菌株(saccharomycescariocanusstrainufrj50791))、sce-jay291vma(酿酒酵母jay291)、sce-yjm789vma(酿酒酵母yjm789菌株)、sce-out7091vma(酿酒酵母out7091)、sce-out7112vma(酿酒酵母out7112)、sjavma(日本裂殖酵母yfs275(schizosaccharomycesjaponicusyfs275))、sex-ifo1128vma(少孢酵母ifo1128菌株(saccharomycesexiguusstrainifo1128))、sherpb2(淡黄毛枝藻utex441菌株(stigeocloniumhelveticumstrainutex441))、sdavma(黛伦恩斯酵母cbs421菌株(saccharomycesdairenensisstraincbs421))、spavma(巴氏酵母ifo11023(saccharomycespastorianusifo11023))、spuprp8(斑点小壶菌(spizellomycespunctatus))、sunvma(单孢酿酒酵母cbs398菌株(saccharomycesunisporusstraincbs398))、tglvma(球有孢圆酵母cbs764菌株(torulasporaglobosastraincbs764))、tprvma(孢圆酵母cbs5080菌株(torulasporapretoriensisstraincbs5080))、ure-1704prp8(非致病性真菌(uncinocarpusreesii))、vpovma(多孢子范氏酵母cbs2163菌株(vanderwaltozymapolysporastraincbs2163))、wivrir1(蝙蝠蛾虹彩病毒(wiseanairidescentvirus))、zrovma(鲁氏酵母cbs688菌株(zygosaccharomycesrouxiistraincbs688))、zbivma(二孢接合酵母cbs702菌株(zygosaccharomycesbisporusstraincbs702))、zbavma(拜氏接合酵母cbs685菌株(zygosaccharomycesbailiistraincbs685))、ap-apse1dpol(豌豆长管蚜次生内共生噬菌体1(acyrthosiphonpisumsecondaryendosymbiotphage1))、ap-apse2dpol(噬菌体apse-2(bacteriophageapse-2))、ap-apse4dpol(暂定种汉氏防卫共生菌5atac菌株噬菌体(candidatushamiltonelladefensastrain5atacbacteriophage))、ap-apse5dpol(噬菌体apse-5)、ap-aaphi23mupf(噬菌体aaphi23)、aaerir2(嗜热菌vf5菌株(aquifexaeolicusstrainvf5))、aave-aac001rir1(瓜类细菌性果斑病菌aac00-1(acidovoraxavenaesubsp.citrulliaac00-1))、aave-aac001aave1721(燕麦细菌性果斑病菌aac00-1)、aave-atcc19860rir1(细菌性果斑病菌atcc19860(acidovoraxavenaesubsp.avenaeatcc19860))、abahyp-02185(鲍氏不动杆菌acicu(acinetobacterbaumanniiacicu))、acerir1(解纤维热酸菌11b(acidothermuscellulolyticus11b))、aehdnab-1(厄氏碱湖生菌mlhe-1(alkalilimnicolaehrlicheimlhe-1))、aehdnab-2(厄氏碱湖生菌mlhe-1)、aehrir1(厄氏碱湖生菌mlhe-1)、mupfmupf(凝聚杆菌噬菌体s1249(aggregatibacterphages1249))、ahadnae-c(嗜盐隐杆藻(aphanothecehalophytica))、ahadnae-n(嗜盐隐杆藻)、alvi-dsm180gyra(光合细菌dsm180(allochromatiumvinosumdsm180))、amamade823(麦氏交替单胞菌(alteromonasmacleodii))、amax-cs328dnax(极大节旋藻cs-328(arthrospiramaximacs-328))、aovdnae-c(卵孢束丝藻(aphanizomenonovalisporum))、aovdnae-n(卵孢束丝藻)、apl-c1dnax(钝顶螺旋藻(arthrospiraplatensis))、aspdnae-c(鱼腥藻pcc7120(anabaenaspeciespcc7120))、arsp-fb24dnab(节杆菌fb24(arthrobacterspeciesfb24))、aspdnae-n(鱼腥藻pcc7120)、avadnae-c(多变鱼腥藻atcc29413(anabaenavariabilisatcc29413))、avinrir1bil(棕色固氮菌(azotobactervinelandii))、avadnae-n(多变鱼腥藻atcc29413)、bce-mco3dnab(伯克霍尔德氏菌mc0-3(burkholderiacenocepaciamc0-3))、bce-pc184dnab(伯克霍尔德氏菌pc184)、bse-mls10tera(还原硒酸盐芽胞杆菌mls10(bacillusselenitireducensmls10))、bsup-m1918rir1(枯草芽孢杆菌m1918前噬菌体(b.subtilism1918prophage))、bsup-spbc2rir1(枯草芽孢杆菌菌株168spβc2前噬菌体)、bcep1808_7358(越南伯克氏菌g4(burkholderiavietnamiensisg4))、cp-p1201thy1(棒状杆菌噬菌体p1201(corynebacteriumphagep1201))、cagrir1(混合菌闭染绿菌(chlorochromatiumaggregatum))、causpovr(嗜热光合绿丝菌j-10-fl(chloroflexusaurantiacusj-10-fl))、cbp-c-strnr(肉毒杆菌噬菌体c-st(clostridiumbotulinumphagec-st))、cbp-d1873rnr(肉毒杆菌噬菌体d)、cbu-dugwaydnab(伯纳特氏立克次氏体达格威5j108-111(coxiellaburnetiidugway5j108-111))、cbu--goatdnab(伯纳特氏立克次氏体msu山羊q177(coxiellaburnetiimsugoatq177))、cbu-rsa334dnab(伯纳特氏立克次氏体rsa334)、cbu-rsa493dnab(伯纳特氏立克次氏体rsa493)、ccehyp1-csp-2(蓝杆藻atcc51142(cyanothecesp.atcc51142))、cchrir1(氯铬绿菌cad3(chlorobiumchlorochromatiicad3))、ccyhyp1-csp-1(蓝杆藻ccy0110)、ccyhyp1-csp-2(蓝杆藻ccy0110)、cfl-dsm20109dnab(产黄纤维单胞菌dsm20109(cellulomonasflavigenadsm20109))、chyrir1(羧基嗜热产氢菌z-2901(carboxydothermushydrogenoformansz-2901))、cklpterm(梭状芽孢杆菌dsm555(clostridiumkluyveridsm555))、cra-cs505dnae-c(柱孢藻cs-505(cylindrospermopsisraciborskiics-505))、cra-cs505dnae-n(柱孢藻cs-505)、cra-cs505gyrb(柱孢藻cs-505)、csp-ccy0110dnae-c(蓝杆藻ccy0110)、csp-ccy0110dnae-n(蓝杆藻ccy0110)、csp-pcc7424dnae-c(蓝杆藻pcc7424)、csp-pcc7424dnae-n(蓝杆藻pcc7424)、csp-pcc7425dnab(蓝杆藻pcc7425)、csp-pcc7822dnae-n(蓝杆藻pcc7822)、csp-pcc8801dnae-c(蓝杆藻pcc8801)、csp-pcc8801dnae-n(蓝杆藻pcc8801)、cthatpasebil(热纤维梭菌(clostridiumthermocellum))、cth-atcc27405tera(热纤维梭菌atcc27405)、cth-dsm2360tera(热纤维梭菌dsm2360)、cwadnab(单细胞集胞藻wh8501(crocosphaerawatsoniiwh8501))、cwadnae-c(单细胞集胞藻wh8501)、cwadnae-n(单细胞集胞藻wh8501)、cwapep(单细胞集胞藻wh8501)、cwarir1(单细胞集胞藻wh8501)、daudrir1(暂定种金矿菌mp104c(candidatusdesulforudisaudaxviatormp104c))、dgednab(中度嗜热菌dsm11300(deinococcusgeothermalisdsm11300))、dha-dcb2rir1(脱卤脱亚硫酸菌dcb-2(desulfitobacteriumhafniensedcb-2))、dha-y51rir1(脱卤脱亚硫酸菌y51)、dpr-mlms1rir1(δ变形菌mlms-1(deltaproteobacteriummlms-1))、drarir1(耐辐射奇球菌r1tigr菌株(deinococcusradioduransr1tigrstrain))、drasnf2-c(耐辐射奇球菌r1tigr菌株)、snf2-nn-term(耐辐射奇球菌r1tigr菌株)、dra-atcc13939snf2(耐辐射奇球菌r1atcc13939布鲁克斯&穆雷菌株(brooks&murraystrain))、udpgd(嗜热网球菌h-6-12(dictyoglomusthermophilumh-6-12))、dvulparb(脱硫弧菌寻常亚种dp4(desulfovibriovulgarissubsp.vulgarisdp4))、ep-min27primase(肠道菌噬菌体min27(enterobacteriaphagemin27))、faldnab(弗兰克氏菌acn14a(frankiaalniacn14a))、fsp-cci3rir1(弗兰克氏种cci3(frankiaspeciescci3))、gobdnae(隐球出芽菌uqm2246(gemmataobscuriglobusuqm2246))、gobhyp(隐球出芽菌uqm2246))、gvidnab(紫色粘杆菌pcc7421(gloeobacterviolaceuspcc7421))、gvirir1-2(紫色粘杆菌pcc7421))、gvirir1-1(紫色粘杆菌pcc7421))、hhaldnab(嗜盐嗜盐红螺菌sl1(halorhodospirahalophilasl1))、kfl-dsm17836dnab(弗拉维达韩国生工菌dsm17836(kribbellaflavidadsm17836))、kradnab(耐辐照动球菌srs30216(kineococcusradiotoleranssrs30216))、llp-ksy1pola(乳球菌噬菌体ksy1(lactococcusphageksy1))、lp-phihsichelicase(利斯顿吕讷噬菌体φhsic(listonellapelagiaphagephihsic))、lsp-pcc8106gyrb(鞘丝藻pcc8106(lyngbyasp.pcc8106))、mp-bednab(伯利恒分枝杆菌噬菌体(mycobacteriophagebethlehem))、mp-begp51(伯利恒分枝杆菌噬菌体))、mp-cateragp206(凯帝分枝杆菌噬菌体(mycobacteriophagecatera))、mp-kbggp53(分枝杆菌噬菌体kbg(mycobacteriumphagekbg))、mp-omegadnab(ω分枝杆菌噬菌体(mycobacteriophageomega))、mp-mcjw1dnab(分枝杆菌噬菌体cjw1))、gp50(分枝杆菌噬菌体u2))、maer-nies843dnab(铜绿微囊藻nies-843(microcystisaeruginosanies-843))、maer-nies843dnae-c(铜绿微囊藻nies-843))、maer-nies843dnae-n(铜绿微囊藻nies-843))、mau-atcc27029gyra(桔橙小单孢菌atcc27029(micromonosporaaurantiacaatcc27029))、mav-104dnab(鸟分枝杆菌104(mycobacteriumavium104))、mav-atcc25291dnab(鸟分枝杆菌亚种atcc25291(mycobacteriumaviumsubsp.aviumatcc25291))、mav-atcc35712dnab(鸟分枝杆菌))、mav-ptdnab(鸟分枝杆菌亚种副结核病k10菌株(mycobacteriumaviumsubsp.paratuberculosisstr.k10))、mbopps1(牛分枝杆菌亚种af2122/97(mycobacteriumbovissubsp.bovisaf2122/97))、mboreca(牛分枝杆菌亚种af2122/97))、mbopps1(牛分枝杆菌亚种af2122/97))、mbo-af2122dnab(牛分枝杆菌亚种af2122/97))、mbo-1173pdnab(牛分枝杆菌bcg巴斯德1173p(mycobacteriumbovisbcgpasteur1173p))、mcamupf(甲基球菌属荚膜巴斯原噬菌体mumc02(methylococcuscapsulatusbathprophagemumc02))、mcarir1(巴斯甲基球菌(methylococcuscapsulatusbath))、mchreca(千田分枝杆菌(mycobacteriumchitae))、mcht-pcc7420dnae-1(土质体微鞘藻pcc7420(microcoleuschthonoplastespcc7420))、mcht-pcc7420dnae-2c(土质体微鞘藻pcc7420))、mcht-pcc7420dnae-2n(土质体微鞘藻pcc7420))、mcht-pcc7420gyrb(土质体微鞘藻pcc7420))、mcht-pcc7420rir1-1(土质体微鞘藻pcc7420))、mcht-pcc7420rir1-2(土质体微鞘藻pcc7420))、mexhelicase(外链甲基杆菌am1(methylobacteriumextorquensam1))、mextrbc(外链甲基杆菌am1))、mfareca(诡诈分枝杆菌(mycobacteriumfallax))、mflgyra(微黄分枝杆菌fla0(mycobacteriumflavescensfla0))、mflreca(微黄分枝杆菌fla0))、mfl-atcc14474reca(微黄分枝杆菌atcc14474))、mfl-pyr-gckdnab(微黄分枝杆菌pyr-gck))、mgagyra(胃分枝杆菌(mycobacteriumgastri))、mgareca(胃分枝杆菌))、mgapps1(胃分枝杆菌))、mgi-pyr-gckdnab(浅黄分枝杆菌pyr-gck(mycobacteriumgilvumpyr-gck))、mgi-pyr-gckgyra(浅黄分枝杆菌pyr-gck))、mgogyra(戈登分枝杆菌(mycobacteriumgordonae))、min-1442dnab(胞内分枝杆菌(mycobacteriumintracellulare))、min-atcc13950gyra(胞内分枝杆菌atcc13950))、mkasgyra(肯沙士分枝杆菌(mycobacteriumkansasii))、mkas-atcc12478gyra(肯沙士分枝杆菌atcc12478))、mle-br4923gyra(麻疯分枝杆菌br4923(mycobacteriumlepraebr4923))、mle-tndnab(麻疯分枝杆菌tn菌株))、mle-tngyra(麻疯分枝杆菌tn))、mlepps1(麻疯分枝杆菌))、mle-tnreca(麻疯分枝杆菌tn菌株))、mmagyra(玛尔摩分枝杆菌(mycobacteriummalmoense))、mmagmagn8951bil(向磁磁螺菌ms-1(magnetospirillummagnetotacticumms-1))、mshreca(西莫得分枝杆菌(mycobacteriumshimodei))、msmdnab-1(包皮垢分枝杆菌mc2155(mycobacteriumsmegmatismc2155))、msmdnab-2(包皮垢分枝杆菌mc2155))、msp-kmsdnab(分枝杆菌kms(mycobacteriumspecieskms))、msp_kmsgyra(分枝杆菌kms))、msp-mcsdnab(分枝杆菌mcs))、msp_mcsgyra(分枝杆菌mcs))、mthereca(耐高温分枝杆菌(mycobacteriumthermoresistibile))、mtupps1(结核分枝杆菌h37rv菌株(mycobacteriumtuberculosisstrainh37rv))、mtu-cdc1551dnab(结核分枝杆菌cdc1551))、mtu-creca(结核分枝杆菌c))、mtu-cphlreca(结核分枝杆菌cphl_a))、mtu-eas054reca(结核分枝杆菌eas054))、mtu-canettireca(结核分枝杆菌卡内蒂菌株(mycobacteriumtuberculosisstraincanetti))、mtu-f11dnab(结核分枝杆菌f11菌株))、mtu-h37radnab(结核分枝杆菌h37ra))、mtu-h37rvdnab(结核分枝杆菌h37rv))、mtu-h37rvreca(结核分枝杆菌h37rv和cdc1551))、mtu-haarlemdnab(结核分枝杆菌哈勒姆菌株(mycobacteriumtuberculosisstr.haarlem))、mtu-r604reca-n(结核分枝杆菌98-r604inh-rif-em))、mtu-k85reca(结核分枝杆菌k85))(mtu-so93reca(结核分枝杆菌so93/卡内蒂亚种))、mtu-t17reca-c(结核分枝杆菌t17))、mtu-t17reca-n(结核分枝杆菌t17))、mtu-t46reca(结核分枝杆菌t46))、mtu-t85reca(结核分枝杆菌t85))、mvandnab(范巴莱尼分枝杆菌pyr-1(mycobacteriumvanbaaleniipyr-1))、mtu-t92reca(结核分枝杆菌t92))、mvangyra(范巴莱尼分枝杆菌pyr-1))、mxarad25(黄色粘球菌dk1622(myxococcusxanthusdk1622))、mxegyra(蟾蜍分枝杆菌imm5024菌株(mycobacteriumxenopistrainimm5024))、naz-0708rir1-2(念珠藻鱼腥藻0708(nostocazollae0708))、naz-0708rir1-1(念珠藻鱼腥藻0708))、nfadnab(皮疽诺卡氏菌ifm10152(nocardiafarcinicaifm10152))、nfanfa15250(皮疽诺卡氏菌ifm10152))、nfarir1(皮疽诺卡氏菌ifm10152))、nosp-ccy9414dnae-n(产泡沫节球藻ccy9414(nodulariaspumigenaccy9414))、npudnab(点型念珠藻(nostocpunctiforme))、npugyrb(点型念珠藻))、npu-pcc73102dnae-c(点型念珠藻pcc73102))、npu-pcc73102dnae-n(点型念珠藻pcc73102))、nsp-js614dnab(诺卡氏菌js614(nocardioidesspeciesjs614))、nsp-js614toprim(诺卡氏菌js614))、nsp-pcc7120dnab(念珠藻pcc7120(nostocspeciespcc7120))、nsp-pcc7120dnae-c(念珠藻pcc7120))、nsp-pcc7120dnae-n(念珠藻pcc7120))、nsp-pcc7120rir1(念珠藻pcc7120))、olidnae-c(沼泽颤藻太阳湖菌株(oscillatorialimneticastr.solarlake))、olidnae-n(沼泽颤藻太阳湖菌株))、pp-phielhelicase(铜绿假单胞菌噬菌体(pseudomonasaeruginosaphagephiel))、pp-phielorf11(铜绿假单胞菌噬菌体))、pp-phielorf40(铜绿假单胞菌噬菌体))、pp-phielorf39(铜绿假单胞菌噬菌体))、pflfhabil(萤光假单胞菌pf-5(pseudomonasfluorescenspf-5))、pma-exh1dnae(玛丽娜佩斯菲尼拉菌ex-h1(persephonellamarinaex-h1))、plutrir1(微黄暗网菌dsm273(pelodictyonluteolumdsm273))、pma-exh1gyra(玛丽娜佩斯菲尼拉菌ex-h1))、pnarir1(极地单胞菌cj2(polaromonasnaphthalenivoranscj2))、posp-js666dnab(极地单胞菌种js666))、puncdnab(多核杆菌qlw-p1dmwa-1(polynucleobactersp.qlw-p1dmwa-1))、posp-js666rir1(极地单胞菌种js666))、pssp-a1-1fha(假单胞菌a1-1(pseudomonasspeciesa1-1))、psyfha(丁香番茄假单胞菌dc3000菌株(pseudomonassyringaepv.tomatostr.dc3000))、rbr-d9gyrb(布鲁克尖头藻d9(raphidiopsisbrookiid9))、rcerir1(森特讷姆红螺菌sw(rhodospirillumcentenumsw))、rer-sk121dnab(红串红球菌sk121(rhodococcuserythropolissk121))、rmadnab(海洋红嗜热盐菌(rhodothermusmarinus))、rma-dsm4252dnae(海洋红嗜热盐菌dsm4252))、rma-dsm4252dnab(海洋红嗜热盐菌dsm4252))、rsprir1(玫瑰变色菌217(roseovariusspecies217))、sap-setp12dpol(沙门氏菌噬菌体setp12(salmonellaphagesetp12))、sap-setp3helicase(沙门氏菌噬菌体setp3))、sap-setp3dpol(沙门氏菌噬菌体setp3))、sap-setp5dpol(沙门氏菌噬菌体setp5))、sarednab(沙蠋盐生孢菌cns-205(salinisporaarenicolacns-205))、reghelicase(阿维链霉菌ma-4680(streptomycesavermitilisma-4680))、sel-pc6301rir1(细长聚球藻pcc6301(synechococcuselongatuspcc6301))、sel-pc7942dnae-c(细长聚球藻pc7942))、sel-pc7942rir1(细长聚球藻pc7942))、sel-pc7942dnae-n(细长聚球藻pc7942))、sel-pcc6301dnae-n(细长聚球藻pcc6301))、sel-pcc6301dnae-c(细长聚球藻pcc6301和pcc7942))、shp-sfv-2a-2457t-nprimase(2a福氏志贺菌2457t菌株(shigellaflexneri2astr.2457t))、seprir1(表皮葡萄球菌rp62a(staphylococcusepidermidisrp62a))、shp-sfv-2a-301primase(2a福氏志贺菌301菌株))、shp-sfv-5primase(5福氏志贺菌8401菌株))、sop-so1dpol(伴突属噬菌体so-1(sodalisphageso-1))、srudnab(鲁波尔极端嗜盐细菌dsm13855(salinibacterruberdsm13855))、spldnax(螺旋藻c1菌株(spirulinaplatensisstrainc1))、srupolbc(鲁波尔极端嗜盐细菌dsm13855))、srurir1(鲁波尔极端嗜盐细菌dsm13855))、sspdnab(集胞藻pcc6803菌株(synechocystisspeciesstrainpcc6803))、sspdnae-n,dnae-n(集胞藻pcc6803菌株))、sspdnae-c,dnae-c(集胞藻pcc6803菌株))、sspdnax(集胞藻pcc6803菌株))、ssp-ja2rir1(聚球藻ja-2-3ba2-13(synechococcusspeciesja-2-3ba2-13))、ssp-ja2dnab(聚球藻ja-2-3ba2-13))、sspgyrb(集胞藻pcc6803菌株))、ssp-ja3dnab(聚球藻ja-3-3ab))、ssp-ja3rir1(聚球藻ja-3-3ab))、ssp-pcc7002dnae-c(集胞藻pcc7002菌株))、ssp-pcc7002dnae-n(集胞藻pcc7002菌株))、ssp-pcc7335rir1(聚球藻pcc7335))、stp-twortorf6(twort葡萄球菌噬菌体(staphylococcusphagetwort))、susp-nbc371dnab(硫氧化单胞菌nbc37-1(sulfurovumsp.nbc37-1))、taq-y51mc23dnae(水生栖热菌y51mc23(thermusaquaticusy51mc23))、teldnae-c(耐热细长聚球藻bp-1(thermosynechococcuselongatusbp-1))、tcu-dsm43183reca(弯曲嗜热单孢菌dsm43183(thermomonosporacurvatadsm43183))、teldnae-n(耐热细长聚球藻bp-1))、taq-y51mc23rir1(水生栖热菌y51mc23))、terdnab-1(红海束毛藻ims101(trichodesmiumerythraeumims101))、terdnab-2(红海束毛藻ims101))、terdnae-2(红海束毛藻ims101))、terdnae-1(红海束毛藻ims101))、terdnae-3c(红海束毛藻ims101))、terdnae-3n(红海束毛藻ims101))、tergyrb(红海束毛藻ims101))、terndse-1(红海束毛藻ims101))、terndse-2(红海束毛藻ims101))、terrir-1(红海束毛藻ims101))、terrir-2(红海束毛藻ims101))、terrir-3(红海束毛藻ims101))、terrir-4(红海束毛藻ims101))、tersnf2(红海束毛藻ims101))、terthyx(红海束毛藻ims101))、tfusreca-1(褐色嗜热菌yx(thermobifidafuscayx))、tfusreca-2(褐色嗜热菌yx))、tfustfu2914(褐色嗜热菌yx))、thsp-k90rir1(嗜盐嗜碱硫氧化菌k90mix(thioalkalivibriosp.k90mix))、tth-dsm571rir1(嗜热无氧细菌短杆菌dsm571(thermoanaerobacteriumthermosaccharolyticumdsm571))、tth-hb27dnae-1,tth(嗜热栖热菌hb27(thermusthermophilushb27))、tth-hb27dnae-2(嗜热栖热菌hb27))、tth-hb27rir1-1(嗜热栖热菌hb27))、tth-hb27rir1-2(嗜热栖热菌hb27))、tth-hb8dnae-1(嗜热栖热菌hb8))、tth-hb8dnae-2(嗜热栖热菌hb8))、tth-hb8rir1-1(嗜热栖热菌hb8))、tth-hb8rir1-2(嗜热栖热菌hb8))、tvudnae-c(嗜热聚球藻(thermosynechococcusvulcanus))、tvudnae-n(嗜热聚球藻))、tyernr-1(黄石嗜热脱硫弧菌dsm11347(thermodesulfovibrioyellowstoniidsm11347))、tyernr-2(黄石嗜热脱硫弧菌dsm11347))、apeape0745(泉生古细菌k1(aeropyrumpernixk1))、cme-boopol-ii(暂定种伯尼嗜酸产甲烷菌6a8(candidatusmethanoregulaboonei6a8))、fac-fer1rir1(嗜酸铁原体分类群97393(ferroplasmaacidarmanustaxon:97393))、facpps1(嗜酸铁原体))、fac-typeirir1(嗜酸铁原体i型))、facpps1(嗜酸铁原体))、hmacdc21(死海盐杆菌atcc43049(haloarculamarismortuiatcc43049))、hmapol-ii(死海盐杆菌atcc43049))、hmapolb(死海盐杆菌atcc43049))、hmatopa(死海盐杆菌atcc43049))、hmu-dsm12286mcm(磨蔻哈迪盐微菌dsm12286(halomicrobiummukohataeidsm12286))、hmu-dsm12286polb(磨蔻哈迪盐微菌dsm12286))、hsa-r1mcm(盐沼盐杆菌r-1(halobacteriumsalinarumr-1))、hsp-nrc1cdc21(嗜盐菌nrc-1(halobacteriumspeciesnrc-1))、hsp-nrc1pol-ii(盐沼盐杆菌nrc-1))、hutmcm-2(犹他盐杆菌dsm12940(halorhabdusutahensisdsm12940))、hutmcm-1(犹他盐杆菌dsm12940))、hwagyrb(沃思碧嗜盐古菌dsm16790(haloquadratumwalsbyidsm16790))、hvopolb(沃氏嗜盐富饶菌ds70(haloferaxvolcaniids70))、hwamcm-1(沃思碧嗜盐古菌dsm16790))、hwamcm-2(沃思碧嗜盐古菌dsm16790))、hwamcm-3(沃思碧嗜盐古菌dsm16790))、hwamcm-4(沃思碧嗜盐古菌dsm16790))、hwapol-ii-1(沃思碧嗜盐古菌dsm16790))、hwapol-ii-2(沃思碧嗜盐古菌dsm16790))、hwapolb-1(沃思碧嗜盐古菌dsm16790))、hwapolb-2(沃思碧嗜盐古菌dsm16790))、hwapolb-3(沃思碧嗜盐古菌dsm16790))、hwarcf(沃思碧嗜盐古菌dsm16790))、hwarir1-1(沃思碧嗜盐古菌dsm16790))、hwarir1-2(沃思碧嗜盐古菌dsm16790))、hwatop6b(沃思碧嗜盐古菌dsm16790))、rpola”(沃思碧嗜盐古菌dsm16790))、maeopol-ii(甲烷超嗜热菌nankai-3(methanococcusaeolicusnankai-3))、maeorfc(甲烷超嗜热菌nankai-3))、maeornr(甲烷超嗜热菌nankai-3))、maeo-n3helicase(甲烷超嗜热菌nankai-3))、udpgd(甲烷超嗜热菌nankai-3))、maeo-n3rtcb(甲烷超嗜热菌nankai-3))、mein-mepep(印发纳斯甲基白豆汤球菌me(methanocaldococcusinfernusme))、mein-merfc(印发纳斯甲基白豆汤球菌me))、memarmcm2(黑海甲烷袋状菌jr1(methanoculleusmarisnigrijr1))、memarpol-ii(黑海甲烷袋状菌jr1))、mesp-fs406polb-1(甲基白豆汤球菌fs406-22(methanocaldococcussp.fs406-22))、mesp-fs406polb-2(甲基白豆汤球菌fs406-22))、mesp-fs406polb-3(甲基白豆汤球菌fs406-22))、msp-fs406-22lhr(甲基白豆汤球菌fs406-22))、mfe-ag86pol-1(发光甲基白豆汤球菌ag86(methanocaldococcusfervensag86))、mfe-ag86pol-2(发光甲基白豆汤球菌ag86))、mhupol-ii(亨氏甲基螺旋菌jf-1(methanospirillumhungateiijf-1))、mjagf-6p(詹氏甲烷球菌(methanococcusjannaschii))、mjahelicase(詹氏甲烷球菌))、mjahyp-1(詹氏甲烷球菌))、mjaif2(詹氏甲烷球菌))、mjaklba(詹氏甲烷球菌))、mjapep(詹氏甲烷球菌))、mjapol-1(詹氏甲烷球菌))、mjapol-2(詹氏甲烷球菌))、mjarfc-1(詹氏甲烷球菌))、mjarfc-2(詹氏甲烷球菌))、mjarfc-3(詹氏甲烷球菌))、mjarnr-1(詹氏甲烷球菌))、mjarnr-2(詹氏甲烷球菌))、mjahyp-2(詹氏甲烷球菌))、mjatfiib(詹氏甲烷球菌))、udpgd(詹氏甲烷球菌))、mjar-gyr(詹氏甲烷球菌))、rpola'(詹氏甲烷球菌))、mjarpola’(詹氏甲烷球菌))、mkacdc48(坎德勒氏甲烷嗜热菌av19(methanopyruskandleriav19))、mkaef2(坎德勒氏甲烷嗜热菌av19))、mkarfc(坎德勒氏甲烷嗜热菌av19))、mkartcb(坎德勒氏甲烷嗜热菌av19))、mkavatb(坎德勒氏甲烷嗜热菌av19))、mthrir1(热自养甲基嗜热杆菌(methanothermobacterthermautotrophicus))、mvu-m7helicase(火山甲基白豆汤球菌m7(methanocaldococcusvulcaniusm7))、mvu-m7pol-1(火山甲基白豆汤球菌m7))、mvu-m7pol-2(火山甲基白豆汤球菌m7))、mvu-m7pol-3(火山甲基白豆汤球菌m7))、udpgd(火山甲基白豆汤球菌m7))、neqpol-c(泉口骑行纳古菌kin4-m(nanoarchaeumequitanskin4-m))、neqpol-n(泉口骑行纳古菌kin4-m))、nma-atcc43099mcm(马氏钠白菌atcc43099(natrialbamagadiiatcc43099))、nma-atcc43099polb-1(马氏钠白菌atcc43099))、nma-atcc43099polb-2(马氏钠白菌atcc43099))、nphcdc21(盐碱单孢菌dsm2160(natronomonaspharaonisdsm2160))、nphpolb-2(盐碱单孢菌dsm2160))、nphpolb-1(盐碱单孢菌dsm2160))、rpola”(盐碱单孢菌dsm2160))、pabcdc21-1(阿氏热球菌(pyrococcusabyssi))、pabcdc21-2(阿氏热球菌))、pabif2(阿氏热球菌))、pabklba(阿氏热球菌))、pablon(阿氏热球菌))、pabmoaa(阿氏热球菌))、pabpol-ii(阿氏热球菌))、pabrfc-1(阿氏热球菌))、pabrfc-2(阿氏热球菌))、pabrir1-1(阿氏热球菌))、pabrir1-2(阿氏热球菌))、pabrir1-3(阿氏热球菌))、pabhyp-2(阿氏热球菌))、pabvma(阿氏热球菌))、parrir1(砷化热棒菌dsm13514(pyrobaculumarsenaticumdsm13514))、pfucdc21(激烈热球菌(pyrococcusfuriosus))、pfuif2(激烈热球菌))、pfuklba(激烈热球菌))、pfulon(激烈热球菌))、pfurfc(激烈热球菌))、pfurir1-1(激烈热球菌))、pfurir1-2(激烈热球菌))、pfuhyp-2(激烈热球菌))、pfutopa(激烈热球菌))、pfuvma(激烈热球菌))、phocdc21-1(霍氏热球菌ot3(pyrococcushorikoshiiot3))、phocdc21-2(霍氏热球菌ot3))、phoif2(霍氏热球菌ot3))、phoklba(霍氏热球菌ot3))、pholhr(霍氏热球菌ot3))、pholon(霍氏热球菌ot3))、poli(霍氏热球菌ot3))、phopol-ii(霍氏热球菌ot3))、phorfc(霍氏热球菌ot3))、phorir1(霍氏热球菌ot3))、phorada(霍氏热球菌ot3))、phovma(霍氏热球菌ot3))、phohyp-2(霍氏热球菌ot3))、phor-gyr(霍氏热球菌ot3))、psp-gbdpol(热球菌种gb-d(pyrococcusspeciesgb-d))、smar1471(海葡萄嗜热菌f1(staphylothermusmarinusf1))、ptovma(干热嗜酸古菌dsm9790(picrophilustorridusdsm9790))、tac-atcc25905vma(嗜酸热原体atcc25905(thermoplasmaacidophilumatcc25905))、smarmcm2(海葡萄嗜热菌f1))、tac-dsm1728vma(嗜酸热原体dsm1728))、tsp-typol-1(成团耐热球菌(thermococcusaggregans))、tsp-typol-2(成团耐热球菌))、tsp-typol-3(成团耐热球菌))、tbapol-ii(嗜压耐热球菌mp(thermococcusbarophilusmp))、tfupol-1(福氏耐热球菌(thermococcusfumicolans))、thypol-1(水热耐热球菌(thermococcushydrothermalis))、tfupol-2(福氏耐热球菌))、thypol-2(水热耐热球菌))、tkocdc21-1(幸田氏耐热球菌kod1(thermococcuskodakaraensiskod1))、tkocdc21-2(幸田氏耐热球菌kod1))、tkohelicase(幸田氏耐热球菌kod1))、tkoif2(幸田氏耐热球菌kod1))、tkoklba(幸田氏耐热球菌kod1))、tkolhr(幸田氏耐热球菌kod1))、psp-kodpol-1(幸田氏耐热球菌kod1))、kodpol-2(幸田氏耐热球菌kod1))、tkopol-ii(幸田氏耐热球菌kod1))、tkorir1-1(幸田氏耐热球菌kod1))、tkorfc(幸田氏耐热球菌kod1))、tkorir1-2(幸田氏耐热球菌kod1))、tkorada(幸田氏耐热球菌kod1))、tkotopa(幸田氏耐热球菌kod1))、tkor-gyr(幸田氏耐热球菌kod1))、tlipol-1(利托氏耐热球菌(thermococcuslitoralis))、tlipol-2(利托氏耐热球菌))、tmapol(马氏耐热球菌(thermococcusmarinus))、ton-na1lhr(奥纳锐斯耐热球菌na1(thermococcusonnurineusna1))、ton-na1pol(奥纳锐斯耐热球菌na1))、tpepol(嗜胨耐热球菌株sm2(thermococcuspeptonophilusstrainsm2))、tsi-mm739lon(芒耐热球菌mm739(thermococcussibiricusmm739))、tsi-mm739pol-1(芒耐热球菌mm739))、tsi-mm739pol-2(芒耐热球菌mm739))、tsi-mm739rfc(芒耐热球菌mm739))、am4rtcb(耐热球菌am4(thermococcussp.am4))(tsp-am4lhr(耐热球菌am4))、tsp-am4lon(耐热球菌am4))、tsp-am4rir1(耐热球菌am4))、tsp-ge8pol-2(耐热球菌ge8(thermococcusspeciesge8))、tsp-ge8pol-1(耐热球菌ge8))、tsp-gtpol-1(耐热球菌gt))、tsp-gtpol-2(耐热球菌gt))、tsp-ogl-p20pol(耐热球菌ogl-20p))、tthipol(嗜硫耐热球菌(thermococcusthioreducens))、tzipol(兹利氏耐热球菌(thermococcuszilligii))、tvovma(火山热原体gss1(thermoplasmavolcaniumgss1))、unc-erspfl(未培养的古细菌gzfos13e1(unculturedarchaeongzfos13e1))、unc-ersrir1(未培养的古细菌gzfos9c4))、unc-metrfsmcm2(未培养的古细菌赖斯集群i(unculturedarchaeonriceclusteri))或unc-ersrnr(未培养的古细菌gzfos10c7))中的一种。
内含肽名称提供了有关生物体和赋予蛋白同源物的蛋白名称的信息,该蛋白同源物在经过充分研究的生物体内携带内含肽。例如,在名称ade-er3prp8中,“ade-er3”是指生物体皮炎组织胞浆菌er-3并且prp8是赋予蛋白同源物的蛋白名称,该蛋白同源物在经过充分研究的生物体内携带内含肽。
内含肽可以具有在美国预授权专利申请公开号us2013-0007919(2013年1月3日公布的2012年9月20日提交的美国申请号13/508,156)、美国专利号2013-0036517(2013年2月7日公布的2012年9月20日提交的美国申请号13/508,234)和美国2013-0071884(2013年3月21日公布的2012年9月20日提交的美国申请号13/508,280)中的任意一个中公开的内含肽的序列,所有这些都通过引用并入本文,如同完整阐述。
内含肽可以是反式剪接的内含肽。反式剪接的内含肽可以是gp41-1、cbu_dnab、mja_gf-6p、mja_hyp-1、mja_if2、mja_pol-1、pab_cdc21-1、pab_if2-n、pab_vma、pho_if2、pho_vma、rma_dnab、sru_dnab、tag_pol-1_tsp-ty_pol-1、ter_rir1-4、tko_if2或tth-hb27_dnae-2内含肽。内含肽可以是gp41-1内含肽。gp41-1内含肽可以含有、基本上由、或由与seqidno:11具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。
反式剪接的内含肽可以包括两个部分。反式剪接的内含肽的一部分可以是n-末端内含肽或n-内含肽(in)。反式剪接的内含肽的另一部分可以是c-末端内含肽或c-内含肽(ic)。反式剪接的内含肽可以包括如下表i中所述的n-内含肽和c-内含肽的组合。与一个反式剪接的内含肽在同一行列出的n-内含肽和c-内含肽序列分别表示第一前体和第二前体中的n-内含肽和c-内含肽的序列。在表i的每个反式剪接的内含肽中,n-内含肽含有、基本上由、或由与表1中相应内含肽在同一行上鉴定的序列具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。类似地,在表i的每个反式剪接的内含肽中,c-内含肽可以含有、基本上由、或由与表1中相应内含肽在同一行上鉴定的序列具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。例如,在以a表示的反式剪接的内含肽中,n-内含肽可以与seqidno:14具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性,并且c-内含肽可以与seqidno:15具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性。
当表1中的每个n-内含肽和c-内含肽与指定的序列具有100%的同一性时,反式剪接的内含肽如下所示:内含肽a为gp41-1,内含肽b为cbu_dnab,内含肽c为mja_gf-6p,内含肽d为mja_hyp-1,内含肽e为mja_if2,内含肽f为mja_pol-1,内含肽g为pab_cdc21-1,内含肽h为pab_if2-n,内含肽i为pab_vma,内含肽j为ph0_if2,内含肽k为pho_vma,内含肽l为rma_dnab,内含肽m为sru_dnab,内含肽n为tag_pol-1_tsp-ty_pol-1,内含肽o为ter_rir1-4,内含肽p为tko_if2,和内含肽q为tth-hb27_dnae-2。在小于100%的同一性下,每个n-内含肽:c内含肽对形成gp41-1、cbu_dnab、mja_gf-6p、mja_hyp-1、mja_if2、mja_pol-1、pab_cdc21-1、pab_if2-n、pab_vma、iph0_if2、pho_vma、rma_dnab、sru_dnab、tag_pol-1_tsp-ty_pol-1、ter_rir1-4、tko_if2和tth-hb27_dnae-2内含肽各自的变体。变体保留了反式剪接的活性,其可以使用双顺反子表达盒进行测试以恢复实施例18中所述的蛋白酶活性。
如本文所用,术语iprotease是内含肽修饰的蛋白酶的替代名称。第一前体可以被称为“ni”,其可以是1)蛋白酶n-末端片段(n-外显肽)与2)反式剪接的内含肽的n-末端部分(in)的框架融合。第二前体可以被称为“ic”,其可以是1)反式剪接的内含肽的c-末端部分(ic)与2)蛋白酶的c-末端片段(c-外显肽)的框架融合。剪接产物可以具有通过肽键无缝连接的氨基外显肽(n)和羧基外显肽(c),且可被称为“nc”。nc可以是通过反式剪接获得的靶蛋白酶。如果n-外显肽包括前结构域(pre-domain),则剪接后的原结构域(pro-domain)的自动切割可以导致有活性的靶蛋白酶。“(ni+ic)预混物”是指处于无活性状态的剪接组分ni和ic的混合物,其可以不具有蛋白酶活性,但可以通过稀释诱导以剪接并产生完全活性的靶蛋白酶。
在一种实施方式中,ni和ic前体可以是原型分子isavinase:s317-gp41-1ni(seqidno:49)和ic(seqidno:50)的可溶性优化版本。在于2013年10月3日提交并作为国际公开号wo2014/055782的于2014年4月4日公布的pct申请pct/us2013/063304中描述了这些前体,其通过引用并入本文,如同完整阐述。
内含肽修饰的蛋白酶可以包括针对可溶性改善而优化的内含肽修饰的蛋白酶组分。优化的内含肽修饰的蛋白酶组分可以具有仅限于内含肽部分的优化,以确保当内含肽自身移除并通过肽键将侧翼蛋白序列无缝连接时,剪接保持靶酶的序列不变。剪接可以将完整的蛋白恢复至完全的蛋白酶活性。内含肽修饰的蛋白酶的第一前体可以含有、基本上由、或由与seqidno:1具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。内含肽修饰的蛋白酶的第二前体可以含有、基本上由、或由与seqidno:2具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。
图1显示了可溶性优化分子的示意图。参考图1,savinase可以在催化残基之间被分割为sav-n和sav-c。参考图1,催化残基可以是:d(asp143)、h(his172)和s(ser326)。这些残基的相对位置用垂直线标记。内含肽部分in和ic可以分别与savinase部分sav-n和sav-c连接。可溶性基因工程改造可以限于内含肽部分。可溶性增强子trx结构域可以通过dpng(asp-pro-asn-gly;seqidno:5)连接体与每个内含肽部分连接。c-内含肽部分(ic)可以具有多个突变以增强可溶性。这些可以是m89δ、l91t、l95t、l122g(根据在融合的in+ic中的位置进行编号)。
被称为ni-dpng-trx的可溶性优化的ni可以具有通过asp-pro-asn-gly(dpng;seqidno:5)连接体与iprotease:s317-gp41-1n中的gp41-1n-内含肽(in;seqidno:14)的c-末端融合的c-末端可溶性增强子硫氧还蛋白(thioredoxin)结构域(seqidno:51)。在被称为trx-dpng-mttic[g122l]的可溶性优化的ic中,硫氧还蛋白结构域(seqidno:5)通过asp-pro-asn-gly(dpng;seqidno:5)连接体n-末端与iprotease:s317-gp41-1c的突变的c-内含肽(ic;seqidno:15)融合。ni中的trx(被称为trx-1)在c-末端比ic中的trx(称为trx-2)长9个aa残基。突变的c-内含肽(seqidno:15),mtt-ic[l122g]可以在位置89、91、95和122处具有四个可溶性增强子突变,如下所示:m89δ、l91t、l95t和l122g(根据在gp41-1的融合的in+ic中的位置进行编号)。
在一种实施方式中,内含肽可以与靶蛋白酶在这样的位置融合,以明显降低或抑制靶蛋白酶的活性。换句话说,与完整的靶蛋白酶相比,包括在第一前体和第二前体中的蛋白酶的片段没有或具有明显降低的蛋白酶活性。明显降低的活性可以是完整的靶蛋白酶的活性的40%至35%、35%至30%、30%至25%、25%至20%、20%至15%、15%至10%、10%至5%、5%至1%、1%至0.5%,或任何两个前述之间的范围内的任何值(含端点)。经内含肽的剪接,可以恢复靶蛋白酶的活性。可以通过实施例2中概述的savinase酶试验分析活性。
一种实施方式包括一种组合物,该组合物含有本文的任何内含肽修饰的蛋白酶的前体(ni和ic)。第一前体ni可以含有、基本上由、或由与seqidno:1的参考序列具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。第一前体可以能够与第二前体进行反式剪接以形成靶蛋白酶。第二前体ic可以含有、基本上由、或由与seqidno:2的参考序列具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。第二前体可以能够与第一前体进行反式剪接以形成靶蛋白酶。第一前体或第二前体的浓度可以在0.01%(v:v)至10%(v:v)的范围内。浓度可以为0.02%(v:v)、0.03%(v:v)、0.04%(v:v)、0.05%(v:v)、0.06%(v:v)、0.07%(v:v)、0.08%(v:v)、0.09%(v:v)、0.1%(v:v)、0.2%(v:v)、0.3%(v:v)、0.4%(v:v)、0.5%(v:v)、0.6%(v:v)、0.7%(v:v)、0.8%(v:v)、1.0%(v:v)、2.0%(v:v)、3.0%(v:v)、4.0%(v:v)、5.0%(v:v)、6.0%(v:v)、7.0%(v:v)、8.0%(v:v)、9.0%(v:v)或10.0%(v:v),或任何两个前述浓度点之间的任何值。第一前体或第二前体的浓度可以在0.3%(v:v)至4%(v:v)的范围内。浓度可以为0.3%(v:v)、0.4%(v:v)、0.5%(v:v)、0.6%(v:v)、0.7%(v:v)、0.8%(v:v)、1.0%(v:v)、2.0%(v:v)、3.0%(v:v)或4.0%(v:v),或任何两个前述浓度点之间的任何值。第一前体或第二前体的浓度可以为至少0.3%(v:v)、至少0.4%(v:v)、至少0.5%(v:v)、至少0.6%(v:v)、至少0.7%(v:v)、至少0.8%(v:v)、至少1.0%(v:v)、至少2.0%(v:v)、至少3.0%(v:v)或至少4.0%(v:v),或至少任何两个前述浓度点之间的任何值。如上所用,浓度10%(v:v)对应于381.00μm的浓度,5%(v:v)对应于190.50μm的浓度,1%(v:v)对应于38.1μm的浓度,0.50%(v:v)对应于19.05μm,且0.1%(v:v)对应于3.81μm。
组合物可以进一步含有至少一种或多种效应物、家庭护理产品的组分或其他成分。一种或多种效应物可以包括一种或多种钠盐补充剂、钾盐补充剂、铵盐补充剂、带电聚合盐补充剂、多元醇补充剂、氯化钠、亚氨基二琥珀酸四钠(tetrasodiumiminodisuccinate)、琥珀酸二钠(disodiumsuccinate)、酒石酸二钠(disodiumtartrate)、乳酸钾、柠檬酸钾、氯化钾、烟酸钠、硫酸铵、硝酸铵、柠檬酸锂、聚天冬氨酸钠(sodiumpolyaspartate)、聚丙烯酸钠(sodiumpolyacrylate)、四乙二醇(tetraethyleneglycol)、聚乙二醇、四甘醇(tetraglycol)、碳酸丙烯酯(propylenecarbonate)、单丙二醇(monopropyleneglycol)、甘油和乙氧基化醇醚(tomadol)。氯化钾浓度可以在0.1m至5.0m的范围内。浓度可以为0.1m、0.2m、0.3m、0.4m、0.5m、0.6m、0.7m、0.8m、0.9m、1.0m、1.1m、1.2m、1.3m、1.4m、1.5m、1.6m、1.7m、1.8m、1.9m、2.0m、2.1m、2.2m、2.3m、2.4m、2.5m、2.6m、2.7m、2.8m、2.9m、3.0m、3.1m、3.2m、3.3m、3.4m、3.5m、3.6m、3.7m、3.8m、3.9m、4.0m、4.1m、4.2m、4.3m、4.4m、4.5m、4.6m、4.7m、4.8m、4.9、5.0m,或任何两个前述浓度点之间的任何值。氯化钾浓度可以在0.5m至2.0m的范围内。浓度可以为0.5m、0.6m、0.7m、0.8m、0.9m、1.0m、1.1m、1.2m、1.3m、1.4m、1.5m、1.6m、1.7m、1.8m、1.9m、2.0m0.5m、0.6m、0.7m、0.8m、0.9m、1.0m、1.1m、1.2m、1.3m、1.4m、1.5m、1.6m、1.7m、1.8m、1.9m、2.0m,或任何两个前述浓度点之间的任何值。本文列出的任何一种效应物的浓度可以与所述氯化钾的浓度相同或相似。
本文的组合物或家庭护理产品的组分可以包括洗涤剂、肥皂、工业清洁剂或碗碟洗涤液中的至少一种。
本文的组合物、家庭护理产品或方法中的洗涤剂可以是任何洗涤剂。洗涤剂可以是任何可以商购获得的洗涤剂。洗涤剂可以是但不限于gresinoltm、seventhgenerationtm、myerstm、tidetm、drefttm、purextm、biokleentm、sunandearthtm、gaintm、alltm、woolitetm、xtratm、fabtm、snuggletm、cheertm、wisktm、surftm、omotm、persiltm、breezetm、skiptm、alatm、rinsotm或greenshieldtm。肥皂可以是任何肥皂。肥皂可以是任何可以商购获得的肥皂。肥皂可以是但不限于ivorytm、dialtm、dovetm、softsoaptm、methodtm、kleenextm或carextm。
工业清洁剂可以是任何工业清洁剂。工业清洁剂可以是任何可以商购获得的工业清洁剂。工业清洁剂可以是但不限于eliminatortm(苛性碱添加剂)、alkazolvtm、ultrazolvtm700、concepttmc20、performtm或reflextmb165。工业清洁剂可以是但不限于苛性碱、naoh(1%)、edta、次氯酸钠或硝酸(1%)。
碗碟洗涤液可以是任何碗碟洗涤液。碗碟洗涤液可以是任何可以商购获得的碗碟洗涤液。碗碟洗涤液可以是但不限于cascadetm、joytm、palmolivetm、gaintm、glotm、dawntm、greenworkstm、ultratm、ziptm、cloroxtm、trimtm或tajtm。
洗涤剂浓度可以在40%(v:v)至100%(v:v)的范围内。浓度可以为40%(v:v)、45%(v:v)、50%(v:v)、55%(v:v)、60%(v:v)、65%(v:v)、70%(v:v)、75%(v:v)、80%(v:v)、90%(v:v)、91%(v:v)、92%(v:v)、93%(v:v)、94%(v:v)、95%(v:v)、96%(v:v)、97%(v:v)、98%(v:v)、99%(v:v)或100%(v:v),或任何两个前述浓度点之间的任何值。洗涤剂浓度可以在65%至100%的范围内。浓度可以为65%(v:v)、70%(v:v)、75%(v:v)、80%(v:v)、90%(v:v)、91%(v:v)、92%(v:v)、93%(v:v)、94%(v:v)、95%(v:v)、96%(v:v)、97%(v:v)、98%(v:v)、99%(v:v)或100%(v:v),或任何两个前述浓度点之间的任何值。本文所列的任何一种肥皂、工业清洁剂或碗碟洗涤液的浓度可以与洗涤剂的浓度相似。本文所列的洗涤剂百分数是指与家庭护理产品或组合物的最终体积相比较的原料洗涤剂的百分数。
本文的家庭护理产品或组合物的组分可以进一步包括选自由:水软化剂、表面活性剂、漂白剂、酶、增白剂、香料、阴离子表面活性剂、非离子表面活性剂、去除硬度离子的助洗剂(builder)、抗再沉积剂(antiredepositionagent)、染料转移抑制剂、去污聚合物、荧光增白剂、酶稳定剂、粘度控制化合物、ph控制化合物、控制过量起泡的肥皂和硅酮、用于微生物控制的防腐剂、用于气味和外观的香水和染料、漂白剂、水、增溶剂、烷基苯磺酸盐、乙氧基化脂肪醇、柠檬酸钠、edta四钠或丙烯酸聚合物、pvpk-30、chromabonds-100、chromabonds-400、sorez100、repel-o-texsrp-6、tinopalcbs-x、氯化钙、四硼酸钠、丙二醇、甲酸钠、柠檬酸钠、单乙醇胺(monoethanolamine)、丙二醇、二甲苯磺酸钠、聚合物和柠檬酸组成的组中的至少一种成分。组合物可以进一步包括作为燃料添加剂的洗涤剂。燃料添加剂可以是长链胺或酰胺。燃料添加剂可以是聚异丁烯胺或聚异丁烯酰胺/琥珀酰亚胺。组合物可以包括作为生物试剂的洗涤剂。生物试剂可用于分离和纯化在生物细胞中发现的完整的膜蛋白。本文所列的任何一种附加成分的浓度可以是本领域已知的任何浓度。
在一种实施方式中,组合物可以含有0.5%(v:v)的第一前体、0.5%(v:v)的第二前体、1mkcl和可变浓度的洗涤剂。洗涤剂的可变浓度可以在65%(v:v)至100%(v:v)的范围内。组合物可以含有38.1μm的第一前体和第二前体的每一种、1mkcl和可变浓度的洗涤剂。
以下示例性的洗涤剂制剂可以是作为原料洗涤剂添加到本文的组合物或家庭产品中以达到所需浓度或以本文的方法提供的洗涤剂。示例性的洗涤剂制剂可以包括aduxol、蓖麻油乙氧基化物(castoroilethoxylate)、脂肪醇、乙氧基化物(ethoxylate)、聚乙二醇、烷氧基羧酸(alkoxylatedcarboxylicacid)、甘油、neodol、mpg、tea、mea、prifactm5908、las酸、柠檬酸、亚硫酸钠、sokalanhp20、sles3eo、dequesttm2010和去离子水。进一步的示例性的洗涤剂可见于richardson等人在1977年4月20日提交的美国申请号05/789,325并在1973年9月19日发行的美国专利号4,115,292、在1967年2月20日发行的booth的美国专利号3,717,630、在1967年7月25日发行的kessler等人的美国专利号3,332,880中,所有这些都通过引用并入本文。示例性的阴离子表面活性剂可见于1976年3月2日发行的gilbert等人的美国专利号3,941,710中,其通过引用并入本文,如同完整阐述。可用于本文的非离子表面活性剂的类别和种类的示例性列表可见于1972年3月23日发行的norris的美国专利号3,664,961中,其通过引用并入本文,如同完整阐述。
使用天然精华增强的液体洗衣洗涤剂的示例性制剂进一步包括非石油源阴离子和非离子表面活性剂,天然存在的助洗剂公开于bastigket等人于2008年5月8日提交的美国申请号12/151,597并于2010年1月19日发行的美国专利号7,648,953中,其通过引用并入本文,如同完整阐述。该专利中公开的制剂包括以下成分:由以下组分组成的表面活性剂混合物:约1重量%至约20重量%的通式r-(och2ch2)x-o-so3m的烷基醚硫酸盐,其中,r为具有约c8至约c20的偶数碳链长度的非石油衍生的脂肪醇,并且其中x为约0.5至约8,并且其中m为碱金属或铵阳离子;约1重量%至约10重量%的通式r-(och2ch2)x-oh的脂肪醇乙氧基化物,其中,r为具有约c10至约c18的偶数碳链长度的非石油衍生的脂肪醇,并且其中x为约0.5至约9;和任选的约1重量%至约10重量%的脂肪酸肥皂;约0.1%至约5%的天然精华(essence);约0.1重量%至约10重量%的助洗剂;和水。
含有阴离子表面活性剂、织物护理剂、阳离子沉积助剂和性能增效剂的液体洗衣洗涤剂组合物的示例性制剂公开于panandiker等人于2009年2月12日提交的欧洲公开号ep2126017a1中,其通过引用并入本文,如同完整阐述。选择该制剂中的性能增效剂使得其不会与阳离子沉积助剂或织物护理剂反应形成凝聚层和/或从溶液中沉淀。液体洗衣洗涤剂制剂含有以下重量百分比的所述组合物:a.1%至80%的阴离子表面活性剂;b.0.1%至10%的织物护理剂;c.0.01%至2%的沉积助剂;和d.0.05%至10%的选自酶、阴离子聚合物和增白剂的性能增效剂。阴离子表面活性剂选自以下的组:c8-c22脂肪酸或其盐;cn-ci8烷基苯磺酸盐;c10-c20支链和随机烷基硫酸盐;ci0-ci8烷基烷氧基硫酸盐,其中,x为1-30;中链支链烷基硫酸盐;中链支链烷基烷氧基硫酸盐;包括1-5个乙氧基单元的ci0-ci8烷基烷氧基羧酸盐;改性烷基苯磺酸盐;c12-c20甲酯磺酸盐;ci0-ci8α-烯烃磺酸盐;c6-c20磺基琥珀酸盐;及其组合。织物护理剂提供了选自以下的组的织物护理益处:织物软化;颜色保护;颜色恢复;绒球/绒毛减少;抗磨损;抗皱;及其组合。织物护理剂选自以下的组:硅氧烷衍生物;油糖衍生物;分散聚烯烃;聚合物胶乳;阳离子表面活性剂;及其组合。酶选自以下的组:蛋白酶;淀粉酶;脂肪酶;纤维素酶;糖酶;木葡聚糖酶;甘露聚糖酶;果胶裂解酶;及其组合。性能增效剂是优选选自以下的组的增白剂:4,4'-双-(2-磺酰苯乙烯基)联苯二钠;苯磺酸;2,2'-(1,2-乙烯二基)双[5-[4-[(2-羟乙基)甲基氨基]-6-(苯基氨基)-1,3,5-三嗪-2-基]氨基]-二钠盐;4,4'-双{[4-苯胺基-6-[双(2-羟乙基)氨基-s-三嗪-2-基]-氨基}-2,2'-芪二磺酸二钠;4,4'-双[(4-苯胺基-6-(n-2-羟乙基-n-甲基氨基)-s-三嗪-2-基)氨基]2,2'-芪二磺酸二钠;4,4'-双{[4-苯胺基-6-甲氨基-s-三嗪-2-基]-氨基}-2,2'-芪二磺酸二钠;4,4”-双[4,6-二-苯胺基-s-三嗪-2-基]-2,2'-芪二磺酸二钠;4,4'-双{[4-苯胺基-6-吗啉代-s-三嗪-2-基}-氨基}-2,2'-芪二磺酸二钠;及其组合。性能增效剂是阴离子分散剂聚合物。组合物可以进一步含有优选选自以下的组的珠光剂:云母;氯氧化铋;鱼鳞;亚烷基二醇单酯和二酯。珠光剂可以选自以下的组:云母;乙二醇双硬脂酸酯;乙二醇单硬脂酸酯;氯氧化铋;及其组合。组合物可以进一步含有选自以下的组的洗衣辅剂:非离子表面活性剂;助洗剂;聚合物去污剂;及其组合。
示例性的洗涤剂制剂可见于2014年2月6日公布的carswel等人的国际公开号wo2014019903中,其通过引用并入本文,如同完整阐述。该申请公开了一种碱性液体洗衣洗涤剂,其含有至少1重量%的三乙醇胺、至少5重量%的非皂基表面活性剂和至少0.5重量%的根据下式(i)的聚酯,其中,r1和r2各自独立地是x-(oc2h4)n-(oc3h6)m,其中x是c1-4烷基,-(oc2h4)基团和-(oc3h6)基团以嵌段的方式排列,并且由-(oc3h6)基团组成的嵌段与coo基团结合或为ho-(c3h6),n基于摩尔平均为12至120,优选为40至50,m基于摩尔平均为1至10,且a基于摩尔平均为4至9。
示例性的洗涤剂制剂可见于2013年10月31日公开的best等人的国际公开号wo2013160023中,其通过引用并入本文,如同完整阐述。该申请公开了一种外部结构化的含水的各向同性的液体洗衣洗涤剂组合物,其含有:至少10重量%的水、至少10重量%的含有阴离子表面活性剂的混合表面活性剂体系、含有0.025至0.15重量%的不溶性纤维素纤维(含有至少50重量%的活性柑橘纤维)的外部结构化体系、-至少0.01重量%的悬浮非粘土固体颗粒,其特征在于,外部结构化体系进一步含有至少0.1重量%的水溶胀性粘土。
示例性的洗涤剂制剂可见于nair等人于1996年10月29日提交的且于1998年3月24日发行的美国专利号5731278中,其通过引用并入本文,如同完整阐述。该参考文献公开了一种液体洗衣洗涤剂,其含有表面活性剂组分、甲酸盐增稠剂组分、选择的香料组分和相对大量的含水液体载体。
示例性的洗涤剂制剂可见于1999年9月30日公布的depoot等人的国际公开号wo1999049009中,其通过引用并入本文,如同完整阐述。该申请公开了一种液体洗衣洗涤剂,其含有hedp、和水溶性和/或可分散的改性的多胺,该多胺具有提供稳定作用的官能化的骨架部分。
示例性的洗涤剂制剂可见于1998年4月2日公布的mciver等人的国际公开号wo1998013461中,其通过引用并入本文,如同完整阐述。该申请公开了一种液体洗衣洗涤剂,其含有以下的一种或多种:增泡剂、助洗剂、去污聚合物、聚丙烯酸酯聚合物、分散剂、染料转移抑制剂、染料、香料、加工助剂、增白剂及其混合物。
示例性的洗涤剂制剂可见于1999年7月21日公布的mciver等人的欧洲公开号ep0929642中,其通过引用并入本文,如同完整阐述。该申请公开了一种液体洗衣洗涤剂,其含有以下的一种或多种:增泡剂、助洗剂、去污聚合物、聚丙烯酸酯聚合物、分散剂、染料转移抑制剂、染料、香料、加工助剂、增白剂及其混合物。
含有阴离子表面活性剂的示例性的液体洗衣洗涤剂制剂可见于2013年1月1日公布的souter等人的欧洲公开号ep2551335中,其通过引用并入本文,如同完整阐述。
示例性的洗涤剂制剂可见于2014年11月27日公布的frankenbach等人的国际公开号wo2014190131中,其通过引用并入本文,如同完整阐述。该申请公开了一种含有支链表面活性剂的液体洗衣洗涤剂。
示例性的洗涤剂制剂可见于2014年4月2日公布的guida等人的欧洲公开号ep2712913中,其通过引用并入本文,如同完整阐述。该公开公开了一种液体洗衣洗涤剂,其含有包括结晶的氢化蓖麻油(hco)的结晶的甘油三酯、表面活性剂和有机非氨基官能醇。
示例性的洗涤剂制剂可见于2014年8月14日公布的zhu等人的中国公开号cn103242973中,其通过引用并入本文,如同完整阐述。该申请公开了一种液体洗衣洗涤剂,其含有0.02-15%的阳离子软化剂、0.01-10%的改性硅油、2-50%的非离子表面活性剂。
示例性的洗涤剂制剂可见于2015年4月8日公布的ellison等人的欧洲公开号ep2855408中,其通过引用并入本文,如同完整阐述。该公开公开了一种液体洗衣洗涤剂,其通过以下方法制备:使一种或多种偶数α-烯烃二聚以生成一种或多种乙烯叉(vinylidene);将乙烯叉加氢甲酰化以生成醇的混合物;并将醇的混合物硫酸化以形成醇硫酸盐。
一种实施方式提供了一种表达盒。表达盒可以包括编码具有增强的可溶性的内含肽修饰的蛋白酶的一种或多种多核苷酸。被编码的内含肽修饰的蛋白酶可以是本文所述的任何一种内含肽修饰的蛋白酶。表达盒可以包括编码第一前体的多核苷酸。表达盒可以包括编码第二前体的多核苷酸。表达盒可以包括编码第一前体和第二前体的多核苷酸。一种或多种多核苷酸可以含有、基本上由、或由与seqidno:3的参考序列具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成,其是编码图1所示的第一前体ni-dpng-trx的序列。一种或多种多核苷酸可以含有、基本上由、或由与seqid:4具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成,其是编码图1所示的第二前体trx-dpng-mtt-ic的序列。
一种或多种多核苷酸可以包括编码本文所述的任何一种靶蛋白酶的序列。一种或多种多核苷酸可以共同编码部分的枯草杆菌蛋白酶carlsberg、枯草杆菌蛋白酶bpn、来自短小芽孢杆菌(cbs)的sapb、来自短小芽孢杆菌ms-1的bpp-a蛋白酶或来自芽孢杆菌b001蛋白酶的aprb。一种或多种多核苷酸可以编码来自地衣芽孢杆菌的蛋白酶(uniprot登录号为:q6pnn5)。一种或多种多核苷酸可以含有、基本上由、或由与seqidno:7具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成,其是编码savinase的序列。
表达盒可以包括编码本文所述的任何一种内含肽的一种或多种多核苷酸。一种或多种多核苷酸可以编码gp41-1、cbu_dnab、mja_gf-6p、mja_hyp-1、mja_if2、mja_pol-1、pab_cdc21-1、pab_if2–n、pab_vma、pho_if2、pho_vma、rma_dnab、sru_dnab、tag_pol-1_tsp-ty_pol-1、ter_rir1-4、tko_if2或tth-hb27_dnae-2内含肽的n-内含肽和c-内含肽部份。一种或多种多核苷酸的两个多核苷酸可以含有、基本上由、或由与seqidno:10具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成,其是编码gp41-1内含肽的序列。一种或多种多核苷酸可以编码内含肽的n-内含肽、内含肽的c-内含肽或两者。一种或多种多核苷酸可以含有、基本上由、或由与seqidno:12具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成,其是编码gp41-1n-内含肽的序列。一种或多种多核苷酸可以含有、基本上由、或由与seqidno:13具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成,其是编码gp41-1c-内含肽的序列。
一种或多种多核苷酸可以进一步编码第一可溶性增强子、第二可溶性增强子或两者。一种或多种多核苷酸可以编码trx结构域。一种或多种多核苷酸的序列可以含有、基本上由、或由与seqidno:52具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成,其是编码包括在图1所示的ni中的trx结构域的序列。一种或多种多核苷酸的序列可以含有、基本上由、或由与seqidno:54具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成,其是编码包括在图1所示的ic中的trx结构域的序列。
一种实施方式包括一种多核苷酸,该多核苷酸含有、基本上由、或由沿其长度与具有本文所述的任何一种序列或其互补的多核苷酸的连续部分具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成。连续部分可以是任何长度,最多为本文所述的序列或其互补的整个长度。
确定两个氨基酸序列或两个核酸序列的同一性百分数可以包括在两个序列中的相应位置比对和比较氨基酸残基或核苷酸。如果两个序列中的所有位置被具有同一性的氨基酸残基或核苷酸占据,则序列被说成是100%同一性。同一性百分数是通过smithwaterman算法(smithtf,watermanms1981“identificationofcommonmolecularsubsequences,”jmolbiol147:195-197,其通过引用并入本文,如同完整阐述)测定的。
在一种实施方式中,反式剪接内含肽技术可用于调节蛋白酶活性。在这种方法中,蛋白酶可以在两个非活性片段中的催化残基之间被分割,它们可以各自地被表达为与反式剪接的内含肽融合。混合可以引发非活性片段的反式-内含肽介导的缔合,通过肽键将非活性部分剪接并与全功能活性蛋白酶无缝连接。
已经在pct/us2013/063304中描述了反式剪接的内含肽调节的液体洗衣蛋白酶savinase的构建、表达和洗涤剂稀释诱导特性,其通过引用并入本文,如同完整阐述。本文描述了将制剂稳定性和洗涤剂稀释诱导的蛋白酶活性改善至商业液体洗衣应用中的相关水平的方法。
一种实施方式提供了调节蛋白酶活性的方法。该方法包括将本文所公开的任何一种或多种内含肽修饰的蛋白酶与家庭护理产品的组分组合,并且可以包括一种或多种效应物以形成混合物。如本文所用,家庭护理产品是指用于清洁或处理家庭或其所容之物的任何产品。本文的家庭护理产品包括用于人的清洁或调理的个人护理产品。混合物中的内含肽修饰的蛋白酶至少在蛋白酶活性或剪接活性方面可以是无活性的。一种或多种效应物可以能够抑制混合物中的内含肽修饰的蛋白酶的剪接。抑制可以是可逆的。引发剪接的步骤可以包括稀释、减少或去除一种或多种效应物。剪接后,可以恢复靶蛋白酶的蛋白酶活性。一种或多种效应物可以是但不限于钠盐补充剂、钾盐补充剂、铵盐补充剂、带电聚合盐补充剂或多元醇补充剂。一种或多种效应物可以选自:氯化钠、亚氨基二琥珀酸四钠、琥珀酸二钠、酒石酸二钠、乳酸钾、柠檬酸钾、氯化钾、烟酸钠、硫酸铵、硝酸铵、柠檬酸锂、聚天冬氨酸钠、聚丙烯酸钠、四乙二醇、聚乙二醇、四甘醇、碳酸丙烯酯、单丙二醇、甘油和乙氧基化醇醚(tomadol)组成的组。一种或多种效应物可以包括氯化钾。氯化钾浓度可以在0.1m至5.0m的范围内。浓度可以为0.1m、0.2m、0.3m、0.4m、0.5m、0.6m、0.7m、0.8m、0.9m、1.0m、1.1m、1.2m、1.3m、1.4m、1.5m、1.6m、1.7m、1.8m、1.9m、2.0m、2.1m、2.2m、2.3m、2.4m、2.5m、2.6m、2.7m、2.8m、2.9m、3.0m、3.1m、3.2m、3.3m、3.4m、3.5m、3.6m、3.7m、3.8m、3.9m、4.0m、4.1m、4.2m、4.3m、4.4m、4.5m、4.6m、4.7m、4.8m、4.9、5.0m,或任何两个前述浓度点之间的任何值。氯化钾浓度可以在0.5m至2.0m的范围内。浓度可以为0.5m、0.6m、0.7m、0.8m、0.9m、1.0m、1.1m、1.2m、1.3m、1.4m、1.5m、1.6m、1.7m、1.8m、1.9m、2.0m0.5m、0.6m、0.7m、0.8m、0.9m、1.0m、1.1m、1.2m、1.3m、1.4m、1.5m、1.6m、1.7m、1.8m、1.9m、2.0m,或任何两个前述浓度点之间的任何值。本文所列的任何一种效应物的浓度可以与氯化钾的浓度相似。
在一种实施方式中,引发的步骤可以包括使用液体稀释混合物。在稀释步骤中添加至混合物中的液体的量可以是满足混合物与液体的比例选自小于或等于以下之一的值的量:1:5(v:v)、1:10(v:v)、1:20(v:v)、1:50(v:v):1:60(v:v)、1:70(v:v)、1:80(v:v)、1:90(v:v)、1:100(v:v)、1:150(v:v)、1:200(v:v)、1:250(v:v)、1:300(v:v)、1:350(v:v)或1:400(v:v),或任何两个前述(包括端值)之间的范围内的任何比例。例如,混合物与液体的比例可以是小于选自1:5至1:10的任何整数或非整数的值。混合物与液体的比例可以等于1:5(v:v)、1:10(v:v)、1:20(v:v)、1:50(v:v):1:60(v:v)、1:70(v:v)、1:80(v:v)、1:90(v:v)、1:100(v:v)、1:150(v:v)、1:200(v:v)、1:250(v:v)、1:300(v:v)、1:350(v:v)或1:400(v:v)或任何两个前述(包括端值)之间的范围内的任何比例。例如,液体与混合物的比例可以是等于1:5至1:10范围内的任何整数或非整数的值。液体可以是水。液体可以是含水缓冲液。家庭护理产品的组分可以是但不限于洗涤剂、肥皂、工业清洁剂或碗碟洗涤液。稀释可以引发内含肽修饰的蛋白酶的剪接和靶蛋白酶的活性恢复。
一种实施方式提供了在液体洗衣洗涤剂中筛选稀释诱导内含肽修饰的蛋白酶活性的效应物的方法。该方法可以包括将液体洗衣洗涤剂与本文所述的第一前体和第二前体中的任意一种混合以获得混合物。该方法可以包括向混合物中加入一种或多种化合物。该方法可以包括使用液体稀释混合物。该方法还可以包括鉴定抑制包括在各自的第一和第二前体中的n-内含肽和c-内含肽的剪接以及靶蛋白酶的活性恢复的化合物。一种实施方式还提供了将一种或多种效应物添加至液体洗衣洗涤剂配制的混合物中以可逆地抑制内含肽修饰的蛋白酶。
一种实施方式提供了家庭护理产品。家庭护理产品可以包括本文公开的任何一种内含肽修饰的蛋白酶和一种或多种效应物。家庭护理产品可以包括碗碟洗涤剂、洗衣洗涤剂、工业清洁剂或无皂肥皂(soaplesssoap)。如本文所用,术语“洗涤剂”是指表面活性剂或表面活性剂的混合物。术语“无皂肥皂”是指具有微酸性ph的无皂液体清洁剂。家庭护理产品可以包括任何其他类型的清洁剂。
家庭护理产品可以包括作为粉末的洗涤剂。家庭护理产品可以包括液体制剂中提供的洗涤剂。液体洗涤剂制剂可以含有水软化剂、表面活性剂、漂白剂、酶、增白剂、香料和一些其他试剂。液体洗涤剂制剂可以含有阴离子和非离子表面活性剂、去除硬度离子的助洗剂、各种抗再沉积剂、防止染料从一种织物脱落并沉积在另一种织物上的染料转移抑制剂、提供织物的屏障的去污聚合物、荧光增白剂、酶稳定剂、粘度控制化合物、ph控制化合物、控制过量起泡的肥皂和硅氧烷、用于微生物控制的防腐剂、用于气味和外观的香料和染料、漂白剂、水、增溶剂或提高性能特点的其他添加剂的组合。液体洗涤剂制剂可以含有阴离子表面活性剂,它们是烷基苯磺酸盐或其他阴离子表面活性剂。液体洗涤剂制剂可以含有乙氧基化脂肪醇(ethoxylatedfattyalcohol)的非离子表面活性剂。液体洗涤剂制剂可以含有助洗剂以除去硬度离子,所述助洗剂选自柠檬酸钠、edta四钠或丙烯酸聚合物。液体洗涤剂制剂可以含有选自pvpk-30、chromabonds-100或chromabonds-400的染料转移抑制剂。液体洗涤剂制剂可以含有选自sorez100聚乙二醇聚酯共聚物或repel-o-texsrp-6、聚乙二醇聚酯的去污聚合物。液体洗涤剂制剂可以含有tinopalcbs-x或任何其他荧光增白剂的荧光增白剂。液体洗涤剂制剂可以含有选自氯化钙、四硼酸钠、丙二醇、甲酸钠、柠檬酸钠或单乙醇胺的酶稳定剂。液体洗涤剂制剂可以含有丙二醇、二甲苯磺酸钠或聚合物的粘度控制化合物。液体洗涤剂制剂可以含有柠檬酸或单乙醇胺的ph控制化合物。
本文的家庭护理产品可以包括一种或多种效应物。一种或多种效应物可以是但不限于氯化钠、亚氨基二琥珀酸四钠、琥珀酸二钠、酒石酸二钠、乳酸钾、柠檬酸钾、氯化钾、烟酸钠、硫酸铵、硝酸铵、柠檬酸锂、聚天冬氨酸钠、聚丙烯酸钠、四乙二醇、聚乙二醇、四甘醇、碳酸丙烯酯、单丙二醇、甘油或乙氧基化醇醚(tomadol)。
一种实施方式提供了将蛋白酶储存在混合物中的方法。该方法可以包括制备本文所述的任何一种内含肽修饰的蛋白酶。该方法可以包括将内含肽修饰的蛋白酶与家庭护理产品和一种或多种效应物组合以形成混合物。家庭护理产品可以选自由:洗涤剂、肥皂、工业清洁剂和碗碟洗涤液组成的组。一种或多种效应物可以被选择为本文公开的任何一种效应物。混合物可以储存任何长的时间。混合物可以储存一小时至一年的时间。混合物可以储存一周、两周、三周、四周、五周、六周、七周或八周。
家庭护理产品可以包括iprotease,其是指本文所述的任何一种内含肽修饰的蛋白酶。家庭护理产品可以包括isavinase。液体洗衣洗涤剂中的isavinase储存稳定性可以与在八周以上的加速老化条件下的抑制剂稳定的savinase相当。对血液、牛奶或油墨染色的织物(empa117)的isavinase染色去除活性可以与酶的等摩尔负载下的内含肽未修饰的savinase相当。
以下列表包括本发明的具体实施方式。但是该列表不是限制性的,并且不排除本文描述的替代实施方式或另外实施方式。在以下实施方式列表中描述的同一性百分数是指沿着参考序列的整个长度的所述序列的同一性。
实施方式
1、一种内含肽修饰的蛋白酶,该内含肽修饰的蛋白酶包括:第一前体和第二前体:所述第一前体包括与反式剪接的内含肽的可溶性增强的n-内含肽融合的靶蛋白酶的n-外显肽,且n-外显肽的羧基末端与可溶性增强的n-内含肽的氨基末端融合;所述第二前体包括与靶蛋白酶的c-外显肽融合的反式剪接的内含肽的可溶性增强的c-内含肽,且可溶性增强的c-内含肽的羧基末端与c-外显肽的氨基末端融合;且在反式剪接之前所述第一前体与所述第二前体是分离的;其中,内含肽修饰的蛋白酶与靶蛋白酶相比具有增强的可溶性和降低的或抑制的活性,且经内含肽修饰的蛋白酶的反式剪接以及n-外显肽与c-外显肽的融合,获得靶蛋白酶活性。
2、根据实施方式1所述的内含肽修饰的蛋白酶,其中,所述可溶性增强的n-内含肽包括n-内含肽和第一可溶性增强子,且所述n-内含肽的羧基末端通过第一连接体与所述第一可溶性增强子融合。
3、根据实施方式1-2中的一项或两项所述的内含肽修饰的蛋白酶,其中,所述可溶性增强的c-内含肽包括c-内含肽和第二可溶性增强子,且所述第二可溶性增强子通过第二连接体与所述c-内含肽的氨基末端融合。
4、根据实施方式2-3中的一项或多项所述的内含肽修饰的蛋白酶,其中,所述第一可溶性增强子或所述第二可溶性增强子的至少一种独立地选自由:硫氧还蛋白结构域trx、小泛素相关蛋白sumo、谷胱甘肽-s转移酶gst、麦芽糖结合蛋白mbp、n利用物质anusa和十七千道尔顿蛋白skp组成的组。
5、根据实施方式2-4中的一项或多项所述的内含肽修饰的蛋白酶,其中,所述第一可溶性增强子和所述第二可溶性增强子的每一种均含有硫氧还蛋白结构域trx。
6、根据实施方式2-5中的一项或多项所述的内含肽修饰的蛋白酶,其中,所述第一可溶性增强子含有、基本上由、或由与seqidno:51的序列具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成,且所述第二可溶性增强子含有、基本上由、或由与seqidno:53的序列具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。
7、根据实施方式2-6中的一项或多项所述的内含肽修饰的蛋白酶,其中,所述第一连接体或所述第二连接体的至少一种为dpng连接体。
8、根据前述实施方式中的一项或多项所述的内含肽修饰的蛋白酶,其中,所述靶蛋白酶为选自由:ec3.4.99蛋白酶、ec3.4.21.62蛋白酶、角蛋白酶、丝氨酸蛋白酶、碱性蛋白酶、酸性蛋白酶、金属蛋白酶、半胱氨酸蛋白酶、天冬氨酸蛋白酶、atp依赖性蛋白酶、枯草杆菌蛋白酶家族的蛋白酶组成的组中的酶。
9、根据前述实施方式中的一项或多项所述的内含肽修饰的蛋白酶,其中,所述靶蛋白酶包括选自由savinase、碱性蛋白酶q6pnn5和酸性蛋白酶b8nly9_pepa_aspen组成的组中的靶蛋白酶。
10、根据前述实施方式中的一项或多项所述的内含肽修饰的蛋白酶,其中,所述靶蛋白酶含有、基本上由、或由与选自由seqidnos:6、8和9组成的组中的序列具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。
11、根据前述实施方式中的一项或多项所述的内含肽修饰的蛋白酶,其中,所述内含肽选自由:gp41-1、cbu_dnab、mja_gf-6p、mja_hyp-1、mja_if2、mja_pol-1、pab_cdc21-1、pab_if2–n、pab_vma、pho_if2、pho_vma、rma_dnab、sru_dnab、tag_pol-1_tsp-ty_pol-1、ter_rir1-4、tko_if2和tth-hb27_dnae-2内含肽组成的组。
12、根据前述实施方式中的一项或多项所述的内含肽修饰的蛋白酶,其中,所述内含肽为gp41-1内含肽并且含有、基本上由、或由与seqidno:11具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。
13、根据前述实施方式中的一项或多项所述的内含肽修饰的蛋白酶,其中,所述n-内含肽含有、基本上由、或由与选自由seqidnos:14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44和46组成的组中的序列具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成且所述c-内含肽含有、基本上由、或由与选自由seqidnos:15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45和47组成的组中的序列具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成,且所述n-内含肽和所述c-内含肽共同形成选自由:gp41-1、cbu_dnab、mja_gf-6p、mja_hyp-1、mja_if2、mja_pol-1、pab_cdc21-1、pab_if2–n、pab_vma、pho_if2、pho_vma、rma_dnab、sru_dnab、tag_pol-1_tsp-ty_pol-1、ter_rir1-4、tko_if2和tth-hb27_dnae-2组成的组中的反式剪接的内含肽或其变体。
14、根据前述实施方式中的一项或多项所述的内含肽修饰的蛋白酶,其中,所述第一前体含有、基本上由、或由与seqidno:1的参考序列具有70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。
15、根据前述实施方式中的一项或多项所述的内含肽修饰的蛋白酶,其中,所述第二前体含有、基本上由、或由与seqidno:2的参考序列具有70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。
16、一种表达盒,该表达盒包括编码内含肽修饰的蛋白酶的第一前体或第二前体的一种或多种多核苷酸,
其中,所述第一前体包括与反式剪接的内含肽的可溶性增强的n-内含肽融合的靶蛋白酶的n-外显肽,且n-外显肽的羧基末端与可溶性增强的n-内含肽的氨基末端融合;所述第二前体包括与靶蛋白酶的c-外显肽融合的反式剪接的内含肽的可溶性增强的c-内含肽,且可溶性增强的c-内含肽的羧基末端与c-外显肽的氨基末端融合;并且在反式剪接之前所述第一前体与所述第二前体是分离的;并且
其中,经所述表达盒的表达,内含肽修饰的蛋白酶与靶蛋白酶相比将具有增强的可溶性和降低的或抑制的活性,且经内含肽修饰的蛋白酶的反式剪接以及n-外显肽与c-外显肽的融合,靶蛋白酶的活性将恢复。
17、根据实施方式16所述的表达盒,其中,编码可溶性增强的n-内含肽的一种或多种多核苷酸包括n-内含肽和第一可溶性增强子,且所述n-内含肽的羧基末端通过第一连接体与所述第一可溶性增强子融合。
18、根据实施方式16-17中的一项或多项所述的表达盒,其中,编码可溶性增强的c-内含肽的一种或多种多核苷酸包括内含肽的c-内含肽和第二可溶性增强子,且所述第二可溶性增强子通过第二连接体与所述c-内含肽的氨基末端融合。
19、根据实施方式16-18中的一项或多项所述的表达盒,其中,一种或多种多核苷酸编码选自由:硫氧还蛋白结构域trx、小泛素相关蛋白sumo、谷胱甘肽-s转移酶gst、麦芽糖结合蛋白mbp、n利用物质anusa和十七千道尔顿蛋白skp组成的组的第一可溶性增强子或第二可溶性增强子。
20、根据实施方式17-19中的一项或多项所述的表达盒,其中,一种或多种多核苷酸编码含有硫氧还蛋白结构域trx的第一可溶性增强子和第二可溶性增强子中的每一种。
21、根据实施方式17-20中的一项或多项所述的表达盒,其中,一种或多种多核苷酸含有、基本上由、或由与seqidno:52具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成。
22、根据实施方式17-21中的一项或多项所述的表达盒,其中,一种或多种多核苷酸含有、基本上由、或由与seqidno:54具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成。
23、根据实施方式17-22中的一项或多项所述的表达盒,其中,一种或多种多核苷酸编码dpng连接体。
24、根据实施方式16-23中的一项或多项所述的表达盒,其中,一种或多种多核苷酸编码选自由:ec3.4.99蛋白酶、ec3.4.21.62蛋白酶、角蛋白酶、丝氨酸蛋白酶、碱性蛋白酶、金属蛋白酶、半胱氨酸蛋白酶、天冬氨酸蛋白酶、atp依赖性蛋白酶和枯草杆菌蛋白酶家族的蛋白酶组成的组中的靶蛋白酶。
25、根据实施方式16-24中的一项或多项所述的表达盒,其中,一种或多种多核苷酸编码选自由:savinase、碱性蛋白酶q6pnn5和酸性蛋白酶b8nly9_pepa_aspen组成的组中的靶蛋白酶。
26、根据实施方式16-25中的一项或多项所述的表达盒,其中,一种或多种多核苷酸编码savinase并且含有、基本上由、或由与seqidno:7具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成。
27、根据实施方式16-26中的一项或多项所述的表达盒,其中,一种或多种多核苷酸编码选自由:gp41-1、cbu_dnab、mja_gf-6p、mja_hyp-1、mja_if2、mja_pol-1、pab_cdc21-1、pab_if2–n、pab_vma、pho_if2、pho_vma、rma_dnab、sru_dnab、tag_pol-1_tsp-ty_pol-1、ter_rir1-4、tko_if2或tth-hb27_dnae-2内含肽组成的组中的内含肽。
28、根据实施方式27所述的表达盒,其中,一种或多种多核苷酸编码gp41-1内含肽并且含有、基本上由、或由与seqidno:10具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成。
29、根据实施方式28所述的表达盒,其中,一种或多种多核苷酸编码gp41-1内含肽的n-内含肽并且含有、基本上由、或由与seqidno:12具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成。
30、根据实施方式28所述的表达盒,其中,一种或多种多核苷酸编码gp41-1内含肽的c-内含肽并且含有、基本上由、或由与seqidno:13具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成。
31、根据实施方式16-30中任意一项或多项所述的表达盒,其中,一种或多种多核苷酸编码所述第一前体并且含有、基本上由、或由与seqidno:3具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成。
32、根据实施方式16-31中任意一项或多项所述的表达盒,其中,一种或多种多核苷酸编码所述第二前体并且含有、基本上由、或由与seqidno:4具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的序列组成。
33、一种组合物,该组合物含有:内含肽修饰的蛋白酶的第一前体和内含肽修饰的蛋白酶的第二前体,所述第一前体包括与反式剪接的内含肽的可溶性增强的n-内含肽融合的靶蛋白酶的n-外显肽,且n-外显肽的羧基末端与可溶性增强的n-内含肽的氨基末端融合;所述第二前体包括与靶蛋白酶的c-外显肽融合的反式剪接的内含肽的可溶性增强的c-内含肽,且可溶性增强的c-内含肽的羧基末端与c-外显肽的氨基末端融合;且在反式剪接之前,所述第一前体与所述第二前体是分离的;其中,内含肽修饰的蛋白酶与靶蛋白酶相比具有增强的可溶性和降低的活性,且经内含肽修饰的蛋白酶的反式剪接以及n-外显肽与c-外显肽的融合,获得靶蛋白酶活性。
34、根据实施方式33所述的组合物,其中,所述可溶性增强的n-内含肽包括n-内含肽和第一可溶性增强子,且所述n-内含肽的羧基末端通过第一连接体与所述第一可溶性增强子融合;和/或所述可溶性增强的c-内含肽包括c-内含肽和第二可溶性增强子,且所述第二可溶性增强子通过第二连接体与所述c-内含肽的氨基末端融合。
35、根据实施方式33-34中的一项或两项所述的组合物,其中,所述第一可溶性增强子或所述第二可溶性增强子选自由:硫氧还蛋白结构域trx、小泛素相关蛋白sumo、谷胱甘肽-s转移酶gst、麦芽糖结合蛋白mbp、n利用物质anusa和十七千道尔顿蛋白skp组成的组。
36、根据实施方式33-35中的一项或多项所述的组合物,其中,所述第一可溶性增强子和所述第二可溶性增强子的每一种均含有硫氧还蛋白结构域trx。
37、根据实施方式34-36中的一项或多项所述的组合物,其中,所述第一可溶性增强子含有、基本上由、或由与seqidno:51具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成;和/或所述第二可溶性增强子含有、基本上由、或由与seqidno:53具有至少90%的同一性的氨基酸序列组成。
38、根据实施方式34-37中的一项或多项所述的组合物,其中,所述第一连接体或所述第二连接体为dpng连接体。
39、根据实施方式33-38中的一项或多项所述的组合物,其中,所述靶蛋白酶为选自由:ec3.4.99蛋白酶、ec3.4.21.62蛋白酶、角蛋白酶、丝氨酸蛋白酶、碱性蛋白酶、酸性蛋白酶、金属蛋白酶、半胱氨酸蛋白酶、天冬氨酸蛋白酶、atp依赖性蛋白酶、枯草杆菌蛋白酶家族的蛋白酶、savinase、碱性蛋白酶q6pnn5和酸性蛋白酶b8nly9_pepa_aspen组成的组中的酶。
40、根据实施方式33-39中的一项或多项所述的组合物,其中,所述靶蛋白酶含有、基本上由、或由与选自由seqidnos:6、8和9组成的组中的序列具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。
41、根据实施方式3-40中的一项或多项所述的组合物,其中,所述n-内含肽含有、基本上由、或由与选自由seqidnos:14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44和46组成的组中的序列具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成,所述c-内含肽含有、基本上由、或由与选自由seqidnos:15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45和47组成的组中的序列具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成,且所述n-内含肽和所述c-内含肽共同形成选自由:gp41-1、cbu_dnab、mja_gf-6p、mja_hyp-1、mja_if2、mja_pol-1、pab_cdc21-1、pab_if2–n、pab_vma、pho_if2、pho_vma、rma_dnab、sru_dnab、tag_pol-1_tsp-ty_pol-1、ter_rir1-4、tko_if2和tth-hb27_dnae-2组成的组中的反式剪接的内含肽或其变体。
42、根据实施方式33-41中的一项或多项所述的组合物,其中,所述第一前体含有、基本上由、或由与seqidno:1具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成;和/或所述第二前体含有、基本上由、或由与seqidno:2具有至少70、72、75、80、85、90、91、92、93、94、95、96、97、98、99或100%的同一性的氨基酸序列组成。
43、一种家庭护理产品,该家庭护理产品含有实施方式1-15中的一项或多项所述的内含肽修饰的蛋白酶、或实施方式33-42中的一项或多项所述的组合物、以及一种或多种效应物。
44、根据实施方式43所述的家庭护理产品,其中,所述一种或多种效应物能够抑制反式剪接。
45、根据实施方式44所述的家庭护理产品,其中,所述抑制是可逆的。
46、根据实施方式43-45中的一项或多项所述的家庭护理产品,所述家庭护理产品进一步含有选自由:洗涤剂、肥皂、工业清洁剂、无皂肥皂和碗碟洗涤液组成的组中的至少一种试剂。
47、根据实施方式43-46中的一项或多项所述的家庭护理产品,其中,所述一种或多种效应物选自由:氯化钠、亚氨基二琥珀酸四钠、琥珀酸二钠、酒石酸二钠、乳酸钾、柠檬酸钾、氯化钾、烟酸钠、硫酸铵、硝酸铵、柠檬酸锂、聚天冬氨酸钠、聚丙烯酸钠、四乙二醇、聚乙二醇、四甘醇、碳酸丙烯酯、单丙二醇、甘油和乙氧基化醇醚(tomadol)组成的组。
48、根据实施方式43-47中的一项或多项所述的家庭护理产品,所述家庭护理产品进一步含有选自由:水软化剂、表面活性剂、漂白剂、酶、增白剂、香料、阴离子表面活性剂、非离子表面活性剂、去除硬度离子的助洗剂、抗再沉积剂、染料转移抑制剂、去污聚合物、荧光增白剂、酶稳定剂、粘度控制化合物、ph控制化合物、控制过量起泡的肥皂和硅酮、用于微生物控制的防腐剂、用于气味和外观的香水和染料、漂白剂、水、增溶剂、烷基苯磺酸盐、乙氧基化脂肪醇、柠檬酸钠、edta四钠或丙烯酸聚合物、pvpk-30、chromabonds-100、chromabonds-400、sorez100、repel-o-texsrp-6、tinopalcbs-x、氯化钙、四硼酸钠、丙二醇、甲酸钠、柠檬酸钠、单乙醇胺、丙二醇、二甲苯磺酸钠、聚合物和柠檬酸组成的组中的至少一种成分。
49、根据实施方式43-48中的一项或多项所述的家庭护理产品,所述家庭护理产品进一步含有选自由:长链胺或酰胺、聚异丁烯胺、聚异丁烯酰胺和琥珀酰亚胺组成的组中的燃料添加剂。
50、根据实施方式43-49中的一项或多项所述的家庭护理产品,所述家庭护理产品进一步含有生物试剂。
51、根据实施方式43-50中任意一项或多项所述的家庭护理产品,其中,一种或多种效应物选自由:氯化钠、亚氨基二琥珀酸四钠、琥珀酸二钠、酒石酸二钠、乳酸钾、柠檬酸钾、氯化钾、烟酸钠、硫酸铵、硝酸铵、柠檬酸锂、聚天冬氨酸钠、聚丙烯酸钠、四乙二醇、聚乙二醇、四甘醇、碳酸丙烯酯、单丙二醇、甘油和乙氧基化醇醚(tomadol)组成的组。
52、一种调节蛋白酶活性的方法,该调节蛋白酶活性的方法包括:
形成实施方式43-51中任意一项或多项所述的家庭护理产品。
53、根据实施方式36所述的方法,其中,形成包括将实施方式33-42中任意一项或多项所述的组合物与一种或多种效应物结合。
54、根据实施方式52-53中的一项或两项所述的方法,所述方法包括引发内含肽修饰的蛋白酶的剪接。
55、根据实施方式52-54中的一项或多项所述的方法,其中,所述一种或多种效应物选自由:钠盐补充剂、钾盐补充剂、铵盐补充剂、带电聚合盐补充剂和多元醇补充剂组成的组。
56、根据实施方式52-55中的一项或多项所述的方法,其中,所述一种或多种效应物选自由:氯化钠、亚氨基二琥珀酸四钠、琥珀酸二钠、酒石酸二钠、乳酸钾、柠檬酸钾、氯化钾、烟酸钠、硫酸铵、硝酸铵、柠檬酸锂、聚天冬氨酸钠、聚丙烯酸钠、四乙二醇、聚乙二醇、四甘醇、碳酸丙烯酯、单丙二醇、甘油和乙氧基化醇醚(tomadol)组成的组。
57、根据实施方式52-56中的一项或多项所述的方法,其中,所述一种或多种效应物含有氯化钾。
58、根据实施方式57所述的方法,其中,所述氯化钾的浓度在0.1m-5.0m的范围内。
59、根据实施方式57或58的一项或两项所述的方法,其中,所述氯化钾的浓度在0.5m-2.0m的范围内。
60、根据实施方式54-59中的一项或多项所述的方法,其中,引发剪接的步骤包括使用液体将混合物稀释成混合物:液体的比例小于或等于选自由混合物比液体为1:5、1:10、1:20、1:50:1:60、1:70、1:80、1:90、1:100、1:150、1:200、1:250、1:300、1:350和1:400组成的组中的一种比例。
61、根据实施方式60所述的方法,其中,所述液体为水和含水缓冲液中的一种。
63、根据实施方式52-61中的一项或多项所述的方法,其中,经所述内含肽修饰的蛋白酶的剪接,获得所述靶蛋白酶的蛋白酶活性。
64、一种在混合物中储存蛋白酶的方法,该方法包括:制备实施方式1-15中任意一项或多项所述的内含肽修饰的蛋白酶或实施方式33-42中任意一项或多项所述的组合物,且将所述内含肽修饰的蛋白酶或所述组合物与选自由:洗涤剂、肥皂、工业清洁剂和碗碟洗涤液组成的组中的至少一种试剂以及一种或多种效应物结合以形成混合物。
本文的其他实施方式可以通过使用本文任何一种或多种其他实施方式的一个或多个元素补充一种实施方式,和/或用本文一种或多种其他实施方式的一个或多个元素替代一种实施方式的一个或多个元素来形成。
实施例
提供以下非限制性的实施例用来说明具体的实施方式。各个实施方式可以用以下一个或多个实施例中的一个或多个细节加以补充,和/或实施方式中的一个或多个元素可以用以下一个或多个实施例中的一个或多个细节代替。
实施例1.用于调节蛋白酶的活性的反式剪接的内含肽技术
反式剪接的内含肽技术可以用于调节蛋白酶活性。在这种方法中,蛋白酶在催化残基之间被分割成两个无活性的片段,其各自被表达为与反式剪接的内含肽融合。在含水环境中混合会触发无活性片段的反式-内含肽介导的结合,无活性部分的剪接和无缝连接以形成具有完全功能的活性蛋白酶。
已经在pct专利申请pct/us2013/063304中描述了反式剪接的内含肽调节的液体洗衣蛋白酶savinase的构建、表达和洗涤剂稀释诱导特性,其通过引用并入本文,如同完整阐述。本文描述了将制剂稳定性和洗涤剂稀释诱导型蛋白酶活性改善到商业液体洗衣应用中的相关水平的方法。
实施例2.可溶性优化的分子
洗涤剂是指液体洗衣洗涤剂。洗涤剂-1和-4是无酶、无荧光增白剂和无着色剂的标准液体洗涤剂制剂。洗涤剂-1可以含有去离子水、neodol25_7、las酸、prifac5908、dequest2010、sles3eo、亚硫酸钠、sokalanhp20、mpg、mea、tea、甘油、tinopal5bmgx和其他常见的液体洗衣洗涤剂组分。洗涤剂-1的ph值为~8.3±0.2,电导率为~3890μsimens。洗涤剂-4的ph值为~7.3±0.2,电导率为~22400μsimens。
洗涤剂-2和-3是具有配制的蛋白酶的无荧光增白剂和着色剂的商业品牌洗涤剂。洗涤剂-2的ph值为~8.8±0.2,电导率为~3900μsimens。洗涤剂-3的ph值为~8.3±0.2,电导率为~7090μsimens。通过在80℃下热处理90min使蛋白酶失活。冷却至室温后,按照如下方法验证不存在蛋白酶:使用去离子水将洗涤剂稀释至50%v/v。向25μl的50%洗涤剂中加入75μl的br缓冲液(加入40mm磷酸氢二钠、40mm乙酸钠、40mm硼酸钠,使用naoh调节ph至9.0)、100μl的faaf-pna底物(500μm),并在37℃下在动力学试验中于400nm下读取吸光度。将试验板密封并在37℃下过夜孵育后检测指示蛋白酶活性的黄色显影。经热处理的洗涤剂没有可测量的蛋白酶活性。未加热的洗涤剂在相同的条件下显示出稳健的和即时的蛋白酶活性。
savinase是来自迟缓芽孢杆菌的细胞外碱性蛋白酶(uniprot登录号为:p29600(http://www.uniprot.org/uniprot/p29600)且是液体洗衣洗涤剂中的常见蛋白酶。本文的实施例使用具有seqidno:6的氨基酸序列的savinase。
savinase酶试验用于savinase酶试验的底物是显色肽底物n-琥珀酰-ala-ala-pro-phe对硝基苯胺(sigma-aldrich)。该底物对枯草杆菌蛋白酶样酶是高度特异的(davis等人,1999),并且它可以支持在细菌悬浮液中进行的酶试验(bonifait等人,2010,其通过引用并入本文,如同完整阐述)。在典型的试验中,将100μl的裂解物或细菌悬浮液加入到20μl的显色底物n-琥珀酰-ala-ala-pro-phe-pna(2mg/ml在50%二甲基甲酰胺中)中,反应混合物在37℃下孵育可变时间,并通过测量415nm处的吸光度来定量pna的释放(bonifait等人,2010)。通过进行高通量蛋白酶活性试验,该方案易于通过自动化以适于支持筛选。蛋白水解活性也可以通过消化azo-酪蛋白来测量(vazquez等人,2004)。在384孔板中将20微升的裂解物与20μl溶于tris-hcl缓冲液(0.1m,ph8.0)的1%(w/v)azo-酪蛋白和0.5mmcacl2在55℃下孵育30min。使用40μl的5%(w/v)三氯乙酸终止反应后,将反应混合物离心,并在340nm处测量上清液的吸光度。
isavinase是指内含肽修饰的蛋白酶体系,其中靶蛋白酶是savinase。更普遍地,本文的任何内含肽修饰的蛋白酶体系,包括isavinase体系,可以被称为iprotease。savinase分子可以通过内含肽修饰的前体的反式剪接产生。在内含肽剪接之前表达的内含肽修饰的前体分子被称为“ni”(代表包括原结构域的savinasen-末端片段(n-外显肽)和末端具有残基316(基于preprosavinase编号)的序列框架与gp41-1内含肽(in)的n-末端部分融合,和ic(代表gp41-1c-末端内含肽(ic)与起始于丝氨酸317的savinase的c-末端部分(c-外显肽)融合。具有无缝连接的氨基外显肽(n)和羧基外显肽(c)的剪接产物被称为“nc”,其在原结构域的自动分裂后产生有活性的酶,iprotease。“(ni+ic)预混物”是指处于无活性状态的剪接合格的ni和ic的混合物,无活性状态不具有蛋白酶活性,但可以通过稀释诱导产生完全活性的iprotease。
本文所述的ni和ic是pct/us2013/063304中描述的原型分子iprotease:s317-gp41-1ni和ic的可溶性优化版本,其通过引用并入本文,如同完整阐述。
具有融合的n-内含肽和c-内含肽部分的野生型gp41-1分割内含肽(长度:375bp)的核苷酸序列如下。c-内含肽部分是粗体的且带有下划线的。
tgtctggacctgaaaacgcaagtgcaaaccccgcaaggcatgaaggaaatctcaaacatccaagtcggtgacctggtgctgtcgaataccggctataacgaagtgctgaatgtttttccgaagagcaaaaagaaatcttacaagatcacgctggaagatggcaaggaaattatttgcagcgaagaacatctgttcccgacccagacgggcgaaatgaatatctccggcggtctgaaagaaggcatgtgtctgtacgtcaaggaa
具有融合的n-和c-内含肽部分的野生型gp41-1分割内含肽的蛋白序列如下。c-内含肽部分是粗体的且带有下划线的。
cldlktqvqtpqgmkeisniqvgdlvlsntgynevlnvfpkskkksykit50ledgkeiicseehlfptqtgemnisgglkegmclyvke
gp41-1序列的图谱如下所示:
在41-1图谱中,n-和c-内含肽部分被融合,并且c-内含肽部分是粗体的且带有下划线的。
用于改善可溶性的分子优化都限于内含肽部分,以确保当内含肽自身移除并无缝连接侧翼蛋白序列以将完整的蛋白主链恢复至完全活性时,在靶酶中的剪接没有痕迹。
图1显示可溶性优化分子的示意图。参考图1,savinase在催化残基之间被分割成sav-n和sav-c。催化残基为:d(asp143)、h(his172)、s(ser326),相对位置用垂直线标记。内含肽部分in和ic分别与savinase部分sav-n和sav-c连接。可溶性工程改造限于内含肽部分。可溶性增强子trx结构域通过dpng(asp-pro-asn-gly(seqidno:5))连接体与每个内含肽部分连接。c-内含肽部分(ic)具有多个突变用于增强可溶性。它们是m89δ、l91t、l95t、l122g(通过在融合的in+ic中的位置编号)。
被称为ni-dpng-trx的可溶性优化的ni具有通过asp-pro-asn-gly(dpng)连接体与iprotease:s317-gp41-1n中的gp41-1n-内含肽(in)的c-末端融合的c-末端可溶性增强子硫氧还蛋白结构域(trx,来自pet32)。在被称为trx-dpng-mttic[g122l]的可溶性优化的ic中,硫氧还蛋白结构域(trx)结构域通过asp-pro-asn-gly(dpng)连接体n末端与iprotease:s317-gp41-1c的突变的c-内含肽(ic)融合。突变的c-内含肽,mtt-ic[l122g]在m89δ、l91t、l95t和l122g(通过在gp41-1的融合的in+ic中的位置编号)位置具有四个可溶性增强子突变。
可溶性优化的iproteaseni(ni-dpng-trx)的核酸序列
可溶性优化的iproteaseni(ni-dpng-trx)的氨基酸序列
可溶性优化的iproteaseic(trx-dpng-mtt-ic)的核酸序列
可溶性优化的iproteaseic(trx-dpng-mtt-ic)的氨基酸序列
iproteaseni的图谱如下:
在iproteaseni的图谱中,proproteases317是proproteasen-末端部分:去除pre-proprotease的27aa残基分泌信号;第一个蛋氨酸被添加至n-末端;并且proproteaseaa残基为残基28至316(pre-proprotease编号)。glfs-n是n-内含肽。dpng是glfs-nn-内含肽和trx之间的asp-pro-asn-gly(seqidno:5)连接体,并且为带有阴影的。trx是来自pet32的可溶性增强子硫氧还蛋白结构域,并以粗体字显示。
iproteaseic的图谱如下:
在iproteaseic的图谱中,trx是来自pet32的可溶性增强子硫氧还蛋白结构域,并以粗体字显示。dpng是trx和glfsc-内含肽之间的asp-pro-asn-gly(seqidno:5)连接体,并且是带有灰色阴影的。glfs-c[m89δ、l91t、l95t、l122g]是glfsc-内含肽的“mtt”变体,并且是带有下划线的。s317protease是以丝氨酸317(s317)开始的proproteasec-末端片段。
实施例3.在大肠杆菌中可溶性优化的ni和ic的表达
为了在大肠杆菌中表达,将可溶性优化的ni和ic的核苷酸序列克隆到iptg诱导型t7启动子下游的petduet1(novagen)表达载体中。在ncoi和psti位点之间克隆ni-dpng-trx,在ndei和kpni位点之间克隆trx-dpng-mtt-ic[l122g]。将质粒引入大肠杆菌菌株bl21gold(de3)(invitrogen)中。
为了表达蛋白,将在lb+羧苄青霉素(100mg/l)中于30℃以300rpm条件下生长过夜的起始孵育物以40倍稀释至新鲜lb+carb100mg/l中,在30℃下以300rpm进一步孵育。当孵育物达到od6000.6时,加入iptg至终浓度0.5mg/l以诱导表达,并且收获t=0时间点样品。在t=1.5、3.0、6.0和22.0小时,取等份(2ml)样品。将细胞在30℃下,在200μl裂解缓冲液中以300rpm离心形成沉淀并裂解1小时,将裂解物分成两等份。裂解缓冲液为[10xfastbreak:mtsb缓冲液(150mmnacl、50mmtrisbase、2mmcacl2,ph=7.5,1mmdtt)]+0.2μl/mlbenzonase(novagen)的1:9混合物。为了确定总蛋白图谱,向20μl细胞裂解液中加入20μl2xsds负载染料+5%βme,将样品在95℃加热5分钟,并在12%sds/page上分离5μl等份样品,并且将凝胶进行考马斯染色。为了确定可溶性蛋白图谱,将裂解物在微量离心机中以5000g旋转5分钟,收集上清可溶部分,并按如上所述处理20μl等份样品。
在iptg诱导的t7启动子驱动的重组蛋白的表达的典型时间过程中,大多数靶蛋白是在诱导的前三个小时期间产生。蛋白表达至相当水平并主要积累至可溶部分。图2a-2b示出了在大肠杆菌中可溶性优化的ni和ic的表达。图2a示出了总蛋白图谱。图2b示出了可溶性蛋白图谱。特别地,图2b示出了可溶性优化的ni和ic的大肠杆菌表达。参考图2a-2b,表达来自大肠杆菌bl21gold(de3)中petduet1的iptg诱导型t7启动子。将过夜孵育物以40倍稀释至新鲜lb+羧苄青霉素(100mg/l)中,并在30℃下,以300rpm生长至od0.6。加入iptg至终浓度0.5mg/l以诱导表达,并在t=0、1.5、3.0、6.0和22.0小时取等份样品。收集细胞,在30℃下,在1/10th孵育体积的裂解缓冲液中以300rpm裂解1小时。参考图2a,为了确定总蛋白图谱,将粗裂解物与等体积的2xsds负载染料+5%βme混合,在95℃下加热5分钟,并在12%sds/page上分离5μl,并将凝胶进行考马斯染色。参考图2b,为了确定可溶性蛋白图谱,将粗裂解物以5000g/5min澄清,并且按如上所述处理上清液部分。观察到ni和ic主要表达于可溶部分。
实施例4.筛选稀释诱导iprotease活性的效应物
目的是开发一种液体洗衣洗涤剂蛋白酶,其可以在制剂中以稳定的无活性状态储存并且在洗涤循环开始时通过稀释于水而活化。概念验证表明,savinase可以被分割为两部分,并表达为内含肽修饰的无活性部分,并将其配制成液体洗衣洗涤剂以通过稀释至水中恢复活性。然而,反式剪接的蛋白酶仍然存在几个挑战以满足应用相关性,主要是稀释反应的稳健性以恢复超过80%的活性(基于与内含肽未修饰的酶相等的摩尔负载量),以及诱导合格状态中的洗涤剂储存稳定性。
主要的技术挑战在于,虽然反式剪接是双分子、缔合驱动和浓度依赖性反应,但该应用需要400倍稀释用于诱导,该诱导可以抵消用于剪接的ni和ic的稀释后缔合。另外,为了使反式剪接稳健,期望稀释诱导的剪接是快速的,并在数分钟内产生与洗衣洗涤剂应用相关的蛋白酶活性。当ni和ic在洗涤剂中分离时,通过稀释可能难以实现这些要求,但是可想而知,当反式剪接的部分预先缔合但是防止剪接时,可以更好地满足这些要求。
反式剪接基本上以与剪接相同的机制进行(perlerfb,2005),主要差异是要求内含肽部分的缔合。使反式剪接的前体结合但防止进展到剪接的一种可能性是使用剪接抑制剂,如zn++、cu++或cistatin,其在剪接开始时在后缔合步骤中起作用(nicholsnm.等人,2003;sunp.等人,2005:zhangl.等人,210;zhangl.等人,2011,所有这些都通过引用并入本文,如同完整阐述)。尽管这些抑制剂可以满足受控的、稀释诱导的剪接的要求,但它们与家庭使用应用不兼容。
作为替代方案,观察到洗涤剂-1抑制ni和ic的制剂中的iprotease活性,但不抑制内含肽未修饰的savinase的活性,这表明在洗涤剂中反式剪接可能被抑制。现代液体洗衣洗涤剂是各种成分的复合制剂,成分包括一种或多种离子和非离子表面活性剂、无机盐、电解质和助剂如增白剂、香料和着色剂以及分散或溶解在通常形成结构化液体的含水介质中的其他化合物,结构化液体可以以不可预测的方式与制剂的蛋白相互作用。抑制可以是结构化液体的特性或个别的洗涤剂组分也可以具有抑制特性。
电解质对蛋白的表面电荷及其等电点的影响在小电解质和聚合电解质的存在下可能是不同的(lyklemaj,2009;martin-molinaa.等人,2003;bestemank.等人2004,所有这些通过引用并入本文,如同完整阐述)。与单价盐相反,通过由抗衡离子相互作用的聚合离子,离子吸附甚至可以导致诸如电荷反转和通过电泳迁移率测量的等电点之间的差异以及零表面电荷的点的影响(bestemank.等人,2004;trulssonm.等人,2006,所有这些都通过引用并入本文,如同完整阐述)。
作为洗涤剂补充剂,研究了具有单价离子的盐,以及具有多个价态的(polyvalent)/多价(multivalent)离子的盐,其使用洗涤剂抑制和稀释诱导试验来影响洗涤剂制剂的ni和ic的稀释诱导型iprotease活性。洗涤剂抑制测量制剂的ni和ic的活性以在洗涤剂中产生iprotease,稀释诱导解决了抑制是否可逆的问题。为了改善试验灵敏度,已经选择了洗涤剂-2。该洗涤剂与ni和ic制剂具有相容性,但粘度较低并且洗涤剂抑制活性低于洗涤剂-1。选择具有洗涤剂相容性的补充剂,并且该补充剂是具有工业质量且成本低的化学品。任意选择与洗涤剂制剂相容的浓度。洗涤剂-2使用具有2x浓度的补充剂的去离子水稀释至50%(v/v),对照组无补充剂。然后,将25μl的ni和25μl的ic依次混合到洗涤剂中,并将样品在20℃下暴露于抑制条件过夜。将样品稀释20倍至相应的洗涤剂+补充剂中(洗涤剂抑制试验)或稀释20倍至去离子水中(稀释诱导试验)。
图3a-3e示出了电解质和多元醇对制剂的反式剪接的ni和ic的洗涤剂抑制和稀释诱导活性的影响。参考这些图,测试的补充剂如下:钠盐补充剂(图3a)、钾盐补充剂(图3b)、铵盐补充剂(图3c)、带电聚合盐补充剂(图3d)和多元醇补充剂(图3e)。使用去离子水将洗涤剂-2稀释至50%(v/v),并加入补充剂至2x浓度(例如,2m用于1m测试)。ni和ic在大肠杆菌中表达,澄清的粗提取物如图2a-2b所述进行制备。然后将25μl的ni和25μl的ic依次混合到有补充剂的洗涤剂中,或者混合到对照中没有补充剂的洗涤剂中,并在20℃下孵育制剂以将样品暴露于抑制条件下过夜。在洗涤剂抑制试验中,将样品稀释20x至具有2x补充剂的洗涤剂中,除了对照是将样品稀释20x至不含补充剂的洗涤剂中。在稀释诱导试验中,稀释20x至去离子水中。通过将100μl的稀释的样品与100μl的faaf-pna底物(500μm)混合并在动力学试验中在400nm处读取吸光度60分钟(图3e,多元醇补充剂)或者直到最高活性样品的吸光度达到接近~2.8的试验线性范围的最大值,测定三个重复样品中的蛋白酶活性,并且如图3a-3e中所示绘制洗涤剂和水中的活性。
经将洗涤剂制剂的ni和ic稀释至水中,各种盐,包括具有各种抗衡离子包括氯离子、硫酸盐、硝酸盐、酒石酸盐、琥珀酸盐、二琥珀酸盐、亚氨基二琥珀酸盐、乳酸盐、柠檬酸盐、聚天冬氨酸盐、聚丙烯酸酯的碱金属(锂、钠、钾)、铵,增加了iprotease活性。当以不同浓度加料时,较高的剂量通常导致更高的活性。
以摩尔计,具有更多正电荷的聚合盐以比单价盐(>0.25m)低得多的浓度(0.02-0.0375m)证明是稀释诱导型iprotease活性的有效增强子。聚丙烯酸钠7k(平均mw7300)和聚丙烯酸酯2k(平均mw2100),每分子聚合物分别携带约100和30个钠抗衡离子。聚天冬氨酸钠,每分子聚合物类似地携带约35个钠阳离子。
列出的电解质和聚电解质是可能适合于控制洗涤剂制剂的ni和ic的活性的化学品的非限制性实例。多元醇,如neodol25-7代表了另一类具有相似特性的潜在添加剂。
现代织物洗涤剂是浓缩的结构化液体。对这种制剂的补充剂的选择将满足多种标准,例如,抑制未稀释的浓缩物中内含肽修饰的蛋白质的活性,经从内含肽修饰的部分稀释时提高酶活性,与液体制剂的相容性,适合作为部分的家庭消费品的目的,包括成本标准。
可想而知地,可能影响制剂的反式剪接的蛋白的总体电荷、电荷分布、水合特性的和/或通过洗涤剂中的其他方式改变蛋白缔合和聚集的补充剂可以对制剂的反式剪接的ni和ic的稀释诱导型iprotease活性产生影响。
本文描述的洗涤剂抑制和稀释诱导试验能够快速筛选这些效应物,并且可以支持用于内含肽修饰的蛋白的活性控制的制剂开发。
实施例5.稀释诱导型iprotease活性的测试效应物
效应物在不同的洗涤剂环境中表现相似
发现在洗涤剂-2中在用于活性增强作用的效应物筛选中鉴定的补充剂与在洗涤剂-1中表现相似。图4a-4b示出了效应物在不同的洗涤剂环境中表现相似。特别地,这些图比较了定量蛋白酶试验中两种洗涤剂中三种小分子效应物,烟酸钠(90mm)、nacl(1m)和kcl(500mm)的稀释诱导特性。试验基本上与筛选相同,但是使用纯化的蛋白代替细胞裂解物,并且在动力学试验中以连续的两倍稀释度测定活性。简言之,将相同摩尔浓度的反式剪接的前体依次加入到盐补充剂的洗涤剂中,在20℃下孵育过夜以使剪接进行,然后将等份样品在试验缓冲液中连续稀释20至1280倍(稀释诱导试验),或稀释20倍至相应的补充剂的洗涤剂中(洗涤剂抑制试验),并通过在动力学试验中测量400nm处的吸光度,使用suc-faaf-pna(500μm)底物测试蛋白酶活性。
参考图4a-4b,洗涤剂-2和洗涤剂-1使用烟酸钠(90mm)或nacl(1m)或使用kcl(500mm)补充,加入ni和ic,并如下比较稀释诱导型iprotease活性:
制剂:将20μl的合适的盐加入至pcr管中的66.7μl的洗涤剂-2或洗涤剂-1中,对照组加入h2o,并通过离心沉淀和涡旋进行混合。将4.42μl的在mtsb缓冲液(50mmtris、150mmnacl、1mmdtt、2mmcacl2,ph=7.5)中的纯化的ic加入到含盐的洗涤剂中,充分混合和搅拌后,将8.86μl的在mtsb缓冲液中的纯化的ni混入到洗涤剂+ic中。ni和ic以等摩尔浓度组合。制剂在20℃下储存过夜以允许剪接的继续进行。
洗涤剂稀释试验如下。在透明的平底96孔板(corning9017,costar)的a行孔中预先负载20μl的1xbr缓冲液ph9.0和160μl的h2o。b-g行预先负载10μl的1xbr缓冲液ph9.0、90μl的h2o。将20μl的制剂加入到a行中,通过移液混合,然后将100μl的该10x稀释的样品加入到b行中的100μl的水和缓冲液中并混合。这导致20x稀释。对于c-g行重复该2x连续稀释过程,从g行中弃去过量的100μl,每孔中最终体积为100μl。a行中的最终洗涤剂浓度,20倍稀释,为3.3%。
洗涤剂抑制试验(在测定中为1:20)如下。在与上述相同的板中,h行预先负载10μl的1xbr缓冲液ph9.0、80μl的100%洗涤剂。将10μl的制剂单独加入到h行中的每个孔中并混合。在抑制试验中20倍稀释的最终洗涤剂浓度为43.3%。
对于两种试验(对于整个96孔板),加入100μl的2xsuc-faaf-pna(在20%dmso中500μm)底物,通过在30℃下读取400nm处的吸光度来测试蛋白酶活性。
在抑制试验中的洗涤剂最终浓度为43.3%,在20倍水稀释试验中为3.3%。参考图4a,洗涤剂-2中的稀释诱导型iprotease使用烟酸钠(90mm)或nacl(1m)或使用kcl(500mm)进行补充。参考图4b,洗涤剂-1中的稀释诱导型iprotease使用烟酸钠(90mm)或nacl(1m)或使用kcl(500mm)进行补充。
参考图4a-4b,与其无盐对照相比,补充盐的洗涤剂在稀释制剂的ni和ic后总是产生更高的活性。参考图4b,在洗涤剂-1中可以实现稀释增强而不失去抑制。然而,可能由于两种洗涤剂的iprotease活性抑制力的固有差异,回收的总活性显著不同,有利于洗涤剂-2(图4b)。这些结果验证了洗涤剂添加剂筛选的效用,以揭示稀释诱导型酶活性的影响因素,并且表明至少一些盐可以是独立于洗涤剂环境的洗涤剂制剂的ni和ic的有效诱导剂。
通过内含肽修饰的前体的缔合引发反式剪接。对于来自集胞藻属(synechocystissp.)pcc6803的dnae分割内含肽的研究表明分割内含肽部分是无序的并且经缔合经历了从无序到有序的构象迁移(zhengy.等人,2012,其通过引用并入本文,如同完整阐述)。最近关于点型念珠藻的模型dnae分割内含肽的缔合的分子机制的报道证实了这一假设,并且显示内含肽部分通过由静电相互作用引发的多步骤过程相互作用,随后将初始无序的内含肽部分压实并稳定到n-内含肽的预折叠区域(shahnh.等人,2013,其通过引用并入本文,如同完整阐述)。这种“捕获和折叠(collapse)”的机制在天然分割的内含肽中可以是常见的,并且可以对影响分子的电荷、电荷分布和水合特性的效应物敏感。
因此,调查了在效应物筛选中鉴定的盐的离子强度的变化对于直接控制不依赖于洗涤剂环境的稀释诱导型iprotease活性的影响以及测试的kcl。
实施例6.kcl具有双重作用,促进剪接的前体的缔合并抑制剪接
反式剪接需要前体的缔合以引发最终产生活性酶的多步骤剪接反应(wuh.等人1998;martin等人,2001;perlerfb.2005,其全部通过引用并入本文,如同完整阐述)。为了更好地理解kcl在什么阶段可以发挥作用,在剪接开始前,将kcl(0.125、0.25、0.5、1.0和2.0m)加入到剪接的前体中,保持ni和ic恒定,并且在剪接完成后在剪接的混合物孵育2小时后加入2m的kcl。
使用0.125、0.25、0.5、1.0和2.0m的浓度递增的kcl分别处理ni和ic的细胞裂解物,并在混合前体之前孵育10min/rt,并孵育额外的2hrs./rt以允许在递增浓度的盐中进行剪接。将样品分割成两等份,一份稀释至br缓冲液ph9.0中(稀释诱导试验),另一份稀释至高盐中(br缓冲液ph9.0中的2m的kcl;盐抑制试验)并测试iprotease。
图5a-5b示出了kcl在含水缓冲液中可逆地抑制iprotease活性。参考这些图,从4m至0.03125m以2倍连续稀释4m的kcl。将两组15μl等份样品分配到pcr管中。一组,加入15μl澄清的ni细胞裂解物,另一组加入15μl澄清的ic细胞裂解物,并将管在室温下孵育10min。预盐化的ni和ic在相同浓度的kcl中混合并在室温下孵育10分钟。图5a示出了稀释诱导试验。参考该图,将连续稀释的kcl中的恒定量的(ni+ic)预混物以20倍稀释至br缓冲液中,并如下测试iprotease活性:将5μl的(ni+ic)预混物加入到95μl的br缓冲液ph9.0中,然后加入100μl的suc-faaf-pna(500μm)底物,并在30℃下在400nm处读取吸光度。
图5b示出了盐抑制试验。参考图5b,将连续稀释的kcl中的恒定量的(ni+ic)预混物以20倍稀释至2mkcl中,并如下测试iprotease活性。在br缓冲液ph9.0中,将5μl的(ni+ic)预混物加入到95μl的2mkcl中,然后加入100μl的suc-faaf-pna(500μm)底物,并在30℃下在400nm处读取吸光度。观察到稀释诱导试验重现了洗涤剂环境中稀释诱导的盐增强作用。参考图5a,观察到在混合之前加入到前体中的盐越多,稀释液中iprotease活性越高。2mkcl中的盐抑制试验揭示了盐浓度和活性之间的另一个相关性。在混合前体之前盐初始浓度高(0.5-2.0mkcl)且在试验中维持高浓度(2.0mkcl)的样品中,iprotease活性被抑制。在混合前体之前盐初始浓度低(0.015-0.025mkcl)的样品中,高盐(2mkcl)中iprotease活性增加,虽然低于其被稀释至缓冲液中的相应等份样品(比较图5a与图5b中相同盐浓度缓冲液的酶活性)。
似乎存在高盐调节的酶活性的转变,其需要在开始剪接之前使用高盐(0.5-2.0mkcl)预处理前体并将盐稀释至低于0.5mkcl以恢复活性。在剪接反应后期加入高盐(2.0mkcl)在很大程度上是无效的,但是在整个剪接反应中保持在高水平(≥0.5mkcl)时是抑制的。
由于反式剪接需要前体缔合用于启动剪接,因此假设kcl可以以两种不同的方式影响该缔合:以浓度依赖性方式可逆地增加缔合但抑制剪接。
反式剪接的蛋白的缔合是浓度驱动的反应,可以想象,其可以通过稀释前体而减少。为了确定2mkcl是否可以促进缔合,将前体在2mkcl中的(ni+ic)预混物中组合,然后在2mkcl中对其进行依次稀释。或者,首先在2mkcl中分别稀释ni和ic,然后将依次稀释的ni与依次稀释的ic组合以产生ni-ic混合物。然后测试样品的稀释诱导型iprotease。
图6a-6b示出了高浓度的kcl促进剪接前体的缔合。图6a示出了br缓冲液中高盐相关的(ni+ic)预混物的连续稀释中的iprotease活性。图6b示出了br缓冲液中ni和ic(ni-ic)的单独的连续稀释的混合中的iprotease活性。
参考图6a,(ni+ic)预混物在所有8个连续稀释中产生iprotease活性,并且活性与预混物中ni和ic的依次减少的量成比例。参考图6b,相反地,在ni-ic混合物的稀释中,iprotease活性在三个稀释步骤后迅速降低至检测水平以下,表明ni和ic浓度降低至缔合和启动剪接所需的水平以下。这些结果与使用高浓度的kcl的剪接前体的预处理促进剪接前体在(ni+ic)预混物中的缔合的解释一致。参考图6a-6b,试验设置如下。设置三组管:对于一组8个pcr管,标记为(ni+ic)1-8,分配40μl的2mkcl。对于分别标记为ni1-8和ic1-8的两组8个pcr管,等量加入20μl的2mkcl。
(1)将50μl澄清的ni裂解物与50μl的4mkcl混合,并将50μl的澄清的ic裂解物与50μl的4mkcl混合,并在室温下孵育10min。
(2)将20μl的预盐化的ni和20μl的预盐化的ic混合到管(ni+ic)1中。对于对照,将20μl预盐化的ni加入到管ni.1中;并将20μl预盐化的ic加入到管ic.1中并混合。
(3)然后将40μl的(ni+ic)1加入到标记为(ni+ic)2的管中并混合;将20μl的ni.1加入到ni.2管中,并将20μl的ic.1加入到ic.2管中,并混合。重复这些步骤以产生多达(ni+ic)1-8、ni1-8和ic1-8的逐步稀释。然后将ni1与ic1、ni2与ic2混合,以构建ni-ic1-8组。
(4)(ni+ic)和ni-ic组在室温下孵育10min。
(5)为了测试稀释诱导型iprotease活性,将5μl等份的(ni+ic)或ni-ic与95μl的br缓冲液ph9.0混合,然后加入100μl的suc-faaf-pna(500μm)底物,并且在30℃下在动力学试验中在400nm处读取吸光度。
图6a示出了在稀释至br缓冲液的高盐相关的(ni+ic)预混物的连续稀释中的iprotease活性。
图6b示出了在br缓冲液中ni和ic(ni-ic)的单独的连续稀释的混合中的iprotease活性。
为了确定iprotease是否通过剪接或经由缔合驱动的蛋白互补产生,对剪接进行了评价。由于考虑到剪接产物可能难以检测,因为其具有蛋白酶活性并且可能自动降解。因此,为了保护剪接产物免受蛋白水解的自动降解,丙氨酸突变了ic中的iprotease的丝氨酸催化残基(s326a)。通过混合等体积的大肠杆菌表达的剪接前体的粗裂解物来测试剪接。制备两种混合,前体进行或不进行盐处理。盐处理是通过在混合前体之前分别混合等体积的ni和ic与等体积的2mkcl。在无盐样品中,在混合之前将等体积的h2o加入到每个前体中。将(ni+ic)和(ni+ics326a)的未盐化的和盐化的混合快速稀释至含水缓冲液中,并在t=0、1min和30min取等份样品到负载染料+5%βme的2xsds中,并在考马斯染色的12%sds/page上分析总蛋白图谱。在稀释至含水缓冲液中后,盐预处理的(ni+ic)混合中的残留的kcl浓度为200mm。
在未盐化的(ni+ic)和(ni+ics326a)的稀释中,在t=30min的剪接反应的晚期,新的蛋白种类以期望的大小聚集用于剪接产物。在(ni+ics326a)的蛋白酶失活的盐化的前体混合中,剪接产物在t=0时立即可见,并保持在相同的高水平,并且没有蛋白水解降解的迹象。在野生型(ni+ic)的盐化的前体混合中,剪接产物检测由于即时蛋白降解而减弱,并且大部分的蛋白由t=30min降解。这些观察结果与将前体混合导致剪接并且在混合前具有等体积的2mkcl的前体的预处理导致剪接和超快速蛋白酶活性的解释一致。
图7a-7b示出了考马斯凝胶在剪接前体ni和ic混合后显示剪接。参考这些图,在剪接前体混合后并在蛋白图谱在考马斯染色的12%sds/page上超过30分钟后评价剪接。为了保护剪接产物免于蛋白水解的自动降解,ic中的丝氨酸催化残基被丙氨酸突变以产生(ics326a)。如图2a(总蛋白图谱)和图2b(可溶性蛋白图谱)所示,制备ni、ic和ics326a的2x浓缩的细胞裂解物。为了制备盐化的预混物,将ni或ic或ics326a分别与等体积的2mkcl混合。在室温下10min后,将1mkcl中的25μl的ni与1mkcl中的25μl的ic混合,并且在另外的样品中将25μl的ni与25μl的ics326a混合。混合后立即将20μl(ni+ic)和(ni+ics326a)用补充有10μl的br缓冲液ph9.0的70μl去离子水稀释,并且在t=0时间点立即取20μl等份样品加入到20μl的负载染料+5%βme的2xsds中,并且样品在95℃/5min加热。进一步地,稀释后1min和30min取出等份样品,并进行类似处理。在加入负载染料之前,稀释样品中残留的kcl浓度为200mm。在12%sds/page上分离15μl样品,并用考马斯染色凝胶。为了制备未盐化的预混物,样品按相同的方案处理,但代替2mkcl,将h2o加入到2x浓缩的细胞裂解物中。图7a示出了将未盐化的预混合物稀释到含水缓冲液中后的蛋白图谱。参考该图,在前体混合后30min至开始剪接,新的蛋白种类以期望的大小聚集在(ni+ic)和ni+ics326a中用于剪接产物。图7b示出了将盐化的预混物稀释至含水缓冲液中后的蛋白图谱。参考该图,在(ni+ic)样品中,在t=0时间点开始剪接后蛋白降解立即可见,并且到t=30min时,大部分蛋白被降解。在蛋白酶失活的(ni+ics326a)预混物中,没有可见的蛋白降解,并且在t=0时检测到大量的剪接产物。标记ni和ic和ics326a的位置。用箭头标记剪接产物的位置。
kcl似乎是ni和ic的有效效应物,促进剪接前体缔合成剪接失活形式,在浓度高于0.5m时抑制iprotease活性,这可以通过降低kcl浓度在0.5m以下来可逆。通过这种措施,kcl是反式剪接调节的iprotease的抑制剂和条件调节剂。
由于包括两个内含肽半段之间的无序结构和电荷分布的共有的结构特征以及反式剪接的内含肽之间缔合的共有机制,分割的内含肽修饰的蛋白片段的高盐控制的缔合可能不限于ni和ic,还可适用于其他分割的内含肽修饰的蛋白,并且可用于组装用于受控活性的两个或多个反式-内含肽修饰的蛋白片段。(zhengy.等人,2012;shahnh.等人,2013,arankoas.等人,2013,所有这些都通过引用并入本文,如同完整阐述)。基于条件调节的盐,如通过kcl调节的iprotease所示,可以提供简单且可能更通用的方法来调节用于受控活性的反式剪接的内含肽修饰的蛋白。该策略的主要优点是依赖于野生型反式剪接的内含肽,并避免了用于靶蛋白活性的条件调节的反式剪接的内含肽的通常进行困难的遗传工程改造(mootz等人,2003;selgrade等人,2013,其通过引用并入本文,如同完整阐述)。
用于稀释诱导型iprotease的效应物筛选显示不同化学特性的化合物、电解质、聚电解质、多元醇、水溶助长剂(甘油、单丙二醇、非离子的exshell(neodol)可以是效应物。可以想象,具有相似理化特性的化学品也可以表现相似。因为效应物似乎比剪接的抑制剂zn++、cu++或cistatin更多且具有不同的化学特性,当需要条件调节靶蛋白的活性时,可能它们可以更好地适应具体应用的需求,包括极端的化学条件(nicholsnm.等人,2003;sunp.等人,2005;zhangl.等人,210;zhangl.等人,2011,所有这些都通过引用并入本文,如同完整阐述)。
实施例7-16描述了在液体洗衣洗涤剂制剂中使用洗涤剂稀释诱导型iprotease活性的效应物,用于稳定储存和蛋白酶活性的稀释诱导的有效恢复。实施例描述了将kcl稳定的(ni+ic)预混物配制成水溶液和液体洗衣洗涤剂,用于稳定储存和蛋白酶活性的快速有效恢复,而不损失清洁效能。
实施例7.稀释诱导型iprotease对savinase的基准
在mtsb缓冲液(50mmtris、150mmnacl、1mmdtt、2mmcacl2,ph7.5)中纯化的ni母液为45mg/ml(855.9微摩尔)。在mtsb缓冲液中纯化的ic母液为33.58mg/ml(1460微摩尔)。iprotease活性基准为1%(vol/vol)savinase(~103g/l,novozymes),纯化的ni和ic的每一种均以38.1μmol浓度使用。用于7x200μl体积制剂的(ni+ic)预混物制备如下:将6.23μl的100mmdtt加入到eppendorf管中的62.3μl的ni中。在室温(rt)下10min后,通过混合加入等体积(68.53μl)的4mkcl,并将样品在室温下孵育10min。将3.64μl的100mmdtt加入到eppendorf管中的36.4μl的ic中并混合。在室温下10min后,加入等体积(40.0μl)的4mkcl,并将样品在室温下孵育10min。为了在2mkcl中制备4x浓缩的(ni+ic)预混物,将在2mkcl中的137μl的ni与2mkcl中的80μl的ic混合,通过移液器混合。在室温下10min后,加入133μl的2mkcl,得到350μl(ni+ic)预混物(4x)。在含水制剂中,将50μl的4x浓缩的(ni+ic)预混物加入到150μl去离子水+kcl中;kcl最终浓度为1m。当制剂被制成洗涤剂时,将50μl的4x浓缩的(ni+ic)预混物加入到150μl的kcl补充的洗涤剂中。制剂中的kcl最终浓度为1m,ni和ic最终浓度各自为38.1μmol。
savinase(~103g/l,抑制剂稳定的母液)基本上被kcl处理为如下的ni和ic。将savinase在mtsb缓冲液中稀释3倍。向mtsb中的60μl的savinase中加入190μl去离子水和250μl的4mkcl,以在2mkcl中制备500μl的4x浓缩的母液,足够用于1%(v/v)savinase的10x200μl制剂。在含水制剂中,将2mkcl预混物中的50μl的4xsavinase加入到150μl去离子水+kcl中;kcl最终浓度为1m。当制剂制成洗涤剂时,将2mkcl预混物中的50μl的4xsavinase加入到150μl的kcl补充的洗涤剂中。savinase为1%,在制剂中的kcl最终浓度为1m。
在96孔板(corning9017,costar,透明的平底)中进行蛋白酶试验。a行孔预先负载20μl1xbr缓冲液ph9.0和160μl的h2o。b-h行预先负载10μl1xbr缓冲液ph9.0、90μl的h2o。将20μl的制剂加入到a行,通过移液器混合,然后将100μl的该10x稀释样品加入到b行中的100μl缓冲液和水中并混合。这导致20倍稀释。对c-h行重复2x连续稀释的这个过程。这是洗涤剂稀释试验。向孔中的100μl样品中加入100μl的2xsuc-faaf-pna(在20%dmso中500μm)底物,并通过在30℃下读取400nm处的吸光度来测试iprotease活性。
图8a-8b示出了稀释诱导型iprotease活性的基准为1%savinase。参考这些图,观察到iprotease(图8a)和savinase(图8b)具有可比的稀释活性图谱。结果证实了具有超过95%效能的iprotease活性的稳健活性和超快速恢复。
实施例8.kcl稳定的(ni+ic)预混物无蛋白酶活性
iprotease活性基准为1%(v/v)savinase。至1mkcl含水缓冲液的(ni+ic)和savinase的制剂与实施例1相同,但在2mkcl中的酶活性测试如下:在透明的平底半容量96孔板中,将a行孔预先负载10μl的1xbr缓冲液ph9.0、50μl的4mkcl和35μl的h2o。b-h行预先负载5μl的1xbr缓冲液ph9.0、25μl的4mkcl和20μl的h2o。将5μl(ni+ic)制剂加入到a行,通过移液器混合,然后将50μl的该20x稀释的样品加入到b行的50μl盐、缓冲液和水中并混合。这些导致40x稀释。对c-h行重复2x连续稀释的过程。通过加入50μl的500μmsuc-faaf-pna(在20%dmso中)底物,将所有样品稀释额外的2x,产生从a行中的40x稀释到h行中的5120x的样品。通过在30℃下在400nm处读取吸光度来测试蛋白酶活性。图9a-9b示出了kcl稳定的(ni+ic)预混物无酶活性。图9a示出了80倍稀释的(ni+ic)预混物的iprotease活性。图9a示出了savinase。观察到1mkcl抑制(ni+ic)预混物的蛋白酶活性,但不抑制内含肽未修饰的参考酶savinase的蛋白酶活性。
实施例9.kcl稳定的(ni+ic)预混物可以储存在含水缓冲液中,而不显著损失活性
如实施例1在2mkcl中制备4x浓缩的(ni+ic)预混物,并在4℃下储存。将4x(ni+ic)预混合物在t=0时配制到含水缓冲液中并且在4℃下储存8周后,并如实施例8所述测试iprotease活性。图10a-10b示出了kcl稳定的(ni+ic)预混物在含水缓冲液中的储存稳定性。参考这些数据,在t=0和t=8周时稀释诱导活性的比较表明稀释诱导型iprotease活性没有损失。在t=8时略高的活性可能是由于试验中的环境温度差异。诱导活性的保留显示了高储存稳定性。
实施例10.可以将kcl稳定的(ni+ic)预混物配制成洗涤剂-1,而不损失稀释诱导活性
这个试验评价了洗涤剂-1制剂的kcl稳定的(ni+ic)预混物的稀释诱导效能。如实施例8所述,纯化的(ni+ic)预混物以基准浓度制备,并将等份样品配制到具有1mkcl的含水缓冲液中或配制到补充有1mkcl的50%洗涤剂-1中。在从80至10240倍稀释至含水缓冲液中的顺序稀释中测量iprotease活性。图11a-11b示出了可以将kcl稳定的(ni+ic)预混物配制成洗涤剂-1,而不损失稀释诱导活性。图11a显示了来自1mkcl含水缓冲液中的稀释活性。11b显示了来自50%洗涤剂-1-1mkcl中的稀释活性。洗涤剂-1制剂的预混物保留了全稀释诱导性。稀释诱导是超快速的并且是高效的。
实施例11.kcl在洗涤剂配制中具有双重作用:稳定(ni+ic)预混物用于配制并维持洗涤剂中的稳定性
图12示出了洗涤剂配制ni和ic的替代方案的比较,并评价了长期稳定性的配制。比较如下的两种配制方案:1)通过将未盐化的ni和ic依次混合到洗涤剂中的配制(洗涤剂+ni+ic),以及2)将kcl稳定的(ni+ic)预混物加入到洗涤剂中的配制。洗涤剂-1以60%(v/v)在两种形式中使用,无需补充(-),或将kcl补充剂加入至1m终浓度(+)。纯化的ni和ic每种均以对于1%(v/v)savinase的基准浓度以每种38.1μmol浓度使用。基本如实施例7所述,制备(ni+ic)预混物并配制到洗涤剂中。制剂在37℃下在加速老化条件下储存,并且在储存中的t=0、1、3、6和8周时通过稀释诱导型iprotease活性超过8周的测试保留来测试储存稳定性。如实施例7所述,在对试验缓冲液从40至5120倍稀释的顺序稀释中测试稀释诱导活性。将1:1280倍稀释的在2min时间点的iprotease活性读数标准化为t=0时无盐的洗涤剂中的(ni+ic)预混物的稀释诱导型iprotease活性,并制图。
结果证实配制1mkcl稳定的(ni+ic)预混物的优点多于通过将ni和ic依次混合到洗涤剂中的配制,并且显示了在洗涤剂中具有1mkcl补充剂对于长期储存的益处。在通过将ni和ic依次混合到不含盐补充剂的洗涤剂中制备的制剂中,iprotease活性低并且快速下降。用1mkcl补充洗涤剂增强活性约3倍,但诱导活性随时间逐渐下降。
(ni+ic)预混物的制剂在t=0时导致高的初始活性,但在未补充的洗涤剂中储存稳定性较弱,并且稀释诱导活性的保留逐渐下降,超过8周约下降50%。当将kcl稳定的(ni+ic)预混物配制到1mkcl补充的洗涤剂中时,获得最佳结果。这种制剂保持稀释诱导的全部活性超过3周,并且在超过8周保持约80%的活性,表明1mkcl在反式剪接的前体的配制中和作为洗涤剂添加剂都是重要的,以维持稀释诱导活性的可逆抑制。
图12示出了kcl在洗涤剂配制中具有双重作用:稳定(ni+ic)预混物用于配制并维持洗涤剂中的稳定性。
实施例12.在加速老化条件下,在洗涤剂-1中,iprotease稳定性与savinase相当
savinase以1%(v/v)使用并配制到补充有1mkcl的60%洗涤剂-1中。纯化的ni和ic以相对于1%(v/v)savinase的等摩尔浓度各自以38.1μmol浓度使用。制备(ni+ic)预混物并配制到补充有kcl至1m终浓度的kcl的60%洗涤剂-1中,如实施例11所述。制剂在37℃下在加速老化条件下储存,并且在储存中的t=0、1、3、6和8周时通过稀释诱导型iprotease活性超过8周的测试保留来测试储存稳定性。图13示出了在加速老化条件下,洗涤剂-1中的iprotease储存稳定性与savinase相当。参考该图,如前所述在顺序稀释至试验缓冲液中测试蛋白酶活性,并将1:1280倍稀释的2min时间点的iprotease和savinase活性读数各自标准化为其t=0时作为100%,并且用曲线图表示残留活性。
iprotease的残留活性图谱与savinase相当。两种制剂在37℃下8周后,在加速老化条件下保持约75-80%的活性。结果证实,反式剪接的内含肽技术可以有效地用于液体洗衣洗涤剂中的蛋白酶稳定。
实施例13.kcl稳定的(ni+ic)预混物与四种不同类型的洗涤剂相容
这个试验评价了用于稀释可诱导型iprotease活性的四种不同类型的液体洗衣洗涤剂:洗涤剂-1、洗涤剂-2、洗涤剂-3和洗涤剂-4。洗涤剂是无酶制剂(洗涤剂-1、洗涤剂-4)或具有通过热处理而失活的配制的蛋白酶的商业品牌洗涤剂(洗涤剂-2、洗涤剂-3)。
iprotease基准为1%(v/v)savinase。如实施例7所述制备kcl稳定的(ni+ic)预混物和1mkcl补充的50%洗涤剂的制剂。使用如实施例8中的相同方案将savinase以1%(v/v)配制到1mkcl补充的50%洗涤剂中。图14示出了洗涤剂配制的kcl稳定的(ni+ic)预混物在不同类型的洗涤剂:洗涤剂-1、洗涤剂-2、洗涤剂-3和洗涤剂-4中保留了完全稀释诱导活性。参考该图,如前所述在顺序稀释至试验缓冲液中测试蛋白酶活性,并用曲线图表示2min时2560倍稀释的iprotease和savinase活性读数。iprotease和savinase活性在所有四种洗涤剂中都是相当的,表明kcl稳定的(ni+ic)预混物的稀释诱导与洗涤剂大体上无关。
实施例14.在四种液体洗衣洗涤剂中iprotease的去污效果与内含肽未修饰的酶savinase相当
评价被制成四种不同类型的洗涤剂的并用于测定蛋白酶活性的制剂(实施例13)的去污活性如下:
污渍布标准如下。瑞士标准局测试织物,empa117用于测试。这种织物是65/35的涤/棉混合物,用血液/牛奶/墨水弄脏。大约重量:165g/m2(4.86盎司/码2)。大约宽度为58英寸(147cm)。将织物圆片冲压出以适合标准的96孔微孔板。冲头尺寸和织物圆直径为5.56mm(7/32;0.22英寸),面积为24.25mm2。
试剂如下:在h2o(1000ml)中120fh缓冲液(法国硬度单位,10x)8mm氯化钙(0.887gcacl2)+4mm氯化镁(0.813gmgcl2·6h2o);200mm硼酸(10x)。在1l的h2o中12.37g硼酸;用naoh将ph调至9.0;和h2o中1.25%洗涤剂。
洗涤:在三个重复的织物片上进行洗涤。在50%洗涤剂母液(dh2o中的洗涤剂)中进行制剂的酶(iprotease和savinase)稀释。对于每种制剂用dh2o进行1:40稀释。按照以下顺序将试剂加入到含有血染色布盘的平底96孔板中:h2o中1.25%洗涤剂20μl;200硼酸20μl;120fh6μl;h2o134μl;和h2o中2.5%制剂20μl。试验总体积为200μl。用箔覆盖96孔板,并在37℃下以200rpm振荡孵育1小时。除去箔,并移去100μl的上清液,然后除去所有液体。
漂洗:将带有被染色的片的板每孔用200μl的dh2o再填充,并用箔密封。使用设定为900rpm的multitron振荡器进行洗涤。将密封板置于平台上并关门,使振荡器旋转起来直至达到890rpm(约60秒),然后移除板。漂洗水被去除并丢弃。共进行3次洗涤。
白光反射率读取。待测织物圆片的直径为5.56mm(面积为24.25mm2)。其中,用于测量白光反射率的内圆的直径约为4.16mm,对应于40像素(面积13.64mm2,对应于1264像素)。而通过对应于测量面积的16位/像素灰度图像来量化光反射率。将成像器预标准化用于成像器像素上的任何光源变化。
图15示出了四种洗衣洗涤剂中洗涤剂配制的iprotease的去污效果与savinase相当。该图显示了洗涤的empa117(血液/牛奶/墨水染色的)织物的白光反射率。每个图是三种被洗涤的织物的反射率的平均值。在每种洗涤剂测试中,iprotease去污与savinase相当。
实施例15.在加速老化条件下,三种洗涤剂中稀释诱导型iprotease活性的稳定性
洗涤剂为:洗涤剂-1、洗涤剂-2和洗涤剂-3。对于基准,使用1%(v/v)savinase,但是不是配制抑制剂稳定的形式,首先使用tube-o-dialyzermedi(4000da;gbiosciences)在4℃下通过使用5lmtsb缓冲液对0.5mlsavinase进行过夜透析的三个步骤去除抑制剂。将mtsb中透析的savinase的体积调整为1.5ml,并使用等体积的4mkcl处理的3μl透析等份样品配制100μl洗涤剂以得到1%(v/v)的savinase。样品处理基本上与实施例8中相同,除了将制剂配制成补充有kcl至1m终浓度的65%洗涤剂。
对于1%(v/v)透析的savinase,以等摩尔浓度使用纯化的ni和ic,各自为38.1μmol浓度。如实施例7中所述制备2mkcl中的(ni+ic)预混物,并将制剂制成补充有kcl至1m终浓度的65%洗涤剂。
洗涤剂制剂在37℃下在加速老化条件下储存。在t=0、1、2、3和4周的5个时间点,如前所述在连续稀释至试验缓冲液中测试残留蛋白酶活性。将每种制剂的1:2560倍稀释的2min读数标准化为t=0作为100%并用曲线图表示。图16示出了在加速老化条件下三种洗涤剂中稀释诱导型iprotease活性的稳定性。数据显示,在三种洗涤剂测试的每种中,以相等剂量的蛋白酶,在加速老化条件下,iprotease相比于不含抑制剂稳定剂的savinase保持更多的活性。
实施例16.加速老化条件下三种洗涤剂中去污活性的稳定性
将实施例15制备的制剂在37℃下在加速老化条件下储存,并且在储存中在t=0、1、2、3和4周的五个时间点评价去污活性。
如实施例15所述,在洗涤测试中使用empa117的血液/牛奶/墨水染色的织物。图17示出了在三种洗涤剂中加速老化条件下的去污稳定性。在每个时间点来自每种制剂进行三次重复洗涤。参考该图,洗涤的empa117织物的白光反射率被标准化为其对应的t=0样品并且用曲线图表示。
参考图16,iprotease在三种洗涤剂的每种中均保留了全部去污活性,与稀释诱导活性的稳定性一致。savinase,没有稳定剂,已经损失初始酶活性的~25-70%之间,也显示出完全去污活性。可能的是,在该试验的范围内,蛋白酶的剂量高于实现最大去污所必需的量。
实施例17.嗜热的顺式内含肽分裂为反式内含肽
嗜热的顺式-内含肽被人工分割为反式剪接的内含肽对。下面按照如newenglandbiolabinteindatabase(http://www.neb.com/neb/inteins.html)中的顺式内含肽的名称列出人工分割的内含肽对的氨基酸序列,随后是剪接的结构域说明符(-n或-c)。分割内含肽部分的缩写标识符在括号中。
1.cbu_dnab(12)n-内含肽(seqidno:16)和cbu_dnab(12)c-内含肽(seqidno:17)
2.mja_gf-6p(44)n-内含肽(seqidno:18)和mja_gf-6p–c(44)c-内含肽(seqidno:19)
3.mja_hyp-1–n(#46-n)(seqidno:20)和mja_hyp-1–c(#46-c)(seqidno:21)
4.mja_if2–n(#47-n)(seqidno:22)和mja_if2–c(#47-c)(seqidno:23)
5.mja_pol-1–n(#50-n)(seqidno:24)和mja_pol-1–c(#50-c)(seqidno:25)
6.pab_cdc21-1–n(#79-n)(seqidno:26)和pab_cdc21-1–c(#79-c)(seqidno:27)
7.pab_if2–n(#81-n)(seqidno:28)和pab_if2–c(#81-c)(seqidno:29)
8.pab_vma-n(#92-n)(seqidno:30)和pab_vma-c(#92-c)(seqidno:31)
9.pho_if2–n(#103-n)(seqidno:32)和pho_if2–c(#103-c)(seqidno:33)
10.pho_vma-n(#110-n)(seqidno:34)和pho_vma-c(#110-c)(seqidno:35)
11.rma_dnab–n(#116-n)(seqidno:36)和rma_dnab–c(#116-c)(seqidno:37)
12.sru_dnab-n(#123-n)(seqidno:38)和sru_dnab-c(#123-c)(seqidno:39)
13.tag_pol-1_tsp-ty_pol-1–n(#128-n)(seqidno:40)和tag_pol-1_tsp-ty_pol-1–c(#128-c)(seqidno:41)
14.ter_rir1-4–n(#135-n)(seqidno:42)和ter_rir1-4–c(#135-c)(seqidno:43)
15.tko_if2–n(#143-n)(seqidno:44)和tko_if2–c(#143-c)(seqidno:45)
16.tth-hb27_dnae-2–n(#150-n)(seqidno:46)和tth-hb27_dnae-2–c(#150-c)(seqidno:46)
使用双顺反子表达盒进一步测试本文所述的反式剪接的内含肽用于蛋白酶活性的恢复。
实施例18.测试savinase中人工分割的内含肽用于蛋白酶活性的恢复的双顺反子表达盒的构建
将savinase在丝氨酸317aa残基(s317)处分割成两部分以产生prosav-n和sav-c外显肽。图18示出了双顺反子表达盒。参考图18,将反式剪接的内含肽-n(in)与产生ni的prosav-n的c-末端在框架中融合,同时将反式剪接的内含肽-c(ic)与产生ic的c-外显肽(sav-c)的n-末端丝氨酸(s317)残基在框架中融合。ni和ic组装成双顺反子表达盒,其编码ni,ni由两个终止密码子终止,随后是移码的核糖体结合位点(seqidno:48)和用于ic表达的前导序列。然后将组装的ni和ic克隆在pbluescript的ecori和xhoi位点之间并在大肠杆菌中表达。这种双顺反子构建体表达具有两个独立的翻译产物(ni和ic)的单一转录体。
实施例19.来自体外反式剪接的内含肽的蛋白酶活性
如上所述制备各种反式剪接的内含肽修饰的分割的savinase的双顺反子构建体。如前所述进行诱导孵育、细胞裂解物的制备和蛋白酶试验。使用测试内含肽gp41-1,之前已经证明,分割的savinaseni和ic单独没有蛋白酶活性,并且剪接失效的ni和ic的混合不能恢复蛋白酶活性。蛋白酶活性需要无活性蛋白酶部分ni和ic的反式剪接介导的连接以及完整蛋白酶序列(nc)的恢复。图19a和19b示出了iptg诱导后3小时来自体外反式剪接的蛋白酶活性。图19a示出了内含肽12、12、44、46、47、50、79、81和103的剪接后的蛋白酶活性。图19b示出了内含肽110、116、123、128、135、143和150的剪接后的蛋白酶活性。构建体之间的蛋白酶活性的变化反映了内含肽剪接中的序列环境特异性差异。
参考文献
arankoas.,oeemigjs.,iwaih.(2013)structuralbasisforproteintrans-splicingbyabacterialintein-likedomainproteinligationwithoutnucleophilicsidechains.febsj.,280(14):3256-69.
bestemank.,zevenbergenm.,heeringh.andlemays.(2004)directobservationofchargeinversionbymultivalentionsasauniversalelectrostaticphenomenon.phys.rev.lett.93,170802.
evanstc.,martind.,kollyr.,panned.,sunl.,ghoshi.,chenl.,liux-q.andxum-q.(2000)proteintrans-splicingandcyclizationbyanaturallysplitinteinfromthednαegeneofsynechocystisspeciespcc6803.j.biol.chem.275,9091-9094.
lyklemaj.(2009)questforion-ioncorrelationsinelectricdoublelayersandoverchargingphenomena.adv.colloidinterfacesci.147,205-213.
martin-molinaa.,quesada-pérezm.,galisteo-gonzálezf.(2003)lookingintoovercharginginmodelcolloidsthroughelectrophoresis:asymmetricelectrolytes.j.chem.phys.118,4183-89.
martindd.,xum-q.andevanstc.(2001)characterizationofanaturallyoccurringtrans-splicinginteinfromsynechocystissp.pcc6803.biochemistry,40,1393-1402.
mootzhd.,blumes.,tyszkiewiczab.andmuirtw.(2003)conditionalproteinsplicing:anewtooltocontrolproteinstructureandfunctioninvitroandinvivo.j.am.chem.soc.125,10561-9.
nicholsnm,bennerjs,martinddandevanstcjr(2003)zincioneffectsonindividulalsspdnaesplicingsteps:regulatingpathwayprogression.biochemistry,42(18),5301-11.
perlerfb.(2005)proteinsplicingmechanismsandapplications.iubmblife,57(7),469-476.
selgradedf.,lohmuellerjj.,lienertf.andsilverp.(2013)proteinscaffold-activatedproteintrans-splicinginmammaliancells.j.am.chem.soc.135,7713-19.
shahnh.,eryilmaze.,cowburnd.andmuirtw.(2013)naturallysplitinteinsassemblethrougha“captureandcollapse”mechanism.j.am.chem.soc.135,18673-81.
shij.andmuirtwj.(2005)developmentofatandemproteintrans-splicingsystembasedonnativeandengineeredsplitinteins.j.am.chem.soc.127,6198-206.
shij.andmuirt.w.(2006)developmentofatandemproteintrans-splicingsystembasedonnativeandengineeredsplitinteins.j.am,chem.soc.,2005,127(17),6198-6206.
sunp.,yes.,ferrandons.,evanst.c.,xum-q.,andraoz.(2005)crystalstructuresofaninteinfromthesplitdnaegeneofsynechocystissp.pcc6803revealthecatalyticmodelwithoutthepenultimatehistidineandthemechanismofzincioninhibitionofproteinsplicing.j.mol.biol.353,1093-1105.
topilinani.andmillskv.(2014)recentadvancesininvivoapplicationsofintein-mediatedproteinsplicing.mobiledna,(5)5.
trulssonm.,jonssonb.,akessont.andforsmanj.(2006)repulsionbetweenoppositelychargedsurfacesinmultivalentelectrolytes.phys.rev.lett.97,068302.
wuh.,huz,andliux-q.(1998)proteintrans-splicingbyasplitinteinencodedinasplitdnaegeneofsynechocystissp.pcc6803.proc.natl.acad.sciusa,95,9226-31.
zhangl.,xiaon.npany.,zhengy.,panz.,luoz.,xux.,liuy.(2010)bindingandinhibitionofcopperionstorecainteinsfrommycobacteriumtuberculosis.chemistry,16(14),4297-306.
zhangl,zhengy.,callahanb,bedfortm.,liuy.(2011)cisplatininhibitsproteinsplicing,suggestinginteinsastherapeutictargetsinmycobacteria.j.biol.chem.286(2),1277-82.
zhengy.,wuq.,xum-q.andliuy.(2012)mutualsynergisticproteinfoldinginsplitintein.biosci.rep.32,433-442.
本申请通篇引用的参考文献和参考文献本身以本文中全部显而易见的目的并入,如同每个参考文献被完整阐述。基于介绍的目的,这些参考文献中的特定文献引用在本文中的特定位置。在特定位置处引用的参考文献表示以该参考文献的教导的方式并入本文。然而,在特定的位置处引用参考文献并不限制以任何目的引用的参考文献的所有教导方式并入本文。
所以应当理解的是,本发明不局限于所公开的具体实施方式,而是意在覆盖在本发明的精神和范围内的,由随附的权利要求书、上述说明书和/或在附图中示出的所限定的所有变型。
sequencelisting
<110>谷万达公司
<120>用于稳定反式剪接的内含肽修饰的蛋白酶的方法和组合物
<130>agr-pt027.1wo
<150>us62/069,653
<151>2014-10-28
<160>57
<170>patentinversion3.5
<210>1
<211>491
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,ni-dpng-trx
<400>1
metalagluglualalysglulystyrleuileglypheasnglugln
151015
glualavalsergluphevalgluglnvalglualaasnaspgluval
202530
alaileleuserglugluglugluvalgluilegluleuleuhisglu
354045
phegluthrileprovalleuservalgluleuserprogluaspval
505560
aspalaleugluleuaspproalailesertyrileglugluaspala
65707580
gluvalthrthrmetalaglnservalprotrpglyileserargval
859095
glnalaproalaalahisasnargglyleuthrglyserglyvallys
100105110
valalavalleuaspthrglyileserthrhisproaspleuasnile
115120125
argglyglyalaserphevalproglygluproserthrglnaspgly
130135140
asnglyhisglythrhisvalalaglythrilealaalaleuasnasn
145150155160
serileglyvalleuglyvalalaproseralagluleutyralaval
165170175
lysvalleuglyalaserglyserglyservalserserilealagln
180185190
glyleuglutrpalaglyasnasnglymethisvalalaasnleuser
195200205
leuglyserproserproseralathrleugluglnalavalasnser
210215220
alathrserargglyvalleuvalvalalaalaserglyasnsergly
225230235240
alaglyserilesertyrproalaargtyralaasnalametalaval
245250255
glyalathraspglnasnasnasnargalaserpheserglntyrgly
260265270
alaglyleuaspilevalalaproglyvalasnvalglnserthrtyr
275280285
proglycysleuaspleulysthrglnvalglnthrproglnglymet
290295300
lysgluileserasnileglnvalglyaspleuvalleuserasnthr
305310315320
glytyrasngluvalleuasnvalpheprolysserlyslyslysser
325330335
tyrlysilethrleugluaspglylysgluileilecyssergluglu
340345350
hisleupheprothrglnthrglyglumetasnileserglyglyleu
355360365
lysgluglymetcysleutyrvallysgluaspproasnglymetser
370375380
asplysileilehisleuthraspaspserpheaspthraspvalleu
385390395400
lysalaaspglyalaileleuvalaspphetrpalaglutrpcysgly
405410415
procyslysmetilealaproileleuaspgluilealaaspglutyr
420425430
glnglylysleuthrvalalalysleuasnileaspglnasnprogly
435440445
thralaprolystyrglyileargglyileprothrleuleuleuphe
450455460
lysasnglygluvalalaalathrlysvalglyalaleuserlysgly
465470475480
glnleulysglupheleuaspalaasnleuala
485490
<210>2
<211>213
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,trx-dpng-mtt-ic
<400>2
metserasplysileilehisleuthraspaspserpheaspthrasp
151015
valleulysalaaspglyalaileleuvalaspphetrpalaglutrp
202530
cysglyprocyslysmetilealaproileleuaspgluilealaasp
354045
glutyrglnglylysleuthrvalalalysleuasnileaspglnasn
505560
proglythralaprolystyrglyileargglyileprothrleuleu
65707580
leuphelysasnglygluvalalaalathrlysvalglyalaleuser
859095
lysglyglnleulysglupheleuaspalaasnleualaaspproasn
100105110
glymetthrlyslysilethrlysileglugluleuaspgluargglu
115120125
leuileaspilegluvalserglyasnhisleuphetyralaasnasp
130135140
ileglythrhisasnserthrtyralaserleuasnglythrsermet
145150155160
alathrprohisvalalaglyalaalaalaleuvallysglnlysasn
165170175
prosertrpserasnvalglnileargasnhisleulysasnthrala
180185190
thrserleuglyserthrasnleutyrglyserglyleuvalasnala
195200205
glualaalathrarg
210
<210>3
<211>1476
<212>dna
<213>artificialsequence
<220>
<223>syntheticconstruct,ni-dpng-trxcodingsequence
<400>3
atggctgaagaagcaaaagaaaaatatttaattggctttaatgagcaggaagctgtcagt60
gagtttgtagaacaagtagaggcaaatgacgaggtcgccattctctctgaggaagaggaa120
gtcgaaattgaattgcttcatgaatttgaaacgattcctgttttatccgttgagttaagc180
ccagaagatgtggacgcgcttgaactcgatccagcgatttcttatattgaagaggatgca240
gaagtaacgacaatggcgcaatcggtaccttggggaattagccgtgtgcaagccccagct300
gcccataaccgtggattgacaggttctggtgtaaaagttgctgtcctcgatacagggata360
tccactcatccagatctaaatattcgtggtggcgcaagctttgtaccaggggaaccgtcg420
actcaagatgggaatgggcatggcacgcatgtggccgggacgatcgctgctttaaacaat480
tcgattggcgttcttggcgtagcgccgagcgctgagctatacgctgttaaagtcctaggg540
gcgagcggttcaggttcggtcagctcgattgcccaaggattggaatgggcagggaacaat600
ggcatgcacgttgctaatttgagtttaggaagcccttcgccaagtgccacacttgagcaa660
gctgttaatagcgcgacttctagaggcgttcttgttgtagcggcatctgggaactcaggt720
gcaggctcaatcagctatccggcgcgctatgcgaacgcaatggcagtcggagctactgat780
caaaacaacaaccgcgctagcttttcacagtatggcgcaggccttgacattgtcgcaccc840
ggggtaaacgtgcagagcacatacccaggttgtctggacctgaaaacgcaagtgcaaacc900
ccgcaaggcatgaaggaaatctcaaacatccaagtcggtgacctggtgctgtcgaatacc960
ggctataacgaagtgctgaatgtttttccgaagagcaaaaagaaatcttacaagatcacg1020
ctggaagatggcaaggaaattatttgcagcgaagaacatctgttcccgacccagacgggc1080
gaaatgaatatctccggcggtctgaaagaaggcatgtgtctgtacgtcaaggaagatcct1140
aatggtatgagcgataaaattattcacctgactgacgacagttttgacacggatgtactc1200
aaagcggacggggcgatcctcgtcgatttctgggcagagtggtgcggtccgtgcaaaatg1260
atcgccccgattctggatgaaatcgctgacgaatatcagggcaaactgaccgttgcaaaa1320
ctgaacatcgatcaaaaccctggcactgcgccgaaatatggcatccgtggtatcccgact1380
ctgctgctgttcaaaaacggtgaagtggcggcaaccaaagtgggtgcactgtctaaaggt1440
cagttgaaagagttcctcgacgctaacctggcctag1476
<210>4
<211>642
<212>dna
<213>artificialsequence
<220>
<223>syntheticconstruct,ni-dpng-trxcodingsequence
<400>4
atgagcgataaaattattcacctgactgacgacagttttgacacggatgtactcaaagcg60
gacggggcgatcctcgtcgatttctgggcagagtggtgcggtccgtgcaaaatgatcgcc120
ccgattctggatgaaatcgctgacgaatatcagggcaaactgaccgttgcaaaactgaac180
atcgatcaaaaccctggcactgcgccgaaatatggcatccgtggtatcccgactctgctg240
ctgttcaaaaacggtgaagtggcggcaaccaaagtgggtgcactgtctaaaggtcagttg300
aaagagttcctcgacgctaacctggccgatcctaatggtatgacgaagaaaattacgaag360
atcgaagaactggatgaacgtgaactgattgacatcgaagttagcggcaaccatctgttt420
tacgcgaatgacattgggacccacaactcaacttatgccagcttaaacggtacatcgatg480
gctactcctcatgttgcaggtgcggccgcccttgttaaacaaaagaacccatcttggtct540
aatgtacaaattcgaaatcatctaaagaatacggcaactagtttaggaagcacgaacttg600
tatggaagcggacttgttaacgcagaagcggcaacgcgttaa642
<210>5
<211>4
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,dpnglinker
<400>5
aspproasngly
1
<210>6
<211>380
<212>prt
<213>bacilluslentus
<220>
<221>misc_feature
<222>(1)..(380)
<223>savinasep29600
<400>6
metlyslysproleuglylysilevalalaserthralaleuleuile
151015
servalalapheserserserilealaseralaalagluglualalys
202530
glulystyrleuileglypheasngluglnglualavalsergluphe
354045
valgluglnvalglualaasnaspgluvalalaileleusergluglu
505560
glugluvalgluilegluleuleuhisgluphegluthrileproval
65707580
leuservalgluleuserprogluaspvalaspalaleugluleuasp
859095
proalailesertyrileglugluaspalagluvalthrthrmetala
100105110
glnservalprotrpglyileserargvalglnalaproalaalahis
115120125
asnargglyleuthrglyserglyvallysvalalavalleuaspthr
130135140
glyileserthrhisproaspleuasnileargglyglyalaserphe
145150155160
valproglygluproserthrglnaspglyasnglyhisglythrhis
165170175
valalaglythrilealaalaleuasnasnserileglyvalleugly
180185190
valalaproseralagluleutyralavallysvalleuglyalaser
195200205
glyserglyservalserserilealaglnglyleuglutrpalagly
210215220
asnasnglymethisvalalaasnleuserleuglyserproserpro
225230235240
seralathrleugluglnalavalasnseralathrserargglyval
245250255
leuvalvalalaalaserglyasnserglyalaglyserilesertyr
260265270
proalaargtyralaasnalametalavalglyalathraspglnasn
275280285
asnasnargalaserpheserglntyrglyalaglyleuaspileval
290295300
alaproglyvalasnvalglnserthrtyrproglyserthrtyrala
305310315320
serleuasnglythrsermetalathrprohisvalalaglyalaala
325330335
alaleuvallysglnlysasnprosertrpserasnvalglnilearg
340345350
asnhisleulysasnthralathrserleuglyserthrasnleutyr
355360365
glyserglyleuvalasnalaglualaalathrarg
370375380
<210>7
<211>1143
<212>dna
<213>bacilluslentus
<220>
<221>misc_feature
<222>(1)..(1143)
<223>p29600codingsequence
<400>7
atgaagaaaccgttggggaaaattgtcgcaagcaccgcactactcatttctgttgctttt60
agttcatcgatcgcatcggctgctgaagaagcaaaagaaaaatatttaattggctttaat120
gagcaggaagctgtcagtgagtttgtagaacaagtagaggcaaatgacgaggtcgccatt180
ctctctgaggaagaggaagtcgaaattgaattgcttcatgaatttgaaacgattcctgtt240
ttatccgttgagttaagcccagaagatgtggacgcgcttgaactcgatccagcgatttct300
tatattgaagaggatgcagaagtaacgacaatggcgcaatcggtaccatggggaattagc360
cgtgtgcaagccccagctgcccataaccgtggattgacaggttctggtgtaaaagttgct420
gtcctcgatacagggatatccactcatccagatctaaatattcgtggtggcgcaagcttt480
gtaccaggggaaccgtcgactcaagatgggaatgggcatggcacgcatgtggccgggacg540
atcgctgctttaaacaattcgattggcgttcttggcgtagcgccgagcgctgagctatac600
gctgttaaagtcctaggggcgagcggttcaggttcggtcagctcgattgcccaaggattg660
gaatgggcagggaacaatggcatgcacgttgctaatttgagtttaggaagcccttcgcca720
agtgccacacttgagcaagctgttaatagcgcgacttctagaggcgttcttgttgtagcg780
gcatctgggaactcaggtgcaggctcaatcagctatccggcgcgctatgcgaacgcaatg840
gcagtcggagctactgatcaaaacaacaaccgcgctagcttttcacagtatggcgcaggc900
cttgacattgtcgcacccggggtaaacgtgcagagcacatacccaggttcaacatatgcc960
agcttaaacggtacatcgatggctactcctcatgttgcaggtgcggccgcccttgttaaa1020
caaaagaacccatcttggtctaatgtacaaattcgaaatcatctaaagaatacggcaact1080
agtttaggaagcacgaacttgtatggaagcggacttgttaacgcagaagcggcaacgcgt1140
taa1143
<210>8
<211>379
<212>prt
<213>bacilluslicheniformis
<220>
<221>misc_feature
<222>(1)..(379)
<223>alkalineproteaseq6pnn5
<400>8
metmetarglyslysserphetrpleuglymetleuthralaphemet
151015
leuvalphethrmetalapheseraspseralaseralaalaglnpro
202530
alalysasnvalglulysasptyrilevalglyphelysserglyval
354045
lysthralaservallyslysaspileilelysgluserglyglylys
505560
valasplysglnpheargileileasnalaalalysalalysleuasp
65707580
lysglualaleulysgluvallysasnaspproaspvalalatyrval
859095
glugluasphisvalalahisalaleualaglnthrvalprotyrgly
100105110
ileproleuilelysalaasplysvalglnalaglnglyphelysgly
115120125
alaasnvallysvalalavalleuaspthrglyileglnalaserhis
130135140
proaspleuasnvalvalglyglyalaserphevalalaglygluala
145150155160
tyrasnthraspglyasnglyhisglythrhisvalalaglythrval
165170175
alaalaleuaspasnthrthrglyvalleuglyvalalaproserval
180185190
serleutyralavallysvalleuasnserserglyserglysertyr
195200205
serglyilevalserglyileglutrpalathrthrasnglymetasp
210215220
valileasnmetserleuglyglyalaserglyserthralametlys
225230235240
glnalavalaspasnalatyralaargglyvalvalvalvalalaala
245250255
alaglyasnserglyserserglyasnthrasnthrileglytyrpro
260265270
alalystyraspservalilealavalglyalavalaspserasnser
275280285
asnargalaserpheserservalglyalagluleugluvalmetala
290295300
proglyalaglyvaltyrserthrtyrprothrasnthrtyralathr
305310315320
leuasnglythrsermetalaserprohisvalalaglyalaalaala
325330335
leuileleuserlyshisproasnleuseralaserglnvalargasn
340345350
argleuserserthralathrtyrleuglyserserphetyrtyrgly
355360365
lysglyleuileasnvalglualaalaalagln
370375
<210>9
<211>404
<212>prt
<213>aspergillusflavus
<220>
<221>misc_feature
<222>(1)..(404)
<223>b8nly9_pepa_aspfn
<400>9
metvalileleuserlysvalalaalavalalavalglyleuserthr
151015
valalaseralaleuprothrglyproserhisserprohisalaarg
202530
argglyphethrileasnglnilethrargglnthralaargvalgly
354045
prolysthralaserpheproalailetyrserargalaleualalys
505560
tyrglyglythrvalproalahisleulysseralavalalasergly
65707580
hisglythrvalvalthrserprogluproasnaspileglutyrleu
859095
thrprovalasnileglyglythrthrleuasnleuasppheaspthr
100105110
glyseralaaspleutrpvalpheserglugluleuprolysserglu
115120125
glnthrglyhisaspvaltyrlysproserglyasnalaserlysile
130135140
alaglyalasertrpaspilesertyrglyaspglyserseralaser
145150155160
glyaspvaltyrglnaspthrvalthrvalglyglyvalthralagln
165170175
glyglnalavalglualaalaserlysileseraspglnphevalgln
180185190
asplysasnasnaspglyleuleuglyleualapheserserileasn
195200205
thrvallysprolysproglnthrthrphepheaspthrvallysasp
210215220
glnleuaspalaproleuphealavalthrleulystyrhisalapro
225230235240
glysertyrasppheglypheileasplysserlysphethrglyglu
245250255
leualatyralaaspvalaspaspserglnglyphetrpglnphethr
260265270
alaaspglytyrservalglylysglyaspalaglnlysalaproile
275280285
thrglyilealaaspthrglythrthrleuvalmetleuaspaspglu
290295300
ilevalaspalatyrtyrlysglnvalglnglyalalysasnaspala
305310315320
seralaglyglytyrvalpheprocysgluthrgluleuprogluphe
325330335
thrvalvalileglysertyrasnalavalileproglylyshisile
340345350
asntyralaproleuglngluglyserserthrcysvalglyglyile
355360365
glnserasnserglyleuglyleuserileleuglyaspvalpheleu
370375380
lysserglntyrvalvalpheaspserglnglyproargleuglyphe
385390395400
alaalaglnala
<210>10
<211>375
<212>dna
<213>artificialsequence
<220>
<223>syntheticconstruct,gp41-1inteincodingsequence
<400>10
tgtctggacctgaaaacgcaagtgcaaaccccgcaaggcatgaaggaaatctcaaacatc60
caagtcggtgacctggtgctgtcgaataccggctataacgaagtgctgaatgtttttccg120
aagagcaaaaagaaatcttacaagatcacgctggaagatggcaaggaaattatttgcagc180
gaagaacatctgttcccgacccagacgggcgaaatgaatatctccggcggtctgaaagaa240
ggcatgtgtctgtacgtcaaggaaatgatgctgaagaaaattctgaagatcgaagaactg300
gatgaacgtgaactgattgacatcgaagttagcggcaaccatctgttttacgcgaatgac360
attctgacccacaac375
<210>11
<211>125
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,gp41-1intein
<400>11
cysleuaspleulysthrglnvalglnthrproglnglymetlysglu
151015
ileserasnileglnvalglyaspleuvalleuserasnthrglytyr
202530
asngluvalleuasnvalpheprolysserlyslyslyssertyrlys
354045
ilethrleugluaspglylysgluileilecysserglugluhisleu
505560
pheprothrglnthrglyglumetasnileserglyglyleulysglu
65707580
glymetcysleutyrvallysglumetmetleulyslysileleulys
859095
ileglugluleuaspgluarggluleuileaspilegluvalsergly
100105110
asnhisleuphetyralaasnaspileleuthrhisasn
115120125
<210>12
<211>264
<212>dna
<213>artificialsequence
<220>
<223>syntheticconstruct,gp41-1n-inteincodingsequence
<400>12
tgtctggacctgaaaacgcaagtgcaaaccccgcaaggcatgaaggaaatctcaaacatc60
caagtcggtgacctggtgctgtcgaataccggctataacgaagtgctgaatgtttttccg120
aagagcaaaaagaaatcttacaagatcacgctggaagatggcaaggaaattatttgcagc180
gaagaacatctgttcccgacccagacgggcgaaatgaatatctccggcggtctgaaagaa240
ggcatgtgtctgtacgtcaaggaa264
<210>13
<211>111
<212>dna
<213>artificialsequence
<220>
<223>syntheticconstruct,gp41-1c-inteincodingsequence
<400>13
atgatgctgaagaaaattctgaagatcgaagaactggatgaacgtgaactgattgacatc60
gaagttagcggcaaccatctgttttacgcgaatgacattctgacccacaac111
<210>14
<211>88
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,gp41-1n-intein
<400>14
cysleuaspleulysthrglnvalglnthrproglnglymetlysglu
151015
ileserasnileglnvalglyaspleuvalleuserasnthrglytyr
202530
asngluvalleuasnvalpheprolysserlyslyslyssertyrlys
354045
ilethrleugluaspglylysgluileilecysserglugluhisleu
505560
pheprothrglnthrglyglumetasnileserglyglyleulysglu
65707580
glymetcysleutyrvallysglu
85
<210>15
<211>37
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,gp41-1c-intein
<400>15
metmetleulyslysileleulysileglugluleuaspgluargglu
151015
leuileaspilegluvalserglyasnhisleuphetyralaasnasp
202530
ileleuthrhisasn
35
<210>16
<211>94
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,cbu_dnab(12)n-intein
<400>16
cysvalthrglyaspthrleuilecysleualaaspglyargargval
151015
proileglnaspleuvalglyhisserprogluvalilealavalasp
202530
asplysglyargleuvalcysalalyssergluvaliletrplysval
354045
glygluargservalphegluilelysleualaserglyargserile
505560
lysalathralagluhisargleuleualaphelysglytrparghis
65707580
vallysaspphelysvalglyaspargleualailealahis
8590
<210>17
<211>40
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,cbu_dnab(12)c-intein
<400>17
metseraspleuphetrpaspargilevalserilegluglulysgly
151015
serglugluvaltyraspleuthrvalprolystyralasertrpleu
202530
alaaspglyvalvalserhisasn
3540
<210>18
<211>96
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,mja_gf-6p(44)n-intein
<400>18
cysleuhisproaspthrtyrvalileleuproaspglyargmetlys
151015
lysilesergluileaspgluaspgluvalleuservalasnpheglu
202530
aspleulysleutyrasnlyslysilelyslysphelyshislysala
354045
prolysileleutyrlysilelysthralaphesergluleuilethr
505560
thrglygluhislysleuphevalvalgluasnglylysilevalglu
65707580
lyscysvallysaspleuasnglysergluleuileglyvalvalarg
859095
<210>19
<211>41
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,mja_gf-6p(44)c-intein
<400>19
metalaaspilevaltrpthrlysphelysileglugluvalgluser
151015
aspvalglutyrvaltyraspleugluvalgluasptyrhisasnphe
202530
ileglyasnleuileileasnhisasn
3540
<210>20
<211>126
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,mja_hyp-1(46)n-intein
<400>20
cysvalproproaspthrleuleuileleugluasnglyphelysarg
151015
ilevalaspilelysvalglyasplysvalleuthrhisgluasnarg
202530
phelyslysvalglulysvaltyrlysargargtyrileglyaspile
354045
ilelysilelysvalargtyrpheproglugluileileleuthrpro
505560
gluhisprovaltyralailelysthrglulysargcysaspglyser
65707580
hisglyilecyslyspheasncysleuthrglntyrthrasnproser
859095
cyslyslysargtyrarglystyrlysargglutrpileilealalys
100105110
aspleulysvalglyaspvalilevaltyrproileproasn
115120125
<210>21
<211>41
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,mja_hyp-1(46)c-intein
<400>21
metglyasntyrleutyralaproileileargileglyargglutyr
151015
tyraspglyphevaltyrasnleugluvalgluaspaspsersertyr
202530
valthrvalserglythrleuhisasn
3540
<210>22
<211>110
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,mja_if2(47)n-intein
<400>22
cysleumetprohisglulysvalleuthrglutyrglygluilelys
151015
ilegluaspleuphelysileglylysgluilevalglulysaspglu
202530
leulysgluilearglysleuasnilelysvalhisthrleuasnglu
354045
asnglygluilelysileileasnalaprotyrvaltrplysleulys
505560
hislysglylysmetilelysvallysleulysasntrphisserile
65707580
thrthrthrprogluhispropheleuthrasnasnglytrpilelys
859095
alagluasnilelyslysglymettyrvalalaileproarg
100105110
<210>23
<211>39
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,mja_if2(47)c-intein
<400>23
metasnilealaphevalgluvalgluaspvalgluileileasptyr
151015
aspglytyrvaltyraspleuthrthrgluthrhisasnpheileala
202530
asnglyilevalvalhisasn
35
<210>24
<211>102
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,mja_pol-1(50)n-intein
<400>24
cyshisprolysglythrlysvalvalvallysglylysglyileval
151015
asnilegluaspvallysgluglyasntyrvalleuglyileaspgly
202530
trpglnlysvallyslysvaltrplystyrglutyrgluglygluleu
354045
ileasnvalasnglyleulyscysthrproasnhislysileproleu
505560
argtyrlysilelyshislyslysileasnlysasnasptyrleuval
65707580
argaspiletyralalysserleuleuthrlysphelysglyglugly
859095
lysleuileleucyslys
100
<210>25
<211>41
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,mja_pol-1(50)c-intein
<400>25
mettyrglyphetyraspleuaspaspvalcysvalserleugluser
151015
tyrlysglygluvaltyraspleuthrleugluglyargprotyrtyr
202530
phealaasnglyileleuthrhisasn
3540
<210>26
<211>122
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,pab_cdc21-1(79)n-intein
<400>26
cysvalasptyrgluthrgluvalvalleuglyasnglygluarglys
151015
lysileglygluilevalgluargalailegluglualaglulysasn
202530
glylysleuglyargvalaspaspglyphetyralaproileaspile
354045
gluvaltyrserleuaspleugluthrleulysvalarglysalaarg
505560
alaasnilealatrplysargthralaprolyslysmetmetleuval
65707580
lysthrargglyglylysargileargvalthrprothrhisprophe
859095
phevalleuglugluglylysvalalametarglysalaargaspleu
100105110
glugluglyasnlysilealathrileglu
115120
<210>27
<211>41
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,pab_cdc21-1(79)c-intein
<400>27
metservalsertrpaspgluvalalagluileleuglutyrglupro
151015
lysaspprotrpvaltyraspleuglnvalproglytyrhisasnphe
202530
leualaasnglyilephevalhisasn
3540
<210>28
<211>111
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,pab_if2(81)n-intein
<400>28
cysleuleuproaspglulysvalvalvalproservalglypheval
151015
thrleulysgluleuphegluthralaserlysvalvalgluargasp
202530
aspglulysgluilearggluleuaspgluargilethrservalasn
354045
glyaspglylysthrglyleuvallysalasertyrvaltrplysval
505560
arghislysglylysvalileargvallysleulysasntrphisgly
65707580
valthrvalthrprogluhispropheleuthrthrlysglytrplys
859095
argalaaspglnleuargproglyasptyrvalalavalproarg
100105110
<210>29
<211>39
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,pab_if2(81)c-intein
<400>29
metthrleuvalpheileprovalgluasnvalgluglugluglutyr
151015
aspglytyrvaltyraspleuthrthrgluthrhisasnpheileala
202530
asnglyileleuvalhisasn
35
<210>30
<211>121
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,pab_vma(92)n-intein
<400>30
cysvalaspglyaspthrleuvalleuthrlysglupheglyleuile
151015
lysilelysaspleutyrlysileleuaspglylysglylyslysthr
202530
valasnglyasngluglutrpthrgluleugluargproilethrleu
354045
tyrglytyrlysaspglylysilevalgluilelysalathrhisval
505560
tyrlysglypheseralaglymetilegluileargthrargthrgly
65707580
arglysilelysvalthrproilehislysleuphethrglyargval
859095
thrlysasnglyleugluilearggluvalmetalalysaspleulys
100105110
lysglyaspargileilevalalalys
115120
<210>31
<211>42
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,pab_vma(92)c-intein
<400>31
metthrhisvalleupheaspgluilevalgluileargtyrileser
151015
gluglyglngluvaltyraspvalthrthrgluthrhisasnpheile
202530
glyglyasnmetprothrleuleuhisasn
3540
<210>32
<211>111
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,pho_if2(103)n-intein
<400>32
cysleuleuproglugluargvalileleuproasptyrglyproile
151015
thrleuglugluleupheasnmetthrlysgluthrvalphelysasp
202530
gluglulysgluvalarglysleuglyileargmetprovalalagly
354045
valaspglyargvalargleuleugluglyprotyrvaltrplysval
505560
argtyrlysglylysmetleuargvallysleulysasptrphisser
65707580
valalavalthrprogluhispropheleuthrthrargglytrpval
859095
argalaaspglnleulysproglyasptyrvalalavalprolys
100105110
<210>33
<211>39
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,pho_if2(103)c-intein
<400>33
metasnphevalpheleuprovalglulysilegluglupheglutyr
151015
aspglytyrvaltyraspvalthrthrgluthrhisasnpheileala
202530
asnglyileleuvalhisasn
35
<210>34
<211>121
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,pho_vma(110)n-intein
<400>34
cysvalaspglyaspthrleuvalleuthrlysglupheglyleuile
151015
lysilelysgluleutyrglulysleuaspglylysglyarglysile
202530
valgluglyasngluglutrpthrgluleuglulysproilethrval
354045
tyrglytyrlysaspglylysilevalgluilelysalathrhisval
505560
tyrlysglyvalserserglymetvalgluileargthrargthrgly
65707580
arglysilelysvalthrproilehisargleuphethrglyargval
859095
thrlysaspglyleuileleulysgluvalmetalamethisvallys
100105110
proglyaspargilealavalvallys
115120
<210>35
<211>42
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,pho_vma(110)c-intein
<400>35
metglnhisileilepheaspgluvalileaspvalargtyrilepro
151015
gluproglngluvaltyraspvalthrthrgluthrhisasnpheval
202530
glyglyasnmetprothrleuleuhisasn
3540
<210>36
<211>96
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,rma_dnab(116)n-intein
<400>36
cysleualaglyaspthrleuilethrleualaaspglyargargval
151015
proilearggluleuvalserglnglnasnpheservaltrpalaleu
202530
asnproglnthrtyrargleugluargalaargvalserargalaphe
354045
cysthrglyilelysprovaltyrargleuthrthrargleuglyarg
505560
serileargalathralaasnhisargpheleuthrproglnglytrp
65707580
lysargvalaspgluleuglnproglyasptyrleualaleuproarg
859095
<210>37
<211>40
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,rma_dnab(116)c-intein
<400>37
metseraspvaltyrtrpaspproilevalserilegluproaspgly
151015
valglugluvalpheaspleuthrvalproglyprohisasnpheval
202530
alaasnaspileilealahisasn
3540
<210>38
<211>89
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,sru_dnab(123)n-intein
<400>38
cysleuglylysglythrprovalmetmettyraspglyargthrlys
151015
provalglulysvalgluvalglyaspargleumetglyaspaspgly
202530
serproargthrvalglnserleualaargglyarggluglnmettyr
354045
trpvalargglnlysargglymetasptyrargvalasngluserhis
505560
ileleuserleulyslysserargarggluglyalaargasparggly
65707580
serilealaaspileservalargasp
85
<210>39
<211>38
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,sru_dnab(123)c-intein
<400>39
mettrpargmetthrglyileaspvalgluproaspglyvalglyasp
151015
tyrpheglyphethrleuaspglyasnglyargpheleuleuglyasp
202530
glythrvalthrhisasn
35
<210>40
<211>95
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,tag_pol-1_tsp-ty_pol-1(128)n-intein
<400>40
cyshisproalaaspthrlysvalilevallysglylysglyileval
151015
asnileseraspvallysgluglyasptyrileleuglyileaspgly
202530
trpglnargvallyslysvaltrplystyrhistyrgluglylysleu
354045
ileasnileasnglyleulyscysthrproasnhislysvalproval
505560
valthrgluasnaspargglnthrargileargaspserleualalys
65707580
serpheleuserglylysvallysglylysileilethrthrlys
859095
<210>41
<211>41
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,tag_pol-1_tsp-ty_pol-1(128)c-intein
<400>41
metasnserphetyrasnleuserthrphegluvalserserglutyr
151015
tyrlysglygluvaltyraspleuthrleugluglyasnprotyrtyr
202530
phealaasnglyileleuthrhisasn
3540
<210>42
<211>107
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,ter_rir1-4(135)n-intein
<400>42
cysleuasplysthralaleuargilepheasnglnglyleuleutyr
151015
alaaspgluvalvalthrproglyserglygluthrvalglyleugly
202530
leuthrvalargasnglyileglyalaserthralailealaasngln
354045
prometgluleuvalgluilelysleualaasnglyarglysleuarg
505560
metthrproasnhisargmetservallysglylystrpilehisala
65707580
cysasnleulysproglymetleuleuasptyrserileglyglutyr
859095
glnlysarggluaspthrleuleuileproleu
100105
<210>43
<211>49
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,ter_rir1-4(135)c-intein
<400>43
metserlyscysvalleuasntyrserprotyrlysilegluserval
151015
asnileglyalavalcysasptyrsertyraspphealaileglugly
202530
ileasnaspasnaspsertrptyrtrpglnglyalaleulysserhis
354045
asn
<210>44
<211>111
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,tko_if2(143)n-intein
<400>44
cysleuleuproaspglulysvalileleuprogluhisglyproile
151015
thrleulysglyleupheaspleualalysgluthrvalvalalaasp
202530
asnglulysgluilearglysleuglyalalysleuthrilevalgly
354045
gluaspglyargleuargvalleugluserprotyrvaltrplysval
505560
arghisargglylysmetleuargvallysleulysasntrphisser
65707580
valservalthrprogluhispropheleuthrthrargglytrpval
859095
argalaaspglnleulysproglyasptyrvalalavalproarg
100105110
<210>45
<211>39
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,tko_if2(143)c-intein
<400>45
metasnleuvalpheileprovalgluaspilegluglupheglutyr
151015
gluglytyrvaltyraspvalthrthrgluthrhisasnphevalala
202530
asnglyileleuvalhisasn
35
<210>46
<211>98
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,tth-hb27_dnae-2(15)n-intein
<400>46
leuproalaargalaargvalvalasptrpcysthrglyargvalval
151015
argvalglygluilevalargglyglualalysglyvaltrpvalval
202530
serleuaspglualaargleuargleuvalproargprovalvalala
354045
alapheproserglylysalaglnvaltyralaleuargthralathr
505560
glyargvalleuglualathralaasnhisprovaltyrthrproglu
65707580
glytrpargproleuglythrleualaproglyasptyrvalalaleu
859095
proarg
<210>47
<211>40
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,tth-hb27_dnae-2(15)c-intein
<400>47
metalagluvaltyrtrpaspargvalglualavalgluproleugly
151015
gluglugluvalpheaspleuthrvalgluglythrhisthrpheval
202530
alagluaspvalilevalhisasn
3540
<210>48
<211>13
<212>dna
<213>artificialsequence
<220>
<223>syntheticconstruct,frameshiftedrbs
<400>48
aaggagatatacc13
<210>49
<211>378
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,isavinases317_gp41-1-ni
<400>49
metalagluglualalysglulystyrleuileglypheasnglugln
151015
glualavalsergluphevalgluglnvalglualaasnaspgluval
202530
alaileleuserglugluglugluvalgluilegluleuleuhisglu
354045
phegluthrileprovalleuservalgluleuserprogluaspval
505560
aspalaleugluleuaspproalailesertyrileglugluaspala
65707580
gluvalthrthrmetalaglnservalprotrpglyileserargval
859095
glnalaproalaalahisasnargglyleuthrglyserglyvallys
100105110
valalavalleuaspthrglyileserthrhisproaspleuasnile
115120125
argglyglyalaserphevalproglygluproserthrglnaspgly
130135140
asnglyhisglythrhisvalalaglythrilealaalaleuasnasn
145150155160
serileglyvalleuglyvalalaproseralagluleutyralaval
165170175
lysvalleuglyalaserglyserglyservalserserilealagln
180185190
glyleuglutrpalaglyasnasnglymethisvalalaasnleuser
195200205
leuglyserproserproseralathrleugluglnalavalasnser
210215220
alathrserargglyvalleuvalvalalaalaserglyasnsergly
225230235240
alaglyserilesertyrproalaargtyralaasnalametalaval
245250255
glyalathraspglnasnasnasnargalaserpheserglntyrgly
260265270
alaglyleuaspilevalalaproglyvalasnvalglnserthrtyr
275280285
proglycysleuaspleulysthrglnvalglnthrproglnglymet
290295300
lysgluileserasnileglnvalglyaspleuvalleuserasnthr
305310315320
glytyrasngluvalleuasnvalpheprolysserlyslyslysser
325330335
tyrlysilethrleugluaspglylysgluileilecyssergluglu
340345350
hisleupheprothrglnthrglyglumetasnileserglyglyleu
355360365
lysgluglymetcysleutyrvallysglu
370375
<210>50
<211>101
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,isavinases317_gp41-1-ic
<400>50
metmetleulyslysileleulysileglugluleuaspgluargglu
151015
leuileaspilegluvalserglyasnhisleuphetyralaasnasp
202530
ileleuthrhisasnserthrtyralaserleuasnglythrsermet
354045
alathrprohisvalalaglyalaalaalaleuvallysglnlysasn
505560
prosertrpserasnvalglnileargasnhisleulysasnthrala
65707580
thrserleuglyserthrasnleutyrglyserglyleuvalasnala
859095
glualaalathrarg
100
<210>51
<211>118
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,trx-1
<400>51
metserasplysileilehisleuthraspaspserpheaspthrasp
151015
valleulysalaaspglyalaileleuvalaspphetrpalaglutrp
202530
cysglyprocyslysmetilealaproileleuaspgluilealaasp
354045
glutyrglnglylysleuthrvalalalysleuasnileaspglnasn
505560
proglythralaprolystyrglyileargglyileprothrleuleu
65707580
leuphelysasnglygluvalalaalathrlysvalglyalaleuser
859095
lysglyglnleulysglupheleuaspalaasnleualaleuglnval
100105110
asplysleualaalaala
115
<210>52
<211>357
<212>dna
<213>artificialsequence
<220>
<223>syntheticconstruct,trx-1codingsequence
<400>52
atgagcgataaaattattcacctgactgacgacagttttgacacggatgtactcaaagcg60
gacggggcgatcctcgtcgatttctgggcagagtggtgcggtccgtgcaaaatgatcgcc120
ccgattctggatgaaatcgctgacgaatatcagggcaaactgaccgttgcaaaactgaac180
atcgatcaaaaccctggcactgcgccgaaatatggcatccgtggtatcccgactctgctg240
ctgttcaaaaacggtgaagtggcggcaaccaaagtgggtgcactgtctaaaggtcagttg300
aaagagttcctcgacgctaacctggccctgcaggtcgacaagcttgcggccgcataa357
<210>53
<211>109
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,trx-2
<400>53
metserasplysileilehisleuthraspaspserpheaspthrasp
151015
valleulysalaaspglyalaileleuvalaspphetrpalaglutrp
202530
cysglyprocyslysmetilealaproileleuaspgluilealaasp
354045
glutyrglnglylysleuthrvalalalysleuasnileaspglnasn
505560
proglythralaprolystyrglyileargglyileprothrleuleu
65707580
leuphelysasnglygluvalalaalathrlysvalglyalaleuser
859095
lysglyglnleulysglupheleuaspalaasnleuala
100105
<210>54
<211>327
<212>dna
<213>artificialsequence
<220>
<223>syntheticconstruct,trx-2codingsequence
<400>54
atgagcgataaaattattcacctgactgacgacagttttgacacggatgtactcaaagcg60
gacggggcgatcctcgtcgatttctgggcagagtggtgcggtccgtgcaaaatgatcgcc120
ccgattctggatgaaatcgctgacgaatatcagggcaaactgaccgttgcaaaactgaac180
atcgatcaaaaccctggcactgcgccgaaatatggcatccgtggtatcccgactctgctg240
ctgttcaaaaacggtgaagtggcggcaaccaaagtgggtgcactgtctaaaggtcagttg300
aaagagttcctcgacgctaacctggcc327
<210>55
<211>5
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,ggggslinker
<400>55
glyglyglyglyser
15
<210>56
<211>6
<212>prt
<213>artificialsequence
<220>
<223>syntehticconstruct,gggggslinker
<400>56
glyglyglyglyglyser
15
<210>57
<211>5
<212>prt
<213>artificialsequence
<220>
<223>syntheticconstruct,ggggglinker
<400>57
glyglyglyglygly
15
- 还没有人留言评论。精彩留言会获得点赞!
- OTUD5的单克隆抗体制备及其在癌症治疗中的用途的制造方法与工艺
- 一种胱氨酸蛋白酶抑制剂C测定试剂盒的制造方法与工艺
- 氮氧化物作为新型组蛋白去乙酰化酶抑制剂的抗肿瘤应用的制造方法与工艺
- 一种硼替佐米水溶性药用组合物及其制备方法和用途与制造工艺
- 初级癌症疗法后使用的含硼蛋白酶体抑制剂的制造方法与工艺
- 斯氏副柔线虫半胱氨酸蛋白酶抑制剂Pj_CPI基因克隆、重组表达方法及应用与制造工艺
- 一种制备自噬小体型瘤苗的培养基添加剂及其制备方法与制造工艺
- 一种高产脯氨酸蛋白酶的黑曲霉菌株的制造方法与工艺
- N-萘烷基取代的含氮杂环衍生物、制备方法及其抗肿瘤用途与制造工艺
- 作为BET蛋白抑制剂的1H‑吡咯并[2,3‑c]吡啶‑7(6H)‑酮和吡唑并[3,4‑c]吡啶‑7(6H)‑酮的制造方法与工艺