一种重组N‑糖酰胺酶F及其编码基因和应用的制作方法
本发明属于生物工程基因领域,涉及一种重组N-糖酰胺酶F(PNGase F)及其编码基因和表达纯化方法和应用。
背景技术:
N-糖酰胺酶F(系统命名:Peptide-N(N-acetyl-β-glucosaminyl)asparagineamidaseF,EC3.5.1.52;通常称PNGaseForglycoamidase,以下简称PNGase F)主要存在于革兰氏阴性菌及一些真核生物的细胞内,能够专一性的断裂糖链和蛋白之间的酰胺键,生成一个完整的糖链和蛋白质,并将蛋白质上的天冬酰胺转化为天冬氨酸。来自于脑膜炎脓杆菌的(F.meningosepticum)N-糖酰胺酶F基因的阅读框中含有1062个碱基,编码一个354个氨基酸的蛋白质,前面的40个氨基酸推测为信号肽,剩余的314个氨基酸为一分子量为34.779kD的蛋白。由于PNGase F能够从糖肽和糖蛋白上面完整切下N连接的寡糖链,将蛋白和糖链完全分开,这对于进行细胞表面糖链结构和功能的分析具有重要意义。且PNGase F对所有类型的N连接的糖蛋白都有作用,可将蛋白质的天冬酰胺残基转化转化为天冬氨酸,从而对糖蛋白进行脱糖基化,这些特性使它成为糖生物学研究中广泛应用的工具酶。
PNGase F最初从脑膜炎脓杆菌中提取得到,但这是一项十分耗资和耗时的工作,步骤繁琐,产率低,每升发酵液只能获得0.1~0.5mg,而且脑膜炎脓杆菌为病原菌不适合大规模发酵。因此,商品化的PNGase F价格非常昂贵,阻碍了PNGase F的广泛应用以及糖蛋白的结构与功能研究。研究者们为了解决这一难题,应用基因工程技术对PNGase F进行了重组表达。最具代表性的是苏移山等2005年在大肠杆菌表达系统BL21(DE3)质粒中进行的表达,得到的PNGase F蛋白活性为200000U/mg,但用LB培养基表达形式为包涵体,变复性条件复杂,难以控制,产量较低,仅为10mg/L;用M9培养基虽然可实现部分可溶表达,然而表达量不高,周期较长,收率也不高。Hua等2014年(Highly efficient production of peptides:N-glycosidase F for N-glycomics analysis,Protein Expression and Purification 97(2014)17-22)在酵母表达系统中表达PNGase F,并进行了5L培养基发酵,产量为800mg/L,活性为172172U/mg,但并未对N-糖酰胺酶F基因在毕赤酵母表达系统中进行优化,在一定程度上影响了N-糖酰胺酶F在毕赤酵母中的表达及最终的活性。
技术实现要素:
为了克服以上缺点,发明人将PNGase F基因在毕赤酵母表达系统中进行了优化,最终得到的密码子优化后的PNGase F,且将优化前后PNGase F基因导入到毕赤酵母宿主菌株中,发明人惊喜地发现,由于优化后PNGase F具有较高的表达量,导致重组PNGase工程菌株进行分泌表达时,由于糖基化修饰程度不一致,从而使得优化后PNGase F活性与优化前相比提高了近50倍,产量提高了4倍。
本发明的一个目的是提供一种编码PNGase F蛋白的DNA序列,其碱基序列如SEQ ID NO:1所示。该序列针对毕赤酵母表达系统进行了密码子优化,其更利于PNGase F在毕赤酵母中表达。
本发明的另一个目的是提供一种PNGase F蛋白,其氨基酸序列如SEQ ID NO:3所示。
本发明的另一个目的是提供一种含有上述编码优化后PNGase F基因的载体,优选的,所述的载体为pAO815,pPIC9,pPIC9K,pPIC3.5,pPIC3.5K,pPICZαA、B、C或pGAPZαA、B、C,更优选为pPIC3.5K,pPICZαA或pGAPZαA。
本发明的另一个目的是提供一种包含上述所述载体的毕赤酵母菌株,优选的,所述毕赤酵母菌株为SMD1168、GS115、KM71、X-33或KM71H,更优选为KM71或X-33菌株。
本发明的另一个目的是提供一种PNGase F蛋白的表达方法,所述方法包含下述步骤:
A.构建含有上述编码PNGase F基因的载体;
B.将步骤A的载体线性化后转入毕赤酵母菌株中,并在合适的条件下培养;
C.回收纯化蛋白质。
上述所述载体优选为pPIC3.5K,pPICZαA或pGAPZαA。
上述所述毕赤酵母菌株优选为KM71或X33菌株。
更优选的,上述所述载体为pPICZαA,并且上述所述毕赤酵母菌株为X33菌株。
本发明的另一个目的是提供一种重组PNGase F蛋白纯化方法,所述纯化方法如下:
A.将PNGase F发酵液低温高速离心收集上清,加入咪唑,氯化钠,磷酸二氢钠,使其上清中终浓度分别为20mM,300mM,20mM,滤膜过滤。
B.首先用平衡缓冲液平衡柱子,接着运用纯化系统将步骤A中预处理获得的PNGase F发酵液通过预装柱或者分离填料,然后运用洗脱缓冲液线性洗脱,收集洗脱峰,即获得了纯化的PNGase F蛋白。所述平衡缓冲液含有20mM磷酸二氢钠、300mM氯化钠、20mM咪唑,pH7.5;所述洗脱缓冲液含有20mM磷酸二氢钠、300mM氯化钠、500mM咪唑,pH7.5。
上述纯化工艺为亲和层析方法,预装柱和填料优选为Histrap crude 5ml FF。
本发明的另一个目的是提供了一种能充分发挥PNGaseF活性的反应缓冲液配方,优选的,包括以下组分:SDS,NP-40,β-ME,磷酸盐,DTT、EDTA。
优选地,上述反应缓冲液中各组分的浓度:SDS(v/v)0.1%-1%,β-ME(v/v)0.1%-1%,DTT(v/v)0.1%-10%,NP-40(v/v)0.1-5%,磷酸盐5-100mM,EDTA1-50mM,pH6.0-8.0。
优选地,上述反应缓冲液中各组分的浓度:SDS(v/v)0.1%-0.5%,β-ME(v/v)0.5%-1%,DTT(v/v)1%-10%,NP-40(v/v)1-5%,磷酸盐20-100mM,EDTA5-50mM,pH6.0-8.0。
优选地,上述反应缓冲液中各组分的浓度:SDS(v/v)0.5%,β-ME(v/v)0.1%,DTT(v/v)1%,NP-40(v/v)1%,磷酸盐50mM,EDTA50mM,pH8.0。
由于本发明对PNGase F的基因序列进行优化,因此性质较以往表达的蛋白有较大改变,与优化前相比活性和产量有大幅提高。发明人同时对其反应缓冲液进行了优化,运用DOE软件对缓冲液中多种成分及配方进行了大量的筛选和优化,最终优化得到了能够最充分发挥PNGase F活性的缓冲液组成,使得最终得到的PNGase F活性优于目前市场上同类产品,具有巨大的市场价值和广阔的应用前景。
附图说明
图1表示优化前后重组PNGase F基因序列对比图。
其中优化前序列对应的为天然PNGase F基因核苷酸序列;优化后序列对应的为本发明的重组PNGase F-opt的基因核苷酸序列,即密码子优化后的序列。
图2-a,2-b为优化前后PNGase F基因在毕赤酵母表达系统中的CAI指数。
其中,图2-a表示天然PNGase F基因核苷酸序列在毕赤酵母表达系统中CAI指数经程序计算为0.72;图2-b表示优化后的本发明的PNGase F-opt密码子在毕赤酵母表达系统中CAI指数经程序计算为0.86。
图3-a,3-b为密码子优化前后PNGase F基因在毕赤酵母表达宿主中最优密码子频率分布区域图。
其中图3-a表示PNGase F天然基因核苷酸序列在毕赤酵母系统中最优密码子频率分布区域图,从图中可以看出:PNGase F天然基因核苷酸序列的低利用率密码子出现百分比为11%;图3-b表示优化后的本发明的PNGase F-opt密码子在毕赤酵母系统中最优密码子频率分布区域图,优化后的本发明的PNGase F-opt密码子序列低利用率密码子出现为0。
图4-a,4-b为密码子优化前后PNGase F基因在毕赤酵母表达系统中平均GC碱基含量分布区域图。
其中,图4-a表示PNGase F天然基因核苷酸序列在毕赤酵母表达系统中平均GC碱基含量为:38.74%;图4-b表示优化后的本发明的PNGase F密码子在毕赤酵母表达系统中平均GC碱基含量为:42.60%。
图5-a,图5-b为密码子优化前后PNGase F基因PCR产物的琼脂糖凝胶电泳图。
其中,图5-a为优化前PNGase F基因PCR产物的琼脂糖凝胶电泳图。泳道1为500bp DNA Ladder;泳道2为两端含有XhoI和XbaI酶切位点的重组PNGase F基因PCR产物。
图5-b为密码子优化后PNGase F-opt基因PCR产物的琼脂糖凝胶电泳图。泳道1为200bp DNA Ladder;泳道2为两端含有XhoI和XbaI酶切位点的重组PNGase F-opt基因PCR产物。
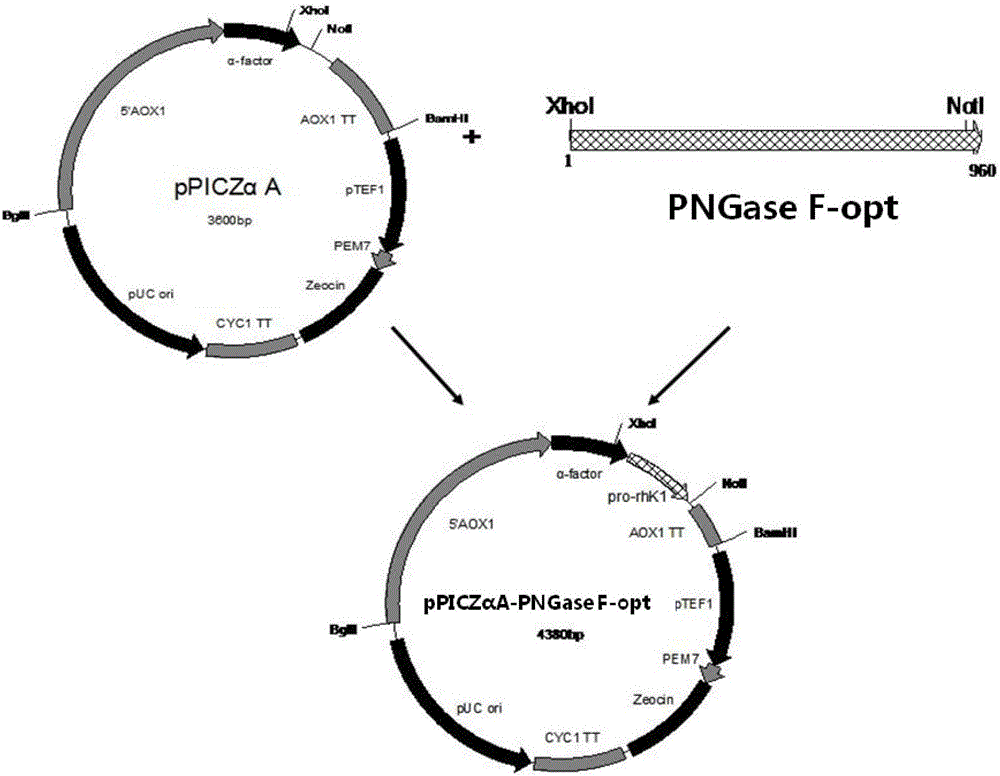
图6-a,6-b为密码子优化前后PNGase F不同表达质粒构建过程图。
图6-a为PNGase F优化前基因表达质粒pPICZ-PNGase F构建过程图;图6-b为优化后PNGase F表达质粒pPICZ-PNGase F-opt构建过程图。
图7-a,7-b为含有密码子优化前PNGase F基因在宿主工程菌中的表达鉴定图。图中箭头所指即为PNGase F蛋白。
图7-a为含有密码子优化前PNGase F基因宿主工程菌株通过甲醇诱导表达一周后,菌液上清SDS-PAGE凝胶电泳图。其中泳道2为10-250KD范围的预染蛋白上样Marker;泳道3-10为通过Zeocin筛选出来的含有密码子优化前PNGase F基因各阳性单克隆宿主工程菌株培养菌液上清。
图7-b为含有密码子优化前PNGase F基因宿主工程菌株通过甲醇诱导表达一周后,菌液上清蛋白免疫印迹图。其中泳道1为10-250KD范围的预染蛋白上样Marker;泳道2-9为通过Zeocin筛选出来的含有密码子优化前PNGase F基因各阳性单克隆宿主工程菌株培养菌液上清。
图8-a,8-b为含有密码子优化后PNGase F-opt基因在宿主工程菌中的表达鉴定图。图中箭头所指即为PNGaseF蛋白。
图8-a为含有密码子优化后PNGase F-opt基因宿主工程菌株通过甲醇诱导表达一周后,菌液上清SDS-PAGE凝胶电泳图。
图8-b为含有密码子优化后PNGase F-opt基因宿主工程菌株通过甲醇诱导表达一周后,菌液上清蛋白免疫印迹图。其中泳道1为10-250KD范围的预染蛋白上样Marker;泳道2为通过Zeocin筛选出来的含有密码子优化后PNGase F-opt基因宿主工程菌株培养菌液上清。
图9为密码子优化前PNGase F发酵液经AKTATM avant150亲和层析纯化色谱图。
图10为PNGase F蛋白经HisTrapcrude FF 5ml预装柱纯化后SDS-PAGE电泳图。其中泳道1为10-250KD范围的预染蛋白上样Marker;泳道2-15为经各纯化分管中PNGase F蛋白。
图11为密码子优化后PNGase F-opt发酵液经AKTATM avant150亲和层析纯化色谱图。
图12为PNGase F-opt蛋白经HisTrapcrude FF 5ml预装柱纯化后SDS-PAGE电泳图。其中泳道1为10-100KD非预染蛋白Marker,泳道2为基因优化前PNGase F蛋白纯化样品,泳道3为密码子优化后PNGase F-opt蛋白纯化样品,图中箭头所指即为PNGase F-opt蛋白。
图13为PNGaseF-opt在优化的各种组成和浓度的反应缓冲液中测定对Rnase B底物的活性。
图13-a为PNGaseF-opt在优化的反应缓冲液中酶切Rnase B的活性,其中泳道1为阴性对照(仅有底物Rnase B,不加N-糖酰胺酶F,箭头所指示的分子量较高蛋白条带为糖基化修饰的Rnase B,较低为未经糖基化修饰,经N-糖酰胺酶F酶切后仅有分子量较低未修饰的Rnase B蛋白条带,分子量较高的糖基化修饰条带消失),泳道2为空白对照1(仅有市售N-糖酰胺酶F,未加Rnase B底物),泳道3为空白对照2(仅有PNGaseF-opt,未加Rnase B底物),泳道4为阳性对照(用市售的N-糖酰胺酶F对Rnase B进行酶切后结果,底物经酶切后仅剩下无糖基化修饰的蛋白条带),泳道5为15-100KD非预染蛋白Marker,泳道6-9对应表1中1-4实验编号。
图13-b为PNGaseF-opt在优化的反应缓冲液中酶切Rnase B的活性,其中泳道4为15-100KD非预染蛋白Marker,其余泳道1-10对应表1中实验编号5-13。
图13-c为PNGaseF-opt在优化的反应缓冲液中酶切Rnase B的活性,其中泳道1为15-100KD非预染蛋白Marker,其余泳道2-8对应表1中实验编号14-20。
图13-d为PNGaseF-opt在优化的反应缓冲液中酶切Rnase B的活性,其中泳道1为15-100KD非预染蛋白Marker,其余泳道2-6对应表1中实验编号21-25;泳道7为PNGase F在优化前的反应缓冲液(1%SDS,2%DTT,100mM磷酸盐,pH7.0)中的活性。
图14为PNGase F和PNGase F-opt在优化后反应缓冲液中酶活性测定结果。
图14-a为PNGase F酶活性测定结果,其中泳道1为阳性对照(市售N-糖酰胺酶F+Rnase B底物),泳道2为阴性对照(仅有Rnase B底物),泳道3为15-100KD非预染蛋白Marker,泳道4-7分别为PNGase F,酶量依次为100ng,50ng,25ng,10ng。
图14-b为PNGase F-opt酶活性测定结果,其中泳道1为空白对照(仅有PNGase F-opt,未加Rnase B底物),泳道2为15-100KD非预染蛋白Marker,泳道3为阴性对照(仅有Rnase B底物,未加PNGase F-opt),泳道4酶量为20ng,泳道5酶量为10ng,泳道6酶量为5ng,泳道7酶量为2ng,泳道8酶量为1ng。
图15为毕赤酵母重组表达的人激肽释放酶(rhKLK1)经PNGase F-opt酶解前后鉴定结果。其中泳道1为毕赤酵母重组表达的人激肽释放酶(中国专利公开号:CN105802989)酶解前,泳道2为10-250KD预染蛋白marker,泳道3为毕赤酵母重组表达的人激肽释放酶酶解后,泳道4为大肠杆菌表达的rhKLK1(中国专利公开号:CN103710367A)。
图16为CHO系统重组表达的人激肽释放酶(rhKLK1)经PNGase F-opt酶解前后鉴定结果。其中泳道1为10-250KD预染蛋白marker,泳道2为CHO系统重组表达的人激肽释放酶(中国专利公开号:CN103710368)酶解前,泳道3为CHO系统重组表达的人激肽释放酶酶解后。
具体实施方式
下面结合具体实施例,进一步阐述本发明,应理解,引用实施例仅用于说明本发明而不用于限制本发明的范围。
实施例1 重组PNGase F密码子优化
发明人根据GenBank已公开的PNGase F的DNA序列(GenBank登录号:AF165910.1),如SEQ ID No:2所示,对该基因进行密码子优化后得到本发明的PNGase F-opt基因,核苷酸序列如SEQ ID No:1所示,氨基酸序列如SEQ ID No:3所示。下面是对PNGase F密码子优化前后各参数对比如下:
1.密码子适应指数(CAI)
由图2-a可知,密码子没有优化前,PNGase F原始基因在毕赤酵母表达系统中密码子适应指数(CAI)为0.72。由图2-b可知,通过密码子优化,PNGase F基因在毕赤酵母表达系统中CAI指数为0.86。通常CAI=1时被认为该基因在该表达系统中是最理想的高效表达状态,CAI指数越低表明该基因在该宿主中表达水平越差,因此可以看出经过了密码子优化后得到的基因序列可以提高PNGase F基因在毕赤酵母表达系统中的表达水平。
2.最优密码子使用频率(FOP)
由图3-a可知,基于毕赤酵母表达载体,密码子没有优化前,PNGase F基因序列的低利用率密码子(利用率低于40%的密码子)出现百分比为11%。这条未进行优化的基因采用串联稀有密码子,这些密码子可能降低翻译效率,甚至能够解散翻译装配物。由图3-b可知,通过密码子优化后,PNGase F基因在毕赤酵母系统中出现低利用率密码子的频率为0。
3. GC碱基含量(GC curve)
GC含量理想分布区域为30%-70%,在这个区域外的出现任何峰都会不同程度地影响转录和翻译效率。由图4-a、图4-b的PNGase F基因的GC碱基平均含量分布区域图对比可知,由图4-a中显示PNGase F基因GC碱基平均含量为38.74%,由图4-b中显示出优化后去除在30%-70%区域外出现的GC含量峰值,最终得到优化后PNGase F的GC碱基平均含量为42.60%。
实施例2:PNGaseF和PNGase F-opt基因的表达质粒构建
将密码子优化前后的PNGase F在5’端引入XhoI酶切位点序列,在3’端引入组氨酸标签(SEQ ID No:4)和XbaI酶切位点序列,并进行全基因合成,将合成的基因片段,构建到pUC57质粒(由南京金斯瑞科技有限公司提供)中,得到一种长期保存质粒,将优化前的质粒记为pUC57-PNGase F,优化后的质粒记为pUC57-PNGase F-opt质粒。
以pUC57-PNGase F和pUC57-PNGase F-opt质粒为模板,进行PCR扩增,所用引物序列如下:
上游引物(SEQ ID No:5):
M13F:CGC CAG GGT TTT CCC AGT CAC GAC
下游引物(SEQ ID No:6):
M13R:AGC GGA TAA CAA TTT CAC ACA GGA
反应总体积50μL,其中浓度为10μmol/L引物各加2.5μL,浓度为10mmol/L的dNTP加1μL,所用DNA聚合酶为Q5(购自New England Biolabs公司),2U/μL,加0.5μL。反应条件为98℃5秒、55℃45秒、72℃30秒,25个循环后,产物经1.0%琼脂糖凝胶电泳分析,结果显示产物大小与预期大小(1000bp)一致(结果如图5-a和图5-b所示)。分别用Xba I(R0145S,购自New England Biolabs公司)和Xho I(R0189S,购自New England Biolabs公司)双酶切后,1%琼脂糖电泳,得到的基因产物用DNA凝胶回收试剂盒(DP214,北京天根生化科技有限公司)纯化。用T4连接酶(M0202S,购自New England Biolabs)连接到pPICZαA质粒(V173-20,购自Invitrogen公司)中,转化到DH5α感受态细胞(CB101,购自北京天根生化科技有限公司)中,在含有博来霉素(购自Invitrogen公司)的LB固体培养基中37℃培养过夜。第二天挑取阳性克隆菌测序,比对,与预期序列完全一致,即得到PNGase F密码子优化前后的表达质粒,分别记为pPICZ-PNGase F和pPICZ-PNGase F-opt(质粒构建如图6-a,6-b所示)。
实施例3:含有重组PNGase F基因不同毕赤酵母宿主工程菌株的构建
YPDS固体培养基配制:Invitrogen公司Easy SelectPichia Expression Kit说明书提供。
1.含有密码子优化前后PNGase F宿主工程菌株的构建
按照Invitrogen公司Easy SelectPichia Expression Kit说明书的方法制备成电感受态细胞。将实施例2得到的质粒pPICZ-PNGase F和pPICZ-PNGase F-opt,用Sac I限制性内切酶(R0156S,购自New England Biolabs)酶切线性化,乙醇沉淀后将线性化载体,电转化进入到毕赤酵母感受态细胞中,涂布于YPDS固体培养基,30℃培养直到转化子长出后。
实施例4:含有密码子优化前后PNGase F基因工程菌株诱导表达及鉴定
BMGY培养基配制:酵母提取物10g/L,蛋白胨20g/L,K2HPO43g/L,KH2PO411.8g/L,YNB 13.4g/L,生物素4×10-4g/L,甘油10g/L。
BMMY培养基配制:酵母提取物10g/L,蛋白胨20g/L,K2HPO43g/L,KH2PO411.8g/L,YNB13.4g/L,生物素4×10-4g/L,甲醇5mL/L。
1.密码子优化前PNGase F宿主工程菌株甲醇诱导表达
挑取实施例3获得的宿主单克隆工程菌于5mL BMGY培养基中,于50mL无菌离心管中30℃,220rpm培养,至OD600=1.0-2.0时,取1mL保存菌种,并将剩余菌液重悬后转移到BMMY中小量诱导表达,每隔24h补加甲醇至终浓度为1%。一周后,离心收集菌液上清,通过SDS-PAGE凝胶电泳和蛋白免疫印迹分析(一抗为Anti-His,M20001,Abmart;二抗为Anti-Rabbit IgG-Peroxidase antibody produced in goat,A0545-1mL,购于Sigma-Aldrich),观察表达产物条带亮度,图7-a,图7-b为含有PNGase F基因工程菌株诱导表达鉴定图。由图7-a和图7-b可知,PNGase F蛋白在工程菌株中得到了表达。
2.密码子优化后PNGase F-opt工程菌株甲醇诱导表达
挑取实施例3获得的宿主单克隆工程菌于5mL BMGY培养基中,于50mL无菌离心管中30℃,220rpm培养,至OD600=1.0-2.0时,取1mL保存菌种,并将剩余菌液重悬后转移到BMMY中小量诱导表达,每隔24h补加甲醇至终浓度为1%。一周后,离心收集菌液上清,通过SDS-PAGE凝胶电泳和蛋白免疫印迹分析,观察表达产物条带亮度,图8-a,图8-b为含有PNGase F-opt基因工程菌株诱导表达鉴定图。由图8-a和图8-b可知,PNGase F-opt蛋白在工程菌株中得到了显著表达。
实施例5:PNGase F和PNGase F-opt重组蛋白的纯化
本专利构建的PNGase F和PNGase F-opt带有6×His-Tag,主要采用亲和层析纯化PNGaseF和PNGase F-opt蛋白。预装柱子选择为HisTrapcrude 5ml FF,具体步骤如下:
1.发酵液的除杂预处理
按实施例4得到PNGase F或PNGase F-opt宿主工程菌株发酵液,12000rpm,15min低温离心收集上清,加入磷酸二氢钠,氯化钠和咪唑,使其在上清中终浓度分别为20mM,300mM,20mM,0.22μm滤膜过滤。
2. Ni亲和层析纯化
运用全自动智能蛋白纯化系统(AKTA avant150,GE healthcare)UNICORN6.1操作软件对预处理获得的PNGase F和PNGase F-opt发酵液进行纯化工艺优化,最终确定为平衡缓冲液为20mM磷酸钠,300mM氯化钠,20mM咪唑,pH7.5,洗脱缓冲液为20mM磷酸钠,300mM氯化钠,500mM咪唑,pH7.5。线性洗脱,收集洗脱峰,PNGase F纯化色谱图及SDS-PAGE纯度鉴定见图9和图10,图10中PNGF为三条蛋白条带,上面两条带为糖基化修饰后的蛋白,分子量较低的条带无糖基化修饰,产量为5mg/L;PNGase F-opt纯化色谱图及SDS-PAGE纯度鉴定图见图11和图12。图10、12显示基因优化后PNGase F蛋白以糖基化修饰程度较高的两条带为主,优化后PNGase F-opt蛋白以糖基化修饰程度较低的条带为主,且产量明显高于优化前,最高可达20mg/L。通过SDS-PAGE电泳确定纯度后合并符合要求的收集管,过滤除菌,即得到符合要求的PNGase F蛋白,进行下一步活性测定试验。
实施例6:PNGase F-opt反应缓冲液的优化
反应缓冲液中对PNGase F-opt酶活性影响因素较多:如变性剂,表面活性剂,还原剂,去垢剂,缓冲液种类,pH值,浓度等等;利用DOE试验设计方法,对上述影响因素进行优化,表1所示为进行优化的反应缓冲液各种配方及浓度,及对应的PNGase F-opt在各缓冲体系下酶活。与优化前体系比较结果,图13所示PNGase F-opt蛋白活性得到了大幅提高,当变性剂浓度较低,缓冲体系浓度适中时酶活力得到最大提升,与缓冲液优化前相比活性提高了220%。
表1:DOE优化反应缓冲液中各组分及其浓度
实施例7:PNGaseF和PNGaseF-opt蛋白酶活性的测定
1.将纯化得到的PNGase F和PNGase F-opt用缓冲液(50mM NaCl,20mM Tris-HCl,5mM EDTA,50%Glycerol,pH7.5)透析,Pierce BCA蛋白浓度测定试剂盒(Cat No:23225)测定蛋白浓度,将PNGase F和PNGase F-opt稀释到1mg/ml;
2. 20μgRnase B中加入1μL糖蛋白变性缓冲液(1%SDS,1%β-ME),100℃加热10min;
3.待溶液冷却至室温后加入2μL 10%NP-40,2μl 10×Glycobuffer2(20mM磷酸盐,10mM EDTA,pH7.0),加入1μL稀释过的PNGase F,37℃准确反应1h;
4.反应液70℃加热10min终止反应,SDS-PAGE鉴定活性;1h能将95%的Rnase B底物切除的PNGase F单位定义为1个活性单位,图14所示为PNGase F和和PNGase F-opt活性鉴定结果,计算得到PNGase F-opt酶比活力为500000U/mg,PNGase F酶比活力为10000U/mg,优化后酶比活力提高了50倍。
实施例8:PNGaseF-opt在毕赤酵母系统表达的蛋白结构分析分析中的应用
抗体和重组蛋白在真核表达系统中表达时极易被糖基化修饰,其中N-糖型修饰为蛋白最常见的修饰类型,此类药物在进行肽图分析前需要先去除糖基化修饰,常用的去除糖基化修饰的方法为酶解法,用N-糖酰胺酶F去除糖基化修饰;在本实施例中,发明人用PNGase F-opt对毕赤酵母系统重组表达的重组人激肽释放酶(rhKLK1)(中国专利公开号:CN105802989)进行N糖基化处理,具体酶解过程参照实施例7中详细步骤;图15所示经N-糖酰胺酶F酶解后rhKLK1为单一的蛋白条带,分子量比酶解前相比明显降低,与大肠杆菌表达的无糖基化修饰的rhKLK1(中国专利公开号:CN103710367A)相比分子量接近,说明rhKLK1的糖蛋白已被完全去除,经酶解后激肽原酶蛋白氨基酸覆盖率完全符合要求,说明本专利中的PNGase F酶可应用于结构分析分析。
实施例9:PNGaseF-opt在CHO系统表达蛋白结构分析中的应用
含有糖基化修饰位点的蛋白在CHO表达系统中表达时会发生N糖基化修饰,分子量远大于理论分子量,且可能会使目的条带在电泳时呈现弥散状态,不利于蛋白分子表征及结构分析。本发明中的PNGase F酶可应用于糖蛋白结构的分析和性质的鉴定,图16所示酶解前CHO系统重组表达的人激肽释放酶(中国专利公开号:CN103710368)分子量非常弥散,且明显高于酶解后,经PNGase F酶解后CHO系统重组表达的人激肽释放酶(中国专利公开号:CN103710368)为均一的蛋白条带,分子量明显低于酶解前,说明此蛋白弥散是完全是由于糖基化修饰引起的。酶解后的CHO系统重组表达的人激肽释放酶(中国专利公开号:CN103710368)因没有糖基化的影响,更加有利于开展质量分析研究以及肽图分析研究等蛋白结构研究。
SEQUENCE LISTING
<110> 江苏众红生物工程创药研究院有限公司
<120> 一种重组N-糖酰胺酶F及其编码基因和应用
<130> 一种重组N-糖酰胺酶F及其编码基因和应用
<160> 6
<170> PatentIn version 3.3
<210> 1
<211> 948
<212> DNA
<213> Pichia pastoris
<400> 1
gccccagctg ataacaccgt taacatcaag acattcgata aggtcaaaaa cgccttcggt 60
gacggattgt ctcaatccgc agaaggtact tttactttcc cagccgacgt tactgcagtc 120
aagaccatta aaatgtttat caagaacgaa tgtcctaaca agacttgcga tgagtgggac 180
agatacgcta acgtttatgt taagaacaag actaccggtg aatggtacga gattggaaga 240
ttcattactc catattgggt tggtactgaa aagttgccta gaggacttga gattgatgtt 300
accgacttca aatcattgct tagtggtaac acagaattga agatctacac tgagacctgg 360
cttgctaaag gtagagagta ctccgttgat ttcgacattg tctatggaac tccagattac 420
aagtattcag ctgttgtccc tgttgtccaa tacaataagt cttccatcga cggtgttcca 480
tacggtaaag ctcatacttt ggccttgaag aagaacattc agttgcctac aaatactgaa 540
aaggcttact tgagaacaac tatctcaggt tggggacacg caaaaccata cgatgctggt 600
agtagaggat gtgctgagtg gtgctttaga acccatacaa ttgccatcaa caactctaac 660
actttccaac accagttggg tgcccttgga tgttctgcaa accctattaa caatcaatcc 720
ccaggaaatt ggaccccaga tagagctggt tggtgcccag gaatggccgt tcctactaga 780
attgacgtct tgaacaattc ccttatcgga tctacctttt cctacgaata taagttccag 840
aactggacaa acaatggtac taatggagat gcattctacg ctatttcaag tttcgttatc 900
gcaaaatcaa acacaccaat tagtgctcct gttgtcacta attaatag 948
<210> 2
<211> 948
<212> DNA
<213> Chryseobacterium meningosepticum
<400> 2
gctccggcag ataatacggt aaatattaaa acattcgaca aagtaaaaaa tgcctttggt 60
gacggattgt cccaaagtgc ggaaggaacc tttacatttc cggccgatgt aacagccgta 120
aaaacgatta agatgttcat taaaaatgaa tgtcctaata aaacttgtga tgaatgggat 180
cgttatgcca atgtttatgt aaaaaataaa acaacaggtg agtggtacga aataggacgc 240
tttattactc catattgggt gggaacggaa aaattacctc gtggactgga aattgatgtt 300
acagatttca aatctttact atccggaaat acagaactta aaatttatac ggagacatgg 360
ctggccaaag gaagagaata cagtgtagat ttcgatattg tatacgggac accggattat 420
aaatattcgg ctgtagtacc tgtagttcag tataacaaat catctattga cggagtccct 480
tatggtaaag cacatacatt ggctttgaaa aagaatatcc agttaccaac aaacacagaa 540
aaagcttatc ttagaactac tatttccgga tggggacatg ctaagccata tgatgcggga 600
agcagaggtt gtgcagaatg gtgcttcaga acacacacta tagcaataaa taattcgaat 660
actttccagc atcagctggg tgctttagga tgttcagcaa accctatcaa taatcagagt 720
ccgggaaatt ggactcccga cagagccggt tggtgcccgg gaatggcagt tccaacacgt 780
atagatgtac tgaataattc tttaataggc agtactttta gttatgaata taaattccag 840
aactggacaa ataacggaac caatggagat gctttttatg caatttccag ttttgtgatt 900
gcaaaaagta atacacctat tagtgctccg gtagttacaa actagtaa 948
<210> 3
<211> 314
<212> PRT
<213> Chryseobacterium meningosepticum
<400> 3
Ala Pro Ala Asp Asn Thr Val Asn Ile Lys Thr Phe Asp Lys Val Lys
1 5 10 15
Asn Ala Phe Gly Asp Gly Leu Ser Gln Ser Ala Glu Gly Thr Phe Thr
20 25 30
Phe Pro Ala Asp Val Thr Ala Val Lys Thr Ile Lys Met Phe Ile Lys
35 40 45
Asn Glu Cys Pro Asn Lys Thr Cys Asp Glu Trp Asp Arg Tyr Ala Asn
50 55 60
Val Tyr Val Lys Asn Lys Thr Thr Gly Glu Trp Tyr Glu Ile Gly Arg
65 70 75 80
Phe Ile Thr Pro Tyr Trp Val Gly Thr Glu Lys Leu Pro Arg Gly Leu
85 90 95
Glu Ile Asp Val Thr Asp Phe Lys Ser Leu Leu Ser Gly Asn Thr Glu
100 105 110
Leu Lys Ile Tyr Thr Glu Thr Trp Leu Ala Lys Gly Arg Glu Tyr Ser
115 120 125
Val Asp Phe Asp Ile Val Tyr Gly Thr Pro Asp Tyr Lys Tyr Ser Ala
130 135 140
Val Val Pro Val Val Gln Tyr Asn Lys Ser Ser Ile Asp Gly Val Pro
145 150 155 160
Tyr Gly Lys Ala His Thr Leu Ala Leu Lys Lys Asn Ile Gln Leu Pro
165 170 175
Thr Asn Thr Glu Lys Ala Tyr Leu Arg Thr Thr Ile Ser Gly Trp Gly
180 185 190
His Ala Lys Pro Tyr Asp Ala Gly Ser Arg Gly Cys Ala Glu Trp Cys
195 200 205
Phe Arg Thr His Thr Ile Ala Ile Asn Asn Ser Asn Thr Phe Gln His
210 215 220
Gln Leu Gly Ala Leu Gly Cys Ser Ala Asn Pro Ile Asn Asn Gln Ser
225 230 235 240
Pro Gly Asn Trp Thr Pro Asp Arg Ala Gly Trp Cys Pro Gly Met Ala
245 250 255
Val Pro Thr Arg Ile Asp Val Leu Asn Asn Ser Leu Ile Gly Ser Thr
260 265 270
Phe Ser Tyr Glu Tyr Lys Phe Gln Asn Trp Thr Asn Asn Gly Thr Asn
275 280 285
Gly Asp Ala Phe Tyr Ala Ile Ser Ser Phe Val Ile Ala Lys Ser Asn
290 295 300
Thr Pro Ile Ser Ala Pro Val Val Thr Asn
305 310
<210> 4
<211> 18
<212> DNA
<213> Pichia pastoris
<400> 4
catcaccatc accatcac 18
<210> 5
<211> 24
<212> DNA
<213> Pichia pastoris
<400> 5
cgccagggtt ttcccagtca cgac 24
<210> 6
<211> 24
<212> DNA
<213> Pichia pastoris
<400> 6
agcggataac aatttcacac agga 24
- 还没有人留言评论。精彩留言会获得点赞!