一种基因敲除方法及其sgRNA片段与应用与流程
本发明涉及基因编辑技术领域,特别是涉及一种基因敲除方法及其sgrna片段与应用。
背景技术:
crispr/cas9基因编辑技术是细菌和古细菌在噬菌体长期选择压力下进化出来的一种有效抵御外源dna和病毒入侵的免疫机制,是由一段rna通过碱基互补配对识别dna,指导cas9核酸内切酶切割识别的外源双链dna,诱发同源重组或非同源末端连接,进而实现在目的dna上进行编辑。crispr-cas系统对dna分子的靶向切割特性,使其可以被用于定向的基因修饰。2012年,doudan研究小组首次将crrnas:tracrrna二元复合体改造为单链rna嵌合体,并指导cas9蛋白在特定位点剪切。目前,crispr-cas9已经在人类细胞、小鼠、斑马鱼、果蝇、拟南芥等生物中成功实现了基因组定点修饰。尽管zfn、和talen技术也具有高效的基因编辑效率,但crispr/cas9定点基因编辑方面有独特优势。首先,crispr/cas9的靶点在基因组中分布频率高,几乎每8bp就有一个靶点,talen的靶点在基因组的分布频率大概是1/125bp,而zfn每500bp才有一个合适的靶点,crispr/cas9系统更容易在需要突变的点附近筛选到高效的靶点。其次,crispr/cas9在引入定点插入时比zfn和talen具有更精确的优势,这是由于cas9切割dna两条链的功能分别属于两个功能域,通过突变其中的一个功能域使cas9变成只切割一条链的切口酶,同时引入修复模板,这样大大降低了天然cas9切割双链引入随机突变的概率。因此,crispr/cas9基因编辑技术在研究基因的功能方面具有得天独厚的优势。
但现有基因编辑技术的编辑效率较低,操作较为复杂,亟待改进。
技术实现要素:
鉴于以上所述现有技术的缺点,本发明的目的在于提供一种基因敲除方法及其sgrna片段与应用,用于解决现有技术中基因编辑的效率较低等问题。
为实现上述目的及其他相关目的,本发明第一方面提供一种基因敲除方法,包括分别获得表达cas9和sgrna的转基因生物,再将两种转基因生物杂交,获得共表达阳性个体。
在本发明的一些实施例中,所述sgrna含有如seqidno.9、seqidno.10所示序列中的至少一种。
在本发明的一些实施例中,所述生物为家蚕,家蚕的中文学名为桑蚕,拉丁学名bombyxmorilinnaeus,是一种以桑叶为食料的鳞翅目泌丝昆虫,属无脊椎动物,节肢动物门蚕蛾科蚕蛾属桑蚕种;将cas9载体和sgrna载体分别导入家蚕胚胎中,得到对应的目标转基因家蚕,将获得的目标转基因家蚕杂交,获得共表达阳性个体。
在本发明的一些实施例中,利用piggybac重组表达载体将cas9基因和sgrna分别整合至家蚕基因组,含有cas9基因的piggybac重组表达载体由含有cas9基因的表达盒插入到载体pbac[3×p3-egfp]的bglⅱ酶切位点得到,含有sgrna的piggybac重组表达载体由含有sgrna的表达盒插入到载体pbac[3×p3-dsred]的bglⅱ酶切位点得到。
在本发明的一些实施例中,包括将表达盒ie1-cas9-sv40插入到最终载体pbac[3×p3-egfp],得到重组表达载体pbac[3×p3-egfp+ie1-cas9-sv40],将上述重组表达载体与辅助质粒混合,将混合液注射至蚕蛾的卵中,孵化得到g0代蚁蚕,蚁蚕羽化成蚕蛾蚕蛾回交或自交,得到g1代蛾圈,取g1代蛾圈中阳性转基因系家蚕,培育至羽化,自交选纯,获得表达cas9蛋白的转基因家蚕;将内源性基因序列接入家蚕内源性u6启动子后,再将sgrna序列连接至u6启动子,获得u6-sgrna表达盒,将u6-sgrna表达盒插入到载体pbac[3×p3-dsred],得到重组表达载体,将上述重组表达载体与辅助质粒混合,将混合液注射至蚕蛾的卵中,孵化得到g0代蚁蚕,蚁蚕羽化成蚕蛾蚕蛾回交或自交,得到g1代蛾圈,取g1代蛾圈中阳性转基因系家蚕,培育至羽化,自交选纯,获得表达sgrna的转基因家蚕;将表达cas9蛋白的转基因家蚕与表达sgrna的转基因家蚕杂交,获得共表达阳性个体。
本发明第二方面提供一种sgrna片段,含有如seqidno.9、seqidno.10所示序列中的至少一种。
本发明第三方面提供含有上述sgrna片段的重组载体、基因表达盒、转基因细胞系或转基因个体。
本发明第四方面提供一种含有上述sgrna片段的crispr/cas9载体,包括cas9表达载体以及含有上述sgrna片段的表达载体,该载体可以为基于piggybac转座载体的cas9蛋白表达载体。
在本发明的一些实施例中,所述cas9表达载体的启动子选自bmnpv极早期启动子ie1。
在本发明的一些实施例中,所述sgrna表达载体的启动子选自u6启动子。
在本发明的一些实施例中,cas9表达载体是以眼和神经特异的启动子3×p3启动的绿色荧光蛋白作为表达盒,绿色荧光蛋白作为筛选标记,筛选标记后含有启动cas9基因表达的ie1启动子;sgrna表达载体是以眼和神经特异的启动子3×p3启动的红色荧光蛋白作为表达盒,红色荧光蛋白作为筛选标记,筛选标记后含有启动sgrna基因表达的u6启动子。
在本发明的一些实施例中,所述cas9表达载体是以眼和神经特异的启动子3×p3启动的加强型绿色荧光蛋白egfp作为表达盒,加强型绿色荧光蛋白作为筛选标记,筛选标记后含有启动cas9基因表达的ie1启动子。
在本发明的一些实施例中,所述sgrna表达载体是含有以眼和神经特异的启动子3×p3启动的红色荧光蛋白作为表达盒,红色荧光蛋白作为筛选标记,筛选标记后含有启动sgrna基因表达的u6启动子。
本发明第五方面提供上述sgrna片段及其crispr/cas9载体在基因敲除中的应用。
如上所述,本发明的一种基因敲除方法及其sgrna片段与应用,具有以下有益效果:本发明简化了载体构建技术,提高了注射效率,对靶标序列的编辑可以在受精完成后持续进行,因而保证了编辑效率。
附图说明
图1显示为表达载体pbac[3×p3-egfp+ie1-cas9-sv40]载体示意图。
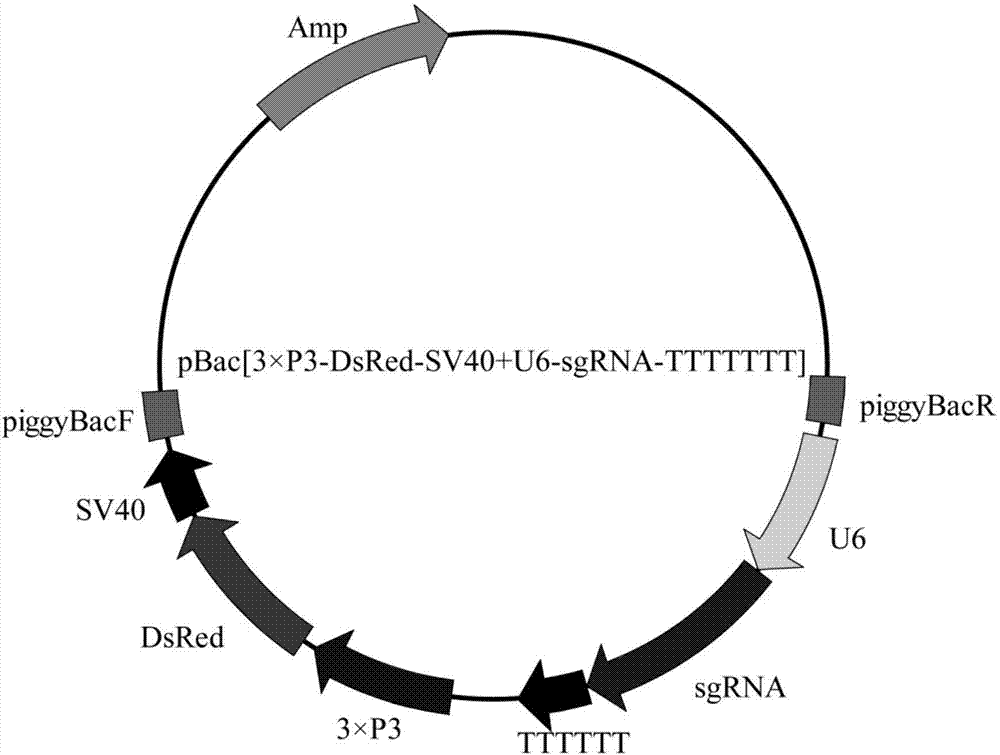
图2显示为表达载体pbac[3×p3-dsred+u6-sgrna]载体示意图。
图3显示为表达载体pbac[3×p3-egfp+ie1-cas9-sv40]获得的表达cas9的转基因家蚕品系各时期荧光图。
图4显示为表达载体pbac[3×p3-dsred+u6-bmdes3sgrna1]获得的表达sgrna的转基因家蚕品系各时期荧光图。
图5显示为表达cas9的转基因家蚕品系cas9蛋白表达图。
图6a显示为cas9表达品系与sgrna1表达品系,在转录水平检测cas9表达水平和在基因组水平u6-sgrna1检测图。
图6b显示为cas9表达品系与sgrna2表达品系,在转录水平检测cas9表达水平和在基因组水平u6-sgrna2检测图。
图7显示为bmdes3基因靶点1的基因编辑示意图。
图8显示为bmdes3基因靶点2的基因编辑示意图。
具体实施方式
以下通过特定的具体实例说明本发明的实施方式,本领域技术人员可由本说明书所揭露的内容轻易地了解本发明的其他优点与功效。本发明还可以通过另外不同的具体实施方式加以实施或应用,本说明书中的各项细节也可以基于不同观点与应用,在没有背离本发明的精神下进行各种修饰或改变。
piggybac转座子是在粉纹夜蛾中发现的一类转座子,piggybac转座系统介导的外源基因插入其能够识别基因组上的ttaa位点并携带外源基因插入,并且可以稳定遗传,被广泛应用于多个物种的转基因研究。在家蚕中,piggybac转座子介导的外源基因插入技术被广泛用于基因功能的研究。
实施例一
基于piggybac转座载体的cas9表达载体pbac[3×p3-egfp+ie1-cas9-sv40]的构建。
图1显示为表达载体pbac[3×p3-egfp+ie1-cas9-sv40]载体示意图。
cas9基因来自载体phsp70-cas9(购自addgene公司,网址:www.addgene.org/),cas9基因序列如seqidno.1所示。
将cas9基因插入到出发载体psl1180的多克隆位点,得到中间载体,然后将完整的含有cas9基因的表达盒ie1-cas9-sv40插入到最终载体pbac[3×p3-egfp]得到重组表达载体。
具体操作方法:
步骤s1、制备载体psl1180[cas9-sv40]
将cas9基因通过claⅰ/xbaⅰ限制性内切酶双酶切载体phsp70-cas9,回收得到cas9基因片段(回收操作按照takara胶回收(小量)试剂盒使用说明进行),将回收片段与相同酶切的psl1180[sv40]载体骨架片段按照takaradnaligationkitver2.1使用说明进行连接,得到psl1180[cas9-sv40]。
步骤s2、制备ie1启动子序列
pcr体外扩增,从bmnpv基因组中扩增ie1启动子序列,并在扩增片段上下游分别添加hindiii限制性内切酶和claⅰ限制性内切酶酶切位点,经t-a克隆到载体pmd19-t获得质粒,将获得的pmd19-t载体质粒用hindiii限制性内切酶和claⅰ限制性内切酶双酶切,回收ie1启动子序列(回收操作按照takara胶回收(小量)试剂盒使用说明进行),启动子ie1的序列如seqidno.2所示。
所需pcr反应程序为:①94℃预变性4min,②94℃变性40s,③55℃退火40s,④72℃延伸40s,⑤返回步骤②进行30个循环,⑥72℃终延伸10min,⑦12℃forever。
pcr扩增上、下游引物分别如seqidno.3、seqidno.4所示:
ie1-f(seqidno.3):5’-cgaagcttttgcagttcgggacataaatg-3’
ie1-r(seqidno.4):5’–cgatcgattagatccctagtcgtttggt-3’
步骤s3、制备中间载体psl1180[ie1-cas9-sv40]
通过hindiii限制性内切酶和claⅰ限制性内切酶双酶切步骤s1所得载体psl1180[cas9-sv40],回收载体骨架(回收操作按照takara胶回收(小量)试剂盒使用说明进行),与步骤s2获得的ie1启动子序列按照takaradnaligationkitver2.1使用说明进行连接,获得中间载体psl1180[ie1-cas9-sv40]。
步骤s4、制备表达盒片段ie1-cas9-sv40
通过ascⅰ限制性内切酶单酶切psl1180[ie1-cas9-sv40],按试剂盒要求回收并补平黏性末端,制得表达盒片段ie1-cas9-sv40。
步骤s5、制备载体骨架
通过bglⅱ限制性内切酶单酶切载体pbac[3×p3-egfp](参见参考文献:tomitam,munetsunah,satot,etal.transgenicsilkwormsproducerecombinanthumantypeiiiprocollagenincocoons.natbiotechnol,2003,21:52~56.),按照takara胶回收(小量)试剂盒使用说明进行回收载体骨架并补平去磷酸化,得到载体骨架片段。
步骤s6、制备目标载体pbac[3×p3-egfp+ie1-cas9-sv40]
将步骤s4制得的表达盒片段ie1-omelo-sv40与步骤s5制得的pbac[3×p3-egfp]载体骨架片段按照takaradnaligationkitver2.1使用说明进行连接,获得目标载体pbac[3×p3-egfp+ie1-cas9-sv40]。
本实施例构建获得的转基因重组载体以眼和神经特异的启动子3×p3启动的增强型绿色荧光蛋白(egfp)的表达盒,增强型绿色荧光蛋白作为筛选标记,其后含有ie1启动子,启动优化后cas9基因的表达。
实施例二
培育cas9基因的转基因系家蚕。
用市购的多化性家蚕品系305作为原始材料,亲代蚕卵经正常桑叶饲养并化蛾交配产卵。
取10nl~15nl浓度为400ng/μl的实施例一所制得重组载体质粒pbac[3×p3-egfp+ie1-cas9-sv40]与辅助质粒pha3pig混合。将混合液分别注射至400粒家蚕品系305雌蛾产下2h~6h的蚕卵中,用无毒胶水封口以后置于25℃、相对湿度为85%的环境中催青孵化。孵化后pbac[3×p3-egfp+ie1-cas9-sv40]获得156头g0代蚁蚕。
所得蚁蚕用桑叶饲养至羽化成蛾,将获得蚕蛾回交或自交,获得注射了pbac[3×p3-egfp+ie1-cas9-sv40]质粒的蛾圈37个。
蛾圈g1代蚕卵,用
将获得的阳性转基因家蚕饲养至羽化后进一步自交选纯,即获得能稳定遗传的在家蚕中全时期全组织表达cas9蛋白的家蚕转基因系,即ie1-cas9-sv40转基因家蚕系。
图3显示为表达载体pbac[3×p3-egfp+ie1-cas9-sv40]获得的表达cas9的转基因家蚕品系各时期荧光图。
图5显示为表达cas9的转基因家蚕品系cas9蛋白表达图。
实施例三
本实施例以家蚕内源性脂肪酸去饱和酶基因bmdes3为例,该基因为家蚕脂肪酸去饱和酶基因家族中的一员,需要说明的是,本发明的敲除方法适用于任何具有特异性适合靶点的单个内源性基因或具有类似结构域的基因家族的多重敲除。bmdes3基因序列如seqidno.5所示。
构建家蚕内源性脂肪酸去饱和酶基因bmdes3的打靶载体。
将设计好的bmdes3特异靶序列接入家蚕内源性u6启动子后,再与sgrna相连,然后将含有完整靶序列的sgrna表达盒插入到最终载体pbac[3×p3-dsred]得到重组表达载体。
图2显示为表达载体pbac[3×p3-dsred+u6-sgrna]载体示意图。
具体操作方法:
步骤s1、制备u6启动子序列
pcr体外扩增,从家蚕dz品系基因组中扩增u6启动子序列,经t-a克隆到载体pmd19-t获得质粒pmd19-t-u6。
所需pcr反应程序为:①94℃预变性4min,②94℃变性40s,③55℃退火40s,④72℃延伸40s,⑤返回步骤②进行30个循环,⑥72℃终延伸10min,⑦12℃forever。
pcr扩增上下游引物分别如seqidno.7、seqidno.8所示:
u6-f(seqidno.7):5’-aggttatgtagtacacat-3’
u6-r(seqidno.8):5’-acttgtagagcacgatat-3’
u6启动子序列如seqidno.6所示。
步骤s2、筛选bmdes3的sgrna靶点序列
根据crisprdirect在线分析工具(http://crispr.dbcls.jp/)预测家蚕内源性去饱和酶基因bmdes3的sgrna序列,同时根据软件分析靶序列在家蚕中的脱靶效率,最后选择高效编缉的sgrna序列,其序列为seqidno.9和seqidno.10。
target1(seqidno.9):ggcttatggcgagtgcataa
target2(seqidno.10):gcgtaactcgggatatgttt
需要说明的是,本实施例选择的sgrna序列是通过与家蚕基因组序列进行blast比对,筛选出的具有最佳特异性的两条序列,以有利于后续的敲除实验,当然,sgrna序列也有其他选择。
步骤s3、制备sgrna表达盒
以u6-bmdes3-f(seqidno.11)和u6-bmdes3-r11(seqidno.12)作为引物,以质粒pmd19-t-u6为模版进行体外扩增,得到扩增产物;将得到的扩增产物稀释50倍后作为模版,再次用引物u6-bmdes3-f(seqidno.11)和引物u6-bmdes3-r2(seqidno.13)进行体外扩增,得到扩增产物;将得到的扩增产物稀释50倍后作为模版,第三次用引物u6-bmdes3-f(seqidno.11)和引物u6-bmdes3-r3(seqidno.14)体外扩增得到扩增产物。扩增产物按照takara胶回收(小量)试剂盒使用说明进行回收,并用ncoi和ecorⅰ双酶切回收产物并补平及去磷酸化。获得表达bmdes3基因target1sgrna的表达盒u6-bmdes3sgrna1。
同理,用引物u6-bmdes3-f(seqidno.11)和引物u6-bmdes3-r21(seqidno.15),以质粒pmd19-t-u6为模版进行体外扩增得到扩增产物;将得到的扩增产物稀释50倍后作为模版,再次用引物u6-bmdes3-f(seqidno.11)和引物u6-bmdes3-r2(seqidno.13)进行体外扩增得到扩增产物;将得到的扩增产物稀释50倍后作为模版,第三次用引物u6-bmdes3-f(seqidno.11)和引物u6-bmdes3-r3(seqidno.14)体外扩增得到扩增产物。扩增产物按照takara胶回收(小量)试剂盒使用说明进行回收,并用ncoi和ecorⅰ双酶切回收产物并补平及去磷酸化。获得表达bmdes3基因target2sgrna的表达盒u6-bmdes3sgrna2。
所需pcr反应程序均为:①94℃预变性4min,②94℃变性40s,③60℃退火40s,④72℃延伸40s,⑤返回步骤②进行30个循环,⑥72℃终延伸10min,⑦12℃forever。
u6-bmdes3-f(seqidno.11):5’-gcgaattcaggttatgtagtacacatt-3’
u6-bmdes3-r11(seqidno.12):
5’-tatttctagctctaaaacttatgcactcgccataagccacttgtagagcacgatatt-3’
u6-bmdes3-r2(seqidno.13):
5’-caagttgataacggactagccttattttaacttgctatttctagctctaaaaca-3’
u6-bmdes3-r3(seqidno.14):
5’-gccatggaaaaaagcaccgactcggtgccactttttcaagttgataacggactag-3’
u6-bmdes3-r21(seqidno.15):
5’-tatttctagctctaaaacaaacatatcccgagttacgcacttgtagagcacgatatt-3’
步骤s4、制备载体骨架
通过bglⅱ限制性内切酶单酶切载体pbac[3×p3-dsred],按照takara胶回收(小量)试剂盒使用说明进行回收载体骨架并补平去磷酸化,得到载体骨架片段。
步骤s5、制备目标载体pbac[3×p3-dsred+u6-bmdes3sgrna1]和pbac[3×p3-dsred+u6-bmdes3sgrna2]
将步骤s3制得的表达盒片段u6-bmdes3sgrna1和u6-bmdes3sgrna2分别与步骤s5制得的pbac[3×p3-dsded]载体骨架片段按照takaradnaligationkitver2.1使用说明进行连接,获得目标载体pbac[3×p3-dsred+u6-bmdes3sgrna1]和pbac[3×p3-dsred+u6-bmdes3sgrna2]。
本实施例构建获得的转基因重组载体以眼和神经特异的启动子3×p3启动的红色荧光蛋白(dsded)的表达盒,红色荧光蛋白作为筛选标记,其后含有以u6启动子启动sgrna的表达盒。
实施例四
培育表达家蚕内源性基因bmdes3的sgrna1和sgrna2的转基因家蚕品系。
用市购的多化性家蚕品系305作为初始材料,亲代蚕卵经正常桑叶饲养至羽化后交配产卵。
取10nl~15nl浓度为400ng/μl的实施例三所制得的重组载体pbac[3×p3-dsred+u6-bmdes3sgrna1]和pbac[3×p3-dsred+u6-bmdes3sgrna2],分别与辅助质粒pha3pig混合。将混合液分别注射至400粒家蚕品系305雌蛾产下2h~6h的蚕卵中,用无毒胶水封口以后置于25℃、相对湿度为85%的环境中催青孵化。孵化后获得注射pbac[3×p3-dsred+u6-bmdes3sgrna1]质粒的g0代蚁蚕79头,注射pbac[3×p3-dsred+u6-bmdes3sgrna2]质粒的g0代蚁蚕93头。
所得蚁蚕用桑叶饲养至羽化成蛾,将获得蚕蛾回交或自交。pbac[3×p3-dsred+u6-bmdes3sgrna1]获得24个蛾圈,pbac[3×p3-dsred+u6-bmdes3sgrna2]获得31个蛾圈。
蛾圈g1代蚕卵,用
将获得的阳性转基因家蚕饲养至羽化成蛾并进一步自交选纯,即获得能稳定遗传的在家蚕中表达家蚕内源性基因bmdes3的sgrna1和sgrna2的转基因家蚕品系,即u6-bmdes3sgrna1转基因家蚕系和u6-bmdes3sgrna2转基因家蚕系。
图4显示为表达载体pbac[3×p3-dsred+u6-bmdes3sgrna1]获得的表达sgrna的转基因家蚕品系各时期荧光图。表达载体pbac[3×p3-dsred+u6-bmdes3sgrna2]获得的转基因家蚕品系各时期荧光图与图4类似。
图6a显示为cas9表达品系与sgrna1表达品系,在转录水平检测cas9表达水平和在基因组水平u6-sgrna1检测图。
图6b显示为cas9表达品系与sgrna2表达品系,在转录水平检测cas9表达水平和在基因组水平u6-sgrna2检测图。
测试例一
将实施例二获得的ie1-cas9-sv40转基因家蚕系和实施例四获得的u6-bmdes3sgrna1转基因家蚕系进行杂交,蚕卵用
测试例二
将实施例二获得的ie1-cas9-sv40转基因家蚕系和实施例四获得的u6-bmdes3sgrna2转基因家蚕系进行杂交,蚕卵用
上述实施例将crispr/cas9基因组定点编辑技术与piggybac转座子介导的基因插入技术相结合,即先分别获得cas9和sgrna的稳定转家蚕基因系,然后通过杂交的方式获得同时表达cas9和sgrna的家蚕个体,可有效提高靶标基因的敲除效率。同现有的直接注射sgrna混合纯化后的cas9蛋白、mrna或者能够转录为cas9mrna的表达载体技术相比,简化了载体构建技术,提高了注射效率,对靶标序列的编辑可以在受精完成后持续进行,因而保证了编辑效率。
综上所述,本发明提供了一种高效、稳定的cas9介导的靶标基因编辑方法。即先分别获得单独表达cas9蛋白和sgrna的转基因家蚕系,然后通过杂交的方式使cas9介导的靶标基因编辑更加高效的进行,避免了现有的注射表达载体后直接筛选阳性个体效率低的问题,共表达阳性个体中靶标基因的敲除效率达100%,从而更好地服务于基因功能研究,并在基因工程育种中发挥重要意义。
本发明适用的物种广泛,目前发现每个物种都具有相应于该物种的u6启动子,均可用于构建pbac[3×p3-dsred+u6-sgrna]表达载体,因而具有在所有物种中均可适用的潜力。
上述实施例仅例示性说明本发明的原理及其功效,而非用于限制本发明。任何熟悉此技术的人士皆可在不违背本发明的精神及范畴下,对上述实施例进行修饰或改变。因此,举凡所属技术领域中具有通常知识者在未脱离本发明所揭示的精神与技术思想下所完成的一切等效修饰或改变,仍应由本发明的权利要求所涵盖。
sequencelisting
<110>西南大学
<120>一种基因敲除方法及其sgrna片段与应用
<130>2017
<160>15
<170>patentinversion3.5
<210>1
<211>4272
<212>dna
<213>artificial
<220>
<223>cas9
<400>1
atggactataaggaccacgacggagactacaaggatcatgatattgattacaaagacgat60
gacgataagatggccccaaagaagaagcggaaggtcggtatccacggagtcccagcagcc120
gacaagaagtacagcatcggcctggacatcggcaccaactctgtgggctgggccgtgatc180
accgacgagtacaaggtgcccagcaagaaattcaaggtgctgggcaacaccgaccggcac240
agcatcaagaagaacctgatcggagccctgctgttcgacagcggcgaaacagccgaggcc300
acccggctgaagagaaccgccagaagaagatacaccagacggaagaaccggatctgctat360
ctgcaagagatcttcagcaacgagatggccaaggtggacgacagcttcttccacagactg420
gaagagtccttcctggtggaagaggataagaagcacgagcggcaccccatcttcggcaac480
atcgtggacgaggtggcctaccacgagaagtaccccaccatctaccacctgagaaagaaa540
ctggtggacagcaccgacaaggccgacctgcggctgatctatctggccctggcccacatg600
atcaagttccggggccacttcctgatcgagggcgacctgaaccccgacaacagcgacgtg660
gacaagctgttcatccagctggtgcagacctacaaccagctgttcgaggaaaaccccatc720
aacgccagcggcgtggacgccaaggccatcctgtctgccagactgagcaagagcagacgg780
ctggaaaatctgatcgcccagctgcccggcgagaagaagaatggcctgttcggaaacctg840
attgccctgagcctgggcctgacccccaacttcaagagcaacttcgacctggccgaggat900
gccaaactgcagctgagcaaggacacctacgacgacgacctggacaacctgctggcccag960
atcggcgaccagtacgccgacctgtttctggccgccaagaacctgtccgacgccatcctg1020
ctgagcgacatcctgagagtgaacaccgagatcaccaaggcccccctgagcgcctctatg1080
atcaagagatacgacgagcaccaccaggacctgaccctgctgaaagctctcgtgcggcag1140
cagctgcctgagaagtacaaagagattttcttcgaccagagcaagaacggctacgccggc1200
tacattgacggcggagccagccaggaagagttctacaagttcatcaagcccatcctggaa1260
aagatggacggcaccgaggaactgctcgtgaagctgaacagagaggacctgctgcggaag1320
cagcggaccttcgacaacggcagcatcccccaccagatccacctgggagagctgcacgcc1380
attctgcggcggcaggaagatttttacccattcctgaaggacaaccgggaaaagatcgag1440
aagatcctgaccttccgcatcccctactacgtgggccctctggccaggggaaacagcaga1500
ttcgcctggatgaccagaaagagcgaggaaaccatcaccccctggaacttcgaggaagtg1560
gtggacaagggcgcttccgcccagagcttcatcgagcggatgaccaacttcgataagaac1620
ctgcccaacgagaaggtgctgcccaagcacagcctgctgtacgagtacttcaccgtgtat1680
aacgagctgaccaaagtgaaatacgtgaccgagggaatgagaaagcccgccttcctgagc1740
ggcgagcagaaaaaggccatcgtggacctgctgttcaagaccaaccggaaagtgaccgtg1800
aagcagctgaaagaggactacttcaagaaaatcgagtgcttcgactccgtggaaatctcc1860
ggcgtggaagatcggttcaacgcctccctgggcacataccacgatctgctgaaaattatc1920
aaggacaaggacttcctggacaatgaggaaaacgaggacattctggaagatatcgtgctg1980
accctgacactgtttgaggacagagagatgatcgaggaacggctgaaaacctatgcccac2040
ctgttcgacgacaaagtgatgaagcagctgaagcggcggagatacaccggctggggcagg2100
ctgagccggaagctgatcaacggcatccgggacaagcagtccggcaagacaatcctggat2160
ttcctgaagtccgacggcttcgccaacagaaacttcatgcagctgatccacgacgacagc2220
ctgacctttaaagaggacatccagaaagcccaggtgtccggccagggcgatagcctgcac2280
gagcacattgccaatctggccggcagccccgccattaagaagggcatcctgcagacagtg2340
aaggtggtggacgagctcgtgaaagtgatgggccggcacaagcccgagaacatcgtgatc2400
gaaatggccagagagaaccagaccacccagaagggacagaagaacagccgcgagagaatg2460
aagcggatcgaagagggcatcaaagagctgggcagccagatcctgaaagaacaccccgtg2520
gaaaacacccagctgcagaacgagaagctgtacctgtactacctgcagaatgggcgggat2580
atgtacgtggaccaggaactggacatcaaccggctgtccgactacgatgtggaccatatc2640
gtgcctcagagctttctgaaggacgactccatcgacaacaaggtgctgaccagaagcgac2700
aagaaccggggcaagagcgacaacgtgccctccgaagaggtcgtgaagaagatgaagaac2760
tactggcggcagctgctgaacgccaagctgattacccagagaaagttcgacaatctgacc2820
aaggccgagagaggcggcctgagcgaactggataaggccggcttcatcaagagacagctg2880
gtggaaacccggcagatcacaaagcacgtggcacagatcctggactcccggatgaacact2940
aagtacgacgagaatgacaagctgatccgggaagtgaaagtgatcaccctgaagtccaag3000
ctggtgtccgatttccggaaggatttccagttttacaaagtgcgcgagatcaacaactac3060
caccacgcccacgacgcctacctgaacgccgtcgtgggaaccgccctgatcaaaaagtac3120
cctaagctggaaagcgagttcgtgtacggcgactacaaggtgtacgacgtgcggaagatg3180
atcgccaagagcgagcaggaaatcggcaaggctaccgccaagtacttcttctacagcaac3240
atcatgaactttttcaagaccgagattaccctggccaacggcgagatccggaagcggcct3300
ctgatcgagacaaacggcgaaaccggggagatcgtgtgggataagggccgggattttgcc3360
accgtgcggaaagtgctgagcatgccccaagtgaatatcgtgaaaaagaccgaggtgcag3420
acaggcggcttcagcaaagagtctatcctgcccaagaggaacagcgataagctgatcgcc3480
agaaagaaggactgggaccctaagaagtacggcggcttcgacagccccaccgtggcctat3540
tctgtgctggtggtggccaaagtggaaaagggcaagtccaagaaactgaagagtgtgaaa3600
gagctgctggggatcaccatcatggaaagaagcagcttcgagaagaatcccatcgacttt3660
ctggaagccaagggctacaaagaagtgaaaaaggacctgatcatcaagctgcctaagtac3720
tccctgttcgagctggaaaacggccggaagagaatgctggcctctgccggcgaactgcag3780
aagggaaacgaactggccctgccctccaaatatgtgaacttcctgtacctggccagccac3840
tatgagaagctgaagggctcccccgaggataatgagcagaaacagctgtttgtggaacag3900
cacaagcactacctggacgagatcatcgagcagatcagcgagttctccaagagagtgatc3960
ctggccgacgctaatctggacaaagtgctgtccgcctacaacaagcaccgggataagccc4020
atcagagagcaggccgagaatatcatccacctgtttaccctgaccaatctgggagcccct4080
gccgccttcaagtactttgacaccaccatcgaccggaagaggtacaccagcaccaaagag4140
gtgctggacgccaccctgatccaccagagcatcaccggcctgtacgagacacggatcgac4200
ctgtctcagctgggaggcgacaaaaggccggcggccacgaaaaaggccggccaggcaaaa4260
aagaaaaagtaa4272
<210>2
<211>638
<212>dna
<213>artificial
<220>
<223>ie1promoter
<400>2
ttgcagttcgggacataaatgtttaaatatatcaatgtctttgtgatgcgcgcgacattt60
ttgtaagttattaataaaatgcaccgacacgttgcccgacattatcattaaatccttggc120
gtagaatttgtcgggtccgttgtccgtgtgcgctagcatgcccgtaacggaccttgagct180
tttggcttcaaaggttttgcgcacagacaaaatgtgccacacttgcagctctgcttgtgt240
acgcgttaccacaaatcccaacggcgcagtgtacttgttatatgtaaataaatctcgata300
aaggcgcggcgcgcgaatgcagctgatcacgtacgctcctcgtgttccgttcaaggacgg360
tgttatcgacctcagattaatatttatcggccgactgttttcgtatccgctcaccaaacg420
ggtttttgcattaacattgtatgtcggcggatgttctgtatctaatttgaataaataaat480
gataaccgcattggttttagagggcataataaaaaaaatattattatcgtgttcgccatt540
ggggcagtataaattgacgttcatgttgaatattgtttcagttgcaagttgacattggcg600
gcgacacgatcgtgaacaaccaaacgactagggatcta638
<210>3
<211>29
<212>dna
<213>artificial
<220>
<223>ie1-f
<400>3
cgaagcttttgcagttcgggacataaatg29
<210>4
<211>28
<212>dna
<213>artificial
<220>
<223>ie1-r
<400>4
cgatcgattagatccctagtcgtttggt28
<210>5
<211>1359
<212>dna
<213>artificial
<220>
<223>bmdes3
<400>5
atggcgcctaaaaatgttgaatatattgaaatagctcatcgaagagcagcagaaaagaaa60
actcatgtcagttttcctcaactcaaatacccctcgctaagggacgaagggttaagagac120
ccggtacaatggttaatcgggaaatccatggacgacggagcagaaggcttatggcgagtg180
cataatgggatttacaatttcaatgatttcctcgagaagcatcccggtggggcggagtgg240
ttggagctctctaagggtaccgatatcacagaagccttcgaaagtcatcacctcaattcg300
tctgtgaataaagttctcgagaaatattacgttagggaagctaaaacacctaggaattcg360
ccttttacttttgaagatgatggattctaccgcacattgaaaagagcggtcgttgaagaa420
ttaaaaaaagttcctaaacatatcccgagttacgctgatatgatcatcgatggtctcttc480
gcgactttgctcatatcttcagcggtttcatgttgggccaacgattattgggtggtaatg540
tcagcgtatttgatcgcctcgttgagtttggcatgggtcacagttgccgctcacaattac600
atccacaggaaaactaattggagaatgtacttgttcaatttgagtttgtggtcttatagg660
gatttcagagtatcacacgccctttctcaccatctttatccgaacacccttatggattta720
gaagttagcggatttgagcctttagtgatttggaatccgaggaaaaagccggtacatgcc780
aaatttgcttttttgattgaacagattacattcccgtttctgttcgttttaaatttcctg840
aagcggttaattttaaatttcctacgcgaaggattcttcaccgagcactaccgttggcat900
gatggtgtagggttcactttacctgtctggatgtggctcgtgagcggttgcagtttctac960
gaagcagcggtgatgtggttgtggattgtgtgcaccgcgagctacatattctttagcatc1020
ggatcgaatgctgctcaccaccatcctgacatctttaaggacggtgaccaagttagggac1080
gttacacctgactggggtatgcacgaactggaggctgtgatggatcgcaccgacattaat1140
ggaaaccttttcaaggtgatgacgtttttcggagaccacgcccttcatcatctgttcccg1200
acattggatcacgccgtgttaccctatctgtatccagttttcttagatttatgtcagaag1260
tatcgcgccaattttagaatgacatcgtcattggatttgtttataggacagattaagatg1320
acgcttaagacggaacctaatatattagataataactaa1359
<210>6
<211>467
<212>dna
<213>artificial
<220>
<223>u6promoter
<400>6
aggttatgtagtacacattgttgtaaatcactgaattgttttagatgattttaacaatta60
gtacttattaatattaaataagtacataccttgagaatttaaaaatcgtcaactataagc120
catacgaatttaagcttggtacttggcttatagataaggacagaataagaattgttaacg180
tgtaagacaaggtcagatagtcatagtgattttgtcaaagtaataacagatggcgctgta240
caaaccataactgttttcatttgtttttatggattttattacaaattctaaaggttttat300
tgttattatttaatttcgttttaattatattatatatctttaatagaatatgttaagagt360
ttttgctctttttgaataatctttgtaaagtcgagtgttgttgtaaatcacgctttcaat420
agtttagtttttttaggtatatatacaaaatatcgtgctctacaagt467
<210>7
<211>18
<212>dna
<213>artificial
<220>
<223>u6-f
<400>7
aggttatgtagtacacat18
<210>8
<211>18
<212>dna
<213>artificial
<220>
<223>u6-r
<400>8
acttgtagagcacgatat18
<210>9
<211>20
<212>dna
<213>artificial
<220>
<223>target1
<400>9
ggcttatggcgagtgcataa20
<210>10
<211>20
<212>dna
<213>artificial
<220>
<223>target2
<400>10
gcgtaactcgggatatgttt20
<210>11
<211>27
<212>dna
<213>artificial
<220>
<223>u6-bmdes3-f
<400>11
gcgaattcaggttatgtagtacacatt27
<210>12
<211>57
<212>dna
<213>artificial
<220>
<223>u6-bmdes3-r11
<400>12
tatttctagctctaaaacttatgcactcgccataagccacttgtagagcacgatatt57
<210>13
<211>54
<212>dna
<213>artificial
<220>
<223>u6-bmdes3-r2
<400>13
caagttgataacggactagccttattttaacttgctatttctagctctaaaaca54
<210>14
<211>55
<212>dna
<213>artificial
<220>
<223>u6-bmdes3-r3
<400>14
gccatggaaaaaagcaccgactcggtgccactttttcaagttgataacggactag55
<210>15
<211>57
<212>dna
<213>artificial
<220>
<223>u6-bmdes3-r21
<400>15
tatttctagctctaaaacaaacatatcccgagttacgcacttgtagagcacgatatt57
- 还没有人留言评论。精彩留言会获得点赞!
- 用CRISPR/Cas9技术易于筛选获得目的基因敲除细胞系的方法及产品与制造工艺
- 一种Candida amazonensis的FLP/FRT基因敲除方法与制造工艺
- 玉米低镁条件下特异性转运镁离子基因ZmMGT10及其应用的制造方法与工艺
- CRISPR‑Cas9特异性敲除猪SLA‑3基因的方法及用于特异性靶向SLA‑3基因的sgRNA与制造工艺
- 利用CRISPR/Cas9敲除人PD‑1基因构建靶向CD19CAR‑T细胞的方法与制造工艺
- 一种构建基因替代小鼠研究野生型和突变体非肌性肌球蛋白重链II功能的方法与制造工艺
- 构建在海马体区域特异性敲除IKKα基因的小鼠模型的方法及打靶载体和试剂盒与制造工艺
- 猪ApoE基因敲除打靶载体及其构建方法和应用与制造工艺
- 一种血小板减少症的斑马鱼模型的制造方法与工艺
- 基于CRISPR技术敲除FUT8基因的方法与制造工艺