一种C3-芳香型吡咯并吲哚类生物碱及其合成方法与流程
本发明属于生物工程领域,具体涉及一种c3-芳香型吡咯并吲哚类生物碱及其酶催化合成方法。
背景技术:
吡咯并吲哚生物碱是一大类重要的活性天然产物,这大类化合物普遍具有优良的生理活性,如抗菌,抗肿瘤,抗疟原虫和胆碱酯酶抑制活性等,部分重要成员也被广泛的用于临床,如:治疗青光眼和胃动力障碍的physostigmine和抗阿尔茨海默病的posiphen等;因此一直以来都是药物化学,天然产物化学和有机化学领域中研究最多的天然产物家族之一。结构上,吡咯并吲哚核心骨架由一个吡咯和吲哚环在2,3位形成的一个稠合的并环结构;根据c3-位取代基的不同,可以分为简单取代(包括无取代基,羟基取代、异戊烯基和甲基取代)和芳香取代(主要为含色氨酸基团)两大类型。其中芳香类型由于为数最多,生物活性最为广泛,对其的化学和生物学研究也是这大类天然产物中最为活跃和重视的。
通过化学合成构建特殊的分子骨架是进行药学研究的重要保障,然而由于吡咯并吲哚中存在一个邻位的c3季碳和c2叔碳手性中心,因此其化学合成极具难度。手性季碳中心由于四周的取代基都为化学惰性的碳原子,难以通过简单的碳碳键的搭建进行构建,并且立体专一性的控制手性中心的形成还需要使用合适的手性催化剂或手性底物来诱导;由于绝大部分的手性催化剂异常复杂,能获得对映体光学纯度不高(e.e.~90%),需要大量实验筛选以及后续进一步纯化,才能获得光学纯的对映体。也正是由于这些原因,构建手性季碳中心一直以来都是有机化学中最为困难的反应之一。目前尽管已有少数c3-芳香型吡咯并吲哚生物碱被化学全合成,但是由于需要立体专一性的构建c2-c3叔碳、季碳手性中心,因此合成路线都很复杂且收率低下;缺乏高效、简单,统一的方法去构建分子骨架及创造其结构多样性。
酶催化反应以其高度区域、立体选择性,以及操作简单、环保安全以及可重复利用等优异的性质,成为工业合成的一个非常瞩目的方面。但是由于c3-芳香型吡咯并吲哚类生物碱结构的特殊性,且酶催化反应具有较高的专一性,按照传统方法,从酶库中随机筛选获得能够用于骨架合成的酶具有很高的挑战性。因此,一种更为可行的思路是通过研究这类天然产物的生物合成机制,从中寻找到合成分子骨架的酶,在此基础上利用酶催化反应,实现分子骨架高效合成和结构多样化。虽然目前简单取代的吡咯并吲哚生物碱的生物合成机制比较清楚,但是芳香型的生物合成机制大部分还未曾揭示,仅c3-c3’偶联型吡咯并吲哚的生物合成机制被解析;其他的如c3与5’,6’,7’位或n原子链接的分子骨架的形成还未有报道。
技术实现要素:
本发明的目的之一在于提供一种c3-芳香型吡咯并吲哚类生物碱,该类化合物具有体外抗神经损伤功能,具有潜在药用价值。
本发明的目的之二在于提供一种酶催化合成c3-芳香型吡咯并吲哚类生物碱的方法,该方法操作简单,催化效率高,所得目标产物光学纯度高,后处理简单,环保安全性好。
为达到上述目的,本发明所采用的技术方案如下:
一种c3-芳香型吡咯并吲哚类生物碱,为nas-1-nas-30,其结构式如下:
第二方面,提供一种酶催化合成c3-芳香型吡咯并吲哚类生物碱的方法,包含如下步骤:
1)将n-boc-色氨酸类似物(i)与另一分子氨基酸甲酯(ii)经缩合反应得到中间体(iii);所得中间体(iii)经过脱除叔丁氧羰基及分子内酯的胺解反应形成含有色氨酸的环二肽底物(iv);
2)含有nascb-p450基因以及菠菜来源的电子传递系统fd/fdr的质粒构建以及改造抗溶菌大肠杆菌gb05dir-t7;
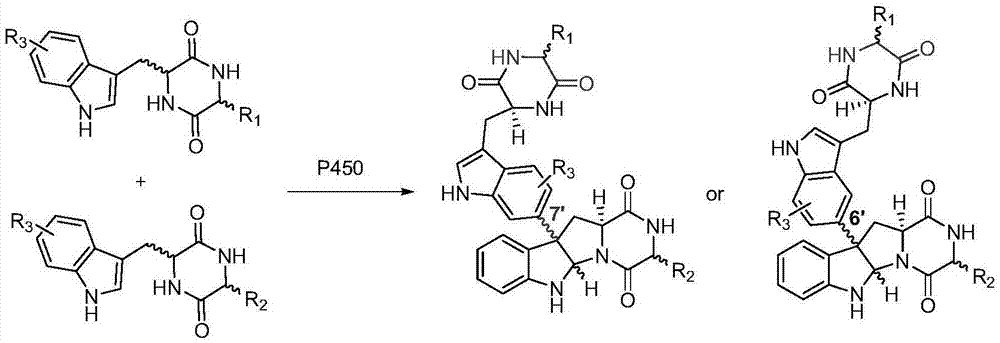
3)将构建好的含有nascb-p450基因以及菠菜来源的电子传递系统fd/fdr的质粒共同转化到改造过后的抗溶菌大肠杆菌gb05dir-t7中,用全菌催化的方法催化环二肽底物(iv)形成不同区域/立体选择性的二聚产物(v)和(vi);
该方法采取以下合成路线:
上式中:
式(i)、式(iii)、式(iv)、式(v)和式(vi)中,r3代表氢,卤素;式(ii)代表20种天然氨基酸的甲酯,其中r1和r2为20种天然氨基酸上а位上的取代部分;式(v)和式(vi)代表不同区域/立体选择性的环二肽二聚产物。
优选地,上述步骤2)中所述的nascb-p450基因来源于斐济海底的链霉菌streptomycessp.cmb-mq030,并在金斯瑞生物科技有限公司,利用optimumgenetm算法,进行大肠杆菌优化表达基因的合成,其核苷酸序列如seqidno:1所示,通过ndei-xhoi两个酶切位点构建在pet21a质粒上,形成nascb-p450-pet21a;菠菜来源的电子传递系统fd/fdr经过与上述nascb-p450相同的大肠杆菌密码子优化后,fd和fdr的核苷酸序列分别如seqidno:2和3所示,fd基因通过ncoi-hindiiii酶切位点构建到prsf-duet上,随后将fdr基因通过ndei-xhoi酶切位点构建到该prsf-duet的另一多克隆微位点区,形成prsf-dute-fd/fdr;
步骤2)中改造抗溶菌大肠杆菌gb05dir-t7的方法如下:
首先,以bl21菌液为模板,用t7-for/t7-rev引物对(seqidno:4-5)进行pcr得到t7-rna聚合酶基因片段;以质粒pib139为模板,用am-for/am-rev引物对(seqidno:6-7)进行pcr得到am(安普霉素)抗性基因片段,然后将这两个片段回收之后,以这两个片段为模板,用t7-for/am-rev引物对pcr获得全长片段;取出低温保存的菌种gb05-dir(该菌通过将recet基因整合到dh10b的基因组上得到的,具体参见junfuetal,full-lengthreceenhanceslinear-linearhomologousrecombinationandfacilitatesdirectcloningforbioprospecting,naturebiotechnology,30(5):440-446.),然后在lb固体培养基平板上划线,37℃培养过夜;挑取单一菌落于5mllb液体培养基中,37℃,220rpm,培养过夜;接种1ml菌液转接至100ml新鲜的lb液体培养基中,同时加入1ml1m阿拉伯糖,37℃220rpm培养,当od600值达到0.6~0.8时,将锥形瓶从摇床中取出,冰浴25min,并且不时缓慢摇匀以保证菌液充分冷却;然后将菌液转移至提前预冷的50ml离心管中,4℃,4000rpm,离心10min,倒掉培养基,收集菌体;先加入1ml预冷的无菌水,用枪尖轻轻吹打,充分重悬菌体,再加入20ml预冷的无菌水,4℃,4000rpm,离心10min,弃去上清,收集菌体;重复上述步骤一次,然后再用20ml冰冷的10%甘油洗涤两次;最后重悬于2ml冰冷的10%甘油中。将重悬的菌体分装到预冷的1.5ml离心管中(50-100μl每管);将装有菌体的离心管用液氮速冻,然后放入-80℃冰箱保存;取一管感受态,然后加入5μl的pcr产物,轻轻混匀,转入预冷的1mm的电转化杯,将电转杯的外部擦干,然后将电转杯放进电转仪,设定参数:电压1.8kv,时间5ms,电转杯间距1mm,启动对细胞的电脉冲,电击结束后,迅速取出电转杯,向电转杯中加入1mllb液体培养基;用移液枪将细胞转移至灭菌的1.5ml离心管中,于37℃220rpm复苏1h,然后离心,倒掉多余的培养基,用剩下的培养基重悬菌体,将菌体均匀涂布在含am抗生素的lb固体平板上,37℃培养过夜;挑取多个单一菌落于5mllb液体培养基中(含am抗生素),37℃,220rpm,培养过夜;然后以菌液为模板,用t7-for/am-rev引物对进行pcr,得到3576bp的条带即为重组菌株gb05dir-t7。
优选地,步骤3)具体实施如下:将prsf-dute-fd/fdr和nascb-p450-pet21a共转化到重组菌株gb05dir-t7中,于37℃培养过夜,挑取单克隆至5ml含有5μlamp、5μlam、5μlkm的lb中于37℃,220转下培养过夜,并转至500ml的lb培养基(含有0.5mlamp、0.5mlam、0.5mlkm)中,37℃220rpm培养,当od600值达到0.8~1.0时,将锥形瓶从摇床中取出,降温至18℃,加入0.5mlala,0.5mlfe2+(0.2mmfeso4),100μliptg(1m),于18℃220rpm下培养18~20h后,4000转下离心收菌,弃去lb培养基,菌体用50ml1×m9培养基重悬后置入无菌的100ml三角瓶中,并投入环二肽底物(iv),于18℃,220rpm下反应24h;之后用等体积乙酸乙酯萃取分液,收集上层有机相,减压蒸馏后用甲醇溶解,选用半制备柱hplc分离纯化。
第三方面,提供上述c3-芳香型吡咯并吲哚类生物碱在制备抗神经损伤药物中的应用。
本发明与现有技术相比具有以下优点:
本发明通过合成一系列含有色氨酸的环二肽底物,结合一步生物酶催化的合成途径,利用酶的高度立体选择性来催化一系列不同区域/立体选择性的c3-芳香型吡咯并吲哚类生物碱,得到的产物具有体外抗神经损伤活性,具有潜在药用价值。该方法原料易得、成本低、避免金属催化剂及大量有机试剂的应用,绿色环保,便于大规模生产c3-芳香型吡咯并吲哚类生物碱,解决了现有技术通过手性拆分造成原料浪费的难题,且相对于金属催化,所有涉及到的催化用酶简单易得,环保安全,操作简单,可应用于大规模工业生产。
附图说明
图1为用全菌催化的方法催化环二肽底物(iv)形成不同区域/立体选择性的二聚产物(v)和(vi)的反应路线图。
图2为二烷基磺酸钠聚丙烯酰胺凝胶(sds-page)电泳检测表达相关蛋白的电泳图。图中泳道1到泳道8蛋白分别为:marker;nascb,44kda;gdh,30.6kda;fdr,80.3kda;fd,31.3kda。
图3为nascb-p450酶体外催化wl-al和其它环二肽底物反应的液相数据(a)cwl-alandcwl-vl,(b)cwl-alandcwl-il,(c)cwl-alandcwl-ll,(d)cwl-alandcwl-ml,(e)cwl-alandcwl-fl。
图4为nascb-p450酶体外催化cwl-pl和其它环二肽底物反应的液相数据:cwl-plandcwl-al(a),cwl-plandcwl-il(b),cwl-plandcwl-ml(c),cwl-plandcwl-vl(d),cwl-plandcwl-ll(e)andcwl-plandcwl-fl(f)。
图5为nascb-p450酶体外催化cwl-vl和其它环二肽底物反应的液相数据:cwl-vlandcwl-il(a),cwl-vlandcwl-ml(b),cwl-vlandcwl-yl(c),cwl-vlandcwd-pl(d),cwl-vlandcwl-ll(e),cwl-vlandcwl-fl(f)andcwl-vlandcwd-pd(g)。
图6为nascb-p450酶体外催化所有底物自身二聚液相结果图:cwl-pl(a),cwl-al(b),cwl-vl(c),7-cl-cwl-pl(d)and7f-cwl-pl(e)。
图7a为化合物nas-10的hresims图;
图7b为化合物nas-10的1hnmr图;
图7c为化合物nas-10的13cnmr图;
图7d为化合物nas-10的hsqc图;
图7e为化合物nas-10的hmbc图;
图7f为化合物nas-10的rosey图。
图8a为化合物nas-11的hresims图;
图8b为化合物nas-11的1hnmr图;
图8c为化合物nas-11的13cnmr图;
图8d为化合物nas-11的hsqc图;
图8e为化合物nas-11的hmbc图;
图8f为化合物nas-11的rosey图。
图9a为化合物nas-12的hresims图;
图9b为化合物nas-12的1hnmr图;
图9c为化合物nas-12的13cnmr图;
图9d为化合物nas-12的hsqc图;
图9e为化合物nas-12的hmbc图;
图9f为化合物nas-12的rosey图。
图10a为化合物nas-27的hresims图;
图10b为化合物nas-27的1hnmr图;
图10c为化合物nas-27的13cnmr图;
图10d为化合物nas-27的hsqc图;
图10e为化合物nas-27的hmbc图;
图10f为化合物nas-27的rosey图。
具体实施方式
通过以下详细说明结合附图可以进一步理解本发明的特点和优点。所提供的实施例仅是对本发明方法的说明,而不以任何方式限制本发明揭示的其余内容。
【实施例1】环二肽底物的合成
例1-1、l-trp-l-gln的合成
合成路线如下:
氮气保护下,在事先烘干的250ml圆底烧瓶中加入谷氨酰胺甲酯100mg(0.46mmol,1.0equiv),60ml无水二氯甲烷,降温至0℃,同时缓慢滴加三乙胺0.28ml(2.06mmol,4.5equiv),之后加入hobt.h2o105.4mg(0.68mmol,1.5equiv),n-boc-色氨酸212.8mg(0.96mmol,2.0equiv),剧烈搅拌均匀后加入edc.hcl131.8mg(0.68mmol,1.5equiv)。滴加完后升至室温反应16小时,反应结束后旋蒸除去溶剂,加入50ml饱和碳酸氢钠水溶液,用乙酸乙酯萃取三次,每次50ml,合并乙酸乙酯相,并用无水硫酸镁干燥1小时,旋蒸除去溶剂既得中间体1,该产物不需纯化直接用于下一步反应。
在事先烘干的100ml圆底烧瓶中加入用20ml无水二氯甲烷溶解的中间体1,降温至0℃,同时缓慢滴加1ml三氟乙酸,之后升至室温反应4h。反应结束后。减压蒸馏除去溶剂及三氟乙酸,并用5ml甲醇溶解,降温至0℃后,逐滴加入1ml氨水溶液。之后升至室温反应10h,过滤得到析出的固体即为产物环二肽底物cw-n(11)。
利用上述例1-1所述方法,合成了以下31个环二肽底物:
【实施例2】关于nascb-p450基因的分子克隆及蛋白表达操作
例2-1、编码nascb-p450酶的基因的获得
通过基因组测序以及分析cmb-mq030的基因组,鉴定出同时具有cdps和nascb-p450基因(seqidno.1)的基因簇来源于斐济海底的链霉streptomycessp.cmb-mq030,并在金斯瑞生物科技有限公司,利用optimumgenetm算法,通过ndei和xhoi两个酶切位点将合成得到如seqidno.1所示的碱基序列构建在pet28a上,获得携带有编码nascb-p450基因的pet28a质粒,即nascb-p450-pet28a。例2-2、表达nascb-p450酶工程菌的获得
取1μlnascb-p450-pet28a质粒加入到放在冰上的入大肠杆菌bl21(de3)感受态细胞中,冰浴30min后于42℃下热击90s,之后放在冰上冰浴2min,加入1mllb培养基,置于37°下培养,1h后涂布于含有50μg/ml卡那霉素抗生素的lb选择性平板上,37℃倒置培养12小时,挑取单克隆,37°下在加入卡那霉素抗生素的的lb长起来的即为表达nascb-p450酶所需的工程菌。
例2-3、酶的纯化
上述例2-2培养的菌体6000rpm,4℃离心10分钟,菌体重悬于15ml破菌缓冲液(25mmhepes,ph7.5,300mmnacl,5mm咪唑,10%甘油),超声破菌,细胞裂解液12000rpm,4℃,离心60分钟。按照每1l菌液用2mlni-ida琼脂糖树脂的比例加入1.6mlni-ida琼脂糖树脂于细胞裂解上清液中,4℃结合1小时。蛋白树脂结合物在重力作用下用不同浓度的咪唑缓冲液a(25mmhepes,ph7.5,300mmnacl,10%甘油)洗脱,收集洗脱液,用12%sds-page胶检测蛋白大小及纯度,将纯度大于90%的蛋白缓冲液合并并回收。纯化之后的nascb-p450酶,按照gehealthcare公司的操作说明,用pd-10的凝胶柱换到蛋白保存缓冲液b(25mmhepes,ph7.5,50mmnacl,10%甘油),然后用10kda(gehealthcare)超滤管浓缩,之后用液氮速冻保存在-80℃,得到目标的p450酶(seqidno:8)。
菠菜来源的电子传递系统fd/fdr经过与上述nascb-p450相同的大肠杆菌密码子优化后,fd通过ecori-hindiii酶切位点构建到含有trx的psj5载体上,fdr通过ecori-hindiii酶切位点构建到含有mbp-tag的psj8载体上,重组质粒转化大肠杆菌bl21(de3)感受态细胞,挑取转化子于含有相应抗性的lb培养基中培养,0.1mmiptg诱导表达,ni-ida纯化得到相应的纯酶,测序得到fd的氨基酸序列如seqidno:9所示,fdr的氨基酸序列如seqidno:10所示。
体外催化所用纯酶的sds-page胶图如图2所示。第1泳道是蛋白marker,第2泳道为nascb-p450蛋白,大小为44kda;第3泳道为葡萄糖脱氢酶(gdh),30.6kda;第4泳道为fdr,大小为80.3kda;第5泳道为fd,蛋白大小为31.3kda,该胶图证明所有体外催化用的蛋白大小均正确,适用于体外催化系统。例2-4、用于全菌催化的工程菌的获得
首先,nascb-p450-pet28a和pet21a分别用ndei-xhoi两个限制性内切酶做酶切,体系如下:质粒25μl,ndei2μl,xhoi2μl,cutsmartbuffer5μl,ddh2o16μl,之后置于37℃下酶切反应3h;跑胶回收后,用t4连接酶进行酶连,体系如下:t4连接酶1μl,t4dnaligasebuffer1μl,nascb-p450基因片段4μl,酶切以后的pet21a片段1μl,ddh2o3μl,之后置于18℃过夜酶连;最后将上述酶连体系转化到dh5a中,挑点酶切验证后,提取质粒即为全菌催化系统用的nascb-p450-pet21a质粒,用类似的方法,fd基因通过ncoi-hindiii酶切位点构建到prsf-duet上,随后将fdr基因通过ndei-xhoi酶切位点构建到该prsf-duet的另一多克隆位点区,形成prsf-dute-fd/fdr。在获得nascb-p450-pet21a和prsf-dute-fd/fdr两个质粒后,各取1μl共同加到重组菌株gb05dir-t7的钙转感受态中,冰浴30min后于42°下热击90s,再冰浴2min,加入1mllb培养基,37°下培养1h,之后涂布于km/amp双重抗性的平板上,37°倒置培养过夜,挑取单克隆至含有km/amp双重抗性的lb中37°培养8h即可获得用于全菌催化的工程菌株。
【实施例3】nascb-p450酶体外和体内催化反应
例3-1、nascb-p450酶体外催化反应
反应体系为500μl,催化用缓冲液为100mmph7.0的heppes缓冲液,其中含有20mmd-葡萄糖,0.2mg/ml葡萄糖脱氢酶,5mm烟酰胺腺嘌呤二核苷酸磷酸(nadp+),环二肽底物2mm以及5%(v/v)助溶剂dmso,最后加入【实施例2】例2-3纯化的p450酶终浓度为0.5mg/ml,置于4℃,静止反应24小时,反应结束后加入等体积的乙酸乙酯萃取三次,合并乙酸乙酯相,hplc分析,分析结果如图3-图6所示,液相分析显示共催化产生了30个产物,且所有产物均已在液相图上标识。
例3-2、nascb-p450全菌催化反应制备化合物
根据体外催化实验结果,将例2-4中所述的全菌催化用的工程菌株转接至500ml的lb培养基(含有0.5mlamp、0.5mlam、0.5mlkm)中,37℃220rpm培养,当od600值达到0.8~1.0时,将锥形瓶从摇床中取出,降温至18℃,加入0.5mlala,0.5mlfe2+(0.2mmfeso4),100μliptg(1m),于18℃220rpm下培养18~20h后,4000转下离心收菌,弃去lb培养基,菌体用50ml1×m9培养基重悬后置入无菌的100ml三角瓶中,并投入环二肽底物(iv),于18℃,220rpm下反应24h;之后用等体积乙酸乙酯萃取分液,收集上层有机相,减压蒸馏后用甲醇溶解,选用半制备柱hplc分离纯化,最终得到了体外催化实验中的30个产物,经结构鉴定后该30个产物结果如下所示:
nas-1-30共有四种不同构型的产物,代表物分别是nas-10、11、12、27,以下表1-4是这4种化合物的核磁共振数据:
表1.化合物nas-10的nmr数据
表2.化合物nas-11的nmr数据
表3.化合物nas-12的nmr数据
表4.化合物nas-27的nmr数据
nas-10的质谱及核磁谱图如图7a-7f所示。
nas-11的质谱及核磁谱图如图8a-8f所示。
nas-12的质谱及核磁谱图如图9a-9f所示。
nas-27的质谱及核磁谱图如图10a-10f所示。
【实施例4】抗神经损伤活性实验:
以谷氨酸盐诱导的pc-12细胞损伤凋亡实验为例,所需材料:培养基,fbs血清,马血清,谷氨酰胺,青霉素/链霉素,96孔板,pbs,cck-8,尼莫地平,谷氨酸(l-glu)等。
样品配置:样品母液浓度为10mm,稀释100倍,即100μm应用液(加药体积20μl,总体系200μl,即样品终浓度为母液稀释1000倍,为10μm)。
方法:1.取对数生长期细胞10000个/孔,160μl/孔,接种于96孔板;2.培养24h后,分别加入待测样品20μl,每个浓度设置4个复孔;同时设置空白组,模型组,阳性药(尼莫地平20μm)组;加药预处理1h后,加入l-glu(15mm),每孔20μl;3.作用24h后,吸弃上清,补加含10%cck-8新培养基,37℃孵育2h后震荡溶解10min,450nm测定各孔吸光值。计算方法:抑制率%=(od空白组-od实验组)/od空白组*100%,实验结果如下表5和表6,实验结果表明nas-1-nas-28对抗老年痴呆作用效果不明显,而对谷氨酸诱导的细胞凋亡有很好的保护作用,尤其是化合物nas-12,27,10and11,展现出了比阳性对照药尼莫地平更好的效果,这四个化合物有一些相同之处,首先上面的环二肽均为cw-p,并且成键方式为c3-c6’,说明cw-p及c3-c6’类型的成键方式对提高活性方面很关键。
表5.化合物nas-1-28抗aβ25-35(a4559)-诱发的pc-12细胞损伤凋亡实验结果
表6.化合物nas-1-28抗谷氨酸盐诱导的pc-12细胞损伤凋亡实验结果
序列表
<110>武汉大学
<120>一种c3-芳香型吡咯并吲哚类生物碱及其合成方法
<160>10
<170>siposequencelisting1.0
<210>1
<211>1212
<212>dna
<213>斐济海底的链霉菌(streptomycessp.cmb-mq030)
<400>1
atgaccaccaccgcaaccctgacctatccgtttcacgattggagtcaggagctgagcccg60
cgctatgcacagctgcgtgcaagcgatgcacctgtgtgcccggtggtgagtgaaggtacc120
ggcgatccgctgtggctggttacccgctatgccaccgccgttaaactgctggaagacagc180
cgcttcagcagtgaagccgcacaagcaagcggtgccccgcgtcaggaacctgttgaactg240
cgcgcaccgggtacacgtggtgacgcaatcgcaatgttacgtgaagccggcctgcgtagc300
gtgttagccgatggtctgggtccgcgtgcagttcgtcgccaccagaagtggattcatgag360
tacgccgaaaccctgattggtgaactggtggatcgtgaaggcacctttgatctggcacgc420
gagtttgcagaacctctgagcagcgcagtggtgagtcgtaccctgctgggcgaactgacc480
agcgatgaacgcgcacgtctggtgggctgggcagacacaggcctgcgcttttgcggcgca540
acccatgaagaacaggttcgcgccttcaccgaaatgcaccgcttttttctggagcatgca600
cgtcgcttagcagccggcccgggcgaacatctgctgaaacacattgccgaggcaccgaca660
ccggcaggtccgttaagcgatgaagccctggccgaagcagccgaactgctggtggttgcc720
ggttttccgaccagcagcggctttctgtgtggtgcactgattacactgctgcgtcacccg780
gaaagcgtgcaggagctgcatacccatccggatcgcgtgccgagcgcagttgaagaactg840
ctgcgccatacaccgctggcaacaggcgccgcaaaacgcatggcaacagaggacctggag900
ctggatggtgttcgtattggtgcaggtgaggtggtgatggttagctttgaggccgttaac960
cgcgacccggatgcctttgaagatccggatcgttttcgcccgggccgcgaaggtccgatg1020
cattttggctttggccgcggccgtcatacctgtccgggtaatcgcctggcccgctgtctg1080
attgaagccaccgttcgtgccgtggcctgtcatccgggcttacgcctggcagtggcaccg1140
gaagaaattcgctggcacgagggtctgtttttccgtcgcccgcgcgcactgcctgccacc1200
tggtaactcgag1212
<210>2
<211>291
<212>dna
<213>菠菜(spinaciaoleracea)
<400>2
gctgcctacaaggtaaccttggtaacacccaccggtaacgtagagtttcaatgcccagac60
gatgtttacatcttggatgctgctgaagaagaaggcattgacttgccttactcatgcaga120
gctgggtcgtgctcttcatgcgccggaaagcttaagacaggtagtcttaaccaagatgat180
cagagttttttggatgacgatcagatcgatgaaggatgggttcttacctgtgctgcttac240
cctgttagtgatgttactattgagacccacaaggaagaggagcttactgcc291
<210>3
<211>942
<212>dna
<213>菠菜(spinaciaoleracea)
<400>3
cagatcgcctctgatgtggaggcacctccacctgctcctgctaaggtagagaaacattca60
aagaaaatggaggaaggcattacagttaacaagtttaagcctaagaccccttacgttgga120
agatgtcttcttaacaccaaaattactggggatgatgcacccggagagacctggcacatg180
gttttttcccatgaaggagagatcccttacagagaagggcaatccgttggggttattcca240
gatggggaagacaagaatggaaagccccataagttgagattgtactcgatcgccagcagt300
gctcttggtgattttggtgatgctaaatctgtttcgttgtgtgtaaaacgactcatctac360
accaatgacgctggagagacgatcaagggagtctgctccaacttcttgtgtgacttgaaa420
cccggtgctgaagtgaagttaacaggaccagttggaaaggagatgctcatgcccaaagac480
cctaacgcgacaattatcatgcttggaactggaacggggattgctcctttccgttcattc540
ttgtggaagatgttcttcgaaaagcatgatgattacaagtttaacggcttggcttggctt600
ttcttgggtgtacccacaagcagttctcttctctacaaagaggaatttgagaagatgaag660
gaaaaggctccagacaacttcaggctggattttgcagtgagcagagagcaaactaacgag720
aaaggggagaagatgtacattcaaacccgaatggcacaatacgcagttgagctatgggaa780
atgttgaagaaagataatacttatgtctacatgtgtggtctcaagggaatggaaaaggga840
attgacgacattatggtttcattggctgctgcagaaggcattgattggattgaatacaag900
aggcagttgaagaaggcagaacaatggaacgttgaagtctac942
<210>4
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>4
atgaccatgattacggattcactggccgtcgtggcccgctccggatttactaactggaag60
aggc64
<210>5
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>5
gcactccaccgctgatgacatgtatatctccttttacgcgaacgcgaagtccgac55
<210>6
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>6
gtcggacttcgcgttcgcgtaaaaggagatatacatgtcatcagcggtggagtgc55
<210>7
<211>63
<212>dna
<213>人工序列(artificialsequence)
<400>7
gccatcaaaaataattcgcgtctggccttcctgtagccatcagccaatcgactggcgagc60
ggc63
<210>8
<211>401
<212>prt
<213>斐济海底的链霉菌(streptomycessp.cmb-mq030)
<400>8
metthrthrthralathrleuthrtyrprophehisasptrpsergln
151015
gluleuserproargtyralaglnleuargalaseraspalaproval
202530
cysprovalvalsergluglythrglyaspproleutrpleuvalthr
354045
argtyralathralavallysleuleugluaspserargpheserser
505560
glualaalaglnalaserglyalaproargglngluprovalgluleu
65707580
argalaproglythrargglyaspalailealametleuarggluala
859095
glyleuargservalleualaaspglyleuglyproargalavalarg
100105110
arghisglnlystrpilehisglutyralagluthrleuileglyglu
115120125
leuvalasparggluglythrpheaspleualaarggluphealaglu
130135140
proleuserseralavalvalserargthrleuleuglygluleuthr
145150155160
seraspgluargalaargleuvalglytrpalaaspthrglyleuarg
165170175
phecysglyalathrhisglugluglnvalargalaphethrglumet
180185190
hisargphepheleugluhisalaargargleualaalaglyprogly
195200205
gluhisleuleulyshisilealaglualaprothrproalaglypro
210215220
leuseraspglualaleualaglualaalagluleuleuvalvalala
225230235240
glypheprothrserserglypheleucysglyalaleuilethrleu
245250255
leuarghisprogluservalglngluleuhisthrhisproasparg
260265270
valproseralavalglugluleuleuarghisthrproleualathr
275280285
glyalaalalysargmetalathrgluaspleugluleuaspglyval
290295300
argileglyalaglygluvalvalmetvalserpheglualavalasn
305310315320
argaspproaspalaphegluaspproaspargpheargproglyarg
325330335
gluglypromethispheglypheglyargglyarghisthrcyspro
340345350
glyasnargleualaargcysleuileglualathrvalargalaval
355360365
alacyshisproglyleuargleualavalalaproglugluilearg
370375380
trphisgluglyleuphepheargargproargalaleuproalathr
385390395400
trp
<210>9
<211>97
<212>prt
<213>菠菜(spinaciaoleracea)
<400>9
alaalatyrlysvalthrleuvalthrprothrglyasnvalgluphe
151015
glncysproaspaspvaltyrileleuaspalaalaglugluglugly
202530
ileaspleuprotyrsercysargalaglysercyssersercysala
354045
glylysleulysthrglyserleuasnglnaspaspglnserpheleu
505560
aspaspaspglnileaspgluglytrpvalleuthrcysalaalatyr
65707580
provalseraspvalthrilegluthrhislysgluglugluleuthr
859095
ala
<210>10
<211>314
<212>prt
<213>菠菜(spinaciaoleracea)
<400>10
glnilealaseraspvalglualaproproproalaproalalysval
151015
glulyshisserlyslysmetglugluglyilethrvalasnlysphe
202530
lysprolysthrprotyrvalglyargcysleuleuasnthrlysile
354045
thrglyaspaspalaproglygluthrtrphismetvalpheserhis
505560
gluglygluileprotyrarggluglyglnservalglyvalilepro
65707580
aspglygluasplysasnglylysprohislysleuargleutyrser
859095
ilealaserseralaleuglyasppheglyaspalalysservalser
100105110
leucysvallysargleuiletyrthrasnaspalaglygluthrile
115120125
lysglyvalcysserasnpheleucysaspleulysproglyalaglu
130135140
vallysleuthrglyprovalglylysglumetleumetprolysasp
145150155160
proasnalathrileilemetleuglythrglythrglyilealapro
165170175
pheargserpheleutrplysmetphepheglulyshisaspasptyr
180185190
lyspheasnglyleualatrpleupheleuglyvalprothrserser
195200205
serleuleutyrlysgluglupheglulysmetlysglulysalapro
210215220
aspasnpheargleuaspphealavalserarggluglnthrasnglu
225230235240
lysglyglulysmettyrileglnthrargmetalaglntyralaval
245250255
gluleutrpglumetleulyslysaspasnthrtyrphetyrmetcys
260265270
glyleulysglymetglulysglyileaspaspilemetvalserleu
275280285
alaalaalagluglyileasptrpileglutyrlysargglnleulys
290295300
lysalagluglntrpasnvalgluvaltyr
305310
- 还没有人留言评论。精彩留言会获得点赞!