阿洛酮糖的生产方法与流程
本发明涉及利用微生物生产阿洛酮糖的方法。
背景技术:
阿洛酮糖(d-psicose)为果糖(d-fructose)的3号碳的差向异构体(epimer),虽然像普通糖类一样具有甜味,但由于无法在人体内代谢,因而属于因卡路里几乎为零而对糖尿病及肥胖患者而言可作为代替白糖的功能性甜味剂来使用的功能性糖。并且,具有通过抑制在肝脏参与脂质合成的酶的活性来减少腹部肥胖的功能,是当前作为糖尿病和动脉硬化治疗剂来进行研究的糖。
像这样,随着阿洛酮糖作为甜味剂备受瞩目,在食品产业领域,研发可有效生产阿洛酮糖的方法的必要性逐渐增加。由于在天然物质内的糖蜜处理过程或葡萄糖异构化反应过程中存在非常少量的阿洛酮糖,因而以往的阿洛酮糖生产主要通过化学过程来实现。比利克(bilik)等人曾研发利用钼酸(molybdicacid)离子的催化作用来由果糖生产阿洛酮糖的技术。麦克唐纳(mcdonald)通过对1,2:4,5-二-o-丙酮缩甘油-β-d-果糖(1,2:4,5-di-o-isopropylidene-bata-d-fructopyranose)进行三步骤化学处理过程来生产了阿洛酮糖。并且,多纳(doner)通过对乙醇、三乙胺及果糖一同进行加热的方法生产了阿洛酮糖。但是,这些通过化学方法生产阿洛酮糖的方法花费很多费用,相反,存在生产效率低、产生大量副产物的问题。
作为基于生物学方法实现的阿洛酮糖生产方法,肯·伊兹莫里(kenizumori)等人证实了可利用微生物细胞反应来从半乳糖醇(galacitol)、d-塔格糖(d-tagatose)或d-塔罗糖醇(d-talitol)等生产阿洛酮糖。但是,这些基质属于自然界中非常稀缺的糖或糖醇,具有成本高的缺点。
酶转换方法有在重组大肠杆菌生产并提炼分离微生物菊苣假单胞菌st-24(pseudomonascichoriist-24)的d-塔格糖-3-差向异构酶(d-tagatose-3-epimerase)来使果糖酶转换成阿洛酮糖的方法,伊兹莫里等人曾利用固定d-塔格糖-3-差向异构酶的反应系统来以约25%的转换率生产了阿洛酮糖。
根据这种现有技术,为了由果糖生产阿洛酮糖,以往以通过提炼酶、固定所提炼的酶来提高阿洛酮糖生产性的方向进行了研究。但这种酶提炼过程实际需要很多时间和费用。
因此,需要一种可通过省略酶提炼步骤来使制造成本变低并以高效率生产阿洛酮糖的菌株及利用其来生产阿洛酮糖的方法。
在韩国公开专利第2006-125971号公开了基于阿洛酮糖差向异构化酶的阿洛酮糖的生产方法。
现有技术文献
专利文献
韩国公开专利第2006-125971号
非专利文献
choijg,juyh,yeomsjandohdk(2011),improvementinthethermostabilityofd-psicose3-epimerasefromagrobacteriumtumefaciensbyrandomandsite-directedmutagenesis,applenvironmicrobiol77(20):7316-20
技术实现要素:
要解决的问题
本发明的目的在于提供可明显改善阿洛酮糖的生产量及生产速度的阿洛酮糖的生产方法。
解决问题的方案
根据本发明的一实施方式,所提供的阿洛酮糖的生产方法包括在微生物内使作为基质的果糖和其差向异构酶在40℃以上的反应温度下发生反应的步骤。
本发明的发明人发现,若在40℃以上的温度下使作为基质的果糖和其差向异构酶进行反应,则可明显增加阿洛酮糖的生产量及生产速度,随着温度的增加,阿洛酮糖的生产量及生产速度也随之增加,由此研究出本发明。
另一方面,差向异构酶等的酶通常在40℃以上的高温下产生热变性,由此失去活性,但本发明在微生物内执行上述反应,因而与以酶的状态露出于外部的情况相比,在外部环境下受到保护,因此能够以即使在高温下也不会产生变性的方式执行上述反应。
由于阿洛酮糖的生产能力随着反应温度的增加而增加,因此,只要在40℃以上且在不使微生物因热量而受损或不使蛋白质或糖变性的范围内,则反应温度不受特殊限制。例如,反应温度可在40℃至50℃、40℃至60℃、40℃至70℃、40℃至80℃、40℃至90℃、45℃至60℃、45℃至70℃、45℃至80℃、45℃至90℃、50℃至70℃、50℃至80℃、50℃至90℃、55℃至60℃、55℃至70℃、55℃至80℃、55℃至90℃、60℃至70℃、60℃至80℃、60℃至90℃、70℃至80℃、70℃至90℃等。优选地,从使上述阿洛酮糖的生产能力极大化的方面出发,反应温度的下限可以是50℃以上,从抑制热损伤及变性的方面出发,反应温度的上限可以是90℃以下。
本说明书中的术语“微生物”可以是可在液体培养基中进行培养的细胞。
上述微生物可内在性表达上述差向异构酶或借助形质转换来表达上述差向异构酶。在此情况下,可通过在微生物内生成差向异构酶来借助上述果糖与差向异构酶的微生物内反应连续执行阿洛酮糖的生产。
在上述微生物被形质转换成编码有差向异构酶的基因来表达差向异构酶的情况下,编码上述差向异构酶的基因可以是编码源于序列1的根癌土壤杆菌(agrobacteriumtumefaciens)的阿洛酮糖-3-差向异构酶的基因或编码源于序列2的粪厌氧棒状菌(anaerostipescaccae)的阿洛酮糖-3-差向异构酶的基因。
源于根癌土壤杆菌的阿洛酮糖-3-差向异构酶可具有序列3的氨基酸序列,源于粪厌氧棒状菌的阿洛酮糖-3-差向异构酶可具有序列4的氨基酸序列。
优选地,从差向异构酶具有更优秀的高温稳定性方面出发,编码差向异构酶的基因可以是编码序列5的阿洛酮糖-3-差向异构酶的氨基酸序列的基因。
上述序列5的氨基酸序列为源于根癌土壤杆菌的阿洛酮糖-3-差向异构酶的氨基酸序列中的第33个氨基酸被亮氨酸取代或第213个氨基酸被半胱氨酸取代的序列,在参考文献1中指出,热稳定性优秀。
本发明的发明人确认了源于梭菌属的阿洛酮糖-3-差向异构酶具有优秀的热稳定性,参照图7,在热稳定性高的源于梭菌属的阿洛酮糖-3-差向异构酶中,一个氨基酸具有与之相对应的序列,或具有所有如上所述的两个序列。
因此,不仅是源于根癌土壤杆菌的阿洛酮糖-3-差向异构酶,源于其他菌株的阿洛酮糖-3-差向异构酶也被确认到与源于上述根癌土壤杆菌的阿洛酮糖-3-差向异构酶的氨基酸序列的第33个氨基酸、第213个氨基酸相对应的氨基酸序列为对提高热稳定性非常重要的序列。
这种氨基酸序列的具体例可有在序列6的氨基酸序列中第32个氨基酸被亮氨酸取代或第196个氨基酸被半胱氨酸取代的序列。序列6为对图7中的用方框所标注的序列进行排列的序列,是在本发明中所使用的根癌土壤杆菌、粪厌氧棒状菌、鲍氏梭菌、源于海氏梭菌(clostridiumhylemonae)的阿洛酮糖-3-差向异构酶氨基酸序列(序列3、序列4、序列9、序列10)的共同碱基序列。因此,上述微生物可以是被编码上述氨基酸序列的基因取代的,但是并不限定于此。
并且,同样,从优秀的高温稳定性、阿洛酮糖的生产能力方面出发,差向异构酶可以是源于梭菌属的阿洛酮糖-3-差向异构酶,编码其的基因可以是编码源于序列7的鲍氏梭菌(clostridiumbolteae)的阿洛酮糖-3-差向异构酶的基因,或可以是编码源于序列8的海氏梭菌(clostridiumhylemonae)的阿洛酮糖-3-差向异构酶的基因。优选地,从使高温稳定性极大化的方面出发,可以是编码源于序列8的海氏梭菌的阿洛酮糖-3-差向异构酶的基因。
源于鲍氏梭菌的阿洛酮糖-3-差向异构酶可具有序列9的氨基酸序列,源于海氏梭菌的阿洛酮糖-3-差向异构酶可具有序列10的氨基酸序列。
在所例示的上述阿洛酮糖-3-差向异构酶中,源于粪厌氧棒状菌的阿洛酮糖-3-差向异构酶、具有在序列6的氨基酸序列中的第32个氨基酸被亮氨酸取代或第196个氨基酸被半胱氨酸取代的序列的阿洛酮糖-3-差向异构酶、源于鲍氏梭菌的阿洛酮糖-3-差向异构酶及源于海氏梭菌的阿洛酮糖-3-差向异构酶等的表示最佳活性的ph为7以下,是较低的。
上述微生物为原核细胞或真核细胞,可在液体培养基中进行培养,并可在上述的高温温度下进行培养。例如,上述微生物可以是细菌、霉或它们的组合。细菌可以是革兰氏阳性细菌、革兰氏阴性细菌或它们的组合,优选地,从提高阿洛酮糖生产率方面出发,可以是革兰氏阳性细菌。革兰氏阴性细菌可以是埃希氏菌属(escherichia)。革兰氏阳性细菌可以是芽孢杆菌属、棒状杆菌属、放线菌属、乳酸菌或它们的组合。霉可以是酵母、克鲁维酵母属或它们的组合。
在本发明的阿洛酮糖的生产方法中,果糖和差向异构酶的反应可在40℃以上的温度执行,优选地,微生物可以是热稳定性高的高温型微生物(thermophiles)。例如,可以是棒状杆菌属、放线菌属,更优选为谷氨酸棒状杆菌,最优选为向谷氨酸棒状杆菌atcc13032导入编码上述差向异构酶的基因的微生物。
埃希氏菌属微生物可以是大肠杆菌,具体地,可以是向dh5α、mg1655、bl21(de)、s17-1、xl1-blue、bw25113或它们的组合导入编码差向异构酶的基因的微生物。
并且,上述大肠杆菌可以是编码内在性6-磷酸果糖激酶的基因及由阿洛糖代谢操纵子(allosemetabolicoperon)构成的一个区域形成非活性化的菌。
编码上述6-磷酸果糖激酶的基因可有具有序列11的核苷酸序列的,6-磷酸果糖激酶可以是具有序列12的氨基酸序列的。
构成上述阿洛糖代谢操纵子的基因为rpib、alsr、alsb、alsa、alsc、alse及alsk,可以是其中的一个以上的基因形成非活性化的。
上述rpib、alsr、alsb、alsa、alsc、alse及alsk基因可有分别具有序列13、序列14、序列15、序列16、序列17、序列18及序列19的核苷酸序列的。
上述rpib、alsr、alsb、alsa、alsc、alse及alsk基因可以是分别编码序列20、序列21、序列22、序列23、序列24、序列25及序列26的氨基酸序列的。
术语“非活性化”意味着上述基因的表达减少或未形成表达的情况。上述“非活性化”可通过本领域公知的方法实现。例如,可通过同源重组(homologuousrecombination)来形成非活性化。上述同源重组的例有转位子突变(transposonmutagenesis)或p1转导。
棒状杆菌属微生物可以是谷氨酸棒状杆菌,具体地,可以是向谷氨酸棒状杆菌atcc13032导入编码差向异构酶的基因的。
棒状杆菌属微生物可以是将内在性d-果糖转换成d–果糖1-磷酸盐并使作为向菌体内进行输送的pts输送系统的ptsf(eiifru、frua、ncgl1861、gi:19553141、ec2.7.1.69)基因缺损或形成非活性化的。
ptsf基因可以是具有序列27的核苷酸序列的,也可以是编码序列28的氨基酸序列的。
由于阿洛酮糖由d-果糖生成,因而若使上述基因缺损或形成非活性化,则可抑制果糖的磷酸化,从而可明显改善阿洛酮糖的生成效率。
并且,棒状杆菌属微生物可以是使编码甘露醇2-脱氢酶(mannitol2-dehydrogenase)的mtld(ncgl0108、gi:19551360、ec1.1.1.67)基因缺损或形成非活性化的。
mtld基因可以是具有序列29的核苷酸序列的,也可以是编码序列30的氨基酸序列的。
上述果糖和其差向异构酶的反应在微生物内执行,因而可在包含果糖的培养基中培养微生物。
上述培养基可以是2yt培养基、lb培养基、tb培养基等的包含酵母提取物和氮源的营养培养基。
培养基所包含的果糖的浓度不受特别限制,例如,可形成1%(w/v)至80%(w/v)的浓度,在以上范围中,例如可形成1%(w/v)至35%(w/v)、10%(w/v)至80%(w/v)、20%(w/v)至80%(w/v)、30%(w/v)至80%(w/v)、40%(w/v)至80%(w/v)等的范围。优选为1%(w/v)至50%(w/v)。
并且,培养基可以是包含碳源、氮源、必要金属离子、维生素等的在本领域通常使用的规定(defined)培养基,碳源包括葡萄糖、甘油等,氮源包括氨、尿素(urea)等,必要金属离子包括钠、钾、钙、镁、锰、钴等。
上述培养可以是连续、半连续或批量(batch)形式的培养。
上述微生物可在包含果糖的培养基中以使菌体的浊度(在600nm吸光度下测定的测定值,以下称为od600)达到0.01至300的浓度接种,例如,菌体的浊度可达到1至300、10至300、20至300、5至300或者40至300。通过使用包含高浓度酶的菌体,可在包含高浓度果糖的培养基中使果糖有效地转换成阿洛酮糖。
上述培养可通过还添加用于诱导编码差向异构酶的基因的表达的物质来执行。
用于诱导基因的表达的物质并不受特殊限制,可以是在本领域通常使用的物质。
在本发明的阿洛酮糖的生产方法中,可在仅包含作为基质的果糖和用于供给辅助因子的无机盐的培养基中执行果糖和差向异构酶的反应。上述无机盐可举锰盐或钴盐。从更加得到改善的阿洛酮糖的生产速度方面出发,优选地使用钴盐,从可将所生产的阿洛酮糖安全地用作食品等的方面出发,优选地使用锰盐。
仅包含果糖和无机盐的培养基可以是果糖和无机盐溶解于溶剂的液体培养基。例如,溶剂可以是水。
在通过利用微生物生产阿洛酮糖时,除阿洛酮糖之外,培养基中还会生成微生物的有机酸等的代谢物,因而培养基有可能逐渐被酸性化。由于本发明的仅包含果糖和无机盐的培养基并不包含缓冲溶液,因而更加优选地,使用最佳活性ph值低的(例如ph达到7以下)阿洛酮糖-3-差向异构酶。
上述微生物的培养物包含阿洛酮糖,回收阿洛酮糖的方法不受特殊限制,可采用本领域公知的方法,例如,可采用离心分离、过滤、结晶化、离子交换色谱法等方法。
具体地,通过对培养物进行离心分离来从微生物中分离培养液,可通过上述回收方法从培养基中分离出阿洛酮糖。
本发明的阿洛酮糖的生产方法还可在上述果糖和差向异构酶发生反应之前包括在未包含上述果糖的培养基中培养上述微生物来以使上述微生物具有休眠细胞的方式进行诱导的步骤。
以具有上述休眠细胞的方式进行的诱导可通过在未包含果糖的培养基中将上述微生物培养到静止期(stationaryphase)来实现。
在本说明书中,休眠细胞(restingcell)意味着处于不再进行增殖的状态的培养细胞。在本说明书中,静止期意味着在培养细胞的过程中经过生长期(exponentialphase)后使细胞的分裂及增殖停止来不再呈现出细胞个体数量的增加并使细胞成分的合成和分解达到均衡的状态。
因此,本发明的休眠细胞意味着处于结束生长并使细胞内的差向异构酶的表达达到充分的状态的细胞,在以使微生物具有休眠细胞的方式进行诱导的情况下,差向异构酶的表达量达到最大值,从而可使阿洛酮糖的生产达到极大化。
除了不包含果糖,未包含果糖的培养基可以是与上述的包含果糖的培养基相同的培养基。
并且,本发明还可包括在上述果糖和其差向异构酶的反应之后回收上述微生物来重新用于使其他基质转换成阿洛酮糖的步骤。
本发明中,在微生物内执行果糖和其差向异构酶的反应,即使暴露在高温下,也可使差向异构酶得到微生物的保护,因此仍然呈现出酶活性,因而可重新进行使用。
即,可在上述反应之后通过回收上述微生物来重新用于使其他基质转换成阿洛酮糖的步骤。
若在可使所分离的微生物的生育得到维持的环境中执行反应,则再使用次数不受限制,可达到数百次以上、数千次以上。
在还包括再使用微生物的步骤的情况下,微生物多次暴露在高温下,因而优选地使用热稳定性高的高温型微生物(thermophiles),以使再使用时的酶活性达到很高水平。
在上述的例示内容中,优选为棒状杆菌属、放线菌属,更优选为谷氨酸棒状杆菌,最优选为向谷氨酸棒状杆菌atcc13032导入编码上述差向异构酶的基因的。
发明的效果
通过本发明的方法,可明显改善阿洛酮糖的生产量及生产速度。
通过本发明的方法,可通过回收微生物来反复用于使果糖转换成阿洛酮糖的转换过程,从而可明显改善工序收益率。
附图说明
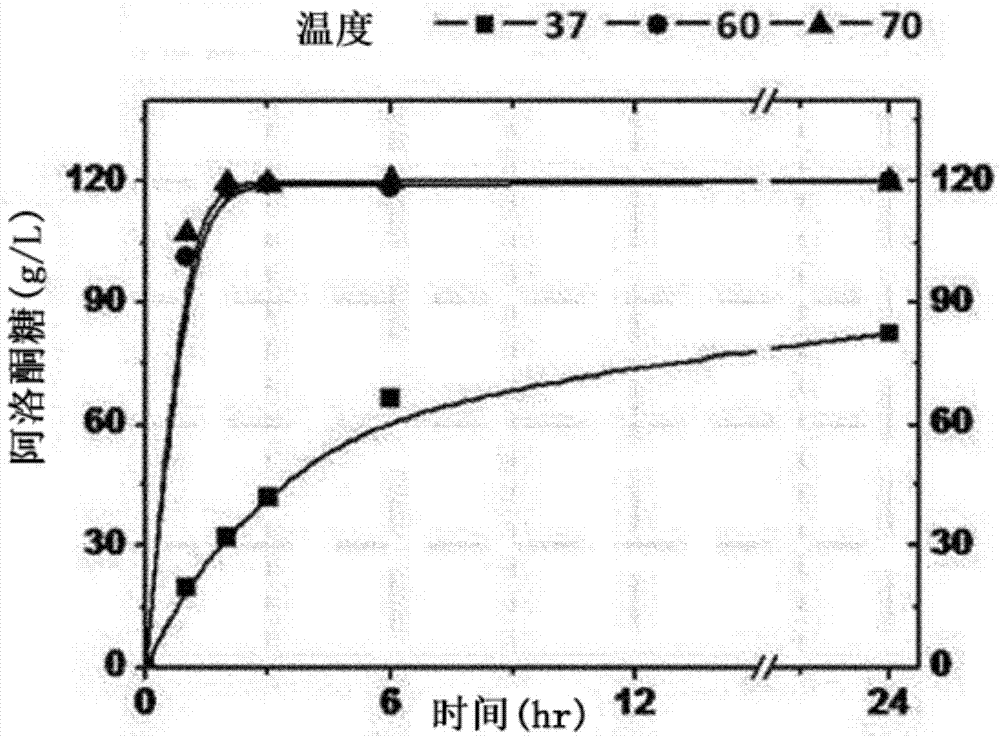
图1为对在导入阿洛酮糖-3-差向异构酶的谷氨酸棒状杆菌形质转换体中基于休眠细胞转换反应(使果糖和差向异构酶发生反应的阿洛酮糖的生产反应)的反应温度来由作为基质的果糖生产阿洛酮糖的生产量进行测定并示出的图。
图2为对在导入阿洛酮糖-3-差向异构酶的大肠杆菌mg1655形质转换体中基于休眠细胞转换反应的反应温度来由作为基质的果糖生产阿洛酮糖的生产量进行测定并示出的图。
图3为对通过利用导入阿洛酮糖-3-差向异构酶的谷氨酸棒状杆菌和大肠杆菌mg1655形质转换体来在60℃的温度下进行3个小时的阿洛酮糖生产休眠细胞转换反应后重新回收菌体并在相同反应条件下进行反应而得的阿洛酮糖的生产量进行测定并示出的图。
图4为示出基于在导入阿洛酮糖-3-差向异构酶的谷氨酸棒状杆菌形质转换体的休眠细胞转换反应所使用的阿洛酮糖生产反应培养基的组成变化的由果糖生产阿洛酮糖的生产量的图。
图5为示出在导入阿洛酮糖-3-差向异构酶的谷氨酸棒状杆菌形质转换体的休眠细胞转换反应时的基于阿洛酮糖-3-差向异构酶的来源菌株及阿洛酮糖生产反应培养基组成的阿洛酮糖的生产量的图。
图6为示出在导入阿洛酮糖-3-差向异构酶的谷氨酸棒状杆菌形质转换体的休眠细胞转换反应时的基于阿洛酮糖-3-差向异构酶的来源菌株及加热时间的阿洛酮糖的生产量的图。
图7为对源于多种菌株的阿洛酮糖-3-差向异构酶的氨基酸序列进行比较的图。
图8为示出基于导入源于梭菌属的阿洛酮糖-3-差向异构酶的谷氨酸棒状杆菌的再使用次数的阿洛酮糖的生产量的图。
具体实施方式
以下,为了更具体地说明本发明,通过实施例来对本发明进行说明。
实施例
实施例1.利用谷氨酸棒状杆菌菌株来由果糖生产阿洛酮糖的过程中基于温度的阿洛酮糖生产速度及生产量变化
(1)重组菌株的制备
通过使作为大肠杆菌-棒杆菌穿梭载体的pces208(j.microbiol.biotechnol.,18:639-647,2008)发生变形来制作插入有终止子(terminator)和lac启动子的psgt208穿梭载体来进行使用。
为了由谷氨酸棒状杆菌生产阿洛酮糖,阿洛酮糖-3-差向异构酶通过将根癌土壤杆菌(agrobacteriumtumefaciensstr.c58;taxid:176299;genbanknid:nc_003062,atcc33970)的dpe基因(agr_l_260,gi:15890243,序列1)导入到已制作的上述psgt208穿梭载体来进行使用。
详细地,利用序列31的引物1和序列32的引物2,来在根癌土壤杆菌基因使dpe基因扩大,使用限制酶kpni和bamhi来进行切割,并向psgt208穿梭载体的相同部位进行插入,从而制作了包含阿洛酮糖-3-差向异构酶的ps208-dpe重组穿梭载体。
之后,为了在谷氨酸棒状杆菌中增加阿洛酮糖-3-差向异构酶的表达量,在ps208-dpe中将lac启动子替换成源于ptrc99a的trc启动子,并将其命名为ps208ct-dpe。
将包含所制作的上述阿洛酮糖
-3-差向异构酶的重组载体ps208-dpe、ps208ct-dpe和作为其阴性对照组的psgt208载体导入到野生型谷氨酸棒状杆菌atcc13032来进行形质转换,将其利用到由果糖生产阿洛酮糖的过程当中。形质转换法采用handbookofcorynebacteriumglutamicum(lothareggeling等,isbn0-8493-1821-1,2005bycrcpress)中的方法。
(2)重组菌株的培养基利用其的阿洛酮糖的生产
为了确保高浓度菌体,将在上述中制备的谷氨酸棒状杆菌形质转换体接种到包含20μg/ml卡那霉素的5ml的lb培养基(difco)来在30℃的温度、250rpm条件下进行种培养,之后接种到包含10g/l葡萄糖及20μg/ml卡那霉素的最小培养基(每1l包含1g的k2hpo4、10g的(nh4)2so4、0.4g的mgso47h2o、20mg的feso47h2o、20mg的mnso45h2o、50mg的nacl、2g的urea、0.1mg的biotin、0.1mg的thiamine)进行了本培养。本培养在具有槽的500ml三角烧瓶以100ml体积在30℃的温度、180rpm条件下进行12个小时,诱导出充分的菌体量并使蛋白质充分表达。
通过对所得到的培养液进行离心分离来去除上层液并回收菌体,在包含作为基质的40%(w/v)果糖的与上述相同的最小培养基中以使菌体浓度达到40od600的方式进行重新悬浮之后,在25℃、30℃、37℃、50℃、60℃或70℃的温度、180rpm的条件下进行了休眠细胞转换反应。
利用高性能液体色谱(hplc)测定了果糖及阿洛酮糖的浓度。高性能液体色谱使用了设置有kromasil5nh2柱(4.6mm×250mm)的scl-10a(日本岛津公司,shimadzu),移动相通过利用75%乙腈来在以1.5ml/分钟流下并在40℃的温度下分离后利用反射指数(ri,reflectiveindex)检测器来进行了分析。在上述条件下,果糖的保留时间(retentiontime)为5.5分钟,阿洛酮糖的保留时间为4.6分钟。
图1示出了测定结果。参照图1,使导入psgt208ct-dpe穿梭载体的谷氨酸棒状杆菌atcc13032菌株在包含40%果糖的培养基进行转换反应的结果,反应温度越高,阿洛酮糖的生产速度越快、生产量越增加。尤其,在50℃、60℃、70℃的温度下发生反应的实验组大约在3个小时的时间内使阿洛酮糖-3-差向异构酶达到酶的反应平衡,生产了约120g/l的阿洛酮糖,这可被视为阿洛酮糖-3-差向异构酶的由果糖转换成阿洛酮糖的转换速度及生产量依赖于温度。
生产量以50℃的温度为起点来急剧增加,这属于比正常酶反应所需的温度明显高的温度,判断在该温度下,酶和基质之间的反应的状态发生变化。
实施例2.在利用大肠杆菌由果糖生产阿洛酮糖的过程中的基于温度的阿洛酮糖生产速度及生产量变化
根据在韩国专利授权号第10-1106253号中的实施例1中所记载的方法,制备了使用向ptrc99a载体导入源于根癌土壤杆菌的阿洛酮糖-3-差向异构酶的ptpe质体进行形质转换的e.colimg1655(δpfka,als2)菌株。
为了阻断阿洛酮糖分解路径,利用了pfka(序列11)和als2(序列14、序列15、序列16、序列17、序列18及序列19)基因缺损的大肠杆菌mg1655。
为了确保高浓度的菌体,将在以上所制备的上述大肠杆菌mg1655形质转换体接种到包含100μg/ml的氨比西林的5ml的lb培养基(difco)并在37℃的温度、250rpm条件下进行种培养,之后接种到包含10g/l葡萄糖及100μg/ml的氨比西林的2yt培养基进行了本培养。本培养在具有槽的500ml三角烧瓶以100ml体积在37℃的温度、180rpm条件下进行12个小时,诱导出充分的菌体量并使蛋白质充分表达。
通过对所得到的培养液进行离心分离来去除上层液并回收菌体,在包含作为基质的40%(w/v)果糖的大肠杆菌最小培养基m9培养基(每1l包含11.3g的m9minimalsalts(difco)、0.1ml的1mcacl2、2ml的1mmgso4、1ml的100mmmnso45h2o)中以使菌体浓度达到40od600的方式进行重新悬浮之后,分别在37℃、60℃或70℃的温度、180rpm的条件下进行了休眠细胞转换反应。果糖及阿洛酮糖的浓度根据在上述实施例1中所记载的方法进行了分析。图2示出测定结果。
参照图2,使导入ptpe载体的大肠杆菌mg1655(δpfka,als2)菌株在包含40%果糖的培养基进行转换反应的结果,也是反应温度越高,阿洛酮糖的生产速度越快,生产量越增加。尤其,与棒状杆菌的实验同样地,在60℃、70℃的温度下发生反应的实验组大约在2个小时的时间内使阿洛酮糖-3-差向异构酶达到酶的反应平衡,生产了约120g/l的阿洛酮糖。
这重新证明了阿洛酮糖-3-差向异构酶的由果糖转换成阿洛酮糖的转换速度及生产量依赖于温度。
以作为代表性的革兰氏阳性菌的棒杆菌和作为代表性的革兰氏阴性菌的大肠杆菌作为对象来在高温下进行休眠细胞转换反应实验的结果,作为糖转换酶的阿洛酮糖-3-差向异构酶在细胞内在极寒外部环境中受到保护,因而与单纯的酶的状态相比,可在高温下不产生热变性并能够以更快的速度生产阿洛酮糖。这种细胞转换反应的优点也可适用于大部分微生物。
实施例3.谷氨酸棒状杆菌形质转换菌体通过在休眠细胞转换反应中的菌体回收及再使用来实现的由果糖的阿洛酮糖的连续生产
在上述实施例1及实施例2的结果中可知,在将阿洛酮糖-3-差向异构酶导入棒杆菌和大肠杆菌形质转换体内并在50℃以上的高温条件下进行转换反应时,在3小时以内使阿洛酮糖的生产量达到最大值之后并不再增加。
即,为了确认阿洛酮糖-3-差向异构酶在达到使阿洛酮糖的生产量达到最大值的反应平衡的3个小时之后还残留多少使果糖转换成阿洛酮糖的活性,通过回收在存在果糖的条件下通过3个小时的休眠细胞转换反应来用于阿洛酮糖生产的菌体,来重新用于阿洛酮糖生产用休眠细胞转换反应中。
上述菌体的再使用休眠细胞转换反应在60℃的温度下反复进行了3次。最初的休眠细胞转换反应标为r0,从之前的反应液中回收菌体来进行第一次再使用的休眠细胞转换反应标为r1,进行第二次再使用的休眠细胞转换反应标为r2,进行第三次再使用的休眠细胞转换反应标为r3。培养条件及分析方法与实施例1相同。图3示出其结果。
参照图3,即使在60℃的高温糖转换反应中也可再使用菌体。但是,越是再使用菌体,酶的活性的一定部分减少。并且,在高温下再使用菌体方面,作为革兰氏阳性菌的谷氨酸棒状杆菌菌株的残留酶活性高于作为革兰氏阴性菌的大肠杆菌mg1655菌株的残留酶活性。
实施例4.在多种休眠细胞转换反应培养基中由果糖生产阿洛酮糖
在上述实施例1中的在谷氨酸棒状杆菌中由果糖生产阿洛酮糖的转换反应中,使用了包含40%果糖的最小培养基(每1l包含1g的k2hpo4、10g的(nh4)2so4、0.4g的mgso47h2o、20mg的feso47h2o、20mg的mnso45h2o、50mg的nacl、2g的urea、0.1mg的biotin、0.1mg的thiamine)。通过使用于该转换反应的培养基的结构成分最小化来制备更加经济、简便的培养基,执行了与在实施例1中使用的培养基之间的阿洛酮糖生产性比较。
参照图4,即使使用包含40%果糖和0.1mmmnso4的磷酸盐缓冲液(phosphatebuffer)(ph7)培养基,即使更加简便地仅使用仅包含40%果糖和0.1mmmnso4的培养基,也在最终生产的阿洛酮糖的生产量方面没有太大差别。上述状态在30℃和60℃的反应温度下均同样被观察到。在未将0.1mmmnso4作为阿洛酮糖-3-差向异构酶的辅助因子来进行添加的情况下,得到了生产量减少的结果。
在上述实施例中,可知用于阿洛酮糖生产用休眠细胞转换反应的培养基的成分仅有作为基质的果糖和作为阿洛酮糖-3-差向异构酶的辅助因子的mnso4即可。
实施例5.包含编码有源于多种菌株的阿洛酮糖-3-差向异构酶氨基酸序列的多核苷酸的重组谷氨酸棒状杆菌的制备
从德国微生物菌种保藏中心(dsmz)购买了粪厌氧棒状菌(anaerostipescaccaedsm14662;taxid:411490)的整体基因。将所购买的整体基因作为模具,以包含阿洛酮糖-3-差向异构酶推定基因(apendonuclease;sequenceid:gb|edr98778.1|;gi:167654649;序列4)的方式将序列33及序列34的引物对作为引物来执行了第一次聚合酶链式反应(pcr)。以扩大的聚合酶链式反应产物作为模具,将与阿洛酮糖-3-差向异构酶基因特殊结合的序列35及序列36的引物对用作引物,来执行了第二次聚合酶链式反应。
通过使用限制酶bamhⅰ和xbaⅰ来将所得到的聚合酶链式反应的产物插入到ps208ct-dpe(在韩国专利申请号10-2013-0060703的实施例1中所记载的载体)的相同酶部位,来制备了重组载体ps208ct-acdpe。
将所制备的ps208ct-acdpe载体导入野生型谷氨酸棒状杆菌atcc13032来进行形质转换,并将其用于由果糖生产阿洛酮糖的生产过程。形质转换法采用在handbookofcorynebacteriumglutamicum(lothareggeling等,isbn0-8493-1821-1,2005bycrcpress)中记载的方法。
所得到的重组谷氨酸棒状杆菌菌株在-80℃的温度下得到保管后用于培养。
包含鲍氏梭菌(clostridiumbolteaeatccbaa-613;taxid:411902)的阿洛酮糖-3-差向异构酶推定基因(hypotheticalproteinclobol_00069;sequenceid:gb|edp19602.1|;gi:15844190;序列9)的质体从韩国益力多乳品有限公司购得。将与阿洛酮糖-3-差向异构酶基因特殊结合的序列37及序列38的引物对用作引物来执行了聚合酶链式反应。
通过使用限制酶kpnⅰ和xbaⅰ来将所得到的聚合酶链式反应的产物插入到ps208ct-dpe(在韩国专利申请号10-2013-0060703的实施例1中所记载的载体)的相同酶部位,来制备了重组载体ps208ct-cbdpe。
通过与上述的内容相同的方法将所制备的重组载体ps208ct-cbdpe载体导入野生型谷氨酸棒状杆菌atcc13032来进行形质转换,并将其用于由果糖生产阿洛酮糖的生产过程。所得到的重组谷氨酸棒状杆菌菌株在-80℃的温度下得到保管后用于培养。
从德国微生物菌种保藏中心(dsmz)购买了海氏梭菌(clostridiumhylemonaedsm15053;taxid:553973)的整体基因。将所购买的整体基因作为模具,以包含阿洛酮糖-3-差向异构酶推定基因(dolicholmonophosphatemannosesynthase;sequenceid:ref|wp_006442985.1|;gi:225161759;序列10)的方式将序列39及序列40的引物对作为引物来执行了第一次聚合酶链式反应。以扩大的聚合酶链式反应产物作为模具,将与阿洛酮糖-3-差向异构酶基因特殊结合的序列41及序列42的引物对用作引物,来执行了第二次聚合酶链式反应。
通过使用限制酶bamhⅰ和xbaⅰ来将所得到的聚合酶链式反应的产物插入到ps208ct-dpe(在韩国专利申请号10-2013-0060703的实施例1中所记载的载体)的相同酶部位,来制备了重组载体ps208ct-chdpe。
通过与上述的内容相同的方法将所制备的ps208ct-chdpe载体导入野生型谷氨酸棒状杆菌atcc13032来进行形质转换,并将其用于由果糖生产阿洛酮糖的生产过程。所得到的重组谷氨酸棒状杆菌菌株在-80℃的温度下得到保管后用于培养。
实施例6.利用导入源于多种菌株的阿洛酮糖-3-差向异构酶的重组谷氨酸棒状杆菌菌株由果糖生产阿洛酮糖
利用在上述实施例5中所制备的谷氨酸棒状杆菌形质转换体来确认了由高浓度果糖生产阿洛酮糖。
将在上述中制备的形质转换体接种到包含20μg/ml卡那霉素的2yt培养基来在30℃的温度、250rpm条件下进行种培养,之后重新将形质转换体接种到包含20μg/ml卡那霉素的2yt培养基来进行本培养。本培养在具有槽的300ml三角烧瓶以60ml体积在30℃的温度、180rpm条件下进行7个小时,诱导出充分的菌体量并使蛋白质充分表达。
通过对所得到的培养液进行离心分离来去除上层液并回收菌体,在包含20μg/ml卡那霉素、作为基质的40%(w/v)果糖和浓度为0.1mm的作为阿洛酮糖-3-差向异构酶的主辅助因子的锰或钴的简单的转换反应培养基中,在55℃的温度下进行了休眠细胞转换反应。果糖和阿洛酮糖的浓度采用在上述实施例1中所记述的方法相同的方法来进行测定。图5示出其结果。atdpe是指当前使用的根癌农杆菌的阿洛酮糖-3-差向异构酶。
参照图5,在所有导入多种阿洛酮糖-3-差向异构酶的重组菌株中,与将锰作为辅助因子相比,当将钴作为辅助因子时,阿洛酮糖的生产速度更快。
导入源于厌氧棒状菌属和土壤杆菌属的阿洛酮糖-3-差向异构酶的重组谷氨酸棒状杆菌的阿洛酮糖生产量在6个小时时达到平衡,但导入源于梭菌的阿洛酮糖-3-差向异构酶的重组谷氨酸棒状杆菌的阿洛酮糖生产量在将锰作为辅助因子的情况下,在3个小时时达到平衡。
因此,不仅可确认到源于梭菌的阿洛酮糖-3-差向异构酶可实现由果糖生产阿洛酮糖,阿洛酮糖的生产速度比源于土壤杆菌属的阿洛酮糖-3-差向异构酶更快。
实施例7.导入源于多种菌株的阿洛酮糖-3-差向异构酶的重组谷氨酸棒状杆菌菌株的高温下的阿洛酮糖持续生产活性维持
在上述实施例1、实施例2的结果中,确认到在以50℃以上的高温条件进行转换反应时,所有阿洛酮糖生产在3个小时以内完成。像这样,在高温条件下,由果糖生产阿洛酮糖的生产速度快,因而需要即使再使用菌体的情况下也能够在长时间的高温条件下维持稳定的阿洛酮糖-3-差向异构酶。因此,确认了源于多种菌株的阿洛酮糖-3-差向异构酶在高温条件下将活性维持到什么程度。
使通过上述实施例1中的技术方法得到的菌体在2yt中进行浮游,来利用振荡培养器在0小时、3小时、6小时、9小时、12小时、24小时的时间内持续施加60℃的热量。在按各种时间施加热量之后,回收菌体并在仅包含20μg/ml卡那霉素、0.1mm锰及40%(w/v)果糖的简单转换反应培养基中进行浮游,重新在60℃的温度下进行3个小时的休眠细胞转换反应。果糖和阿洛酮糖的浓度通过与上述实施例1中的技术方法相同的方法进行了测定。图6示出其结果。
参照图6,导入以往使用的源于土壤杆菌属的阿洛酮糖-3-差向异构酶和源于厌氧棒状菌属的阿洛酮糖-3-差向异构酶的重组谷氨酸棒状杆菌在受到3个小时的60℃的热量之后,几乎无法进行阿洛酮糖生产。相反,导入源于梭菌属的阿洛酮糖-3-差向异构酶的重组谷氨酸棒状杆菌在受到24个小时的热量后也能够维持阿洛酮糖生产,因而与源于土壤杆菌属的阿洛酮糖-3-差向异构酶相比,更加有利于高温条件下的阿洛酮糖的生产。
当对于源于根癌土壤杆菌的阿洛酮糖-3-差向异构酶的热稳定性起到重要作用的序列(参考文献1)中的第33个氨基酸或第213个氨基酸分别被亮氨酸、半胱氨酸取代时,在50℃的温度下的酶的半寿命分别达到3.3倍、7.2倍,在两个均被取代时,达到29.9倍。
源于根癌土壤杆菌的阿洛酮糖-3-差向异构酶的氨基酸序列与源于梭菌属的阿洛酮糖-3-差向异构酶的氨基酸序列之间的比较在图7中示出,参照图7,热稳定性高的源于梭菌属的阿洛酮糖-3-差向异构酶使得一个氨基酸具有与之相对应的序列或具有所有两个序列。
实施例8.导入源于梭菌属的阿洛酮糖-3-差向异构酶的重组谷氨酸棒状杆菌菌体通过在休眠细胞转换反应中的菌体回收及再使用来实现由果糖生产阿洛酮糖
由于在上述实施例7中确认到源于梭菌属的阿洛酮糖-3-差向异构酶在高温中具有高稳定性,因而在导入源于梭菌属的阿洛酮糖-3-差向异构酶的两个重组菌株中使代表性地导入海氏梭菌的阿洛酮糖-3-差向异构酶的重组谷氨酸棒状杆菌菌体在高温下进行休眠细胞转换反应之后,确认了菌体再使用效果。
在使通过与在上述实施例3中所记述的方法相同的方法得到的菌体在60℃的温度下进行3个小时的休眠细胞转换反应之后,重新回收菌体来实施相同的休眠细胞转换反应,菌体共被再使用3次(与上述实施例3中的条件相同的条件进行实验)。最初的休眠细胞转换反应标为r0,从之前的反应液中回收菌体来进行第一次再使用的休眠细胞转换反应标为r1,进行第二次再使用的休眠细胞转换反应标为r2,进行第三次再使用的休眠细胞转换反应标为r3。图8示出其结果。
参照图8,在高温条件下多次再使用菌体的过程中,导入梭菌属的阿洛酮糖-3-差向异构酶来制备的菌体以阿洛酮糖生产量没有任何减少情况的方式维持规定量。可结合上述实施例7的结果来确认到,酶本身的热稳定性在高温条件下持续进行糖转换反应,在菌体的再使用过程中也非常有利。
<110>庆尚大学校产学协力团
<120>阿洛酮糖的生产方法
<130>khp172110383.6
<160>42
<170>kopatentin2.0
<210>1
<211>870
<212>dna
<213>根癌土壤杆菌
<400>1
atgaaacacggcatctattattcttactgggaacatgagtggagcgccaagttcggtccc60
tatatcgagaaggtcgccaagctcggtttcgacatcatcgaagtcgccgcccaccatatc120
aacgaatacagcgacgccgaactcgcgaccatcaggaagagcgcgaaggataacggcatc180
atcctcaccgccggcatcggtccgtcgaaaaccaagaacctgtcgtcggaagatgctgcg240
gtgcgtgcggccggcaaggcgttctttgaaagaaccctttcgaacgtcgccaagctcgat300
atccacaccatcggcggcgcattgcattcctattggccaatcgattattcgcagcccgtc360
gacaaggcaggcgattatgcgcgcggcgtcgagggtatcaacggcattgccgatttcgcc420
aatgatctcggcatcaacctgtgcatcgaagtcctcaaccgctttgaaaaccacgtcctc480
aacacggcggcggaaggcgtcgcttttgtgaaggatgtcggcaagaacaatgtgaaagtc540
atgctggataccttccacatgaacatcgaggaagacagtttcggtgacgccatccgcacg600
gccggcccgcttctggggcacttccataccggtgaaagcaatcgccgcgtaccgggcaag660
ggcagaatgccgtggcacgaaatcggccttgcgctgcgtgatatcaactacaccggcgcg720
gtaatcatggagcctttcgtcaagacaggcggcaccatcggctcggatatcaaggtgtgg780
cgcgacctgagcggtggcgccgacatcgcgaaaatggatgaagatgcccgcaatgcgctg840
gcattctcccgcttcgttcttggtggctaa870
<210>2
<211>858
<212>dna
<213>粪厌氧棒状菌
<400>2
atgaaaaataaattcggagttgacagttttatttggactgaatccttttctaaaaaagat60
ttatggatcatccccaaggcaaaagaactgggatttgaagtcatcgactttgcgatctcc120
aacccattcacattccctgtagagaaagtgaaggcagagctagagagagtgggaatcgac180
tgtgtctgcactaccacgctgacacctgaaaccaatccgatttctccggatgccgagatc240
cgtgcggcaggcgtaaaagccatgaaaaaatgtgtggatatctgcaacgaactgggtgca300
ccgatcttaggcggtgtaaattatgcaggctggggatatctgacgaagaagccaaggacc360
gaggaagagtggaactggggcgtagagtgcatgagggaagttgccgagtacgcaaagcaa420
accggagatgttaccatctgtgtggaatgtgtcaacagatttgaaacccacttcttaaac480
attgcggaagatgcagtggccttctgtaaggatgttggaacaggaaatgtcaaggttcat540
ctcgactgcttccatatgatcagagaagaaaagagctttgcaggggcagtaaagacctgc600
ggcaaagaatatctcggatacattcatgtcaatgaaaacgacagaggtattcctggaaca660
gggcttgtaccgtttaaagaatttttcaatgcattagtagagatcgggtatgacggacct720
ttggtgatcgaatcttttgatccgagctttgaagaactgtccggcaactgtgcgatctgg780
agaaaacttgccgatactggagaagaacttgcgattgaagggctgaaaaatctgaaagcc840
atcgctgctgagatataa858
<210>3
<211>289
<212>prt
<213>根癌土壤杆菌
<400>3
metlyshisglyiletyrtyrsertyrtrpgluhisglutrpserala
151015
lyspheglyprotyrileglulysvalalalysleuglypheaspile
202530
ilegluvalalaalahishisileasnglutyrseraspalagluleu
354045
alathrilearglysseralalysaspasnglyileileleuthrala
505560
glyileglyproserlysthrlysasnleusersergluaspalaala
65707580
valargalaalaglylysalaphephegluargthrleuserasnval
859095
alalysleuaspilehisthrileglyglyalaleuhissertyrtrp
100105110
proileasptyrserglnprovalasplysalaglyasptyralaarg
115120125
glyvalgluglyileasnglyilealaaspphealaasnaspleugly
130135140
ileasnleucysilegluvalleuasnargphegluasnhisvalleu
145150155160
asnthralaalagluglyvalalaphevallysaspvalglylysasn
165170175
asnvallysvalmetleuaspthrphehismetasnileglugluasp
180185190
serpheglyaspalaileargthralaglyproleuleuglyhisphe
195200205
histhrglygluserasnargargvalproglylysglyargmetpro
210215220
trphisgluileglyleualaleuargaspileasntyrthrglyala
225230235240
valilemetgluprophevallysthrglyglythrileglyserasp
245250255
ilelysvaltrpargaspleuserglyglyalaaspilealalysmet
260265270
aspgluaspalaargasnalaleualapheserargphevalleugly
275280285
gly
<210>4
<211>285
<212>prt
<213>粪厌氧棒状菌
<400>4
metlysasnlyspheglyvalaspserpheiletrpthrgluserphe
151015
serlyslysaspleutrpileileprolysalalysgluleuglyphe
202530
gluvalileaspphealaileserasnprophethrpheprovalglu
354045
lysvallysalagluleugluargvalglyileaspcysvalcysthr
505560
thrthrleuthrprogluthrasnproileserproaspalagluile
65707580
argalaalaglyvallysalametlyslyscysvalaspilecysasn
859095
gluleuglyalaproileleuglyglyvalasntyralaglytrpgly
100105110
tyrleuthrlyslysproargthrglugluglutrpasntrpglyval
115120125
glucysmetarggluvalalaglutyralalysglnthrglyaspval
130135140
thrilecysvalglucysvalasnargphegluthrhispheleuasn
145150155160
ilealagluaspalavalalaphecyslysaspvalglythrglyasn
165170175
vallysvalhisleuaspcysphehismetilearggluglulysser
180185190
phealaglyalavallysthrcysglylysglutyrleuglytyrile
195200205
hisvalasngluasnaspargglyileproglythrglyleuvalpro
210215220
phelysgluphepheasnalaleuvalgluileglytyraspglypro
225230235240
leuvalilegluserpheaspproserpheglugluleuserglyasn
245250255
cysalailetrparglysleualaaspthrglyglugluleualaile
260265270
gluglyleulysasnleulysalailealaalagluile
275280285
<210>5
<211>289
<212>prt
<213>根癌土壤杆菌
<400>5
metlyshisglyiletyrtyrsertyrtrpgluhisglutrpserala
151015
lyspheglyprotyrileglulysvalalalysleuglypheaspile
202530
leugluvalalaalahishisileasnglutyrseraspalagluleu
354045
alathrilearglysseralalysaspasnglyileileleuthrala
505560
glyileglyproserlysthrlysasnleusersergluaspalaala
65707580
valargalaalaglylysalaphephegluargthrleuserasnval
859095
alalysleuaspilehisthrileglyglyalaleuhissertyrtrp
100105110
proileasptyrserglnprovalasplysalaglyasptyralaarg
115120125
glyvalgluglyileasnglyilealaaspphealaasnaspleugly
130135140
ileasnleucysilegluvalleuasnargphegluasnhisvalleu
145150155160
asnthralaalagluglyvalalaphevallysaspvalglylysasn
165170175
asnvallysvalmetleuaspthrphehismetasnileglugluasp
180185190
serpheglyaspalaileargthralaglyproleuleuglyhisphe
195200205
histhrglyglucysasnargargvalproglylysglyargmetpro
210215220
trphisgluileglyleualaleuargaspileasntyrthrglyala
225230235240
valilemetgluprophevallysthrglyglythrileglyserasp
245250255
ilelysvaltrpargaspleuserglyglyalaaspilealalysmet
260265270
aspgluaspalaargasnalaleualapheserargphevalleugly
275280285
gly
<210>6
<211>269
<212>prt
<213>未知
<220>
<223>阿洛酮糖-3-差向异构酶
<400>6
metlyshisglyiletyrtyralatyrtrpthrglutrpseralalys
151015
tyrlyslystyrileglulysvalalalysleuglypheaspileile
202530
gluilealaalaalaleuglutyrseraspaspleugluleulyslys
354045
alalysaspasnglyileileleuthralaglytyrglyprothrlys
505560
asnleusergluaspalagluvalargalaalaalaleuphephelys
65707580
argleuleuaspileleualagluleuaspilehisileileglygly
859095
alaleutyrsertyrtrpprovalasppheserasnasplysglyasp
100105110
trpalatrpglyvalgluglymetarggluleualaaspphealaasp
115120125
aspileasnleuglymetgluvalleuasnargphegluserhisile
130135140
leuasnthralagluglualavalalaphevallysaspvalglyser
145150155160
asnvallysvalmetleuaspthrphehismetasnileglugluser
165170175
phealaglyalaileargthralaglyaspleuleuglyhisphehis
180185190
thrglygluasnasnargleuvalproglylysglyargileprotrp
195200205
lysgluileglyasnalaleuargaspileasntyraspglyalaala
210215220
valmetgluprophevallysserglyglythrileglyseraspile
225230235240
lysvaltrpargaspleuserglyalaaspglualaalaleuaspasp
245250255
aspalaargalaleugluphealaarghisvalleugly
260265
<210>7
<211>876
<212>dna
<213>鲍氏梭菌
<400>7
atgaaatatggtatttattttgcttattggacgaaggaatggtttgctgattataagaag60
tatatggataaggtgtctgctttggggtttgatgtcctggaaatttcctgtgcagccctc120
agagatgtatacacaaccaaggaacagctgattgagctacgtgaatacgccaaagaaaaa180
gggcttgttctaacagctggttatggacctactaaagcagaaaatctgtgttcagaagac240
ccggaggcagtgagacgggccatgacattcttcaaggacctgcttccaaagctgcagtta300
atggatatccatatcctgggagggggattatattcctactggcccgtggattttaccatt360
aataatgacaagcagggagaccgggccagggctgtcaggaatctgagggaattgtccaaa420
acagcggaggaatgtgacgtggtgcttggaatggaggtactgaaccgctatgaggggtat480
attcttaatacctgtgaagaggcaattgattttgtcgatgagattggaagcagccatgta540
aaaatcatgctggatactttccatatgaatattgaagagacaaatatggctgatgcaatc600
cgcaaggcgggagacaggctgggacatctccatctgggagaacagaaccgcctggtgccg660
ggaaaaggcagcctgccatgggctgagatagggcaggcgctccgtgatattaactatcag720
ggagccgctgtcatggaaccttttgtcatgcagggagggaccatcggttctgagataaag780
gtatggagagacatggtgccggatctttctgaggaagcactggacagggatgcaaagggt840
gcgctggaattctgcaggcatgtgtttggtatctaa876
<210>8
<211>870
<212>dna
<213>海氏梭菌
<400>8
atgaaacatggtatctattatgcatactgggaacaagaatgggcggccgactacaagcgc60
tatgttgaaaaggtggcaaagcttgggtttgacattctggagatcggcgctgggccgctg120
ccggaatacgcagagcaggatgtgaaggaactgaagaaatgtgcgcaggacaatgggatc180
acgctgacggccggatatggtccgacgttcaaccacaatatcggttcttcagacgccggg240
gtaagggaagaggcgctggaatggtataagaggttatttgaagtgctggcagagcttgat300
atccacctgatcggaggggcgctctattcttactggcctgtcgattttgcaaacgccgat360
aaaacggaagactggaagtggagtgtagagggcatgcagaggctggcgccggccgcggcc420
aaatatgacatcaacctgggcatggaagttctgaaccggtttgagagccatatcctgaat480
acagccgaggaaggtgtgaagtttgtagaggaagtcggcatggacaacgtaaaggtcatg540
ctggatacattccatatgaatatagaagagcaaagcataggcggcgcgatccgccgggca600
ggaaaactgctcgggcatttccacaccggagaatgcaaccgcatggtgcccgggaaggga660
cgtattccatggcgtgagataggggatgctctccgtgatatcggatatgacggaactgct720
gtaatggagccgttcgttcgcatgggaggacaggtcggcgctgatatcaaggtgtggaga780
gacataagccgtggagcagacgaggcacagcttgacgatgacgcgcgccgtgcgctggag840
ttccagagatatatgctggagtggaagtaa870
<210>9
<211>291
<212>prt
<213>鲍氏梭菌
<400>9
metlystyrglyiletyrphealatyrtrpthrlysglutrppheala
151015
asptyrlyslystyrmetasplysvalseralaleuglypheaspval
202530
leugluilesercysalaalaleuargaspvaltyrthrthrlysglu
354045
glnleuilegluleuargglutyralalysglulysglyleuvalleu
505560
thralaglytyrglyprothrlysalagluasnleucyssergluasp
65707580
proglualavalargargalametthrphephelysaspleuleupro
859095
lysleuglnleumetaspilehisileleuglyglyglyleutyrser
100105110
tyrtrpprovalaspphethrileasnasnasplysglnglyasparg
115120125
alaargalavalargasnleuarggluleuserlysthralagluglu
130135140
cysaspvalvalleuglymetgluvalleuasnargtyrgluglytyr
145150155160
ileleuasnthrcysgluglualaileaspphevalaspgluilegly
165170175
serserhisvallysilemetleuaspthrphehismetasnileglu
180185190
gluthrasnmetalaaspalailearglysalaglyaspargleugly
195200205
hisleuhisleuglygluglnasnargleuvalproglylysglyser
210215220
leuprotrpalagluileglyglnalaleuargaspileasntyrgln
225230235240
glyalaalavalmetgluprophevalmetglnglyglythrilegly
245250255
sergluilelysvaltrpargaspmetvalproaspleusergluglu
260265270
alaleuaspargaspalalysglyalaleugluphecysarghisval
275280285
pheglyile
290
<210>10
<211>289
<212>prt
<213>海氏梭菌
<400>10
metlyshisglyiletyrtyralatyrtrpgluglnglutrpalaala
151015
asptyrlysargtyrvalglulysvalalalysleuglypheaspile
202530
leugluileglyalaglyproleuproglutyralagluglnaspval
354045
lysgluleulyslyscysalaglnaspasnglyilethrleuthrala
505560
glytyrglyprothrpheasnhisasnileglyserseraspalagly
65707580
valarggluglualaleuglutrptyrlysargleuphegluvalleu
859095
alagluleuaspilehisleuileglyglyalaleutyrsertyrtrp
100105110
provalaspphealaasnalaasplysthrgluasptrplystrpser
115120125
valgluglymetglnargleualaproalaalaalalystyraspile
130135140
asnleuglymetgluvalleuasnargphegluserhisileleuasn
145150155160
thralaglugluglyvallysphevalglugluvalglymetaspasn
165170175
vallysvalmetleuaspthrphehismetasnileglugluglnser
180185190
ileglyglyalaileargargalaglylysleuleuglyhisphehis
195200205
thrglyglucysasnargmetvalproglylysglyargileprotrp
210215220
arggluileglyaspalaleuargaspileglytyraspglythrala
225230235240
valmetgluprophevalargmetglyglyglnvalglyalaaspile
245250255
lysvaltrpargaspileserargglyalaaspglualaglnleuasp
260265270
aspaspalaargargalaleuglupheglnargtyrmetleuglutrp
275280285
lys
<210>11
<211>963
<212>dna
<213>大肠杆菌
<400>11
atgattaagaaaatcggtgtgttgacaagcggcggtgatgcgccaggcatgaacgccgca60
attcgcggggttgttcgttctgcgctgacagaaggtctggaagtaatgggtatttatgac120
ggctatctgggtctgtatgaagaccgtatggtacagctagaccgttacagcgtgtctgac180
atgatcaaccgtggcggtacgttcctcggttctgcgcgtttcccggaattccgcgacgag240
aacatccgcgccgtggctatcgaaaacctgaaaaaacgtggtatcgacgcgctggtggtt300
atcggcggtgacggttcctacatgggtgcaatgcgtctgaccgaaatgggcttcccgtgc360
atcggtctgccgggcactatcgacaacgacatcaaaggcactgactacactatcggtttc420
ttcactgcgctgagcaccgttgtagaagcgatcgaccgtctgcgtgacacctcttcttct480
caccagcgtatttccgtggtggaagtgatgggccgttattgtggagatctgacgttggct540
gcggccattgccggtggctgtgaattcgttgtggttccggaagttgaattcagccgtgaa600
gacctggtaaacgaaatcaaagcgggtatcgcgaaaggtaaaaaacacgcgatcgtggcg660
attaccgaacatatgtgtgatgttgacgaactggcgcatttcatcgagaaagaaaccggt720
cgtgaaacccgcgcaactgtgctgggccacatccagcgcggtggttctccggtgccttac780
gaccgtattctggcttcccgtatgggcgcttacgctatcgatctgctgctggcaggttac840
ggcggtcgttgtgtaggtatccagaacgaacagctggttcaccacgacatcatcgacgct900
atcgaaaacatgaagcgtccgttcaaaggtgactggctggactgcgcgaaaaaactgtat960
taa963
<210>12
<211>320
<212>prt
<213>大肠杆菌
<400>12
metilelyslysileglyvalleuthrserglyglyaspalaprogly
151015
metasnalaalaileargglyvalvalargseralaleuthrglugly
202530
leugluvalmetglyiletyraspglytyrleuglyleutyrgluasp
354045
argmetvalglnleuaspargtyrservalseraspmetileasnarg
505560
glyglythrpheleuglyseralaargpheproglupheargaspglu
65707580
asnileargalavalalailegluasnleulyslysargglyileasp
859095
alaleuvalvalileglyglyaspglysertyrmetglyalametarg
100105110
leuthrglumetglypheprocysileglyleuproglythrileasp
115120125
asnaspilelysglythrasptyrthrileglyphephethralaleu
130135140
serthrvalvalglualaileaspargleuargaspthrserserser
145150155160
hisglnargileservalvalgluvalmetglyargtyrcysglyasp
165170175
leuthrleualaalaalailealaglyglycysgluphevalvalval
180185190
progluvalglupheserarggluaspleuvalasngluilelysala
195200205
glyilealalysglylyslyshisalailevalalailethrgluhis
210215220
metcysaspvalaspgluleualahispheileglulysgluthrgly
225230235240
arggluthrargalathrvalleuglyhisileglnargglyglyser
245250255
provalprotyraspargileleualaserargmetglyalatyrala
260265270
ileaspleuleuleualaglytyrglyglyargcysvalglyilegln
275280285
asngluglnleuvalhishisaspileileaspalailegluasnmet
290295300
lysargprophelysglyasptrpleuaspcysalalyslysleutyr
305310315320
<210>13
<211>450
<212>dna
<213>大肠杆菌
<400>13
atgaaaaagattgcatttggctgtgatcatgtcggtttcattttaaaacatgaaatagtg60
gcacatttagttgagcgtggcgttgaagtgattgataaaggaacctggtcgtcagagcgt120
actgattatccacattacgccagtcaagtcgcactggctgttgctggcggagaggttgat180
ggcgggattttgatttgtggtactggcgtcggtatttcgatagcggcgaacaagtttgcc240
ggaattcgcgcggtcgtctgtagcgaaccttattccgcgcaactttcgcggcagcataac300
gacaccaacgtgctggcttttggttcacgagtggttggcctcgaactggcaaaaatgatt360
gtggatgcgtggctgggcgcacagtacgaaggcggtcgtcatcaacaacgcgtggaggcg420
attacggcaatagagcagcggagaaattga450
<210>14
<211>891
<212>dna
<213>大肠杆菌
<400>14
atgagccagtcagagtttgattcagcgcttccgaacggtatagggttagcgccttacctg60
cgaatgaagcaggaaggaatgacagaaaatgaaagccgcatcgtggagtggttactcaaa120
cccggtaacctgagttgtgcacccgcaattaaagatgtcgcagaagctctggcggtatct180
gaagcgatgatagttaaggtatcaaagctgctggggtttagcggctttcgtaacttacgc240
agtgcgctggaagattatttttctcagtcagaacaggtattgccttccgagttggctttt300
gatgaagcgccgcaggatgtggtgaataaggtatttaacatcactttacgcaccattatg360
gaaggtcagtcgatcgtcaacgttgatgagatccaccgtgccgcccgctttttctatcag420
gccagacagcgggatttgtacggtgccggaggatcaaatgctatctgtgctgatgtacag480
cacaagttcttgcgcattggcgtacgctgtcaggcctatcctgatgctcacatcatgatg540
atgtccgcttcgttgttacaggaaggagatgttgtgctggtagtgacccattccgggcga600
accagtgatgtaaaagcggccgtagaactggcaaaaaagaacggggcaaagattatttgt660
ataacccatagctaccattcaccgatagcgaaactggccgattatattatttgctcacca720
gccccggaaacgccgttattaggtcgtaatgcctcggcaagaatattacaactaactttg780
ctggacgctttttttgtctctgtcgcccagctcaacattgaacaagctaatattaatatg840
caaaaaaccggcgcaattgttgatttcttctcaccaggcgcgctgaaataa891
<210>15
<211>936
<212>dna
<213>大肠杆菌
<400>15
atgaataaatatctgaaatatttcagcggcacactcgtgggcttaatgttgtcaaccagc60
gcttttgctgccgccgaatatgctgtcgtattgaaaaccctctccaacccattttgggta120
gatatgaaaaaaggcattgaagatgaagcaaaaacactgggcgtcagcgttgatattttt180
gcctctccttcagaaggcgattttcaatctcaattgcagttatttgaagatctcagtaat240
aaaaattacaaaggtatcgccttcgctccattatcctcagtgaatctggtcatgcctgtc300
gcccgcgcatggaaaaaaggcatttatctggttaatctcgatgaaaaaatcgacatggat360
aatctgaaaaaagctggcggcaatgtggaagcttttgtcaccaccgataacgttgctgtc420
ggggcgaaaggcgcgtcgttcattattgacaaattgggcgctgaaggtggtgaagtcgca480
atcattgagggtaaagccggtaacgcctccggtgaagcgcgtcgtaatggtgccaccgaa540
gccttcaaaaaagcaagccagatcaagcttgtcgccagccagcctgccgactgggaccgc600
attaaagcactggatgtcgccactaacgtgttgcaacgtaatccgaatattaaagcgatc660
tattgcgcgaatgacacgatggcaatgggtgttgctcaggcagtcgcaaacgccggaaaa720
acgggaaaagtgctggtcgtcggtacagatggcattccggaagcccgcaaaatggtggaa780
gccggacaaatgaccgcgacggttgcccagaacccggcggatatcggcgcaacgggtctg840
aagctgatggttgacgctgagaaatccggcaaggttatcccgctggataaagcaccggaa900
tttaaactggtcgattcaatcctggtcactcaataa936
<210>16
<211>1533
<212>dna
<213>大肠杆菌
<400>16
atggccacgccatatatatcgatggcggggatcggcaagtcctttggtccggttcacgca60
ttaaagtcggttaatttaacggtttatcctggtgaaatacatgcattactaggagaaaat120
ggcgcgggtaaatccacgctaatgaaagttttatccggaatacatgagccgaccaaaggc180
accattaccattaataacattagctataacaagctggatcataaattagcggcacaactc240
ggtatcgggattatttatcaggaactcagcgttattgatgaattaaccgtactggaaaat300
ttatatattggtcgtcatctgacgaaaaaaatctgtggcgtcaatattatcgactggcga360
gaaatgcgtgtccgcgccgccatgatgttattacgcgtgggcttgaaagttgatctagat420
gagaaagtggcgaatttatctatcagccacaagcagatgctagaaattgccaaaacgctg480
atgctcgatgccaaagtcatcatcatggatgaacccacctcctcactcaccaataaagag540
gtggactatctgtttctgatcatgaatcagttgcgtaaagagggtacggccatcgtctat600
atctcgcataagttggcggaaattcgccgtatttgcgaccgctatacggtgatgaaagac660
ggcagcagcgtttgcagcggcatagtaagcgatgtgtcaaatgacgatatcgtccgtctg720
atggtaggccgcgaactgcaaaaccgttttaacgcgatgaaggagaatgtcagcaacctt780
gcgcacgaaacggtttttgaggtgcggaacgtcaccagtcgtgacagaaaaaaggtccgg840
gatatctcatttagcgtctgccggggagaaatattaggctttgccggactggtcggttcc900
ggacgtactgaactgatgaattgtctgtttggcgtggataaacgcgctggcggagaaatc960
cgtcttaatggcaaagatatctctccacgttcacccctggatgccgtgaaaaaagggatg1020
gcttacatcactgaaagccgccgggataacggttttttccccaacttttccatcgctcag1080
aacatggcgatcagccgcagtctgaaagacggcggctataaaggcgcgatgggcttgttt1140
catgaagttgacgagcaacgtaccgctgaaaatcaacgcgaactgctggcgctgaaatgt1200
cattcggtaaaccagaatatcaccgaactctccgggggaaatcagcagaaagtcctgatc1260
tccaaatggctgtgctgttgcccggaagtgattattttcgatgaacctacccgcggcatc1320
gacgttggcgcgaaagccgaaatttacaaagtgatgcgccaactggcggacgacggaaaa1380
gtcatcctgatggtgtcatctgaactacctgaaattatcaccgtctgcgaccgcatcgcc1440
gtgttctgcgaaggacgactgacgcaaatcctgacgaatcgcgatgacatgagcgaagag1500
gagattatggcatgggctttaccacaagagtaa1533
<210>17
<211>981
<212>dna
<213>大肠杆菌
<400>17
atgggctttaccacaagagtaaaaagcgaagcgagcgagaagaaaccgttcaactttgcg60
ctgttctgggataaatacggcaccttttttatcctggcgatcatcgtcgccatctttggt120
tcgctgtcaccagaatattttctgaccaccaataatattacccagatttttgttcaaagc180
tccgtgacggtattgatcggcatgggcgagtttttcgctatcctggtcgctggtatcgac240
ctctcggttggcgcgattctggcgctttccggtatggtgaccgccaaactgatgttggca300
ggtgttgacccgtttctcgcagcgatgattggcggtgtactggttggcggcgcactgggg360
gcgatcaacggctgcctggtcaactggacggggctacacccgttcatcatcacccttggc420
accaacgcgattttccgtgggatcacgctggtgatctccgatgccaactcggtatacggc480
ttctcatttgacttcgtgaacttctttgccgccagcgtaattgggatacctgtccccgtt540
atcttctcactaattgtcgcgctcatcctttggtttctgacaacgcgtatgcggctcggg600
cgcaacatctacgcactgggcggcaacaaaaattcggcgttctattccgggattgacgtg660
aaattccacatcctggtggtgtttatcatctccggtgtttgtgcaggtctggcaggcgtc720
gtctcaactgcacgactcggtgccgcagaaccgcttgccggtatgggttttgaaacctat780
gccattgccagcgccatcattggcggcaccagtttcttcggcggcaaggggcgcattttc840
tctgtggtgattggcgggttgatcatcggcaccatcaacaacggtctgaatattttgcag900
gtacaaacctattaccaactggtggtgatgggcggattaattatcgcggctgtcgccctt960
gaccgtcttatcagtaagtaa981
<210>18
<211>696
<212>dna
<213>大肠杆菌
<400>18
atgaaaatctccccctcgttaatgtgtatggatctgctgaaatttaaagaacagatcgaa60
tttatcgacagccatgccgattacttccacatcgatatcatggacggtcactttgtcccc120
aatctgacactctcaccgttcttcgtaagtcaggttaaaaaactggcaactaaaccgctc180
gactgtcatctgatggtgacgcggccgcaggattacattgctcaactggcgcgtgcggga240
gcagatttcatcactctgcatccggaaaccatcaacggccaggcgttccgcctgattgat300
gaaatccgccgtcatgacatgaaagtggggctgatccttaacccggagacgccagttgag360
gccatgaaatactatatccataaggccgataaaattacggtcatgactgtcgatcccggc420
tttgccggacaaccgttcattcctgaaatgctggataaacttgccgaactgaaggcatgg480
cgtgaacgagaaggtctggagtacgaaattgaggtggacggttcctgcaaccaggcaact540
tacgaaaaactgatggcggcaggggcggatgtctttatcgtcggcacttccggcctgttt600
aatcatgcggaaaatatcgacgaagcatggagaattatgaccgcgcagattctggctgca660
aaaagcgaggtacagcctcatgcaaaaacagcataa696
<210>19
<211>930
<212>dna
<213>大肠杆菌
<400>19
atgcaaaaacagcataacgtcgtagcgggcgtggatatgggggcaacgcatatccgcttt60
tgtctgcggacagcagaaggtgaaacgctacactgcgaaaaaaagcggaccgcagaagtc120
attgctcccggcctggtgtcgggtatcggcgaaatgattgacgagcaactcaggcgcttt180
aacgctcgctgtcatggtctggtgatgggatttccggcgctggtcagtaaagataaacgc240
accattatttctacgcctaacctgccgttaacagcggcggatttatatgatctcgccgat300
aagctcgaaaatacgctgaattgtccggttgagttttcccgcgacgttaacctgcaactc360
tcctgggacgtagtagaaaaccgccttacgcaacaactggttctggcggcctatctcggt420
acggggatggggttcgcagtgtggatgaacggtgcgccgtggacgggtgcacacggtgtg480
gcaggcgaactgggtcatatccccctgggagatatgacccaacactgcgcgtgtggcaat540
cctgggtgcctggaaaccaattgctctggaatggcgctaagacgctggtacgaacaacag600
ccccgaaattacccattgcgcgatcttttcgtccatgcggaaaacgcccctttcgtccag660
agtctgcttgaaaacgcggcacgggccattgccaccagcattaatctgttcgatcccgat720
gcggtgatcctgggcggtggcgtgatggatatgcccgccttcccacgcgagactctcgtt780
gccatgacccaaaagtacctgcgccgtccactgccgcatcaggtcgtgcgctttattgcc840
gcctcatcttctgactttaatggcgctcagggtgcagcaatattggcgcatcaacgtttt900
ttgccacagttctgtgctaaagccccatga930
<210>20
<211>149
<212>prt
<213>大肠杆菌
<400>20
metlyslysilealapheglycysasphisvalglypheileleulys
151015
hisgluilevalalahisleuvalgluargglyvalgluvalileasp
202530
lysglythrtrpsersergluargthrasptyrprohistyralaser
354045
glnvalalaleualavalalaglyglygluvalaspglyglyileleu
505560
ilecysglythrglyvalglyileserilealaalaasnlyspheala
65707580
glyileargalavalvalcyssergluprotyrseralaglnleuser
859095
argglnhisasnaspthrasnvalleualapheglyserargvalval
100105110
glyleugluleualalysmetilevalaspalatrpleuglyalagln
115120125
tyrgluglyglyarghisglnglnargvalglualailethralaile
130135140
gluglnargargasn
145
<210>21
<211>296
<212>prt
<213>大肠杆菌
<400>21
metserglnserglupheaspseralaleuproasnglyileglyleu
151015
alaprotyrleuargmetlysglngluglymetthrgluasngluser
202530
argilevalglutrpleuleulysproglyasnleusercysalapro
354045
alailelysaspvalalaglualaleualavalserglualametile
505560
vallysvalserlysleuleuglypheserglypheargasnleuarg
65707580
seralaleugluasptyrpheserglnsergluglnvalleuproser
859095
gluleualapheaspglualaproglnaspvalvalasnlysvalphe
100105110
asnilethrleuargthrilemetgluglyglnserilevalasnval
115120125
aspgluilehisargalaalaargphephetyrglnalaargglnarg
130135140
aspleutyrglyalaglyglyserasnalailecysalaaspvalgln
145150155160
hislyspheleuargileglyvalargcysglnalatyrproaspala
165170175
hisilemetmetmetseralaserleuleuglngluglyaspvalval
180185190
leuvalvalthrhisserglyargthrseraspvallysalaalaval
195200205
gluleualalyslysasnglyalalysileilecysilethrhisser
210215220
tyrhisserproilealalysleualaasptyrileilecysserpro
225230235240
alaprogluthrproleuleuglyargasnalaseralaargileleu
245250255
glnleuthrleuleuaspalaphephevalservalalaglnleuasn
260265270
ilegluglnalaasnileasnmetglnlysthrglyalailevalasp
275280285
phepheserproglyalaleulys
290295
<210>22
<211>311
<212>prt
<213>大肠杆菌
<400>22
metasnlystyrleulystyrpheserglythrleuvalglyleumet
151015
leuserthrseralaphealaalaalaglutyralavalvalleulys
202530
thrleuserasnprophetrpvalaspmetlyslysglyilegluasp
354045
glualalysthrleuglyvalservalaspilephealaserproser
505560
gluglyasppheglnserglnleuglnleuphegluaspleuserasn
65707580
lysasntyrlysglyilealaphealaproleuserservalasnleu
859095
valmetprovalalaargalatrplyslysglyiletyrleuvalasn
100105110
leuaspglulysileaspmetaspasnleulyslysalaglyglyasn
115120125
valglualaphevalthrthraspasnvalalavalglyalalysgly
130135140
alaserpheileileasplysleuglyalagluglyglygluvalala
145150155160
ileilegluglylysalaglyasnalaserglyglualaargargasn
165170175
glyalathrglualaphelyslysalaserglnilelysleuvalala
180185190
serglnproalaasptrpaspargilelysalaleuaspvalalathr
195200205
asnvalleuglnargasnproasnilelysalailetyrcysalaasn
210215220
aspthrmetalametglyvalalaglnalavalalaasnalaglylys
225230235240
thrglylysvalleuvalvalglythraspglyileproglualaarg
245250255
lysmetvalglualaglyglnmetthralathrvalalaglnasnpro
260265270
alaaspileglyalathrglyleulysleumetvalaspalaglulys
275280285
serglylysvalileproleuasplysalaprogluphelysleuval
290295300
aspserileleuvalthrgln
305310
<210>23
<211>326
<212>prt
<213>大肠杆菌
<400>23
metglyphethrthrargvallysserglualaserglulyslyspro
151015
pheasnphealaleuphetrpasplystyrglythrphepheileleu
202530
alaileilevalalailepheglyserleuserproglutyrpheleu
354045
thrthrasnasnilethrglnilephevalglnserservalthrval
505560
leuileglymetglygluphephealaileleuvalalaglyileasp
65707580
leuservalglyalaileleualaleuserglymetvalthralalys
859095
leumetleualaglyvalasppropheleualaalametileglygly
100105110
valleuvalglyglyalaleuglyalaileasnglycysleuvalasn
115120125
trpthrglyleuhispropheileilethrleuglythrasnalaile
130135140
pheargglyilethrleuvalileseraspalaasnservaltyrgly
145150155160
pheserpheaspphevalasnphephealaalaservalileglyile
165170175
provalprovalilepheserleuilevalalaleuileleutrpphe
180185190
leuthrthrargmetargleuglyargasniletyralaleuglygly
195200205
asnlysasnseralaphetyrserglyileaspvallysphehisile
210215220
leuvalvalpheileileserglyvalcysalaglyleualaglyval
225230235240
valserthralaargleuglyalaalagluproleualaglymetgly
245250255
phegluthrtyralailealaseralaileileglyglythrserphe
260265270
pheglyglylysglyargilepheservalvalileglyglyleuile
275280285
ileglythrileasnasnglyleuasnileleuglnvalglnthrtyr
290295300
tyrglnleuvalvalmetglyglyleuileilealaalavalalaleu
305310315320
aspargleuileserlys
325
<210>24
<211>326
<212>prt
<213>大肠杆菌
<400>24
metglyphethrthrargvallysserglualaserglulyslyspro
151015
pheasnphealaleuphetrpasplystyrglythrphepheileleu
202530
alaileilevalalailepheglyserleuserproglutyrpheleu
354045
thrthrasnasnilethrglnilephevalglnserservalthrval
505560
leuileglymetglygluphephealaileleuvalalaglyileasp
65707580
leuservalglyalaileleualaleuserglymetvalthralalys
859095
leumetleualaglyvalasppropheleualaalametileglygly
100105110
valleuvalglyglyalaleuglyalaileasnglycysleuvalasn
115120125
trpthrglyleuhispropheileilethrleuglythrasnalaile
130135140
pheargglyilethrleuvalileseraspalaasnservaltyrgly
145150155160
pheserpheaspphevalasnphephealaalaservalileglyile
165170175
provalprovalilepheserleuilevalalaleuileleutrpphe
180185190
leuthrthrargmetargleuglyargasniletyralaleuglygly
195200205
asnlysasnseralaphetyrserglyileaspvallysphehisile
210215220
leuvalvalpheileileserglyvalcysalaglyleualaglyval
225230235240
valserthralaargleuglyalaalagluproleualaglymetgly
245250255
phegluthrtyralailealaseralaileileglyglythrserphe
260265270
pheglyglylysglyargilepheservalvalileglyglyleuile
275280285
ileglythrileasnasnglyleuasnileleuglnvalglnthrtyr
290295300
tyrglnleuvalvalmetglyglyleuileilealaalavalalaleu
305310315320
aspargleuileserlys
325
<210>25
<211>309
<212>prt
<213>大肠杆菌
<400>25
metglnlysglnhisasnvalvalalaglyvalaspmetglyalathr
151015
hisileargphecysleuargthralagluglygluthrleuhiscys
202530
glulyslysargthralagluvalilealaproglyleuvalsergly
354045
ileglyglumetileaspgluglnleuargargpheasnalaargcys
505560
hisglyleuvalmetglypheproalaleuvalserlysasplysarg
65707580
thrileileserthrproasnleuproleuthralaalaaspleutyr
859095
aspleualaasplysleugluasnthrleuasncysprovalgluphe
100105110
serargaspvalasnleuglnleusertrpaspvalvalgluasnarg
115120125
leuthrglnglnleuvalleualaalatyrleuglythrglymetgly
130135140
phealavaltrpmetasnglyalaprotrpthrglyalahisglyval
145150155160
alaglygluleuglyhisileproleuglyaspmetthrglnhiscys
165170175
alacysglyasnproglycysleugluthrasncysserglymetala
180185190
leuargargtrptyrgluglnglnproargasntyrproleuargasp
195200205
leuphevalhisalagluasnalaprophevalglnserleuleuglu
210215220
asnalaalaargalailealathrserileasnleupheaspproasp
225230235240
alavalileleuglyglyglyvalmetaspmetproalapheproarg
245250255
gluthrleuvalalametthrglnlystyrleuargargproleupro
260265270
hisglnvalvalargpheilealaalaserserserasppheasngly
275280285
alaglnglyalaalaileleualahisglnargpheleuproglnphe
290295300
cysalalysalapro
305
<210>26
<211>309
<212>prt
<213>大肠杆菌
<400>26
metglnlysglnhisasnvalvalalaglyvalaspmetglyalathr
151015
hisileargphecysleuargthralagluglygluthrleuhiscys
202530
glulyslysargthralagluvalilealaproglyleuvalsergly
354045
ileglyglumetileaspgluglnleuargargpheasnalaargcys
505560
hisglyleuvalmetglypheproalaleuvalserlysasplysarg
65707580
thrileileserthrproasnleuproleuthralaalaaspleutyr
859095
aspleualaasplysleugluasnthrleuasncysprovalgluphe
100105110
serargaspvalasnleuglnleusertrpaspvalvalgluasnarg
115120125
leuthrglnglnleuvalleualaalatyrleuglythrglymetgly
130135140
phealavaltrpmetasnglyalaprotrpthrglyalahisglyval
145150155160
alaglygluleuglyhisileproleuglyaspmetthrglnhiscys
165170175
alacysglyasnproglycysleugluthrasncysserglymetala
180185190
leuargargtrptyrgluglnglnproargasntyrproleuargasp
195200205
leuphevalhisalagluasnalaprophevalglnserleuleuglu
210215220
asnalaalaargalailealathrserileasnleupheaspproasp
225230235240
alavalileleuglyglyglyvalmetaspmetproalapheproarg
245250255
gluthrleuvalalametthrglnlystyrleuargargproleupro
260265270
hisglnvalvalargpheilealaalaserserserasppheasngly
275280285
alaglnglyalaalaileleualahisglnargpheleuproglnphe
290295300
cysalalysalapro
305
<210>27
<211>2067
<212>dna
<213>棒状杆菌
<400>27
atgaatagcgtaaataattcctcgcttgtccggctggatgtcgatttcggcgactccacc60
acggatgtcatcaacaaccttgccactgttattttcgacgctggccgagcttcctccgcc120
gacgcccttgccaaagacgcgctggatcgtgaagcaaagtccggcaccggcgttcctggt180
caagttgctatcccccactgccgttccgaagccgtatctgtccctaccttgggctttgct240
cgcctgagcaagggtgtggacttcagcggacctgatggcgatgccaacttggtgttcctc300
attgcagcacctgctggcggcggcaaagagcacctgaagatcctgtccaagcttgctcgc360
tccttggtgaagaaggatttcatcaaggctctgcaggaagccaccaccgagcaggaaatc420
gtcgacgttgtcgatgccgtgctcaacccagcaccaaaaaccaccgagccagctgcagct480
ccggctgcggcggcggttgctgagagtggggcggcgtcgacaagcgttactcgtatcgtg540
gcaatcaccgcatgcccaaccggtatcgcacacacctacatggctgcggattccctgacg600
caaaacgcggaaggccgcgatgatgtggaactcgttgtggagactcagggctcttccgct660
gtcaccccagtcgatccgaagatcatcgaagctgccgacgccgtcatcttcgccaccgac720
gtgggagttaaagaccgcgagcgtttcgctggcaagccagtcattgaatccggcgtcaag780
cgcgcgatcaatgagccagccaagatgatcgacgaggccatcgcagcctccaagaaccca840
aacgcccgcaaggtttccggttccggtgtcgcggcatctgctgaaaccaccggcgagaag900
ctcggctggggcaagcgcatccagcaggcagtcatgaccggcgtgtcctacatggttcca960
ttcgtagctgccggcggcctcctgttggctctcggcttcgcattcggtggatacgacatg1020
gcgaacggctggcaagcaatcgccacccagttctctctgaccaacctgccaggcaacacc1080
gtcgatgttgacggcgtggccatgaccttcgagcgttcaggcttcctgttgtacttcggc1140
gcagtcctgttcgccaccggccaagcagccatgggcttcatcgtggcagccctgtctggc1200
tacaccgcatacgcacttgctggacgcccaggcatcgcgccgggcttcgtcggtggcgcc1260
atctccgtcaccatcggcgctggcttcattggtggtctggttaccggtatcttggctggt1320
ctcattgccctgtggattggctcctggaaggtgccacgcgtggtgcagtcactgatgcct1380
gtggtcatcatcccgctacttacctcagtggttgttggtctcgtcatgtacctcctgctg1440
ggtcgcccactcgcatccatcatgactggtttgcaggactggctatcgtcaatgtccgga1500
agctccgccatcttgctgggtatcatcttgggcctcatgatgtgtttcgacctcggcgga1560
ccagtaaacaaggcagcctacctctttggtaccgcaggcctgtctaccggcgaccaagct1620
tccatggaaatcatggccgcgatcatggcagctggcatggtcccaccaatcgcgttgtcc1680
attgctaccctgctgcgcaagaagctgttcaccccagcagagcaagaaaacggcaagtct1740
tcctggctgcttggcctggcattcgtctccgaaggtgccatcccattcgccgcagctgac1800
ccattccgtgtgatcccagcaatgatggctggcggtgcaaccactggtgcaatctccatg1860
gcactgggcgtcggctctcgggctccacacggcggtatcttcgtggtctgggcaatcgaa1920
ccatggtggggctggctcatcgcacttgcagcaggcaccatcgtgtccaccatcgttgtc1980
atcgcactgaagcagttctggccaaacaaggccgtcgctgcagaagtcgcgaagcaagaa2040
gcacaacaagcagctgtaaacgcataa2067
<210>28
<211>688
<212>prt
<213>棒状杆菌
<400>28
metasnservalasnasnserserleuvalargleuaspvalaspphe
151015
glyaspserthrthraspvalileasnasnleualathrvalilephe
202530
aspalaglyargalaserseralaaspalaleualalysaspalaleu
354045
aspargglualalysserglythrglyvalproglyglnvalalaile
505560
prohiscysargserglualavalservalprothrleuglypheala
65707580
argleuserlysglyvalasppheserglyproaspglyaspalaasn
859095
leuvalpheleuilealaalaproalaglyglyglylysgluhisleu
100105110
lysileleuserlysleualaargserleuvallyslysasppheile
115120125
lysalaleuglnglualathrthrgluglngluilevalaspvalval
130135140
aspalavalleuasnproalaprolysthrthrgluproalaalaala
145150155160
proalaalaalaalavalalagluserglyalaalaserthrserval
165170175
thrargilevalalailethralacysprothrglyilealahisthr
180185190
tyrmetalaalaaspserleuthrglnasnalagluglyargaspasp
195200205
valgluleuvalvalgluthrglnglyserseralavalthrproval
210215220
aspprolysileileglualaalaaspalavalilephealathrasp
225230235240
valglyvallysasparggluargphealaglylysprovalileglu
245250255
serglyvallysargalaileasngluproalalysmetileaspglu
260265270
alailealaalaserlysasnproasnalaarglysvalserglyser
275280285
glyvalalaalaseralagluthrthrglyglulysleuglytrpgly
290295300
lysargileglnglnalavalmetthrglyvalsertyrmetvalpro
305310315320
phevalalaalaglyglyleuleuleualaleuglyphealaphegly
325330335
glytyraspmetalaasnglytrpglnalailealathrglnpheser
340345350
leuthrasnleuproglyasnthrvalaspvalaspglyvalalamet
355360365
thrphegluargserglypheleuleutyrpheglyalavalleuphe
370375380
alathrglyglnalaalametglypheilevalalaalaleusergly
385390395400
tyrthralatyralaleualaglyargproglyilealaproglyphe
405410415
valglyglyalaileservalthrileglyalaglypheileglygly
420425430
leuvalthrglyileleualaglyleuilealaleutrpileglyser
435440445
trplysvalproargvalvalglnserleumetprovalvalileile
450455460
proleuleuthrservalvalvalglyleuvalmettyrleuleuleu
465470475480
glyargproleualaserilemetthrglyleuglnasptrpleuser
485490495
sermetserglyserseralaileleuleuglyileileleuglyleu
500505510
metmetcyspheaspleuglyglyprovalasnlysalaalatyrleu
515520525
pheglythralaglyleuserthrglyaspglnalasermetgluile
530535540
metalaalailemetalaalaglymetvalproproilealaleuser
545550555560
ilealathrleuleuarglyslysleuphethrproalagluglnglu
565570575
asnglylyssersertrpleuleuglyleualaphevalserglugly
580585590
alaileprophealaalaalaasppropheargvalileproalamet
595600605
metalaglyglyalathrthrglyalailesermetalaleuglyval
610615620
glyserargalaprohisglyglyilephevalvaltrpalaileglu
625630635640
protrptrpglytrpleuilealaleualaalaglythrilevalser
645650655
thrilevalvalilealaleulysglnphetrpproasnlysalaval
660665670
alaalagluvalalalysglnglualaglnglnalaalavalasnala
675680685
<210>29
<211>1512
<212>dna
<213>棒状杆菌
<400>29
atgaacaccccactccagctcaacactgaaaacctgcaggaaatcgcttcgacttccgga60
gtgcagatcccagcgttcaaccgcgctgacgtcgccccgggcattgtccacttcggtgtt120
ggcggattccatcgcgctcaccaagcgatgtacctcaatgaattgatgaatgagggcaag180
gccttggattggggcatcatcggcatgggtgtcatgccttccgatgtgcgcatgcgcgat240
gccctggccagccaagatcacctttataccctgaccactaaagctcctgatggaactctt300
gatcaaaaaatcatcggatccatcattgactacgtgttcgctcccgaggacccagcacgg360
gccgttgcaaccctcgcgcaggactccatccgcattgtttccctcacggtgactgaaggc420
ggatacaacatcgatccggcgacagaagatttcgaccacaccaaccctcgaatcgttgct480
gaccgcgaagccctgcaggcgggcgatacttccactttgcagaccttctttgggttgatc540
actgccgcattgatttcccgaaaagaatcaggatctacgccatttaccatcatgagctgc600
gataacatccaaggcaacggcgatctggctaagcgtttcttcctcgccttcgcacattcc660
gtgtcttctgagctcggcgaatgggtggaaaacaacgtggccttccccaactccatggtg720
gaccgcatcacccctgaaaccaccgacggcgaccgcgatgacatcaaggaaatcggctac780
atcgatgcgtggccagtggtttctgaagatttcacccaatgggtcctcgaggatgccttc840
acccagggccgccccgcgtacgaggaggttggcgtgcaggtcgtctccgacgtggagcct900
tatgaattaatgaagctgcgcctgctcaacgcctcccaccagggactttgctacttcggc960
cacttggctggccaccacatggtccacgacgtcatggcggatacccgcttccaggatttc1020
ctcctggcttacatggagcgcgaagccacccctaccctcaaggaacttccaggtgtcgat1080
ctagatgcttatcgacgccaactcatcgcgcgattcggcaacgccgcagtcaaagacacc1140
gtaccgcgcctgtgtgcggaatcctccgaccgcattccaaagtggctgttgccagtcgta1200
cgcgaaaacctcgcagcaggccgcgacgtcacactttctgcagccatcgtcgcatcctgg1260
gcgcgctacgcagaaggcaccgacgagcagggcaacccaataaagattgttgaccgtttg1320
agtgagcgcgtccaagaaaacgcatcaggaaatcgcaccgatattttgtcattcatccgc1380
gaccgtggaatcttcggagacttggtcgatgctgaaccattcaccaaggcatactccgag1440
acactgtcctcccttcatgaccgtggcgcggaagcaaccatcgatgcacttcttacgcag1500
gtaactgtctaa1512
<210>30
<211>503
<212>prt
<213>棒状杆菌
<400>30
metasnthrproleuglnleuasnthrgluasnleuglngluileala
151015
serthrserglyvalglnileproalapheasnargalaaspvalala
202530
proglyilevalhispheglyvalglyglyphehisargalahisgln
354045
alamettyrleuasngluleumetasngluglylysalaleuasptrp
505560
glyileileglymetglyvalmetproseraspvalargmetargasp
65707580
alaleualaserglnasphisleutyrthrleuthrthrlysalapro
859095
aspglythrleuaspglnlysileileglyserileileasptyrval
100105110
phealaprogluaspproalaargalavalalathrleualaglnasp
115120125
serileargilevalserleuthrvalthrgluglyglytyrasnile
130135140
aspproalathrgluasppheasphisthrasnproargilevalala
145150155160
aspargglualaleuglnalaglyaspthrserthrleuglnthrphe
165170175
pheglyleuilethralaalaleuileserarglysgluserglyser
180185190
thrprophethrilemetsercysaspasnileglnglyasnglyasp
195200205
leualalysargphepheleualaphealahisservalserserglu
210215220
leuglyglutrpvalgluasnasnvalalapheproasnsermetval
225230235240
aspargilethrprogluthrthraspglyaspargaspaspilelys
245250255
gluileglytyrileaspalatrpprovalvalsergluaspphethr
260265270
glntrpvalleugluaspalaphethrglnglyargproalatyrglu
275280285
gluvalglyvalglnvalvalseraspvalgluprotyrgluleumet
290295300
lysleuargleuleuasnalaserhisglnglyleucystyrphegly
305310315320
hisleualaglyhishismetvalhisaspvalmetalaaspthrarg
325330335
pheglnasppheleuleualatyrmetgluargglualathrprothr
340345350
leulysgluleuproglyvalaspleuaspalatyrargargglnleu
355360365
ilealaargpheglyasnalaalavallysaspthrvalproargleu
370375380
cysalagluserseraspargileprolystrpleuleuprovalval
385390395400
arggluasnleualaalaglyargaspvalthrleuseralaalaile
405410415
valalasertrpalaargtyralagluglythraspgluglnglyasn
420425430
proilelysilevalaspargleusergluargvalglngluasnala
435440445
serglyasnargthraspileleuserpheileargaspargglyile
450455460
pheglyaspleuvalaspalagluprophethrlysalatyrserglu
465470475480
thrleuserserleuhisaspargglyalaglualathrileaspala
485490495
leuleuthrglnvalthrval
500
<210>31
<211>53
<212>dna
<213>人工序列
<220>
<223>引物
<400>31
gcaggtaccaggaggtaataaatatgaaacacggcatctattattcttactgg53
<210>32
<211>32
<212>dna
<213>人工序列
<220>
<223>引物
<400>32
gctggatccttagccaccaagaacgaagcggg32
<210>33
<211>23
<212>dna
<213>人工序列
<220>
<223>引物
<400>33
ccacggaactttcggctgttttg23
<210>34
<211>22
<212>dna
<213>人工序列
<220>
<223>引物
<400>34
cttccgtaggtgccacaatatc22
<210>35
<211>44
<212>dna
<213>人工序列
<220>
<223>引物
<400>35
cgaggatccaggaggtaataatatgaaaaataaattcggagttg44
<210>36
<211>28
<212>dna
<213>人工序列
<220>
<223>引物
<400>36
gtctagattatatctcagcagcgatggc28
<210>37
<211>53
<212>dna
<213>人工序列
<220>
<223>引物
<400>37
cggtaccaggaggtaataatatgaaatatggtatttattttgcttattggacg53
<210>38
<211>32
<212>dna
<213>人工序列
<220>
<223>引物
<400>38
ctctagattagataccaaacacatgcctgcag32
<210>39
<211>24
<212>dna
<213>人工序列
<220>
<223>引物
<400>39
cggactatctgaacgaactgacgg24
<210>40
<211>23
<212>dna
<213>人工序列
<220>
<223>引物
<400>40
ctccataagcatcctcctcatcc23
<210>41
<211>45
<212>dna
<213>人工序列
<220>
<223>引物
<400>41
cgaggatccaggaggtaataatatgaaacatggtatctattatgc45
<210>42
<211>27
<212>dna
<213>人工序列
<220>
<223>引物
<400>42
gtctagattacttccactccagcatat27
-
 0198117... 来自[中国] 2023年01月09日 19:02文章很不错
0198117... 来自[中国] 2023年01月09日 19:02文章很不错