基于RNA聚合酶的体外转录机器快速多重检测NTPs的方法及试剂盒与应用与流程
本发明属于技术领域,涉及一种基于RNA聚合酶的体外转录机器快速多重检测NTPs的方法及试剂盒与应用,用于对ATP、CTP、UTP和GTP进行超快速、高选择性、高灵敏和高通量的检测。
背景技术:
三磷酸核苷(nucleoside triphosphates,NTPs,又称核苷三磷酸)是生命体内最基本、最重要的功能分子,由一分子核糖、一分子碱基和三分子磷酸组成。根据碱基类别,NTPs被分为三磷酸腺苷(adenosine triphosphate,ATP)、三磷酸胞苷(cytidine triphosphate,CTP)、三磷酸尿苷(uridine triphosphate,UTP)和三磷酸鸟苷(guanosine triphosphate,GTP)。NTPs作为核酸分子的原料,不仅确保核酸的正常复制、修复、转录和逆转录,还在几乎所有的细胞结构、新陈代谢、信号转导等方面起着重要作用(Y.Zhou,et al.Chem.Soc.Rev.2011,40,2222-2235)。NTPs水平的变化或不平衡会导致生理反应异常、代谢紊乱、细胞凋亡或坏死,并与许多疾病如癌症、帕金森氏病等密切相关(D.Davalos,et al.Nat.Neurosci.2005,8,752)。同时,多种抗癌和抗病毒药物作为核苷酸类似物,以干扰NTPs的生物合成、竞争性参与RNA合成或代谢活动、扰乱NTPs水平等机制来实现其药理作用(L.P.Jordheim,et al.Biochim.Biophys.Acta.2007,1776,138)。因此,发展对NTPs的多重体外检测和超快速分析方法具有重大需求和意义,可以为研究生理与病理的基本机制、关联信号通路(S.Seino,et al.Diabetologia 2012,55,2096)、研发抗癌药物及载药系统(R.Mo,et al.Nat.Commun.2014,5,3364)和监测药物治疗效果等提供必要、便捷和高效的研究技术和手段。
然而发展NTPs的多重体外检测和超快速分析方法却是十分困难和具有挑战性的。众多结构相似的核苷酸衍生物,例如脱氧核苷三磷酸(deoxyribonucleoside triphosphates,dNTPs),二磷酸核苷(nucleoside diphosphates,NDPs)和单磷酸核苷(nucleoside monophosphates,NMPs),会对NTPs的检测产生干扰和假阳性,从而使检测方法缺乏对NTPs的选择性。而要同时满足NTPs的选择性、多种NTPs的同步检测、超快速(小于5分钟)、简便(无需大型仪器)和高灵敏(nM)的检测要求更是难上加难。目前基于分离的方法如色谱、毛细管电泳及质谱联用,虽然可以实现对多种NTPs的同步检测,但面临细胞基质和基线噪音的干扰,样品前处理复杂耗时,运行时间长(大于2小时),灵敏度相对有限(低于10pmol或50nM),且依赖于昂贵的大型仪器和专门训练的技术人员,在临床分析应用受到极大限制(S.Cohen,et al.J.Chromatogr.B 2009,877,3831)。而基于分子间的相互作用,结合荧光法(Z.Xu,et al.Chem.Soc.Rev.2010,39,1457)、电化学法(L.Lu,et al.Biosens.Bioelectron.2015,63,14–20)、化学发光法(R.Freeman,et al.J.Am.Chem.Soc.2011,133,11597)和比色法(D.Cheng,et al.Chem.Commun.2015,51,8544)等已经发展了各种各样的传感识别手段,其中荧光分析法因操作简单、响应快速、高灵敏和高通量的特点在临床样品分析独具优势。具体地,目前NTPs的传感识别原理分为六类:一是有机小分子荧光探针(X.Li,et al.Angew.Chem.Int.Ed.2014,53,7809),其优点是响应快,但合成步骤极为复杂,细胞毒性、灵敏度(μM)和选择性都较差;二是基因编码的荧光蛋白或多肽(H.Yaginuma,et al.Sci.Rep.2014,4,6522),虽生物相容性好,但表达和纯化步骤复杂,灵敏度和选择性也都较差;三是体外筛选的适配体(A.B.Iliuk,et al.Anal.Chem.2011,83,4440),ATP适配体的本质是脱氧核糖核酸(deoxyribonucleic acid,DNA),对二磷酸腺苷(adenosine diphosphate,ADP)和一磷酸腺苷(adenosine monophosphate,AMP)的选择性较差,GTP的适配体是核糖核酸(ribonucleic acid,RNA),稳定性不好,且适配体与目标分子的孵育时间过长(大于30分钟),而UTP与CTP的适配体却鲜有报道;四是基于特异性识别ATP或GTP的脱氧核酶(A.Sreedhara,et al.J.Am.Chem.Soc.2004,126,3454;S.A.Mcmanus,et al.J.Am.Chem.Soc.2013,135,7181),但灵敏度(μM)非常差;五是基于ATP依赖的萤光素酶(P.E.Stanley,et al.Biolumin.Chemilumin.1989,4,289),其活性容易受到环境因素的影响,化学反应发出的荧光不稳定,30分钟后光强可下降10%,并且通用性差,无法检测CTP、UTP和GTP;六是基于ATP依赖的T4 DNA连接酶(L.M.Lu,et al.J.Am.Chem.Soc.2011,133,11686),连接时间至少半小时,且同样无法用于CTP、UTP和GTP的检测。综上所述,现有的传感方法均是检测ATP或GTP的单一目标物传感器,且在选择性、灵敏度、反应时间和简便性上只能满足其中的一两个方面,无法对受到外界刺激时NTP的含量和关联性变化进行快速检测,难以满足未来科学研究和临床分析对快速多重检测NTPs的需求。
因此,迫切需要发展新的传感识别机制,突破现有技术对选择性、多重分析能力、灵敏度和反应时间的限制,开发出同时具备超快速、高选择性、高灵敏、高通量、低成本和实用性强的NTPs的多重检测方法。
技术实现要素:
本发明的目的是提供一种基于RNA聚合酶的体外转录机器快速多重检测NTPs的方法及试剂盒与应用,实现了对四种不同的NTPs进行超快速准确定量、高选择性、高灵敏度、高通量、低成本、便于临床和研究应用的检测。
为了实现上述目的,本发明采用以下技术方案:
一种基于RNA聚合酶的体外转录机器快速多重检测NTPs的方法,包括:
1)预杂交形成双链DNA的模板链和编码链,在RNA聚合酶启动子序列之后的转录区域的模板链上设置与待检测的目标NTP的碱基类型互补的碱基作为识别位点,其余的转录序列则不含有识别位点,所述编码链上与识别位点互补的碱基是目标位点,其后是能被分子信标(molecular beacon,MB)识别的信号序列;
2)在RNA聚合酶和过量的其他非目标NTPs存在时,加入目标NTP使转录反应顺利通过识别位点,得到包含目标NTP和能被分子信标识别的信号序列的转录产物RNA,从而使目标NTP转化为携带信号序列的RNA;
3)通过转录产物RNA的信号序列与分子信标的结合,实现对目标NTP的检测。
进一步地,所述NTPs包括四种NTP:ATP、CTP、UTP和GTP,所述目标NTP为四种NTP中的一种或任意两种的组合。
进一步地,所述目标NTP为ATP时,模板链的识别位点设置成T,其余的转录序列不含有T,此时编码链上的目标位点为A,其余与转录序列互补的部分不含有A。优选地,ATP的模板链从转录起始到识别位点之后的相邻碱基之间的最优序列结果是(5'→3')CCCCTG(粗体为识别位点),剩余模板链的序列可由任意不含T的碱基组成。
进一步地,所述目标NTP为CTP时,模板链的识别位点设置成G,其余的转录序列不含有G,此时编码链上的目标位点为C,其余与转录序列互补的部分不含有C。优选地,CTP的模板链从转录起始到识别位点之后的相邻碱基之间的最优序列结果是(5'→3')CCCCTGT(粗体为识别位点),剩余模板链的序列可由任意不含G的碱基组成。
进一步地,所述目标NTP为UTP时,模板链的识别位点设置成A,其余的转录序列不含有A,此时编码链上的识别位点为T,其余与转录序列互补的部分不含有T。优选地,UTP的模板链从转录起始到识别位点之后的相邻碱基之间的最优序列结果是(5'→3')CCCCGAG(粗体为识别位点),剩余模板链的序列可由任意不含A的碱基组成。
进一步地,所述目标NTP为GTP时,模板链的识别位点设置成C,其余的转录序列不含有C,此时编码链上的识别位点为G,其余与转录序列互补的部分不含有G。优选地,GTP的模板链从转录起始到识别位点之后的相邻碱基之间的最优序列结果是(5'→3')GCT(粗体为识别位点),剩余模板链的序列可由任意不含C的碱基组成。
进一步地,为节约实验成本,可使信号序列由两种非目标NTPs组成,从而使其中的两种目标NTPs在检测时可以共享一种分子信标。例如ATP和CTP的信号序列可设计为UGUUGUGGUGUGUUGGUU;UTP和GTP的信号序列可设计为ACAACACCACACAACCAA。并且信号序列的位置因在目标位点之后,便于在无需改变分子信标的情况下即可探索和优化识别位点的位置及前后碱基的组成,观测其对荧光曲线和信号窗口的影响,以此设计出性能最优的模板序列。
进一步地,所述目标NTP为四种NTP中任意两种的组合时,每种目标NTP的模板链的非识别位点碱基类型不包括自身的识别位点碱基类型及另外一种目标NTP的识别位点碱基类型;两者对应的转录产物RNA的信号序列各不相同,且对应的分子信标的环部序列和荧光基团标记也各不相同,以实现对不同模板链的转录产物RNA的分别响应。
进一步地,本发明所用的RNA聚合酶可包括各种类别和来源的RNA聚合酶,例如:噬菌体RNA聚合酶(T7 RNA聚合酶,T3 RNA聚合酶,SP6 RNA聚合酶,K11 RNA聚合酶和BA14 RNA聚合酶),原核生物RNA聚合酶(大肠杆菌RNA聚合酶),真核生物的RNA聚合酶(RNA聚合酶Ⅰ,RNA聚合酶Ⅱ和RNA聚合酶Ⅲ)。
进一步地,所述分子信标是一条呈发夹结构的短链DNA,一端标记荧光基团,另一端标记猝灭基团,所述荧光基团为FAM,猝灭基团为DABCYL或BHQ1或TAMRA;或者所述荧光基团为ROX,猝灭基团为BHQ2。
进一步地,步骤3)中还可包括:根据转录产物RNA的信号序列与分子信标结合时生成的实时荧光上升曲线对目标NTP进行定量检测。
进一步地,上述定量检测中进行定量的方法包括:根据实时荧光曲线上升的绝对速率或相对速率与目标NTP的浓度或绝对含量建立线性关系;或根据荧光强度值与目标NTP的浓度或绝对含量建立线性关系。
本发明还提供了一种基于RNA聚合酶的体外转录机器快速多重检测NTPs的试剂盒,包括:四种NTP套装,反应缓冲液,四种NTP标准品,RNA酶抑制剂以及RNA聚合酶,四种NTP套装又包括四种NTP的模板链、编码链和分子信标的DNA干粉,所述四种NTP包括ATP、CTP、UTP和GTP,所述模板链上设有待检测的目标NTP碱基类型的互补碱基作为识别位点,其余的转录序列则不含有识别位点,所述编码链包含能被分子信标(molecular beacon,MB)识别的信号序列。
进一步地,1x反应缓冲液的组成为100μg/mL牛血清白蛋白(bovine serum albumin,BSA),40mM Tris-HCl(pH 7.9),6mM MgCl2,2mM亚精胺,10mM DTT。
本发明可用于逻辑门的构建。当利用CCCCTG作为模板时,可以构建ATP和CTP的与门,只有体系中同时存在ATP和CTP(GTP、UTP和聚合酶等过量)时,转录反应才能顺利通过识别位点而转录成携带信号序列的RNA,使分子信标产生荧光信号;而当利用CCCCTA和CCCCGA同时作为模板时,则可以构建ATP和CTP的或门,体系中只要存在ATP或CTP的一种或两种(GTP、UTP和聚合酶等过量),就可以转录出携带信号序列的RNA,使分子信标产生荧光信号。
本发明还可用于检测样品中NTP/[NDP+NMP]的比值。当将模板链上的识别位点设置在起始转录位点时,由于起始转录位点可以掺入NDP和NMP,其反应效率与NTP一致,此时若反应体系中同时存在与目标NTP具有相同碱基的NDP和NMP时,则所测结果为[NTP+NDP+NMP]的总含量。而当将识别位点设置在转录区域中间如优化的模板序列时,只有目标NTP才能参与转录反应,NDP和NMP由于缺少r-磷酸基团而无法参与转录延伸反应,因此所测结果为NTP的含量,将[NTP+NDP+NMP]的总含量减去NTP的含量即可得[NDP+NMP]的含量,,最终计算得到样品NTP/NDP+NMP的比值,从而可以实时监测实际样品中NTP/[NDP+NMP]的值,由此反映细胞的生理状态、监测治疗效果、揭示功能机制。
本发明首次提出基于RNA聚合酶的体外转录机器快速多重检测NTP的方法,与现有技术相比具有以下明显优点:
(1)通用性强,是目前唯一一种可以同时用于四种NTP的传感检测方法。通过简单的改变模板链和编码链的序列、其余过量NTP的组成和相匹配的分子信标,即可实现对ATP、CTP、UTP和GTP的检测。
(2)反应体系简单,无需复杂的探针设计和合成步骤。反应所用的组分均为商业化试剂,反应时只需将各组分简单混合即可。
(3)一锅法的检测模式,有利于反应过程的即时监测。通过分子信标与转录产物RNA的结合所导致的荧光基团的信号恢复,实现了对待测NTP所参与的转录反应的即时快速监测,无需复杂的分离和纯化步骤,简化操作。而且荧光标记基团的荧光信号比化学发光更加稳定。
(4)超快速检测,极大将检测时间缩短至1分钟之内,使得检测更加高效快捷。
(5)高灵敏度,可检测浓度低至1nM,绝对含量低至20fmol的待测NTP样品,极大地缩减了对实际样品数量的要求,节省样品。
(6)宽的定量范围,可检测1~10000nM的浓度范围(绝对含量0.02~200pmol),且线性相关系数均大于0.99。
(7)高选择性,首次通过利用RNA聚合酶的活性中心、Watson-Crick碱基配对原则和来自酸酐键裂解的转录驱动力分别识别核糖、碱基和磷酸三个部分,从而为分子机器提供一种三重保障选择性的识别元件,并通过对转录序列的筛选和优化,有效避免了dNTPs、NDPs和NMPs的干扰,使得检测具有更高的可靠性和准确性。
(8)高通量检测,根据荧光检测仪器的通量可同时进行多个反应离心管或96孔板的检测,实现对多组样品的高通量检测,加样可以用排枪加样,易于自动化。
附图说明
图1本发明基于RNA聚合酶的体外转录机器检测ATP的原理示意图。
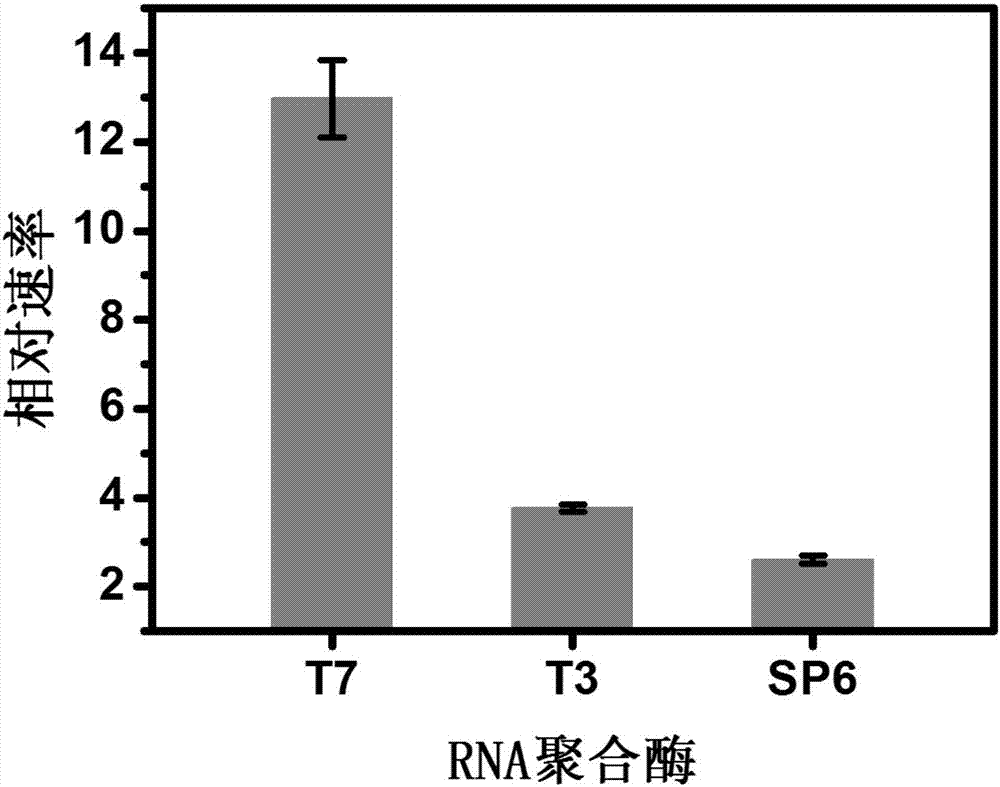
图2本发明实施例1中对三种商业RNA聚合酶的性能比较。
图3本发明实施例2中对NTPs的模板链上识别位点附近的序列优化结果,其中(a)(b)ATP的模板序列优化;(c)CTP的模板序列优化;(d)UTP的模板序列优化;(e)(f)GTP的模板序列优化。
图4本发明实施例3中对NTPs的检测范围与标准曲线结果,其中:(a)标准品为ATP;(b)标准品为CTP;(c)标准品为UTP;(d)标准品为GTP。
图5本发明实施例4中NTPs的选择性实验结果,其中:(a)对ATP的选择性;(b)对CTP的选择性;(c)对UTP的选择性;(d)对GTP的选择性。
图6本发明实施例6中逻辑门的构建原理图。
具体实施方式
下面结合附图,通过具体实施例进一步阐述本发明。本领域的技术人员应当理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。
本发明的技术方案是,根据RNA聚合酶的活性中心、Watson-Crick碱基配对原则和来自酸酐键裂解的转录驱动力分别识别核糖、碱基和磷酸三个部分,从而为分子机器提供一种三重保障选择性的识别元件,通过在模板链上设置待测NTP的互补碱基作为识别位点,在RNA聚合酶和其他三种非目标NTPs过量存在时,将目标NTP通过转录反应而转化为RNA分子,进而通过分子信标(molecular beacon,MB)的环部序列对转录产物RNA的信号序列进行特异性识别和结合,使得分子信标的构象发生变化而得以恢复荧光信号,从而通过荧光信号实时指示体外转录机器的动力学作用过程并对目标待测的NTP进行快速准确的定量检测。
所述RNA聚合酶,又称转录酶,是催化以DNA为模板、NTPs为底物、通过磷酸二酯键合成RNA过程的酶。作为模板的双链DNA中,指导RNA合成的一股链叫做模板链(template strand)或反义链(antisense strand),与此相对的另一股链叫做编码链(coding strand)或有意义链(sense strand),除了用T代替U外,编码链的其余碱基序列与转录产物RNA序列相同。RNA聚合酶辨认和结合的DNA部位称为启动子(promoter),且对启动子具有高度专一性。RNA聚合酶可以完整的双链DNA为模板,也可以包含启动子双链的不完整双链DNA为模板。RNA聚合酶催化的转录过程可划分为三步:(1)启动子DNA结合RNA聚合酶,并与转录5’端首位核苷酸形成起始复合物;(2)转录延伸,以首位核苷酸的3’-OH逐渐加入NTPs形成磷酸二酯键,从5’向3’端合成RNA;(3)转录终止。RNA聚合酶结构域上重要的氨基酸对NTPs起着特异性区分作用,可有效区分NTPs和dNTPs。
本发明所用的RNA聚合酶可由任何一种类别和来源的RNA聚合酶参与,例如:噬菌体RNA聚合酶(T7 RNA聚合酶,T3 RNA聚合酶,SP6 RNA聚合酶,K11 RNA聚合酶和BA14 RNA聚合酶),原核生物RNA聚合酶(大肠杆菌RNA聚合酶),真核生物的RNA聚合酶(RNA聚合酶Ⅰ,RNA聚合酶Ⅱ和RNA聚合酶Ⅲ,现对噬菌体RNA聚合酶及其启动子序列列举如下,但不限于以下方案:
1)T7 RNA聚合酶,高度特异识别T7启动子,序列如下:
编码链(5'→3'):TAATACGACTCACTATAGGGAGA(下划线为固定序列,加粗部分为转录生成RNA的起始序列,且可根据待测NTP和检测需要来进行设计和优化,下同)
2)T3 RNA聚合酶,高度特异识别T3启动子,序列如下:
编码链(5'→3'):AATTAACCCTCACTAAAGGGAGA
3)SP6 RNA聚合酶,高度特异识别SP6启动子,序列如下:
编码链(5'→3'):ATTTAGGTGACACTATAGAAGAA
4)K11 RNA聚合酶,高度特异识别K11启动子,序列如下:
编码链(5'→3'):AATTAGGGCACACTATAGGGAGA
5)BA14 RNA聚合酶,高度特异识别BA14启动子,序列如下:
编码链(5'→3'):TAATACGACTCACTAATGCGAGA
所述DNA转录模板与核苷酸之间的碱基互补配对原则是指模板链的碱基与NTP的碱基之间存在的一一对应的关系,即腺嘌呤(adenine,A)一定与尿嘧啶(uracil,U)配对(在DNA中一定与胸腺嘧啶(Thymine,T)配对),鸟嘌呤(Guanine,G)一定与胞嘧啶(Cytosine,C)配对,反之亦然。碱基互补配对原则确保体外转录机器对NTPs的碱基具有特异性识别和选择作用。
所述模板链上的识别位点是指根据待测定NTP的碱基类型和碱基互补配对原则,在模板链上设置与其配对的碱基作为识别位点。该识别位点是人为设置的,其数目和位置均可根据需要调动。与此相反的是非识别位点,指排除识别位点碱基类型的其余三种碱基类型,非识别位点不能与待测定NTP互补配对。模板链上除识别位点之外的其他碱基均为非识别位点。
本发明中识别位点的个数与转录一条RNA产物所消耗的待测NTP的个数呈正相关,进而RNA产物的数量与体系中待测NTP的数量呈正相关,以此为依据对待测NTP进行定量。识别位点的个数至少为1,多则不限,但随着识别位点个数的增多,体系对待测NTP的检测范围和检测限会向高浓度偏移,灵敏度也随之降低。为追求高的灵敏度,本发明中识别位点的典型个数设置为1。另外,识别位点前后的碱基类型及数目可以根据需要来设计和优化,以最大程度的提高转录效率和信号窗口,降低反应阴性溶液的背景信号。
所述转录产物RNA是指在目标NTP和其余三种过量的NTPs存在下,由RNA聚合酶催化的转录反应顺利通过识别位点,将目标NTP分子通过磷酸二酯键合成到转录产物RNA分子内部,并且将模板链上识别位点之后的碱基序列包括能被分子信标识别的信号序列依据碱基互补配对原则而完整转录所得到的RNA链。
所述分子信标是一条呈发夹结构的短链DNA,一端标记荧光基团,另一端标记猝灭基团。荧光基团和猝灭基团选自常用的FRET基团对,如荧光基团FAM和猝灭基团DABCYL或BHQ1或TAMRA,荧光基团ROX和猝灭基团BHQ2等。转录产物RNA的信号序列可通过碱基互补配对原则与分子信标结合,导致分子信标的构象发生变化,由初始茎部结合和荧光基团被猝灭基团猝灭的状态变为茎部打开和荧光基团荧光信号恢复的状态,从而生成实时荧光上升曲线来对RNA聚合酶催化、待测NTP参与的体外转录机器的运转过程进行实时反应。
本发明的定量模式可选用以下任意一种,但不限于以下方法:
(1)根据荧光上升速率或相对速率与待测NTP的浓度或绝对含量建立线性关系。荧光曲线随时间呈现直线上升阶段的斜率值,即为荧光上升速率。相对速率等于含目标物体系的荧光上升速率与不含目标物的空白溶液的荧光上升速率的比值。通过取反应前1分钟内的荧光上升速率或相对速率,可将检测时间极大缩短至1分钟。以下实验结果默认采用此定量方法。
(2)根据荧光强度值与待测NTP的浓度或绝对含量建立线性关系。具体可以根据同一时间点的荧光强度,也可根据荧光平台值。
本发明对待测NTP的检测模式可以分为两类:第一类,每个反应离心管中只检测一种NTP;第二类,每个反应离心管中同时检测两种NTPs。现列举检测思路如下,但不限于以下方案:
(1)每个反应离心管中只检测一种NTP,可同时进行多个反应离心管中不同种类NTPs的检测。
(2)每个反应离心管中同时检测两种NTPs,并可进行任意两种NTPs的组合。这里以ATP与CTP组合为例进行说明:ATP模板链的非识别位点仅由C、A组成,不设置G;与此同时,CTP模板链的非识别位点也仅由C、A组成,不设置T,但两者的信号序列和分子信标的环部设计不相同,荧光基团也用不同种类来分别标记两者的分子信标,以实现对不同模板链的转录产物的分别响应。如此,对于ATP的检测,转录仅消耗GTP、UTP和目标物ATP,不消耗CTP,产生的转录产物RNA仅能识别其相应的分子信标,产生相应的荧光信号,而对CTP的检测毫无影响。同样CTP的检测也不会影响ATP的检测。在双荧光通道的检测模式下,实现同一反应体系中对两种NTPs的同时检测,互不干扰。
下述实施例中所述实验方法,如无特殊说明,均为常规方法;所述试剂和材料,如无特殊说明,均可从商业途径获得。
实施例1<以ATP为待测目标物,比较三种商业RNA聚合酶对体外转录机器的性能影响>
根据图1的原理示意图,以ATP为模型,在其余NTPs过量存在的情况下,向基于RNA聚合酶的体外转录机器中加入ATP时,转录反应将顺利通过识别位点,合成包括待测ATP和后续信号序列的转录产物RNA,该转录产物RNA的信号序列被分子信标的环部识别,使分子信标的构象发生变化而导致荧光基团的信号得以恢复,从而将ATP以1:1的信号模式转换为RNA和分子信标的荧光信号。
为比较三种常见的商业RNA聚合酶对该体外转录机器的性能影响,现选用T7 RNA聚合酶、T3 RNA聚合酶和SP6 RNA聚合酶,分别将模板链和编码链的启动子区域设计为各自的启动子序列,识别位点前后碱基与信号序列完全一致。三种RNA聚合酶各自拥有空白体系和目标物体系,通过综合比较各自空白体系的荧光上升速率、目标物体系的荧光上升速率以及由目标物与空白比值所得的相对速率,荧光上升速率和相对速率均较大者视为性能较优的RNA聚合酶。
检测具体步骤如下:
如无特殊说明,反应体积均为20μL,实验条件均为1x反应缓冲液[100μg/mL BSA,40mM Tris-HCl(pH 7.9),6mM MgCl2,2mM亚精胺,10mM DTT],反应时加入用1x反应缓冲液溶解的预杂交的模板链和编码链(浓度比1:1)、用1x反应缓冲液溶解的预退火的分子信标(终浓度均为200nM),并加入终浓度各0.5mM的其余三种非待测NTPs,另加入RNase inhibitor(终浓度1U/μL)以避免转录产物RNA的降解,最后加入与模板启动子相匹配的RNA聚合酶,在37℃进行温育。以上所述即为空白体系组成。向反应中加入ATP(终浓度为1μM),则为目标物体系。同一反应原则上保证至少三份平行样。
检测时使用的仪器为Rotor-Gene Q实时荧光定量PCR分析仪,采用488nm氩离子激光器光源,一次最多可同时分析96个样品,可检测大量实验样品。荧光检测时的参数设置:Green通道,37℃,5s/cycle,200cycles,即每5s检测一次荧光强度。
模板链与编码链的预杂交、分子信标的预退火条件:90℃90s,70℃90s,50℃90s,37℃180s。
定量步骤:(a)取各反应从起始到1分钟内荧光曲线随时间呈现直线上升阶段的斜率值,即为荧光上升速率。(b)目标物体系与空白体系的荧光上升速率之比,即为相对速率,其中空白体系的相对速率为1。(c)以不同种类的RNA聚合酶为x轴,其目标物体系和空白体系的荧光上升速率为左y轴,相对速率为右y轴,分别做柱状与折线图,如图2所示。
在本实施例中,编码链、模板链、分子信标的序列如下:
T7 RNA聚合酶体系
编码链5’-TAATACGACTCACTATAGGGATTGTTGTGGTGTGTTGGTT-3’
模板链5’-AACCAACACACCACAACAATCCCTATAGTGAGTCGTATTA-3’
T3 RNA聚合酶体系
编码链5’-AATTAACCCTCACTAAAGGGATTGTTGTGGTGTGTTGGTT-3’
模板链5’-AACCAACACACCACAACAATCCCTTTAGTGAGGGTTAATT-3’
SP6 RNA聚合酶体系
编码链5’-ATTTAGGTGACACTATAGGGATTGTTGTGGTGTGTTGGTT-3’
模板链5’-AACCAACACACCACAACAATCCCTATAGTGTCACCTAAAT-3’
以上三类RNA聚合酶的体系均用下述分子信标
分子信标5’-FAM-CCTGGCAACCAACACACCACAACATGCCAGG-DABCYL-3’
(编码链中斜体对应于转录序列,下划线为目标位点,粗体对应于RNA的信号序列可被分子信标识别。模板链中斜体为转录序列,下划线为识别位点。分子信标中下划线为茎部序列,粗体为用于识别RNA信号序列的环部。)
其余NTP组成为:CTP、UTP、GTP,T7 RNA聚合酶、T3 RNA聚合酶和SP6 RNA聚合酶的浓度均为2U/μL。Rotor-Gene Q的Gain设为7。
从图2可以看出,T7 RNA聚合酶的相对速率远大于T3 RNA聚合酶和SP6 RNA聚合酶,即T7 RNA聚合酶的性能最优。因此以下实施例中默认所用的RNA聚合酶为T7 RNA聚合酶。
实施例2<基于RNA聚合酶的体外转录机器中不同NTPs的模板链的序列优化>
在该实施例中,我们首先以ATP为目标物,当改变目标位点T前后的碱基类型时,发现可以显著改变目标体系和空白体系的荧光上升速率,并由此影响相对速率的大小。而相对速率作为信号窗口的指示,其数值越大,则表明信号窗口越大,转录机器的性能更优。在对ATP的模板转录区域序列进行优化时,主要分为三步:(1)固定识别位点及上游序列相同,比较识别位点下游相邻碱基类型;(2)将下游相邻碱基设为最优碱基,改变和比较识别位点上游相邻碱基的类型;(3)将上下游相邻碱基均设为最优碱基,比较转录起始模板碱基C的个数。并且将ATP的检测原理(图1)和上述模板优化的规律,分别扩展至CTP、UTP和GTP的检测及模板转录区域的序列优化(图3)。
检测具体步骤如下:
如无特殊说明,反应体积均为20μL,实验条件均为1x反应缓冲液[100μg/mL BSA,40mM Tris-HCl(pH 7.9),6mM MgCl2,2mM亚精胺,10mM DTT],反应时加入用1x反应缓冲液溶解的预杂交的模板链和编码链(浓度比1:1)、用1x反应缓冲液溶解的预退火的分子信标(终浓度均为200nM),并加入终浓度各0.5mM的其余三种非待测NTPs,另加入RNase inhibitor(终浓度1U/μL)以避免转录产物RNA的降解,最后加入一定浓度的T7 RNA聚合酶,在37℃进行温育。以上所述即为空白体系组成。向反应中加入一定浓度的目标NTP,则为目标物体系。同一反应原则上保证至少三份平行样。
检测时使用的仪器为Rotor-Gene Q实时荧光定量PCR分析仪,采用488nm氩离子激光器光源,一次最多可同时分析96个样品,可检测大量实验样品。荧光检测时的参数设置:Green通道,37℃,5s/cycle,200cycles,即每5s检测一次荧光强度。
模板链与编码链的预杂交、分子信标的预退火条件:90℃90s,70℃90s,50℃90s,37℃180s。
定量步骤:(a)取各反应从起始到1分钟内荧光曲线随时间呈现直线上升阶段的斜率值,即为荧光上升速率。(b)目标物体系与空白体系的荧光上升速率之比,即为相对速率,其中空白体系的相对速率为1。(c)以某一组模板链的含目标物体系的荧光上升速率为参照值,对同一目标NTP的不同模板序列的目标体系和空白体系的荧光上升速率均除以同一实验条件下该参照序列的目标体系的荧光上升速率,由此得到归一化的荧光上升速率;将不同模板序列的相对速率均除以同一条件下该参照序列的相对速率,由此得到归一化的相对速率;(d)以不同目标NTP的模板序列为x轴,其目标物体系和空白体系的归一化的荧光上升速率为左y轴,归一化的相对速率为右y轴,分别做柱状与折线图。
1.ATP的模板序列优化
在该实施例中,作为归一化参照序列的编码链、模板链、分子信标的序列如下:
编码链5’TAATACGACTCACTATAGGGACTGTTGTGGTGTGTTGGTT-3’
模板链5’-AACCAACACACCACAACAGTCCCTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCAACCAACACACCACAACATGCCAGG-DABCYL-3’
(编码链中斜体对应于转录序列,下划线为包含识别位点的优化区域(可变区),粗体对应于RNA的信号序列可被分子信标识别。模板链中斜体为转录序列,下划线为包含识别位点的优化区域(可变区)。分子信标中下划线为茎部序列,粗体为识别RNA信号序列的环部。下同)
其中包含识别位点的模板序列优化区域(可变区)从3’末端起始转录点至5’方向如下:
CCCTG、CCCTA、CCCTC、CCCATG、CCCGTG、CCCCTG、CCCCCTG、CCCCCCTG、CCCCCCCTG(转录起始区连续C序列可以缩写为(C)n,n为C的个数)
ATP的模板序列优化结果如图3(a)和(b)所示,不同的荧光上升速率和相对速率反映了RNA聚合酶对不同碱基类型的不同亲和力及对转录反应动力学和保真性的影响。
(1)当识别位点T的下游相邻碱基为G时具有更大的相对速率,相比之下当下游相邻碱基为A时,目标体系的荧光上升速率较低导致相对速率较低,这表明RNA聚合酶对UTP的亲和力非常弱,对A碱基的亲和力较强,使得RNA聚合酶在模板链上的迁移减缓,从而使后续转录反应的速率降低;而当下游相邻碱基为C时,空白体系的荧光上升速率较大导致相对速率减小,这表明RNA聚合酶对GTP有着非常强的亲和力,与模板链C的亲和作用较弱,RNA聚合酶在模板链上的迁移非常快,使得在没有目标物ATP存在下,识别位点的错误掺入率增加,从而影响了识别位点的保真性,使相对速率降低。
(2)在固定下游相邻碱基为G时,比较识别位点上游相邻碱基发现,C碱基比G和A更优,这是因为RNA聚合酶对模板G和A的亲和力较强,从而使其在模板链上的迁移速率大大降低,与此同时相邻碱基为A时,UTP的掺入会增加识别位点处的错误掺入率,从而使其空白体系的荧光上升速率增加,导致其相对速率比模板G更低。
(3)由于RNA聚合酶对GTP作为起始聚合核苷酸具有非常强的偏好性,因此固定识别位点上下游碱基分别为C和G,比较模板链起始转录区C碱基的个数发现4个连续的C最佳。随着C的继续增加,虽然目标体系的荧光上升速率大大增加,但空白体系的荧光上升速率同时急剧增大,导致相对速率反而减小。
综上所述,ATP的最优模板序列(可变区3’→5’)为CCCCTG。
2.标准品CTP的检测及其标准曲线和检测限
在该实施例中,作为归一化参照序列的编码链、模板链、分子信标的序列如下:
编码链5’-TAATACGACTCACTATAGGGGACATGTTGTGGTGTGTTGGTT-3’
模板链5’-AACCAACACACCACAACATGTCCCCTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCAACCAACACACCACAACATGCCAGG-DABCYL-3’
其中包含识别位点的模板序列优化区域(可变区)从3’末端起始转录点至5’方向如下:
CCCCTGT、CCCCTGA、CCCCTGC、CCCCAGT、CCCCGT、CCCCCTGT(转录起始区连续C序列可以缩写为(C)n,n为C的个数)
CTP的模板序列优化结果如图3(c)所示:
(1)当识别位点G的下游相邻碱基为T时具有更大的相对速率,相比之下当下游相邻碱基为A时,目标体系的荧光上升速率较低导致相对速率较低,这表明RNA聚合酶对UTP的亲和力非常弱,对A碱基的亲和力较强,使得RNA聚合酶在模板链上的迁移减缓,从而使后续转录反应的速率降低;而当下游相邻碱基为C时,空白体系的荧光上升速率较大导致相对速率减小,这表明RNA聚合酶对GTP有着非常强的亲和力,与模板链C的亲和作用较弱,RNA聚合酶在模板链上的迁移非常快,使得在没有目标物CTP存在下,识别位点的错误掺入率增加,从而影响了识别位点的保真性,使相对速率降低。这一点与ATP的模板优化结果非常一致。
(2)在固定下游相邻碱基为T时,比较识别位点上游相邻碱基发现,T碱基比C和A更优,这是因为RNA聚合酶对模板A的亲和力较强,从而使其在模板链上的迁移速率大大降低,与此同时相邻碱基为A时,UTP的掺入会增加识别位点处的错误掺入率,从而使其空白体系的荧光上升速率增加,导致其相对速率比模板G更低。而当相邻碱基为C时,虽然目标体系的荧光上升速率比碱基为A时较大,但由于RNA聚合酶在此位点迁移过快,而使识别位点的错误掺入率有所增加,从而也使相对速率较低。
(3)与ATP的模板优化结果相同,CTP的模板链起始转录区也是4个连续的C碱基最佳。随着C的继续增加,虽然目标体系的荧光上升速率大大增加,但空白体系的荧光上升速率同时急剧增大,导致相对速率反而减小。
综上所述,CTP的最优模板序列(可变区3’→5’)为CCCCTGT。
3.标准品UTP的检测及其标准曲线和检测限
在该实施例中,作为归一化参照序列的编码链、模板链、分子信标的序列如下:
编码链5’-TAATACGACTCACTATAGGGGCTCACAACACCACACAACCAA-3’
模板链5’-TTGGTTGTGTGGTGTTGTGAGCCCCTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCTTGGTTGTGTGGTGTTGTAGCCAGG-DABCYL-3’
其中包含识别位点的模板序列优化区域(可变区)从3’末端起始转录点至5’方向如下:
CCCCGAG、CCCCGAT、CCCCGAC、CCCCTAG、CCCCAG(转录起始区连续C序列可以缩写为(C)n,n为C的个数)
UTP的模板序列优化结果如图3(d)所示,与ATP和CTP所不同的是UTP作为目标NTP时的空白体系荧光上升速率较大,表明UTP的识别位点处本身容易掺入错误碱基,造成较差的保真性。由于不同序列的相对速率较为接近,因此主要通过比较空白体系的荧光上升速率来选择最优的模板序列。
(1)当识别位点A的下游相邻碱基为G时空白体系的荧光上升速率较低,相比之下当下游相邻碱基为T或C时,由于RNA聚合酶在模板链上的迁移速率过快,使得空白体系的荧光上升速率也较大,识别位点处的保真性降低。
(2)在固定下游相邻碱基为G时,比较识别位点上游相邻碱基发现,G碱基比C和T更优,这是因为RNA聚合酶对模板G的亲和力较强,从而使其在模板链上的迁移速率大大降低,也使识别位点处错配的风险降低。而当上游碱基为C和T时,RNA聚合酶的迁移过快,会使识别位点的错误掺入率大大增加,从而也使相对速率较低。
综上所述,UTP的最优模板序列(可变区3’→5’)为CCCCGAG。
4.标准品GTP的检测及其标准曲线和检测限
在该实施例中,作为归一化参照序列的编码链、模板链、分子信标的序列如下:
编码链5’-TAATACGACTCACTATAGGACACAACACCACACAACCAA-3’
模板链5’-TTGGTTGTGTGGTGTTGTGTCCTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCTTGGTTGTGTGGTGTTGTAGCCAGG-DABCYL-3’
其中包含识别位点的模板序列优化区域(可变区)从3’末端起始转录点至5’方向如下:CCT、CCG、CCA、CG、GCT、TCT、ACT
GTP的模板序列优化结果如图3(e)和(f)所示,由于GTP作为目标物时,识别位点为C,为了保证体系的灵敏度,需要降低起始转录区域C即识别位点的个数,如1~2。
(1)当识别位点C的下游相邻碱基为T时空白体系的荧光上升速率较低,相对速率较大,为最优碱基;相比之下当下游相邻碱基为G或A时,由于RNA聚合酶在模板链上的迁移速率过慢,使得目标体系的荧光上升速率也较小,从而相对速率较低。
(2)在固定下游相邻碱基为T时,比较识别位点上游相邻碱基发现,G碱基比C和T更优,此时模板链与RNA聚合酶的结合力强弱成为转录速率的决定因素,当模板起始碱基为G时,RNA聚合酶对模板G的强亲和力使得其与模板链的结合力增强,从而使目标体系的荧光上升速率较大;而模板起始碱基为T和A时,由于RNA聚合酶较弱的亲和力,从而使转录反应和荧光上升速率较低。
(3)尽管起始CG和CCT也有较大的相对速率,但由于转录起始核苷酸并不要求GTP酸酐键的断裂和磷酸二酯键的形成,因此GMP和GDP也可以代替GTP参与转录反应,从而使体系缺乏对GTP的选择性。
综上所述,GTP的最优模板序列(可变区3’→5’)为GCT。
实施例3<基于RNA聚合酶的体外转录机器和优化的模板序列对标准品NTPs的检测及其标准曲线>
根据上述NTPs的转录区域模板序列优化结果,采用各NTPs的最优模板序列进行标准品NTPs的浓度梯度检测并绘制标准曲线,以获得其线性范围和检测限。
检测具体步骤如下:
如无特殊说明,反应体积均为20μL,实验条件均为1x反应缓冲液[100μg/mL BSA,40mM Tris-HCl(pH 7.9),6mM MgCl2,2mM亚精胺,10mM DTT],反应时加入用1x反应缓冲液溶解的预杂交的模板链和编码链(浓度比1:1)、用1x反应缓冲液溶解的预退火的分子信标(终浓度均为200nM),并加入终浓度各0.5mM的其余三种非待测NTP,另加入RNase inhibitor(终浓度1U/μL)以避免转录产物RNA的降解,最终加入一定浓度的T7 RNA聚合酶,在37℃进行温育。以上所述即为反应空白体系组成。向反应中加入一定浓度的待测NTP,则为反应目标体系。同一反应原则上保证至少三份平行样。
检测时使用的仪器为Rotor-Gene Q实时荧光定量PCR分析仪,采用488nm氩离子激光器光源,一次最多可同时分析96个样品,可检测大量实验样品。荧光检测时的参数设置:Green通道,37℃,5s/cycle,200cycles,即每5s检测一次荧光强度。
模板链与编码链的预杂交、分子信标的预退火条件:90℃90s,70℃90s,50℃90s,37℃180s。
定量步骤:(a)取各反应从起始到1分钟内荧光曲线随时间呈现直线上升阶段的斜率值,即为荧光上升速率。空白体系荧光上升速率的平均值加上3倍标准差的数值所对应的最低浓度即为检测限。(b)目标体系与空白体系的荧光上升速率之比,即为相对速率,其中空白体系的相对速率为1。(c)将各目标体系待测NTP的绝对含量或浓度为x轴,其相对速率为y轴,做一次函数关系图。(d)线性拟合,得到标准曲线、线性范围和线性相关系数。
1.标准品ATP的检测及其标准曲线和检测限
在该实施例中,编码链、模板链、分子信标的序列如下:
编码链5’-TAATACGACTCACTATAGGGGACTGTTGTGGTGTGTTGGTT-3’
模板链5’-AACCAACACACCACAACAGTCCCCTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCAACCAACACACCACAACATGCCAGG-DABCYL-3’
(编码链中斜体对应于转录序列,下划线为目标位点,粗体对应于RNA的信号序列可被分子信标识别。模板链中斜体为转录序列,下划线为识别位点。分子信标中下划线为茎部序列,粗体为用于识别RNA信号序列的环部。下同)
体系中分别加入不同绝对含量的ATP(pmol):0,0.02,0.1,0.2,1,2,8,12,20,200。
ATP的标准曲线如图4(a)所示,线性范围是0.02~20pmol(1~1000nM),线性相关系数R2为0.998,检测限低于0.02pmol(1nM)。
2.标准品CTP的检测及其标准曲线和检测限
在该实施例中,编码链、模板链、分子信标的序列如下:
编码链5’-TAATACGACTCACTATAGGGGACATGTTGTGGTGTGTTGGTT-3’
模板链5’-AACCAACACACCACAACATGTCCCCTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCAACCAACACACCACAACATGCCAGG-DABCYL-3’
体系中分别加入不同绝对含量的CTP(pmol):0,0.2,0.4,1,2,10,20,40,80,200。
CTP的标准曲线如图4(b)所示,线性范围是0.2~80pmol(10~4000nM),线性相关系数R2为0.994,检测限低于0.2pmol(10nM)。
3.标准品UTP的检测及其标准曲线和检测限
在该实施例中,编码链、模板链、分子信标的序列如下:
编码链5’-TAATACGACTCACTATAGGGGCTCACAACACCACACAACCAA-3’
模板链5’-TTGGTTGTGTGGTGTTGTGAGCCCTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCTTGGTTGTGTGGTGTTGTAGCCAGG-DABCYL-3’
体系中分别加入不同绝对含量的UTP(pmol):0,0.1,0.2,0.4,1,2,8,16,20,40,80,200。
UTP的标准曲线如图4(c)所示,线性范围是0.1~80pmol(5~4000nM),线性相关系数R2为0.994,检测限低于0.1pmol(5nM)。
4.标准品GTP的检测及其标准曲线和检测限
在该实施例中,编码链、模板链、分子信标的序列如下:
编码链5’-TAATACGACTCACTATACGACACAACACCACACAACCAA-3’
模板链5’-TTGGTTGTGTGGTGTTGTGTCGTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCTTGGTTGTGTGGTGTTGTAGCCAGG-DABCYL-3’
体系中分别加入不同绝对含量的GTP(pmol):0,0.02,0.2,1,2,8,12,20,60,100,200。
GTP的标准曲线如图4(d)所示,线性范围是0.2~200pmol(10~10000nM),线性相关系数R2为0.990,检测限为0.02pmol(1nM)。
实施例4<基于RNA聚合酶的体外转录机器对NTPs检测的选择性>
在该实施例中,由于相对速率将空白体系荧光上升速率的扣除,已经排除了不同碱基类型对目标碱基的干扰。而基于RNA聚合酶对同一碱基类型的不同类核苷酸的区别能力,可能对目标NTP的检测造成干扰的物质是同一碱基类型的dNTP,NDP和NMP。为了证明基于RNA聚合酶的体外转录机器对NTP具有非常好的选择性,向各NTP检测体系中加入与目标NTP等浓度的干扰物质,根据各体系相对速率的大小和目标NTP回收率的大小,验证本发明对NTP检测的选择性,具体步骤如下:
如无特殊说明,反应体积均为20μL,实验条件均为1x反应缓冲液[100μg/mL BSA,40mM Tris-HCl(pH 7.9),6mM MgCl2,2mM亚精胺,10mM DTT],反应时加入预杂交的模板链和编码链、预退火的分子信标(终浓度均为200nM),并加入终浓度各0.5mM的其余三种非待测NTP,另加入RNase inhibitor(终浓度1U/μL)以避免转录产物RNA的降解,最终加入一定浓度的T7 RNA聚合酶,在37℃进行温育。以上所述即为反应空白体系组成。向反应中加入400nM的待测NTP,则为反应目标体系。向反应中加入400nM的干扰物质如dNTP,NDP和NMP,则为反应干扰体系。向反应中同时加入400nM的待测NTP和400nM的干扰物质,则为反应混合体系。同一反应原则上保证至少三份平行样。
检测时使用的仪器为Rotor-Gene Q实时荧光定量PCR分析仪,采用488nm氩离子激光器光源,一次最多可同时分析96个样品,可检测大量实验样品。荧光检测时的参数设置:Green通道,37℃,5s/cycle,200cycles,即每5s检测一次荧光强度。
模板链与编码链的预杂交、分子信标的预退火条件:90℃90s,70℃90s,50℃90s,37℃180s。
数据处理步骤:(a)取各反应从起始到1分钟内荧光曲线随时间呈现直线上升阶段的斜率值,即为荧光上升速率。各反应体系与空白体系的荧光上升速率之比,即为相对速率。(b)反应混合体系与反应目标物体系的荧光上升速率之比,即为待测NTP的回收率。(c)以目标NTP及其干扰物质为x轴,其各体系的相对速率为左y轴,待测NTP的回收率为右y轴,做柱状图。
1.ATP的选择性
在该实施例中,编码链、模板链、分子信标的序列如下:
编码链5’-TAATACGACTCACTATAGGGGACTGTTGTGGTGTGTTGGTT-3’
模板链5’-AACCAACACACCACAACAGTCCCCTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCAACCAACACACCACAACATGCCAGG-DABCYL-3’
各反应体系中分别加入400nM的ATP、dATP、ADP、AMP、ATP+dATP、ATP+ADP、ATP+AMP。
ATP的选择性实验结果如图5(a)所示,dATP、ADP和AMP的相对速率接近于1,ATP的相对速率明显高于三者,回收率均控制在100±10%,证明本发明对ATP具有非常好的选择性。
2.CTP的选择性
在该实施例中,编码链、模板链、分子信标的序列如下:
编码链5’-TAATACGACTCACTATAGGGGACATGTTGTGGTGTGTTGGTT-3’
模板链5’-AACCAACACACCACAACATGTCCCCTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCAACCAACACACCACAACATGCCAGG-DABCYL-3’
各反应体系中分别加入400nM的CTP、dCTP、CDP、CMP、CTP+dCTP、CTP+CDP、CTP+CMP。
CTP的选择性实验结果如图5(b)所示,dCTP、CDP和CMP的相对速率接近于1,CTP的相对速率明显高于三者,回收率均控制在100±10%,证明本发明对CTP具有非常好的选择性。
3.UTP的选择性
在该实施例中,编码链、模板链、分子信标的序列如下:
编码链5’-TAATACGACTCACTATAGGGGCTCACAACACCACACAACCAA-3’
模板链5’-TTGGTTGTGTGGTGTTGTGAGCCCCTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCTTGGTTGTGTGGTGTTGTAGCCAGG-DABCYL-3’
各反应体系中分别加入400nM的UTP、dTTP、dUTP、UDP、UMP、UTP+dTTP、UTP+dUTP、UTP+UDP、UTP+UMP。
UTP的选择性实验结果如图5(c)所示,dTTP、dUTP、UDP和UMP的相对速率接近于1,UTP的相对速率明显高于四者,回收率均控制在100±10%,证明本发明对UTP具有非常好的选择性。
4.GTP的选择性
在该实施例中,编码链、模板链、分子信标的序列如下:
编码链5’-TAATACGACTCACTATACGACACAACACCACACAACCAA-3’
模板链5’-TTGGTTGTGTGGTGTTGTGTCGTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCTTGGTTGTGTGGTGTTGTAGCCAGG-DABCYL-3’
各反应体系中分别加入400nM的GTP、dGTP、GDP、GMP、GTP+dGTP、GTP+GDP、GTP+GMP。
GTP的选择性实验结果如图5(d)所示,dGTP、GDP和GMP的相对速率接近于1,GTP的相对速率明显高于三者,回收率均控制在100±10%,证明本发明对GTP具有非常好的选择性。
实施例5<基于RNA聚合酶的体外转录机器对实际样品中NTPs的检测>
在该实施例中,实际样品可为体外培养的组织样品、细胞裂解液、脑脊液或血清等,既可检测NTPs的正常含量水平、也可用作外界刺激或药物处理后NTPs的含量变化。检测时,根据实施例1做各NTPs的标准曲线和线性拟合关系式,并将实际样品稀释一定倍数使其相对速率落在相应NTPs的线性范围内,根据所得线性拟合的一次函数关系式,将实际样品的相对速率值代入获得相应NTPs的绝对含量或浓度。
样品加标回收率:取两份相同样品,其中一份加入定量的目标NTP的标准物质;两份同时按实施例1进行分析,加标的一份所得的结果减去未加标一份所得的结果,其差值同加入标准物质的理论值之比即为样品加标回收率。
以A549人小非肺癌细胞的细胞裂解液为例,具体实施步骤如下:
(1)细胞培养与裂解液的制备:A549细胞在含有10%胎牛血清、1%青霉素-链霉素的Ham’s F-12K(Kaighn’s)培养液中和37℃、5%CO2的条件下培养。每2-3天传代一次。当细胞处于指数生长期时,用胰酶消化并分散在冰冷PBS中,用TC 10自动细胞计数器和台盼染料对细胞计数和求比活率。然后3000g 4℃离心5min,细胞团用500μL 60%甲醇悬浮,95℃3min,超声30s。提取物离心16000g 4℃5min。用3kDa超滤管在14000g 4℃超滤2h。滤液在真空70℃条件下离心蒸发溶剂1h,所得颗粒再次悬浮于一定体积的无酶水中。存储于-80℃以备检测。
(2)对细胞提取物进行不同稀释倍数的探索,使其落在标准曲线的线性区间内。根据实施例1中所描述的步骤做标准曲线、待测样品、加标样品的检测。这里用104个细胞即可对A549细胞进行准确定量,且检测限低至102~103个细胞。结果如下:
实施例6<基于RNA聚合酶的体外转录机器用于逻辑门的构建>
基于RNA聚合酶的体外转录机器不仅可以用于实际样品中NTPs的检测,还可用于逻辑门的构建。由于其多重分析能力和简便的组装特点,通过改变模板链和编码链以及体系中非目标NTPs的组成,即可将机器转变为不同的逻辑门。
在该实施例中,以ATP和CTP的“与”门和“或”门的构建为代表进行阐述,如图6所示。
空白体系中含有模板链与编码链的双链DNA、分子信标、反应缓冲液、T7 RNA聚合酶、RNAP inhibitor和过量的GTP与UTP。
当ATP或CTP的物质的量大于0.02pmol时,逻辑输入为1,当n小于0.02pmol时,逻辑输入为0;当体系的相对速率大于1.0时,逻辑输出为1,当相对速率小于或等于1.0时,逻辑输出为0。
(1)ATP和CTP的“与”门
在该实施例中,编码链、模板链、分子信标的序列如下:
编码链5’-GATCACTAATACGACTCACTATAGGGGACTGTTGTGGTGTGTTGGTT-3’
模板链5’-AACCAACACACCACAACAGTCCCCTATAGTGAGTCGTATTAGTGATC-3’
分子信标5’-FAM-CCTGGCAACCAACACACCACAACATGCCAGG-DABCYL-3’
ATP和CTP的“与”门所对应的真值表为:只有当ATP和CTP的逻辑输入值均为1时,体系的逻辑输出为1;其余输入值时体系的逻辑输出均为0.
(2)ATP和CTP的“或”门
在该实施例中,编码链、模板链、分子信标的序列如下:
编码链-1 5’-TAATACGACTCACTATAGGGGATTGTTGTGGTGTGTTGGTT-3’
模板链-1 5’-AACCAACACACCACAACAATCCCCTATAGTGAGTCGTATTA-3’
编码链-2 5’-TAATACGACTCACTATAGGGGCTTGTTGTGGTGTGTTGGTT-3’
模板链-2 5’-AACCAACACACCACAACAAGCCCCTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCAACCAACACACCACAACATGCCAGG-DABCYL-3’
ATP和CTP的“或”门所对应的真值表为:只有当ATP和CTP的逻辑输入值均为0时,体系的逻辑输出为0;其余输入值时体系的逻辑输出均为1。
实施例7<基于RNA聚合酶的体外转录机器用于NTP/[NDP+NMP]的实时监测>
本发明还可用于NTP/[NDP+NMP]的实时监测。当将模板链上的识别位点设置在起始转录位点时,由于起始转录位点可以掺入NDP和NMP,其反应效率与NTP一致,此时若反应体系中同时存在与目标NTP具有相同碱基的NDP和NMP时,则所测结果为[NTP+NDP+NMP]的总和。而当将识别位点设置在转录区域中间如优化的模板序列时,只有目标NTP才能参与转录反应,NDP和NMP由于缺少r-磷酸基团而无法参与转录延伸反应,因此所测结果为NTP的值,将[NTP+NDP+NMP]减去NTP即可得[NDP+NMP]的含量,从而可以实时监测实际样品中NTP/[NDP+NMP]的值,由此反映细胞的生理状态、监测治疗效果、揭示功能机制。
在该实施例中,以GTP/[GDP+GMP]的计算为例:
编码链、模板链、分子信标的序列如下
编码链-1 5’-TAATACGACTCACTATACGACACAACACCACACAACCAA-3’
模板链-1 5’-TTGGTTGTGTGGTGTTGTGTCGTATAGTGAGTCGTATTA-3’
编码链-2 5’-TAATACGACTCACTATAGCACAACACCACACAACCAA-3’
模板链-2 5’-TTGGTTGTGTGGTGTTGTGTGCTATAGTGAGTCGTATTA-3’
分子信标5’-FAM-CCTGGCTTGGTTGTGTGGTGTTGTAGCCAGG-DABCYL-3’
空白体系中含有模板链与编码链的双链DNA、分子信标、反应缓冲液、T7 RNA聚合酶、RNAP inhibitor和过量的ATP、CTP与UTP。在上述空白体系中加入标准品GTP,或GTP、GDP和GMP的混合物,或包含有GTP、GDP和GMP的实际样品,即为反应目标体系。
GTP/[GDP+GMP]的定量方法如下:(1)利用编码链-1和模板链-1,参照实施例3中所述步骤绘制GTP的标准曲线,并参照实施例5中所述由标准曲线测得样品混合体系中GTP的含量;(2)利用编码链-2和模板链-2参照实施例3中所述步骤绘制GTP的标准曲线,并参照实施例5中所述由标准曲线测得样品混合体系中[GTP+GDP+GMP]的总量;(3)将计算所得[GTP+GDP+GMP]的总量减去计算所得GTP的含量,即得[GDP+GMP]的总量,从而可获得GTP/[GDP+GMP]的结果。
综上所述,本发明基于RNA聚合酶的体外转录机器为四种不同的NTP提供了一种超快速、操作简单、定量准确、高选择性、高灵敏度、高通量、低成本的检测方法,是目前唯一一种兼具以上优点的方法,且易于开发试剂盒,极大地方便了研究应用和临床检测,具有非常大的科学意义和商业价值。
- 还没有人留言评论。精彩留言会获得点赞!
- 检测血清中HSP60 mRNA转录水平的RT-PCR引物、评估高血糖人群并发肠癌易感性的试剂 ...的制作方法
- 检测血清中NOX1 mRNA转录水平的RT-PCR引物、评估高血糖人群并发肠癌易感性的试剂盒 ...的制作方法
- 一种新型聚合酶链反应微流体芯片控制系统及其制备方法
- 一种基于聚合酶链式反应产物测序序列分型的实现方法和系统的制作方法
- 一种聚合酶链式反应基因扩展仪的校准系统的制作方法
- 一种基于led的数字聚合酶链式反应pcr多窗口多路检测方法
- 一种循环肿瘤细胞的富集方法
- 一种实时荧光rt-pcr检测猪流感病毒的试剂盒的制作方法
- 实时荧光定量rt-pcr检测人细胞角蛋白19试剂盒的制作方法
- 实时荧光定量逆转录聚合酶链反应检测鸡高迁移率族蛋白b1的试剂盒的制作方法