多重实时荧光PCR检测Cr(VI)还原复合菌群六种功能基因的方法与流程
本发明涉及微生物检测技术领域,具体地说,涉及多重实时荧光PCR检测Cr(VI)还原复合菌群六种功能基因的方法。
背景技术:
在重金属污染中Cr(VI)因其毒性大、氧化性强以及致癌性高,对人类和动植物的生命安全造成了非常大的威胁,其中Cr(VI)土壤污染问题最突出。据统计,我国受Cr(VI)严重污染的土壤面积就高达500万平方米,污染土方量约1500万立方。因此,对我国Cr(VI)污染土壤进行合理有效的修复已经迫在眉睫。
目前微生物还原Cr(VI)成为Cr(VI)污染土壤修复的热点。以Pannonibacter phragmitetus BB为主,Shewanella oneidensis MR-1和Pseudomonas putida F1的复合Cr(VI)还原菌群可以克服温度、pH、有机污染物和重金属等诸多环境因素的影响,增强还原群落刚性,提高还原效率,是目前较为高效的Cr(VI)土壤的修复方法之一。但是,传统方法在复合菌群中无法检测单一菌株的生理生长状态和Cr(VI)还原基因的表达,严重阻碍了菌群Cr(VI)还原协同机理的研究以及中试和场地微生物修复工程中菌群配方的调控。因此,亟需开发一种新的基于核酸水平的检测方法。
对于复合菌群中微生物,Naoki M.等(Fukuma N M,Koike S,Kobayashi Y.Monitoring of gene expression in Fibrobacter succinogenes S85 under the co-culture with non-fibrolytic ruminal bacteria.[J].Archives of Microbiology,2015,197(2):269-276.)采用16s rDNA的拷贝数和16s rRNA/rDNA的比值分别表示瘤胃中复合微生物的生长状况与代谢活性。针对基因表达的检测,常规的方法主要有Northern blotting、in situ hydridisation、RNAse protection assays、cDNA arrays和reverse transcription real-time polymerase chain reaction(RT-qPCR)。Northern blotting虽然可检测目的RNA的大小,但其操作繁琐,耗时耗力;in situ hydridisation操作复杂;RNAse protection assays主要适用于绘制转录起始和终止位点以及内含子/外显子边界,以及用于区分相似大小的相关mRNA,对于表达的定量并不适用;cDNA arrays成本高,适用性有限。以上技术还有一个共同的缺点:敏感性低,对于微量的mRNA无法有效的检测。RT-qPCR是用于检测目的基因表达量的最敏感和最灵活的方法,可用于比较不同样本群体中mRNA的水平检测,表征mRNA表达模式,区分密切相关的mRNA以及分析RNA结构。但是针对单一基因的RT-qPCR,一次只能检测一种基因的表达,同时又存在操作繁琐、费时费力等缺点,尤其是需要同时检测多个基因的情况时。为了探明菌株之间的相互影响,必须要涉及到同时检测多个基因,多重RT-qPCR技术可以实现同时检测多个基因,同时具有高特异性和敏感性、检测时间短、检测成本低等优点,是一种较为理想的检测方法。
本发明选择以下六种功能基因作为目的基因:在单一菌株情况下Cr(VI)还原基因HcrR的表达使Pannonibacter phragmitetus BB有很好的Cr(VI)还原能力,因此,可以应用HcrR作为核酸标签,表征菌株Cr(VI)还原效率。同时,核蛋白体核糖核酸16s亚基基因16s rDNA(BB)为该菌株生命必须基因,因此可用作表征该菌株生理生长状态的标签。Shewanella oneidensis MR-1菌株同样能够高效还原Cr(VI),其细胞色素酶MtrC和OmcA等协同还原Cr(VI),但剔除OmcA相关基因对还原效果影响更大。因此,本发明将OmcA作为该菌株Cr(VI)还原效率表征标签。同样,16s rDNA(MR-1)作表征Shewanella oneidensis MR-1生理生长状态的标签。Pseudomonas putida F1菌株也对Cr(VI)具有一定还原作用,其ChrR酶可以有效地还原Cr(VI),因此本发明将基因ChrR作为Cr(VI)还原效率表征的标签,以16s rDNA(F1)作表征Pseudomonas putida F1生理生长状态的标签。以此开发一种实时荧光PCR技术同时检测Cr(VI)还原复合菌群六种功能基因表达的方法,有助于提高基因检测水平,在微生物联合修复Cr(VI)污染土壤的研究、中试和工程中具有广阔的应用前景。
技术实现要素:
本发明的目的是提供一种利用多重实时荧光定量PCR技术检测Cr(VI)还原复合菌群六种功能基因的方法。
为了实现本发明目的,本发明的用于多重实时荧光PCR检测Cr(VI)还原复合菌群六种功能基因的引物组合,包括:
用于扩增保藏编号为CGMCC No.3052的Pannonibacter phragmitetus BB菌的Cr(VI)还原功能基因HcrR及其核蛋白体核糖核酸16s亚基基因16s rDNA(BB)的引物,用于扩增保藏编号为ATCC No.700550的Shewanella oneidensis MR-1菌的Cr(VI)还原酶基因OmcA及其核蛋白体核糖核酸16s亚基基因16s rDNA(MR-1)的引物,用于扩增保藏编号为ATCC No.700007的Pseudomonas putida F1菌的Cr(VI)还原酶基因ChrR及其核蛋白体核糖核酸16s亚基基因16s rDNA(F1)的引物;
引物序列如下:
HcrR-F:5'-GGGACAACTACACCGCAGAC-3'
HcrR-R:5'-AGCCAGAGCGATGCCGAA-3'
OmcA-F:5'-ATTGGCTATGGCGGTAAGGTA-3'
OmcA-R:5'-AACAGATCGGCATTAGCAGGT-3'
ChrR-F:5'-CAAGCTCAATGACAAGACG-3'
ChrR-R:5'-GCCCTGTTCAACTTCACCCAT-3'
16s rDNA(BB)-F:5'-TTCACAACACGAGCTGACGA-3'
16s rDNA(BB)-R:5'-CAACGCGAAGAACCTTACCT-3'
16s rDNA(MR-1)-F:5'-ACGTGCTACAATGGCGAGTA-3'
16s rDNA(MR-1)-R:5'-TTCATGGAGTCGAGTTGCAG-3'
16s rDNA(F1)-F:5'-CGCGTGTGTGAAGAAGGTCT-3'
16s rDNA(F1)-R:5'-CACAGAGTTAGCCGGTGCTT-3'。
本发明中,P.phragmitetus BB为申请人前期从铬渣堆场土壤中分离得到,该菌现保藏于中国微生物菌种保藏管理委员会普通微生物中心,地址北京市朝阳区北辰西路1号院3号,中国科学院微生物研究所,邮编100101,保藏编号CGMCC No.3052,保藏日期2009年5月5日。
本发明还提供含有所述引物组合的用于多重实时荧光PCR检测Cr(VI)还原复合菌群六种功能基因的试剂盒。
本发明涉及的试剂如下:荧光检测PCR反应液,阴性对照品,阳性对照品,定量PCR标准品;
①所述荧光检测PCR反应液包含FastStart Essential DNA Green Master(Roche,Germany)、六种目的功能基因的引物;
②所述阴性对照品为无RNase酶的水;
③所述阳性对照品:分别携带HcrR、16s rDNA(BB)、OmcA、16s rDNA(MR-1)、ChrR、16s rDNA(F1)六种基因的pGM-Simple-T重组质粒;
④所述检测PCR标准品由以下6种标准品组成:
HcrR标准品、16s rDNA(BB)标准品、OmcA标准品、16s rDNA(MR-1)标准品、ChrR标准品、16s rDNA(F1)标准品;上述六种基因标准品的序列分别如SEQ ID NO:13-18所示。
本发明还提供一种多重实时荧光PCR检测Cr(VI)还原复合菌群六种功能基因的方法,该方法基于多重实时荧光PCR检测技术,利用所述引物组合或试剂盒对Cr(VI)还原复合菌群六种功能基因进行多重实时荧光PCR检测。
前述的方法包含两个PCR反应体系,其中一个体系用于检测Pannonibacter phragmitetus BB菌的Cr(VI)还原功能基因HcrR、Shewanella oneidensis MR-1菌的Cr(VI)还原酶基因OmcA及Pseudomonas putida F1菌的Cr(VI)还原酶基因ChrR,另一个体系用于检测上述三种菌的核蛋白体核糖核酸16s亚基基因。
前述方法包括以下步骤:
1)提取待测样品的总RNA,反转录成cDNA;
2)以上述cDNA为模板,分两管进行实时荧光定量PCR扩增;
3)分析PCR扩增产物。
步骤2)其中一个管的PCR扩增体系为:2×FastStart Essential DNA Green Master 25μL,HcrR、OmcA、ChrR各基因上下游引物的终浓度分别为0.05μM、0.2μM、0.2μM,cDNA模板1μL,RNase/DNase Free dH2O至总体积50μL;
另一个管的PCR扩增体系为:2×FastStart Essential DNA Green Master 25μL,16s rDNA(BB)、16s rDNA(MR-1)、16s rDNA(F1)各基因上下游引物的终浓度分别为0.2μM、0.1μM、0.2μM,cDNA模板1μL,RNase/DNase Free dH2O至总体积50μL。
步骤2)的PCR扩增程序为:95℃预变性600s;95℃变性10s,58℃退火10s,72℃延伸15s,35个循环;熔解曲线程序为初始阶段:60℃60s,最终阶段:60℃以0.1℃/s的变化率上升到90℃,从60℃开始收集荧光信号。
步骤3)根据目的基因的扩增产物在熔解曲线中对应的峰值高低及有无来判定待测样品;如果目的基因相应扩增产物产生特异性熔解峰,即对应的Tm值与理论Tm值一致,则表明待测样品中含有该基因。
HcrR的理论Tm值为87.67±0.26℃、OmcA的理论Tm值为84.79±0.12℃、ChrR的理论Tm值为82.51±0.22℃、16s rDNA(BB)的理论Tm值为83.46±0.21℃、16s rDNA(MR-1)的理论Tm值为86.50±0.19℃、16s rDNA(F1)的理论Tm值为80.47±0.23℃。
本发明方法的具体步骤可如下:
检测时根据实际情况设立阳性对照和阴性对照。
DNA标准品用无菌双蒸水稀释为1.0×101~1.0×107copies/μL。
总RNA的提取:利用试剂盒RNAprep Pure Cell/Bacteria Kit(天根生化科技(北京)有限公司),从过夜培养的菌液中提取细菌总RNA。
逆转录反应:在RNase-Free的0.2mL PCR管中依次加入上一步骤中制备的总RNA模板1μL,使用TIANScript RT kit(天根(北京))进行RT反应。待反转录反应结束后,所得反应液即为cDNA模板。
核酸的检测(多重荧光PCR反应):取1~1.5μL待测cDNA为模板进行多重荧光PCR反应,其中Cr(VI)还原基因的反应体系和16srDNA的反应体系均为2×FastStart Essential DNA Green Master 25μL,HcrR、16s rDNA(BB)、OmcA、16s rDNA(MR-1)、ChrR、16s rDNA(F1)各基因上下游引物的终浓度分别为0.05μM、0.2μM、0.2μM、0.1μM、0.2μM、0.2μM,cDNA模板1μL,RNase/DNase Free dH2O至总体积50μL;反应参数为95℃预变性600s,95℃变性10s,58℃退火10s,72℃延伸15s,35个循环;熔解曲线程序为初始阶段:60℃60s,最终阶段:60℃以0.1℃/s的变化率上升到90℃,从60℃开始收集荧光信号。
标准曲线的绘制:分别取目的基因的重组质粒系列标准品(1×101~1×107copies/μL)进行实时荧光检测PCR反应,其中反应体系均为2×FastStart Essential DNA Green Master 10μL,某特定基因上下游引物分别1μL(终浓度0.5μM),系列标准品模板1μL,RNase/DNase Free dH2O至总体积20μL;反应参数为95℃预变性600s,95℃变性10s,58℃退火10s,72℃延伸15s,35个循环。扩增完成后,以Cq值为纵坐标,log10(q)(q为标准品系列浓度)为横坐标,Cq值与log10(q)呈线性关系,制作标准曲线。
荧光检测结果报告:
通过目的基因的扩增产物在熔解曲线中对应的峰值高低及有无来判定检测样品。如果目的基因相应扩增产物产生特异性熔解峰,即对应的Tm值(理论Tm值HcrR为87.67±0.26℃、OmcA为84.79±0.12℃、ChrR为82.51±0.22℃、16s rDNA(BB)为83.46±0.21℃、16s rDNA(MR-1)为86.50±0.19℃、16s rDNA(F1)为80.47±0.23℃),如果两个值具有一致性,则检测结果对应于该基因扩增产物存在,即待测样品中有该基因的存在或者该基因的表达存在。
如果检测结果为基因存在或表达,根据所获得的标准曲线及待测样品Cq值,计算待测基因的模板量或mRNA初始量,即待测目的基因的拷贝数或表达量。
本发明具有以下优点:
利用实时荧光检测PCR技术,采用六种特定基因的特异性引物,开发研制出一种利用实时荧光PCR技术同时检测来自三株菌株的六种特定基因表达的方法。本发明通过两个PCR反应可以从样品中同时检测出六种基因的表达情况,比传统的普通PCR和单重荧光PCR方法更简便快捷,并且能够对目的基因的表达情况进行实时准确检测。本发明提供的试剂盒可以快速检测三种Cr(VI)还原菌的还原基因表达情况及16s rRNA基因表达情况,确定外部环境改变对三种Cr(VI)还原菌的Cr(VI)还原功能和生长状况的影响。本发明提供了一种操作简单、快捷,成本低廉的多重荧光PCR技术检测六种功能基因表达动态变化的方法,检测方法灵敏性和特异性高,检测结果准确。
附图说明
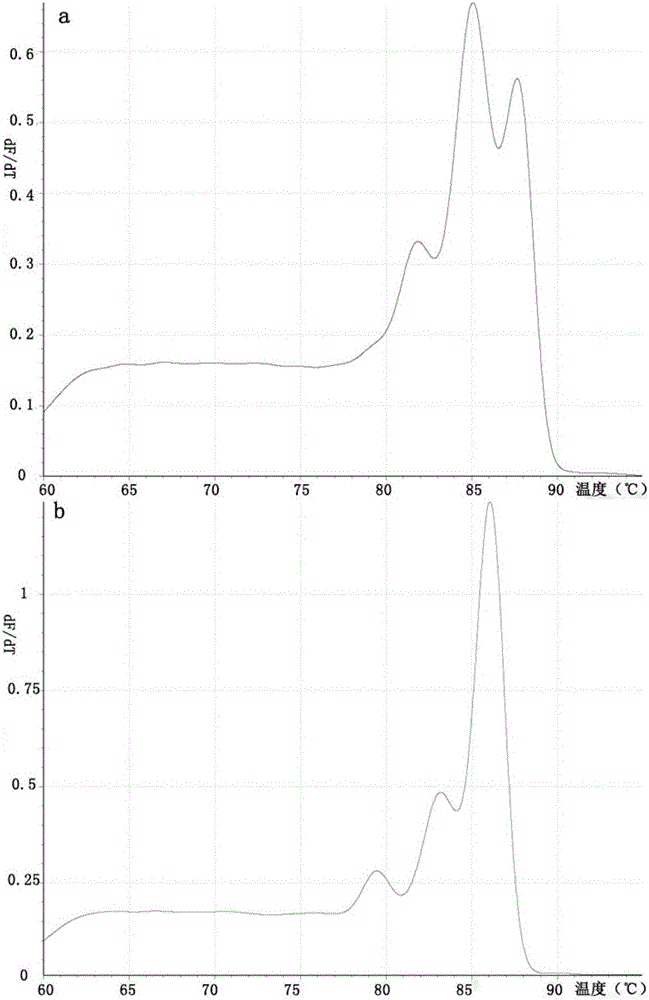
图1为本发明实施例2中六种基因多重PCR体系中的特异性熔解峰。其中:a为三种Cr(VI)还原基因在多重PCR体系中的特异性熔解峰,从左至右依次为:ChrR、OmcA、HcrR;b为16s rDNA在多重PCR体系中的特异性熔解峰,从左至右依次为:16s rDNA(F1)、16s rDNA(BB)、16s rDNA(MR-1)。
图2为本发明实施例5中含有六种基因(HcrR、OmcA、ChrR、16s rDNA(BB)、16s rDNA(MR-1)、16s rDNA(F1))的核酸样品混合进行多重实时荧光检测PCR检测结果。其中:a为Cr(VI)还原基因检测图,从左至右依次是ChrR、OmcA、HcrR;b为16s rDNA检测图,从左至右依次为:16s rDNA(F1)、16s rDNA(BB)、16s rDNA(MR-1)。
图3为本发明实施例6中成功表达目的基因的样本定量检测结果。
具体实施方式
以下实施例用于说明本发明,但不用来限制本发明的范围。若未特别指明,实施例均按照常规实验条件,如Sambrook等分子克隆实验手册(Sambrook J&Russell DW,Molecular Cloning:a Laboratory Manual,2001),或按照制造厂商说明书建议的条件。
实施例1质粒标准品的制备
第一步:引物设计与合成
按照基因的DNA序列设计相应的引物,所设计的HcrR、OmcA、ChrR、16s rDNA(BB)、16s rDNA(MR-1)、16s rDNA(F1)六种基因普通PCR引物(表1)由上海生工生物公司合成,合成量为2OD。
表1本发明中所涉及的普通PCR引物
第二步:细菌总RNA提取
将含有特定基因的三种菌株培养液取1mL置于1.5mL离心管中,利用RNAprep Pure Cell/Bacteria Kit(天根(北京))提取得到细菌总RNA。
第三步:反转录合成cDNA
反转录反应:在RNase-Free的0.2mL PCR管仲依次加入上一步骤中制备的总RNA模板1μL,按照TIANScript RT Kit(天根(北京))的操作说明进行RT反应,待反转录反应结束后,所得反应液即为cDNA模板。
第四步:普通PCR扩增
以第三步中合成的cDNA为模板,利用第一步中合成的六种基因标准品引物分别进行PCR扩增。普通PCR反应体系为2×Taq PCR MasterMix 12.5μL,上下游引物各1μL(终浓度0.4μM),cDNA模板1μL,dd H2O至总体积25μL。反应参数为:95℃预变性5min,95℃变性30s,58℃退火30s,72℃延伸42s,72℃延伸7min。反应在PCR扩增仪(Eppendorf,Germany)中进行。
第五步:质粒标准品制备
将第四步获得的扩增产物进行琼脂糖凝胶电泳,按照DNA Gel Extraction Kit(天根(北京))操作回收目的片段,将回收产物连接到质粒载体pGM-Simple-T上,通过大肠杆菌DH5α感受态转化,涂布到含有氨苄青霉素(100μg/mL)的固体平板上,挑选阳性克隆子(白色菌落)接种到含100μg/mL氨苄青霉素的3-5mL LB液体培养基中培养过夜,取菌液提取质粒(具体操作步骤按E.Z.N.A.TM Plasmid Mini Kits Ⅰ(OMAGA,America)说明书)进行鉴定,重组质粒序列委托上海生工生物公司进行序列测定。利用Nanodrop ND-1000检测重组质粒浓度为167.1ng/μL、81.7ng/μL、68.3ng/μL、149.7ng/μL、75.1ng/μL、144.2ng/μL,分别为对应HcrR、OmcA、ChrR、16s rDNA(BB)、16srDNA(MR-1)、16s rDNA(F1),按照如下经验公式转化为拷贝数后,以无菌双蒸水将重组质粒按照1.0×107~1.0×101copies/μL 10倍梯度稀释制备质粒标准品。
实施例2多重实时荧光PCR检测六种特定基因的特异性实验
单个基因在多重体系中的特异性检测:
按照美国罗氏公司LightCycler Nano 32孔荧光检测聚合酶链反应(PCR)仪反应体系,对于Cr(VI)还原基因配制4份相同的多重PCR反应液,分别单独加入1μL的已知量的HcrR、OmcA、ChrR的标准品质粒,设立一份阴性对照;对于16s rDNA配制4份相同的多重PCR反应液,分别单独加入1μL的已知量的16s rDNA(BB)、16s rDNA(MR-1)、16s rDNA(F1)的标准品质粒,设立一份阴性对照。
Cr(VI)还原基因反应体系为2×FastStart Essential DNA Green Master 25μL,HcrR、OmcA、ChrR各基因上下游引物的终浓度分别为0.05μM、0.2μM、0.2μM,cDNA模板1μL,RNase/DNase Free dH2O至总体积50μL;16s rDNA反应体系为2×FastStart Essential DNA Green Master 25μL,16s rDNA(BB)、16s rDNA(MR-1)、16s rDNA(F1)各基因上下游引物的终浓度分别为0.2μM、0.1μM、0.2μM,cDNA模板1μL,RNase/DNase Free dH2O至总体积50μL。反应程序为95℃预变性600s,95℃变性10s,58℃退火10s,72℃延伸15s,35个循环;熔解曲线程序为初始阶段:60℃60s,最终阶段:60℃以0.1℃/s的变化率上升到90℃,从60℃开始收集荧光信号。
同时进行六种基因的多重实时荧光检测PCR特异性检测试验。
特异性实验结果显示:在两个多重反应体系中单独加入(六种基因)的质粒标准品进行实时荧光检测PCR反应,每种基因的扩增片段在其相应目的片段Tm值位置都有特异性目的峰的产生(图1),可见本发明中构建的多重实时荧光检测PCR反应体系特异性较好。各个基因的Tm值范围HcrR为87.67±0.26℃、OmcA为84.79±0.12℃、ChrR为82.51±0.22℃、16s rDNA(BB)为83.46±0.21℃、6s rDNA(MR-1)为86.50±0.19℃、16s rDNA(F1)为80.47±0.23℃。
实施例3多重实时荧光PCR检测六种特定基因的稳定性实验
将六种基因分别进行如下操作:
取3份已知的质粒标准品和一份阴性对照品(无RNase酶的水),分别进行批内、批间重复试验。批内重复试验是将3份样品同时进行实时荧光检测PCR实验;批间重复试验在3次不同的时间(间隔3天)进行实时荧光检测PCR实验。几种基因的批内、批间重复试验的平均变异系数CV均小于1%。
实验结果表明,本发明的检测方法具有良好的稳定性。
实施例4多重实时荧光PCR检测六种特定基因的敏感性实验
以10倍系列稀释的六种基因质粒标准品作为模板,每个稀释度设立3个重复,分别进行实时荧光检测PCR、普通PCR检测敏感度,比较敏感性。
实验结果表明,本发明可检测出50copies/μL的基因表达量,而常规PCR只能检测到1×104copies/μL的基因表达量。可见,本发明检测基因表达的敏感性感高于常规PCR。
实施例5多重实时荧光PCR检测技术的应用
第一步:细菌总RNA提取
总RNA提取:将含有HcrR、16s rDNA(BB)、OmcA、16s rDNA(MR-1)、ChrR、16s rDNA(F1)六种基因的Pannonibacter phragmitetus BB、Shewanella oneidensis MR-1和Pseudomonas putida F1菌株分别在含有Cr(VI)的LB培养基中过夜培养,利用试剂盒RNAprep Pure Cell/Bacteria Kit(天根生化科技(北京)有限公司)提取细菌总RNA。
第二步:反转录合成cDNA
逆转录反应:在RNase-Free的0.2mL PCR管仲依次加入上一步骤中制备的总RNA模板1μL,使用TIANScript RT kit(天根(北京))进行RT反应。待反转录反应结束后,所得反应液即为cDNA模板。
第三步:多重荧光检测PCR扩增
多重荧光检测PCR反应体系(总反应体积50μL):包括第三步合成的cDNA和第一步中的基因引物,最后加RNase/DNase Free dH2O至反应体系总体积50μL。同时设一阳性对照(以质粒标准品为模板)和一阴性对照(无RNase酶的水)。Cr(VI)还原基因的反应体系和16s rDNA的反应体系均为2×FastStart Essential DNA Green Master 25μL,HcrR、16s rDNA(BB)、OmcA、16s rDNA(MR-1)、ChrR、16s rDNA(F1)各基因上下游引物的终浓度分别为0.05μM、0.2μM、0.2μM、0.1μM、0.2μM、0.2μM,cDNA模板1μL,RNase/DNase Free dH2O至总体积50μL;反应参数为95℃预变性600s,95℃变性10s,58℃退火10s,72℃延伸15s,35个循环;熔解曲线程序为初始阶段:60℃60s,最终阶段:60℃以0.1℃/s的变化率上升到90℃,从60℃开始收集荧光信号。
第四步:检测结果判定
根据LightCycler Nano 32孔荧光检测聚合酶链反应(PCR)仪所自动生成的熔解曲线图(图2),进行扩增结果判定。
Tm值范围:HcrR为87.67±0.26℃、OmcA为84.79±0.12℃、ChrR为82.51±0.22℃、16s rDNA(BB)为83.46±0.21℃、6s rDNA(MR-1)为86.50±0.19℃、16s rDNA(F1)为80.47±0.23℃。
结果显示,本发明能很好地同时通过PCR扩增出对应的六种基因扩增产物,进而确定特定基因的表达状况,并通过熔解曲线峰不同,将六种产物明显区分开来,阴性对照无非特异熔解峰,证明测定结果可靠。
从图2可以得出以下结论:六种基因同时通过两个多重实时荧光PCR反应,扩增出相应目的产物,并能在熔解曲线上,通过Tm值得不同,将各种基因有效区分开来,实验结果与阳性对照品产生的熔解曲线很吻合,而阴性对照熔解曲线上没有特异性熔解峰,可以很直观通过熔解曲线来判定实验结果。证明所检测的基因为对应的目的基因。
实施例6对三种菌株联合培养的六种特定基因的检测及准确定量分析
第一步:样品准备
将Pannonibacter phragmitetus BB、Shewanella oneidensis MR-1和Pseudomonas putida F1接种到同一LB培养基(含有Cr(VI))中联合过夜培养得混合菌液。
第二步:细菌总RNA提取
总RNA提取:将过夜培养的菌液,利用试剂盒RNAprep Pure Cell/Bacteria Kit(天根生化科技(北京)有限公司)提取细菌总RNA。
第三步:反转录合成cDNA
逆转录反应:在RNase-Free的0.2mL PCR管仲依次加入上一步骤中制备的总RNA模板1μL,使用TIANScript RT kit(天根(北京))进行RT反应。待反转录反应结束后,所得反应液即为cDNA模板。
第四步:普通PCR检测和多重荧光检测PCR扩增
1、普通PCR检测:
以上一步骤中合成的cDNA为模板,利用第一步中合成的六种基因标准品引物分别进行PCR扩增。普通PCR反应体系为2×Taq PCR MasterMix 12.5μL,上下游引物各1μL(终浓度0.4μM),cDNA模板1μL,dd H2O至总体积25μL。反应参数为:95℃预变性5min,95℃变性30s,58℃退火30s,72℃延伸42s,72℃延伸7min。反应在PCR扩增仪(Eppendorf,Germany)中进行。
2、多重荧光检测PCR过程同实施例5中的第三步。
第五步:检测结果判定
普通PCR结果通过琼脂糖电泳进行检测;多重荧光PCR检测结果同实施例5中第四步。
普通PCR法检测与本发明检测结果如下:普通PCR琼脂糖凝胶电泳结果中,HcrR、MtrC和16s rDNA(F1)这三种基因的条带模糊不清。多重荧光PCR检测中均扩增出相应目的产物,并能在熔解曲线上,通过Tm值得不同,将各种基因有效区分开来,实验结果与阳性对照品产生的熔解曲线很吻合,而阴性对照熔解曲线上没有特异性熔解峰,可以很直观通过熔解曲线来判定实验结果。
本发明检测效果明显好于普通PCR检测效果,可以清晰地检出各个基因,减少二次PCR验证及并不需要PCR扩增后续处理,大大节约时间,提高工作效率。
第六步:对检测结果进行准确定量
分别取HcrR、16s rDNA(BB)、OmcA、16s rDNA(MR-1)、ChrR、16s rDNA(F1)重组质粒系列标准品1.0×107~1.0×101copies/μL进行单重实时PCR反应检测绘制标准曲线,其中反应体系为2×FastStart Essential DNA Green Master 10μL,某特定基因上下游引物分别1μL(终浓度0.5μM),系列标准品模板1μL,RNase/DNase Free dH2O至总体积20μL;反应程序为95℃预变性600s,95℃变性10s,58℃退火10s,72℃延伸15s,35个循环。
扩增完成后,以Cq值为纵坐标,log10(q)(q为标准品系列浓度)为横坐标,Cq值与log10(q)呈线性关系,制作标准曲线。根据所获得的标准曲线及待测样品Cq值,计算待测样品copies/μL。通过结果分析所测样品中Cr(VI)还原基因HcrR、OmcA、ChrR的cDNA大多都在105~107copies/μL,16s rDNA(BB)、16s rDNA(MR-1)、16s rDNA(F1)的cDNA均在108~109copies/μL。其中OmcA阳性样本准确定向见图3,其标准曲线上的Cq值为19.823,通过Cq值与log10(q)(q为标准品系列浓度)的线性关系,计算出的cDNA的拷贝数为104.864copies/μL左右。
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之做一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
序列表
<110> 中南大学
<120> 多重实时荧光PCR检测Cr(VI)还原复合菌群六种功能基因的方法
<130> KHP171110622.1
<160> 18
<170> PatentIn version 3.3
<210> 1
<211> 20
<212> DNA
<213> 人工序列
<400> 1
ctgcagccgt tcgaggttat 20
<210> 2
<211> 20
<212> DNA
<213> 人工序列
<400> 2
aatggtgacg gaccgcatac 20
<210> 3
<211> 21
<212> DNA
<213> 人工序列
<400> 3
ccagaatcag gcaatagcat t 21
<210> 4
<211> 21
<212> DNA
<213> 人工序列
<400> 4
aatgcaaagc cacgatcact a 21
<210> 5
<211> 21
<212> DNA
<213> 人工序列
<400> 5
cagtgatgcg gtgttgttcg t 21
<210> 6
<211> 21
<212> DNA
<213> 人工序列
<400> 6
tcagaccgcc ctgttcaact t 21
<210> 7
<211> 20
<212> DNA
<213> 人工序列
<400> 7
gctggaaacg gctgctaata 20
<210> 8
<211> 21
<212> DNA
<213> 人工序列
<400> 8
aaaggcagtt ccgaggttga g 21
<210> 9
<211> 21
<212> DNA
<213> 人工序列
<400> 9
agtacggccg caaggttaaa a 21
<210> 10
<211> 21
<212> DNA
<213> 人工序列
<400> 10
cccacttctt ttgcagccca c 21
<210> 11
<211> 20
<212> DNA
<213> 人工序列
<400> 11
ctaataccgc atacgtccta 20
<210> 12
<211> 21
<212> DNA
<213> 人工序列
<400> 12
caccaccctc taccgtactc t 21
<210> 13
<211> 413
<212> DNA
<213> 人工序列
<400> 13
ctgcagccgt tcgaggttat cgtcgtgacc aatccggaag tccgggcgaa aattcaggaa60
attgcctgga accaggcgca ggtgaccgaa gggtcgcacc tgctcgtgtt tgcggcctgg120
gacaactaca ccgcagaccg catcaacatg atgttcgacc tcgtcaacga ggaacgcggc180
ttcaccagcg aaggctggga aaactaccgg cagatgctgc tcaacaccta tgtgccgcgc240
gatgccgaga ccaactatca gcacgccgcc cgtcaggcct atatcgcctt cggcatcgct300
ctggctgctg cggccttcga aggcgttgat tccaccccga tggaaggttt tgacgcggac360
aagctcgacg agattctggg cctgcgcgcc aagggtatgc ggtccgtcac cat413
<210> 14
<211> 443
<212> DNA
<213> 人工序列
<400> 14
gctggaaacg gctgctaata ccgcatgcgc cctacggggg aaagatttat cgccgaagga60
ttggcccgcg tctgattagc tagttggtga ggtaatggct caccaaggcg acgatcagta120
gctggtctga gaggatgatc agccacactg ggactgagac acggcccaga ctcctacggg180
aggcagcagt ggggaatatt ggacaatggg cgcaagcctg atccagccat gccgcgtgag240
tgatgaaggc cctagggttg taaagctctt tcagcgagga ggataatgac gttactcgca300
gaagaagccc cggctaactt cgtgccagca gccgcggtaa tacgaagggg gctagcgttg360
ttcggaatca ctgggcgtaa agcgcacgta ggcggacttt taagtcaggg gtgaaatccc420
ggggctcaac ctcggaactg cct443
<210> 15
<211> 659
<212> DNA
<213> 人工序列
<400> 15
ccagaatcag gcaatagcat tgaatttact tatatgatcc atgctatcca taaaggtggc60
gagcgtcata ccttcgatgc taccggtgca caagtgcctg ccccatataa aattattggc120
tatggcggta aggtaatcga ttatggcaaa gtgcattacc cccaaaaacc agccgcagat180
tgtgcagcct gtcacgttga aggcgctggc gcacctgcta atgccgatct gttcaaagca240
gatttaagca atcaagcatg tattggctgt cacactgaaa aaccatctgc tcaccatagc300
agcactgatt gtatggcttg ccacaatgca accaagcctt acggcggtac gggaagtgca360
gctaaacgtc atggcgatgt aatgaaagct tataacgata gccttggtta taaagcgaaa420
ttcagcaaca ttggtattaa aaataatgcc ctaacattcg atgtacaaat tcttgataat480
aaagatcaac ctatcggcaa ggaatttatt tcggatccga gtgcatacac taaatcgagt540
atctatttct catggggaat agataaagat taccctgctt ataccgcagg tagcagatat600
agtgatcgtg gctttgcatt atcaaattcg aaggtttcaa cttacaacga agcaactaa659
<210> 16
<211> 550
<212> DNA
<213> 人工序列
<400> 16
agtacggccg caaggttaaa actcaaatga attgacgggg gcccgcacaa gcggtggagc60
atgtggttta attcgatgca acgcgaagaa ccttacctac tcttgacatc cacggaagac120
tgcagagatg cggttgtgcc ttcgggaacc gtgagacagg tgctgcatgg ctgtcgtcag180
ctcgtgttgt gaaatgttgg gttaagtccc gcaacgagcg caacccctat ccttatttgc240
cagcacgtaa tggtgggaac tctagggaga ctgccggtga taaaccggag gaaggtgggg300
acgacgtcaa gtcatcatgg cccttacgag tagggctaca cacgtgctac aatggcgagt360
acagagggtt gcaaagccgc gaggtggagc taatctcaca aagctcgtcg tagtccggat420
tggagtctgc aactcgactc catgaagtcg gaatcgctag taatcgtgga tcagaatgcc480
acggtgaata cgttcccggg ccttgtacac accgcccgtc acaccatggg agtgggctgc540
aaaagaagtg550
<210> 17
<211> 351
<212> DNA
<213> 人工序列
<400> 17
gcagtgatgc ggtgttgttc gtcaccccgg aatacaaccg ctcggtgcca ggctgcctga60
aaaatgccat cgatgtgggt tcgcgtcctt acgggcaaag tgcctggagc ggcaagccga120
cggcggtggt gagtgtgtcg ccgggggcga ttggtggctt tggcgccaac catgcggtgc180
gccagtcgct ggtgtttctc gacatgccct gcatgcagat gcccgaggct taccttggcg240
gtgcggcgag cttgttcgag gattcgggca agctcaatga caagacgcga ccgttcttgc300
aggcgtttgt cgacaggttt gcgtcatggg tgaagttgaa cagggcggtc t351
<210> 18
<211> 504
<212> DNA
<213> 人工序列
<400> 18
ctaataccgc atacgtccta cgggagaaag caggggacct tcgggccttg cgctatcaga60
tgagcctagg tcggattagc tagttggtgg ggtaatggct caccaaggcg acgatccgta120
actggtctga gaggatgatc agtcacactg gaactgagac acggtccaga ctcctacggg180
aggcagcagt ggggaatatt ggacaatggg cgaaagcctg atccagccat gccgcgtgtg240
tgaagaaggt cttcggattg taaagcactt taagttggga ggaagggcag taagctaata300
ccttgctgtt ttgacgttac cgacagaata agcaccggct aactctgtgc cagcagccgc360
ggtaatacag agggtgcaag cgttaatcgg aattactggg cgtaaagcgc gcgtaggtgg420
tttgttaagt tggatgtgaa agccccgggc tcaacctggg aactgcatcc aaaactggca480
agctagagta cggtagaggg tggt504
- 还没有人留言评论。精彩留言会获得点赞!
- 一种转基因鲑鱼AquAdvantage品系特异性实时荧光PCR检测引物、检测方法和试剂盒与流程
- 犬圆环病毒的实时荧光PCR检测用引物及检测方法与流程
- 用于荧光学图像的检测和实时显示的荧光学系统的制作方法与工艺
- 用于荧光学图像的检测和实时显示的荧光学系统的制作方法与工艺
- 利用单拷贝核基因检测食品和饲料中牛源性成分的实时荧光PCR检测方法与流程
- H10N7禽流感病毒的实时荧光PCR检测试剂盒及检测方法与流程
- 多重实时荧光PCR检测Cr(VI)还原复合菌群六种功能基因的方法与流程
- 刺苋的实时荧光PCR鉴定方法与流程
- 一种猪肺炎支原体的实时荧光PCR检测试剂盒及其用途的制作方法与工艺
- 实时荧光PCR特异性检测远志的引物及检测方法与流程
- 一种特异性引物检测鼠源性成分的方法与制造工艺
- 一组用于驴源性的实时荧光PCR检测的特异性引物和探针的制造方法与工艺
- 一组用于鸡源性实时荧光PCR检测的特异性引物和探针的制造方法与工艺
- 一种燕窝制品的实时荧光定量PCR鉴定试剂盒及其鉴定方法与制造工艺
- 一种具有移液功能的荧光定量PCR仪的制造方法与工艺
- 一种基于多灾种实时耦合的化工园区实时定量风险评估方法与制造工艺
- 一种实时荧光定量PCR检测方法及其标准品和检测试剂盒与制造工艺
- 检测外周血CAR‑T细胞的TaqMan实时荧光定量PCR试剂盒的制造方法与工艺
- 一种实时荧光定量PCR检测土壤西瓜枯萎病菌的方法及引物与制造工艺
- 一种基于反射镜的荧光定量PCR的检测系统的制造方法与工艺
- 一种基于荧光定量PCR鉴定啮齿类螺杆菌的引物、试剂盒及鉴定方法与流程
- 一种检测包含潜伏期结核感染的试剂盒及其引物对和探针的制造方法与工艺
- 用于甜瓜果实基因PCR表达分析的内参基因及该内参基因的稳定性验证方法与流程
- 一种特异性引物检测鼠源性成分的方法与制造工艺
- 一组用于驴源性的实时荧光PCR检测的特异性引物和探针的制造方法与工艺
- 一组用于鸡源性实时荧光PCR检测的特异性引物和探针的制造方法与工艺
- 一种燕窝制品的实时荧光定量PCR鉴定试剂盒及其鉴定方法与制造工艺
- 一种具有移液功能的荧光定量PCR仪的制造方法与工艺
- 一种基于多灾种实时耦合的化工园区实时定量风险评估方法与制造工艺
- 一种实时荧光定量PCR检测方法及其标准品和检测试剂盒与制造工艺