特异靶向拟南芥ILK2基因的sgRNA序列及其应用的制作方法
本发明属于基因工程技术领域,具体而言,涉及一种特异靶向拟南芥ilk2基因的sgrna序列及其应用。
背景技术:
zfn、talen和crispr基因打靶技术是基因编辑的核心技术,相比于前两种技术繁琐的构建程序,比如每一个位点需要构建一对相应的核酸酶,并且高度依赖目标上下游序列,crispr/cas9系统对特异位点的识别靠小的sgrna的引导,sgrna区可以由一系列针对不同基因的多个sgrna组成,每个针对特异位点的sgrna只有二十个碱基,整个载体较小,criepr载体更加容易构建,其基因编辑也更加高效。
crispr/cas9原是细菌和古细菌应对外源病毒侵害的防御系统,现已广泛应用于人类细胞、小鼠、斑马鱼、水稻等进行基因改造。它由导向序列sgrna和核酸酶cas9两种元件组成,sgrna可通过碱基互补识别基因组中特定序列从而引导核酸酶cas9对其进行切割造成双链dna断裂(double-strandbreaks,dsb),当宿主细胞通过非同源重组方式进行修复,发生非同源重组末端连接(non-homologousendjoining,nhej),就可能造成移码突变(frameshiftmutation),导致基因沉默(multiplexgenomeengineeringusingcrispr/cassystems.science.congetal.2013)。
这一技术由于能快速、简便、高效地靶向基因组任何基因,从而引起了广泛的关注,但是,随后的研究发现,该系统也存在一定的局限性,就是在非目标区域会产生dna的突变即脱靶效应,这在一定程度上阻碍了该技术的广泛应用(high-frequencyoff-targetmutagenesisinducedbycrispr-casnucleasesinhumancells.naturebiotechnology.fuetal,2013;high-throughputprofilingofoff-targetdnacleavagerevealsrna-programmedcas9nucleasespecificity.naturebiotechnology.pattanayaketal,2013)。麻省理工学院张峰团队使用双切刻技术将cas9的特异性提高了50倍以上,稳固了cas9在基因打靶技术领域的地位(doublenickingbyrna-guidedcrisprcas9forenhancedgenomeeditingspecificity.cell.ranetal,2013)。借助于这一技术,动植物基因功能研究及育种等领域都将得到迅猛的发展。精确靶向目标基因是crispr-cas9能否特异性沉默目标基因的先决条件,无论是脱靶还是错误靶向,都会影响crispr-cas9对目标基因的特异性沉默。因此,设计、制备出特异性靶向目标基因序列的sgrna成为了crispr-cas9基因沉默技术的关键。
拟南芥基因ilk2编码蛋白属于整合素连接蛋白激酶家族,具有丝氨酸和苏氨酸蛋白激酶活性。能与mapk、pkb、gsk3等多条信号通路和细胞骨架蛋白相互作用,参与调控细胞周期g1/s/g2期,抑制细胞凋亡等过程。在拟南芥中相关家族蛋白参与多种生物与非生物逆境,在介导细胞内环境稳态和植物免疫等方面有重要作用(theraf-likekinaseilk1andthehighaffinityk+transporterhak5arerequiredforinnateimmunityandabioticstressresponse.plantphysiology.elizabethetal,2016)。因此,在基因组水平上对拟南芥整合素连接蛋白激酶家族相关基因进行敲除或修饰,有助于解析相关基因的功能,为农作物抗性育种提供理论支持和实践参考。
技术实现要素:
本发明的目的在于提供一种特异靶向拟南芥整合素连接蛋白激酶家族基因ilk2的sgrna导向序列及其应用。本发明利用编码这种sgrna序列的dna构建植物表达载体,该表达载体能够表达出这种sgrna,这种sgrna能够特异性地识别拟南芥ilk2基因。本发明还利用这对sgrna引导cas9核酸酶(即cas9切割酶)对拟南芥ilk2基因进行准确、高效地靶向修饰。
具体地,本发明的目的是通过以下技术方案来实现的:
本发明的目的是提供一种敲除拟南芥ilk2基因的方法,提供了敲除拟南芥ilk2基因的sgrna,及用于敲除拟南芥ilk2基因的载体。
本发明的敲除拟南芥ilk2基因的sgrna序列,其序列如seqidno.1所示。
本发明的用于编码本发明的敲除拟南芥ilk2基因的sgrna的dna序列,所述dna序列如seqidno.2所示。
本发明的用于敲除拟南芥ilk2基因的载体,所述载体为含有能表达特异性靶向ilk2基因第一外显子的sgrna的dna序列(序列如seqidno.2所示)和cas9蛋白dna序列的植物表达载体。
优选的,sgrna的dna序列上游为拟南芥u6启动子,cas9蛋白dna序列上游为拟南芥u1启动子,筛选标记为潮霉素抗性。
本发明的靶向敲除拟南芥ilk2基因的载体构建方法,按以下步骤进行:
(1)确定ilk2基因的合适的靶标序列,所述靶标序列如seqidno.3所示;
(2)将靶标序列去除末端agg获得sgrna编码序列,其dna序列见seqidno.2所示;将seqidno.2序列反向互补获得sgrna的反向互补序列,其dna序列见seqidno.4所示;
(3)在sgrna编码序列的5端引入4个额外的碱基gatt,获得seqidno.5序列;在sgrna编码序列的反向互补序列的5端引入4个额外的碱基aaac,获得seqidno.6序列;人工合成seqidno.5和seqidno.6的序列后并将其制备成oligo二聚体;
(4)将制备好的oligo二聚体连接至载体cas9的u6启动子下游,然后转化大肠杆菌dh5α获得cas9-ilk2ko载体。
本发明还涉及一对用于鉴定所述sgrna序列敲除效果的引物对,所述引物对的序列如seqidno.7和seqidno.8所示。
与现有技术相比,本发明具有如下有益效果:
1)敲除载体cas9-ilk2ko转化拟南芥后,即可实现高效、快速、特异对拟南芥ilk2基因进行敲除。
2)可直接用此做研究材料来探讨ilk2基因的功能和作用机理。
附图说明
通过阅读参照以下附图对非限制性实施例所作的详细描述,本发明的其它特征、目的和优点将会变得更明显:
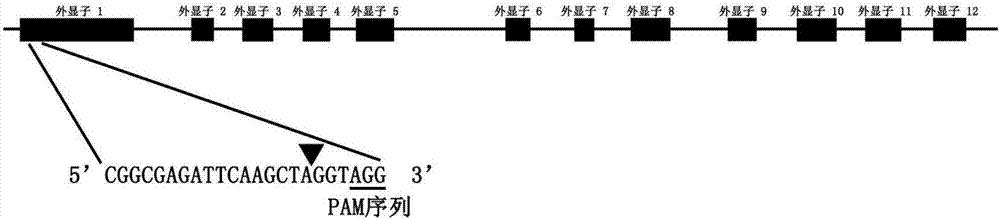
图1为sgrna靶点设计结果示意图,其中箭头处代表cas9酶切位点。
图2为农杆菌介导的拟南芥遗传转化得到的t1代植株抽提dna后进行pcr扩增,将pcr产物克隆后的部分测序结果(突变序列)。
具体实施方式
下面结合实施例对本发明进行详细说明。以下实施例将有助于本领域的技术人员进一步理解本发明,但不以任何形式限制本发明。应当指出的是,对本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干调整和改进。这些都属于本发明的保护范围。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。
cas9载体构建方法:patubq-cas9-tubq(applicationofthecrispr-cassystemforefficientgenomeengineeringinplants.maoyeta1,2013)由生工生物工程(上海)股份有限公司合成,通过双酶切(ecori,hindiii),连接(t4dna连接酶)到载体pcambia1300上,命名为cas9。bbsi内切酶购自neb公司,货号r0539s。dna寡核苷酸均由生工生物工程(上海)股份有限公司合成。
实施例1、拟南芥ilk2基因目标靶序列的获得
在phytozome网站(https://phytozome.jgi.doe.gov/pz/portal.html)获取ilk2基因第一外显子的编码区序列,根据设计用于敲除的sgrna原则,确定出目标靶序列,位于第一外显子,见图1,序列如下:
seqidno.3∶5’-cggcgagattcaagctaggtagg-3’
靶向编辑此dna序列的sgrna序列如下所示:
seqidno.1:5’-cggcgagauucaagcuaggu-3’
sgrna设计原则如下:
1.sgrna的长度,一般应为20nt左右
2.gc%含量最佳为40%~60%,
3.sgrna靶向基因的结合位置需尽量靠近基因编码区的atg下游,一般位于第一或第二外显子。
4.sgrna的序列与脱靶位点的匹配数尽可能低。
5.如采用cas9单切口酶,设计paired-grna需考虑成对sgrna的间距。
6.如果构建u6或t7启动子驱动sgrna的表达载体,需考虑sgrna的5′碱基为g或gg,以提高其转录效率。
实施例2、拟南芥cas9-ilk2ko载体的构建
(1)sgrna序列:目标靶序列去除3端pam序列agg得到sgrna的编码序列seqidno.2:5’-cggcgagattcaagctaggt-3’。
(2)sgrna编码序列的反向互补序列:将sgrna序列反向互补获得反向互补序列seqidno.4:5’-acctagcttgaatctcgccg-3’。
(3)合成单链dna寡核苷酸:根据所选择的cas9载体的bbsi酶切位点,在sgrna序列的3端引入4个额外的碱基gatt,获得seqidno.5序列;在sgrna反向互补序列的5端引入4个额外的碱基aaac,获得seqidno.6序列。将seqidno.5和seqidno.6的序列交由生工生物工程(上海)股份有限公司合成进行人工合成分别得到单链dna寡核苷酸forwardoligo与reverseoligo。
(4)oligo二聚体的制备:
将合成的oligo加水溶解至10μm,按下列反应体系混合后,95℃加热3分钟,然后以约0.2℃/秒缓慢降至20℃,反应在pcr仪上进行。
反应体系为:
ddh2o:8ul
forwardoligo:1μl
reverseoligo:1μl
(5)酶切cas9载体:
载体cas9有bbsi酶切位点,用bbsi酶切。
酶切体系:
bbsi:1μl
10×nebuffer:2μl
质粒:1μl
ddh2o:16μl
酶切条件为:37℃酶切1h
(6)构建cas9-ilk2ko载体:
将酶切后的cas9载体与步骤(4)制得的oligo二聚体利用t4连接酶进行连接。
连接体系为:
oligo二聚体:2μl
cas9载体:2μl
10×nebt4dnaligasebuffer:1μl
t4dnaligase:1μl
ddh2o:4μl
连接条件为:16℃连接过夜
(7)转化大肠杆菌:
将50μl感受态大肠杆菌dh5α加入(6)中的连接体系中混匀;混合液冰浴30min,42℃热刺激90s后,冰浴10min;加入800μllb液体培养基,37℃、200rpm摇床培养90min使菌体复苏;培养结束后,常温3000rpm离心1min收集菌体;在超净台上吸去上清,剩余约0.1ml时,使用移液枪混匀,接入带有kan抗性的lb固体平板上,用无菌三角棒涂布均匀;37℃过夜培养;挑取菌落接种于lb液体+kan的培养基,37℃、200rpm培养12h后,提取质粒。通过pcr检测获得的重组质粒,并将pcr检验正确的重组质粒送至上海博尚生物技术有限公司测序进行序列验证,最后获得目标载体cas9-ilk2ko。
实施例3、cas9-ilk2ko转农杆菌eha105
(1)农杆菌感受态细胞制备:
挑取农杆菌eha105单菌落接种于5mlyeb培养基中,28℃、200rpm摇床培养过夜,按1∶100的比例接种于50mlyeb培养基中扩大培养,28℃继续培养约6-7h至od600=0.4-0.6。将菌液置于冰上30min;5000rpm,4℃离心5min,弃上清,将菌体悬于10ml0.15mcacl2中;5000rpm,4℃离心5min,弃上清,菌体用1ml20mmcacl2轻轻悬浮,每管50μl分装,加入终浓度为20%的无菌甘油,-70℃保存。
(2)农杆菌的转化及鉴定:
将2μl载体cas9-ilk2kodna加入50μl农杆菌感受态中,混匀,冰浴30min,液氮冷冻4min,37℃水浴6min,加入1mlyeb培养基,28℃、200rpm摇床培养4h使菌体复苏。培养结束后,常温3000rpm离心1min收集菌体;在超净台上吸去上清,剩余约0.1ml时,使用移液枪混匀,接入带有kan抗性的yeb固体平板上,用无菌三角棒涂布均匀;28℃过夜培养;2天后,挑取单菌落接种于yeb液体+kan的培养基,28℃、200rpm培养12h后,进行pcr鉴定。
实施例4、含cas9-ilk2ko载体的农杆菌eha105转化拟南芥
(1)拟南芥前处理(可选步骤):
当拟南芥主花序长至5cm长左右的时候,将其剪掉,一周后花序再次长出,此时花序较多并且生长状态基本相同。
(2)农杆菌扩大培养:
将含cas9-ilk2ko载体的农杆菌eha105接种到100mlyeb培养基中,28℃、200rpm摇床培养18h。
(3)富集并重悬农杆菌菌体:
将(2)中的农杆菌菌液常温3000rpm离心10min收集菌体,去上清,用5%蔗糖溶液重悬菌体,od600=0.8-1.0。加入silwetl-77至终浓度为0.05%。
(4)侵染拟南芥:
拟南芥前一天浇水促进其生长。将拟南芥主花序浸入农杆菌菌液中30s,并轻柔搅动,使整个花序覆盖一层农杆菌菌液。
(5)暗处理:
用保鲜膜包裹整个拟南芥植株,保持花序湿润。用黑塑料袋包裹整盆拟南芥,放在暗处遮光生长24h。
(6)结束侵染:
去除遮盖物之后,放在长日照下正常培养生长。当角果变黄时候,可以收种,用含有潮霉素的固体ms培养基筛选阳性苗。
实施例5、拟南芥转基因植株的鉴定
(1)拟南芥dna的提取
将适量的经潮霉素筛选的拟南芥植株叶片放入1.5ml的离心管中,加入400μl的ctab抽提缓冲液,用蓝色小棒研磨成浆状,加入等体积酚∶氯仿∶异戊醇(25∶24∶1),猛烈摇匀,12000rpm,离心10min。取上清至新的离心管中,加入2倍体积的无水乙醇和0.1倍体积的3m醋酸钠(ph5.2),剧烈混匀使dna成团,放入-20℃沉淀2h以上。随后12000rpm,离心10min。弃上清,沉淀用70%乙醇洗涤一次后室温晾干,加适量的te或超纯水溶解。
(2)拟南芥转基因植株的鉴定
以抽提的拟南芥dna为模板,使用序列分别如seqidno.7和seqidno.8所示的引物checkf和checkr进行pcr扩增。
pcr反应体系为:
pcrbuffer:2.5μl
taq:0.5μl
dna:2μl
dntp:2μl
checkf:1μl
checkr:1μl
ddh2o:16μl
pcr反应条件为:
1.94℃5min
2.94℃变性30s
52℃退火30s
72℃延伸60s
35个循环
3.72℃10min
将pcr产物送至上海博尚生物技术有限公司进行序列测定并进行序列分析,可以发现转基因植株的目标靶序列出现了插入和缺失,将会产生移码突变,产生不具有功能的蛋白,达到敲除基因的目的;见图2。
以上对本发明的具体实施例进行了描述。需要理解的是,本发明并不局限于上述特定实施方式,本领域技术人员可以在权利要求的范围内做出各种变形或修改,这并不影响本发明的实质内容。
sequencelisting
<110>上海交通大学
<120>特异靶向拟南芥ilk2基因的sgrna序列及其应用
<130>dag29521
<160>8
<170>patentinversion3.5
<210>1
<211>20
<212>rna
<213>arabidopsisthaliana(l.)heynh.
<400>1
cggcgagauucaagcuaggu20
<210>2
<211>20
<212>dna
<213>artificialsequence
<220>
<223>artificialsequence
<400>2
cggcgagattcaagctaggt20
<210>3
<211>23
<212>dna
<213>arabidopsisthaliana(l.)heynh.
<400>3
cggcgagattcaagctaggtagg23
<210>4
<211>20
<212>dna
<213>artificialsequence
<220>
<223>artificialsequence
<400>4
acctagcttgaatctcgccg20
<210>5
<211>24
<212>dna
<213>artificialsequence
<220>
<223>artificialsequence
<400>5
gattcggcgagattcaagctaggt24
<210>6
<211>24
<212>dna
<213>artificialsequence
<220>
<223>artificialsequence
<400>6
aaacacctagcttgaatctcgccg24
<210>7
<211>20
<212>dna
<213>artificialsequence
<220>
<223>artificialsequence
<400>7
ttgatggacttgtgaacgct20
<210>8
<211>20
<212>dna
<213>artificialsequence
<220>
<223>artificialsequence
<400>8
cgagaagtcaagtcaggcaa20
- 还没有人留言评论。精彩留言会获得点赞!
- 采用寡核苷酸介导的基因修复提高靶向基因修饰的效率的方法和组合物与流程
- CRISPR/Cas9靶向敲除人ezrin基因增强子关键区的方法及其特异性gRNA与流程
- CRISPR/Cas9 靶向敲除人PD‑1基因及其特异性gRNA的制作方法与工艺
- 靶向人TSPAN8基因的siRNA及其应用的制作方法与工艺
- 靶向人TSPAN8基因的siRNA及其应用的制作方法与工艺
- CRISPR/Cas9靶向敲除人乙肝病毒P基因及其特异性gRNA的制作方法与工艺
- 一种特异靶向人ABCG2基因的sgRNA导向序列及其应用的制作方法与工艺
- 一种利用CRISPR‑Cas9靶向敲除MSTN基因的方法与流程
- CRISPR/Cas9靶向敲除人CNE10基因及其特异性gRNA的制作方法与工艺
- 用于猪APN基因编辑的靶向sgRNA、修饰载体及其制备方法和应用与流程