扩增PKD1外显子超长片段的PCR引物、检测PKD1基因突变的试剂盒及应用的制作方法
本发明涉及遗传病基因检测领域,更具体地,涉及一种扩增pkd1外显子超长片段的pcr引物、检测pkd1基因突变的试剂盒,以及所述pcr引物和试剂盒作为成人型多囊肾病致病基因检测试剂的应用。
背景技术:
多囊肾病(polycystickidneydisease,pkd)是一种双侧肾脏实质发生多个囊样肿块的遗传性肾脏疾病。根据发病年龄和遗传的特点,可以分为两种类型:常染色体隐性遗传型(婴儿型)多囊肾;常染色体显性遗传型(成年型)多囊肾。成人型多囊肾较为常见,人群中的发病率为1/400-1/1000;成人型多囊肾为常染色体显性遗传病,其特点是具有家族聚集性,男女均可发病,大部分患者在30-50岁才出现典型症状,故称“成人型”。成人型多囊肾的病理特征是双侧肾脏肿大,皮质、髓质出现多个大小不一的囊泡,且进行性增大,引起肾脏结构和功能损害。成人型多囊肾起病隐匿,患者早期临床症状轻,甚至无症状,多数在无意发现腹部有包块,或者体检中发现。随着年龄增长囊肿逐渐增大后,肾实质减少肾功能出现损害,临床症状明显。本病常累及肾外多个器官,患者常伴有多囊肝、高血压、多囊脾脏、多囊胰腺、颅内动脉瘤等。约50%的患者在60岁时进入终末期肾功能衰竭。目前尚无任何有效方法可以阻止多囊肾病的发展,成人型多囊肾家系成员非常关注尚无症状的幼年兄弟姐妹或子女的将来患病的风险,及患病人员生育下一代的问题。目前多囊肾病的诊断主要依据是双肾的超声、ct等影像学检查。基因层面的确诊对明确多囊肾病类型、早期干预及产前诊断预防该病发生起着更为关键的作用。
成人型多囊肾主要是由pkd1和pkd2基因突变致病,其中约85-90%的患者为pkd1基因突变导致的,约10-15%为pkd2基因突变,pkd1是成人型多囊肾的主要致病基因。pkd1基因编码的蛋白为多囊蛋白1(pc1),是11次跨膜大分子蛋白,主要表达在肾小管上皮细胞的细胞膜表面,负责将细胞外环境信号传递给胞内信号分子。pkd1基因位于染色体16p13.3区,基因体长达52kb,共有46个外显子,编码区长达12906bp。研究统计表明,成人型多囊肾基因缺陷以点突变致病为主,并且没有明显的突变热点或者热区,整个基因外显子区及内含子剪切位点附近均可能发生突变而致病。pkd1基因存在6个高度同源的假基因,与pkd1基因1-33外显子序列同源性高达97.7%,普通的pcr难以避免假基因的干扰。另外,pkd1基因结构复杂,部分区域gc含量超过70%,增加了pcr和sanger测序的难度。以上pkd1的基因结构特点和突变特点,给pkd1基因检测带来的巨大的困难,突变检出率较低。
根据pkd1基因的结构特点和突变类型,在新技术不断革新的背景下,pkd1基因突变的检测方法也在不断地创新与优化,先后提出的检测方法有:lr-pcr,变性高效液相色谱(dhplc)、多重连接探针扩增(mlpa)、lr-pcr-高通量测序(ngs)、外显子捕获-高通量测序(ngs)。
传统的巢式pcr-sanger测序,先用若干个长片段pcr分别扩增pkd1基因的各个区域,然后针对每一个外显子单独pcr扩增,再测序。虽然pcr结合sanger测序被认为是检测基因突变的经典方法,但是对于由46个外显子构成、编码区长达12906bp的pkd1基因而言,传统的巢式pcr-sanger测序技术耗费大量人力,成本高昂,效率低下。
巢式pcr-dhplc方法,根据双链dna解链特征的差异,可以对pkd1基因巢式pcr产物进行初步筛查,快速筛查有无突变。该方法的局限性是只能提供有无突变,不能得出具体的突变类型,不能直接检测纯合突变,并且结果判读容易出错。mlpa方法主要用于对pkd1基因大片段的缺失、重复或者重排,此类变异只占到pkd1异常患者的1-3%。
由于高度同源的假基因和富含gc含量区域的存在,常规的高通量测序(ngs)方法设计的引物panel在pkd1基因外显子上的覆盖度很低(<70%),达不到预期目的。us7553644b2、cn104531883a公开了一种检测pkd1基因突变的检测方法和试剂盒,先利用5-8对lr-pcr引物组,扩增pkd1基因第2_46外显子;然后以纯化的lr-pcr产物为模板,通过扩增子的方法或者酶切的方法构建文库。这种方法,已经应用到iontorrent或者illumina公司高通量测序平台。该方法具有高通量、快速、低成本的优点,可实现多样本、多基因的同时检测。该方法的缺点是:需要用到利用5-8对lr-pcr引物扩增,其中任何一个lr-pcr扩增失败,均需要重新制备,并且lr-pcr产物需要切胶纯化并精确定量;这种方法前期处理的工作量大,程序复杂。
因此,本领域仍然需寻求一种新的检测pkd1基因突变的方法,提高诊断准确率,简化操作流程,提高时效性。
技术实现要素:
本发明的目的是提供一种扩增pkd1外显子超长片段的pcr引物、检测pkd1基因突变的试剂盒,以及所述pcr引物和试剂盒作为成人型多囊肾病致病基因检测试剂的应用。通过先获得超长片段lr-pcr产物,再以lr-pcr产物为模板,进行多重pcr扩增,扩增产物利用高通量半导体测序技术进行测序,寻找致病突变,明确成人型多囊肾的遗传学病因,为临床提供遗传学和分子生物学的理论依据。
根据本发明的第一方面,本发明提供一种扩增pkd1外显子超长片段的pcr引物,所述pkd1外显子为第2至第46外显子,该pcr引物为具有如seqidno:1和seqidno:2所示碱基序列的引物。
长片段pcr(longrange-pcr,lr-pcr)是指扩增长度大于5kb的pcr,与常规的短片段pcr相比,最大的区别在于,其受dna模板的二级结构、引物的特异性和退火温度、以及不理想的循环条件等因素影响较大,以至于超长片段pcr的扩增难度较大。另外,pkd1基因结构复杂,除了存在多个高度同源的假基因外,其部分区域gc含量超过75%,增加了pcr扩增的难度。通过本发明提供的引物,可以完整扩增得到pkd1第2至第46外显子的超长片段。
根据本发明的第二方面,本发明提供一种检测pkd1基因突变的试剂盒,该试剂盒包括权利要求1所述的pcr引物,以及能够分别检测pkd1基因第2至第46外显子的扩增引物组合。
pkd1-e2_46_fl_forwordprimer:5′-gttcatttgggtcttccatcagaaagt-3′(seqidno:1)
pkd1_e2_46_fl_reverseprimer:5′-ggctagaaaccgtccaatactgctgt-3′(seqidno:2)
所述能够分别检测pkd1基因第2至第46外显子的扩增引物组合可以根据本发明的常规方法设计得到。优选地,经过本发明的发明人大量实验,为避免引物间相互干扰,所述能够分别检测pkd1基因第2至第46外显子的扩增引物组合包括两个引物池,分别为引物池1和引物池2,所述引物池1包括具有seqidno:3至seqidno:108所示碱基序列的引物,所述引物池2包括具有序列表seqidno:109至seqidno:214所示碱基序列的引物。每个引物池中各引物浓度为100-1000nm。
根据本发明,为便于操作,该试剂盒还优选包括:
用于所述扩增引物组合进行多重pcr反应的试剂;和/或
用于处理多重pcr扩增产物以使得多重pcr扩增产物能用于高通量测序的试剂。
其中,所述用于所述扩增引物组合进行多重pcr反应的试剂包括但不限于为dna聚合酶、pcr反应缓冲液和dntps。所述dna聚合酶优选为扩增效率高、高保真性、延伸性能好、适用于gc含量高的dna聚合酶。
常用的高通量测序平台是illumiahiseq/miseq和iontorrentpgm/proton测序仪。优选的,高通量测序仪为iontorrent测序平台。iontorrent半导体测序平台结合扩增子测序技术,与其他测序技术相比,具有通量选择灵活、测序周期短、运行成本低的特点,非常适合于单基因遗传病的基因诊断。
根据本发明,所述试剂盒还优选包括用于从样品中提取基因组dna的试剂。
上述试剂可配制也可商购获得。配制方法或商购途径为本领域技术人员公知。
根据本发明的第三方面,本发明提供上述pcr引物和/或上述试剂盒作为成人型多囊肾病致病基因检测试剂的应用。
具体地,可通过如下方法利用本发明的试剂盒检测成人型多囊肾致病基因-pkd1基因突变:
(1)取受检者样本,如外周血、唾液、组织器官,提取基因组dna;
(2)采用高保真、高效的dna聚合酶,利用优选的引物浓度、反应温度、反应时间,扩增覆盖pkd1基因第2至第46外显子的长片段;
(3)对步骤(2)得到的长片段pcr产物纯化;
(4)利用引物池1和引物池2的dna文库从iontorrent高通量测序平台自动合成覆盖pkd1基因第2至第46外显子的扩增引物文库,以上述lr-pcr产物为模板,在适于扩增目的dna片段的条件下,采用多重pcr技术对45个外显子进行靶向扩增;
(5)对步骤(4)得到的多重pcr扩增产物进行酶切,消化引物序列;
(6)对步骤(5)得到的酶切产物加标签序列(barcode接头)。优选8个碱基的核苷酸序列,如ionxpressbarcodeadapters33-48kit。所述标签只是为了区别每个样品,以便用于同时检测多个样品,本领域技术人员可以根据需要以及通常的引物设计原则选择使用不同的标签,而实现本发明的目的;
(7)对步骤(6)得到的连接产物进行纯化;
(8)对步骤(7)得到的纯化产物用通用引物进行二次扩增;优选的,该通用引物来自于高通量测序建库试剂盒;
(9)对步骤(8)得到的二次扩增产物进行,进一步纯化、去除大片段dna,选择保留文库扩增片段,并进行精确的浓度测定,即完成待测样本目标区域文库扩增;
(10)对步骤(9)所得到的文库进行等量混合,经过乳液pcr扩增反应(onetouch)后,将待测序目的片段进一步扩增并连接于isp珠子,得到反应液;
(11)对步骤(10)得到的isp珠子连接的扩增产物富集、纯化;
(12)将步骤(11)得到的产物负载加入芯片,上ionpgm测序仪进行测序;
(13)将步骤(12)得到的测序信息进行生物信息学比对处理,排除多态性变异,获取每个样本pkd1基因的突变;
(14)对步骤(13)得到的突变位点进行pcr-sanger测序法验证。
本发明具有以下优点:
(1)采用基于超长片段pcr产物为模板的高通量测序技术,每个待测样本只需要经过一个超长片段pcr反应,pcr产物只需简单纯化或者不经纯化即可以用于后续的文库构建。为了完成对pkd1基因高覆盖度,现有技术采用的多管长片段pcr方法需要在反应之后切胶纯化、精确定量,操作复杂;并且,在多管长片段pcr方法中,其中任何一个lr-pcr扩增失败,均需要重新制备,这种方法前期处理的工作量大,程序复杂。本发明简化了操作流程,降低了成本,缩短了实验周期;具有扩增效率高、特异性强、适用于高通量的优点,可实现多样本、多个外显子的同时检测。
(2)设计的引物密集,扩增产物覆盖范围广,能够在一次测序反应中同时检测pkd1基因第2至第46外显子及毗邻区域,覆盖度为96.7%,针对hgmd和pkdmutationdatabase(http://pkdb.mayo.edu)已报道的致病突变的覆盖大于95%。
(3)利用高通量测序,特别是iontorrent高通量测序平台,读长长达300bp,准确性高(99.9%);采用iontorrent测序平台,能根据不同芯片数据量的大小,调整检测样本数量,在保证平均测序深度在200×的前提下,ion316tm芯片检测样本量可以多达到100个,大大降低了扩增反应的成本;整个检测流程(从提取dna、文库构建、测序反应和数据分析判读)能够在2.5天内完成,提高了时效性,尤其适合于临床诊断服务。
(4)通过对目标扩增区域的测序和数据判读,能够准确识别与疾病相关的突变,判断疾病种类和病因,为临床提供及时可靠的检测报告。本发明高通量测序检测到的点突变、微小缺失/重复,经过sanger测序法验证;对高通量测序深度达到100×的点突变,本发明的准确度超过99%。
本发明的其它特征和优点将在随后具体实施方式部分予以详细说明。
附图说明
通过结合附图对本发明示例性实施方式进行更详细的描述。
图1示出了引物组合的设计。以人类基因组(hg19)chr16:2089483-2119546序列为参考序列,覆盖pkd1基因第1内含子/第2外显子毗邻位置(ivs2-168)到3’-端非翻译区(c.*1267)位置,将该段序列为customreference,针对外显子及毗邻区域设计多重pcr引物。引物池1和引物池2对于目标区域的覆盖为96.74%,缺失区域占3.26%。
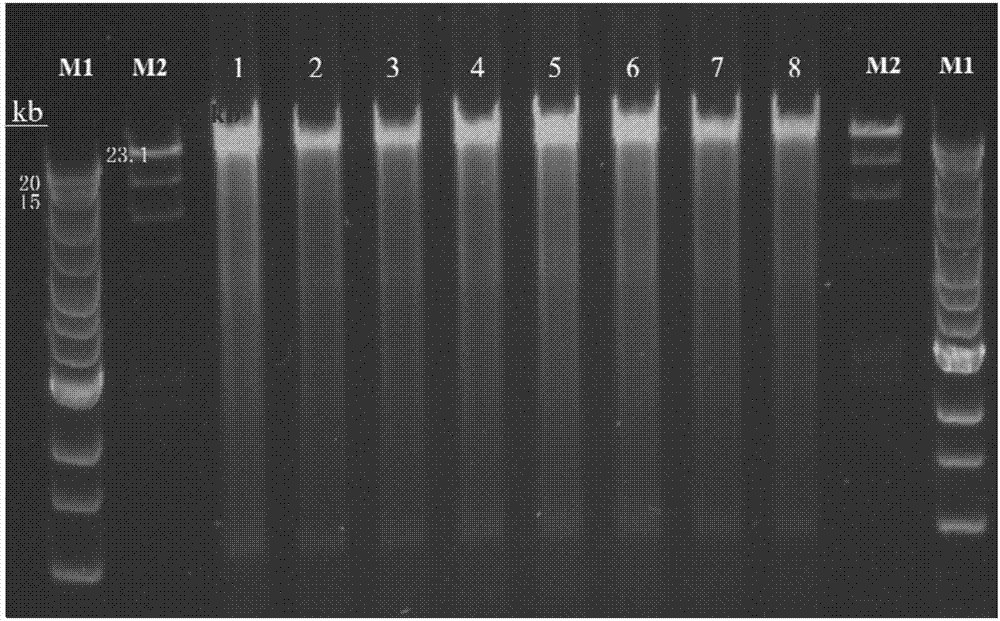
图2示出了8个样本的超长片段pcr产物的电泳结果。目的条带大小为30064bp,电泳结果与预期一致。
图3示出了一个样本的iontorrentpgm测序的整体覆盖度和深度,图中显示所有pkd1基因e2_46外显子区平均覆盖深度达到1200*,除外显子部分区域因高gc含量不能完全检测外,目标区域的覆盖度完全。
图4示出了一个样本的iontorrentpgm测序结果中pkd1基因e2_46各外显子区的覆盖图。
图5①-⑧为8个pkd1基因突变检测为阳性的样本的igv视图(上)和sanger测序验证结果(下)。其中,图5①为c.6832g>a杂合突变的igv视图和验证结果;图5②为c.12053_12054inst杂合突变的igv视图和验证结果;图5③为c.10540c>t杂合突变的igv视图和验证结果;图5④为c.9713-1g>a杂合突变的igv视图和验证结果;图5⑤为c.11537g>c杂合突变的igv视图和验证结果;图5⑥为c.3349c>t杂合突变的igv视图和验证结果;图5⑦为c.4669c>t杂合突变的igv视图和验证结果;图5⑧为c.9013c>t杂合突变的igv视图和验证结果。
具体实施方式
下面将参照附图更详细地描述本发明的优选实施方式。
所用到的试剂:
kodfxneodna聚合酶,ionampliseqlibrarykit2.0,ionpgmtemplateot2200kitv3,ionpgmsequencing200kitv2,ionxpressbarcordeadaptor33-48kit,ion316chipkitv2bc。
1、提取待检测的样本全基因组dna;
使用omega“基因组dna抽提试剂盒”提取外周血dna,同时去除rna。提取的dna总体积约为100ul,浓度在20-200ng/μl;a260/280在1.8-2.0之间。
2、超长片段pcr扩增pkd1基因第2至第46外显子;
pcr扩增试剂选用kodfxneo(东洋纺,toyobolifescience)dna聚合酶,以基因组dna为模板最大可扩增40kb的片段,具有高扩增效率、高保真性的特点,并且适用于高gc含量片段的扩增。
扩增程序:
pcr产物经琼脂糖凝胶电泳,由图2可见,检测扩增片段大小与预期目的片段大小一致。
3、超长片段pcr产物的纯化
pcr产物使用
4、以长片段pcr产物为模板,经多重pcr扩增目标区域
取纯化的长片段pcr产物,稀释500倍作为使用液,用两个引物组(引物池1和引物池2)同时扩增。每个引物池中引物的浓度为400nm。
扩增程序:
5、消化引物序列
pcr扩增结束,离心,将每个样本的引物池1和引物池2两管反应液合并为一管,然后加入2μlfupareagent,总体系达到22μl,涡旋混匀。
反应条件:50℃,10min;55℃,15min,60℃,20min;4℃保持
6、barcode连接至扩增产物并纯化
反应条件:22℃,30min;72℃,15min;4℃保持
产物使用
7、文库扩增、纯化和测定
扩增程序:
pcr扩增产物需经过两次纯化,分别去除基因组dna和pcr反应中的各类离子、引物、酶。纯化步骤按照ionampliseqlibrarypreparation操作手册进行。纯化产物,经qubit2.0fluorometer定量,即制备成片段大小200bp左右的文库。大于0.1ng/μl的文库可以进入后续乳液pcr反应。
8、乳液pcr反应(采用ionpgmtemplateot2200kitv3):
将构建好的文库,按照分子量和浓度等比例混合,配制成2pg/μl的文库混合液,上述反应体系加入onetouch2中进行乳液pcr反应,制备template-positiveot2200ionsphereparticles。
9、高通量测序(采用ionsequencing200kitv2):
乳液pcr反应完成后,连接有测序模板的ionsphereparticles,经过onetouches富集、纯化。加入测序引物,退火处理,模板与引物结合,然后加入dna聚合酶,室温孵育5min后,将反应液载入iontorrent316v2芯片,转移至ionpgm测序仪进行测序。
结果分析
torrentsuite对原始的测序数据提取、清除接头序列、滤过质量差的读常;结果与人类基因组参考序列hg19进行序列比对、及snvs和indel提取,测序数据经过coverageanalysis和variantcaller分析,得到碱基序列和突变位点信息(vcf文件和bam、bai文件);经过ionreporter在线软件注释后,得到pkd1基因突变信息。针对得到的突变位点,采用pcr-sanger测序法进行验证。图3和图4显示一个样本的iontorrentpgm测序的覆盖度和深度,图中显示所有pkd1基因e2_46外显子区平均覆盖深度达到1200*,除极少部分区域因高gc含量不能完全覆盖外,其余所有外显子的覆盖率为100%。图5①-⑧为8个pkd1基因突变检测为阳性的样本的igv视图(上)和sanger测序验证结果(下)。图5①为c.6832g>a杂合突变的igv视图和验证结果;图5②为c.12053_12054inst杂合突变的igv视图和验证结果;图5③为c.10540c>t杂合突变的igv视图和验证结果;图5④为c.9713-1g>a杂合突变的igv视图和验证结果;图5⑤为c.11537g>c杂合突变的igv视图和验证结果;图5⑥为c.3349c>t杂合突变的igv视图和验证结果;图5⑦为c.4669c>t杂合突变的igv视图和验证结果;图5⑧为c.9013c>t杂合突变的igv视图和验证结果。高通量测序筛出的pkd1基因突变结果与sanger测序的结果是一致的,说明本发明的方法是可行的。
受屏幕分辨率和附图尺寸所限,图1、图5①-图5⑧中部分字体显示不清,但并不影响本领域技术人员对本发明技术方案和技术效果的理解。
以上已经描述了本发明的各实施例,上述说明是示例性的,并非穷尽性的,并且也不限于所披露的各实施例。在不偏离所说明的各实施例的范围和精神的情况下,对于本技术领域的普通技术人员来说许多修改和变更都是显而易见的。
sequencelisting
<110>郑州大学第一附属医院
<120>扩增pkd1外显子超长片段的pcr引物、检测pkd1基因突变的试剂盒及应用
<130>1700032
<160>214
<170>patentinversion3.3
<210>1
<211>27
<212>dna
<213>人工序列
<400>1
gttcatttgggtcttccatcagaaagt27
<210>2
<211>26
<212>dna
<213>人工序列
<400>2
ggctagaaaccgtccaatactgctgt26
<210>3
<211>20
<212>dna
<213>人工序列
<400>3
ggatggcctctggcctacta20
<210>4
<211>22
<212>dna
<213>人工序列
<400>4
gattcggcaaagctgatggaag22
<210>5
<211>18
<212>dna
<213>人工序列
<400>5
gggctggcatagaccctt18
<210>6
<211>19
<212>dna
<213>人工序列
<400>6
cctggctgggaaggacaga19
<210>7
<211>19
<212>dna
<213>人工序列
<400>7
cctgacaacagctcaggca19
<210>8
<211>18
<212>dna
<213>人工序列
<400>8
ggaagacgtgctggagga18
<210>9
<211>20
<212>dna
<213>人工序列
<400>9
cagctagcagccttccacat20
<210>10
<211>19
<212>dna
<213>人工序列
<400>10
tcgaggctctcgtcactct19
<210>11
<211>18
<212>dna
<213>人工序列
<400>11
gcccacactgaccgttga18
<210>12
<211>19
<212>dna
<213>人工序列
<400>12
gcgggactgtccaccattg19
<210>13
<211>19
<212>dna
<213>人工序列
<400>13
gctgtgagggtgggaggat19
<210>14
<211>19
<212>dna
<213>人工序列
<400>14
gctcgcagacgtagctgtg19
<210>15
<211>19
<212>dna
<213>人工序列
<400>15
ccagtctgttcgtcctggt19
<210>16
<211>18
<212>dna
<213>人工序列
<400>16
ttggcgagacccacagtg18
<210>17
<211>21
<212>dna
<213>人工序列
<400>17
cccaccaggtcatggtattcc21
<210>18
<211>21
<212>dna
<213>人工序列
<400>18
gcagacgtgaaagctcagaga21
<210>19
<211>20
<212>dna
<213>人工序列
<400>19
ctggagccaacatctgcttg20
<210>20
<211>22
<212>dna
<213>人工序列
<400>20
ctgaggagatgcagggaacaga22
<210>21
<211>18
<212>dna
<213>人工序列
<400>21
acggccaggatgtcctca18
<210>22
<211>19
<212>dna
<213>人工序列
<400>22
tccagggttgggcctcaag19
<210>23
<211>20
<212>dna
<213>人工序列
<400>23
cacaacctctcctgcagctt20
<210>24
<211>20
<212>dna
<213>人工序列
<400>24
ggcagtgctaccactgagaa20
<210>25
<211>21
<212>dna
<213>人工序列
<400>25
cgtgtactgcagggagtccta21
<210>26
<211>25
<212>dna
<213>人工序列
<400>26
tgactttccaggaatttaaagccca25
<210>27
<211>20
<212>dna
<213>人工序列
<400>27
ggtggaccatcaacgacaag20
<210>28
<211>20
<212>dna
<213>人工序列
<400>28
ctcgggcagcatgaagcaga20
<210>29
<211>22
<212>dna
<213>人工序列
<400>29
ccgtgaactacaacgtaaccgt22
<210>30
<211>24
<212>dna
<213>人工序列
<400>30
ttacctcccaacagacagggaaac24
<210>31
<211>20
<212>dna
<213>人工序列
<400>31
ctcagtgctgctacacctgt20
<210>32
<211>20
<212>dna
<213>人工序列
<400>32
gctgcgtcaggttctcgaag20
<210>33
<211>23
<212>dna
<213>人工序列
<400>33
ggtgttctttacacgtgggactt23
<210>34
<211>21
<212>dna
<213>人工序列
<400>34
gaaggtccacgtgatgttgtc21
<210>35
<211>22
<212>dna
<213>人工序列
<400>35
caacagtggagcatgtgtacct22
<210>36
<211>22
<212>dna
<213>人工序列
<400>36
aggtccagtcgaagaggtagtg22
<210>37
<211>22
<212>dna
<213>人工序列
<400>37
gcgcattacttcaccagcatct22
<210>38
<211>23
<212>dna
<213>人工序列
<400>38
cattggcagcagagatgttgttg23
<210>39
<211>20
<212>dna
<213>人工序列
<400>39
gaggtcacccacgcttacaa20
<210>40
<211>24
<212>dna
<213>人工序列
<400>40
cggaaggtgtaagagatggtagga24
<210>41
<211>18
<212>dna
<213>人工序列
<400>41
caggccgtggttagggat18
<210>42
<211>18
<212>dna
<213>人工序列
<400>42
gctgcccagcatgttggt18
<210>43
<211>23
<212>dna
<213>人工序列
<400>43
ggtgtcgtatacacttggtcctt23
<210>44
<211>20
<212>dna
<213>人工序列
<400>44
cacctgcacatccacttcca20
<210>45
<211>22
<212>dna
<213>人工序列
<400>45
ccctcatgtcaccatggtcttc22
<210>46
<211>23
<212>dna
<213>人工序列
<400>46
cagcaggatctgaaaatggacca23
<210>47
<211>19
<212>dna
<213>人工序列
<400>47
cccgtttctcccacagctt19
<210>48
<211>18
<212>dna
<213>人工序列
<400>48
agtcgccctggaccttct18
<210>49
<211>18
<212>dna
<213>人工序列
<400>49
gctggtgctggaggttca18
<210>50
<211>19
<212>dna
<213>人工序列
<400>50
ctgggctcatctgtgtcct19
<210>51
<211>20
<212>dna
<213>人工序列
<400>51
ctccaacctggtgagcttct20
<210>52
<211>22
<212>dna
<213>人工序列
<400>52
cggtactcagtctggtaggtga22
<210>53
<211>18
<212>dna
<213>人工序列
<400>53
cgcctggtgcccatcatt18
<210>54
<211>19
<212>dna
<213>人工序列
<400>54
gggtgttctctgggctcat19
<210>55
<211>21
<212>dna
<213>人工序列
<400>55
ttggaaggtggcaaaccggat21
<210>56
<211>21
<212>dna
<213>人工序列
<400>56
gccctccaagtacacgtagga21
<210>57
<211>20
<212>dna
<213>人工序列
<400>57
gcacgtacgttcagcaacaa20
<210>58
<211>20
<212>dna
<213>人工序列
<400>58
gtgcattcgaagtgcacctt20
<210>59
<211>23
<212>dna
<213>人工序列
<400>59
cgaggagttctgtgtctacaagg23
<210>60
<211>21
<212>dna
<213>人工序列
<400>60
gtgatggccaaagacctacga21
<210>61
<211>22
<212>dna
<213>人工序列
<400>61
gcacgtcatcgagtactcgttg22
<210>62
<211>23
<212>dna
<213>人工序列
<400>62
gggtcactgggatttatctctgg23
<210>63
<211>22
<212>dna
<213>人工序列
<400>63
gcaagaacatcacggagactct22
<210>64
<211>22
<212>dna
<213>人工序列
<400>64
ctgcgttcacacaggacagaac22
<210>65
<211>19
<212>dna
<213>人工序列
<400>65
ccatcggagacagcatcct19
<210>66
<211>26
<212>dna
<213>人工序列
<400>66
ggagaaaagggatggtaatagggaag26
<210>67
<211>18
<212>dna
<213>人工序列
<400>67
gtcccaggcctacaacct18
<210>68
<211>24
<212>dna
<213>人工序列
<400>68
gattggagtccaccagaaagatga24
<210>69
<211>21
<212>dna
<213>人工序列
<400>69
caactacaccgtctccaccaa21
<210>70
<211>23
<212>dna
<213>人工序列
<400>70
cgtatagttgagctgcagatgca23
<210>71
<211>22
<212>dna
<213>人工序列
<400>71
gggtgtctctggtgaaatttgc22
<210>72
<211>21
<212>dna
<213>人工序列
<400>72
gctccgagtgtaggtagactg21
<210>73
<211>20
<212>dna
<213>人工序列
<400>73
gggcatcatgggtcagcatt20
<210>74
<211>21
<212>dna
<213>人工序列
<400>74
gtcctcctcgctgaagtactg21
<210>75
<211>22
<212>dna
<213>人工序列
<400>75
ctatcctgagaaggcacagctt22
<210>76
<211>23
<212>dna
<213>人工序列
<400>76
gacgaggatctcgtacttgaagc23
<210>77
<211>20
<212>dna
<213>人工序列
<400>77
cacaggccaaagctgagatg20
<210>78
<211>21
<212>dna
<213>人工序列
<400>78
cgcacaaacctttgttgtcgt21
<210>79
<211>22
<212>dna
<213>人工序列
<400>79
cgccttcttcctggtcaatgac22
<210>80
<211>23
<212>dna
<213>人工序列
<400>80
agatgtgcttgtcaaagaagcca23
<210>81
<211>22
<212>dna
<213>人工序列
<400>81
acatctggctctccatatggga22
<210>82
<211>19
<212>dna
<213>人工序列
<400>82
ggctcagcctggacacatg19
<210>83
<211>20
<212>dna
<213>人工序列
<400>83
gtgtccagcgtggttgtcta20
<210>84
<211>21
<212>dna
<213>人工序列
<400>84
tgctcaagtgtgttgaccctt21
<210>85
<211>20
<212>dna
<213>人工序列
<400>85
cgcagcttggactcaagaca20
<210>86
<211>20
<212>dna
<213>人工序列
<400>86
ctgggcttccgagcaaacct20
<210>87
<211>22
<212>dna
<213>人工序列
<400>87
cgtccattgtgggtagcaatct22
<210>88
<211>22
<212>dna
<213>人工序列
<400>88
cctgctggatcaggtcttcatc22
<210>89
<211>20
<212>dna
<213>人工序列
<400>89
gcccaggttaacatgggctt20
<210>90
<211>21
<212>dna
<213>人工序列
<400>90
gaagcagagacagacctgtga21
<210>91
<211>20
<212>dna
<213>人工序列
<400>91
ccagcttcctggcctcattc20
<210>92
<211>23
<212>dna
<213>人工序列
<400>92
ctaccagggtgtcatcttcatcc23
<210>93
<211>19
<212>dna
<213>人工序列
<400>93
ccaaagccctgctgtcact19
<210>94
<211>20
<212>dna
<213>人工序列
<400>94
gtctggctggactaaaggca20
<210>95
<211>22
<212>dna
<213>人工序列
<400>95
caggaaacactcctgttgggtt22
<210>96
<211>23
<212>dna
<213>人工序列
<400>96
cactcctggagaactactccctt23
<210>97
<211>18
<212>dna
<213>人工序列
<400>97
cttcctgcagctgcacaa18
<210>98
<211>18
<212>dna
<213>人工序列
<400>98
agagggtgcgggtcagta18
<210>99
<211>17
<212>dna
<213>人工序列
<400>99
cctctgctcacctcggt17
<210>100
<211>17
<212>dna
<213>人工序列
<400>100
agcaggcacacctgtgg17
<210>101
<211>18
<212>dna
<213>人工序列
<400>101
ccagtggacccgtttcgt18
<210>102
<211>22
<212>dna
<213>人工序列
<400>102
acgagaaatctgtctgcttgca22
<210>103
<211>20
<212>dna
<213>人工序列
<400>103
ggccatcctggtaggtgact20
<210>104
<211>21
<212>dna
<213>人工序列
<400>104
ctcggcaggacacagggtaga21
<210>105
<211>19
<212>dna
<213>人工序列
<400>105
ccttgcgtggagagctgta19
<210>106
<211>18
<212>dna
<213>人工序列
<400>106
gcggctccatcccttcaa18
<210>107
<211>18
<212>dna
<213>人工序列
<400>107
gccctgctcacccagttt18
<210>108
<211>18
<212>dna
<213>人工序列
<400>108
atgggccacgggaagatc18
<210>109
<211>20
<212>dna
<213>人工序列
<400>109
cttccatcagctttgccgaa20
<210>110
<211>21
<212>dna
<213>人工序列
<400>110
ggagggcagaagggatattgg21
<210>111
<211>19
<212>dna
<213>人工序列
<400>111
actgggcagggctcaggaa19
<210>112
<211>21
<212>dna
<213>人工序列
<400>112
gtgggcagctgaaaaggacac21
<210>113
<211>18
<212>dna
<213>人工序列
<400>113
ccacgaaggcctgcttca18
<210>114
<211>20
<212>dna
<213>人工序列
<400>114
ctagctggccagaggccaga20
<210>115
<211>19
<212>dna
<213>人工序列
<400>115
gacagacgtgcaggtggaa19
<210>116
<211>19
<212>dna
<213>人工序列
<400>116
gggtgtcaacggtcagtgt19
<210>117
<211>20
<212>dna
<213>人工序列
<400>117
ccctcggacacggagatctt20
<210>118
<211>24
<212>dna
<213>人工序列
<400>118
gcttcagagatctcccaacctatg24
<210>119
<211>20
<212>dna
<213>人工序列
<400>119
gtggtgtaacaccgacctgt20
<210>120
<211>18
<212>dna
<213>人工序列
<400>120
tggcgagtgcacagtgag18
<210>121
<211>21
<212>dna
<213>人工序列
<400>121
gacagtttccaccggagtctt21
<210>122
<211>20
<212>dna
<213>人工序列
<400>122
gctcctgggtcccaaattcg20
<210>123
<211>20
<212>dna
<213>人工序列
<400>123
gagacctgtcctcacagcaa20
<210>124
<211>18
<212>dna
<213>人工序列
<400>124
ccctgacgtgcagccatt18
<210>125
<211>18
<212>dna
<213>人工序列
<400>125
gctccttcctgtctgcca18
<210>126
<211>18
<212>dna
<213>人工序列
<400>126
ctgcaagccaacgaggtc18
<210>127
<211>20
<212>dna
<213>人工序列
<400>127
ttgcagccacggaacagctc20
<210>128
<211>18
<212>dna
<213>人工序列
<400>128
ttggtgggcacgtagagg18
<210>129
<211>19
<212>dna
<213>人工序列
<400>129
ccctgggagaccaacgata19
<210>130
<211>18
<212>dna
<213>人工序列
<400>130
tggagcctcggccatact18
<210>131
<211>21
<212>dna
<213>人工序列
<400>131
gtccaggaggcgacaggctaa21
<210>132
<211>24
<212>dna
<213>人工序列
<400>132
acattgaagaccacgttctggaag24
<210>133
<211>20
<212>dna
<213>人工序列
<400>133
caccttcacctctgccttct20
<210>134
<211>20
<212>dna
<213>人工序列
<400>134
gcagaccctgcatcctgttc20
<210>135
<211>18
<212>dna
<213>人工序列
<400>135
cctgtcccggttcactca18
<210>136
<211>20
<212>dna
<213>人工序列
<400>136
ctcagagcctgaaaggcagt20
<210>137
<211>20
<212>dna
<213>人工序列
<400>137
ctgaccgtgctggcatctaa20
<210>138
<211>20
<212>dna
<213>人工序列
<400>138
ctcgaggcataggtgtggtt20
<210>139
<211>18
<212>dna
<213>人工序列
<400>139
gcgcctggaggtcaacaa18
<210>140
<211>18
<212>dna
<213>人工序列
<400>140
ccacggtcactgtgcagt18
<210>141
<211>18
<212>dna
<213>人工序列
<400>141
ggcccactacctcttcga18
<210>142
<211>18
<212>dna
<213>人工序列
<400>142
cccgagctgcacaaactg18
<210>143
<211>21
<212>dna
<213>人工序列
<400>143
ccaggctcctatcttgtgaca21
<210>144
<211>20
<212>dna
<213>人工序列
<400>144
ccaccctaacggtgaagtca20
<210>145
<211>21
<212>dna
<213>人工序列
<400>145
ctattcctgggtgctctgtga21
<210>146
<211>20
<212>dna
<213>人工序列
<400>146
ggcagtccagctgtaggaga20
<210>147
<211>18
<212>dna
<213>人工序列
<400>147
cggcacctaccatgtgca18
<210>148
<211>23
<212>dna
<213>人工序列
<400>148
gaagctatgggtggtaaatggct23
<210>149
<211>20
<212>dna
<213>人工序列
<400>149
cctgcacttggtcaccatga20
<210>150
<211>22
<212>dna
<213>人工序列
<400>150
gggaagaccatggtgacatgag22
<210>151
<211>20
<212>dna
<213>人工序列
<400>151
gggtctcagccacgtacaac20
<210>152
<211>20
<212>dna
<213>人工序列
<400>152
ccagctcacgtggtttttgc20
<210>153
<211>18
<212>dna
<213>人工序列
<400>153
tcgcctacgcctggtact18
<210>154
<211>19
<212>dna
<213>人工序列
<400>154
ctctgcagggccacatact19
<210>155
<211>20
<212>dna
<213>人工序列
<400>155
ggcctaccactgggactttg20
<210>156
<211>21
<212>dna
<213>人工序列
<400>156
tccaagtagttgcgctgtgat21
<210>157
<211>24
<212>dna
<213>人工序列
<400>157
tgctttgtgtttgtcgtgtcattt24
<210>158
<211>19
<212>dna
<213>人工序列
<400>158
gtctgaccacacgcggtat19
<210>159
<211>21
<212>dna
<213>人工序列
<400>159
cgggctctgctttaaaactgg21
<210>160
<211>18
<212>dna
<213>人工序列
<400>160
ctgggctgtccaaggcaa18
<210>161
<211>20
<212>dna
<213>人工序列
<400>161
ttggagtgtgtgtcctgcaa20
<210>162
<211>21
<212>dna
<213>人工序列
<400>162
gtcttgttgctgaacgtacgt21
<210>163
<211>18
<212>dna
<213>人工序列
<400>163
cggcgagggatacacctt18
<210>164
<211>19
<212>dna
<213>人工序列
<400>164
ctccgtgacgtcacagagt19
<210>165
<211>18
<212>dna
<213>人工序列
<400>165
ctgtggtcgccctcaaca18
<210>166
<211>19
<212>dna
<213>人工序列
<400>166
gctgcactcacctcgttca19
<210>167
<211>22
<212>dna
<213>人工序列
<400>167
gcccatgaagaaacagctcagt22
<210>168
<211>21
<212>dna
<213>人工序列
<400>168
gctggatgtcatccacagtgt21
<210>169
<211>19
<212>dna
<213>人工序列
<400>169
aggtgaggacccgtgtaga19
<210>170
<211>18
<212>dna
<213>人工序列
<400>170
gcggcacctgtgatgttg18
<210>171
<211>21
<212>dna
<213>人工序列
<400>171
tccctctacctccctgtctct21
<210>172
<211>18
<212>dna
<213>人工序列
<400>172
gcatgaggatgcgcatga18
<210>173
<211>18
<212>dna
<213>人工序列
<400>173
cctggccaacctcagtga18
<210>174
<211>18
<212>dna
<213>人工序列
<400>174
cggcctgtgtctggaatg18
<210>175
<211>20
<212>dna
<213>人工序列
<400>175
gtcaccctggacagcagcaa20
<210>176
<211>21
<212>dna
<213>人工序列
<400>176
atggaagccctacgagaaacg21
<210>177
<211>22
<212>dna
<213>人工序列
<400>177
actacctgtctgaggaacctga22
<210>178
<211>20
<212>dna
<213>人工序列
<400>178
ggcaatgctgacccatgatg20
<210>179
<211>21
<212>dna
<213>人工序列
<400>179
tctgaacctctccagccactt21
<210>180
<211>23
<212>dna
<213>人工序列
<400>180
gtcactcacaggaaacacaaagc23
<210>181
<211>22
<212>dna
<213>人工序列
<400>181
cctggtgacctacatggtcatg22
<210>182
<211>23
<212>dna
<213>人工序列
<400>182
tgcaaaaactgccttgttctgac23
<210>183
<211>20
<212>dna
<213>人工序列
<400>183
ctgggtagcgtgtggaagat20
<210>184
<211>23
<212>dna
<213>人工序列
<400>184
gccagtcattgaccaggaagaag23
<210>185
<211>20
<212>dna
<213>人工序列
<400>185
cgagtaaggcctcgttccat20
<210>186
<211>20
<212>dna
<213>人工序列
<400>186
ggatgcgagtgaaacggcta20
<210>187
<211>20
<212>dna
<213>人工序列
<400>187
cctgctgcgttctcctcatc20
<210>188
<211>23
<212>dna
<213>人工序列
<400>188
gagaaaaaggatggccaggtaga23
<210>189
<211>20
<212>dna
<213>人工序列
<400>189
ctgaggaggtggcttctcat20
<210>190
<211>22
<212>dna
<213>人工序列
<400>190
tgcacagtgtcttgagtccaag22
<210>191
<211>20
<212>dna
<213>人工序列
<400>191
gggctgtgtgtgtgacacat20
<210>192
<211>22
<212>dna
<213>人工序列
<400>192
tgatgctgagaaggatttggca22
<210>193
<211>22
<212>dna
<213>人工序列
<400>193
ccctatgcctcctgtacctcta22
<210>194
<211>24
<212>dna
<213>人工序列
<400>194
agaggttcagagaagtgaagtggt24
<210>195
<211>20
<212>dna
<213>人工序列
<400>195
tgcctctcacaggtctgtct20
<210>196
<211>20
<212>dna
<213>人工序列
<400>196
tgatggaggcctgtagccta20
<210>197
<211>22
<212>dna
<213>人工序列
<400>197
ggaagccctgtacttctcactg22
<210>198
<211>23
<212>dna
<213>人工序列
<400>198
gtgggaggtgggagacaagagac23
<210>199
<211>20
<212>dna
<213>人工序列
<400>199
gctggagcgtgtgactgatg20
<210>200
<211>27
<212>dna
<213>人工序列
<400>200
caacaagaggaacgatttaagtcttgg27
<210>201
<211>19
<212>dna
<213>人工序列
<400>201
gcgtctacgccaaggacaa19
<210>202
<211>18
<212>dna
<213>人工序列
<400>202
agggagctcccacctgtt18
<210>203
<211>17
<212>dna
<213>人工序列
<400>203
tgccacccgctcctact17
<210>204
<211>17
<212>dna
<213>人工序列
<400>204
gcgtaccgaggtgagca17
<210>205
<211>17
<212>dna
<213>人工序列
<400>205
ccacaggtgtgcctgct17
<210>206
<211>19
<212>dna
<213>人工序列
<400>206
tcgaagctagtgaagcggc19
<210>207
<211>22
<212>dna
<213>人工序列
<400>207
gctgcaagcagacagatttctc22
<210>208
<211>22
<212>dna
<213>人工序列
<400>208
ctgagctgagctgagctaagac22
<210>209
<211>20
<212>dna
<213>人工序列
<400>209
cagctcgtgtcttcctgtgt20
<210>210
<211>22
<212>dna
<213>人工序列
<400>210
aactccaccatctcgtagtcct22
<210>211
<211>19
<212>dna
<213>人工序列
<400>211
ccagttccgccacaaagtc19
<210>212
<211>18
<212>dna
<213>人工序列
<400>212
cctctgtggcctggttga18
<210>213
<211>19
<212>dna
<213>人工序列
<400>213
ctggagcagcagctgcaca19
<210>214
<211>19
<212>dna
<213>人工序列
<400>214
ggtgtccactccgactcca19
- 还没有人留言评论。精彩留言会获得点赞!
- 一种引物组及其在扩增siv/shiv基因组中的应用与试剂盒的制作方法
- 检测f8基因突变的扩增引物、试剂盒及方法
- 拟茎点霉rpb2基因扩增引物及其设计方法和应用
- 一种马来穿山甲线粒体Cox3基因序列的扩增引物及鉴定方法
- 拟茎点霉gpdh基因扩增引物及其设计方法和应用
- 甲型流感病毒全基因组扩增方法及使用的复合引物和试剂盒的制作方法
- 用于检测2型糖尿病视网膜病变的miR-2116-5p分子标记物及其扩增引物和应用
- 用于检测2型糖尿病视网膜病变的miR-3197分子标记物及其扩增引物和应用
- 一种用于鱼类线粒体全基因组扩增的引物、试剂盒及扩增方法
- 用Alu重复序列基因扩增引物且无需DNA抽提的检测血浆游离DNA的定量PCR方法