一种定向改造的氨基葡萄糖合酶突变体及其应用的制作方法
本发明属于工业生物技术领域,具体涉及一种定向改造的氨基葡萄糖合酶突变体及其应用。
背景技术:
氨基葡萄糖(glucosamine,glcn),简称氨糖,存在多种有机体中,能够作为蛋白聚糖与糖蛋白的主要成分,在细胞内通过氨基化生成6-磷酸葡萄糖。n-乙酰化氨基葡萄糖(glcnac)在医药、食品以及日化等领域有着广泛的应用。传统生产工艺主要采用虾蟹来源的甲壳素为原料,较多采用酸水解进行制备,反应需要浓酸和高温条件,对环境污染严重,设备要求高,且生产成本偏高。随着代谢工程与合成生物学的迅速发展,生物发酵法运用在工业上的生产也日趋成熟,以葡萄糖为原料直接进行氨糖的转化,具有原料成本低、提取简单、产品纯度高、过程污染少等优势。
氨基葡萄糖合酶(简称氨糖合酶,gls)是glcn生物合成的关键酶,能够催化6-磷酸果糖生成glcn-6-p,由谷氨酰胺作为氨基供体。该步是glcn合成途径中第一个限速反应,gls严重受该合成途径代谢产物glcn-6-p的反馈抑制,因而glcn-6-p在正常的代谢合成中无法大量积累。为降低这种产物抑制作用,在大肠杆菌e.coli体系中通常采用共表达氨糖乙酰化酶基因,将代谢体系中的glcn-6-p转化为刺激性较小的glcnac-6-p,产物glcn-6-p和glcnac-6-p均可以从胞内转移到胞外,并经去磷酸化作用形成glcn和glcnac,而glcnac能在酸性条件下去乙酰化再次转化为glcn。
gls的生物合成能力偏低、对产物耐受能力较差是影响产量水平提升的主要原因。尽管目前工业生产上使用的菌株已远远突破上述水平,其中大都经过系统的代谢工程技术改造和发酵优化控制,但从有关文献分析,其改造的重点之一是氨糖合酶。
技术实现要素:
本发明旨在提供一种通过基因工程的方法克隆表达氨糖合酶以及通过易错pcr对氨基葡萄糖合酶进行定向改造,从而构建高产氨糖的工程菌的方法。该方法是一种简便、有效地获得dna序列变异的技术,主要是针对特定的基因,这在遗传和基因改良研究中具有巨大的应用前景。该方法构建的工程菌在工业生产中周期短、能耗少、污染小,完全适合于工业化生产的要求。
本发明为探索gls新的改造策略,首先筛选出较优的表达质粒和表达宿主,进而利用易错pcr对gls进行非理性改造,通过培养基中外加氨糖作为筛选压力,筛选出耐受性提升的突变株。此外,本发明也通过共表达氨基葡萄糖乙酰化酶gnal的方式,削弱glcn积累对宿主细胞的抑制效应,考察突变株的合成能力。
一种定向改造的氨基葡萄糖合酶突变体,其氨基酸序列如序列表seqidno.1所示。
上述定向改造的氨基葡萄糖合酶突变体的核苷酸序列如序列表seqidno.2所示。
包含所述氨基葡萄糖合酶突变体基因的工程菌。
上述定向改造的氨基葡萄糖合酶突变体在氨基葡萄糖生物合成中的应用,将突变体通过共表达氨基葡萄糖乙酰化酶,将发酵体系中的氨基葡萄糖转化为乙酰化氨基葡萄糖,从而减少产物的抑制效应,提升氨基葡萄糖产量。
一种通过基因工程手段定向改造氨基葡萄糖合酶的方法,包括克隆表达氨基葡萄糖合酶基因,并采用连续易错pcr,建立突变体文库进行高通量筛选,获得发酵性能提升的氨基葡萄糖合酶突变体m15-9。
所述定向改造的氨基葡萄糖合酶编码基因相对于天然酶60位的丙氨酸变为丝氨酸,128位的缬氨酸变为丙氨酸,352位的天冬氨酸变成了丙氨酸,354位的精氨酸变成半胱氨酸,422位的异亮氨酸变成蛋氨酸,432位的亮氨酸变成了缬氨酸,471位的天冬氨酸变成了谷氨酸,556位的亮氨酸变成了脯氨酸,567位的的缬氨酸变为谷氨酸。上述突变是多轮累积的结果,结构分析结果显示突变酶相对于野生酶疏水性明显增加,导致酶活性中心的残基向里或侧面聚集,同时突变酶的氢键加强,另外带负电荷的氨基酸明显减少,变成了中性氨基酸,减弱了酶分子的极性,更利于酶与底物结合的区域;以上变化改变了酶的空间构象,增加了酶与底物结合的区域,提高了酶与底物的结合效率。
与现有技术相比,本发明具有如下有益效果:本发明通过基因工程的方法克隆表达氨糖合酶,采用基因工程手段克隆表达氨糖合酶,通过不同表达质粒和宿主的优化筛选出最优表达体系,以大肠杆菌e.coli为例,成功构建获得e.colirosetta-gami(de3)-pet-24a-gls工程菌,检测其氨糖产量为1.63g/l。为提升重组菌对氨糖的耐受能力,采用易错pcr技术对氨糖合酶进行定向进化改造,通过高通量筛选方法从2700余株突变株中筛选获得一株高产glcn的菌株,glcn产量达到3.57g/l,相比出发菌株提高1.19倍。由于glcn的积累对于氨糖合酶重组菌生长及代谢活动具有一定的抑制作用,本发明进一步通过共表达氨糖乙酰化酶gnal将glcn转化为乙酰化氨糖glcnac从而减少产物的抑制效应,结果表明gnal与gls串联表达能显著提升菌株的发酵能力,glcn及glcnac累积产量达到7.83g/l,相比单独表达gls时提升2.19倍。经过初步摇瓶发酵优化及5l罐发酵验证,结果显示氨糖发酵水平基本稳定,经过22h发酵最高产量可达到9.85g/l。
附图说明
图1为gls基因扩增产物电泳图;
图中的m为marker,lane1和lane2为pcr扩增获得的gls基因,在1800bp左右能看到目的条带。
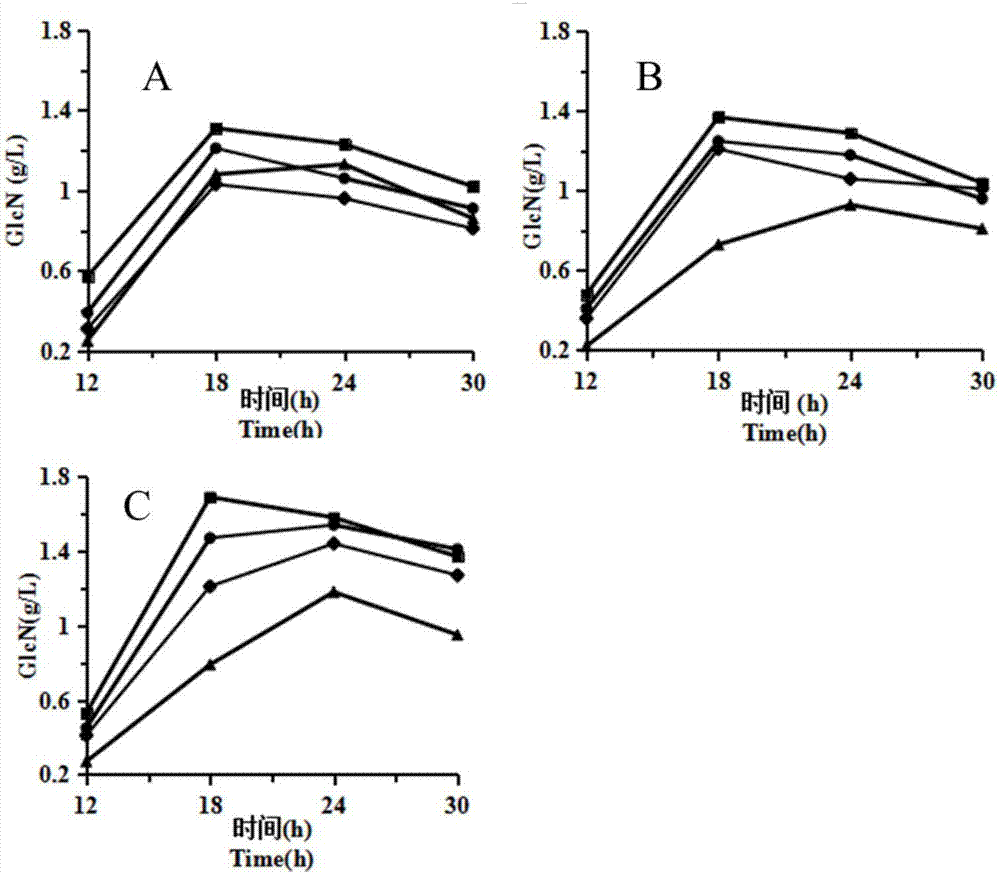
图2为表达质粒和表达宿主的优化图;
图中,●:pet-28a,■:pet-24a,▲:prsfduet-1,◆:pet-22b,图a为e.colibl21(de3)的表达宿主;图b为e.colibl21(de3)plss;图c为e.colirosetta-gami(de3)。
图3为突变株的glcn产量;
图中,●:原始菌株;■:第一轮最优突变株;▲:第二轮最优突变株;◆:第三轮最优突变株。
图4为野生酶和m15-9突变酶疏水性的对比;
图中,(a)为野生酶,(b)为m15-9突变酶。
图5为两种酶氢键的对比;
图中,(a)为野生酶,(b)为m15-9突变酶。
图6为两种酶电荷的对比;
图中(a)为野生酶,(b)为m15-9突变酶。
图7为共表达重组菌的glcn产量;
图中1为gls未突变重组菌;2为gls突变后的重组菌;3为突变后的gls与gnal共表达的重组菌。
图8为7l发酵液中的glcn和glcnac产量
图中●:glcnac,▲:glcn,■:glcnac+glcnac,◆:od600,◇:ph。
具体实施方式
下面对本发明的具体实施方式进行详细描述,但应当理解本发明的保护范围并不受具体实施方式的限制。
实施例1
根据gls基因设计引物5’端的引物为gls(1),3‘端的引物为gls(2)。根据gnal基因设计引物5’端的引物为gnal(1),3’端的引物为gnal(2),本发明中所涉及到的引物序列如表1所示。
表1本实验所用的引物
以引物gls(1)和gls(2)对gls基因进行克隆,反应采用extaq聚合酶进行扩增,体系为体系为50μl,pcr反应条件为:94℃预变性10min;94℃变性30s,62℃退火45s,72℃延伸60s,35个循环;72℃终延伸10min。pcr反应结束后,将pcr产物进行1.0%的琼脂糖凝胶电泳(图1),对目的片段进行胶回收。将gls和pmd19-t连接构建克隆质粒,将克隆质粒和表达质粒分别用bamhi和ecori进行酶切,然后通过t4连接酶反应构建重组表达质粒。
实施例2
选择四种不同的表达质粒,分别是pet-22b,pet-24a,pet-28a及prsfduet-1,表达质粒均采用ecori和bamhi进行酶切。通过t4连接酶将gls连接到表达质粒上,分别构建重组质粒pet-22b-gls,pet-24a-gls,pet-28a-gls和prsfduet-1-gls。以e.colirosetta-gami(de3),e.colibl21(de3),e.colibl21(de3)plss作为表达宿主,建立了不同的gls重组表达系统,经筛选获得的阳性克隆加入iptg进行诱导表达,结果表明以pet-24a作为表达质粒时具有最高氨糖合成能力,在e.colibl21(de3),e.colibl21(de3)plss和e.colirosetta-gami(de3)分别达到1.31g/l,1.37g/l及1.63g/l(图2)。
实施例3
为提升氨糖合酶的氨糖耐受能力和氨糖产生能力,采用易错pcr方法对氨糖合酶进行定向进化改造。经过优化的易错pcr反应体系如表2所示:
表2易错pcr的反应体系
pcr反应条件为:94℃预变性10min;94℃变性30s,56℃退火45s,72℃延伸60s,30个循环;72℃终延伸10min。pcr反应结束后,将pcr产物进行1.0%的琼脂糖凝胶电泳,对目的片段割胶回收,用elutionbuffer复溶。将pcr产物进行1.0%的琼脂糖凝胶电泳,对目的片段进行割胶,通过胶回收试剂盒回收。将gls和pet-24a用bamhi和ecori进行双酶切,然后通过t4连接酶反应构建pet-24a-gls重组质粒。
实施例4
将突变的重组质粒转化到e.colirosetta-gami(de3),转接到5ml含有25g/l氨糖的lb培养基中进行驯化培养,在37℃,220rpm的条件下培养10h。将培养后的菌液涂布于含有25g/l氨糖的lb固体平板上,放置在37℃培养箱中培养12h,实时监测菌落的生长情况。挑取转化子接入含有3%葡萄糖的tb液体培养基,在96孔深孔培养板中进行培养,同时对每个挑取的菌株在孔板上做上标。每孔对应一个阳性转化子,同时预留2孔作为对照,一孔接入出发菌株,一孔只添加培养基不接入任何菌种,培养条件为37℃、220rpm。培养至od600为0.6-0.8时加入终浓度为0.5mm的iptg进行产酶诱导,培养条件为25℃、220rpm。发酵24h后测定氨糖含量。
发酵结束后取出深孔板进行离心,然后向深孔板里面加入100μl乙酰丙酮试剂,然后将孔板固定在90℃的水浴锅中水浴1h,取出待其冷却后加入1ml的96%的乙醇,然后加入100μl的dmab试剂,然后室温显色1h后用排枪从深孔板中对应吸出200μl样品到96孔板中,然后将96孔板放在酶标仪上测量530nm处的分光光度值,筛选出高于出发菌株的编号并再次从平板上挑取出进行培养测量glcn产量。
实验以pet-24a-gls作为模板,采用易错pcr技术对gls进行体外突变时,应用高浓度glcn进行驯化以筛选突变株,在96孔板上进行高通量筛选。第一轮易错pcr从1152株克隆中筛选出产量性状得到提升的突变株22株,经复筛验证,其中的编号m4-27突变株glcn生成量为2.84g/l,产量提升了74.2%。提取该突变株的质粒作为模板,进行下一轮突变和筛选,从864株克隆中筛选出突变株15株,产量最高的一株为3.39g/l(编号m6-9);第三轮实验从691株克隆中筛选出突变株20株,最高产量达到3.57g/l(编号m15-9),其产量累计提升119%(图3)。
实施例5
将原始菌株m0-0和最优突变株m15-9接种到lb的种子培养基中,并转接到tb(3%葡萄糖)发酵培养基中在37℃、220r/min的条件下发酵至菌液的od600至0.6-0.8时,向发酵液中添加终浓度为0.5mmol/l的iptg并转移到25℃进行诱导,发酵18h后测定产量。野生菌及突变菌各项指标对比如表3所示。
表3菌株摇瓶发酵指标对比
本实施例通过调控反应体系中的mncl2和dgtp的含量控制碱基突变率为2-3‰,使每一轮的突变株中均可以获得阳性突变。每一轮突变的碱基和氨基酸位点如表4所示,多轮优势积累最终获得一株稳定高产的菌株m15-9,该突变酶成熟蛋白的核苷酸序列如seqidno.2所示,氨基酸序列如seqidno.1所示。该氨糖合酶野生酶氨基酸序列如seqidno.3所示;氨基酸序列如seqidno.4所示。
表4突变碱基和氨基酸
氨糖合酶的模型构建:通过与蛋白质数据库(pdb)内同源性高的蛋白序列分析,利用在线软件swiss-model进行同源建模,获得氨糖合酶的立体结构模型。通过序列和结构分析得知氨糖合酶的三维结构活性中心
①疏水性的对比
在酶的氨基酸残基变化后,导致如图4b左下角区域中的突变酶相对于野生酶有明显疏水性的增加,导致酶活性中心的残基向里或侧面聚集,改变了酶的空间构象,增加了酶与底物结合的区域,提高了酶的结合效率。
②氢键的对比
如图5的(a)中的野生酶中含有5个氢键,图5的(b)中含有7个氢键,活性中心的氢键加强。氢键及疏水相互作用在维系酶分子三维构象的稳定性中起着至关重要的作用。活性中心周围由氢键连接起来增强了非极性基团之间的疏水相互作用,也使得酶分子三维构象更加稳定。残基的改变导致氢键的增减和相互作用影响了酶的空间结构,从而影响了酶分子的三维构象,提升了酶和底物的催化效率。
③电荷的对比
在酶的氨基酸残基变化后,导致如图6的(b)左下角区域中的突变酶相对于野生酶带负电荷的氨基酸明显减少,变成了中性氨基酸,减弱了酶分子的极性,更利于酶与底物结合的区域,提高了酶与底物的结合效率。
实施例6
由于乙酰氨基葡萄糖对细胞刺激性小,且在下游提取过程中,用弱酸进行脱乙酰化即可转化为glcn,所以发酵产物通常是乙酰氨基葡萄糖。以引物gnal(1)和gnal(2)对gnal基因进行扩增,将pcr产物进行1.0%的琼脂糖凝胶电泳,对目的片段割胶回收,然后将gnal和pet-24a-gls用hindiii和xhoi进行双酶切,然后通过t4连接酶反应构建pet-24a-gls-gnal重组质粒。并将重组质粒导入到e.colirosetta-gami(de3)中,将验证成功后的阳性克隆子接入发酵培养基,在37℃、250r/min的条件下发酵至菌液的od600至0.6~0.8时,向发酵液中添加终浓度为0.5mmol/l的iptg并转移到25℃进行诱导,发酵18h后测定glcn和glcnac的含量,结果显示,glcn及glcnac累积产量达到8.57g/l,相比单独表达gls时产量提升2.4倍(图7)。
在7l数控式发酵罐中进行分批补糖发酵实验,初始装液量控制在4l。接种量5%,初始培养温度37℃,诱导后在25℃下进行培养。由于发酵过程中产酸,实验通过补加氨水调节ph。图8为7l发酵罐上的分批发酵过程曲线。16h时生物量od600达到最高,而glcn、glcnac浓度要到20h后才达到最高,分别为3.93g/l、5.86g/l,二者在22h累计达到最高,为9.79g/l。
以上公开的仅为本发明的具体实施例,但是,本发明并非局限于此,任何本领域的技术人员能思之的变化都应落入本发明的保护范围。
sequencelisting
<110>扬州日兴生物科技股份有限公司;江南大学
<120>一种定向改造的氨基葡萄糖合酶突变体及其应用
<130>1111
<160>4
<170>patentinversion3.5
<210>1
<211>600
<212>prt
<213>e.coli
<400>1
metcysglyilevalglytyrileglyglnleuaspalalysgluile
151015
leuleulysglyleuglulysleuglutyrargglytyraspserala
202530
glyilealavalalaasngluglnglyilehisvalphelysglulys
354045
glyargilealaaspleuarggluvalvalaspserasnvalgluala
505560
lysalaglyileglyhisthrargtrpalathrhisglygluproser
65707580
tyrleuasnalahisprohisglnseralaleuglyargphethrleu
859095
valhisasnglyvalilegluasntyrvalglnleulysglnglutyr
100105110
leuglnaspvalgluleulysseraspthraspthrgluvalvalala
115120125
glnvalilegluglnphevalasnglyglyleugluthrglugluala
130135140
phearglysthrleuthrleuleulysglysertyralailealaleu
145150155160
pheaspasnaspasnarggluthrilephevalalalysasnlysser
165170175
proleuleuvalglyleuglyaspthrpheasnvalvalalaserasp
180185190
alametalametleuglnvalthrasnglutyrvalgluleumetasp
195200205
lysglumetvalilevalthraspaspglnvalvalilelysasnleu
210215220
aspglyaspvalilethrargalasertyrilealagluleuaspala
225230235240
seraspileglulysglythrtyrprohistyrmetleulysgluthr
245250255
aspgluglnprovalvalmetarglysileileglnthrtyrglnasp
260265270
gluasnglylysleuservalproglyaspilealaalaalavalala
275280285
glualaaspargiletyrileileglycysglythrsertyrhisala
290295300
glyleuvalglylysglntyrileglumettrpalaasnvalproval
305310315320
gluvalhisvalalasergluphesertyrasnmetproleuleuser
325330335
lyslysproleupheilepheleuserglnserglygluthralaala
340345350
sercysalavalleuvalglnvallysalaleuglyhislysalaleu
355360365
thrilethrasnvalproglyserthrleuserargglualaasptyr
370375380
thrleuleuleuhisalaglyprogluilealavalalaserthrlys
385390395400
alatyrthralaglnilealavalleualavalleualaservalala
405410415
alaasplysasnglymetasnileglypheaspleuvallysgluval
420425430
glyilealaalaasnalametglualaleucysaspglnlysaspglu
435440445
metglumetilealaargglutyrleuthrvalserargasnalaphe
450455460
pheileglyargglyleuglutyrphevalcysvalgluglyalaleu
465470475480
lysleulysgluilesertyrileglnalagluglyphealaglygly
485490495
gluleulyshisglythrilealaleuilegluglnglythrproval
500505510
phealaleualathrglngluhisvalasnleuserileargglyasn
515520525
vallysgluvalalaalaargglyalaasnthrcysileileserleu
530535540
lysglyleuaspaspalaaspaspargphevalproprogluvalasn
545550555560
proalaleualaproleugluservalvalproleuglnleuileala
565570575
tyrtyralaalaleuhisargglycysaspvalasplysproargasn
580585590
leualalysservalthrvalglu
595600
<210>2
<211>1803
<212>dna
<213>e.coli
<400>2
atgtgcggcattgtgggttatatcggccagctggatgcaaaagaaattctgctgaaaggt60
ctggaaaaactggaatatcgcggttacgatagcgcgggcattgctgtcgcgaacgaacaa120
ggtatccatgtgttcaaagaaaaaggccgtattgccgatctgcgcgaagtggttgactca180
aacgtggaagccaaagcaggcatcggtcatacccgttgggcaacgcacggcgaaccgagt240
tacctgaatgctcatccgcaccagtccgcgctgggtcgttttaccctggtgcacaacggc300
gttattgaaaattatgtgcagctgaaacaagaatacctgcaagatgttgaactgaaaagc360
gataccgacacggaagtcgtggctcaggttatcgaacaatttgtcaatggcggtctggaa420
accgaagaagcgttccgcaaaaccctgacgctgctgaaaggttcttatgctattgcgctg480
tttgataacgacaatcgtgaaacgatcttcgtggccaaaaacaaatcaccgctgctggtt540
ggcctgggtgataccttcaatgtcgtggcatcggacgcgatggcgatgctgcaggtgacg600
aacgaatatgttgaactgatggataaagaaatggttattgtcaccgatgaccaagttgtc660
atcaaaaatctggatggtgacgtgattacgcgtgcaagctacatcgctgaactggatgcg720
tctgacattgaaaaaggcacctatccgcattacatgctgaaagaaacggatgaacagccg780
gtggttatgcgcaaaattatccagacctatcaagatgaaaacggtaaactgagcgttccg840
ggcgatattgcggcggcagtcgctgaagcggaccgtatctatattatcggctgtggcacg900
tcttaccatgcgggtctggtgggcaaacagtatattgaaatgtgggccaacgtgccggtt960
gaagtccacgtggcaagtgaattttcctacaatatgccgctgctgagtaaaaaaccgctg1020
tttattttcctgagccagtctggcgaaaccgccgcttcctgcgcagttctggtccaagtg1080
aaagccctgggtcataaagcactgaccatcacgaatgtgccgggctcaaccctgtcgcgt1140
gaagctgattatacgctgctgctgcacgcgggtccggaaattgccgttgcaagcaccaaa1200
gcgtatacggcacagatcgcagtcctggcagtgctggcttctgtggctgcggataaaaac1260
ggtatgaatatcggctttgacctggttaaagaagtgggcattgccgcaaacgcgatggaa1320
gccctgtgcgatcagaaagacgaaatggaaatgattgctcgtgaatatctgaccgtgagt1380
cgcaatgcctttttcatcggccgtggtctggagtattttgtttgtgtcgaaggtgccctg1440
aaactgaaagaaatttcctacatccaggctgaaggcttcgcgggcggtgaactgaaacat1500
ggtaccattgcgctgatcgaacagggcaccccggtctttgctctggcgacgcaagaacac1560
gttaacctgtcaattcgcggtaatgttaaagaagtcgctgcgcgtggcgcaaacacctgc1620
attatctcgctgaaaggtctggatgacgcggatgaccgctttgtcccgccggaagtgaat1680
ccggcactggcaccgctggagagtgtcgtgccgctgcagctgatcgcgtattacgccgca1740
ctgcatcgcggctgtgatgttgacaaaccgcgtaacctggccaaaagcgtgaccgttgaa1800
taa1803
<210>3
<211>600
<212>prt
<213>e.coli
<400>3
metcysglyilevalglytyrileglyglnleuaspalalysgluile
151015
leuleulysglyleuglulysleuglutyrargglytyraspserala
202530
glyilealavalalaasngluglnglyilehisvalphelysglulys
354045
glyargilealaaspleuarggluvalvalaspalaasnvalgluala
505560
lysalaglyileglyhisthrargtrpalathrhisglygluproser
65707580
tyrleuasnalahisprohisglnseralaleuglyargphethrleu
859095
valhisasnglyvalilegluasntyrvalglnleulysglnglutyr
100105110
leuglnaspvalgluleulysseraspthraspthrgluvalvalval
115120125
glnvalilegluglnphevalasnglyglyleugluthrglugluala
130135140
phearglysthrleuthrleuleulysglysertyralailealaleu
145150155160
pheaspasnaspasnarggluthrilephevalalalysasnlysser
165170175
proleuleuvalglyleuglyaspthrpheasnvalvalalaserasp
180185190
alametalametleuglnvalthrasnglutyrvalgluleumetasp
195200205
lysglumetvalilevalthraspaspglnvalvalilelysasnleu
210215220
aspglyaspvalilethrargalasertyrilealagluleuaspala
225230235240
seraspileglulysglythrtyrprohistyrmetleulysgluthr
245250255
aspgluglnprovalvalmetarglysileileglnthrtyrglnasp
260265270
gluasnglylysleuservalproglyaspilealaalaalavalala
275280285
glualaaspargiletyrileileglycysglythrsertyrhisala
290295300
glyleuvalglylysglntyrileglumettrpalaasnvalproval
305310315320
gluvalhisvalalasergluphesertyrasnmetproleuleuser
325330335
lyslysproleupheilepheleuserglnserglygluthralaasp
340345350
serargalavalleuvalglnvallysalaleuglyhislysalaleu
355360365
thrilethrasnvalproglyserthrleuserargglualaasptyr
370375380
thrleuleuleuhisalaglyprogluilealavalalaserthrlys
385390395400
alatyrthralaglnilealavalleualavalleualaservalala
405410415
alaasplysasnglyileasnileglypheaspleuvallysgluleu
420425430
glyilealaalaasnalametglualaleucysaspglnlysaspglu
435440445
metglumetilealaargglutyrleuthrvalserargasnalaphe
450455460
pheileglyargglyleuasptyrphevalcysvalgluglyalaleu
465470475480
lysleulysgluilesertyrileglnalagluglyphealaglygly
485490495
gluleulyshisglythrilealaleuilegluglnglythrproval
500505510
phealaleualathrglngluhisvalasnleuserileargglyasn
515520525
vallysgluvalalaalaargglyalaasnthrcysileileserleu
530535540
lysglyleuaspaspalaaspaspargphevalleuprogluvalasn
545550555560
proalaleualaproleuvalservalvalproleuglnleuileala
565570575
tyrtyralaalaleuhisargglycysaspvalasplysproargasn
580585590
leualalysservalthrvalglu
595600
<210>4
<211>1803
<212>dna
<213>e.coli
<400>4
atgtgcggcattgtgggttatatcggccagctggatgcaaaagaaattctgctgaaaggt60
ctggaaaaactggaatatcgcggttacgatagcgcgggcattgctgtcgcgaacgaacaa120
ggtatccatgtgttcaaagaaaaaggccgtattgccgatctgcgcgaagtggttgacgca180
aacgtggaagccaaagcaggcatcggtcatacccgttgggcaacgcacggcgaaccgagt240
tacctgaatgctcatccgcaccagtccgcgctgggtcgttttaccctggtgcacaacggc300
gttattgaaaattatgtgcagctgaaacaagaatacctgcaagatgttgaactgaaaagc360
gataccgacacggaagtcgtggttcaggttatcgaacaatttgtcaatggcggtctggaa420
accgaagaagcgttccgcaaaaccctgacgctgctgaaaggttcttatgctattgcgctg480
tttgataacgacaatcgtgaaacgatcttcgtggccaaaaacaaatcaccgctgctggtt540
ggcctgggtgataccttcaatgtcgtggcatcggacgcgatggcgatgctgcaggtgacg600
aacgaatatgttgaactgatggataaagaaatggttattgtcaccgatgaccaagttgtc660
atcaaaaatctggatggtgacgtgattacgcgtgcaagctacatcgctgaactggatgcg720
tctgacattgaaaaaggcacctatccgcattacatgctgaaagaaacggatgaacagccg780
gtggttatgcgcaaaattatccagacctatcaagatgaaaacggtaaactgagcgttccg840
ggcgatattgcggcggcagtcgctgaagcggaccgtatctatattatcggctgtggcacg900
tcttaccatgcgggtctggtgggcaaacagtatattgaaatgtgggccaacgtgccggtt960
gaagtccacgtggcaagtgaattttcctacaatatgccgctgctgagtaaaaaaccgctg1020
tttattttcctgagccagtctggcgaaaccgccgattcccgcgcagttctggtccaagtg1080
aaagccctgggtcataaagcactgaccatcacgaatgtgccgggctcaaccctgtcgcgt1140
gaagctgattatacgctgctgctgcacgcgggtccggaaattgccgttgcaagcaccaaa1200
gcgtatacggcacagatcgcagtcctggcagtgctggcttctgtggctgcggataaaaac1260
ggtattaatatcggctttgacctggttaaagaactgggcattgccgcaaacgcgatggaa1320
gccctgtgcgatcagaaagacgaaatggaaatgattgctcgtgaatatctgaccgtgagt1380
cgcaatgcctttttcatcggccgtggtctggattattttgtttgtgtcgaaggtgccctg1440
aaactgaaagaaatttcctacatccaggctgaaggcttcgcgggcggtgaactgaaacat1500
ggtaccattgcgctgatcgaacagggcaccccggtctttgctctggcgacgcaagaacac1560
gttaacctgtcaattcgcggtaatgttaaagaagtcgctgcgcgtggcgcaaacacctgc1620
attatctcgctgaaaggtctggatgacgcggatgaccgctttgtcctgccggaagtgaat1680
ccggcactggcaccgctggtgagtgtcgtgccgctgcagctgatcgcgtattacgccgca1740
ctgcatcgcggctgtgatgttgacaaaccgcgtaacctggccaaaagcgtgaccgttgaa1800
taa1803
- 还没有人留言评论。精彩留言会获得点赞!
- 一种氨基葡萄糖‑6磷酸合成酶突变体的制作方法与工艺
- 海洋α‑环糊精葡萄糖基转移酶转化淀粉制备α‑环糊精的方法与流程
- 一种高效合成乙酰氨基葡萄糖的重组枯草芽孢杆菌的制作方法与工艺
- N‑乙酰‑D‑氨基葡萄糖及其药物可接受盐在制备治疗病毒性肝炎药物中的应用及药物的制作方法与工艺
- N‑乙酰‑D‑氨基葡萄糖在制备治疗脱发和乌发药物、保健品中的应用及药物的制作方法与工艺
- 一种提高N‑乙酰氨基葡萄糖产量的发酵方法与流程
- 提高乙酰氨基葡萄糖产量的重组枯草芽孢杆菌及其构建方法与流程
- 一种提高乙酰氨基葡萄糖产量的重组枯草芽孢杆菌及其构建方法与流程
- 高产乙酰氨基葡萄糖的重组枯草芽孢杆菌及其构建方法与流程
- 微生物发酵生产N‑乙酰‑D‑氨基葡萄糖和/或D‑氨基葡萄糖盐的方法与流程
- 一种乙酰氨基葡萄糖苷酶检测试剂、反应垫、其制备方法及试剂盒与流程
- 一种氨基葡萄糖‑6磷酸合成酶突变体的制作方法与工艺
- 海洋α‑环糊精葡萄糖基转移酶转化淀粉制备α‑环糊精的方法与流程
- 一种高效合成乙酰氨基葡萄糖的重组枯草芽孢杆菌的制作方法与工艺
- N‑乙酰‑D‑氨基葡萄糖及其药物可接受盐在制备治疗病毒性肝炎药物中的应用及药物的制作方法与工艺
- N‑乙酰‑D‑氨基葡萄糖在制备治疗脱发和乌发药物、保健品中的应用及药物的制作方法与工艺
- 提高乙酰氨基葡萄糖产量的重组枯草芽孢杆菌及其构建方法与流程
- 一种提高乙酰氨基葡萄糖产量的重组枯草芽孢杆菌及其构建方法与流程
- 高产乙酰氨基葡萄糖的重组枯草芽孢杆菌及其构建方法与流程
- 微生物发酵生产N‑乙酰‑D‑氨基葡萄糖和/或D‑氨基葡萄糖盐的方法与流程