一种基于格氏嗜盐碱杆菌Argonaute靶向激活基因转录的方法与流程
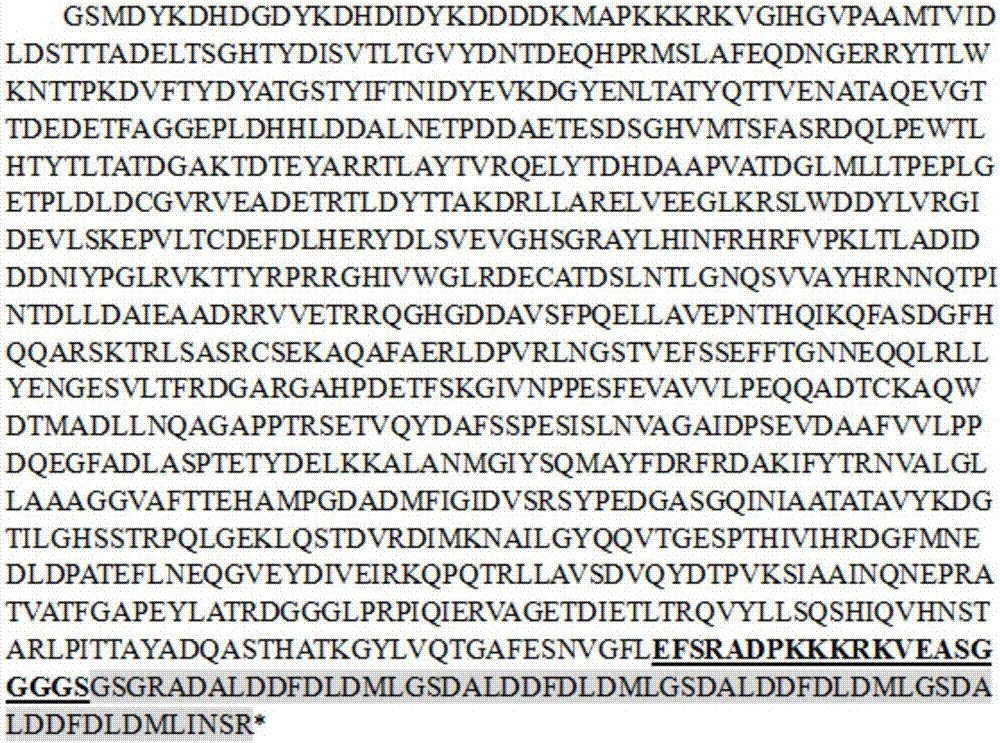
本发明属于生物技术领域。更具体地,涉及一种基于格氏嗜盐碱杆菌argonaute(ngago)靶向激活基因转录的方法。
背景技术:
基因编辑技术指能够让人类对目标基因进行“编辑”,实现对特定dna片段的敲除、加入等。在基础研究、基因治疗和遗传改良等方面展示出了巨大的潜力。因此,基因组编辑工具具有极大的理论和临床应用价值。
目前,基于zfn和cas9基因编辑工具的针对艾滋病和肿瘤的治疗新手段已经进入临床实验。并且,初期临床实验结果证明了其有效性。相对于zfn的基于蛋白与dna识别的基因组编辑工具,cas9作为基于rna与dna碱基配对识别机制,具有易于操作、利于大批量构建等优点。但由于cas9需要rna具有特定二级结构并且识别dna位点必须具有pam结构,因此对识别位点具有一定选择性,在一定程度上限制了其应用的范围。
另外,基于dna与dna识别的、无二级结构需求的格氏嗜盐碱杆菌(natronobacteriumgregoryi)argonaute(ngago)基因组编辑工具,具有更加广泛的应用前景。但是,目前对于ngago的基因组编辑功能,大家仍持有怀疑态度,因为国内国际上的众多科学家都无法重复这个发现。鉴于基于ngago的基因组编辑工具具有更加广泛的潜在应用前景,国内国际上众多科学家都试图开发这款工具,但都以失败告终。
技术实现要素:
本发明要解决的技术问题是克服现有ngago基因组编辑工具技术和应用的缺陷及不足,我们的研究发现,格氏嗜盐碱杆菌(natronobacteriumgregoryi)argonaute(ngago)具有结合导向dna的能力,并具有识别靶点dna的能力,但缺乏dna切割能力。因此,本发明将ngago与vp64构建成融合蛋白,该蛋白在细胞内具有激活内源基因的能力。本发明解决了近期大家对ngago应用的问题,并为后期开发为靶向基因组编辑工具和其它工具提供了支撑。
本发明的目的是提供一种ngago-vp64融合蛋白及其在基因编辑或在制备ngago基因组编辑工具方面的应用。
本发明另一目的是提供一种基于格氏嗜盐碱杆菌argonaute(ngago)靶向激活基因转录的方法。
本发明上述目的通过以下技术方案实现:
一种ngago-vp64融合蛋白,是由vp64与ngago融合表达获得。具体是将vp64融合到ngago的c末端(记为ngc)或n末端(记为ngn),并进行人源化密码子优化所得。
所述融合蛋白的序列如seqidno.1或seqidno.2所示。
基于所述ngago-vp64融合蛋白的结构进行的类似蛋白序列改变且具有相同功能的蛋白,也应在本发明范围之内。
一种表达ngago-vp64融合蛋白的质粒,是将ngago-vp64融合蛋白基因克隆到表达载体获得。
所述表达载体包括,但不局限于瞬时表达载体、病毒载体,真核表达载体和原核表达载体,具体如表达载体pcdna3.1。
一种导向dna(gdna),为seqidno.3~12所示序列片段中的任一种或几种的混合物。
优选地,所述导向dna为seqidno.4、seqidno.7、seqidno.9、seqidno.12任一所示序列片段,或seqidno.3~7所示序列片段的混合物,或seqidno.8~12所示序列片段的混合物。
上述ngago-vp64融合蛋白、质粒或导向dna在基因编辑方面的应用,以及在制备ngago基因编辑工具方面的应用,都应在本发明的保护范围之内。
具体地,所述基因编辑是指激活内源基因的表达。其应用物种,不仅仅限制于人类细胞,还包括其他物种。
一种基于格氏嗜盐碱杆菌argonaute靶向激活基因转录的ngago基因编辑工具,包括上述ngago-vp64融合蛋白或质粒,还包括上述导向dna(gdna)。
一种基于格氏嗜盐碱杆菌argonaute靶向激活内源基因转录的方法,是上述ngago基因编辑工具与转染试剂共转染细胞。
具体地,该方法包括如下步骤:
s1.细胞传代培养至指数增长期,转染前0.5~2小时(优选1小时),将细胞培养基换为无血清培养基,孵育0.5~2小时(优选1小时);
s2.将ngago-vp64融合蛋白质粒和导向dna稀释到无血清培养基中,转染试剂稀释到无血清培养基中,然后将两者混合,室温静置20~30分钟(优选25分钟)后,加入细胞中,2~4小时(优选3小时)后加入血清至终浓度为8~15%(优选10%);
s3.第二天重复转染,但只需转染导向dna(gdna)。
其中,步骤s2所述ngago-vp64融合蛋白的序列如seqidno.1或seqidno.2所示。
优选地,步骤s2所述导向dna为seqidno.4、seqidno.7、seqidno.9、seqidno.12任一所示序列片段,或seqidno.3~7所示序列片段的混合物,或seqidno.8~12所示序列片段的混合物。
优选地,步骤s2中ngago-vp64融合蛋白:导向dna:无血清培养基=0.5~2μg:0.5~2μl:100μl。
更优选地,步骤s2中ngago-vp64融合蛋白:导向dna:无血清培养基=1μg:1μl:100μl。
基于现有技术的研究结果以及我们自己前期的探索研究,我们推测目前无法将细菌ago开发为基因组编辑工具的可能原因为两点:(1)ngago在哺乳动物细胞内不具有结合导向dna并具有识别靶点基因组dna的能力;(2)ngago在哺乳动物细胞内具有结合导向dna并具有识别靶点基因组dna的能力,但不具有dna切割活性。
为了验证原因(2),我们将ngago与转录激活蛋白vp64进行融合表达,并在哺乳细胞内表达。研究结果发现,ngago与vp64的融合蛋白可以激活细胞内基因nanog,进而证实ngago在哺乳动物细胞内具有结合导向dna并具有识别靶点基因组dna的能力,但不具有dna切割活性。
本发明具有以下有益效果:
本发明证明了ngago具有结合导向dna,并具有识别靶点基因组dna的能力。首次发现ngago与vp64构建成的融合蛋白,在细胞内具有激活内源基因的能力。在基于该发明的基础上,可以进一步将ngago与dna切割酶融合(例如iseci和foki)并开发为靶向基因组编辑工具或其它工具。
本发明解决了近期大家对ngago应用的怀疑观望问题,并为后期开发为靶向基因组编辑工具和其它工具提供了支撑。
附图说明
图1为ngc和ngn的基因结构示意图。
图2为ngago-vp64(ngc)的序列组成;黑色非加粗部分为ngago蛋白序列,加粗且下划线部分为核定位信号,灰色阴影部分为vp64序列。
图3为vp64-ngago(ngn)的序列组成;黑色非加粗部分为ngago蛋白序列;加粗且下划线部分为核定位信号,灰色阴影部分为vp64序列。
图4为ngago与vp64融合蛋白可激活细胞内nanog基因转录;(a)qpcr分析ngc激活细胞内nanog基因转录水平,(b)qpcr分析ngn激活细胞内nanog基因转录水平;ngc代表vp64融合到ngago的c端;ngn代表vp64融合到ngago的n端;n1、n2、n3、n4、n5、n6、n7、n8、n9、n10代表针对nanog的不同导向dna。n1-5代表n1、n2、n3、n4和n5的等体积混合物;n6-10代表n6、n7、n8、n9和n10的等体积混合物;blank代表转染时不加入ngc、ngn或者导向dna。*代表显著性差异p值小于0.01。
图5为ngc和ngn都可以激活细胞nanog基因转录;ngc代表vp64融合到ngago的c端;ngn代表vp64融合到ngago的n端;n10代表针对nanog的导向dna;ng代表没有融合vp64的ngago基因;blank代表转染时不加入ngc、ngn或者导向dna。*代表显著性差异p值小于0.01。
具体实施方式
以下结合说明书附图和具体实施例来进一步说明本发明,但实施例并不对本发明做任何形式的限定。除非特别说明,本发明采用的试剂、方法和设备为本技术领域常规试剂、方法和设备。
除非特别说明,以下实施例所用试剂和材料均为市购。
以下实施例以nanog基因为例,进行利用ngc(将vp64融合到ngago的c末端)和ngn(将vp64融合到ngago的n末端)激活细胞内基因研究。实施例1构建ngago-vp64融合蛋白
1、将vp64与ngago融合表达
为了防止蛋白相对位置对蛋白活性的影响,分别将vp64融合到ngago的c末端(ngc)和n末端(ngn),蛋白序列分别如seqidno.1和seqidno.2所示(基因结构示意图如图1所示,序列组成分别如图2和图3所示)。
2、将该蛋白序列进行人源化密码子优化后,分别克隆到哺乳动物表达载体pcdna3.1上。质粒经测序验证正确后,质粒大提后用于后续实验。
实施例2导向dna合成与磷酸化
1、设计导向dna序列,如表1所示:
表1
导向dna(gdna)长度为24bp,合成后用1xt4pnk缓冲液(neb)溶解至100μm。每1nmolgdna用5单位t4pnk进行磷酸化,37℃孵育过夜。然后储存至-20℃,备用。
实施例3由ngago-vp64融合蛋白和导向dna构成的ngago基因组编辑工具及其应用
1、一种基于格氏嗜盐碱杆菌argonaute靶向激活基因转录的ngago基因组编辑工具,包括实施例1所述的ngago-vp64融合蛋白和实施例2所述的导向dna。
2、ngago基因组编辑工具激活基因转录实验
(1)细胞培养与转染
转染前一天,对细胞进行传代培养,使转染时细胞为指数增长期。以细胞hela为例,12孔板中每孔可铺细胞10万。转染前一小时,将细胞培养基换为无血清培养基孵育1小时。利用lipofactamine2000转染试剂进行转染。
以12孔板中每孔的量为例,将1μg融合蛋白质粒与1μlgdna稀释到100μlopti-mem中;将2μllipofactamine2000稀释到100μlopti-mem中,室温静置5分钟。然后将两者混合,室温静置25分钟后,加入细胞中,3小时后加入血清至终浓度为10%。
第二天重复转染过程,但只需转染gdna即可。转染后48小时,细胞用于后续分析。
(2)rna提取、cdna反转录以及实时定量pcr检测
转染48小时后,细胞用trizol(thermoscientific)进行裂解,经氯仿进一步裂解后,12000rpm离心15分钟,取上清并于等体积异丙醇混合。12000rpm离心15分钟,去上清,并用75%乙醇清洗沉淀,得rna沉淀。rna沉淀用depc水溶解后定量。
取2μgrna进行cdna反转录,反应体系为mmlv逆转录酶和随机引物(thermoscientific),共20μl。反转录完成后,将cdna稀释4倍,用于实时定量pcr分析。所用试剂为takarasybg试剂盒,仪器为bioradcfx96。基因表达分析利用2-δδct方法。
其中,用于qpcr的引物如表2所示:
表2qpcr引物
(3)实验结果
图4中a图所示结果显示,位点n2、n5、n7、n10、n1-5、n6-10可以显著激活nanog基因转录;b图所示结果显示,位点n9、n10可以显著激活nanog基因转录。图5所示结果显示,ngc和ngn都可以激活细胞nanog基因转录。
sequencelisting
<110>深圳大学
<120>一种基于格氏嗜盐碱杆菌argonaute靶向激活基因转录的方法
<130>
<160>16
<170>patentinversion3.3
<210>1
<211>1009
<212>prt
<213>ngago-vp64(ngc)
<400>1
glysermetasptyrlysasphisaspglyasptyrlysasphisasp
151015
ileasptyrlysaspaspaspasplysmetalaprolyslyslysarg
202530
lysvalglyilehisglyvalproalaalametthrvalileaspleu
354045
aspserthrthrthralaaspgluleuthrserglyhisthrtyrasp
505560
ileservalthrleuthrglyvaltyraspasnthraspgluglnhis
65707580
proargmetserleualaphegluglnaspasnglygluargargtyr
859095
ilethrleutrplysasnthrthrprolysaspvalphethrtyrasp
100105110
tyralathrglyserthrtyrilephethrasnileasptyrgluval
115120125
lysaspglytyrgluasnleuthralathrtyrglnthrthrvalglu
130135140
asnalathralaglngluvalglythrthraspgluaspgluthrphe
145150155160
alaglyglygluproleuasphishisleuaspaspalaleuasnglu
165170175
thrproaspaspalagluthrgluseraspserglyhisvalmetthr
180185190
serphealaserargaspglnleuproglutrpthrleuhisthrtyr
195200205
thrleuthralathraspglyalalysthraspthrglutyralaarg
210215220
argthrleualatyrthrvalargglngluleutyrthrasphisasp
225230235240
alaalaprovalalathraspglyleumetleuleuthrproglupro
245250255
leuglygluthrproleuaspleuaspcysglyvalargvalgluala
260265270
aspgluthrargthrleuasptyrthrthralalysaspargleuleu
275280285
alaarggluleuvalglugluglyleulysargserleutrpaspasp
290295300
tyrleuvalargglyileaspgluvalleuserlysgluprovalleu
305310315320
thrcysaspglupheaspleuhisgluargtyraspleuservalglu
325330335
valglyhisserglyargalatyrleuhisileasnphearghisarg
340345350
phevalprolysleuthrleualaaspileaspaspaspasniletyr
355360365
proglyleuargvallysthrthrtyrargproargargglyhisile
370375380
valtrpglyleuargaspglucysalathraspserleuasnthrleu
385390395400
glyasnglnservalvalalatyrhisargasnasnglnthrproile
405410415
asnthraspleuleuaspalaileglualaalaaspargargvalval
420425430
gluthrargargglnglyhisglyaspaspalavalserpheprogln
435440445
gluleuleualavalgluproasnthrhisglnilelysglnpheala
450455460
seraspglyphehisglnglnalaargserlysthrargleuserala
465470475480
serargcysserglulysalaglnalaphealagluargleuasppro
485490495
valargleuasnglyserthrvalgluphesersergluphephethr
500505510
glyasnasngluglnglnleuargleuleutyrgluasnglygluser
515520525
valleuthrpheargaspglyalaargglyalahisproaspgluthr
530535540
pheserlysglyilevalasnproprogluserphegluvalalaval
545550555560
valleuprogluglnglnalaaspthrcyslysalaglntrpaspthr
565570575
metalaaspleuleuasnglnalaglyalaproprothrargserglu
580585590
thrvalglntyraspalapheserserprogluserileserleuasn
595600605
valalaglyalaileaspprosergluvalaspalaalaphevalval
610615620
leuproproaspglngluglyphealaaspleualaserprothrglu
625630635640
thrtyraspgluleulyslysalaleualaasnmetglyiletyrser
645650655
glnmetalatyrpheaspargpheargaspalalysilephetyrthr
660665670
argasnvalalaleuglyleuleualaalaalaglyglyvalalaphe
675680685
thrthrgluhisalametproglyaspalaaspmetpheileglyile
690695700
aspvalserargsertyrprogluaspglyalaserglyglnileasn
705710715720
ilealaalathralathralavaltyrlysaspglythrileleugly
725730735
hisserserthrargproglnleuglyglulysleuglnserthrasp
740745750
valargaspilemetlysasnalaileleuglytyrglnglnvalthr
755760765
glygluserprothrhisilevalilehisargaspglyphemetasn
770775780
gluaspleuaspproalathrglupheleuasngluglnglyvalglu
785790795800
tyraspilevalgluilearglysglnproglnthrargleuleuala
805810815
valseraspvalglntyraspthrprovallysserilealaalaile
820825830
asnglnasngluproargalathrvalalathrpheglyalaproglu
835840845
tyrleualathrargaspglyglyglyleuproargproileglnile
850855860
gluargvalalaglygluthraspilegluthrleuthrargglnval
865870875880
tyrleuleuserglnserhisileglnvalhisasnserthralaarg
885890895
leuproilethrthralatyralaaspglnalaserthrhisalathr
900905910
lysglytyrleuvalglnthrglyalaphegluserasnvalglyphe
915920925
leuglupheserargalaaspprolyslyslysarglysvalgluala
930935940
serglyglyglyglyserglyserglyargalaaspalaleuaspasp
945950955960
pheaspleuaspmetleuglyseraspalaleuaspasppheaspleu
965970975
aspmetleuglyseraspalaleuaspasppheaspleuaspmetleu
980985990
glyseraspalaleuaspasppheaspleuaspmetleuileasnser
99510001005
arg
<210>2
<211>1034
<212>prt
<213>vp64-ngago(ngn)
<400>2
metserargalaaspprolyslyslysarglysvalglualasergly
151015
glyglyglyserglyserglyargalaaspalaleuaspasppheasp
202530
leuaspmetleuglyseraspalaleuaspasppheaspleuaspmet
354045
leuglyseraspalaleuaspasppheaspleuaspmetleuglyser
505560
aspalaleuaspasppheaspleuaspmetleuileasnserarglys
65707580
leuglyserleuvalproserseraspproleuvalthralaalaser
859095
valleuglupheglnserthrglyproglysermetasptyrlysasp
100105110
hisaspglyasptyrlysasphisaspileasptyrlysaspaspasp
115120125
asplysmetalaprolyslyslysarglysvalglyilehisglyval
130135140
proalaalametthrvalileaspleuaspserthrthrthralaasp
145150155160
gluleuthrserglyhisthrtyraspileservalthrleuthrgly
165170175
valtyraspasnthraspgluglnhisproargmetserleualaphe
180185190
gluglnaspasnglygluargargtyrilethrleutrplysasnthr
195200205
thrprolysaspvalphethrtyrasptyralathrglyserthrtyr
210215220
ilephethrasnileasptyrgluvallysaspglytyrgluasnleu
225230235240
thralathrtyrglnthrthrvalgluasnalathralaglngluval
245250255
glythrthraspgluaspgluthrphealaglyglygluproleuasp
260265270
hishisleuaspaspalaleuasngluthrproaspaspalagluthr
275280285
gluseraspserglyhisvalmetthrserphealaserargaspgln
290295300
leuproglutrpthrleuhisthrtyrthrleuthralathraspgly
305310315320
alalysthraspthrglutyralaargargthrleualatyrthrval
325330335
argglngluleutyrthrasphisaspalaalaprovalalathrasp
340345350
glyleumetleuleuthrprogluproleuglygluthrproleuasp
355360365
leuaspcysglyvalargvalglualaaspgluthrargthrleuasp
370375380
tyrthrthralalysaspargleuleualaarggluleuvalgluglu
385390395400
glyleulysargserleutrpaspasptyrleuvalargglyileasp
405410415
gluvalleuserlysgluprovalleuthrcysaspglupheaspleu
420425430
hisgluargtyraspleuservalgluvalglyhisserglyargala
435440445
tyrleuhisileasnphearghisargphevalprolysleuthrleu
450455460
alaaspileaspaspaspasniletyrproglyleuargvallysthr
465470475480
thrtyrargproargargglyhisilevaltrpglyleuargaspglu
485490495
cysalathraspserleuasnthrleuglyasnglnservalvalala
500505510
tyrhisargasnasnglnthrproileasnthraspleuleuaspala
515520525
ileglualaalaaspargargvalvalgluthrargargglnglyhis
530535540
glyaspaspalavalserpheproglngluleuleualavalglupro
545550555560
asnthrhisglnilelysglnphealaseraspglyphehisglngln
565570575
alaargserlysthrargleuseralaserargcysserglulysala
580585590
glnalaphealagluargleuaspprovalargleuasnglyserthr
595600605
valgluphesersergluphephethrglyasnasngluglnglnleu
610615620
argleuleutyrgluasnglygluservalleuthrpheargaspgly
625630635640
alaargglyalahisproaspgluthrpheserlysglyilevalasn
645650655
proprogluserphegluvalalavalvalleuprogluglnglnala
660665670
aspthrcyslysalaglntrpaspthrmetalaaspleuleuasngln
675680685
alaglyalaproprothrargsergluthrvalglntyraspalaphe
690695700
serserprogluserileserleuasnvalalaglyalaileasppro
705710715720
sergluvalaspalaalaphevalvalleuproproaspglnglugly
725730735
phealaaspleualaserprothrgluthrtyraspgluleulyslys
740745750
alaleualaasnmetglyiletyrserglnmetalatyrpheasparg
755760765
pheargaspalalysilephetyrthrargasnvalalaleuglyleu
770775780
leualaalaalaglyglyvalalaphethrthrgluhisalametpro
785790795800
glyaspalaaspmetpheileglyileaspvalserargsertyrpro
805810815
gluaspglyalaserglyglnileasnilealaalathralathrala
820825830
valtyrlysaspglythrileleuglyhisserserthrargprogln
835840845
leuglyglulysleuglnserthraspvalargaspilemetlysasn
850855860
alaileleuglytyrglnglnvalthrglygluserprothrhisile
865870875880
valilehisargaspglyphemetasngluaspleuaspproalathr
885890895
glupheleuasngluglnglyvalglutyraspilevalgluilearg
900905910
lysglnproglnthrargleuleualavalseraspvalglntyrasp
915920925
thrprovallysserilealaalaileasnglnasngluproargala
930935940
thrvalalathrpheglyalaproglutyrleualathrargaspgly
945950955960
glyglyleuproargproileglnilegluargvalalaglygluthr
965970975
aspilegluthrleuthrargglnvaltyrleuleuserglnserhis
980985990
ileglnvalhisasnserthralaargleuproilethrthralatyr
99510001005
alaaspglnalaserthrhisalathrlysglytyrleuvalgln
101010151020
thrglyalaphegluserasnvalglypheleu
10251030
<210>3
<211>24
<212>dna
<213>gdna序列n1
<400>3
cacacacacccacacgagatgggc24
<210>4
<211>24
<212>dna
<213>gdna序列n2
<400>4
ggaagaagctaaagagccagaggg24
<210>5
<211>24
<212>dna
<213>gdna序列n3
<400>5
atgagaatttcaataacctcagga24
<210>6
<211>24
<212>dna
<213>gdna序列n4
<400>6
gtcccgctctgttgcccaggctgg24
<210>7
<211>24
<212>dna
<213>gdna序列n5
<400>7
cagacacccaccaccatgcgtggc24
<210>8
<211>24
<212>dna
<213>gdna序列n6
<400>8
tcccaatttactgggattacaggg24
<210>9
<211>24
<212>dna
<213>gdna序列n7
<400>9
ctgatttaaaagttggaaacgtgg24
<210>10
<211>24
<212>dna
<213>gdna序列n8
<400>10
tctagttccccacctagtctgggt24
<210>11
<211>24
<212>dna
<213>gdna序列n9
<400>11
ggattaactgagaattcacaaggg24
<210>12
<211>24
<212>dna
<213>gdna序列n10
<400>12
ccgccaggaggggtgggtctaagg24
<210>13
<211>20
<212>dna
<213>基因nanog正向引物
<400>13
tgaacctcagctacaaacag20
<210>14
<211>20
<212>dna
<213>基因nanog反向引物
<400>14
tggtggtaggaagagtaaag20
<210>15
<211>17
<212>dna
<213>内参β-actin正向引物
<400>15
gttgtcgacgacgagcg17
<210>16
<211>17
<212>dna
<213>内参β-actin反向引物
<400>16
gcacagagcctcgcctt17
- 还没有人留言评论。精彩留言会获得点赞!
- 靶向PD-1的多肽及其应用的制造方法与工艺
- 靶向同位素生产系统的制造方法与工艺
- 在制备68GA‑螯合物官能化的靶向剂中用作金属抑制剂的单糖、二糖或多糖的制造方法与工艺
- 用于引导和靶向按需物质递送的纳米结构的载体的制造方法与工艺
- 通过PROSTRATIN靶向K‑RAS介导的信号转导通路和恶性肿瘤的制造方法与工艺
- 肿瘤靶向核磁共振/荧光双模态成像造影剂及其制备和应用的制造方法与工艺
- pH氧化还原双敏感的PAMAM靶向纳米给药载体及其制备方法与制造工艺
- 具有卵巢肿瘤靶向D构型多肽及其基因递释系统的制造方法与工艺
- 靶向survivin纳米抗体及其制备方法和应用与制造工艺
- 用于PSMA靶向的成像和放射疗法的金属/放射性金属标记的PSMA抑制物的制造方法与工艺
- 阻断UBE2M介导Neddylation修饰在肾细胞癌治疗中的应用的制造方法与工艺
- 无载体共组装肿瘤靶向抗癌纳米药物及其制备方法与应用与流程
- 一种用于判断靶向单克隆抗体治疗肿瘤疗效的预测分子的制造方法与工艺
- 一种靶向CD47的免疫检查点抑制剂药物组合物及其制备方法与流程
- 具有淋巴靶向功能的生物自组装纳米晶注射剂及制备方法与流程
- 用于检测肝癌的基因标志物及其用途的制造方法与工艺
- 特异性shRNA筛选及其靶向Ang‑2基因抑制肺癌细胞的验证方法与流程
- 一种多功能自动化生物细胞提取分离控制系统及采用该系统实现的细胞提取分离方法与流程
- 一种融合蛋白及光敏剂复合物及其制备方法和应用与流程
- 具有可视化和显示光斑边界功能的光动力治疗系统的制造方法与工艺
- 肿瘤靶向核磁共振/荧光双模态成像造影剂及其制备和应用的制造方法与工艺
- pH氧化还原双敏感的PAMAM靶向纳米给药载体及其制备方法与制造工艺
- 具有卵巢肿瘤靶向D构型多肽及其基因递释系统的制造方法与工艺
- 靶向survivin纳米抗体及其制备方法和应用与制造工艺
- 用于PSMA靶向的成像和放射疗法的金属/放射性金属标记的PSMA抑制物的制造方法与工艺
- 一种线粒体靶向的粘度荧光探针及其制备方法和应用与制造工艺
- 一种兼具荧光可视、磁靶向和pH敏多功能的复合空心微球药物载体及其制备方法与制造工艺
- 一种用于修饰微泡的多肽以及靶向GBM的药物制剂的制造方法与工艺
- 白果内酯作为脑靶向增效剂在制备防治脑肿瘤药物的应用的制造方法与工艺
- 机器人辅助精确靶向穿刺软组织目标柔性针夹紧固定装置的制造方法
- 一种用于判断靶向单克隆抗体治疗肿瘤疗效的预测分子的制造方法与工艺
- 一种检测肺癌相关基因热点突变的试剂盒及其应用的制造方法与工艺
- 一种为肺癌提供离体个体化药物测试的方法及培养基与流程
- 一种谷胱甘肽/超声双重刺激响应纳米聚合物囊泡、其制备方法及应用与流程
- 一种多重刺激响应交联型聚合物纳米水凝胶、其制备方法及应用与流程
- 一种靶向CD47的免疫检查点抑制剂药物组合物及其制备方法与流程
- 具有淋巴靶向功能的生物自组装纳米晶注射剂及制备方法与流程
- 用于检测肝癌的基因标志物及其用途的制造方法与工艺
- 特异性shRNA筛选及其靶向Ang‑2基因抑制肺癌细胞的验证方法与流程
- 一种具有靶向功能仿病毒结构高分子囊泡及其制备与应用的制造方法与工艺