一种促进微生物细胞转运葡萄糖、木糖和阿拉伯糖的方法及其在生物基产品发酵中的应用与流程
技术领域
本发明属于生物技术领域,促进微生物细胞转运葡萄糖、木糖和阿拉伯糖的方法。主要涉及转运蛋白(GLT-1,XYT-1,XAT-1,LAT-1和MtLAT-1)和编码基因以及在导入微生物菌株后使其获得或提高葡萄糖、木糖或阿拉伯糖转运能力及其在微生物发酵生产生物基产品中的应用。
背景技术:
生物质是地球上数量最大的可再生资源,同时也是分布最为广泛的碳水化合物。面临能源危机及资源短缺,利用可再生的生物质生产生物能源为人类可持续发展提供了希望。生物质主要由纤维素,半纤维素及木质素构成,其降解物主要包括葡萄糖,木糖,阿拉伯糖等单糖及部分寡糖。
为提高对生物质降解物的利用率,研究者对多种微生物进行了代谢工程改造。其中工程酿酒酵母具有易培养,产率高,对代谢抑制物及乙醇具有较高的耐受性以及研究背景清楚,遗传操作简单的特点,成为了利用生物质降解物进行乙醇发酵的主要菌株。但是由于很多发酵工业重要微生物如酿酒酵母不能利用木糖及阿拉伯糖,使其有效的利用生物质降解物进行生物基化学品(乙醇、丁醇等)发酵生产受到了限制。随后,虽然研究者的不断努力,将真菌或细菌的代谢途径经过遗传改造的导入构建工程菌株(如工程酿酒酵母),使其能够的以木糖及阿拉伯糖等为碳源进行乙醇等生物基化学品发酵,但仍然存在着诸多的问题,例如,细胞内氧化还原代谢不平衡,戊糖代谢缓慢,缺乏有效的戊糖转运蛋白等等。此外,碳源阻遏效应存在于几乎所有的微生物,使工程菌株(如工程酿酒酵母)在利用生物质降解物的混合糖进行发酵的过程中,不能同时利用全糖中的各种成分而导致发酵时间的延长,发酵效率的降低。在利用混合糖进行发酵时,当葡萄糖用尽时,其发酵液中的乙醇含量已达到较高的浓度,将会极大的降低戊糖的发酵率。近期有研究表明,葡萄糖的阻遏效应主要发生在糖转运,因此,为了提高酿酒酵母对全糖的利用率,寻找木糖或阿拉伯糖转运蛋白成了研究的热点。
在五碳糖转运蛋白方面,过去的研究多集中在利用己糖转运蛋白等底物广谱性转运蛋白来转运五碳糖,而对于特异性五碳糖转运蛋白(Pentose Specific Transporter)的研究才刚刚起步。2011年,美国伊利诺伊大学赵慧民教授实验室分别从粗糙脉孢菌和毕赤酵母中鉴定了两个木糖特异转运蛋白(An25和Xyp29)(Du et al.2010.Discovery and characterization of novel d-xylose-specific transporters from Neurospora crassa and Pichia stipitis.Mol Biosyst,6(11):2150-2156)。作为半纤维素的主要成分之一,阿拉伯糖的利用对木质纤维素彻底转化利用至关重要,国际上关于阿拉伯糖转运蛋白研究逐渐开始涉及,2011年,芬兰VTT科学家Richard实验室从Ambrosiozyme monospora酵母中克隆两个阿拉伯糖特异性转运转运(LAT1,LAT2),但该阿拉伯糖转运蛋白亲和力不高(Verho et al.2011.Cloning of two genes(LAT1,2)encoding specific L-arabinose transporters of the L-arabinose fermenting yeast Ambrosiozyma monospora.Appl Biochem Biotechnol,164(5):604-611.),寻找亲和力更高的阿拉伯糖转运蛋白是研究阿拉伯糖转化利用的核心问题之一。
不同于细菌木寡糖转运蛋白,丝状真菌的糖转运蛋白根据转运能量的来源可以将转运蛋白分为初级主动转运蛋白(primary active transporters)和次级转运蛋白(secondary transporters)。初级主动转运蛋白通过利用ATP水解、光子吸收、电子流、底物脱羧或甲基转移反应等释放的能量实现转运过程,典型代表是ATP结合盒(ATP binding cassette,ABC)超家族;而次级转运蛋白则是利用由于物质在膜内外浓度不同造成的电化学渗透势能来转运底物,典型代表是主要协助转运蛋白超家族(major facilitator superfamily,MFS)(详见参考文献:孙林峰,王佳伟,颜宁,MFS超家族转运蛋白结构与分子机制的研究.生命科学,第23卷第11期,1052-1056.)。依据转运的机制,MFS又分为简单转运蛋白(Uniporter),又称协助扩散蛋白(Facilitated Diffusion Protein),同向转运蛋白(Symporter)以及反向转运蛋白(Antiporter)。Uniporter,依靠底物浓度梯度驱动转运,转运蛋白主要起着协助运输的作用。Symporter,向同一个方向同时转运两种及以上的底物,并以其中一种底物的电化学梯度作为推动力,常见的有sugar/H+,glucose/Na+,phosphate/H+,nucleoside/H+以及nitrate/H+等等。Antiporter,向反方向协同转运两种及以上的底物,驱动力来源和Symporter一样,这一类里很多都是drag/H+反向转运蛋白。真菌中的糖转运蛋白主要是Uniporter和Symporter/H+类型(Reddy et al.2012.A collection of programs for the study of transport protein evolution.FEBS J,279(11):2036-2046)。早在1974年已有报道指出,脉孢菌在主动转运糖进入细胞的同时会协同转运质子(Slayman et al.1974.Depolarization of the plasma membrane of Neurospora during active transport of glucose:evidence for a proton-dependent cotransport system.Proc Natl Acad Sci USA,71(5):1935-1939.),但在分子水平上哪些糖转运蛋白的作用,导致了表观上电势的变化至今仍没有被阐释。此外,由于自然界中脉孢菌能生长在各种干枯的木质纤维上,可以预见其糖转运蛋白家族具有很高的功能多样性。同时,在长期的进化适应过程中,细胞面对不同的生长环境,如酸性-碱性,碳充足—碳匮乏,会有很好的策略来协同这些转运蛋白之间的转运工作,以使得其能高效地吸收外界的营养。全面系统的了解糖转运蛋白动力类型和生化特征,对于研究丝状真菌环境适应性,理清纤维素降解菌利用木质纤维素的机制有重要意义。更进一步地,这种高效的糖转运吸收方式,有可能对改造工程菌(包括酵母,曲霉,木霉)以充分利用培养基中的残糖发酵,提高糖的转化利用率有一定的指导意义。
技术实现要素:
本发明第一方面,提供了一种分离的糖转运蛋白多肽,所述的多肽选自下组:
(a)具有SEQ ID NO.:10(MtLAT-1)、8(LAT-1)、6(XAT-1)、4(XYT-1)、或2(GLT-1)中任一条所示氨基酸序列的多肽;
(b)将SEQ ID NO.:10、8、6、4、或2中任一条所述氨基酸序列的多肽经过一个或几个氨基酸残基的取代、缺失或添加而形成的、或是添加信号肽序列后形成的、具有戊糖和/或己糖转运活性的衍生多肽;
(c)序列中含有(a)或(b)中所述多肽序列的衍生多肽;
(d)氨基酸序列与SEQ ID NO.:10、8、6、4、或2中任意一条所述氨基酸序列的同源性≥85%(较佳地≥90%、更佳地≥95%、98%),并具有戊糖和/或己糖转运活性的衍生多肽;
其中,所述的戊糖和/或己糖转运活性是指将戊糖和/或己糖自细胞外转运至细胞内。
在另一优选例中,(d)中所述的衍生多肽来源于以下一种或多种菌株:嗜热毁丝菌(Myceliophthora thermophila)、球毛壳菌(Chaetomium globosum)、柄孢霉(Podospora anserina)、稻瘟病菌(Magnaporthe oryzae)、玉米赤霉(Gibberella zeae)、尖孢镰刀菌(Fusarium oxysporum)、产黄青霉(Penicillium chrysogenum)、土曲霉(Aspergillus terreus)、鞭毛藻丛赤壳菌(Nectria haematococca)、斯梭孢壳霉(Thielavia terrestris)、绿色木霉(Trichoderma virens)、肉原毛平革菌(Phanerochaete carnosa)、黑曲霉(Aspergillus niger)、里氏木霉(Trichoderma reesei)、树干毕赤酵母(Scheffersomyces stipitis)、乳酸克鲁维酵母(Kluyveromyces lactis)。
在另一优选例中,所述的细胞为微生物细胞。
在另一优选例中,所述的多肽具有将选自以下一种或多种糖类自细胞外转运至细胞内的活性:阿拉伯糖、木糖、和葡萄糖。
在另一优选例中,所述的戊糖包括阿拉伯糖、或木糖;
在另一优选例中,所述的己糖包括葡萄糖。
在另一优选例中,序列如SEQ ID NO.:10所示的多肽具有将阿拉伯糖自细胞外转运至细胞内的活性;和/或
序列如SEQ ID NO.:8所示的多肽,具有阿拉伯糖自细胞外转运至细胞内的活性;和/或
序列如SEQ ID NO.:6所示的多肽,具有将木糖和/或阿拉伯糖自细胞外转运至细胞内的活性;和/或
序列如SEQ ID NO.:4所示的多肽,具有将木糖自细胞外转运至细胞内的活性;和/或
序列如SEQ ID NO.:2所示的多肽,具有将葡萄糖自细胞外转运至细胞内的活性。
本发明第二方面,提供了一种分离的多核苷酸,所述的多核苷酸为选自下组的序列:
(A)编码本发明第一方面所述多肽的核苷酸序列;
(B)编码SEQ ID NO.:10、8、6、4、或2中任一所示多肽的核苷酸序列;
(C)如SEQ ID NO.:9、7、5、3、或1中任一所示的核苷酸序列;
(D)与(A)-(C)任一所述的核苷酸序列互补的核苷酸序列。
本发明第三方面,提供了一种载体,所述的载体含有本发明第二方面所述的多核苷酸。
在另一优选例中,所述载体包括表达载体、穿梭载体、整合载体。
本发明第四方面,提供了一种宿主细胞,所述的宿主细胞含有本发明第三方面所述的载体,和/或所述的宿主细胞的染色体整合有外源的本发明第二方面所述的多核苷酸。
在另一优选例中,所述的宿主细胞包括真核或原核细胞,优选为真核细胞。
在另一优选例中,所述的宿主细胞表达一个或多个本发明第一方面所述的多肽。
在另一优选例中,所述的宿主细胞包括酵母(Saccharomyces)属、克鲁维酵母菌属、梭状芽胞杆菌属、或丝状真菌。
在另一优选例中,所述的酵母属包括的酿酒酵母(Saccharomyces cerevisiae)、摩纳酵母(Saccharomyces monacensis)、贝酵母(Saccharomyces bayanus)、巴氏酵母(Saccharomyces pastorianus)、卡氏酵母(Saccharomyces carlsbergensis)、裂殖酵母(Saccharomyces pombe);
所述克鲁维酵母菌属(Kluyveromyces)包括马克斯克鲁维酵母(Kluyveromyces marxiamus)、乳酸克鲁维酵母(Kluyveromyces lactis)、脆壁克鲁维酵母(Kluyveromyces fragilis),树干毕赤酵母(Pichia stipites)、休哈塔假丝酵母(Candida shehatae)、热带假丝酵母(Candida tropicalis)、运动发酵单孢菌(Zymomonas mobilis);
所述梭状芽孢杆菌属(Clostridium sp.)包括梭热杆菌(Clostridium thermocellum)、拜氏梭菌(Clostridium beijerinckii)、丙酮丁醇梭杆菌(Clostridium acetobutylicum)、热乙酸菌(Moorella thermoacetica)、大肠杆菌(Escherichia coli)、产酸克雷伯氏菌(Klebsiella oxytoca)、厌氧杆菌(Thermoanaerobacterium saccharolyticu)或枯草杆菌(Bacillus subtilis);
所述丝状真菌包括嗜热侧孢霉(Sporotrichum thermophile)、粗糙脉孢菌(Neurospora crassa)。
本发明第五方面,提供了本发明第一方面所述多肽、本发明第二方面所述多核苷酸、本发明第三方面所述载体或本发明第四方面所述宿主细胞的用途,(i)用于将戊糖和/或己糖自细胞外转运至细胞内;(ii)用于制备乙醇。
本发明第六方面,提供了一种制备乙醇和/或促进宿主细胞转运戊糖和/或己糖的方法,包括步骤:在戊糖或己糖的存在下,培养本发明第四方面所述的宿主细胞。
在另一优选例中,所述的方法还包括将培养物中的乙醇分离和提纯的步骤。
在另一优选例中,当本发明第四方面所述宿主细胞的表达SEQ ID NO.:10、或8所示的多肽时,所述的戊糖为阿拉伯糖。
在另一优选例中,当本发明第四方面所述的宿主细胞表达SEQ ID NO.:6所示的多肽时,所述的戊糖为阿拉伯糖、和/或木糖。
在另一优选例中,当本发明第四方面所述的宿主细胞表达SEQ ID NO.:4所示的多肽时,所述的戊糖为木糖。
在另一优选例中,当本发明第四方面所述的宿主细胞表达SEQ ID NO.:2所示的多肽时,所述的己糖为葡萄糖。
本发明第七方面一种制备重组乙醇发酵菌株的方法,包括步骤:将本发明第三方面所述的载体转入到出发菌株,从而获得重组乙醇发酵菌株。
在另一优选例中,所述重组乙醇发酵菌株的乙醇发酵活性是出发菌株的1.2-5倍,较佳地,为1.5-2倍。
在另一优选例中,所述的重组乙醇发酵菌株为以戊糖(如阿拉伯糖和/或木糖)和/或己糖(如葡萄糖)为碳源的菌株。
在另一优选例中,所述的出发菌株包括酿酒酵母,例如酿酒酵母BSW2AP、酿酒酵母EBY.VW4000,优选为酿酒酵母BSW2AP菌株
本专利的目的在于提供了五种新的糖转运蛋白,其具体核苷酸序列及其编码蛋白的氨基酸残基序列如下
a.葡萄糖转运蛋白基因GLT-1(NCU01633,来源于粗糙脉胞孢霉,Neurospora crassa)的核苷酸序列如序列表中SEQ ID NO.1所示;
葡萄糖转运蛋白基因GLT-1(NCU01633,来源于粗糙脉胞孢霉,Neurospora crassa)编码蛋白的氨基酸残基序列如序列表中SEQ ID NO.2;
b.木糖转运蛋白基因XYT-1(NCU05627,来源于粗糙脉胞孢霉,Neurospora crassa)的核苷酸序列如序列表中SEQ ID NO.3所示;
木糖转运蛋白基因XYT-1(NCU05627,来源于粗糙脉胞孢霉,Neurospora crassa)编码蛋白的氨基酸残基序列如序列表中SEQ ID NO.4
c.木糖及阿拉伯糖转运蛋白基因XAT-1(NCU01132,来源于粗糙脉胞孢霉,Neurospora crassa)的核苷酸序列如序列表中SEQ ID NO.5所示;
木糖及阿拉伯糖转运蛋白基因XAT-1(NCU01132,来源于粗糙脉胞霉,Neurospora crassa)编码蛋白的氨基酸残基序列如序列表中SEQ ID NO.6;
d.阿拉伯糖转运蛋白基因LAT-1(NCU02188,来源于粗糙脉胞孢霉,Neurospora crassa)的核苷酸序列如序列表中SEQ ID NO.7所示;
阿拉伯糖转运蛋白基因LAT-1(NCU02188,来源于粗糙脉胞孢霉,Neurospora crassa)编码蛋白的氨基酸残基序列如序列表中SEQ ID NO.8;
e.阿拉伯糖转运蛋白基因MtLAT-1(MYCTH_95427,来源于嗜热毁丝霉,Myceliophora thermophila)的核苷酸序列如序列表中SEQ ID NO.9所示;
阿拉伯糖转运蛋白基因MtLAT-1(MYCTH_95427,来源于嗜热毁丝霉,Myceliophora thermophila)编码蛋白的氨基酸残基序列如序列表中SEQ ID NO.10。
此外,还包含了与以上所述五个转运蛋白氨基酸残基序列全长或局部结构域同源度在75%以上的蛋白;该同源度在75%以上的蛋白来自于但不限于以下菌:嗜热毁丝菌(Myceliophthora thermophila)、球毛壳菌(Chaetomium globosum)、柄孢霉(Podospora anserina)、稻瘟病菌(Magnaporthe oryzae)、玉米赤霉(Gibberella zeae)、尖孢镰刀菌(Fusarium oxysporum)、产黄青霉(Penicillium chrysogenum)、土曲霉(Aspergillus terreus)、鞭毛藻丛赤壳菌(Nectria haematococca)、斯梭孢壳霉(Thielavia terrestris)、绿色木霉(Trichoderma virens)、肉原毛平革菌(Phanerochaete carnosa)、黑曲霉(Aspergillus niger)、里氏木霉(Trichoderma reesei)、树干毕赤酵母(Scheffersomyces stipitis)、乳酸克鲁维酵母(Kluyveromyces lactis)等。
所应用的微生物包括但不限于以下菌:酵母属(Saccharomyces sp.)的酿酒酵母(Saccharomyces cerevisiae)、摩纳酵母(Saccharomyces monacensis)、贝酵母(Saccharomyces bayanus)、巴氏酵母(Saccharomyces pastorianus)、卡氏酵母(Saccharomyces carlsbergensis)、裂殖酵母(Saccharomyces pombe),克鲁维酵母菌属(Kluyveromyces sp.)的马克斯克鲁维酵母(Kluyveromyces marxiamus)、乳酸克鲁维酵母(Kluyveromyces lactis)、脆壁克鲁维酵母(Kluyveromyces fragilis),树干毕赤酵母(Pichia stipites)、嗜热侧孢霉(Sporotrichum thermophile)、休哈塔假丝酵母(Candida shehatae)、热带假丝酵母(Candida tropicalis)、粗糙脉孢菌(Neurospora crassa)、运动发酵单孢菌(Zymomonas mobilis)、梭状芽孢杆菌(Clostridium sp.)、(Clostridium phytofermentans)、梭热杆菌(Clostridium thermocellum)、拜氏梭菌(Clostridium beijerinckii)、丙酮丁醇梭杆菌(Clostridium acetobutylicum)、热乙酸菌(Moorella thermoacetica)、大肠杆菌(Escherichia coli)、产酸克雷伯氏菌(Klebsiella oxytoca)、厌氧杆菌(Thermoanaerobacterium saccharolyticu)和枯草杆菌(Bacillus subtilis)。
另一方面,本发明还提供一种使微生物获得或提高对葡萄糖、木糖或阿拉伯糖利用的方法,是将上述转运蛋白导入微生物细胞(如上述所列)中,所得微生物工程菌株可以将葡萄糖、木糖或阿拉伯糖从微生物胞外转运至胞内,从而提高微生物对葡萄糖、木糖或阿拉伯糖的利用效率和相应发酵生产生物基产品的能力。
应理解,在本发明范围内中,本发明的上述各技术特征和在下文(如实施例)中具体描述的各技术特征之间都可以互相组合,从而构成新的或优选的技术方案。限于篇幅,在此不再一一累述。
附图说明
图1为糖转运蛋白的在酿酒酵母中的绿色荧光定位图;
图2为携带GLT-1基因的重组表达质粒pRS426-LAT的物理图谱;
图3为GLT-1对葡萄糖的转运的测定;
图4为含有GLT-1的酿酒酵母EGLT在葡萄糖平板上的生长;
图5为XYT-1对木糖转运能力的测定;
图6A为XAT-1对木糖转运能力的测定;
图6B为XAT-1对阿拉伯糖转运能力的测定;
图7为LAT-1对阿拉伯糖转运能力的测定;
图8为LAT-1对阿拉伯糖转运类型的测定;
图9为MtLAT-1对阿拉伯糖转运能力的测定;
图10为MtLAT-1对阿拉伯糖转运类型的测定;
图11为携带XYT-1的酿酒酵母XXYT在木糖上的生长曲线;
图12为携带LAT-1基因的重组表达质粒p426LAT的物理图谱;
图13为携带MtLAT-1基因的重组表达质粒p426MtLAT的物理图谱;
图14为表达LAT-1或MtLAT-1的酿酒酵母BSWLAT有氧条件下在阿拉伯糖上的生长曲线(A)及L-阿拉伯糖消耗曲线(B);
图15为携带LAT-1或MtLAT-1的酿酒酵母BSWLAT厌氧条件下在阿拉伯糖上的生长曲线(A),L-阿拉伯糖消耗曲线(B)和乙醇生产曲线(C)。
具体实施方式
本发明人经过广泛而深入的研究,首次发现并鉴定了数个能够赋予微生物细胞戊糖或己糖(尤其是戊糖特异性)利用能力的糖转运蛋白。利用本发明蛋白可以使酵母等微生物以阿拉伯糖、木糖为碳源进行发酵,从而使微生物对碳源的利用不受葡萄糖的阻遏效应影响,从而更高效、更经济地获得如乙醇等的生物基产品。在此基础上,完成了本发明。
定义
如本文所用,术语“活性多肽”、“本发明多肽及其衍生多肽”、“本发明转运蛋白”、“戊糖和/或己糖转运蛋白”、“SEQ ID NO.:10、8、6、4、或2所示多肽”,均指具有将戊糖和/或己糖自细胞外向细胞内转运活性的MtLAT-1(SEQ ID NO.:10)、LAT-1(SEQ ID NO.:8)、XAT-1(SEQ ID NO.:6)、XYT-1(SEQ ID NO.:4)、或GLT-1(SEQ ID NO.:2)所示的多肽及其衍生多肽。
如本文所用,术语“戊糖”指的是含有5个碳原子的糖类,通常,所述的戊糖包括五碳醛糖(如核糖、来苏糖、阿拉伯糖、木糖)和五碳酮糖(如核酮糖、木酮糖),优选地,所述的戊糖包括阿拉伯糖、木糖等可用于微生物发酵的五碳糖。
如本文所用,术语“己糖”指的是含有6个碳原子的糖类,通常,所述的己糖包括葡萄糖、半乳糖、甘露糖、果糖等。优选地,所述的己糖为葡萄糖等可用于微生物发酵的六碳糖。
分离的多肽及编码多核苷酸
如本文所用,“分离的多肽”是指所述多肽基本上不含天然与其相关的其它蛋白、脂类、糖类或其它物质。本领域的技术人员能用标准的蛋白质纯化技术纯化所述多肽。基本上纯的多肽在非还原聚丙烯酰胺凝胶上能产生单一的主带。所述多肽的纯度还可以用氨基酸序列进行进一步分析。
本发明的活性多肽可以是重组多肽、天然多肽、合成多肽。本发明的多肽可以是天然纯化的产物,或是化学合成的产物,或使用重组技术从原核或真核宿主(例如,细菌、酵母、植物)中产生。根据重组生产方案所用的宿主,本发明的多肽可以是糖基化的,或可以是非糖基化的。本发明的多肽还可包括或不包括起始的甲硫氨酸残基。
本发明还包括所述多肽的片段、衍生物和类似物。如本文所用,术语“片段”、“衍生物”和“类似物”是指基本上保持所述多肽相同的生物学功能或活性的多肽。
本发明的多肽片段、衍生物或类似物可以是(i)有一个或多个保守或非保守性氨基酸残基(优选保守性氨基酸残基)被取代的多肽,而这样的取代的氨基酸残基可以是也可以不是由遗传密码编码的,或(ii)在一个或多个氨基酸残基中具有取代基团的多肽,或(iii)成熟多肽与另一个化合物(比如延长多肽半衰期的化合物,例如聚乙二醇)融合所形成的多肽,或(iv)附加的氨基酸序列融合到此多肽序列而形成的多肽(如前导序列或分泌序列或用来纯化此多肽的序列或蛋白原序列,或与抗原IgG片段的形成的融合蛋白)。根据本文的教导,这些片段、衍生物和类似物属于本领域熟练技术人员公知的范围。
本发明多肽具有将戊糖和/或己糖自细胞外向细胞内转运的活性,优选地,所述的多肽具有将选自以下一种或多种糖类自细胞外转运至细胞内的活性:阿拉伯糖、木糖、和葡萄糖。
例如:
序列如SEQ ID NO.:10所示的多肽或其衍生多肽具有将阿拉伯糖自细胞外转运至细胞内的活性;和/或
序列如SEQ ID NO.:8所示的多肽或其衍生多肽,具有阿拉伯糖自细胞外转运至细胞内的活性;和/或
序列如SEQ ID NO.:6所示的多肽或其衍生多肽,具有将木糖和/或阿拉伯糖自细胞外转运至细胞内的活性;和/或
序列如SEQ ID NO.:4所示的多肽或其衍生多肽,具有将木糖自细胞外转运至细胞内的活性;和/或
序列如SEQ ID NO.:2所示的多肽或其衍生多肽,具有将葡萄糖自细胞外转运至细胞内的活性。
本发明多肽优选序列为SEQ ID NO.:10、8、6、4、或2所示的多肽,该术语还包括具有与所示多肽具有相同或相似功能的这些多肽的变异形式及衍生多肽。这些变异形式包括(但并不限于):一个或多个(通常为1-50个,较佳地1-30个,更佳地1-20个,最佳地1-10个)氨基酸的缺失、插入和/或取代,以及在C末端和/或N末端添加一个或数个(通常为20个以内,较佳地为10个以内,更佳地为5个以内)氨基酸。例如,在本领域中,用性能相近或相似的氨基酸进行取代时,通常不会改变蛋白质的功能。又比如,在C末端和/或N末端添加一个或数个氨基酸通常也不会改变蛋白质的功能。本发明还提供所述多肽的类似物或活性衍生物。这些类似物与天然本发明多肽的差别可以是氨基酸序列上的差异,也可以是不影响序列的修饰形式上的差异,或者兼而有之。这些多肽包括天然或诱导的遗传变异体。诱导变异体可以通过各种技术得到,如通过辐射或暴露于诱变剂而产生随机诱变,还可通过定点诱变法或其他已知分子生物学的技术。类似物还包括具有不同于天然L-氨基酸的残基(如D-氨基酸)的类似物,以及具有非天然存在的或合成的氨基酸(如β、γ-氨基酸)的类似物。应理解,本发明的多肽并不限于上述例举的代表性的多肽。
修饰(通常不改变一级结构)形式包括:体内或体外的多肽的化学衍生形式如乙酰化或羧基化。修饰还包括糖基化,如那些在多肽的合成和加工中或进一步加工步骤中进行糖基化修饰而产生的多肽。这种修饰可以通过将多肽暴露于进行糖基化的酶(如哺乳动物的糖基化酶或去糖基化酶)而完成。修饰形式还包括具有磷酸化氨基酸残基(如磷酸酪氨酸,磷酸丝氨酸,磷酸苏氨酸)的序列。还包括被修饰从而提高了其抗蛋白水解性能或优化了溶解性能的多肽。
本发明的多核苷酸可以是DNA形式或RNA形式。DNA形式包括cDNA、基因组DNA或人工合成的DNA。DNA可以是单链的或是双链的。DNA可以是编码链或非编码链。编码成熟多肽的编码区序列可以与SEQ ID NO:1所示的编码区序列相同或者是简并的变异体。如本文所用,“简并的变异体”在本发明中是指具有SEQ ID NO.:9、7、5、3、或1所示的编码区序列有差别的核酸序列。
编码SEQ ID NO.:10、8、6、4、或2的成熟多肽的多核苷酸包括:只编码成熟多肽的编码序列;成熟多肽的编码序列和各种附加编码序列;成熟多肽的编码序列(和任选的附加编码序列)以及非编码序列。
术语“编码多肽的多核苷酸”可以是包括编码此多肽的多核苷酸,也可以是还包括附加编码和/或非编码序列的多核苷酸。
本发明还涉及上述多核苷酸的变异体,其编码与本发明有相同的氨基酸序列的多肽或多肽的片段、类似物和衍生物。此多核苷酸的变异体可以是天然发生的等位变异体或非天然发生的变异体。这些核苷酸变异体包括取代变异体、缺失变异体和插入变异体。如本领域所知的,等位变异体是一个多核苷酸的替换形式,它可能是一个或多个核苷酸的取代、缺失或插入,但不会从实质上改变其编码的多肽的功能。
本发明还涉及与上述的序列杂交且两个序列之间具有至少50%,较佳地至少70%,更佳地至少80%相同性的多核苷酸。本发明特别涉及在严格条件(或严紧条件)下与本发明所述多核苷酸可杂交的多核苷酸。在本发明中,“严格条件”是指:(1)在较低离子强度和较高温度下的杂交和洗脱,如0.2×SSC,0.1%SDS,60℃;或(2)杂交时加有变性剂,如50%(v/v)甲酰胺,0.1%小牛血清/0.1%Ficoll,42℃等;或(3)仅在两条序列之间的相同性至少在90%以上,更好是95%以上时才发生杂交。并且,可杂交的多核苷酸编码的多肽与SEQ ID NO.:10、8、6、4、或2所示的成熟多肽有相同的生物学功能和活性。
本发明多肽的核苷酸全长序列或其片段通常可以用PCR扩增法、重组法或人工合成的方法获得。对于PCR扩增法,可根据本发明所公开的有关核苷酸序列,尤其是开放阅读框序列来设计引物,并用市售的cDNA库或按本领域技术人员已知的常规方法所制备的cDNA库作为模板,扩增而得有关序列。当序列较长时,常常需要进行两次或多次PCR扩增,然后再将各次扩增出的片段按正确次序拼接在一起。
一旦获得了有关的序列,就可以用重组法来大批量地获得有关序列。这通常是将其克隆入载体,再转入细胞,然后通过常规方法从增殖后的宿主细胞中得到有关序列。
应用PCR技术扩增DNA/RNA的方法被优选用于获得本发明的基因。特别是很难从文库中得到全长的cDNA时,可优选使用RACE法(RACE-cDNA末端快速扩增法),用于PCR的引物可根据本文所公开的本发明的序列信息适当地选择,并可用常规方法合成。可用常规方法如通过凝胶电泳分离和纯化扩增的DNA/RNA片段。
载体
本发明也涉及包含本发明的多核苷酸的载体,以及用本发明的载体或多肽编码序列经基因工程产生的宿主细胞,以及经重组技术产生本发明所述多肽的方法。
本发明中,将编码本发明多肽多核苷酸序列插入到重组表达载体中。术语“重组表达载体”指本领域熟知的细菌质粒、噬菌体、酵母质粒、植物细胞病毒、哺乳动物细胞病毒如腺病毒、逆转录病毒或其他载体。只要能在宿主体内复制和稳定,任何质粒和载体都可以用。表达载体的一个重要特征是通常含有复制起点、启动子、标记基因和翻译控制元件。
本领域的技术人员熟知的方法能用于构建含本发明多肽编码DNA序列和合适的转录/翻译控制信号的表达载体。这些方法包括体外重组DNA技术、DNA合成技术、体内重组技术等。所述的DNA序列可有效连接到表达载体中的适当启动子上,以指导mRNA合成。这些启动子的代表性例子有:大肠杆菌的lac或trp启动子;λ噬菌体PL启动子;真核启动子包括CMV立即早期启动子、HSV胸苷激酶启动子、早期和晚期SV40启动子、反转录病毒的LTRs和其他一些已知的可控制基因在原核或真核细胞或其病毒中表达的启动子。表达载体还包括翻译起始用的核糖体结合位点和转录终止子。
此外,表达载体优选地包含一个或多个选择性标记基因,以提供用于选择转化的宿主细胞的表型性状,如真核细胞培养用的二氢叶酸还原酶、新霉素抗性以及绿色荧光蛋白(GFP),或用于大肠杆菌的四环素或氨苄青霉素抗性。
包含上述的适当DNA序列以及适当启动子或者控制序列的载体,可以用于转化适当的宿主细胞,以使其能够表达蛋白质。
本发明的宿主细胞优选为对糖类具有利用能力并能够进一步产生乙醇等生物基发酵产品的宿主细胞。优选包括酵母属(Saccharomyces)的酿酒酵母(Saccharomyces cerevisiae)、摩纳酵母(Saccharomyces monacensis)、贝酵母(Saccharomyces bayanus)、巴氏酵母(Saccharomyces pastorianus)、卡氏酵母(Saccharomyces carlsbergensis)、裂殖酵母(Saccharomyces pombe),克鲁维酵母菌属(Kluyveromyces sp.)的马克斯克鲁维酵母(Kluyveromyces marxiamus)、乳酸克鲁维酵母(Kluyveromyces lactis)、脆壁克鲁维酵母(Kluyveromyces fragilis),树干毕赤酵母(Pichia stipites)、嗜热侧孢霉(Sporotrichum thermophile)、休哈塔假丝酵母(Candida shehatae)、热带假丝酵母(Candida tropicalis)、粗糙脉孢菌(Neurospora crassa)、运动发酵单孢菌(Zymomonas mobilis)、梭状芽孢杆菌属(Clostridium sp.)的梭热杆菌(Clostridium thermocellum)、拜氏梭菌(Clostridium beijerinckii)、丙酮丁醇梭杆菌(Clostridium acetobutylicum)、热乙酸菌(Moorella thermoacetica)、大肠杆菌(Escherichia coli)、产酸克雷伯氏菌(Klebsiella oxytoca)、厌氧杆菌(Thermoanaerobacterium saccharolyticu)或枯草杆菌(Bacillus subtilis)
本领域一般技术人员都清楚如何选择适当的载体、启动子、增强子和宿主细胞。
用重组DNA转化宿主细胞可用本领域技术人员熟知的常规技术进行。可选用如下的DNA转染方法:磷酸钙共沉淀法,常规机械方法如显微注射、电穿孔、脂质体包装等。
获得的转化子可以用常规方法培养,表达本发明的基因所编码的多肽。根据所用的宿主细胞,培养中所用的培养基可选自各种常规培养基。在适于宿主细胞生长的条件下进行培养。当宿主细胞生长到适当的细胞密度后,用合适的方法(如温度转换或化学诱导)诱导选择的启动子,将细胞再培养一段时间。
本发明方法获得的重组多肽可以在细胞膜上跨膜表达。
应用
利用编码本发明转运蛋白的多核苷酸可以制备一种新的重组菌株,所述的菌株可以利用原先具有或不具有戊糖或己糖转运能力的出发菌株,通过导入本发明载体,赋予出发菌株新的或更强的戊糖或己糖转运能力,从而提高碳源的利用率。
下述实施例中所用方法如无特别说明均为常规方法,具体步骤可参见:《Molecular Cloning:A Laboratory Manual》(Sambrook,J.,Russell,David W.,Molecular Cloning:A Laboratory Manual,3rd edition,2001,NY,Cold Spring Harbor)。
所出现的百分比浓度如无特别说明均为质量百分浓度。
实施例中描述到的各种生物材料的取得途径仅是提供一种实验获取的途径以达到具体公开的目的,不应成为对本发明生物材料来源的限制。事实上,所用到的生物材料的来源是广泛的,任何不违反法律和道德伦理能够获取的生物材料都可以按照实施例中的提示替换使用。
所用引物均由金唯智生物科技有限公司合成。
本发明中,葡萄糖,木糖和阿拉伯糖购自sigma试剂公司;
间苯腙氯羰氰(CCCP)购自sigma公司;
同位素标记的葡萄糖,木糖和阿拉伯糖均购自美国Radiolabeled化工有限公司。
实施例1.GLT-1是葡萄糖转运蛋白,使微生物获得转运和利用葡萄糖能力
一、GLT-1基因表达载体的构建
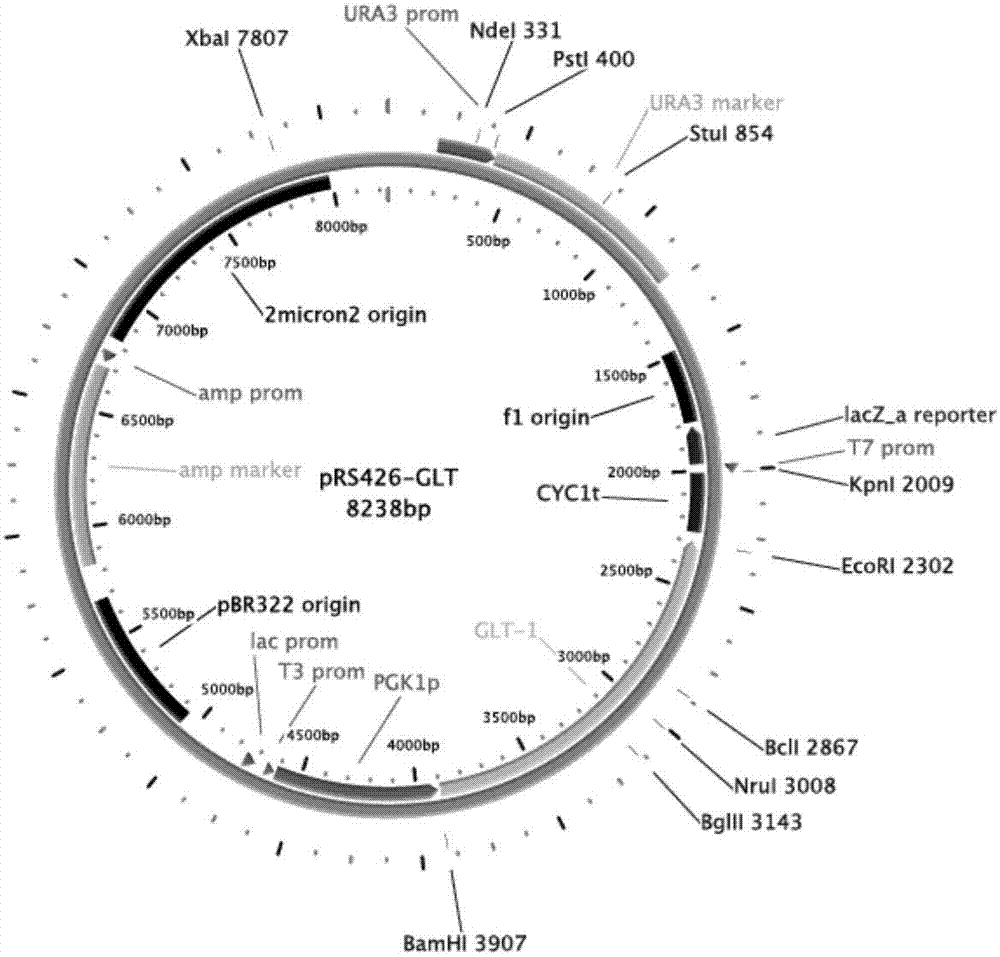
利用引物GLT-F(序列:5’-CGCGGATCCATGGGTCTCTTCTCGAAAAAGTC-3’)(SEQ ID NO.:11)和GLT-R(序列:5’-CCGGAATTCCTAAACCTCTCCATGGCTTGAGG-3’)(SEQ ID NO.:12)从粗糙脉孢菌的cDNA中PCR扩增GLT-1基因的编码阅读框,PCR反应体系为:5×phusion HF buffer 10μL,10mMdNTPs 1μL,GLT-F 2.5μL,GLT-R 2.5μL,cDNA 1μL,Phusion DNA polymerase0.5μL,水32.5μL。PCR反应条件为:先98℃30s;然后98℃10s,65℃30s,72℃1.5min,35个循环;最后72℃10min,4℃10min。PCR反应结束后,使用限制性内切酶BamHI和EcoRI将PCR产物和质粒pRS426-PGK1[该质粒构建根据参考文献(Galazka,J.M.,et al.,2010.Cellodextrin transport in yeast for improved biofuel production.Science.330,84-86.)],进行双酶切,并将两种双酶切产物进行连接,将连接产物用限制性内切酶进行酶切鉴定,再进行测序,测序结果表明GLT-1基因的核苷酸序列如序列表中序列1所示,表明获得了序列及插入位置正确的携带GLT-1基因的重组表达质粒,命名为pRS426-GLT,其物理图谱如图2。将质粒pRS426-GLT和pRS426-PGK1转入酿酒酵母EBY.VW4000(Wieczorke,R.,et al.,1999.Concurrent knock-out of at least 20transporter genes is required to block uptake of hexoses in Saccharomyces cerevisiae.FEBS Lett.464,123-128.)中,分别命名为EGLT和E426。
二、GLT-1对葡萄糖的转运的测定
1、分别挑取EGLT和对照菌株E426单克隆,将其接种在10mL以2%麦芽糖为碳源的SC-URA培养基(配方:无氨基酵母氮源6.7g/L,酵母合成缺失培养基补充物1.4g/L,麦芽糖20g/L,亮氨酸,组氨酸和色氨酸各20mg/L)中30℃培养过夜(10-12小时)至菌体浓度为1.5-2.0(OD600);
2、离心收集菌体(4000rpm,5min)后,用冰预冷assay buffer(100mM Tris-Citrate buffer pH 5.0)洗三次,重悬至OD600为20;
3、将菌体分装至1.5mL离心管(100ul/管)中,其中三个用于烘干称其干重,其余置于冰上,以备使用。
4、反应前将菌体于30摄氏度,放置5min。
5、100ul细胞悬液加入50ul不同浓度同位素标记的葡萄糖溶液(糖浓度:400mM,250mM,100mM,50mM,10mM,5mM),反应120s后加入1mL冰水终止反应,
6、10000rpm离心1min来收集细胞,冰水洗涤两次,离心,去上清。
7、用500mL,0.1mM NaOH重选菌体,转移至装有3mL Ultima Gold scintillatioin fluid小瓶中,测定其放射量,并计算单位细胞干重单位时间的转运量。
最终结果如图3所示。酿酒酵母EBY.VW4000缺失17个己糖转运蛋白和3个麦芽糖/葡萄糖转运蛋白,而失去了转运葡萄糖的能力。由于GLT-1的导入,重组酵母EGLT恢复了对葡萄糖的转运能力,其中如图3,GLT对葡萄糖的亲和力Km为18.42±3.38mM,最大转运速率为30.75±1.34mmol/h/gram DCW。
三、GLT-1使微生物具有利用葡萄糖的能力
1、分别挑取EGLT和对照菌株E426单克隆,将其接种在10mL以2%麦芽糖为碳源的SC-URA培养基(配方:无氨基酵母氮源6.7g/L,酵母合成缺失培养基补充物1.4g/L,麦芽糖20g/L,亮氨酸,组氨酸和色氨酸各20mg/L)中30℃培养过夜(10-12小时)至菌体浓度为1.5-2.0;
2、离心收集菌体(4000rpm,5min)后,用冰预冷双蒸水洗三次,重悬至OD600为2;
3、等梯度稀释菌夜(100,10-1,10-2,10-3,10-4,10-5);
4、将等梯度稀释的酵母点在以2%麦芽糖为碳源的SC-URA平板中(配方:无氨基酵母氮源6.7g/L,酵母合成缺失培养基补充物1.4g/L,麦芽糖20g/L,亮氨酸,组氨酸和色氨酸各20mg/L,2%琼脂),置于30℃培养箱中培养。
结果如图4所示,相比对照菌株(E426),重组酵母(EGLT)快速利用葡萄糖的能力。说明GLT-1的导入,酿酒酵母恢复了以葡萄糖为碳源进行生长的能力。
综合所述,GLT-1对葡萄糖具有较高的转运能力(30.75±1.34mmol/h/gramDCW)和亲和性(Km为18.42±3.38mM),并且GLT-1导入酿酒酵母后,可使其恢复利用葡萄糖为碳源的能力。
实施例2.XYT-1是木糖转运蛋白,使微生物获得转运木糖能力
一、XYT-1基因表达载体的构建
利用引物XYT-F(序列:5’-GGACTAGTATGGTTCTGGGGAAAAAGTCAATC-3’)(SEQ ID NO.:13)和XYT-R(序列:5’-CCCAAGCTTCTAAACCCTATGGTTAATAACCTT-3’)(SEQ ID NO.:14)从粗糙脉孢菌的cDNA中PCR扩增XYT-1基因的编码阅读框,PCR反应体系为:5×phusion HF buffer 10μL,10mM dNTPs 1μL,XYT-F 2.5μL,XYT-R 2.5μL,cDNA 1μL,Phusion DNA polymerase 0.5μL,水32.5μL。PCR反应条件为:先98℃30s;然后98℃10s,60℃30s,72℃1.5min,35个循环;最后72℃10min,4℃10min。PCR反应结束后,使用限制性内切酶SpeI和HindIII将PCR产物和质粒pRS426-PGK1[该质粒构建根据参考文献(Galazka,J.M.,et al.,2010.Cellodextrin transport in yeast for improved biofuel production.Science.330,84-86.)],进行双酶切,并将两种双酶切产物进行连接,将连接产物用限制性内切酶进行酶切鉴定,再进行测序,测序结果表明XYT-1基因的核苷酸序列如序列表中序列3所示,表明获得了序列及插入位置正确的携带XYT-1基因的重组表达质粒,命名为pRS426-XYT。将质粒pRS426-XYT和pRS426-PGK1转入酿酒酵母EBY.VW4000(Wieczorke,R.,et al.,1999.Concurrent knock-out of at least 20transporter genes is required to block uptake of hexoses in Saccharomyces cerevisiae.FEBS Lett.464,123-128.)中,分别命名为EXYT和E426。
二、XYT-1对木糖的转运的测定
XYT-1对木糖的转运能力的测定参见实施例1,其结果如图5所示。由于XYT-1的导入,重组酵母EXYT具有木糖的转运能力,XYT-1对木糖的亲和力Km为7.58±0.60mM,最大转运速率为49.61±1.20μmol/h/gram DCW。
实施例3.XAT-1是木糖和阿拉伯糖转运蛋白,使微生物获得转运木糖和阿拉伯糖能力
一、XAT-1基因表达载体的构建
利用引物XAT-F(序列:5’-CGCGGATCCATGAAGCCATTTCTGGGGCTC-3’)(SEQ ID NO.:15)和XAT-R(序列:5’-CCCAAGCTTCTACGACTCCCGATTACCTCCAT-3’)(SEQ ID NO.:16)从粗糙脉孢菌的cDNA中PCR扩增XAT-1基因的编码阅读框,PCR反应体系为:5×phusion HF buffer 10μL,10mM dNTPs 1μL,XAT-F 2.5μL,XAT-R2.5μL,cDNA 1μL,Phusion DNA polymerase 0.5μL,水32.5μL。PCR反应条件为:先98℃30s;然后98℃10s,65℃30s,72℃1.5min,35个循环;最后72℃10min,4℃10min。PCR反应结束后,使用限制性内切酶BamHI和HindIII将PCR产物和质粒pRS426-PGK1[该质粒构建根据参考文献(Galazka,J.M.,et al.,2010.Cellodextrin transport in yeast for improved biofuel production.Science.330,84-86.)],进行双酶切,并将两种双酶切产物进行连接,将连接产物用限制性内切酶进行酶切鉴定,再进行测序,测序结果表明XAT-1基因的核苷酸序列如序列表中序列5所示,表明获得了序列及插入位置正确的携带XAT-1基因的重组表达质粒,命名为pRS426-XAT。将质粒pRS426-XAT和pRS426-PGK1转入酿酒酵母EBY.VW4000(Wieczorke,R.,et al.,1999.Concurrent knock-out of at least 20transporter genes is required to block uptake of hexoses in Saccharomyces cerevisiae.FEBS Lett.464,123-128.)中,分别命名为EXAT和E426。
二、XAT-1对木糖和阿拉伯糖转运的测定
XAT-1对木糖和阿拉伯糖的转运能力的测定参见实施例1,其结果如图6所示。由于XAT-1的导入,重组酵母EXAT具有木糖和阿拉伯糖的转运能力,其中,XAT-1对木糖的亲和力Km为18.17±3.23mM,最大转运速率为54.11±3.83μmol/h/gram DCW(图6A);XAT-1对阿拉伯糖的亲和力Km为61.93±17.68mM,大转运速率为65.84±11.76μmol/h/gram DCW(图6B)。
实施例4.LAT-1是阿拉伯糖转运蛋白,使微生物获得转运阿拉伯糖转运能力
一、LAT-1基因表达载体的构建
利用引物ELAT-F(序列:5’-CGCGGATCCATGGGGCTCGGGCTTAAGCTAC-3’)(SEQ ID NO.:17)和ELAT-R(序列:5’-CGGAATTCCTAAACCTTCTCATGCTCATGCAC-3’)(SEQ ID NO.:18)从粗糙脉孢菌的cDNA中PCR扩增LAT-1基因的编码阅读框,PCR反应体系为:5×phusion HF buffer 10μL,10mM dNTPs 1μL,ELAT-F 2.5μL,ELAT-R 2.5μL,cDNA 1μL,Phusion DNA polymerase 0.5μL,水32.5μL。PCR反应条件为:先98℃30s;然后98℃10s,65℃30s,72℃1.5min,35个循环;最后72℃10min,4℃10min。PCR反应结束后,使用限制性内切酶BamHI和HindIII将PCR产物和质粒pRS426-PGK1[该质粒构建根据参考文献(Galazka,J.M.,et al.,2010.Cellodextrin transport in yeast for improved biofuel production.Science.330,84-86.)],进行双酶切,并将两种双酶切产物进行连接,将连接产物用限制性内切酶进行酶切鉴定,再进行测序,测序结果表明LAT-1基因的核苷酸序列如序列表中序列7所示,表明获得了序列及插入位置正确的携带LAT-1基因的重组表达质粒,命名为pRS426-LAT。将质粒pRS426-LAT和pRS426-PGK1转入酿酒酵母EBY.VW4000(Wieczorke,R.,et al.,1999.Concurrent knock-out of at least 20transporter genes is required to block uptake of hexoses in Saccharomyces cerevisiae.FEBS Lett.464,123-128.)中,分别命名为ELAT和E426。
二、LAT-1对阿拉伯糖转运的测定
LAT-1对木糖和阿拉伯糖的转运能力的测定参见实施例1,其结果如图7所示。由于LAT-1的导入,重组酵母ELAT具有阿拉伯糖的转运能力,LAT-1对阿拉伯糖的亲和力Km为25.12±2.98mM,最大转运速率为116.7±4.06mmol/h/gram DCW。
三、LAT-1转运类型的测定
真菌中的糖转运蛋白主要是Uniporter和Symporter/H+类型,Uniporter,依靠底物浓度梯度驱动转运,转运蛋白主要起着协助运输的作用。Symporter/H+,向同一个方向同时转运糖类及H+,并以H+的电化学梯度作为推动力。为了鉴定LAT-1的转运类型设计了如下实验:
其中,CCCP(间苯腙氯羰氰)具有破坏细胞质子梯度的作用,因此不同CCCP浓度下,测定LAT-1对阿拉伯糖的转运能力,从而鉴定LAT-1的转运类型。
1、挑取ELAT单克隆,将其接种在10mL以2%麦芽糖为碳源的SC-URA培养基(配方:无氨基酵母氮源6.7g/L,酵母合成缺失培养基补充物1.4g/L,麦芽糖20g/L,亮氨酸,组氨酸和色氨酸各20mg/L)中30℃培养过夜(10-12小时)至菌体浓度为1.5-2.0(OD600);
2、离心收集菌体(4000rpm,5min)后,用冰预冷assay buffer(100mM Tris-Citrate buffer pH 5.0)洗三次,重悬至OD600为20;
3、将菌体分装至1.5mL离心管(100ul/管)中,每管50μL,其中三个用于烘干称其干重,其余置于冰上,以备使用。
4、反应前加入1μL不同浓度的CCCP(终浓度分别为:0μM,0.5μM,1.0μM,25μM,50μM),于30℃培养箱中,放置10min。
5、50ul细胞悬液加入50ul 50mM同位素标记的阿拉伯糖溶液,于30℃下反应120s后,加入1mL冰水终止反应,
6、10000rpm离心1min来收集细胞,冰水洗涤两次,离心,去上清。
7、用500mL 0.1mM NaOH重选菌体,转移至装有3mL Ultima Gold scintillatioin fluid小瓶中,测定其放射量,并计算其单位干重单位时间的转运量。
其实验结果如图8所示,随着CCCP浓度的增高,ELAT对阿拉伯糖的转运能力逐渐下降,说明LAT-1的转运类型为Symporter/H+,当CCCP浓度分别25μM时,其对阿拉伯糖的转运速率降到了最低,此时LAT-1依靠细胞内外的阿拉伯糖浓度梯度为动力,转运阿拉伯糖。有实验证明当转运蛋白的转运类型为Symporter/H+时,会有较大的转运速率,具有更好的应用前景。
实施例5.MtLAT-1是阿拉伯糖转运蛋白,使微生物获得转运阿拉伯糖能力
一、MtLAT-1基因表达载体的构建
利用引物MtLAT-F(序列:5’-CGCGGATCCATGAAGCTGCCCACGATTTAC-3’)(SEQ ID NO.:19)和MtLAT-R(序列:5’-CCGGAATTCTTAAACCTTCTCCTGCTCGCC-3’)(SEQ ID NO.:20)从嗜热毁丝霉的cDNA中PCR扩增MtLAT-1基因的编码阅读框,PCR反应体系为:5×phusion HF buffer 10μL,10mM dNTPs 1μL,MtLAT-F 2.5μL,MtLAT-R 2.5μL,cDNA 1μL,Phusion DNA polymerase 0.5μL,水32.5μL。PCR反应条件为:先98℃30s;然后98℃10s,67℃30s,72℃1.5min,35个循环;最后72℃10min,4℃10min。PCR反应结束后,使用限制性内切酶BamHI和EcoRI将PCR产物和质粒pRS426-PGK1[该质粒构建根据参考文献(Galazka,J.M.,et al.,2010.Cellodextrin transport in yeast for improved biofuel production.Science.330,84-86.)],进行双酶切,并将这两种双酶切产物进行连接,将连接产物用限制性内切酶进行酶切鉴定,再进行测序,测序结果表明MtLAT-1基因的核苷酸序列如序列表中序列9所示,表明获得了序列及插入位置正确的携带MtLAT-1基因的重组表达质粒,命名为pRS426-MtLAT。将质粒pRS426-MtLAT和pRS426-PGK1转入酿酒酵母EBY.VW4000(Wieczorke,R.,et al.,1999.Concurrent knock-out of at least 20transporter genes is required to block uptake of hexoses in Saccharomyces cerevisiae.FEBS Lett.464,123-128.)中,分别命名为EMtLAT和E426。
二、MtLAT-1阿拉伯糖转运的测定
MtLAT-1对阿拉伯糖的转运能力的测定参见实施例1,其结果如图9所示。由于MtLAT-1的导入,重组酵母EMtLAT具有木糖和阿拉伯糖的转运能力,其中,MtLAT-1对阿拉伯糖的亲和力Km为10.29±0.35mM,大转运速率为10.29±3.6mmol/h/gram DCW。
三、MtLAT-1转运类型的测定
实验方法参加实施例4所述,其实验结果如图10,实验说明MtLAT-1对阿拉伯糖的转运类型为Symporter/H+;MtLAT-1的转运速率随CCCP浓度的增加而降低,当CCCP为25μM时,其转运速率降到了最低。
实施例6.XYT-1促进酿酒酵母木糖生长和乙醇发酵
将实施例2所述的质粒pRS426-XYT和pRS426-PGK1转入含有毕赤酵母木糖代谢途径的酿酒酵母EBY.VW4000(Wieczorke,R.,et al.,1999.Concurrent knock-out of at least 20transporter genes is required to block uptake of hexoses in Saccharomyces cerevisiae.FEBS Lett.464,123-128.)中,分别命名为XXYT和X426。
分别挑取XXYT和对照菌株X426单克隆,将其接种在10mL以2%麦芽糖为碳源的SC-URA培养基(配方:无氨基酵母氮源6.7g/L,酵母合成缺失培养基补充物1.4g/L,麦芽糖20g/L,亮氨酸,组氨酸和色氨酸各20mg/L)中30℃培养过夜(10-12小时),之后分别转入以2%木糖为碳源40mL SC-URA培养基中,最终OD600为1,100mL三角瓶中培养(250rpm,30℃),每个一段时间取样并测其OD600。
结果如图11所示:相对于对照菌株X426,XXYT具有利用木糖生长的能力。结果说明XYT-1具有转运木糖的能力,同时可以让酿酒酵母重新获得利用木糖为碳源生长的能力。
实施例7.LAT-1和MtLAT-1促进酿酒酵母阿拉伯糖生长和乙醇发酵
LAT-1为阿拉伯糖转运蛋白,本实施例的目的是通过LAT-1基因在酿酒酵母中的表达来确定LAT-1的功能及对重组酿酒酵母的作用。
一、LAT-1及MtLAT-1基因表达载体的构建
利用引物KanMX-F(序列:5’-GGGAATTCCATATGGATCTGTTTAGCTTGCCTCGTC-3’)(SEQ ID NO.:21)和KanMX-R(序列:5’-ATGGGCCCCGACACTGGATGGCGGCGTTAG-3’)(SEQ ID NO.:22)从质粒pUG-6(Gueldener,U,et al.,2002.A second set of loxP marker cassettes for Cre-mediated multiple gene knockouts in budding yeast.Nucleic Acids Res.30(6))中PCR扩增遗传霉素G418的抗性基因kanMX的编码阅读框,PCR的反应体系为:5×phusion HF buffer 10μL,10mM dNTPs 1μL,KanMX-F 2.5μL,KanMx-R 2.5μL,pUG6质粒1μL,Phusion DNA polymerase 0.5μL,水32.5μL。PCR反应条件为:先98℃30s;然后98℃10s,60℃30s,72℃1.0min,35个循环;最后72℃10min,4℃10min。PCR反应结束后,使用限制性内切酶NdeI和ApaI将PCR产物和质粒pRS426-PGK1[该质粒构建根据参考文献(Galazka,J.M.,et al.,2010.Cellodextrin transport in yeast for improved biofuel production.Science.330,84-86.)略有改动],进行双酶切,并将两种双酶切产物进行连接,将连接产物用限制性内切酶进行酶切鉴定,再进行测序验证,KanMX基因的重组表达质粒,命名为p426kanmx4。使用引物ELAT-F(序列:5’-CGCGGATCCATGGGGCTCGGGCTTAAGCTAC-3’)(SEQ ID NO.:17)和ELATF-R(序列:5’-CGGAATTCCTAAACCTTCTCATGCTCATGCAC-3’)(SEQ ID NO.:18)从粗糙脉孢菌的cDNA中PCR扩增LAT-1基因的编码阅读框,PCR反应体系为:5×phusion HF buffer 10μL,10mM dNTPs 1μL,LAT-F 2.5μL,LAT-R2.5μL,cDNA 1μL,Phusion DNA polymerase 0.5μL,水32.5μL。PCR反应条件为:先98℃30s;然后98℃10s,64℃30s,72℃1.5min,35个循环;最后72℃10min,4℃10min。PCR反应结束后,使用限制性内切酶SpeI和EcoRI将PCR产物和质粒pRS426-PGK1URA::KanMX进行双酶切,并将两种双酶切产物进行连接,将连接产物用限制性内切酶进行酶切鉴定,再进行测序,测序结果表明LAT-1基因的核苷酸序列如序列表中序列7所示,表明获得了序列及插入位置正确的携带LAT-1基因的重组表达质粒,命名为p426LAT,其物理图谱如图12所示。
使用引物MtLAT-F(序列:5’-CGCGGATCCATGAAGCTGCCCACGATTTAC-3’)(SEQ ID NO.:19)和MtLATF-R(序列:5’-CCGGAATTCTTAAACCTTCTCCTGCTCGCCGAC-3’)(SEQ ID NO.:20)从嗜热毁丝霉的cDNA中PCR扩增MtLAT-1基因的编码阅读框,PCR反应体系为:5×phusion HF buffer 10μL,10mM dNTPs 1μL,LAT-F 2.5μL,LAT-R 2.5μL,cDNA 1μL,Phusion DNA polymerase 0.5μL,水32.5μL。PCR反应条件为:先98℃30s;然后98℃10s,64℃30s,72℃1.5min,35个循环;最后72℃10min,4℃10min。PCR反应结束后,使用限制性内切酶BamHI和EcoRI将PCR产物和质粒pRS426-PGK1URA::KanMX进行双酶切,并将两种双酶切产物进行连接,将连接产物用限制性内切酶进行酶切鉴定,再进行测序,测序结果表明MtLAT-1基因的核苷酸序列如序列表中序列7所示,表明获得了序列及插入位置正确的携带MtLAT-1基因的重组表达质粒,命名为p426MtLAT,其物理图谱如图13所示。
将质粒p426LAT,p426MtLAT和p426kanmx转入酿酒酵母BSW2AP(Wang,et al.,2013.Improvement of L-Arabinose Fermentation by Modifying the Metabolic Pathway and Transport in Saccharomyces cerevisiae.Biomed Res Int.)中。
二、LAT-1及MtLAT-1促进重组酿酒酵母在L-arabinose中的生长。
分别挑取分别含有p426LAT,p426MtLAT和p426kanmx的酿酒酵母转化子单克隆,将其接种在50mL以1.5%L-阿拉伯糖及0.5%D-葡萄糖为碳源的SC-URA-LEU培养基(配方:无氨基酵母氮源6.7g/L,酵母合成缺失培养基补充物1.4g/L,麦芽糖20g/L,组氨酸和色氨酸各20mg/L,同时添加400μg/mL G418)中30℃培养过夜(10-12小时),之后分别转入以2%L-阿拉伯糖为碳源40mL SC-URA-LEU培养基中(添加400μg/mL G418),最终OD600为1.0,分别在250mL三角瓶(视为有氧培养)及100mL厌氧瓶(视为限氧培养)中培养(250rpm,25℃),每个一段时间取样并测其OD600。结果如图14和图15所示。
在图14中,在菌株BSW2AP中过表达LAT-1及MtLAT-1后,在有氧条件下相对于对照菌株具有较快的生长速率,其L-阿拉伯糖的消耗速率也明显加快。说明在有氧条件下,LAT-1及MtLAT-1可以加强微生物对阿拉伯糖的利用,促进微生物的生长。
在图15中,菌株BSW2AP过表达LAT-1及MtLAT-1后,在限氧条件下相对于对照菌株具有较快的生长速率,其L-阿拉伯糖的消耗速率及乙醇的生产速率也明显加快。说明在限氧条件下,LAT-1及MtLAT-1可以加强微生物对阿拉伯糖的利用,促进微生物的生长,有利用乙醇的生产。
相对于对照菌株BSW426,BSWLAT在50小时后具有较快的生长,说明在限氧的条件下,LAT-1的表达有利用阿拉伯糖发酵酿酒酵母对阿拉伯糖的利用,促进酵母的代谢。
在本发明提及的所有文献都在本申请中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
序列表
<110> 中国科学院天津工业生物技术研究所
<120> 一种促进微生物细胞转运葡萄糖、木糖和阿拉伯糖的方法及其在生物基产品
发酵中的应用
<130> P2018-0213
<150> 201410051718.3
<151> 2014-02-16
<160> 22
<170> PatentIn version 3.5
<210> 1
<211> 1599
<212> DNA
<213> 粗糙脉胞孢霉(Neurospora crassa)
<400> 1
atg ggt ctc ttc tcg aaa aag tcg gct gcg ccg cag acc caa tca caa 48
Met Gly Leu Phe Ser Lys Lys Ser Ala Ala Pro Gln Thr Gln Ser Gln
1 5 10 15
gat gag atc gat ctc gct gct gag cag aag gtc act ttc cgt gcc gtc 96
Asp Glu Ile Asp Leu Ala Ala Glu Gln Lys Val Thr Phe Arg Ala Val
20 25 30
ttc ctc ggt gtt gtc gcc tcc gta ggt ggc ttc atg ttt ggc tac gtc 144
Phe Leu Gly Val Val Ala Ser Val Gly Gly Phe Met Phe Gly Tyr Val
35 40 45
agt ggt caa att tct ggt ttc ttc gac atg gaa gac ttc ggt cgt cgg 192
Ser Gly Gln Ile Ser Gly Phe Phe Asp Met Glu Asp Phe Gly Arg Arg
50 55 60
ttc ggt aac tac caa gac gcg gat ggc tgg gtc ttc tca gca tac cgc 240
Phe Gly Asn Tyr Gln Asp Ala Asp Gly Trp Val Phe Ser Ala Tyr Arg
65 70 75 80
cag ggt gct att gtc gcc cta ctc cct gct ggt gcc ctt ctt ggt tcg 288
Gln Gly Ala Ile Val Ala Leu Leu Pro Ala Gly Ala Leu Leu Gly Ser
85 90 95
ctc gtt gcc ggt aga att gcg gat acc ctt ggt cgc cgt atc gcc atc 336
Leu Val Ala Gly Arg Ile Ala Asp Thr Leu Gly Arg Arg Ile Ala Ile
100 105 110
tct gcg tcc gcc ctt ttc tcc tgc atc gga aca att atc gag atc gcc 384
Ser Ala Ser Ala Leu Phe Ser Cys Ile Gly Thr Ile Ile Glu Ile Ala
115 120 125
tcc acc acg cac tgg gcc cag ttt gcg gtc ggt cgt ctt atc acc ggt 432
Ser Thr Thr His Trp Ala Gln Phe Ala Val Gly Arg Leu Ile Thr Gly
130 135 140
att ggt atc ggt gct ctc tcc gtc gtc gtc ccg atg tac cag tct gag 480
Ile Gly Ile Gly Ala Leu Ser Val Val Val Pro Met Tyr Gln Ser Glu
145 150 155 160
tcc gcg ccc gcc atc ctc cgt ggt atc ctc gtc tcg tgc tac cag ctc 528
Ser Ala Pro Ala Ile Leu Arg Gly Ile Leu Val Ser Cys Tyr Gln Leu
165 170 175
ttc atc act ctt ggt atc tgg acc gct gag atg atc aac tac ggt act 576
Phe Ile Thr Leu Gly Ile Trp Thr Ala Glu Met Ile Asn Tyr Gly Thr
180 185 190
cac gac ctc agc aac tcc gcc tct tgg cgt att ccc aac ggt atc tcc 624
His Asp Leu Ser Asn Ser Ala Ser Trp Arg Ile Pro Asn Gly Ile Ser
195 200 205
ttc ctc tgg gct ttg gtt ctc ggt ggc gga ata ttg ttc ctt cct gag 672
Phe Leu Trp Ala Leu Val Leu Gly Gly Gly Ile Leu Phe Leu Pro Glu
210 215 220
tct ccc cgt tat gcc tac cgt gtt ggt cgc gag gac gag gct cgc aac 720
Ser Pro Arg Tyr Ala Tyr Arg Val Gly Arg Glu Asp Glu Ala Arg Asn
225 230 235 240
acc att gcc cgc ctt gcc ggt ctc gag ccc agc gcc cgc tct gtc aac 768
Thr Ile Ala Arg Leu Ala Gly Leu Glu Pro Ser Ala Arg Ser Val Asn
245 250 255
atg caa atc gat gag atc cgt atg aag ctt gag gag gag aag gct ggt 816
Met Gln Ile Asp Glu Ile Arg Met Lys Leu Glu Glu Glu Lys Ala Gly
260 265 270
gcc gac acc aag tgg tac gag atc ttc gga cct gct ctg ttg cgc cgc 864
Ala Asp Thr Lys Trp Tyr Glu Ile Phe Gly Pro Ala Leu Leu Arg Arg
275 280 285
acc ctt atc ggt atc att ctt cag tct ggc cag cag ctt act ggt gcc 912
Thr Leu Ile Gly Ile Ile Leu Gln Ser Gly Gln Gln Leu Thr Gly Ala
290 295 300
aac ttc ttc ttc tac tac gga acc acg att ttc aag gct act ggt ctt 960
Asn Phe Phe Phe Tyr Tyr Gly Thr Thr Ile Phe Lys Ala Thr Gly Leu
305 310 315 320
agc gac tct tac gtt acc cag atc att ctt ggt tcc gtc aac gct gga 1008
Ser Asp Ser Tyr Val Thr Gln Ile Ile Leu Gly Ser Val Asn Ala Gly
325 330 335
tgc act gtt gct ggt ctc tgg gtt gtc aag aat gtt ggc cgc cgt aag 1056
Cys Thr Val Ala Gly Leu Trp Val Val Lys Asn Val Gly Arg Arg Lys
340 345 350
gcc ctc atc ggt ggt gcc ctc tgg atg acc atg tgc ttc ttg gtc tac 1104
Ala Leu Ile Gly Gly Ala Leu Trp Met Thr Met Cys Phe Leu Val Tyr
355 360 365
tct ttc gtc gga aga ttt gtg ctc gac ccc gtc aac ccg gct agc act 1152
Ser Phe Val Gly Arg Phe Val Leu Asp Pro Val Asn Pro Ala Ser Thr
370 375 380
cct cag gcc ggc aac gtc ctc att gtc ttc tcc tgc ttc ttc atc gtc 1200
Pro Gln Ala Gly Asn Val Leu Ile Val Phe Ser Cys Phe Phe Ile Val
385 390 395 400
gcc ttt gcc acc act tgg ggt cct ctc gtc tgg gcc gtc gtt gct gag 1248
Ala Phe Ala Thr Thr Trp Gly Pro Leu Val Trp Ala Val Val Ala Glu
405 410 415
ctc tac cct gct cgc tac cgt gct cct gcc atg gcc ttg gcc acc gct 1296
Leu Tyr Pro Ala Arg Tyr Arg Ala Pro Ala Met Ala Leu Ala Thr Ala
420 425 430
tcc aac tgg ctg tgg aac ttc ctc atg tcc ctc ttc acg cgc ccc atc 1344
Ser Asn Trp Leu Trp Asn Phe Leu Met Ser Leu Phe Thr Arg Pro Ile
435 440 445
acc gac tcc att ggc tac ttc tat ggc ttg gtg ttc gcc gga tgc tgc 1392
Thr Asp Ser Ile Gly Tyr Phe Tyr Gly Leu Val Phe Ala Gly Cys Cys
450 455 460
ctt gcc ctc gcc gct ttc gtt tgg ctc ttt gtg atc gag tcc aag gac 1440
Leu Ala Leu Ala Ala Phe Val Trp Leu Phe Val Ile Glu Ser Lys Asp
465 470 475 480
cgc acc ctt gag gag atc gag acc atg tac aac cag aag gtc agc cct 1488
Arg Thr Leu Glu Glu Ile Glu Thr Met Tyr Asn Gln Lys Val Ser Pro
485 490 495
agg cac tcc acc cac tgg cac gct gag gtc cct tcg gga ccg cgg gat 1536
Arg His Ser Thr His Trp His Ala Glu Val Pro Ser Gly Pro Arg Asp
500 505 510
gcg gag gag aag ccc gag gtt cac agt ggt tct gcg aca acc tca agc 1584
Ala Glu Glu Lys Pro Glu Val His Ser Gly Ser Ala Thr Thr Ser Ser
515 520 525
cat gga gag gtt tag 1599
His Gly Glu Val
530
<210> 2
<211> 532
<212> PRT
<213> 粗糙脉胞孢霉(Neurospora crassa)
<400> 2
Met Gly Leu Phe Ser Lys Lys Ser Ala Ala Pro Gln Thr Gln Ser Gln
1 5 10 15
Asp Glu Ile Asp Leu Ala Ala Glu Gln Lys Val Thr Phe Arg Ala Val
20 25 30
Phe Leu Gly Val Val Ala Ser Val Gly Gly Phe Met Phe Gly Tyr Val
35 40 45
Ser Gly Gln Ile Ser Gly Phe Phe Asp Met Glu Asp Phe Gly Arg Arg
50 55 60
Phe Gly Asn Tyr Gln Asp Ala Asp Gly Trp Val Phe Ser Ala Tyr Arg
65 70 75 80
Gln Gly Ala Ile Val Ala Leu Leu Pro Ala Gly Ala Leu Leu Gly Ser
85 90 95
Leu Val Ala Gly Arg Ile Ala Asp Thr Leu Gly Arg Arg Ile Ala Ile
100 105 110
Ser Ala Ser Ala Leu Phe Ser Cys Ile Gly Thr Ile Ile Glu Ile Ala
115 120 125
Ser Thr Thr His Trp Ala Gln Phe Ala Val Gly Arg Leu Ile Thr Gly
130 135 140
Ile Gly Ile Gly Ala Leu Ser Val Val Val Pro Met Tyr Gln Ser Glu
145 150 155 160
Ser Ala Pro Ala Ile Leu Arg Gly Ile Leu Val Ser Cys Tyr Gln Leu
165 170 175
Phe Ile Thr Leu Gly Ile Trp Thr Ala Glu Met Ile Asn Tyr Gly Thr
180 185 190
His Asp Leu Ser Asn Ser Ala Ser Trp Arg Ile Pro Asn Gly Ile Ser
195 200 205
Phe Leu Trp Ala Leu Val Leu Gly Gly Gly Ile Leu Phe Leu Pro Glu
210 215 220
Ser Pro Arg Tyr Ala Tyr Arg Val Gly Arg Glu Asp Glu Ala Arg Asn
225 230 235 240
Thr Ile Ala Arg Leu Ala Gly Leu Glu Pro Ser Ala Arg Ser Val Asn
245 250 255
Met Gln Ile Asp Glu Ile Arg Met Lys Leu Glu Glu Glu Lys Ala Gly
260 265 270
Ala Asp Thr Lys Trp Tyr Glu Ile Phe Gly Pro Ala Leu Leu Arg Arg
275 280 285
Thr Leu Ile Gly Ile Ile Leu Gln Ser Gly Gln Gln Leu Thr Gly Ala
290 295 300
Asn Phe Phe Phe Tyr Tyr Gly Thr Thr Ile Phe Lys Ala Thr Gly Leu
305 310 315 320
Ser Asp Ser Tyr Val Thr Gln Ile Ile Leu Gly Ser Val Asn Ala Gly
325 330 335
Cys Thr Val Ala Gly Leu Trp Val Val Lys Asn Val Gly Arg Arg Lys
340 345 350
Ala Leu Ile Gly Gly Ala Leu Trp Met Thr Met Cys Phe Leu Val Tyr
355 360 365
Ser Phe Val Gly Arg Phe Val Leu Asp Pro Val Asn Pro Ala Ser Thr
370 375 380
Pro Gln Ala Gly Asn Val Leu Ile Val Phe Ser Cys Phe Phe Ile Val
385 390 395 400
Ala Phe Ala Thr Thr Trp Gly Pro Leu Val Trp Ala Val Val Ala Glu
405 410 415
Leu Tyr Pro Ala Arg Tyr Arg Ala Pro Ala Met Ala Leu Ala Thr Ala
420 425 430
Ser Asn Trp Leu Trp Asn Phe Leu Met Ser Leu Phe Thr Arg Pro Ile
435 440 445
Thr Asp Ser Ile Gly Tyr Phe Tyr Gly Leu Val Phe Ala Gly Cys Cys
450 455 460
Leu Ala Leu Ala Ala Phe Val Trp Leu Phe Val Ile Glu Ser Lys Asp
465 470 475 480
Arg Thr Leu Glu Glu Ile Glu Thr Met Tyr Asn Gln Lys Val Ser Pro
485 490 495
Arg His Ser Thr His Trp His Ala Glu Val Pro Ser Gly Pro Arg Asp
500 505 510
Ala Glu Glu Lys Pro Glu Val His Ser Gly Ser Ala Thr Thr Ser Ser
515 520 525
His Gly Glu Val
530
<210> 3
<211> 1647
<212> DNA
<213> 粗糙脉胞孢霉(Neurospora crassa)
<400> 3
atg gtt ctg ggg aaa aag tca atc aaa atc aat ggc gcc gac att ggc 48
Met Val Leu Gly Lys Lys Ser Ile Lys Ile Asn Gly Ala Asp Ile Gly
1 5 10 15
gtc gaa gcc atc ctt ctc ggt gcc gtc acc gcg ata gga ggc ttc ctt 96
Val Glu Ala Ile Leu Leu Gly Ala Val Thr Ala Ile Gly Gly Phe Leu
20 25 30
ttc ggc tat gac acc ggt cag atc tcg ggc atg ctt cta ttc agc gac 144
Phe Gly Tyr Asp Thr Gly Gln Ile Ser Gly Met Leu Leu Phe Ser Asp
35 40 45
ttc aag aac aga ttt ggc cag atc acc caa cca gat gga tca aag gaa 192
Phe Lys Asn Arg Phe Gly Gln Ile Thr Gln Pro Asp Gly Ser Lys Glu
50 55 60
ttc gag tcc atc atc cag tca ctg cta gta tct ctt atg agt atc ggt 240
Phe Glu Ser Ile Ile Gln Ser Leu Leu Val Ser Leu Met Ser Ile Gly
65 70 75 80
aca ctc ctc ggt tct ttg act tca tca tac acc gcc acc tgg tgg ggc 288
Thr Leu Leu Gly Ser Leu Thr Ser Ser Tyr Thr Ala Thr Trp Trp Gly
85 90 95
cgc cgc aag tcc ctt aca ttc ggc gtc ggt ctc ttc atc atc ggc aac 336
Arg Arg Lys Ser Leu Thr Phe Gly Val Gly Leu Phe Ile Ile Gly Asn
100 105 110
atc att cag atc acc gca atg cac tcc tgg gta cac atg atg atg ggc 384
Ile Ile Gln Ile Thr Ala Met His Ser Trp Val His Met Met Met Gly
115 120 125
cgc ttc gtc gcc ggt cta ggc gtc ggt acc ctc tcc gtc ggt gtg ccc 432
Arg Phe Val Ala Gly Leu Gly Val Gly Thr Leu Ser Val Gly Val Pro
130 135 140
atg ttc caa tcc gag tgc tcc cct aga gaa atc cgt ggt gcc gtc gtc 480
Met Phe Gln Ser Glu Cys Ser Pro Arg Glu Ile Arg Gly Ala Val Val
145 150 155 160
gcc tct tac caa ctg ctc atc acc ttt ggt atc ctc atc gcc aac att 528
Ala Ser Tyr Gln Leu Leu Ile Thr Phe Gly Ile Leu Ile Ala Asn Ile
165 170 175
gtc aac tac ggc gtg cgc gag atc cag gag cag gac gct tcc tgg cgt 576
Val Asn Tyr Gly Val Arg Glu Ile Gln Glu Gln Asp Ala Ser Trp Arg
180 185 190
atc gtc att ggt ttg ggt atc ttt ttc tcg ctt ccg ctt ggc gtg gga 624
Ile Val Ile Gly Leu Gly Ile Phe Phe Ser Leu Pro Leu Gly Val Gly
195 200 205
gtg ctg ttg gtc ccc gaa agc cca agg tgg ctg gcg tcc aag gag gat 672
Val Leu Leu Val Pro Glu Ser Pro Arg Trp Leu Ala Ser Lys Glu Asp
210 215 220
tgg gag gga gcc cgc atg tcg atg gcg aga ctg aga ggc ttg aag cat 720
Trp Glu Gly Ala Arg Met Ser Met Ala Arg Leu Arg Gly Leu Lys His
225 230 235 240
gac ccg cat aac gag ctg gtg gag gat gat atg aag gag atg cgg gag 768
Asp Pro His Asn Glu Leu Val Glu Asp Asp Met Lys Glu Met Arg Glu
245 250 255
gtt ttg gag aaa gag cgg act gcg gcc gtg gga act tgg aag gag tgt 816
Val Leu Glu Lys Glu Arg Thr Ala Ala Val Gly Thr Trp Lys Glu Cys
260 265 270
ttc atc ccc aac aag aac ggt gtt ccc aag cag gtg tac cgc aca ttc 864
Phe Ile Pro Asn Lys Asn Gly Val Pro Lys Gln Val Tyr Arg Thr Phe
275 280 285
ctg ggt atc ggc atc cac ttc ctg caa cag tgg act ggc gtc aac tac 912
Leu Gly Ile Gly Ile His Phe Leu Gln Gln Trp Thr Gly Val Asn Tyr
290 295 300
ttc ttc tac tat ggc gcc acc atc ttc cag tcg gcg ggt atc aag gac 960
Phe Phe Tyr Tyr Gly Ala Thr Ile Phe Gln Ser Ala Gly Ile Lys Asp
305 310 315 320
ccc att caa aca cag ctc atc ctc ggc gct gtc aac gtc ttc tca acc 1008
Pro Ile Gln Thr Gln Leu Ile Leu Gly Ala Val Asn Val Phe Ser Thr
325 330 335
ctg ttc ggt ctc tgg gtc gtc gag cgc ttc gga cgc aga tgg ccg ctg 1056
Leu Phe Gly Leu Trp Val Val Glu Arg Phe Gly Arg Arg Trp Pro Leu
340 345 350
ttc atc ggt gct atc tgg cag gcg tct tgg ctg gcc gtg ttc gct tcc 1104
Phe Ile Gly Ala Ile Trp Gln Ala Ser Trp Leu Ala Val Phe Ala Ser
355 360 365
atg ggc acc gct ctc gaa ccc gac caa aac aag gcg tcg ggt att gtc 1152
Met Gly Thr Ala Leu Glu Pro Asp Gln Asn Lys Ala Ser Gly Ile Val
370 375 380
atg atc gtc tcg gcc gca ctc ttc atc gct tcc ttt gct tgc acc tgg 1200
Met Ile Val Ser Ala Ala Leu Phe Ile Ala Ser Phe Ala Cys Thr Trp
385 390 395 400
ggt ccc att gct tgg gtc gtc atc ggc gag agt ttc cct ctg cgc acc 1248
Gly Pro Ile Ala Trp Val Val Ile Gly Glu Ser Phe Pro Leu Arg Thr
405 410 415
cgt gcc aag cag gct tcg ctt gcc acg gct ggt aac tgg ctt gga aac 1296
Arg Ala Lys Gln Ala Ser Leu Ala Thr Ala Gly Asn Trp Leu Gly Asn
420 425 430
ttc atg atc tcc ttc ctc acc ccc ctc gcc acc gat ggc atc ggc tat 1344
Phe Met Ile Ser Phe Leu Thr Pro Leu Ala Thr Asp Gly Ile Gly Tyr
435 440 445
gcc tac ggc tac gtc ttt gta gcc acc aac atc atg ggc gca ctt ttg 1392
Ala Tyr Gly Tyr Val Phe Val Ala Thr Asn Ile Met Gly Ala Leu Leu
450 455 460
gtc tgg ttc ttc ctg tac gag tcc acc agc ttg tcc ctc gag aac gtc 1440
Val Trp Phe Phe Leu Tyr Glu Ser Thr Ser Leu Ser Leu Glu Asn Val
465 470 475 480
gac ctc atg tac agc gag ccg ggc atc aag ccg tgg aac tcg cac aag 1488
Asp Leu Met Tyr Ser Glu Pro Gly Ile Lys Pro Trp Asn Ser His Lys
485 490 495
tgg atg ccg cct ggc tac atc acg cgc atg cag cgc gat gat gag tac 1536
Trp Met Pro Pro Gly Tyr Ile Thr Arg Met Gln Arg Asp Asp Glu Tyr
500 505 510
ttc cac cat cct gag aag ggt ggt tcg gat ggt ctt gaa atc acg aat 1584
Phe His His Pro Glu Lys Gly Gly Ser Asp Gly Leu Glu Ile Thr Asn
515 520 525
gga agc aag gag atg cac ccc cac gag gag agg gaa gag aag gtt att 1632
Gly Ser Lys Glu Met His Pro His Glu Glu Arg Glu Glu Lys Val Ile
530 535 540
aac cat agg gtt tag 1647
Asn His Arg Val
545
<210> 4
<211> 548
<212> PRT
<213> 粗糙脉胞孢霉(Neurospora crassa)
<400> 4
Met Val Leu Gly Lys Lys Ser Ile Lys Ile Asn Gly Ala Asp Ile Gly
1 5 10 15
Val Glu Ala Ile Leu Leu Gly Ala Val Thr Ala Ile Gly Gly Phe Leu
20 25 30
Phe Gly Tyr Asp Thr Gly Gln Ile Ser Gly Met Leu Leu Phe Ser Asp
35 40 45
Phe Lys Asn Arg Phe Gly Gln Ile Thr Gln Pro Asp Gly Ser Lys Glu
50 55 60
Phe Glu Ser Ile Ile Gln Ser Leu Leu Val Ser Leu Met Ser Ile Gly
65 70 75 80
Thr Leu Leu Gly Ser Leu Thr Ser Ser Tyr Thr Ala Thr Trp Trp Gly
85 90 95
Arg Arg Lys Ser Leu Thr Phe Gly Val Gly Leu Phe Ile Ile Gly Asn
100 105 110
Ile Ile Gln Ile Thr Ala Met His Ser Trp Val His Met Met Met Gly
115 120 125
Arg Phe Val Ala Gly Leu Gly Val Gly Thr Leu Ser Val Gly Val Pro
130 135 140
Met Phe Gln Ser Glu Cys Ser Pro Arg Glu Ile Arg Gly Ala Val Val
145 150 155 160
Ala Ser Tyr Gln Leu Leu Ile Thr Phe Gly Ile Leu Ile Ala Asn Ile
165 170 175
Val Asn Tyr Gly Val Arg Glu Ile Gln Glu Gln Asp Ala Ser Trp Arg
180 185 190
Ile Val Ile Gly Leu Gly Ile Phe Phe Ser Leu Pro Leu Gly Val Gly
195 200 205
Val Leu Leu Val Pro Glu Ser Pro Arg Trp Leu Ala Ser Lys Glu Asp
210 215 220
Trp Glu Gly Ala Arg Met Ser Met Ala Arg Leu Arg Gly Leu Lys His
225 230 235 240
Asp Pro His Asn Glu Leu Val Glu Asp Asp Met Lys Glu Met Arg Glu
245 250 255
Val Leu Glu Lys Glu Arg Thr Ala Ala Val Gly Thr Trp Lys Glu Cys
260 265 270
Phe Ile Pro Asn Lys Asn Gly Val Pro Lys Gln Val Tyr Arg Thr Phe
275 280 285
Leu Gly Ile Gly Ile His Phe Leu Gln Gln Trp Thr Gly Val Asn Tyr
290 295 300
Phe Phe Tyr Tyr Gly Ala Thr Ile Phe Gln Ser Ala Gly Ile Lys Asp
305 310 315 320
Pro Ile Gln Thr Gln Leu Ile Leu Gly Ala Val Asn Val Phe Ser Thr
325 330 335
Leu Phe Gly Leu Trp Val Val Glu Arg Phe Gly Arg Arg Trp Pro Leu
340 345 350
Phe Ile Gly Ala Ile Trp Gln Ala Ser Trp Leu Ala Val Phe Ala Ser
355 360 365
Met Gly Thr Ala Leu Glu Pro Asp Gln Asn Lys Ala Ser Gly Ile Val
370 375 380
Met Ile Val Ser Ala Ala Leu Phe Ile Ala Ser Phe Ala Cys Thr Trp
385 390 395 400
Gly Pro Ile Ala Trp Val Val Ile Gly Glu Ser Phe Pro Leu Arg Thr
405 410 415
Arg Ala Lys Gln Ala Ser Leu Ala Thr Ala Gly Asn Trp Leu Gly Asn
420 425 430
Phe Met Ile Ser Phe Leu Thr Pro Leu Ala Thr Asp Gly Ile Gly Tyr
435 440 445
Ala Tyr Gly Tyr Val Phe Val Ala Thr Asn Ile Met Gly Ala Leu Leu
450 455 460
Val Trp Phe Phe Leu Tyr Glu Ser Thr Ser Leu Ser Leu Glu Asn Val
465 470 475 480
Asp Leu Met Tyr Ser Glu Pro Gly Ile Lys Pro Trp Asn Ser His Lys
485 490 495
Trp Met Pro Pro Gly Tyr Ile Thr Arg Met Gln Arg Asp Asp Glu Tyr
500 505 510
Phe His His Pro Glu Lys Gly Gly Ser Asp Gly Leu Glu Ile Thr Asn
515 520 525
Gly Ser Lys Glu Met His Pro His Glu Glu Arg Glu Glu Lys Val Ile
530 535 540
Asn His Arg Val
545
<210> 5
<211> 1662
<212> DNA
<213> 粗糙脉胞孢霉(Neurospora crassa)
<400> 5
atg aag cca ttt ctg ggg ctc aga ggt cag ccc ctc aac ctg gcc gtg 48
Met Lys Pro Phe Leu Gly Leu Arg Gly Gln Pro Leu Asn Leu Ala Val
1 5 10 15
ggc gca gtt gcc gga tgt gat ttt ctt ctt ttc gga tac gat cag ggt 96
Gly Ala Val Ala Gly Cys Asp Phe Leu Leu Phe Gly Tyr Asp Gln Gly
20 25 30
gtt atg ggc gga att ctg act ctc ccc gag ttc ctc ggt tac ttt gaa 144
Val Met Gly Gly Ile Leu Thr Leu Pro Glu Phe Leu Gly Tyr Phe Glu
35 40 45
caa atc aat cca gac gca ccg ggc ctc aca cca cac gaa tct tct atg 192
Gln Ile Asn Pro Asp Ala Pro Gly Leu Thr Pro His Glu Ser Ser Met
50 55 60
cga tct act tac caa ggc atc tcc gtc gct tcc tac aac ctg gga tgt 240
Arg Ser Thr Tyr Gln Gly Ile Ser Val Ala Ser Tyr Asn Leu Gly Cys
65 70 75 80
ttt atc ggc gcc atc atc act atc ttc att ggc aat ccc tgg ggc cgt 288
Phe Ile Gly Ala Ile Ile Thr Ile Phe Ile Gly Asn Pro Trp Gly Arg
85 90 95
aag aaa ata ata ttg ctc ggg act tct atc atg att gtc ggt gcc att 336
Lys Lys Ile Ile Leu Leu Gly Thr Ser Ile Met Ile Val Gly Ala Ile
100 105 110
ctt cag gcc agc gct act act cta ggc cat ttc atc att ggg cgt atc 384
Leu Gln Ala Ser Ala Thr Thr Leu Gly His Phe Ile Ile Gly Arg Ile
115 120 125
att acc ggc att gga aac gga gga aac aca tct act gtc cct act tgg 432
Ile Thr Gly Ile Gly Asn Gly Gly Asn Thr Ser Thr Val Pro Thr Trp
130 135 140
caa tcc gag act tcg aaa gca cac aag cga ggc aag atg gtc atg atc 480
Gln Ser Glu Thr Ser Lys Ala His Lys Arg Gly Lys Met Val Met Ile
145 150 155 160
gaa gga tct ctc gtc acc gcc ggc atc atg ttg agt tac tgg att gac 528
Glu Gly Ser Leu Val Thr Ala Gly Ile Met Leu Ser Tyr Trp Ile Asp
165 170 175
ctc ggg tta agt ttt gcc cct ggc tca gtt gct tgg cgc ttc cct ctt 576
Leu Gly Leu Ser Phe Ala Pro Gly Ser Val Ala Trp Arg Phe Pro Leu
180 185 190
gcc ttc cag atc atc ttc tgc atc ctc atc ctc atc ttt att ccc ttc 624
Ala Phe Gln Ile Ile Phe Cys Ile Leu Ile Leu Ile Phe Ile Pro Phe
195 200 205
ctg ccc gaa tca cct cgt tgg ttg gtt ttc aaa ggc cgc gac gcg gaa 672
Leu Pro Glu Ser Pro Arg Trp Leu Val Phe Lys Gly Arg Asp Ala Glu
210 215 220
gcc aag gaa atc ctg gcc gcc ctc aac gac gtg gaa ctt gac gat ccc 720
Ala Lys Glu Ile Leu Ala Ala Leu Asn Asp Val Glu Leu Asp Asp Pro
225 230 235 240
att gtc gac acc gag ttc cat ttc atc cac gat acg gtt gtc gag atg 768
Ile Val Asp Thr Glu Phe His Phe Ile His Asp Thr Val Val Glu Met
245 250 255
tcc aaa ggc agc ttc aag gac ctc ttc acc atg gac aag gac cgc aac 816
Ser Lys Gly Ser Phe Lys Asp Leu Phe Thr Met Asp Lys Asp Arg Asn
260 265 270
ttc cac cgc act ctg ctg gca tac ctc aac cag gtt ttc cag cag atc 864
Phe His Arg Thr Leu Leu Ala Tyr Leu Asn Gln Val Phe Gln Gln Ile
275 280 285
tct ggt ata aac ctc atc acc tac tat gcc gcc gtc atc tac agc gga 912
Ser Gly Ile Asn Leu Ile Thr Tyr Tyr Ala Ala Val Ile Tyr Ser Gly
290 295 300
ctt ggc atg tcc gac ttc ctg gcc cgt ctc ctc gcc gcc ctc aac ggc 960
Leu Gly Met Ser Asp Phe Leu Ala Arg Leu Leu Ala Ala Leu Asn Gly
305 310 315 320
acc gag tac ttc att gcc tcg tgg cca gcc gtc ttc ctt gtc gag cgc 1008
Thr Glu Tyr Phe Ile Ala Ser Trp Pro Ala Val Phe Leu Val Glu Arg
325 330 335
gtc ggc cgc cgc aag ctc atg ttg ttt ggt gcc atc ggc caa gcg gcc 1056
Val Gly Arg Arg Lys Leu Met Leu Phe Gly Ala Ile Gly Gln Ala Ala
340 345 350
acc atg gcc atc ctg gca ggc gtc aac tcc cgg cca gac gac aag cca 1104
Thr Met Ala Ile Leu Ala Gly Val Asn Ser Arg Pro Asp Asp Lys Pro
355 360 365
tac caa att gcg gga atc gtc ttt ctg ttc gtc ttc aac acg gtt ttc 1152
Tyr Gln Ile Ala Gly Ile Val Phe Leu Phe Val Phe Asn Thr Val Phe
370 375 380
gcg gtc ggc tgg ctc ggc atg tcc tgg ctc tac cca gcc gag atc gtg 1200
Ala Val Gly Trp Leu Gly Met Ser Trp Leu Tyr Pro Ala Glu Ile Val
385 390 395 400
ccg ctc cgc atc cgc gcc ccg gct aac gcg cta tcc acg tcg gcg aac 1248
Pro Leu Arg Ile Arg Ala Pro Ala Asn Ala Leu Ser Thr Ser Ala Asn
405 410 415
tgg atc ttc aac ttt atg gtt gtc atg atc act ccg gtt gcg ttc aac 1296
Trp Ile Phe Asn Phe Met Val Val Met Ile Thr Pro Val Ala Phe Asn
435 440 445
aaa atc aag tat cag acg tac atc atc ttc gcc gtc atc aac gcc ttt 1344
Lys Ile Lys Tyr Gln Thr Tyr Ile Ile Phe Ala Val Ile Asn Ala Phe
450 455 460
att gtt cca gtc gtc tat ttc ttc tat ccg gaa acg gcg tgc cgg tca 1392
Ile Val Pro Val Val Tyr Phe Phe Tyr Pro Glu Thr Ala Cys Arg Ser
450 455 460
cta gag gaa atg gat atg atc ttc cac aag gtt gat ggg tgg aag ggg 1440
Leu Glu Glu Met Asp Met Ile Phe His Lys Val Asp Gly Trp Lys Gly
465 470 475 480
tac ttc acg gta gtg cac cag gcc aag gtt gag cct aaa tgg tac gac 1488
Tyr Phe Thr Val Val His Gln Ala Lys Val Glu Pro Lys Trp Tyr Asp
485 490 495
aag gat ggc cag cgc att ggc ggg gct gac ttt gag aag act gct ggg 1536
Lys Asp Gly Gln Arg Ile Gly Gly Ala Asp Phe Glu Lys Thr Ala Gly
500 505 510
tac cag agc cac tcg att ccg gag tcg tct gga ttt gaa aag ccg acg 1584
Tyr Gln Ser His Ser Ile Pro Glu Ser Ser Gly Phe Glu Lys Pro Thr
515 520 525
aag gcg cat gtt gag tcg cct agg gcg gat gac ggg atc act tcg tcg 1632
Lys Ala His Val Glu Ser Pro Arg Ala Asp Asp Gly Ile Thr Ser Ser
530 535 540
agt agt gat gga ggt aat cgg gag tcg tag 1662
Ser Ser Asp Gly Gly Asn Arg Glu Ser
545 550
<210> 6
<211> 553
<212> PRT
<213> 粗糙脉胞孢霉(Neurospora crassa)
<400> 6
Met Lys Pro Phe Leu Gly Leu Arg Gly Gln Pro Leu Asn Leu Ala Val
1 5 10 15
Gly Ala Val Ala Gly Cys Asp Phe Leu Leu Phe Gly Tyr Asp Gln Gly
20 25 30
Val Met Gly Gly Ile Leu Thr Leu Pro Glu Phe Leu Gly Tyr Phe Glu
35 40 45
Gln Ile Asn Pro Asp Ala Pro Gly Leu Thr Pro His Glu Ser Ser Met
50 55 60
Arg Ser Thr Tyr Gln Gly Ile Ser Val Ala Ser Tyr Asn Leu Gly Cys
65 70 75 80
Phe Ile Gly Ala Ile Ile Thr Ile Phe Ile Gly Asn Pro Trp Gly Arg
85 90 95
Lys Lys Ile Ile Leu Leu Gly Thr Ser Ile Met Ile Val Gly Ala Ile
100 105 110
Leu Gln Ala Ser Ala Thr Thr Leu Gly His Phe Ile Ile Gly Arg Ile
115 120 125
Ile Thr Gly Ile Gly Asn Gly Gly Asn Thr Ser Thr Val Pro Thr Trp
130 135 140
Gln Ser Glu Thr Ser Lys Ala His Lys Arg Gly Lys Met Val Met Ile
145 150 155 160
Glu Gly Ser Leu Val Thr Ala Gly Ile Met Leu Ser Tyr Trp Ile Asp
165 170 175
Leu Gly Leu Ser Phe Ala Pro Gly Ser Val Ala Trp Arg Phe Pro Leu
180 185 190
Ala Phe Gln Ile Ile Phe Cys Ile Leu Ile Leu Ile Phe Ile Pro Phe
195 200 205
Leu Pro Glu Ser Pro Arg Trp Leu Val Phe Lys Gly Arg Asp Ala Glu
210 215 220
Ala Lys Glu Ile Leu Ala Ala Leu Asn Asp Val Glu Leu Asp Asp Pro
225 230 235 240
Ile Val Asp Thr Glu Phe His Phe Ile His Asp Thr Val Val Glu Met
245 250 255
Ser Lys Gly Ser Phe Lys Asp Leu Phe Thr Met Asp Lys Asp Arg Asn
260 265 270
Phe His Arg Thr Leu Leu Ala Tyr Leu Asn Gln Val Phe Gln Gln Ile
275 280 285
Ser Gly Ile Asn Leu Ile Thr Tyr Tyr Ala Ala Val Ile Tyr Ser Gly
290 295 300
Leu Gly Met Ser Asp Phe Leu Ala Arg Leu Leu Ala Ala Leu Asn Gly
305 310 315 320
Thr Glu Tyr Phe Ile Ala Ser Trp Pro Ala Val Phe Leu Val Glu Arg
325 330 335
Val Gly Arg Arg Lys Leu Met Leu Phe Gly Ala Ile Gly Gln Ala Ala
340 345 350
Thr Met Ala Ile Leu Ala Gly Val Asn Ser Arg Pro Asp Asp Lys Pro
355 360 365
Tyr Gln Ile Ala Gly Ile Val Phe Leu Phe Val Phe Asn Thr Val Phe
370 375 380
Ala Val Gly Trp Leu Gly Met Ser Trp Leu Tyr Pro Ala Glu Ile Val
385 390 395 400
Pro Leu Arg Ile Arg Ala Pro Ala Asn Ala Leu Ser Thr Ser Ala Asn
405 410 415
Trp Ile Phe Asn Phe Met Val Val Met Ile Thr Pro Val Ala Phe Asn
420 425 430
Lys Ile Lys Tyr Gln Thr Tyr Ile Ile Phe Ala Val Ile Asn Ala Phe
435 440 445
Ile Val Pro Val Val Tyr Phe Phe Tyr Pro Glu Thr Ala Cys Arg Ser
450 455 460
Leu Glu Glu Met Asp Met Ile Phe His Lys Val Asp Gly Trp Lys Gly
465 470 475 480
Tyr Phe Thr Val Val His Gln Ala Lys Val Glu Pro Lys Trp Tyr Asp
485 490 495
Lys Asp Gly Gln Arg Ile Gly Gly Ala Asp Phe Glu Lys Thr Ala Gly
500 505 510
Tyr Gln Ser His Ser Ile Pro Glu Ser Ser Gly Phe Glu Lys Pro Thr
515 520 525
Lys Ala His Val Glu Ser Pro Arg Ala Asp Asp Gly Ile Thr Ser Ser
530 535 540
Ser Ser Asp Gly Gly Asn Arg Glu Ser
545 550
<210> 7
<211> 1557
<212> DNA
<213> 粗糙脉胞孢霉(Neurospora crassa)
<400> 7
atg ggg ctc ggg ctt aag cta ccg aca atc tac aat gtc cac ctt gtg 48
Met Gly Leu Gly Leu Lys Leu Pro Thr Ile Tyr Asn Val His Leu Val
1 5 10 15
gct atc att gcc acc ttg ggt ggt gca ctc ttc ggc ttt gat atc tcc 96
Ala Ile Ile Ala Thr Leu Gly Gly Ala Leu Phe Gly Phe Asp Ile Ser
20 25 30
tcc atg tcc gcc atc gtc gtg acc gac caa tac ctc acc tac ttc aac 144
Ser Met Ser Ala Ile Val Val Thr Asp Gln Tyr Leu Thr Tyr Phe Asn
35 40 45
aac ccc cat gat atc atc caa gga gcc atc ggc tct gcc ctt gct gct 192
Asn Pro His Asp Ile Ile Gln Gly Ala Ile Gly Ser Ala Leu Ala Ala
50 55 60
ggc tcc gtc gtc ggt tcc gcc atc gcc ggt cct ctt tcc gac aag atc 240
Gly Ser Val Val Gly Ser Ala Ile Ala Gly Pro Leu Ser Asp Lys Ile
65 70 75 80
ggt cgt cgt gac tcc atc ttt ttc gcc tgc ttc ttc tgg ctc att ggt 288
Gly Arg Arg Asp Ser Ile Phe Phe Ala Cys Phe Phe Trp Leu Ile Gly
85 90 95
acc tcc gtc cag gtt gcc tgc aag aac tat ggc cag ctc atc gcc ggc 336
Thr Ser Val Gln Val Ala Cys Lys Asn Tyr Gly Gln Leu Ile Ala Gly
100 105 110
cgt gtg ctc aac ggc ttt acc gtc ggc atc act tcc tcc cag gtt ccc 384
Arg Val Leu Asn Gly Phe Thr Val Gly Ile Thr Ser Ser Gln Val Pro
115 120 125
gtg tac ctt gcc gag atc gcc aag gca gag aag cgt ggt tcc ttg gtc 432
Val Tyr Leu Ala Glu Ile Ala Lys Ala Glu Lys Arg Gly Ser Leu Val
130 135 140
atc atc cag caa ctc gcc atc gag ttt ggt atc ttg atc atg tac ttt 480
Ile Ile Gln Gln Leu Ala Ile Glu Phe Gly Ile Leu Ile Met Tyr Phe
145 150 155 160
atc ggc tac ggc tgt gcg tcg atc gag ggc cct gct tcg ttc cgg acc 528
Ile Gly Tyr Gly Cys Ala Ser Ile Glu Gly Pro Ala Ser Phe Arg Thr
165 170 175
gct tgg ggc att cag ttt atc cct tgc ttt ttc ctc atg gtc ggt ctt 576
Ala Trp Gly Ile Gln Phe Ile Pro Cys Phe Phe Leu Met Val Gly Leu
180 185 190
ccc ttc ttg cct agg tcg ccc aga tgg ctg gcc aag gtc ggt agg gac 624
Pro Phe Leu Pro Arg Ser Pro Arg Trp Leu Ala Lys Val Gly Arg Asp
195 200 205
cag gag gcc att gct gtc ctg gct aac atc cag gct gat ggc aac gtt 672
Gln Glu Ala Ile Ala Val Leu Ala Asn Ile Gln Ala Asp Gly Asn Val
210 215 220
gat gac ccg aga gtc gtt gct gag tgg gag gag att gtc acc gtt atg 720
Asp Asp Pro Arg Val Val Ala Glu Trp Glu Glu Ile Val Thr Val Met
225 230 235 240
aac gcc gag cgt gag gcc ggt aag gga tgg agg aag ttt gtc aag aac 768
Asn Ala Glu Arg Glu Ala Gly Lys Gly Trp Arg Lys Phe Val Lys Asn
245 250 255
ggc atg tgg aag cga acc atg gct ggc atg act gta cag gct tgg cag 816
Gly Met Trp Lys Arg Thr Met Ala Gly Met Thr Val Gln Ala Trp Gln
260 265 270
caa ctc gcc ggc gcc aac gta atc gtc tac tac cta acc tac atc gcc 864
Gln Leu Ala Gly Ala Asn Val Ile Val Tyr Tyr Leu Thr Tyr Ile Ala
275 280 285
caa atg gcc gga ctc aca ggc aac gtc gcc atg gta acc tcg ggc atc 912
Gln Met Ala Gly Leu Thr Gly Asn Val Ala Met Val Thr Ser Gly Ile
290 295 300
caa tac gcc gtt ttc atc atc ttc acc ggc gtc atg tgg ctc ttc atc 960
Gln Tyr Ala Val Phe Ile Ile Phe Thr Gly Val Met Trp Leu Phe Ile
305 310 315 320
gac aag acc ggt cgt cgc acc ctt tta gtt tac ggc gcc ttg gga atg 1008
Asp Lys Thr Gly Arg Arg Thr Leu Leu Val Tyr Gly Ala Leu Gly Met
325 330 335
gcc ttc tgc cac ttt gtc gtc ggc ggc gtc atg ggc gcg cac cac gac 1056
Ala Phe Cys His Phe Val Val Gly Gly Val Met Gly Ala His His Asp
340 345 350
aac gtt ccg gac ggc gtc ggc ggc aac gcc aac att gtc att agc gtg 1104
Asn Val Pro Asp Gly Val Gly Gly Asn Ala Asn Ile Val Ile Ser Val
355 360 365
cac aag ggc gcg ccc gcc aac acg gtc atc ctg ttc tcg tac ctg ctc 1152
His Lys Gly Ala Pro Ala Asn Thr Val Ile Leu Phe Ser Tyr Leu Leu
370 375 380
att gtc gtc tac gcc ttg acg ctc gct ccc gtc tgc tgg atc tac gcc 1200
Ile Val Val Tyr Ala Leu Thr Leu Ala Pro Val Cys Trp Ile Tyr Ala
385 390 395 400
gcc gag gtc tgg tcg ttg ggc act cgc gct acg ggc atg tcc atg gct 1248
Ala Glu Val Trp Ser Leu Gly Thr Arg Ala Thr Gly Met Ser Met Ala
405 410 415
gcc atg tcc aac tgg gtg ttc aac ttt gcg ctg ggc atg ttc acg ccg 1296
Ala Met Ser Asn Trp Val Phe Asn Phe Ala Leu Gly Met Phe Thr Pro
420 425 430
ccg gcg ttt gtc aat att acg tgg aag ctg ttt atc att ttc ggg gtg 1344
Pro Ala Phe Val Asn Ile Thr Trp Lys Leu Phe Ile Ile Phe Gly Val
435 440 445
ctt tgc gtc acg gcg gcg gtc tgg ttc tgg ttg ttt tac ccg gag acg 1392
Leu Cys Val Thr Ala Ala Val Trp Phe Trp Leu Phe Tyr Pro Glu Thr
450 455 460
tgt ggt aag acg ctg gag gag att gag atc ctg ttt ggt gat cag ggt 1440
Cys Gly Lys Thr Leu Glu Glu Ile Glu Ile Leu Phe Gly Asp Gln Gly
465 470 475 480
cct aag ccg tgg aag acg aag aag ggc gag tcg aga ctt acg gcg gag 1488
Pro Lys Pro Trp Lys Thr Lys Lys Gly Glu Ser Arg Leu Thr Ala Glu
485 490 495
att gag gct gtc aag gcg agg aag acg gtg gag cac gag att gag gtg 1536
Ile Glu Ala Val Lys Ala Arg Lys Thr Val Glu His Glu Ile Glu Val
500 505 510
cat gag cat gag aag gtt tag 1557
His Glu His Glu Lys Val
515
<210> 8
<211> 518
<212> PRT
<213> 粗糙脉胞孢霉(Neurospora crassa)
<400> 8
Met Gly Leu Gly Leu Lys Leu Pro Thr Ile Tyr Asn Val His Leu Val
1 5 10 15
Ala Ile Ile Ala Thr Leu Gly Gly Ala Leu Phe Gly Phe Asp Ile Ser
20 25 30
Ser Met Ser Ala Ile Val Val Thr Asp Gln Tyr Leu Thr Tyr Phe Asn
35 40 45
Asn Pro His Asp Ile Ile Gln Gly Ala Ile Gly Ser Ala Leu Ala Ala
50 55 60
Gly Ser Val Val Gly Ser Ala Ile Ala Gly Pro Leu Ser Asp Lys Ile
65 70 75 80
Gly Arg Arg Asp Ser Ile Phe Phe Ala Cys Phe Phe Trp Leu Ile Gly
85 90 95
Thr Ser Val Gln Val Ala Cys Lys Asn Tyr Gly Gln Leu Ile Ala Gly
100 105 110
Arg Val Leu Asn Gly Phe Thr Val Gly Ile Thr Ser Ser Gln Val Pro
115 120 125
Val Tyr Leu Ala Glu Ile Ala Lys Ala Glu Lys Arg Gly Ser Leu Val
130 135 140
Ile Ile Gln Gln Leu Ala Ile Glu Phe Gly Ile Leu Ile Met Tyr Phe
145 150 155 160
Ile Gly Tyr Gly Cys Ala Ser Ile Glu Gly Pro Ala Ser Phe Arg Thr
165 170 175
Ala Trp Gly Ile Gln Phe Ile Pro Cys Phe Phe Leu Met Val Gly Leu
180 185 190
Pro Phe Leu Pro Arg Ser Pro Arg Trp Leu Ala Lys Val Gly Arg Asp
195 200 205
Gln Glu Ala Ile Ala Val Leu Ala Asn Ile Gln Ala Asp Gly Asn Val
210 215 220
Asp Asp Pro Arg Val Val Ala Glu Trp Glu Glu Ile Val Thr Val Met
225 230 235 240
Asn Ala Glu Arg Glu Ala Gly Lys Gly Trp Arg Lys Phe Val Lys Asn
245 250 255
Gly Met Trp Lys Arg Thr Met Ala Gly Met Thr Val Gln Ala Trp Gln
260 265 270
Gln Leu Ala Gly Ala Asn Val Ile Val Tyr Tyr Leu Thr Tyr Ile Ala
275 280 285
Gln Met Ala Gly Leu Thr Gly Asn Val Ala Met Val Thr Ser Gly Ile
290 295 300
Gln Tyr Ala Val Phe Ile Ile Phe Thr Gly Val Met Trp Leu Phe Ile
305 310 315 320
Asp Lys Thr Gly Arg Arg Thr Leu Leu Val Tyr Gly Ala Leu Gly Met
325 330 335
Ala Phe Cys His Phe Val Val Gly Gly Val Met Gly Ala His His Asp
340 345 350
Asn Val Pro Asp Gly Val Gly Gly Asn Ala Asn Ile Val Ile Ser Val
355 360 365
His Lys Gly Ala Pro Ala Asn Thr Val Ile Leu Phe Ser Tyr Leu Leu
370 375 380
Ile Val Val Tyr Ala Leu Thr Leu Ala Pro Val Cys Trp Ile Tyr Ala
385 390 395 400
Ala Glu Val Trp Ser Leu Gly Thr Arg Ala Thr Gly Met Ser Met Ala
405 410 415
Ala Met Ser Asn Trp Val Phe Asn Phe Ala Leu Gly Met Phe Thr Pro
420 425 430
Pro Ala Phe Val Asn Ile Thr Trp Lys Leu Phe Ile Ile Phe Gly Val
435 440 445
Leu Cys Val Thr Ala Ala Val Trp Phe Trp Leu Phe Tyr Pro Glu Thr
450 455 460
Cys Gly Lys Thr Leu Glu Glu Ile Glu Ile Leu Phe Gly Asp Gln Gly
465 470 475 480
Pro Lys Pro Trp Lys Thr Lys Lys Gly Glu Ser Arg Leu Thr Ala Glu
485 490 495
Ile Glu Ala Val Lys Ala Arg Lys Thr Val Glu His Glu Ile Glu Val
500 505 510
His Glu His Glu Lys Val
515
<210> 9
<211> 1575
<212> DNA
<213> 嗜热毁丝霉(Myceliophora thermophila)
<400> 9
atg aag ctg ccc acg att tac aat gtc cag ctc gtg gcc atc atc gcg 48
Met Lys Leu Pro Thr Ile Tyr Asn Val Gln Leu Val Ala Ile Ile Ala
1 5 10 15
acc ctc ggt ggc atg ctg ttc ggg ttc gac att tct tcc atg tcg gcc 96
Thr Leu Gly Gly Met Leu Phe Gly Phe Asp Ile Ser Ser Met Ser Ala
20 25 30
atc gtc gtg acc gac cag tac att gag tac ttc aac aac cct acc ggg 144
Ile Val Val Thr Asp Gln Tyr Ile Glu Tyr Phe Asn Asn Pro Thr Gly
35 40 45
gtt atc caa ggc gcc atc ggg tcg gct ctc gcg gcc ggc tcg gtc gtc 192
Val Ile Gln Gly Ala Ile Gly Ser Ala Leu Ala Ala Gly Ser Val Val
50 55 60
ggt tct gcc gtg gca ggt ccg ctg tcc gac tgg atg ggc cgg cgc gac 240
Gly Ser Ala Val Ala Gly Pro Leu Ser Asp Trp Met Gly Arg Arg Asp
65 70 75 80
tcc atc atg ttt gcc tgc ttg ttc tgg ctc gtc ggg acg gcg gtc cag 288
Ser Ile Met Phe Ala Cys Leu Phe Trp Leu Val Gly Thr Ala Val Gln
85 90 95
gtg gcg acc cag aac gtc ggc cag ctc atc gcc ggc cgc gtg ctc aac 336
Val Ala Thr Gln Asn Val Gly Gln Leu Ile Ala Gly Arg Val Leu Asn
100 105 110
ggc ttc acc gtc ggc atc acg tcg tcc cag gtg ccc gtc tac ctg gcc 384
Gly Phe Thr Val Gly Ile Thr Ser Ser Gln Val Pro Val Tyr Leu Ala
115 120 125
gag atc gcc aag gcc gag aag cgc ggc tcc atc gtc atc atc cag cag 432
Glu Ile Ala Lys Ala Glu Lys Arg Gly Ser Ile Val Ile Ile Gln Gln
130 135 140
ctg gcc gtc gag ttc ggc atc ctc atc atg tac ttc atc ggc tac ggc 480
Leu Ala Val Glu Phe Gly Ile Leu Ile Met Tyr Phe Ile Gly Tyr Gly
145 150 155 160
tgc gcc tcc atc gag ggc acc ggc tcc ttc cgc acc gcc tgg ggc acc 528
Cys Ala Ser Ile Glu Gly Thr Gly Ser Phe Arg Thr Ala Trp Gly Thr
165 170 175
cag ttc atc ccc tgc gtc ttc ctc atg ctc ggc ctc ccc ttc ctc ccg 576
Gln Phe Ile Pro Cys Val Phe Leu Met Leu Gly Leu Pro Phe Leu Pro
180 185 190
cgc tcg ccc cga tgg ctc gcc aag gtc ggc cgc gac aag gag gcc atc 624
Arg Ser Pro Arg Trp Leu Ala Lys Val Gly Arg Asp Lys Glu Ala Ile
195 200 205
gag acc ctc gcc aac atc cag gcc gac ggc aac acc cag gac tcc cgc 672
Glu Thr Leu Ala Asn Ile Gln Ala Asp Gly Asn Thr Gln Asp Ser Arg
210 215 220
gtc att gcc gag tgg gag gag atc cag acc gtc atg cag gcc gag cgc 720
Val Ile Ala Glu Trp Glu Glu Ile Gln Thr Val Met Gln Ala Glu Arg
225 230 235 240
gag gcc ggc cgc ggc tgg agg aag ttc ttg ctc aac ggc atg tgg aag 768
Glu Ala Gly Arg Gly Trp Arg Lys Phe Leu Leu Asn Gly Met Trp Lys
245 250 255
cgt acc ctg gcc ggc atg tcg gtc cag gcg tgg cag caa ctg gct ggc 816
Arg Thr Leu Ala Gly Met Ser Val Gln Ala Trp Gln Gln Leu Ala Gly
260 265 270
gcc aac gtg atc gtt tac tac ctg act tac atc gct cag atg gct ggt 864
Ala Asn Val Ile Val Tyr Tyr Leu Thr Tyr Ile Ala Gln Met Ala Gly
275 280 285
ctg acc ggc gac gtc gcc atg gtt acg tcg ggt atc caa tat gcc gtc 912
Leu Thr Gly Asp Val Ala Met Val Thr Ser Gly Ile Gln Tyr Ala Val
290 295 300
ttc atc gtc ttc acc ggc atc atg tgg ctc ttc atc gac aag acc ggc 960
Phe Ile Val Phe Thr Gly Ile Met Trp Leu Phe Ile Asp Lys Thr Gly
305 310 315 320
cgc cgc acg ctg ctc atc tgg ggc gcc ctc ggc atg ggc ttc tgc cac 1008
Arg Arg Thr Leu Leu Ile Trp Gly Ala Leu Gly Met Gly Phe Cys His
325 330 335
ttc gtc gtc ggc ggc gtc atg ggc gcg cac tcg acc tac cac ccc gag 1056
Phe Val Val Gly Gly Val Met Gly Ala His Ser Thr Tyr His Pro Glu
340 345 350
ggc gtg ggc aac ccg ccc aac ggc aac atc gtc atc gcc gtc aac aag 1104
Gly Val Gly Asn Pro Pro Asn Gly Asn Ile Val Ile Ala Val Asn Lys
355 360 365
ggc gcg ccg gcc aac acg gtc atc acc ttc tcg tac ctg ctc atc gtc 1152
Gly Ala Pro Ala Asn Thr Val Ile Thr Phe Ser Tyr Leu Leu Ile Val
370 375 380
gtc tac gcg ctg acg ctg gcc ccg gtc tgc tgg atc tac gcg gcc gag 1200
Val Tyr Ala Leu Thr Leu Ala Pro Val Cys Trp Ile Tyr Ala Ala Glu
385 390 395 400
gtc tgg tcg ctg ggc acg cgg gcg acg ggc atg tcc ctg gcg gcc atg 1248
Val Trp Ser Leu Gly Thr Arg Ala Thr Gly Met Ser Leu Ala Ala Met
405 410 415
agc aac tgg gtc ttc aac ttc gcc ctc ggc atg ttc acc ccg ccg ggc 1296
Ser Asn Trp Val Phe Asn Phe Ala Leu Gly Met Phe Thr Pro Pro Gly
420 425 430
ttc gtc aac atc acc tgg aag ctc ttc atc atc ttc ggc gtc ctc tgc 1344
Phe Val Asn Ile Thr Trp Lys Leu Phe Ile Ile Phe Gly Val Leu Cys
435 440 445
gtc acc gcc gcc gcc tgg ttc ttc ctg ctc tgc ccg gag acg tgc ggc 1392
Val Thr Ala Ala Ala Trp Phe Phe Leu Leu Cys Pro Glu Thr Cys Gly
450 455 460
aag acg ctc gag gag atc gag ctc ctc ttc tcc ggc ccg gac gcc ccg 1440
Lys Thr Leu Glu Glu Ile Glu Leu Leu Phe Ser Gly Pro Asp Ala Pro
465 470 475 480
cac ccc tgg aac acc agg aag ggc gac tcc cgc ctc gcc gcc gag atc 1488
His Pro Trp Asn Thr Arg Lys Gly Asp Ser Arg Leu Ala Ala Glu Ile
485 490 495
gcc gcc gtc gag gcc cgc cgg cgc gag aag acc gag gcc ggc gag gtg 1536
Ala Ala Val Glu Ala Arg Arg Arg Glu Lys Thr Glu Ala Gly Glu Val
500 505 510
gag gcc gtg ccc tcc gtc ggc gag cag gag aag gtt taa 1575
Glu Ala Val Pro Ser Val Gly Glu Gln Glu Lys Val
515 520
<210> 10
<211> 524
<212> PRT
<213> 嗜热毁丝霉(Myceliophora thermophila)
<400> 10
Met Lys Leu Pro Thr Ile Tyr Asn Val Gln Leu Val Ala Ile Ile Ala
1 5 10 15
Thr Leu Gly Gly Met Leu Phe Gly Phe Asp Ile Ser Ser Met Ser Ala
20 25 30
Ile Val Val Thr Asp Gln Tyr Ile Glu Tyr Phe Asn Asn Pro Thr Gly
35 40 45
Val Ile Gln Gly Ala Ile Gly Ser Ala Leu Ala Ala Gly Ser Val Val
50 55 60
Gly Ser Ala Val Ala Gly Pro Leu Ser Asp Trp Met Gly Arg Arg Asp
65 70 75 80
Ser Ile Met Phe Ala Cys Leu Phe Trp Leu Val Gly Thr Ala Val Gln
85 90 95
Val Ala Thr Gln Asn Val Gly Gln Leu Ile Ala Gly Arg Val Leu Asn
100 105 110
Gly Phe Thr Val Gly Ile Thr Ser Ser Gln Val Pro Val Tyr Leu Ala
115 120 125
Glu Ile Ala Lys Ala Glu Lys Arg Gly Ser Ile Val Ile Ile Gln Gln
130 135 140
Leu Ala Val Glu Phe Gly Ile Leu Ile Met Tyr Phe Ile Gly Tyr Gly
145 150 155 160
Cys Ala Ser Ile Glu Gly Thr Gly Ser Phe Arg Thr Ala Trp Gly Thr
165 170 175
Gln Phe Ile Pro Cys Val Phe Leu Met Leu Gly Leu Pro Phe Leu Pro
180 185 190
Arg Ser Pro Arg Trp Leu Ala Lys Val Gly Arg Asp Lys Glu Ala Ile
195 200 205
Glu Thr Leu Ala Asn Ile Gln Ala Asp Gly Asn Thr Gln Asp Ser Arg
210 215 220
Val Ile Ala Glu Trp Glu Glu Ile Gln Thr Val Met Gln Ala Glu Arg
225 230 235 240
Glu Ala Gly Arg Gly Trp Arg Lys Phe Leu Leu Asn Gly Met Trp Lys
245 250 255
Arg Thr Leu Ala Gly Met Ser Val Gln Ala Trp Gln Gln Leu Ala Gly
260 265 270
Ala Asn Val Ile Val Tyr Tyr Leu Thr Tyr Ile Ala Gln Met Ala Gly
275 280 285
Leu Thr Gly Asp Val Ala Met Val Thr Ser Gly Ile Gln Tyr Ala Val
290 295 300
Phe Ile Val Phe Thr Gly Ile Met Trp Leu Phe Ile Asp Lys Thr Gly
305 310 315 320
Arg Arg Thr Leu Leu Ile Trp Gly Ala Leu Gly Met Gly Phe Cys His
325 330 335
Phe Val Val Gly Gly Val Met Gly Ala His Ser Thr Tyr His Pro Glu
340 345 350
Gly Val Gly Asn Pro Pro Asn Gly Asn Ile Val Ile Ala Val Asn Lys
355 360 365
Gly Ala Pro Ala Asn Thr Val Ile Thr Phe Ser Tyr Leu Leu Ile Val
370 375 380
Val Tyr Ala Leu Thr Leu Ala Pro Val Cys Trp Ile Tyr Ala Ala Glu
385 390 395 400
Val Trp Ser Leu Gly Thr Arg Ala Thr Gly Met Ser Leu Ala Ala Met
405 410 415
Ser Asn Trp Val Phe Asn Phe Ala Leu Gly Met Phe Thr Pro Pro Gly
420 425 430
Phe Val Asn Ile Thr Trp Lys Leu Phe Ile Ile Phe Gly Val Leu Cys
435 440 445
Val Thr Ala Ala Ala Trp Phe Phe Leu Leu Cys Pro Glu Thr Cys Gly
450 455 460
Lys Thr Leu Glu Glu Ile Glu Leu Leu Phe Ser Gly Pro Asp Ala Pro
465 470 475 480
His Pro Trp Asn Thr Arg Lys Gly Asp Ser Arg Leu Ala Ala Glu Ile
485 490 495
Ala Ala Val Glu Ala Arg Arg Arg Glu Lys Thr Glu Ala Gly Glu Val
500 505 510
Glu Ala Val Pro Ser Val Gly Glu Gln Glu Lys Val
515 520
<210> 11
<211> 32
<212> DNA
<213> 人工序列(artificial sequence)
<400> 11
cgcggatcca tgggtctctt ctcgaaaaag tc 32
<210> 12
<211> 32
<212> DNA
<213> 人工序列(artificial sequence)
<400> 12
ccggaattcc taaacctctc catggcttga gg 32
<210> 13
<211> 32
<212> DNA
<213> 人工序列(artificial sequence)
<400> 13
ggactagtat ggttctgggg aaaaagtcaa tc 32
<210> 14
<211> 33
<212> DNA
<213> 人工序列(artificial sequence)
<400> 14
cccaagcttc taaaccctat ggttaataac ctt 33
<210> 15
<211> 30
<212> DNA
<213> 人工序列(artificial sequence)
<400> 15
cgcggatcca tgaagccatt tctggggctc 30
<210> 16
<211> 32
<212> DNA
<213> 人工序列(artificial sequence)
<400> 16
cccaagcttc tacgactccc gattacctcc at 32
<210> 17
<211> 31
<212> DNA
<213> 人工序列(artificial sequence)
<400> 17
cgcggatcca tggggctcgg gcttaagcta c 31
<210> 18
<211> 32
<212> DNA
<213> 人工序列(artificial sequence)
<400> 18
cggaattcct aaaccttctc atgctcatgc ac 32
<210> 19
<211> 30
<212> DNA
<213> 人工序列(artificial sequence)
<400> 19
cgcggatcca tgaagctgcc cacgatttac 30
<210> 20
<211> 30
<212> DNA
<213> 人工序列(artificial sequence)
<400> 20
ccggaattct taaaccttct cctgctcgcc 30
<210> 21
<211> 36
<212> DNA
<213> 人工序列(artificial sequence)
<400> 21
gggaattcca tatggatctg tttagcttgc ctcgtc 36
<210> 22
<211> 30
<212> DNA
<213> 人工序列(artificial sequence)
<400> 22
atgggccccg acactggatg gcggcgttag 30
- 还没有人留言评论。精彩留言会获得点赞!