苯丙酮尿症相关基因突变检测试剂盒的制作方法
本发明涉及一种苯丙酮尿症相关基因突变检测试剂盒。
背景技术:
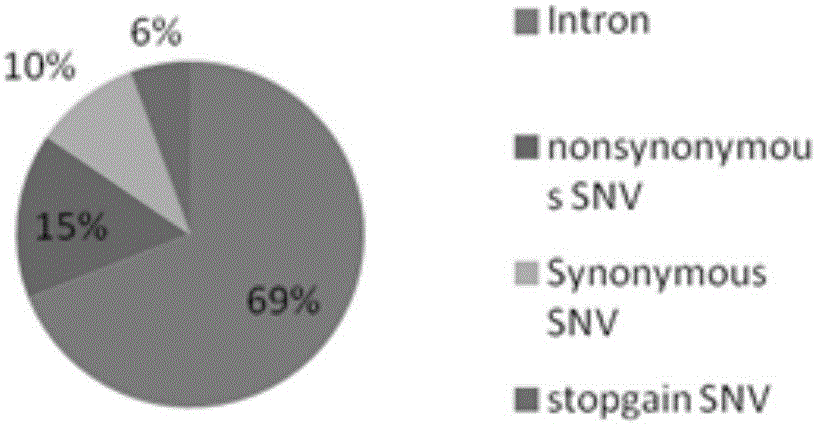
:苯丙酮尿症(Phenylketonuria,PKU)是最常见的氨基酸代谢障碍性疾病之一,属常染色体隐性遗传病。该病患儿出生时正常。通常3~6个月时始出现症状,1岁时症状明显,表现如下。①神经系统:智力发育落后最为突出,智商低于正常,有行为异常,如兴奋不安、忧郁等。可有癫痫小发作,少数呈现肌张力增高和腱反射亢进。②皮肤:患儿在出生数月后因黑色素合成不足,头发由黑变黄,皮肤白皙。皮肤湿疹较常见。③体味:由于尿和汗液中排出较多苯乙酸,可有明显鼠尿臭味。苯丙氨酸(phenylalanine,Phe)是人体必需的氨基酸之一,正常小儿每日需要量为200-500mg,其中1/3供机体合成组织蛋白;2/3则通过肝细胞中苯丙氨酸-4-羟化酶(PhenylalanineHydroxylase,PAH)的作用转化为酪氨酸,供合成甲状腺素、多巴胺、肾上腺素和黑色素等之用。在Phe的羟化过程中,除了PAH之外,还必需有辅酶四氢生物蝶呤(tetrahydrobiopterin,BH4)的参与,人体内的BH4来源于鸟苷三磷酸(GTP),在其转化和再生过程中参与作用的酶有鸟苷三磷酸环化水合酶(GCH1)、6-丙酮酰四氢蝶呤合成酶(PTS)、二氢生物蝶呤还原酶(QDPR)和蝶呤-4-甲醇胺脱水酶(PCBD1)等。上述五种酶的编码基因PAH、GCH1、PTS、QDPR、PCBD1,分别位于12q22-q24.2、14q22.1-q22.2、11q22.3、4p15.31和10q22,任一基因的突变都可能导致相关酶的活性缺陷,从而导致体内苯丙氨酸代谢紊乱[1]。本病按酶缺陷的不同可大致分为经典型和非经典型PKU两类,下面简单概述这两类PKU的发病原因。1.经典型PKU。绝大多数苯丙酮尿症病例是由于肝细胞先天性PAH缺陷所致的经典型PKU。PAH基因突变使酶蛋白合成不足或蛋白稳定性降低从而损伤酶活性,抑制苯丙氨酸转化为酪氨酸,故苯丙氨酸在血、脑脊液、各种组织和尿液中浓度显著增高。这种高浓度的苯丙氨酸竞争性地阻抑脑细胞代谢所必需的酪氨酸、色氨酸和支链氨基酸等通过脑实质的细胞质膜,使脑细胞不能正常合成神经递质;高浓度的Phe还抑制多核糖体的聚合,干扰脑组织中蛋白合成和髓鞘形成过程。因此,过量的Phe及其中间代谢产物可直接对处于生长发育阶段的婴幼儿神经及生理功能产生危害作用,尤其是导致患儿的智力发育障碍及语言发育迟缓等,严重者甚至危及生命。2.非经典型PKU。该类型仅占苯丙酮尿症病例的1%-3%,是由于鸟苷三磷酸环化水合酶(GCH1)、6-丙酮酰四氢蝶呤合成酶(PTS)、二氢生物蝶呤还原酶(QDPR)或蝶呤-4-甲醇胺脱水酶(PCBD1)基因突变导致相关酶缺乏使苯丙氨酸合成原料辅酶四氢生物蝶呤(BH4)缺乏所致,BH4是苯丙氨酸、酪氨酸和色氨酸等芳香氨基酸在羟化过程中所必须的辅酶,缺乏时不仅苯丙氨酸不能氧化成酪氨酸,而且造成多巴胺、5-羟色胺等重要的神经递质缺乏,加重神经系统的功能损伤。故BH4缺乏型PKU的病情一般较严重,治疗亦不易,如不经治疗,常在幼儿期死亡[2]。目前针对引起苯丙酮尿症的相关基因突变,多采用聚合酶链式反应(PCR)扩增目的基因的外显子及启动子区序列,然后应用Sanger测序的方法进行检测从而获得突变位点。该方法是参考GeneBank目标基因序列,设计引物分别扩增基因的外显子区和启动子区,采用Sanger测序的方法进行测序。其他方法还包括PCR-STR连锁分析(聚合酶链式反应-短串链重复序列连锁分析)、PCR-SSCP分析(聚合酶链式反应-单链构象的多态性分析)等,但多数只对经典型PKU的致病基因PAH进行突变检测。由于多个基因突变均可导致疾病发生,传统的Sanger测序等技术已经远远不能满足医学研究和临床应用的需求。而二代测序的方法应运而生,其具有通量高,速度快,价格低等优势,并且可以同时分析大量样本而得到广泛应用[3,4]。多重PCR富集二代测序技术是针对目的区域设计扩增引物,通过PCR扩增达到富集目的片段的目的,对研究者感兴趣的基因组区域进行富集。技术实现要素:本发明的目的在于克服现有技术存在的以上问题,提供一种苯丙酮尿症相关基因突变检测试剂盒,本试剂盒采用多重PCR富集二代测序全面系统的检测PAH、GCH1、PTS、QDPR及PCBD1共5个基因的全部外显子及调控区的点突变。本试剂盒旨在应用于生物学研究和分子诊断由上述基因缺陷导致的苯丙酮尿症。针对苯丙酮尿症相关基因的全外显子突变检测体系的设计方案如下:1.针对PAH、GCH1、PTS、QDPR、PCBD1共5个基因转录起始点上游500bp,5’UTR,编码区,剪切点8bp内含子序列,转录终止子设计引物;2.通过PCR将扩增出的特异目标序列,加上index序列和Illumina测序接头;3.模板分子与芯片测序通道上的引物结合,通过桥式PCR生成簇,以供测序反应;4.将测得序列与参考序列比对,从而找出突变位点,并对突变位点进行功能注释;5.对所有明确与疾病发生相关的致病突变进行Sanger测序验证。为实现上述技术目的,达到上述技术效果,本发明通过以下技术方案实现:一种苯丙酮尿症相关基因突变检测试剂盒,其特征在于,包括:ddH2O,10×buffer(Takara),2×GCbufferI,dNTP,MgCl2,HSTaq,多重PCR引物混合液,IlluminaI5/I7引物。进一步的,所述多重PCR引物混合液的引物序列如SQEIDNO.:1至SQEIDNO.:118所示。本发明的有益效果是:1、检测范围广:全面检测5个基因缺陷导致的苯丙酮尿症,对基因转录起始点上游500bp,5’UTR,编码区,剪切点8bp内含子序列,转录终止子进行测序检测,预计突变检出率可达95%以上,能够更好地应用于临床。2、通量高:多重PCR富集二代测序技术可实现同时筛查数百个患者标本的苯丙酮尿症相关5个基因的点突变检测。3、检测方法快速、准确:多重PCR富集二代测序技术,无需复杂的建库过程,而且利用IlluminaMiseq测序仪的2×250bp测序模式对目的片段实现双向测序验证,大大提高二代测序的数据准确度。4、检测准确性高:二代测序所有目标区段平均有效测序深度>300x,所有片段测序深度>=20x,且对所有致病性突变进行Sanger测序验证,保证数据准确性可达99%以上。5、成本低廉:相较于传统的Sanger测序,针对目标区域采用多重PCR富集二代测序方法的成本将大幅降低。附图说明图1为SNV在基因区域上的分布统计;图2为SNV功能分类;图3为Sanger测序验证图;图4为实验检测22个样本平均测序深度(横坐标为样本,纵坐标为平均测序深度);图5为碱基覆盖深度比例(横坐标为样本,纵坐标为覆盖比例)。具体实施方式下面将参考附图并结合实施例,来详细说明本发明。利用本发明所述基因点突变检测方法对22个苯丙酮尿症患者样本进行检测。一、检测样本将待测样本浓度标化至20ng/μl,样本体积要求20μl。二、检测方法目标区域富集测序方法检测点突变主要包括以下步骤:本实验所需的试剂包括以下:ddH2O、10×buffer(Takara)、2×GCbufferI、dNTP、MgCl2、HSTaq、多重PCR引物混合液、IlluminaI5/I7引物。A.反应体系配置a)10×多重PCR反应体系配制:10μl体系:PCR反应条件设置如下:95℃2min;11个循环扩增(94℃20s,63℃-0.5℃每循环40s和72℃90s);24个循环扩增(95℃20s,65℃30s和72℃60s),在72℃延伸2min,4℃保存。b)GC多重PCR反应体系配制:PCR反应条件设置如下:95℃2min;35个循环扩增(95℃20s,68℃60s和72℃2min),4℃保存。B.将PCR产物按照比例混合并将得到的混合产物按体积比样本:磁珠=1:1使用PCRcleanbeads进行纯化。C.I5/I7IndexPrimer对纯化产物进行加IndexPCR扩增10×反应体系配制:10μl体系:PCR反应条件设置如下:95℃2min;12个循环扩增(95℃20s,60℃30s和72℃60s),在72℃延伸2min,4℃保存。D.将PCR产物按照体积比样本:磁珠=1:1使用PCRcleanbeads进行纯化。E.按照2100bioanalyzer标准操作流程对PCR产物进行片段分析,确定产物扩增sizeF.样本文库进行上机测序前定量使用QubitSpectrophotometer对文库进行精确定量,换算浓度(nM)G.将定量好的文库混合上机测序,最终文库上样量11pM,按照MiSeqBenchtopSequencer标准操作流程进行测序,双向测序,两端各产生250bp序列,及两端各8bp的index标签序列。H.通过生物信息的方法对各突变进行功能注释。通过生物信息的数据分析可得到目标区域的所有SNVs。1)SNV基因区域分布统计这些SNVs位点相对于基因的位置分类如下表所示,包括外显子突变(Exonic)、剪切位点突变(Splicing)、non-codingRNA、基因非翻译区域5’端突变(5’UTR)、基因非翻译区域3’端突变(3’UTR)、内含子突变(Intronic)、基因上游突变(Upstream)、基因下游突变(Downstream)、基因间区突变(Intergenic)等,参照图1所示。表1SNV基因区域分布统计GeneRegionSNVcountPercentageIntronic912.68%Exonic230.99%5’UTR1216.90%3’UTR79.86%Splicing57.04%Upstream1622.54%Total71100.00%2)SNV功能类型分类相对于其他的突变类型,突变发生在外显子区域有可能直接导致最终产物---蛋白质发生序列以及结构变化,而这种变化最有可能是导致性状改变的分子机理。根据不同的变异类型,可以分为FrameshiftDeletion、FrameshiftInsertion、NonFrameshiftDeletion、NonFrameshiftInsertion、NonSynonymousSNV、StopGainSNV、SynonymousSNV。表2SNV功能类型分类统计表FunctionSNVcountPercentageIntron4969.01%nonsynonymousSNV1115.49%SynonymousSNV79.86%stopgainSNV45.63%TOTAL71100.00%3)SNV功能注释SNV的详细注释可以使研究变得更加容易,通过这些注释,包括保守性分析,1000genome、ESP6500、HGMD数据库注释,GO和KEGGPathway分析,通常可以迅速的将相关SNV的范围缩小到几十、甚至几个,在保证研究准确性的前提下,极大的提高了研究效率(部分展示见表3)。表3SNV注释统计三、数据分析表根据上述突变SNV功能注释,22个苯丙酮尿症患者突变检测如下表-4示,95%的患者找到疾病相关突变,且经Sanger测序验证均为真实存在(图2为部分测序图展示)。所有样本所有目标区段平均有效测序深度>300x,所有片段测序深度>=20x,均达到测序质量要求(图3-图4)。表4以上所述仅为本发明的优选实施例而已,并不用于限制本发明,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。<110>王瑞天昊生物医药科技(苏州)有限公司<120>苯丙酮尿症相关基因突变检测试剂盒<160>118<170>PatentInversion3.5<210>1<211>24<212>DNA<213>ArtificialSequence<223>人工序列<400>1gcacctaatccccataacttccag24<210>2<211>29<212>DNA<213>ArtificialSequence<223>人工序列<400>2gctaatgtatttctaaggcagtgagagga29<210>3<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>3ctctttctctggaggcccaaat22<210>4<211>19<212>DNA<213>ArtificialSequence<223>人工序列<400>4cacgaggggcgttactgtg19<210>5<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>5tcgcactagggaggagaagaga22<210>6<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>6agagtcccggaagtgcctaaac22<210>7<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>7caagccccgtcgattagaca20<210>8<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>8ttgctctttacctgctgtgctc22<210>9<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>9ctacagctagagggcacgtgaa22<210>10<211>23<212>DNA<213>ArtificialSequence<223>人工序列<400>10gcacacggtacaggtctcagtaa23<210>11<211>26<212>DNA<213>ArtificialSequence<223>人工序列<400>11aaaagcaaatccacataaatttgaca26<210>12<211>24<212>DNA<213>ArtificialSequence<223>人工序列<400>12tggcatcttcctgactatttgaag24<210>13<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>13gcctgttccagatcctgtgttc22<210>14<211>23<212>DNA<213>ArtificialSequence<223>人工序列<400>14tgtccatggaggtttaacaggaa23<210>15<211>27<212>DNA<213>ArtificialSequence<223>人工序列<400>15ttgctgttattttatgaagacagtgtg27<210>16<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>16ctcctcaccctccccattct20<210>17<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>17ggtaagaggaagggaggggagt22<210>18<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>18ctgggatccccacttctgatct22<210>19<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>19ttctctcttcccctcaacaagc22<210>20<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>20cccccattcaaagcattcataa22<210>21<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>21ctcaatcctcccccaactttct22<210>22<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>22ctgccctgcttgagacacctat22<210>23<211>21<212>DNA<213>ArtificialSequence<223>人工序列<400>23gaaaagatggcgctcattgtg21<210>24<211>25<212>DNA<213>ArtificialSequence<223>人工序列<400>24tggtgatgagctttgagttttcttt25<210>25<211>21<212>DNA<213>ArtificialSequence<223>人工序列<400>25ctccctgggctcaactcattt21<210>26<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>26tctttctgcccattcctcatgt22<210>27<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>27tttcaaagacctgagggccata22<210>28<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>28aggttggttctgtggttccaat22<210>29<211>27<212>DNA<213>ArtificialSequence<223>人工序列<400>29cttttaaatctatccttggttcctgtg27<210>30<211>29<212>DNA<213>ArtificialSequence<223>人工序列<400>30ggtgacacataaaattaaccatcatagag29<210>31<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>31ccagagctagtggctcaccttt22<210>32<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>32gatgagagaaggggcacaaatg22<210>33<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>33cgattactgagaaaccgagtgg22<210>34<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>34ctttctccaaatggtgcccttc22<210>35<211>23<212>DNA<213>ArtificialSequence<223>人工序列<400>35tgcttttcggactttttctgatg23<210>36<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>36gcccacttatcccctagtgctt22<210>37<211>29<212>DNA<213>ArtificialSequence<223>人工序列<400>37tccacaggctgttgtcttattagtaaatg29<210>38<211>32<212>DNA<213>ArtificialSequence<223>人工序列<400>38aatatttatagtggtggttgaaaaagaaacaa32<210>39<211>19<212>DNA<213>ArtificialSequence<223>人工序列<400>39ccgcccgcacgctctagca19<210>40<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>40ccagcaggccggcggagaag20<210>41<211>19<212>DNA<213>ArtificialSequence<223>人工序列<400>41gggccgctcgcccttccag19<210>42<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>42ccggccacagcccattgtcc20<210>43<211>23<212>DNA<213>ArtificialSequence<223>人工序列<400>43cgcttcgaggtctgcggctaaac23<210>44<211>19<212>DNA<213>ArtificialSequence<223>人工序列<400>44aacccctccgggcgtctcc19<210>45<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>45tttatggcccggcggctcag20<210>46<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>46gcgcccagaccgcgtctttc20<210>47<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>47cggtcccctcgccctgtgaa20<210>48<211>24<212>DNA<213>ArtificialSequence<223>人工序列<400>48tgatgtgacacttgccccgacagc24<210>49<211>24<212>DNA<213>ArtificialSequence<223>人工序列<400>49ttttaaattgctgggaaacaacaa24<210>50<211>26<212>DNA<213>ArtificialSequence<223>人工序列<400>50tggacatgttactaaagcaagcctct26<210>51<211>21<212>DNA<213>ArtificialSequence<223>人工序列<400>51agcagatgagggcaggtccta21<210>52<211>25<212>DNA<213>ArtificialSequence<223>人工序列<400>52tcacaaagaaggcactgtattttca25<210>53<211>29<212>DNA<213>ArtificialSequence<223>人工序列<400>53gagcaagactctgtctcaaaaataaaaat29<210>54<211>28<212>DNA<213>ArtificialSequence<223>人工序列<400>54tgaggaaggcttatcaattttacttcaa28<210>55<211>27<212>DNA<213>ArtificialSequence<223>人工序列<400>55tgagaagcactaaatagcaagatcact27<210>56<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>56gcttccagctgtttgtgtcaga22<210>57<211>28<212>DNA<213>ArtificialSequence<223>人工序列<400>57aattgaaaatggaatgtacaaacaagac28<210>58<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>58caaaccagcagctgtctactcc22<210>59<211>24<212>DNA<213>ArtificialSequence<223>人工序列<400>59tggtgtgggattatttgaaagcaa24<210>60<211>24<212>DNA<213>ArtificialSequence<223>人工序列<400>60ccacaacagatgcctcacatgatt24<210>61<211>18<212>DNA<213>ArtificialSequence<223>人工序列<400>61tgggtgcagcaaaccaac18<210>62<211>21<212>DNA<213>ArtificialSequence<223>人工序列<400>62ctggtgactcaatcggatgaa21<210>63<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>63tcaattcctgctccttttggtc22<210>64<211>23<212>DNA<213>ArtificialSequence<223>人工序列<400>64aagggtgtctctcgctatctgaa23<210>65<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>65acagggtccccacccaccctga22<210>66<211>21<212>DNA<213>ArtificialSequence<223>人工序列<400>66acggccaccaccttccgtgct21<210>67<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>67ggccgagcaccgcagacagc20<210>68<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>68cgtccgaagccccacgaagc20<210>69<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>69aaggaacagagaagggggtttg22<210>70<211>23<212>DNA<213>ArtificialSequence<223>人工序列<400>70tgtgtccgtaagttttcccattc23<210>71<211>23<212>DNA<213>ArtificialSequence<223>人工序列<400>71tgtgcttggatgttgatctgttg23<210>72<211>25<212>DNA<213>ArtificialSequence<223>人工序列<400>72gcaatactgactggaacagttttca25<210>73<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>73ggatgaaggcaaatgtgcaa20<210>74<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>74gggggccaccaaatattaca20<210>75<211>24<212>DNA<213>ArtificialSequence<223>人工序列<400>75ggagtcaatgatattttcccttgg24<210>76<211>27<212>DNA<213>ArtificialSequence<223>人工序列<400>76aacttcagaaatcacacaacaatcaaa27<210>77<211>27<212>DNA<213>ArtificialSequence<223>人工序列<400>77catgaaattttattgtttgcattttga27<210>78<211>23<212>DNA<213>ArtificialSequence<223>人工序列<400>78cgtacaacaatcacgtgttgacc23<210>79<211>25<212>DNA<213>ArtificialSequence<223>人工序列<400>79tctgtcctggtatccttttcctgtg25<210>80<211>26<212>DNA<213>ArtificialSequence<223>人工序列<400>80cagattaaaagacctccagcaatcca26<210>81<211>19<212>DNA<213>ArtificialSequence<223>人工序列<400>81ccccgcggagacccagcag19<210>82<211>19<212>DNA<213>ArtificialSequence<223>人工序列<400>82agcgcgctaggcggcagca19<210>83<211>19<212>DNA<213>ArtificialSequence<223>人工序列<400>83cgccgccatcctgctcctg19<210>84<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>84gccccgaggcccagcacttg20<210>85<211>21<212>DNA<213>ArtificialSequence<223>人工序列<400>85cacccggagggactgtaactt21<210>86<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>86tgccaaaacagtggtcgaaaat22<210>87<211>26<212>DNA<213>ArtificialSequence<223>人工序列<400>87cgagcattaatttgtgaagtgtaagc26<210>88<211>21<212>DNA<213>ArtificialSequence<223>人工序列<400>88caggatctgtgtctggcgatg21<210>89<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>89ccgagacgagacaaaccacact22<210>90<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>90agggcagcacctgtcacaaata22<210>91<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>91tacagccagtggtcacctctcc22<210>92<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>92ggccaactcccctcatttctat22<210>93<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>93cctagtgcaaacccaatccttg22<210>94<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>94tgctagcctgtgttggcttacc22<210>95<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>95agtcctcatcccatgaaagtgc22<210>96<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>96gctcaccttttggtttctttcc22<210>97<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>97cagtcagacaaacggtcacctg22<210>98<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>98agattgcatgtccttgctgtcc22<210>99<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>99accacagcaggcagagaatagg22<210>100<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>100gggcacctgttgttagaaagga22<210>101<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>101acagaaaacacaaggctggaca22<210>102<211>21<212>DNA<213>ArtificialSequence<223>人工序列<400>102cgtcactgtgaaggtcagcat21<210>103<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>103ttgctgtgggatgtgccagt20<210>104<211>21<212>DNA<213>ArtificialSequence<223>人工序列<400>104tgtggggtttagggtcctggt21<210>105<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>105aagactttcccccgccccttcc22<210>106<211>24<212>DNA<213>ArtificialSequence<223>人工序列<400>106gctccctgaaacaagcacggagga24<210>107<211>21<212>DNA<213>ArtificialSequence<223>人工序列<400>107gcttgcttccagcactttcct21<210>108<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>108tgttgatctcggtgatggtctc22<210>109<211>26<212>DNA<213>ArtificialSequence<223>人工序列<400>109gcaataaattgcacatattcaacgtg26<210>110<211>23<212>DNA<213>ArtificialSequence<223>人工序列<400>110ccccaaaacagttatcctgccta23<210>111<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>111gccaggcaagcactgatgtact22<210>112<211>20<212>DNA<213>ArtificialSequence<223>人工序列<400>112tgtgcaccatattcgcagca20<210>113<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>113tttgtaaggtgaccccatcagc22<210>114<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>114ctaaaggttggagcccctctgt22<210>115<211>23<212>DNA<213>ArtificialSequence<223>人工序列<400>115cagagttcgcagtatttggatgg23<210>116<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>116cgtctaggccttgaattggtga22<210>117<211>21<212>DNA<213>ArtificialSequence<223>人工序列<400>117agggtaagggctcctcagctc21<210>118<211>22<212>DNA<213>ArtificialSequence<223>人工序列<400>118gcagctagtgactccctcctgt22当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1