腈水解酶XiNit1及其编码基因和应用的制作方法
本发明属于生物
技术领域:
,具体涉及腈水解酶xinit1及其编码基因和应用。
背景技术:
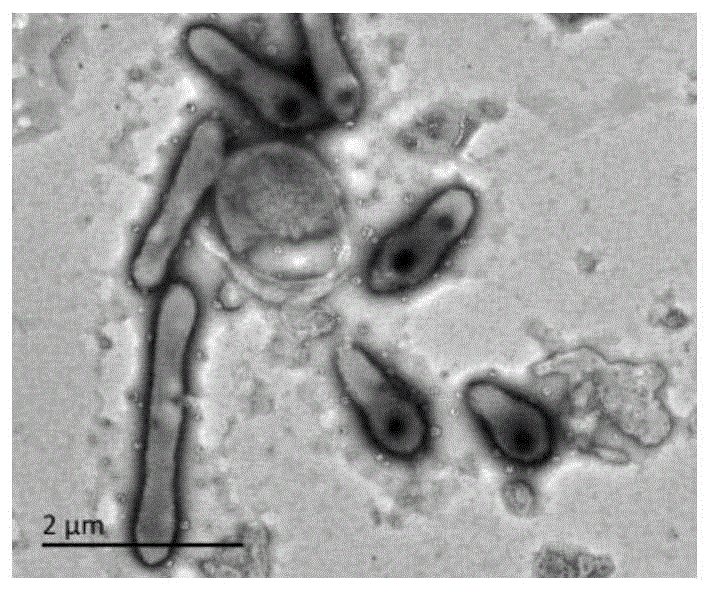
:腈类化合物是一种重要的有机化工原料,同时也是一种有刺激性气味的有毒物质。在合成橡胶、石油炼制过程的污水中含有众多不同种类的腈类化合物,丙烯腈就是其中的一种重要有毒成分。相比于物理法、化学法在处理腈类化合物过程中的副产物,人们更倾向于采用对环境无害的生物法处理污水中的腈类化合物。生物法相较于物理法和化学法而言,具有反应条件温和、运行稳定且成本低廉的特点。自19世纪以来,利用生物法处理废水中的有机物已成为世界各国的主要方法。利用微生物的代谢功能将废水中的腈类和酚类等有机物降解为水和二氧化碳等,从而达到废水的净化效果。世界上第一株腈水解酶是在19世纪60年代在大麦叶中被发现的,后来证明其在真菌、细菌甚至古菌中也广泛存在。目前人类已从众多不同属种的微生物中发现腈水解酶的存在,例如红细菌、诺卡氏菌、节杆菌等。腈水解酶(nitrilase,ec3.5.5.1)是属于腈水解酶超家族的一类重要的工业用酶,不仅可以降解水体或土壤中的腈类污染物,还可以将廉价的腈类化合物水解转化为高附加值的工业、食品和医药等产品的中间体,如:丙烯酸、烟酸和手性扁桃酸等。其催化过程温和环保,属于绿色化工。因此,开发具有高活性、高稳定性和极端条件耐受性的腈水解酶,具有重要的应用价值。根据目前的报道,从一种古菌:火球菌pyrococcussp.中克隆表达的腈水解酶具有较高的温度稳定性,其最适温度为60-90℃,但ph范围较窄(6-8),且对底物的降解效率非常低。根据底物范围,可以将腈水解酶分为三大类:脂肪腈水解酶、芳香腈水解酶和芳基乙腈类腈水解酶。一般来说,现有报道的腈水解酶只能专一的降解一类腈化物。因此,寻找更为优质的腈水解酶,成为了研究的热点。技术实现要素:本发明的目的是提供腈水解酶xinit1及其编码基因和应用。本发明提供的蛋白质,来源于辛芳芳菌(xinfangfangiasp.),是一种腈水解酶,命名为xinit1蛋白,是如下(a1)或(a2)或(a3)或(a4)或(a5)或(a6)或(a7):(a1)由序列表中序列1所示的氨基酸序列组成的蛋白质;(a2)由序列表中序列3所示的氨基酸序列组成的蛋白质;(a3)含有(a1)的融合蛋白;(a4)在(a1)的末端连接含有标签的短肽得到的融合蛋白;(a5)在(a1)的末端连接标签得到的融合蛋白;(a6)将(a1)或(a2)或(a3)或(a4)或(a5)经过一个或几个氨基酸残基的取代和/或缺失和/或添加具有腈水解酶功能的由其衍生的蛋白质。(a7)来源于辛芳芳菌,且与(a1)具有99%以上同源性,且具有腈水解酶功能的蛋白质。标签具体如表1所示。表1标签的序列标签残基序列poly-arg5-6(通常为5个)rrrrrpoly-his2-10(通常为6个)hhhhhhflag8dykddddkstrep-tagii8wshpqfekc-myc10eqkliseedlha9ypydvpdyaxinit1蛋白的编码基因也属于本发明的保护范围。所述基因为如下1)至4)中任一所述的dna分子:1)编码区如序列表中序列2所示的dna分子;2)编码区如序列表中序列4中第1-891位核苷酸所示的dna分子;3)在严格条件下与1)或2)限定的dna序列杂交且编码具有腈水解酶功能蛋白的dna分子;4)来源于辛芳芳菌,且与1)限定的dna序列具有90%以上同源性且编码具有腈水解酶功能蛋白的dna分子。上述严格条件可为用6×ssc,0.5%sds的溶液,在65℃下杂交,然后用2×ssc,0.1%sds和1×ssc,0.1%sds各洗膜一次。含有所述基因的重组表达载体、表达盒或重组微生物均属于本发明的保护范围。本发明还保护xinit1蛋白作为腈水解酶的应用。应用xinit1蛋白作为腈水解酶时,采用的温度为30-80℃,采用的ph为3-11。应用xinit1蛋白作为腈水解酶时,采用的温度为55℃,采用的ph为7。本发明还保护xinit1蛋白在降解腈类化合物中的应用。应用xinit1蛋白降解腈类化合物时,采用的温度为30-80℃,采用的ph为3-11。应用xinit1蛋白降解腈类化合物时,采用的温度为55℃,采用的ph为7。本发明还保护xinit1蛋白在制备腈水解酶中的应用。本发明还保护一种用于降解腈类化合物的制剂,其活性成分为xinit1蛋白。以上任一所述腈类化合物为脂肪族腈类、芳香族腈类或酰基腈类。本发明提供的腈水解酶xinit1具有较高的酶活力,宽泛的底物,并且反应温度宽泛,反应ph宽泛,对于金属离子、金属离子螯合剂、还原剂、有机溶剂和表面活性剂均具有高耐受性。辛芳芳菌(xinfangfangiasp.)dly26已于2019年1月2日保藏于中国典型培养物保藏中心(地址:中国,武汉,武汉大学;邮编:430072),保藏编号为cctccno:m2019001。本发明对于相关化工领域、环境污染治理领域等,具有重大的应用前景。附图说明图1为菌株dly26的照片。图2为系统发生树。图3为细菌生长曲线。图4为气相色谱法检测的色谱图。图5为振荡培养的各个时间点丙烯腈的降解率。图6为superdextm20010/60凝胶层析柱进行分离纯化的色谱图。图7为xinit1蛋白溶液的电泳图。图8为检测最适ph时的相对酶活结果。图9为检测ph稳定性时的相对酶活结果。图10为检测最适反应温度时的相对酶活结果。图11为检测温度稳定性时的相对酶活结果。具体实施方式以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。以下实施例中的定量试验,均设置三次重复实验,结果取平均值。实施例1、辛芳芳菌dly26的分离、鉴定和保藏一、分离取约1ml活性污泥样品(取自石化炼油化污水处理系统),用无菌蒸馏水依次稀释至10倍体积、20倍体积、50倍体积、100倍体积和1000倍体积。采用涂布平板法将100μl稀释后样本涂布于固体培养基表面,30℃培养,每天观察表面菌落的生长情况。挑取颜色不同、形态差异的菌落,通过三区划线法进行纯化和培养,直至在培养基表面生长出能够观察到形态、大小等特征都相同的单菌落为止。二、鉴定将纯化获得的菌株接种于tsa平板,30℃培养24h,然后使用透射电子显微镜观察细胞的形态、大小等特征(菌株dly26的照片见图1)。从培养24h的tsa平板表面挑取单菌落,按照标准方法进行革兰氏染色处理,使用倒置荧光显微镜观察细菌的革兰氏染色情况。制备半固体培养基tsb,采用穿刺接种法观察细菌的运动性和好氧性情况。以液体培养基tsb为基础:配制不同nacl浓度的培养基体系(nacl梯度为0、1.0、2.0、3.0、4.0、5.0、6.0、7.0、8.0及9.0g/100ml),接种后置于30℃的摇床中振荡培养3-5d,使用uv-2450分光光度计在od600处检测细菌的其生长情况,以确定最佳盐度生长范围;配制ph值不同的液体培养基(ph梯度设置为4.0、5.0、6.0、7.0、8.0、9.0、10.0、11.0),接种后置于30℃的摇床中振荡培养3-5d,以培养条件相同的不接菌培养基作为空白对照,以确定最佳生长ph范围;设置温度梯度为4℃、20℃、28℃、30℃、35℃、37℃、40℃、45℃、50℃,以确定最佳生长温度范围。以液体碳源培养基为基础,添加浓度的不同碳水化合物作为唯一的碳源,检测细菌的同化作用。参照常见细菌系统鉴定手册中的方法,检测细菌的硝酸盐还原、亚硝酸盐还原能力,反硝化能力以及脂酶、接触酶的有无。使用海博革兰氏阴性细菌鉴定系统(青岛,中国)进行细菌的voges-proskauer测试(vp测试),检测细菌的硫化氢(h2s)产生、脲酶(ure)、明胶(gel)水解情况。也可以用于评价其他生理生化特征的有无,例如通过对精氨酸(adh)、山梨醇(sor)、蔗糖(sac)等的降解情况,以检验精氨酸酶、山梨醇脱氢酶、蔗糖酶的存在情况。以x.solizqbwt、r.megalophilusja194t和fal.halotoleransja744t作为参考菌株。菌株dly26和参考菌株的结果见表2和表3和表4。表2生化特征dly26zqbwtja194tja744t温度范围4-4526-354-454-40最适温度30303030ph范围5-116-123-113-11最适ph7.07.07.07.0盐度范围(%)0-5.00-5.00-5.00-5.0最适盐度(%)1.0000细胞大小(μm)1.2-2.0*,0.4-0.61.5-2.6*,0.7-0.91.2-1.0*,1.5-2.02.0-5.0*,1.0-1.2鞭毛---+细胞外形rodrodovalrodovalrod运动性---+gc含量(%)63.967.066.6774-76表3表4onpgadhldhodccith2sureindvpgelrhamelamyoxdly26+++++w+------+dly30++--+w+--+---+ja194t-++++w+------+ja744t+++++w+------+注:onpg:β-半乳糖苷酶;adh:精氨酸;ldh:赖氨酸;odc:鸟氨酸;cit:柠檬酸盐;h2s:产硫化氢试验;ure:脲酶;ind:吲哚;gel:明胶;rha:鼠李糖;mel:蜜二糖;amy:苦杏仁甙;ox:氧化酶。利用细菌基因组dna提取试剂盒,提取菌株dly26的基因组dna,并采用通用引物27f和1492r进行16srdna扩增。扩增产物采用琼脂糖凝胶电泳法分析产物条带,获得目标条带并使用天根dna纯化试剂盒dp209进行纯化。将纯化产物连接到载体pmd19-t中,利用pmd19-t克隆试剂盒,将目的载体转化至大肠杆菌dh5α。将重组菌送往青岛擎科生物技术有限公司进行测序,获得16srrna序列。利用genbank数据库检索每个相关基因的16srrna的序列,使用mega版本7.0构建菌株的系统发育树。使用近邻连接法(nj)进行细菌的系统发育分析。基于1000次重复,使用bootstrap分析确定系统发生树的拓扑结构。见图2。以上鉴定结果表明,菌株dly26属于红细菌科(rhodobacteraceae),辛芳芳菌属(xinfangfangia)。三、保藏辛芳芳菌(xinfangfangiasp.)dly26已于2019年1月2日保藏于中国典型培养物保藏中心(地址:中国,武汉,武汉大学;邮编:430072),保藏编号为cctccno:m2019001。实施例2、辛芳芳菌dly26对丙烯腈的降解作用na2hpo4/nah2po4缓冲液的配方:磷酸氢二钠14.2g、磷酸二氢钠12.2g,溶于800mlddh2o,用ddh2o定容至1l,调ph至7.0。活化培养基的配方:胰蛋白胨大豆肉汤培养基(tsb)30g,用ddh2o溶解并定容至1l,调ph至7.0。基础培养基:磷酸氢二钠2.0g、磷酸二氢钾1.0g、酵母粉0.5g、硫酸镁0.2g、硫酸亚铁0.03g,溶于800mlna2hpo4/nah2po4缓冲液,然后加入甘油3.25ml并混匀,然后用na2hpo4/nah2po4缓冲液定容至1l。一、制备种子液将辛芳芳菌dly26接种至活化培养基,30℃、150rpm振荡培养,获得种子液。种子液的od600nm值=0.6-0.8。二、制作细胞干重标准曲线1、取步骤一制备的种子液,按10%接种量接种至活化培养基,然后30℃、150rpm振荡培养48h。2、完成步骤1后,取样40ml,用活化培养基调整od600nm值,得到od600nm值不同的各个菌液。3、取步骤2得到的菌液,8000rpm离心15min,收集菌体沉淀。4、取步骤3得到的菌体沉淀,用生理盐水充分洗涤,然后60℃烘干,测量干重。5、制作od600nm值与生物量(以细胞干重计)的标准曲线。标准曲线方程:y=2.895x-0.036;r2=0.9988;x为od600nm值,y为细胞干重(单位:g/l)。三、制作细菌生长曲线以及检测丙烯腈的降解率1、取步骤一制备的种子液,按10%接种量接种至含10mm丙烯腈(作为唯一氮源)的基础培养基,然后30℃、150rpm振荡培养,每隔12h检测od600nm值。结果表明,振荡培养48h后od600nm值达到最大值。2、取步骤1中振荡培养48h的培养体系,8000rpm离心15min,收集菌体沉淀,洗涤2-3次(每次洗涤的方法均为:用na2hpo4/nah2po4缓冲液悬浮菌体,然后8000rpm离心15min,收集菌体),用na2hpo4/nah2po4缓冲液悬浮菌体,得到od600nm值=0.43的菌悬液。3、取100ml含20mm丙烯腈的基础培养基,加入5ml步骤2制备的菌悬液,然后30℃、150rpm振荡培养104h。0-8h每隔2h取样一次,9-32h每隔12h取样一次,33-104h每隔24h取样一次;每次取样5ml;每次取样后在培养体系中加入丙烯腈并使其在体系中的浓度为20mm。4、制作细菌生长曲线从步骤3的取样样本中取3ml,测od600nm值,根据步骤二的标准曲线,计算振荡培养的各个时间点每升培养体系中的生物量(生物量以细胞干重计)。结果见图3。在培养期间,细菌的生物量先升高后降低。培养4h时达到了最大值。培养4h以后,随着培养时间的延长,菌体生物量呈现降低的趋势(原因应该在于:随着丙烯腈产物丙烯酸的逐渐积累,对细菌的危害毒力也越来越大,细菌的死亡率逐渐大于生长率)。5、检测丙烯腈的降解率从步骤3的取样样本中取1ml,8000rpm离心2min,取上清液,向上清液中加入0.1ml2m盐酸水溶液终止反应,然后取200μl,加入800μl乙酸乙酯并混匀,然后静置30min,收集乙酸乙酯相。采用气相色谱法检测乙酸乙酯相中的丙烯酸含量。气相色谱法相关参数:色谱柱为极性hp-ffap毛细管柱(30m×320μm×0.25μm),进样口温度为260℃,柱箱温度为190℃,fid检测器温度为260℃,氢气流速为40ml/min,空气流速为450ml/min,氮气尾吹气流速为40ml/min,进样量为0.2μl,分流比为50:1。采用乙酸乙酯作为溶剂,配制各个浓度的丙烯酸标准品溶液。采用气相色谱法(参数同上)检测丙烯酸标准品溶液。制作峰面积与丙烯酸浓度的标准曲线。y=2.9507x+6.9485,相关系数r2=99.17%;y代表峰面积,x代表丙烯酸浓度(单位为mmol/l)。将乙酸乙酯标准品、丙烯腈标准品和丙烯酸标准品按照以上参数进行气相色谱法检测,色谱图见图4。乙酸乙酯的出峰时间为1.387min、丙烯腈的出峰时间为2.187min、丙烯酸的出峰时间为4.522min。根据标准曲线和峰面积计算丙烯酸的产量,进一步计算振荡培养的各个时间点丙烯腈的降解率,结果见图5。培养时间为0-32小时,丙烯腈的降解率逐步提高。培养时间为32-104小时,丙烯腈的降解率基本不变,维持在79.4%左右。实施例3、腈水解酶(xinit1蛋白)的制备经过大量序列分析、比对和功能验证,从辛芳芳菌dly26中发现一个新蛋白,将其命名为xinit1蛋白,如序列表的序列1所示。将辛芳芳菌dly26中编码xinit1蛋白的基因命名为xinit1基因,其编码框如序列表的序列2所示。一、构建重组质粒1、以辛芳芳菌dly26的基因组dna为模板,采用dn1-f和dn1-r组成的引物对进行pcr扩增,回收pcr扩增产物。dn1-f:5’-caccatgctggcggaagg-3’;dn1-r:5’-cggccagctcgtcatgac-3’。2、取步骤1得到的pcr扩增产物,与pde1载体连接,得到重组质粒pde1-xinit1。pde1载体(pde1vector)为pde1directionalexpressionkit的组件。pde1directionalexpressionkit:北京擎科新业生物技术有限公司,产品目录号tsv-e1。经测序,重组质粒pde1-xinit1中具有序列表的序列4所示的dna分子(其中第1-891位核苷酸为开放阅读框),表达序列表的序列3所示的融合蛋白。序列表的序列3中,第2-7位氨基酸残基组成his6标签,第21-296位氨基酸残基组成xinit1蛋白。二、制备重组菌将重组质粒pde1-xinit1导入大肠杆菌bl21(de3),得到重组菌甲。将pde1载体导入大肠杆菌bl21(de3),得到重组菌乙。三、表达蛋白1、将重组菌接种至含50μg/ml卡那霉素的液体lb培养基,37℃、150rpm振荡培养12小时,得到种子液。2、将1体积份种子液接种至99体积份含50μg/ml卡那霉素的液体lb培养基中,37℃、200rpm振荡培养至od600nm值约为0.6,此时加入iptg诱导剂并使其在体系中的浓度为0.5mmol/l,然后25℃、200rpm振荡培养6h(诱导表达),然后4℃、8000×g离心10min,收集菌体沉淀。3、取步骤2得到的沉淀,用磷酸钠缓冲液(0.1m,ph7.2)洗涤,然后悬浮于磷酸钠缓冲液(0.1m,ph7.2)并进行超声破碎(超声破碎参数:功率200w,每超声4s停6s,总时间为40min),然后4℃、10000×g离心20min,收集上清液。重组菌甲进行上述步骤得到的上清液,命名为粗酶液甲。重组菌乙进行上述步骤得到的上清液,命名为粗酶液乙。四、纯化蛋白取步骤三得到的粗酶液甲,用孔径为0.22μm的微滤滤膜过滤,收集滤液。取滤液,采用superdextm20010/60凝胶层析柱进行分离纯化。将superdextm20010/60凝胶层析柱连接到快速蛋白液相系统,以1×pbs缓冲液(ph8.0)作为流动相,流速为0.1ml/min。收集保留体积为18-20ml对应洗脱峰的过柱后溶液,即为xinit1蛋白溶液。色谱图见图6。xinit1蛋白溶液的电泳图见图7,仅一条蛋白带,且符合预估分子量(约29.6kd)。实施例4、腈水解酶(xinit1蛋白)的酶学性质无氨水:将去离子水煮沸后,保持沸腾状态15min。本实施例中的水,均指的是无氨水。苯酚钠溶液的配制:苯酚25g,溶于800ml水,然后加入78ml4.0m氢氧化钠水溶液,然后用水定容至1l。亚硝基铁氰化钠溶液的配制:1g亚硝基铁氰化钠溶解于100ml水,得到母液,使用前用水稀释至100倍体积,得到亚硝基铁氰化钠溶液。次氯酸钠溶液的配制:19.4ml市售10%次氯酸钠原液,用水定容至1l。氨标准液的配制:原料为氯化铵和水,制备不同氨(nh3)浓度的氨标准液。3-氰基吡啶溶液:3-氰基吡啶浓度为500mm,余量为水。反应原理:腈水解酶可以将1分子3-氰基吡啶降解为1分子烟酸和1分子氨,因此可以通过测定氨来测定酶活。显色原理:在碱性条件下,亚硝基铁氰化钠可催化氨与苯酚和次氯酸钠反应,生成蓝色可溶性物质,该物质在630nm处有最大吸收值。一、ph对腈水解酶活性的影响1、最适ph取实施例3制备的xinit1蛋白溶液,用缓冲液稀释至2倍体积,将稀释液作为供试液。检测方法:取离心管,加入供试液1ml、3-氰基吡啶溶液0.1ml、缓冲液3.9ml,30℃反应1h,然后加入0.5ml2mhcl水溶液以终止反应,然后取样1ml(称为取样液)进行后续的显色反应;取10ml离心管,依次加入取样液1ml、苯酚钠溶液2ml、亚硝基铁氰化钠溶液3ml、次氯酸钠溶液3ml,然后用水补足至10ml,37℃反应15min,然后测定od630nm值。分别采用如下缓冲液:ph3.0的柠檬酸盐缓冲液、ph4.0的柠檬酸盐缓冲液、ph5.0的柠檬酸盐缓冲液、ph6.0的柠檬酸盐缓冲液、ph6.0的磷酸盐缓冲液、ph7.0的磷酸盐缓冲液、ph8.0的磷酸盐缓冲液、ph8.0的碳酸盐缓冲液、ph9.0的碳酸盐缓冲液、ph10.0的碳酸盐缓冲液、ph11.0的碳酸盐缓冲液。柠檬酸盐缓冲液的配方见表5。磷酸盐缓冲液的配方见表6。碳酸盐缓冲液的配方见表7。表5ph0.1m柠檬酸水溶液(ml)0.1m柠檬酸钠水溶液(ml)3.037.22.84.026.213.85.016.423.66.07.632.4表6ph0.2m磷酸氢二钠水溶液(ml)0.2m磷酸二氢钠水溶液(ml)6.061.5438.57.03051958.0473.526.5表7ph0.1m碳酸钠水溶液(ml)0.1m碳酸氢钠水溶液(ml)8.0504509.015035010.030020011.045050xinit1蛋白的最适ph为7。将采用最适ph对应的缓冲液时的od630nm值作为100%,计算采用各种缓冲液时的相对值,作为相对酶活。结果见图8。2、ph稳定性取实施例3制备的xinit1蛋白溶液,用缓冲液稀释至2倍体积,然后4℃孵育12h。孵育0时刻作为供试液0,孵育12h后作为供试液12。检测方法:取离心管,加入供试液1ml、3-氰基吡啶溶液0.1ml、磷酸钠缓冲液(100mm、ph7.2)3.9ml,30℃反应1h,然后加入0.5ml2mhcl水溶液以终止反应,然后取样1ml(称为取样液)进行后续的显色反应;取10ml离心管,依次加入取样液1ml、苯酚钠溶液2ml、亚硝基铁氰化钠溶液3ml、次氯酸钠溶液3ml,然后用水补足至10ml,37℃反应15min,然后测定od630nm值。以供试液0的od630nm值作为100%,分别计算各个供试液12的相对值,作为相对酶活。结果见图9。xinit1蛋白在ph7时酶活力最稳定。二、温度对腈水解酶活性的影响1、最适反应温度取实施例3制备的xinit1蛋白溶液,用磷酸钠缓冲液(100mm、ph7.2)稀释至2倍体积,然后作为供试液。检测方法:取离心管,加入供试液1ml、3-氰基吡啶溶液0.1ml、磷酸钠缓冲液(100mm、ph7.2)3.9ml,反应1h(反应温度分别设置为30、40、50、55、60、70、80℃),然后加入0.5ml2mhcl水溶液以终止反应,然后取样1ml(称为取样液)进行后续的显色反应;取10ml离心管,依次加入取样液1ml、苯酚钠溶液2ml、亚硝基铁氰化钠溶液3ml、次氯酸钠溶液3ml,然后用水补足至10ml,37℃反应15min,然后测定od630nm值。最适反应温度为55℃。将采用最适反应温度时的od630nm值作为100%,计算采用各种反应温度时的相对值,作为相对酶活。结果见图10。2、温度稳定性取实施例3制备的xinit1蛋白溶液,用磷酸钠缓冲液(100mm、ph7.2)稀释至2倍体积,然后孵育(孵育温度分别设置为40、50、60、70、80℃;孵育时间分别设置为0、30、60、90、120min),然后作为供试液。检测方法:取离心管,加入供试液1ml、3-氰基吡啶溶液0.1ml、磷酸钠缓冲液(100mm、ph7.2)3.9ml,30℃反应1h,然后加入0.5ml2mhcl水溶液以终止反应,然后取样1ml(称为取样液)进行后续的显色反应;取10ml离心管,依次加入取样液1ml、苯酚钠溶液2ml、亚硝基铁氰化钠溶液3ml、次氯酸钠溶液3ml,然后用水补足至10ml,37℃反应15min,然后测定od630nm值。以孵育0h时刻供试液相应的od630nm值作为100%,分别计算其它孵育时间供试液的相对酶活。结果见图11。xinit1蛋白酶活性稳定温度为50℃。三、金属离子及化学试剂对腈水解酶活性的影响取实施例3制备的xinit1蛋白溶液,用磷酸钠缓冲液(100mm、ph7.0)稀释至2倍体积,然后作为供试液。检测方法:取离心管,加入供试液1ml、3-氰基吡啶溶液0.1ml、化合物水溶液10μl或有机溶剂0.25ml,用磷酸钠缓冲液(100mm、ph7.0)补足至5ml,55℃反应1h,然后加入0.5ml2mhcl水溶液以终止反应,然后取样1ml(称为取样液)进行后续的显色反应;取10ml离心管,依次加入取样液1ml、苯酚钠溶液2ml、亚硝基铁氰化钠溶液3ml、次氯酸钠溶液3ml,然后用水补足至10ml,37℃反应15min,然后测定od630nm值。以不加入化合物水溶液/有机溶剂时相应的od630nm值作为100%,分别计算加入各种化合物水溶液/有机溶剂后的相对酶活。部分化合物分别见表8。该部分化合物在5ml体系中的浓度分别为1mm或5mm。结果见表8。部分化合物分别见表9。该部分化合物在5ml体系中的浓度为1mm。结果见表9。有机溶剂见表10。结果见表10。表8表9化合物相对酶活sds(表面活性剂)97(±3)%edta(金属离子螯合剂)102(±1)%l-谷胱甘肽(还原剂)92(±1)%l-半胱氨酸(还原剂)96(±2)%表10有机溶剂相对酶活异丙醇127(±3.1)%曲拉通113(±2.5)%甲醇116(±3.4)%丙三醇132(±3.2)%正己烷137(±3.7)%乙醇153(±4.8)%二氯甲烷132(±1.0)%正庚烷129(±1.1)%乙醚113(±1.7)%乙酸乙酯109(±4.6)%dmso31(±5.3)%span80129(±1.9)%丙酮107(±1.3)%四、腈水解酶的底物特异性取实施例3制备的xinit1蛋白溶液,用磷酸钠缓冲液(100mm、ph7.0)稀释至2倍体积,然后作为供试液。检测方法:取离心管,加入供试液1ml、底物水溶液、用磷酸钠缓冲液(100mm、ph7.0)补足至5ml,55℃反应1h,然后加入0.5ml2mhcl水溶液以终止反应,然后取样1ml(称为取样液)进行后续的显色反应;取10ml离心管,依次加入取样液1ml、苯酚钠溶液2ml、亚硝基铁氰化钠溶液3ml、次氯酸钠溶液3ml,然后用水补足至10ml,37℃反应15min,然后测定od630nm值。以3-氰基吡啶作为底物时相应的od630nm值作为100%,分别计算对于各种底物的相对酶活。底物分别见表11。底物在5ml体系中的浓度为10mm。结果见表11。月桂腈、3-苯基丙腈、4-氯丁腈、2-甲基戊二腈、戊腈、丙烯腈、亚氨基二乙腈、丁二腈、己二腈和癸二腈属于脂肪族腈类。苯甲腈、3-氰基吡啶和3-氰基噻吩属于芳香族腈类。1,2-苯基乙二腈属于酰基腈类。表11月桂腈(dodecanenitile)348(士4.9)%3-苯基丙腈(3-phenylpropionitrile)381(土5.6)%4-氯丁腈(4-chlorobutyronitrile)392(土5.8)%2-甲基戊二腈(2-methylglutarontrile)380(土5.5)%戊腈(pentanenitile)371(土5.4)%丙烯腈(acrylonitrile)324(士4.4)%亚氨基二乙腈(iminodiacetonitrile)331(土4.6)%丁二腈(succinonitrile)315(土4.3)%己二腈(dicyanobutane)329(土4.5)%苯甲腈(benzonitile)296(土3.9)%3-氰基噻吩(thiophene-3-carbonitile)334(土4.6)%1,2-苯基乙二腈(1,2-phenylenediacetonitrile)329(土4.5)%癸二腈(decanedinitile)345(土4.4)%五、酶活力的测定酶活(1u)定义为:每1min形成1μmol·l-1氨所需的酶量。检测方法:取离心管,加入供试液1ml、3-氰基吡啶溶液0.1ml、磷酸钠缓冲液(100mm、ph7.0)3.9ml,55℃反应1h,然后加入0.5ml2mhcl水溶液以终止反应,然后取样1ml(称为取样液)进行后续的显色反应;取10ml离心管,依次加入取样液1ml、苯酚钠溶液2ml、亚硝基铁氰化钠溶液3ml、次氯酸钠溶液3ml,然后用水补足至10ml,37℃反应15min,然后测定od630nm值。标准曲线的绘制:取离心管,加入1ml氨标准溶液,苯酚钠溶液2ml、亚硝基铁氰化钠溶液3ml、次氯酸钠溶液3ml,然后用水补足至10ml,37℃反应15min,然后测定od630nm值。标准曲线回归方程为:y=0.1024x+0.0265,r2=0.9953;y为od630nm值,x为氨浓度(单位为μg/ml)。根据od630nm值和标准曲线回归方程检测供试液中的氨含量,从而计算酶活。采用实施例3制备的粗酶液甲作为供试液,酶活为20.5u/ml。采用实施例3制备的粗酶液乙作为供试液,酶活为0u/ml。采用实施例3制备的xinit1蛋白溶液作为供试液,检测单位体积供试液的酶活。检测实施例3制备的xinit1蛋白溶液中的蛋白浓度。将单位体积供试液的酶活除以单位体积供试液的蛋白含量,得到蛋白比活力,数值为187u/mg。sequencelisting<110>中国石油大学(华东)<120>腈水解酶xinit1及其编码基因和应用<130>gncyx191305<160>4<170>patentinversion3.5<210>1<211>276<212>prt<213>辛芳芳菌(xinfangfangiasp.)<400>1metargalaalaleuleuglnleuasnvalseraspaspproalaala151015asnleualaalathrleualaleuvalaspargalavalalaglygly202530alaalaphevalleuthrproglumetthrasnglyleuserserser354045argasphisglnargalavaltyrargprogluaspglnaspgluthr505560leualaalaleuargalalysalaalaglnalaglyiletrpleuleu65707580ileglyserleuglyleuthrthrthraspalaaspglyargpheala859095asnargserpheleuileglyproaspglyalailevalalaargtyr100105110asplysilehismetpheaspvalasnvalseraspthrgluvaltyr115120125arggluseralaglytyrargproglyhisglnalavalleualaglu130135140thrprophealalysileglymetservalcystyraspleuargphe145150155160prohisleutyrargargleualalysalaglyalaglnileleuthr165170175valproalaalapheasnhisilethrglyalaalahistrpgluval180185190leuleuargalaargalailegluthrglycysphevalleualapro195200205alaglnthrglyphehisalaglualahisglylysglyargargthr210215220hisglyhisserleualavalalaprotrpglygluvalleualaasp225230235240alaglythrgluproglyvalthrphevalaspleuaspleusergln245250255valgluargalaargglyargvalproserileasphisaspargala260265270ilegluglypro275<210>2<211>831<212>dna<213>辛芳芳菌(xinfangfangiasp.)<400>2atgcgcgcggcccttctgcagctgaatgtctcggacgatcccgccgccaatctggccgcg60acgctggcgctggtcgatcgggcggtggcggggggcgcggctttcgtgctgacgcccgag120atgaccaatggcctgtcttcctcgcgcgatcaccagcgcgcggtctaccggcccgaagat180caggacgaaaccctggccgccctgcgcgccaaggcggcgcaggccgggatctggctgctg240atcggatcgctgggcctgaccacgaccgatgccgacgggcgctttgccaaccggtccttc300ctgatcgggccggatggcgccatcgtggcgcgctatgacaagatccacatgttcgacgtg360aacgtgtcggacaccgaagtctaccgcgaaagcgcgggctaccggccgggccatcaggcg420gttctggccgagacgcctttcgccaagatcgggatgagcgtctgctacgacctgcgcttc480ccgcatctgtaccgccgtctggccaaggccggggcgcagatcctgaccgtgccggcagcc540ttcaaccacatcaccggggccgcgcattgggaggtgctgctgcgggcgcgtgcgatcgag600accggctgtttcgtgctggccccggcgcaaaccggctttcacgccgaagcgcatggcaag660ggacgccgcacccatggccattccctggccgtcgcgccctggggcgaggttctggccgat720gccggcaccgagccgggggtcaccttcgtcgatctggacctgtcgcaagtggagcgcgcg780cggggccgtgtgccctcgatcgaccatgaccgcgcgatcgaggggccatga831<210>3<211>296<212>prt<213>artificialsequence<400>3methishishishishishisargglyserprophethrhishisala151015glyglyargleumetargalaalaleuleuglnleuasnvalserasp202530aspproalaalaasnleualaalathrleualaleuvalaspargala354045valalaglyglyalaalaphevalleuthrproglumetthrasngly505560leuserserserargasphisglnargalavaltyrargprogluasp65707580glnaspgluthrleualaalaleuargalalysalaalaglnalagly859095iletrpleuleuileglyserleuglyleuthrthrthraspalaasp100105110glyargphealaasnargserpheleuileglyproaspglyalaile115120125valalaargtyrasplysilehismetpheaspvalasnvalserasp130135140thrgluvaltyrarggluseralaglytyrargproglyhisglnala145150155160valleualagluthrprophealalysileglymetservalcystyr165170175aspleuargpheprohisleutyrargargleualalysalaglyala180185190glnileleuthrvalproalaalapheasnhisilethrglyalaala195200205histrpgluvalleuleuargalaargalailegluthrglycysphe210215220valleualaproalaglnthrglyphehisalaglualahisglylys225230235240glyargargthrhisglyhisserleualavalalaprotrpglyglu245250255valleualaaspalaglythrgluproglyvalthrphevalaspleu260265270aspleuserglnvalgluargalaargglyargvalproserileasp275280285hisaspargalailegluglypro290295<210>4<211>918<212>dna<213>artificialsequence<400>4atgcatcatcatcatcatcaccgcggatcgcccttcacccaccatgctggcggaaggctg60atgcgcgcggcccttctgcagctgaatgtctcggacgatcccgccgccaatctggccgcg120acgctggcgctggtcgatcgggcggtggcggggggcgcggctttcgtgctgacgcccgag180atgaccaatggcctgtcttcctcgcgcgatcaccagcgcgcggtctaccggcccgaagat240caggacgaaaccctggccgccctgcgcgccaaggcggcgcaggccgggatctggctgctg300atcggatcgctgggcctgaccacgaccgatgccgacgggcgctttgccaaccggtccttc360ctgatcgggccggatggcgccatcgtggcgcgctatgacaagatccacatgttcgacgtg420aacgtgtcggacaccgaagtctaccgcgaaagcgcgggctaccggccgggccatcaggcg480gttctggccgagacgcctttcgccaagatcgggatgagcgtctgctacgacctgcgcttc540ccgcatctgtaccgccgtctggccaaggccggggcgcagatcctgaccgtgccggcagcc600ttcaaccacatcaccggggccgcgcattgggaggtgctgctgcgggcgcgtgcgatcgag660accggctgtttcgtgctggccccggcgcaaaccggctttcacgccgaagcgcatggcaag720ggacgccgcacccatggccattccctggccgtcgcgccctggggcgaggttctggccgat780gccggcaccgagccgggggtcaccttcgtcgatctggacctgtcgcaagtggagcgcgcg840cggggccgtgtgccctcgatcgaccatgaccgcgcgatcgaggggccatgagccaggatc900gtcatgacgagctggccg918当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1