一种重组荞麦谷氧还蛋白及其制备方法和应用与流程
本发明涉及生物技术和医药保健品领域,具体涉及谷氧还蛋白,更具体地说属于一种重组荞麦谷氧还蛋白(recombinant buckwheat glutaredoxin,简称为rbGrx)及其制备方法和应用。
背景技术:
生物体中一些蛋白质可以在半胱氨酸残基活性位点和对应二硫化物的半胱氨酸之间快速进行可逆的巯基和二硫键反应,从而调节细胞内含巯基的蛋白的氧化还原电势,二硫键的可逆还原反应主要通过一系列含双巯基(C-X-X-C)功能区为活性位点的巯基氧化还原酶介导完成。生物体内负责巯基氧化还原的系统主要有硫氧还蛋白系统(Thioredoxin system)和谷氧还蛋白系统(Glutaredoxin system)。Grx与谷胱甘肽(GSH)、谷胱甘肽还原酶(glutathione reductase,GR)和NAD(P)H共同构成Grx系统,通过可逆的氧化还原修饰调节细胞的谷胱甘肽化、去谷胱甘肽化以及二硫键的形成和还原,以维持细胞氧化还原稳态。Grx按活性位点所含半胱氨酸的数量可分为单巯基(C-X-X-S)和双巯基(C-X-X-C)形式。Grx在细胞内行使多种生物学功能,包括体内抗氧化胁迫、调节转录因子、还原脱氢抗坏血酸、抑制细胞凋亡、参与硫代谢等过程。谷氧还蛋白已在水稻、菠菜、拟南芥、杨树等植物中发现,但是荞麦谷氧还蛋白(rbGrx)基因的鉴定以及蛋白的表达纯化和功能分析尚未见报道。
技术实现要素:
本发明的目的在于提供一种重组荞麦谷氧还蛋白及其制备方法,以及该蛋白在催化巯基转移反应中的应用。
本发明的另一目的在于提供一种重组荞麦谷氧还蛋白晶体及其结晶方法,以及重组荞麦谷氧还蛋白晶体及其结构的用途。
为实现上述目的本发明提供如下技术方案:
一种荞麦谷氧还蛋白(rbGrx)基因,其核苷酸序列为SEQ ID NO:1。该基因是通过分析荞麦全基因序列筛选获得的。与来自不同种植物的Grx蛋白的序列进行比对发现,rbGrx与其他植物来源的Grx有序列同源性,rbGrx为植物中谷氧还蛋白家族,并且属于家族分类中第一类蛋白。
一种重组荞麦谷氧还蛋白,其氨基酸序列为SEQ ID NO:2。它由124个氨基酸组成,相对分子量为13500道尔顿。它能够还原蛋白二硫键的同时还可还原谷胱甘肽化的蛋白。
一种重组荞麦谷氧还蛋白的制备方法,包括如下步骤:
(1)将rbGrx基因构建到pGEX-6p-1载体上,转化到大肠杆菌BL21(DE3)中,诱导表达连接有GST的融合蛋白;
(2)通过Glutathione-sepharose亲和柱分离纯化GST-rbGrx蛋白,使用ProScission蛋白酶酶切结合在亲和柱上的GST融合蛋白,然后将酶切后的rbGrx蛋白使用阴离子交换柱与凝胶柱进一步纯化,获得重组rbGrx蛋白。
所述的rbGrx蛋白具有很高的巯基转移酶活性,酶比活力为300U/mg。最适pH为8.5,最适温度为45℃。该蛋白可在催化巯基转移反应中应用,在医疗保健和食品开发等方面也具有实用价值。
一种重组荞麦谷氧还蛋白晶体的结晶方法,步骤包括:将重组rbGrx浓缩至6–7mg/ml,在18-25摄氏度下用悬滴法获得晶体;结晶化溶液的条件为:18%-25%PEG3350(W/V),pH 4.5-pH 5.0,0.1M CH3COONa。
所述的重组荞麦谷氧还蛋白晶体,为rbGrx蛋白与谷胱甘肽(GSH)的复合物晶体;具有P2(1)2(1)2的空间群,晶胞参数约为:α=β=γ=90°;
所述rbGrx晶体的结构,整体呈一个类似于椭球形的立体结构,由6个α螺旋,包围在4个β折叠周围形成;β1,2,4与3反向平行;其中α螺旋1,即含Glu12-Ser28氨基酸区段,β折叠1,即含Leu31-Ser35氨基酸区段,α螺旋2,即含Gly40-Leu52氨基酸区段,β折叠2,即含Gln57-Glu60氨基酸区段,α螺旋3,即含Leu61-Gln63氨基酸区段,α螺旋4,即含Gly67-Thr78氨基酸区段,β折叠3,即含Asn85-Ile88氨基酸区段,β折叠4,即含Lys91-Gly94氨基酸区段,α螺旋5,即含Cys96-Asp105氨基酸区段,α螺旋6,即含Lys107-Cys115氨基酸区段。
基于rbGrx蛋白与GSH复合体的晶体结构,可清晰分辨出与催化活性相关的重要氨基酸残基区域,包括K36,C39,Q71,T82VP84和G95CD97。从而可以有目的调控其稳定性及酶的活力,如突变C39A后其失去巯基转移酶的活性。同时,其结构可为药物设计和食品开发提供理论指导。
附图说明
图1为来自不同种植物的Grx蛋白的序列比对图
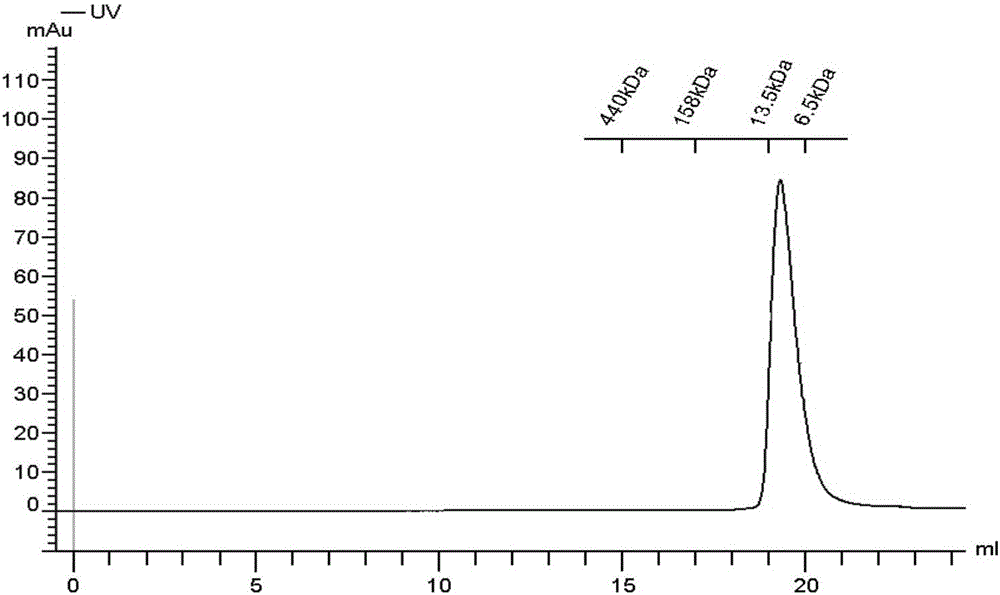
图2 rbGrx蛋白经过凝胶柱纯化的峰图,凝胶分离显示rbGrx以单体形式存在,并且均一性良好。
图3 rbGrx蛋白的酶活测定图,图中:(A)rbGrx酶活力测定图,其中含圆球曲线为阴性对照,含方块曲线为rbGrx酶活曲线;(B)Cys39突变为Ala后活性明显降低;(C)温度对酶活力的影响;(D)pH对酶活力的影响。
图4 rbGrx蛋白的晶体结构图
图5 rbGrx蛋白的催化活性中心结构图
具体实施方式
实施例1 荞麦谷氧还蛋白基因的筛选与分析
荞麦谷氧还蛋白(rbGrx)基因,其核苷酸序列为SEQ ID NO:1。该基因是通过分析荞麦全基因序列筛选获得的。rbGrx与来自不同种植物的Grx蛋白的序列比对(见图1),其中包括九种不同植物的12种Grx序列。其中PoGRXC1使用来自美国国立卫生院数据库2E7P_A,PoGRXS14来自美国国立卫生院数据库Accession:2LKU_A,PoGRXS12来自美国国立卫生院数据库Accession:3FZ9_A,AtGRXS16来自美国国立卫生院数据库Accession:2LWF_A,AtGRXcp来自美国国立卫生院数据库Accession:3IPZ_A,AtGRXC5来自美国国立卫生院数据库Accession:3RHB_A,OrGRX(Oryza sativa)来自美国国立卫生院数据库Accession:CAA54397,CuGRX(Cucumis sativus)来自美国国立卫生院数据库Accession:NP_001274139,IpGRX(Ipomoea batatas)来自美国国立卫生院数据库Accession:ABP48736,ThGRX(Theobroma cacao)来自美国国立卫生院数据库Accession:XP_007036260,RiGRX(Ricinus communis)来自美国国立卫生院数据库Accession:CAA89699。
通过比对分析发明人发现,rbGrx与其他植物来源的Grx有序列同源性,rbGrx为植物中谷氧还蛋白家族,并且属于家族分类中第一类蛋白。图中用…表示在相对应区段的氨基酸缺失。在说明书及权利要求书中具体的氨基酸位置均是以rbGrx的序列进行说明。
实施例2 重组荞麦谷氧还蛋白基因在大肠杆菌中的表达及纯化。
通过基因工程技术将rbGrx基因构建到pGEX-6p-1载体上。将克隆构建的融合蛋白表达质粒转化入BL21(DE3)中,先接种于5mL的LB培养基于37℃过夜培养,12小时后,转接到800mL的LB培养基中扩大培养,37℃下在摇瓶中培养至OD为0.4-0.6左右,4小时后降低培养温度至16℃,然后加入终浓度为0.5mM的IPTG(异丙基-β-D硫代半乳糖苷)进行诱导表达,大约16-20小时后离心收菌。用50mM的HEPES缓冲液(pH 7.8,含200mM NaCl)悬浮菌体,使用低温超高压细胞破碎仪裂解细胞,通过离心分离出不溶性沉淀,将高速离心(约13,000rmp)后获得的上清通过Glutathione-sepharose亲和柱来初步分离纯化GST-rbGrx蛋白。含有GST标签的蛋白可以结合到Glutathione-Sepharose亲和柱,使用50mM HEPEs(pH7.8),500mM NaCl与200mM NaCl缓冲液交替将杂蛋白洗净后,使用ProScission蛋白酶酶切结合在亲和柱上的GST融合蛋白,此过程大约20小时,然后将酶切下来的rbGrx蛋白使用阴离子交换柱与凝胶柱进一步纯化出rbGrx蛋白(见图2),重组蛋白rbGrx纯度达98%以上。
实施例3 重组荞麦谷氧还蛋白rbGrx酶活的测定
蛋白的Grx活力测定:反应体系为500μL,其中包含HEPEs(50mmol/L,pH 7.0),EDTA(1mmol/L),GSH(1mmol/L),NADPH(0.25mmol/L),1U GR(2μmol/L)。将反应物混合进行预保温3min后加入终浓度0.5mmol/L HED(双二羟基乙基二硫醚)启动反应,监测待测物340nm(εNADPH=6220M-1cm-1)处NADPH吸光度值的变化。Grx活力定义为在25℃条件下,每分钟消耗1μmol/L NADPH所需要酶的量即一个活力单位。经过测定得到的rbGrx酶的比活力为300U/mg±15.0(见图3)。
实施例4 荞麦谷氧还蛋白的结晶以及晶体结构解析
重组荞麦谷氧还蛋白的结晶方法,步骤包括:将重组rbGrx浓缩至7mg/ml,在18摄氏度下用悬滴法获得晶体;结晶化溶液的条件为:22%PEG3350(W/V),pH 4.5,0.1M CH3COONa。
rbGrx衍射数据的收集、处理和结构解析:
使用上海同步辐射光源18U线站的X光光源收集衍射数据,rbGrx与GSH复合物晶体衍射分辨率可达2.0埃。利用HKL2000(Otwinowski 1997)处理衍射数据,发现rbGrx与GSH复合物蛋白晶体具有P2(1)2(1)2空间群,晶胞参数为:α=β=γ=90°。
使用PHENIX程序包进行结构解析,利用软件COOT手工进行模型的搭建和修正。进行模拟退火、限制性原子偏移位置修正、能量最小化修正、温度因子等修正后,通过带有sigma A权重因子的2Fo-Fc差值电子图来添加溶剂分子。用软件PROCHECK对模型的几何可靠性进行确认。所有的与结构相关的图像均通过软件PyMOL来进行展示和制作。最终获得结构的R因子为16.8%,自由R因子为21.6%(见图4)。
所述的rbGrx的晶体具有P2(1)2(1)2的空间群,晶胞参数约为:α=β=γ=90°。
其中所述的rbGrx晶体的结构由6个α螺旋,包围在4个β折叠周围形成整体结构,β1,2,4与3反向平行。其中α螺旋1,即含Glu12-Ser28氨基酸区段,β折叠1,即含Leu31-Ser35氨基酸区段,α螺旋2,即含Gly40-Leu52氨基酸区段,β折叠2,即含Gln57-Glu60氨基酸区段,α螺旋3,即含Leu61-Gln63氨基酸区段,α螺旋4,即含Gly67-Thr78氨基酸区段,β折叠3,即含Asn85-Ile88氨基酸区段,β折叠4,即含Lys91-Gly94氨基酸区段,α螺旋5,即含Cys96-Asp105氨基酸区段,α螺旋6,即含Lys107-Cys115氨基酸区段。
基于rbGrx蛋白与GSH复合体的晶体结构,可清晰分辨出与催化活性相关的重要氨基酸残基区域,包括K36,C39,Q71,T82VP84和G95CD97(见图5)。从而可以有目的调控其稳定性及酶的活力,如突变C39A后其失去巯基转移酶的活性。同时,其结构可为药物设计和食品开发提供理论指导。
- 还没有人留言评论。精彩留言会获得点赞!