产生ω‑3脂肪酸的酶和方法与流程
本申请是申请日为2009年11月17日,申请号为200980154876.9,标题为“产生ω-3脂肪酸的酶和方法”的专利申请的分案申请。本发明涉及在重组细胞(例如酵母或植物细胞)中合成长链多不饱和脂肪酸(特别是二十碳五烯酸、二十二碳五烯酸和二十二碳六烯酸)的方法。本发明还提供产生长链多不饱和脂肪酸的重组细胞或植物。此外,本发明涉及一组新的酶,它们具有去饱和酶或延长酶活性,可用于合成长链多不饱和脂肪酸的方法。具体地,本发明提供具有新活性的ω3去饱和酶、δ5延长酶和δ6去饱和酶。本发明还提供用于以多种基因瞬时和/或稳定转化细胞(特别是植物细胞)的方法和dna构建体。
背景技术:
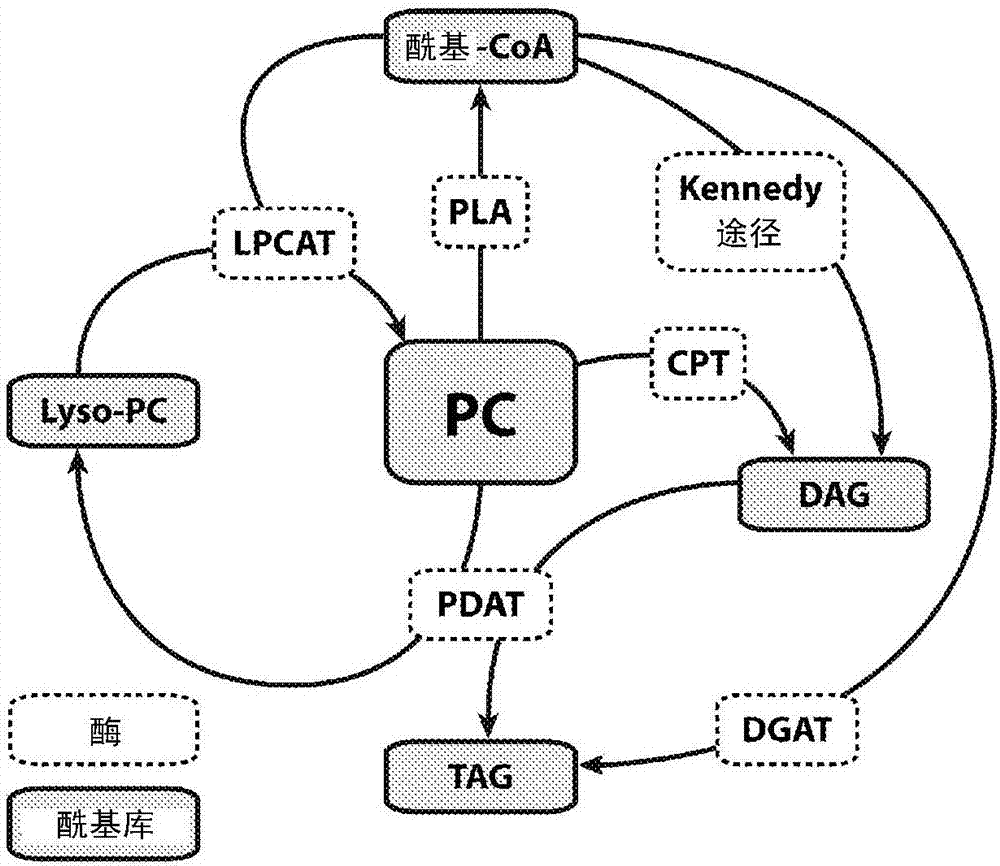
:现在广泛认为ω-3长链多不饱和脂肪酸(lc-pufa和vlc-pufa)是关乎人和动物健康的重要化合物。可从膳食来源或通过转换亚油酸(la,18:2ω6)或α-亚麻酸(ala,18:3ω3)而获得这些脂肪酸,la和ala被认为是人类膳食中的必需脂肪酸。尽管人和许多其他脊椎动物能够将植物来源的la或ala转换为vlc-pufa,但这种转换的速率很慢。此外,大多数现代社会的饮食不平衡,其中至少90%的多不饱和脂肪酸(pufa)是ω6脂肪酸,而非理想的4:1或更低的ω6:ω3脂肪酸的比例(trautwein,2001)。对于人类,vlc-pufa例如二十碳五烯酸(epa,20:5ω3)和二十二碳六烯酸(dha,22:6ω3)的直接来源主要是来自鱼类或鱼油。因此,健康专家们推荐将含有高水平vlc-pufa的鱼类常规纳入人类饮食。例如,来源于鱼类的vlc-pufa油被越来越多地添加到食品和婴儿配方奶中。不过,鉴于全球和国家鱼业的下滑,需要找到这些有益健康的油类的替代来源。与动物不同,高等植物不能合成链长超过18个碳原子的多不饱和脂肪酸。具体地,农作物和园艺植物以及其他被子植物不具有合成长链ω3脂肪酸例如衍生自ala的epa、二十二碳五烯酸(dpa,22:5ω3)和dha所需的酶。因此,植物生物技术的一个重要目的是通过遗传工程学方法改造出产生大量vlc-pufa的农作物,由此提供这些化合物的替代来源。vlc-pufa生物合成途径生物体(例如微藻类、苔藓和真菌)中vlc-pufa的生物合成通常是一系列的依赖氧的去饱和作用和延长反应(图1)。这些生物体中产生epa的最常见的途径包括δ6-去饱和作用、δ6-延长作用和δ5-去饱和作用(称为δ6-去饱和作用途径),较不常见的途径则使用δ9-延长作用、δ8-去饱和作用和δ5-去饱和作用(称为δ9-去饱和作用途径)。这些连续的去饱和作用和延长反应可以从ω6脂肪酸底物la开始,见图1左上部分(ω6),或从ω3底物ala开始,见图1右下部分(ω3)。如果起始的δ6-去饱和作用在ω6底物la上进行,该系类三种酶的vlc-pufa产物会是ω6脂肪酸ara。合成vlc-pufa的生物体可利用ω3-去饱和酶将ω6脂肪酸转换为ω3脂肪酸,如图1所示的将花生四烯酸(ara,20:4ω6)转换为epa的δ17-去饱和酶步骤。ω3-去饱和酶家族的一些成员可作用于从la到ara的大量底物。植物ω3-去饱和酶通常特异性催化由la到ala的δ15-去饱和作用,而真菌和酵母ω3-去饱和酶对由ara到epa的δ17-去饱和作用具有特异性(pereira等,2004a;zank等,2005)。一些报道提示可能存在非特异性的ω3-去饱和酶,可将多种ω6底物转换为相应的ω3产物(zhang等,2007)。其他ω3-去饱和酶可对ω3底物具有偏向性(sayanova等,2003)。在这些生物体中,epa转换为dha相对简单,包括通过δ5-延长作用由epa产生dpa,随后通过δ4-去饱和作用产生dha(图1)。相反,哺乳动物则利用所谓的“sprecher”途径,通过不依赖于δ4去饱和酶的3个分开的反应将dpa转换为dha(sprecher等,1995)。前端去饱和酶通常存在于植物、苔藓、微藻类和低等动物(例如秀丽隐杆线虫(caenorhabditiselegans))中,主要接受与磷酯酰胆碱(pc)底物的sn-2位置发生酯化的脂肪酸底物。这些去饱和酶因此称为酰基-pc脂质连接的前端去饱和酶(domergue等,2003)。相反,高等动物前端去饱和酶通常接受酰基辅酶a底物,其中脂肪酸底物连接于coa而非pc(domergue等,2005)。pufa和vlc-pufa延长反应各自包括由多组分蛋白复合体催化的4个步骤:首先,缩合反应给脂肪酸加上来自丙二酰-coa的2c单元,导致形成β-酮酰基中间体,然后通过nadph将其还原,随后脱水产生烯醇基中间体,该中间体最后被再次还原,产生延长的脂肪酸。通常认为这4个反应中的缩合步骤具有底物特异性,而其他步骤则不具有。在实践中,这意味着,只要引入对vlc-pufa具有特异性的缩合酶(典型地称为“延长酶”),植物中的天然延长机制便能够延长vlc-pufa,只不过植物中的天然延长机制延长非天然vlc-pufa底物的效率可能是低的。2007年鉴定并表征了酵母延长作用循环脱水酶(denicandweissman,2007)。在植物、苔藓和微藻类中,vlc-pufa去饱和作用天然发生在主要属于酰基-pc库的脂肪酸底物,而延长作用则发生在酰基-coa库的底物。脂肪酸从酰基-pc分子转移至coa载体是由磷脂酶类(pla)进行的,而酰基-coa脂肪酸转移至pc载体是由溶血卵磷脂酰基转移酶类(lpcat)进行的(图2)(singh等,2005)。在去饱和作用可以跟上延长作用之前必需发生酰基交换(反之亦然),由此引起的流量下降可通过使用对酰基辅酶a底物具有特异性的去饱和酶而克服(hoffmann等,2008)。遗传工程化产生vlc-pufa绝大多数vlc-pufa代谢工程一直采用需氧δ6-去饱和作用/延长作用途径进行。1996年首次报道了使用来自蓝细菌集胞藻(synechocystis)的δ6-去饱和酶在烟草中生物合成γ-亚麻酸(gla,18:3ω6)(reddyandthomas,1996)。最近,已经在农作物例如向日葵(73%gla;knauf等,2006)和大豆(28%gla;sato等,2004)中产生gla。由于涉及更多的去饱和作用和延长作用步骤,因此产生vlc-pufa例如epa和dha涉及更复杂的工程化。qi等(2004)最早报道了在陆生植物中产生epa,其将编码来自球等鞭金藻(isochrysisgalbana)的δ9-延长酶的基因、编码来自小眼虫(euglenagracilis)的δ8-去饱和酶的基因、和编码来自高山被孢霉(mortierellaalpina)的δ5-去饱和酶的基因引入拟南芥,产生高达3%的epa。随后abbadi等(2004)报道了使用编码来自小立碗藓(physcomitrellapatens)的δ6-去饱和酶和δ6-延长酶的基因和编码来自三角褐指藻(phaeodactylumtricornutum)的δ5-去饱和酶的基因在亚麻子中产生了高达0.8%的epa。wo04/017467首次报道了产生dha,并迄今为止其报道的产生vlc-pufa的水平是最高的,其描述通过引入编码异丝水霉(saprolegniadiclina)δ6-去饱和酶、高山被孢霉δ6-去饱和酶、高山被孢霉δ5-去饱和酶、异丝水霉δ4-去饱和酶、异丝水霉δ17-去饱和酶、高山被孢霉δ6-延长酶和路氏巴夫藻(pavlovalutheri)δ5-延长酶的基因,在大豆胚(而非种子)中产生了3%的dha。在也产生dha的胚中,最高epa水平是19.6%,说明epa转换为dha的效率低下(wo2004/071467)。这一发现与robert等(2005)发表的结果类似,后者的从epa到dha的流量低下,其使用鮐(daniorerio)δ5/6-去饱和酶、秀丽隐杆线虫δ6-延长酶、和盐生巴夫藻(pavlovasalina)δ5-延长酶和δ4-去饱和酶,在拟南芥中产生了3%epa和0.5%dha。也在2005,wuetal.报道使用畸雌腐霉(pythiumirregulare)δ6-去饱和酶、破囊壶菌(thraustochytrid)δ5-去饱和酶、小立碗藓δ6-延长酶、金盏花(calendulaofficianalis)δ12-去饱和酶、破囊壶菌δ5-延长酶、致病疫霉(phytophthorainfestans)δ17-去饱和酶、虹鱒(oncorhynchusmykiss)vlc-pufa延长酶、破囊壶菌δ4-去饱和酶和破囊壶菌lpcat,在芥菜(brassicajuncea)中产生了25%ara、15%epa和1.5%dha(wu等,2005)。因此仍需要在重组细胞中,特别是在油料种子植物的种子中更加高效地产生lc-pufa。技术实现要素:本发明人首次鉴定到了能够在重组细胞中将epa高效转换为dpa的δ5延长酶。因此,本发明还提供重组细胞,优选地是植物细胞,更优选地是植物种子细胞,所述细胞包含编码具有δ5延长酶活性的脂肪酸延长酶的外源性多核苷酸,其中当所述延长酶由所述细胞、优选植物细胞中的所述外源性多核苷酸表达时,所述延长酶对epa具有活性,以至少60%、至少65%、至少70%或至少75%的效率产生dpa。在一个实施方式中,所述延长酶包含seqidno:6所示的氨基酸序列、其生物学活性片段、或与seqidno:6具有至少47%相同性的氨基酸序列。在另一个实施方式中,所述细胞还包含编码以下各项的外源性多核苷酸:i)δ8去饱和酶和/或δ6去饱和酶,ii)δ9延长酶和/或δ6延长酶,iii)δ5去饱和酶,和iv)任选存在的δ4去饱和酶和/或ω3去饱和酶,其中每一多核苷酸可操纵地连接于一或多种能够在所述细胞中指导所述多核苷酸表达的启动子。本发明人还鉴定到了具有新特性的ω3去饱和酶。所述ω3去饱和酶可用于为产生epa、下游脂肪酸dpa和dha、以及其他ω3vlc-pufa而设计的重组途径中。因此,本发明提供重组细胞,优选地是植物细胞,更优选地是植物种子细胞,所述细胞包含编码具有ω3去饱和酶活性的脂肪酸去饱和酶的外源性多核苷酸,其中当所述去饱和酶由所述细胞中的所述外源性多核苷酸表达时,所述去饱和酶能够进行以下至少一种去饱和作用:将ara去饱和化为epa、将dgla去饱和化为eta、将gla去饱和化为sda、将ara去饱和化为epa并将dgla去饱和化为eta、将ara去饱和化为epa并将gla去饱和化为sda、或所有这三种去饱和作用。所述去饱和酶优选地是前端去饱和酶。在另一个实施方式中,所述去饱和酶对酰基链中具有至少三个碳碳双键的c20脂肪酸、优选ara具有δ17去饱和酶活性。在另一个实施方式中,所述去饱和酶对酰基链中具有三个碳碳双键的c18脂肪酸、优选gla具有δ15去饱和酶活性。所述去饱和酶优选地对酰基辅酶a底物的活性高于对相应的酰基-pc底物的活性。在一个实施方式中,所述酰基辅酶a底物是ara-coa,而所述酰基-pc底物包含位于的pc的sn-2位置的ara。在另一个实施方式中,所述细胞是植物细胞,且当所述去饱和酶由所述细胞中的所述外源性多核苷酸表达时,所述去饱和酶对ara具有活性,以至少40%的效率产生epa。在一个具体的实施方式中,所述去饱和酶包含seqidno:15、17或20所示的氨基酸序列、其生物学活性片段、或与seqidno:15具有至少35%相同性的氨基酸序列、与seqidno:17具有至少60%相同性的氨基酸序列和/或与seqidno:20具有至少60%相同性的氨基酸序列。此外,本发明人鉴定到了编码一种δ6去饱和酶的基因,所述δ6去饱和酶在植物和/或酵母中对ω3脂肪酸底物的转换效率高于对ω6脂肪酸底物的转换效率。该δ6去饱和酶还表现出δ8去饱和酶活性。与对ω3去饱和的脂肪酸底物不具有偏向性的去饱和酶相比,在植物的重组lc-pufa途径中使用该δ6去饱和酶或其他对ω3去饱和的脂肪酸底物具有高特异性的去饱和酶提高了epa、dpa和dha的水平。因此,本发明还提供重组细胞,优选地是植物细胞,更优选地是植物种子细胞,所述细胞包含编码具有δ6去饱和酶活性的脂肪酸去饱和酶的外源性多核苷酸,其中所述去饱和酶进一步具有至少两种、优选全部三种以下特性:i)对作为脂肪酸底物的ala的δ6去饱和酶活性高于对la的δ6去饱和酶活性,优选地在植物细胞中是这样,ii)对作为脂肪酸底物的ala-coa的δ6去饱和酶活性高于对作为脂肪酸底物的连接于pc的sn-2位置的ala的δ6去饱和酶活性,优选地在植物细胞中是这样,和iii)对etra具有δ8去饱和酶活性,优选地在植物细胞中是这样。本发明还提供重组细胞,优选地是植物细胞,更优选地是植物种子细胞,所述细胞包含编码具有δ6去饱和酶活性的脂肪酸去饱和酶的外源性多核苷酸,其中所述去饱和酶对ω3底物的活性高于对相应的ω6底物的活性,且其中当所述去饱和酶由所述细胞中的所述多核苷酸表达时,所述去饱和酶对ala具有活性,以至少5%、至少7.5%、或至少10%的效率产生sda,或当所述去饱和酶在酵母细胞中表达时,其以至少35%的效率产生sda。在一个实施方式中,所述去饱和酶对脂肪酸底物ala的δ6去饱和酶活性高于对la的δ6去饱和酶活性,优选地在植物细胞中是这样。所述δ6去饱和酶优选地对ala底物的δ6去饱和酶活性是对la的至少大约2倍、至少3倍、至少4倍、或至少5倍,优选地在植物细胞中是这样。在另一个实施方式中,所述δ6去饱和酶对作为脂肪酸底物的ala-coa的活性高于对作为脂肪酸底物的连接至pc的sn-2位置的ala的活性,优选地在植物细胞中是这样。所述δ6去饱和酶优选地对作为脂肪酸底物的ala-coa的δ6去饱和酶活性是对作为脂肪酸底物的连接至pc的sn-2位置的ala的δ6去饱和酶活性的至少大约5倍或至少10-倍,优选地在植物细胞中是这样。所述δ6去饱和酶优选地是前端去饱和酶。在另一个实施方式中,本发明的细胞还包含编码以下各项的外源性多核苷酸:i)δ6延长酶,ii)δ5去饱和酶,iii)δ5延长酶,和iv)任选存在的δ4去饱和酶和/或ω3去饱和酶,其中每一多核苷酸可操纵地连接于一或多种能够在所述细胞中指导所述多核苷酸表达的启动子。本发明细胞中的所述δ6去饱和酶优选地对eta不具有可检测到的δ5去饱和酶活性。所述δ6去饱和酶优选地包含seqidno:10所示的氨基酸序列、其生物学活性片段、或与seqidno:10具有至少77%相同性的氨基酸序列。在另一个实施方式中,所述δ6去饱和酶包含seqidno:8所示的氨基酸序列、其生物学活性片段、或与seqidno:8具有至少67%相同性的氨基酸序列并具有δ8去饱和酶活性。本发明人还发现,表达δ9延长酶、δ8去饱和酶和δ5去饱和酶的重组细胞能够更高效地将脂肪酸底物转换为epa、dpa和dha。因此,本发明还提供重组细胞,优选地是植物细胞,更优选地是植物种子细胞,所述细胞包含编码以下各项的外源性多核苷酸:i)δ9延长酶,ii)δ8去饱和酶,iii)δ5去饱和酶,iv)任选存在的δ5延长酶,和v)如果存在δ5延长酶,则任选存在的δ4去饱和酶,其中每一多核苷酸可操纵地连接于一或多种能够在所述细胞中指导所述多核苷酸表达的启动子,且其中所述细胞内的总脂肪酸有至少15%、至少20%、或至少25%在它们的酰基链中包含至少20个碳和至少3个碳碳双键。在一个实施方式中,本发明细胞内脂肪酸中的ara、epa、dpa和dha总计占所述细胞内的总脂肪酸的至少15%、至少20%、或至少25%。在进一步的实施方式中,本发明的细胞将油酸转换为二十碳烯酸(c20:1)的能力与野生型植物相比是降低的,和/或在所述细胞中有低于5%的油酸被转换为二十碳烯酸。在一个具体的实施方式中,所述细胞内的总脂肪酸具有低于1%的c20:1。在进一步的实施方式中,本发明的细胞与野生型细胞相比具有降低的内源性δ15去饱和酶活性,和/或所述细胞中有低于10%的la被转换为ala。在一个具体的实施方式中,所述内源性δ15去饱和酶对酰基-pc底物的活性高于对相应的酰基辅酶a底物的活性,优选地其中所述酰基是la。在进一步的实施方式中,本发明的细胞与缺乏所述外源性多核苷酸的相应细胞相比,其将gla转换为sda和/或将ara转换为epa的转换是增加的。在一个实施方式中,本发明的细胞内脂肪酸中的dha的量占所述细胞内的总脂肪酸的至少3%、至少5%、或至少10%。在另一个实施方式中,在本发明的细胞内,la转换为ara和/或ala转换为epa的转换效率是至少80%或至少90%。在一个实施方式中,所述δ9延长酶包含seqidno:22所示的氨基酸序列、其生物学活性片段、或与seqidno:22具有至少80%相同性的氨基酸序列。在进一步的实施方式中,所述δ8去饱和酶包含seqidno:24所示的氨基酸序列、其生物学活性片段、或与seqidno:24具有至少80%相同性的氨基酸序列。在另一个实施方式中,所述δ5去饱和酶包含seqidno:26或seqidno:13所示的氨基酸序列、其生物学活性片段、或与seqidno:26和/或seqidno:13具有至少80%相同性的氨基酸序列。本发明人已经获得的结果表明,可以利用表达所述δ6-去饱和酶、δ6延长酶、δ5去饱和酶、δ5延长酶、和δ4去饱和酶的一组基因、或类似的(特别是其中所述去饱和酶对酰基辅酶a底物具有活性的)一组基因,来合成显著水平的epa、dpa和dha。因此,本发明还提供重组细胞,优选地是植物细胞,更优选地是植物种子细胞,所述细胞包含编码以下各项的外源性多核苷酸:i)δ6延长酶和/或δ9延长酶,ii)δ6去饱和酶和/或δ8去饱和酶,iii)δ5去饱和酶,iv)δ5延长酶,v)δ4去饱和酶,和vi)任选存在的二酰基甘油酰基转移酶,其中每一多核苷酸可操纵地连接于一或多种能够在所述细胞中指导所述多核苷酸表达的启动子,特征在于具有一种或多种或全部以下特性:a)将ala转换为epa、dpa或dha的转换效率是至少17.3%、或至少23%,b)将ala转换为dpa或dha的转换效率是至少15.4%、或至少21%,c)将ala转换为dha的效率是至少9.5%、或至少10.8%,和d)将epa转换为dha的效率是至少45%、或至少50%,且优选地进一步特征在于所述细胞内总脂肪酸中有至少4%是dha。优选地,掺入到细胞内的三酰基甘油中的总脂肪酸中有至少6%、至少11%或至少15%是dha。在一个实施方式中,dha构成所述细胞内sda、eta、epa、dpa和dha的总和的20-65%,优选地为40-65%。优选地,细胞内的ω3脂肪酸中的0.1-25%是sda、0.1-10%是eta、0.1-60%是epa、0.1-50%是dpa、且30-95%是dha,更优选地,所述细胞内的ω3脂肪酸中的0.1-25%是sda、0.1-10%是eta、0.1-50%是epa、0.1-50%是dpa和40-95%是dha。所述δ4去饱和酶优选地包含seqidno:73所示的氨基酸序列、其生物学活性片段、或与seqidno:73具有至少80%相同性的氨基酸序列。在另一个方面,本发明提供重组细胞,优选地是植物细胞,更优选地是植物种子细胞,所述细胞包含编码以下各项的外源性多核苷酸:i)δ6延长酶和/或δ9延长酶,ii)δ6去饱和酶和/或δ8去饱和酶,iii)δ5去饱和酶,iv)δ5延长酶,和v)任选存在的二酰基甘油酰基转移酶其中每一多核苷酸可操纵地连接于一或多种能够在所述细胞中指导所述多核苷酸表达的启动子,特征在于具有一种或多种或全部以下特性:a)将ala转换为epa或dpa的转换效率是至少17.3%、或至少23%,和b)将ala转换为dpa的效率是至少15.4%、或至少21%,且优选地进一步特征在于所述细胞内的总脂肪酸中有至少4%是dpa。优选地,掺入到细胞内的三酰基甘油中的总脂肪酸中有至少6%、至少11%或至少15%是dpa。优选地,dpa构成细胞内sda、eta、epa和dpa的总量的20-65%,更优选地40-65%。优选地,细胞内的ω3脂肪酸中的0.1-35%是sda、0.1-15%是eta、0.1-60%是epa、而30-75%是dpa,更优选地,细胞内的ω3脂肪酸中的0.1-35%是sda、0.1-15%是eta、0.1-50%是epa而40-75%是dpa。在一个实施方式中,所述δ6延长酶包含seqidno:4所示的氨基酸序列、其生物学活性片段、或与seqidno:4具有至少55%相同性的氨基酸序列。在另一个实施方式中,所述δ6去饱和酶包含seqidno:8所示的氨基酸序列、其生物学活性片段、或与seqidno:8具有至少67%相同性的氨基酸序列。在一个实施方式中,所述δ5去饱和酶包含seqidno:26所示的氨基酸序列、其生物学活性片段、或与seqidno:26具有至少80%相同性的氨基酸序列。在另一个实施方式中,所述δ5延长酶包含seqidno:6所示的氨基酸序列、其生物学活性片段、或与seqidno:6具有至少47%相同性的氨基酸序列。在一个实施方式中,所述二酰基甘油酰基转移酶包含seqidno:75或seqidno:108所示的氨基酸序列、其生物学活性片段、或与seqidno:75和/或seqidno:108具有至少80%相同性的氨基酸序列。本发明显然涵盖任何两种、三种、四种或全部上述酶的组合。在另一个实施方式中,本发明的细胞、优选地植物细胞、且更优选地植物种子细胞,还包含编码以下各项的外源性多核苷酸:i)δ17去饱和酶,ii)δ15去饱和酶,和/或iii)δ12去饱和酶其中每一多核苷酸可操纵地连接于一或多种能够在所述细胞中指导所述多核苷酸表达的启动子,在另一个实施方式中,本发明的细胞中由外源性多核苷酸表达的一种或多种或全部所述去饱和酶对酰基辅酶a底物的活性高于对相应的酰基-pc底物的活性。在具体的实施方式中,所述δ6去饱和酶和δ5去饱和酶、δ5去饱和酶和δ4去饱和酶、δ6去饱和酶和δ4去饱和酶、或全部三种所述δ6去饱和酶、δ5去饱和酶和δ4去饱和酶、或在每一上述这些组合的基础上额外加上任何δ17去饱和酶、δ15去饱和酶和/或δ12去饱和酶,均对它们的酰基辅酶a底物的活性高于对相应的酰基-pc底物的活性。在这一实施方式中,细胞中由外源性多核苷酸表达的其他去饱和酶对酰基辅酶a底物的活性可高于或不高于对相应的酰基-pc底物的活性。可以理解,每种酶的优选的酰基辅酶a底物是不同的。本发明还提供重组细胞,优选地是植物细胞,更优选地是植物种子细胞,所述细胞包含编码具有δ6延长酶和δ9延长酶活性的脂肪酸延长酶的外源性多核苷酸,其中所述延长酶的δ6延长酶活性高于δ9延长酶活性。在一个实施方式中,所述延长酶转换sda以产生eta的效率是至少50%或至少60%,和/或转换ala以产生etra的效率是至少6%或至少9%。优选地,所述延长酶具有的δ6延长酶活性是δ9延长酶活性的至少大约6.5倍。在另一个实施方式中,所述延长酶不具有可检测到的δ5延长酶活性。延长酶优选地包含seqidno:4所示的氨基酸序列、其生物学活性片段、或与seqidno:4具有至少55%相同性的氨基酸序列。在另一个实施方式中,所述细胞还包含编码以下各项的外源性多核苷酸:i)δ8去饱和酶和/或δ6去饱和酶,ii)δ5去饱和酶,iii)δ5延长酶,和iv)任选存在的δ4去饱和酶和/或ω3去饱和酶,其中每一多核苷酸可操纵地连接于一或多种能够在所述细胞中指导所述多核苷酸表达的启动子。本发明还提供重组细胞,优选地是植物细胞,更优选地是植物种子细胞,所述细胞包含编码具有δ5去饱和酶活性的脂肪酸去饱和酶的外源性多核苷酸,其中所述去饱和酶包含seqidno:13所示的氨基酸序列、其生物学活性片段、或与seqidno:13具有至少53%相同性的氨基酸序列。本发明还提供重组细胞,优选地是植物细胞,更优选地是植物种子细胞,所述细胞包含编码具有δ9延长酶活性的脂肪酸延长酶的外源性多核苷酸,其中所述延长酶包含seqidno:28、94和96中任一序列所示的氨基酸序列、其生物学活性片段、与seqidno:28具有至少81%相同性的氨基酸序列、或与seqidno:94和/或seqidno:96具有至少50%相同性的氨基酸序列。在一个实施方式中,所述δ9延长酶包含seqidno:94或seqidno:96所示的氨基酸序列、其生物学活性片段、或与seqidno:94和/或seqidno:96具有至少50%相同性的氨基酸序列,且其中所述延长酶对ω6底物的活性高于对相应的ω3底物的活性。更优选地,所述δ9延长酶对ω6底物(例如la)的活性是对相应的ω3底物(例如ala)的活性的至少2倍,更优选地至少4倍。在另一个方面,本发明提供一种重组细胞,其包含编码二酰基甘油酰基转移酶的外源性多核苷酸,其中所述二酰基甘油酰基转移酶包含seqidno:108所示的氨基酸序列、其生物学活性片段、或与seqidno:108具有至少54%相同性的氨基酸序列。在本发明细胞的一个实施方式中,所述去饱和酶和/或延长酶、或多种去饱和酶和/或延长酶,可纯化自微藻类。优选的微藻类是巴夫藻(pavlovaspp)、塔胞藻(pyramimonasspp)和微胞藻(micromonasspp)。在优选的实施方式中,本发明的细胞是真核细胞。例如所述细胞可以是植物细胞、哺乳动物细胞、昆虫细胞、真菌细胞或酵母细胞。所述细胞可以是组织培养物中的细胞、体外的细胞和/或分离的细胞。在一个实施方式中,所述细胞在植物中和/或是成熟的植物种子细胞。所述植物可以是田间的植物或作为植物部分收获的植物,或所述种子可以是收获的种子。在一个具体的实施方式中,所述植物或植物种子分别是油料种子植物或油料种子。本领域人员可以理解,可以在同一细胞中共表达一或多种所定义的延长酶和/或去饱和酶。在进一步的实施方式中,本发明的细胞能够合成长链多不饱和脂肪酸(lc-pufa),其中所述衍生自不能合成所述lc-pufa的细胞。本发明人还发现,共表达沉默阻抑物(silencingsuppressor)可升高植物细胞内脂肪酸生物合成酶类的水平,特别是在从最初的转化植物重复繁殖数代之后。因此,在优选的实施方式中,本发明的细胞,优选地植物细胞,更优选地植物贮藏器官细胞或种子细胞,包含编码沉默阻抑物的外源性多核苷酸。优选地,编码沉默阻抑物的外源性多核苷酸可操纵地连接于植物贮藏器官特异性启动子。在一个实施方式中,所述植物贮藏器官特异性启动子是种子特异性启动子,或是优先在发育的种子中表达的子叶-特异性启动子或胚乳-特异性启动子。本发明还提供获得细胞、优选地植物细胞、更优选地植物种子细胞的方法,所述的细胞能够合成一或多种长链多不饱和脂肪酸(lc-pufa),所述方法包括a)将编码脂肪酸ω3去饱和酶活性的外源性多核苷酸引入细胞,优选地是不能合成所述lc-pufa的细胞,其中所述多核苷酸可操纵地连接于能够在所述细胞中指导所述多核苷酸表达的启动子,b)在所述细胞中表达所述外源性多核苷酸,c)分析所述细胞的脂肪酸组成,和d)选择能够进行以下至少一种去饱和作用的细胞:将ara去饱和化为epa、将dgla去饱和化为eta、将gla去饱和化为sda、将ara去饱和化为epa并将dgla去饱和化为eta、将ara去饱和化为epa并将gla去饱和化为sda、或所有这三种去饱和作用。在一个实施方式中,所选的细胞是本发明的细胞。具体地,所述细胞可进一步包含本申请所描述的去饱和酶和延长酶的组合。本发明还提供获得细胞、优选地植物细胞、更优选地植物种子细胞的方法,所述的细胞能够合成一或多种长链多不饱和脂肪酸(lc-pufa),所述方法包括a)将编码脂肪酸δ5延长酶的外源性多核苷酸引入细胞,优选地是不能合成所述lc-pufa的细胞,其中所述多核苷酸可操纵地连接于能够在所述细胞中指导所述多核苷酸表达的启动子,b)在所述细胞中表达所述外源性多核苷酸,c)分析所述细胞的脂肪酸组成,和d)选择其中所述δ5延长酶对epa具有活性并以至少60%、至少65%、至少70%或至少75%的效率产生dpa的细胞。在本发明方法的一个实施方式中,所选的细胞是本发明的细胞。具体地,所述细胞可进一步包含本申请所描述的去饱和酶和延长酶的组合。本发明还提供获得细胞、优选地植物细胞、更优选地植物种子细胞的方法,所述的细胞能够合成一或多种长链多不饱和脂肪酸(lc-pufa),所述方法包括a)将编码脂肪酸δ6去饱和酶的外源性多核苷酸引入细胞,优选地是不能合成所述lc-pufa的细胞,其中所述多核苷酸可操纵地连接于能够在所述细胞中指导所述多核苷酸表达的启动子,b)在所述细胞中表达所述外源性多核苷酸,c)分析所述细胞的脂肪酸组成,和d)选择具有至少两种、优选全部三种以下特性的细胞:i)对作为脂肪酸底物的ala的δ6去饱和酶活性高于对la的δ6去饱和酶活性,优选地在植物细胞中是这样,ii)对作为脂肪酸底物的ala-coa的δ6去饱和酶活性高于对作为脂肪酸底物的连接于pc的sn-2位置的ala的δ6去饱和酶活性,优选地在植物细胞中是这样,和iii)对ala具有δ6去饱和酶活性并对etra具有δ8去饱和酶活性,优选地在植物细胞中是这样。本发明还提供获得细胞、优选地植物细胞、更优选地植物种子细胞的方法,所述的细胞能够合成一或多种长链多不饱和脂肪酸(lc-pufa),所述方法包括a)将编码脂肪酸δ6去饱和酶的外源性多核苷酸引入细胞,优选地是不能合成所述lc-pufa的细胞,其中所述多核苷酸可操纵地连接于能够在所述细胞中指导所述多核苷酸表达的启动子,b)在所述细胞中表达所述外源性多核苷酸,c)分析所述细胞的脂肪酸组成,和d)选择具有δ6去饱和酶活性的细胞,所述δ6去饱和酶对ω3底物的活性高于对相应的ω6底物的活性,且所述δ6去饱和酶对ala具有活性并以至少5%、至少7.5%、或至少10%的效率产生sda,或当其在酵母细胞中表达时以至少35%的效率产生sda。在一个实施方式中,所选的细胞是本发明的细胞。具体地,所述细胞可进一步包含本申请所描述的去饱和酶和延长酶的组合。本发明还提供获得细胞、优选地植物细胞、更优选地植物种子细胞的方法,所述的细胞能够合成一或多种长链多不饱和脂肪酸(lc-pufa),所述方法包括a)将编码以下各项的外源性多核苷酸引入细胞,优选地是不能合成所述lc-pufa的细胞:i)δ9延长酶,ii)δ8去饱和酶,iii)δ5去饱和酶,iv)任选存在的δ5延长酶,和v)如果存在δ5延长酶,则任选存在的δ4去饱和酶,其中每一多核苷酸可操纵地连接于一或多种能够在所述细胞中指导所述多核苷酸表达的启动子,b)在所述细胞中表达所述外源性多核苷酸,c)分析所述细胞的脂肪酸组成,和d)选择其中总脂肪酸有至少15%、至少20%或至少25%在它们的酰基链中包含至少20个碳和至少3个碳碳双键的细胞。在一个实施方式中,所选的细胞是本发明的细胞。本发明还提供获得细胞、优选地植物细胞、更优选地植物种子细胞的方法,所述的细胞能够合成一或多种长链多不饱和脂肪酸(lc-pufa),所述方法包括a)将编码以下各项的外源性多核苷酸引入细胞,优选地是不能合成所述lc-pufa的细胞:i)δ6延长酶和/或δ9延长酶,ii)δ6去饱和酶和/或δ8去饱和酶,iii)δ5去饱和酶,iv)δ5延长酶,v)δ4去饱和酶,和vi)任选存在的二酰基甘油酰基转移酶,其中每一多核苷酸可操纵地连接于一或多种能够在所述细胞中指导所述多核苷酸表达的启动子,b)在所述细胞中表达所述外源性多核苷酸,c)分析所述细胞的脂肪酸组成,和d)选择具有一种或多种或全部以下特性的细胞:1)将ala转换为epa、dpa或dha的转换效率是至少17.3%、或至少23%;2)将ala转换为dpa或dha的转换效率是至少15.4%、或至少21%;3)将ala转换为dha的效率是至少9.5%、或至少10.8%;和4)将epa转换为dha的效率是至少45%、或至少50%;且优选地进一步特征在于所述细胞内总脂肪酸中有至少4%是dha。在另一方面,本发明提供获得细胞、优选地植物细胞、更优选地植物种子细胞的方法,所述的细胞能够合成一或多种长链多不饱和脂肪酸(lc-pufa),所述方法包括a)将编码以下各项的外源性多核苷酸引入细胞,优选地是不能合成所述lc-pufa的细胞:i)δ6延长酶和/或δ9延长酶,ii)δ6去饱和酶和/或δ8去饱和酶,iii)δ5去饱和酶,iv)δ5延长酶,和v)任选存在的二酰基甘油酰基转移酶其中每一多核苷酸可操纵地连接于一或多种能够在所述细胞中指导所述多核苷酸表达的启动子,b)在所述细胞中表达所述外源性多核苷酸,c)分析所述细胞的脂肪酸组成,和d)选择具有一种或多种或全部以下特性的细胞:a)将ala转换为epa或dpa的转换效率是至少17.3%、或至少23%,和b)将ala转换为dpa的效率是至少15.4%、或至少21%,且优选地进一步特征在于所述细胞内的总脂肪酸中有至少4%是dpa。在本发明的方法的一个实施方式中,所述外源性多核苷酸稳定整合到所述细胞的基因组中。在另一个实施方式中,所述方法还包括从步骤a)的细胞再生转化植物的步骤。在进一步的实施方式中,所述外源性多核苷酸在所述细胞中瞬时表达。在一个实施方式中,所述细胞是植物的叶细胞。本发明还提供选择参与脂肪酸去饱和作用的核酸分子的方法,包括:i)获得可操纵地连接于启动子的核酸分子,所述核酸分子编码多肽,所述多肽可能是脂肪酸去饱和酶;ii)将所述核酸分子引入细胞,其中所述启动子在所述细胞中具有活性;iii)在所述细胞中表达所述核酸分子;iv)分析所述细胞中的脂肪酸组成;和v)基于以下事实选择参与脂肪酸去饱和作用的核酸分子:所述多肽具有ω3去饱和酶活性并能够进行以下至少一种去饱和作用:将ara去饱和化为epa、将dgla去饱和化为eta、将gla去饱和化为sda、将ara去饱和化为epa并将dgla去饱和化为eta、将ara去饱和化为epa并将gla去饱和化为sda、或所有这三种去饱和作用。在所述方法的一个实施方式中,所述多肽的氨基酸序列与seqidno:15具有至少35%的相同性、与seqidno:17具有至少60%的相同性、和/或与seqidno:20具有至少60%的相同性。本发明还提供选择参与脂肪酸延长作用的核酸分子的方法,包括:i)获得可操纵地连接于启动子的核酸分子,所述核酸分子编码多肽,所述多肽可能是脂肪酸延长酶,ii)将所述核酸分子引入细胞,其中所述启动子在所述细胞中具有活性,iii)在所述细胞中表达所述核酸分子;iv)分析所述细胞中的脂肪酸组成;和v)基于以下事实选择参与脂肪酸延长作用的核酸分子:所述多肽具有δ5延长酶活性且其转换epa产生dpa的效率是至少60%、至少65%、至少70%或至少75%。在一个实施方式中,所述多肽的氨基酸序列与seqidno:6具有至少47%的相同性。本发明还提供选择参与脂肪酸去饱和作用的核酸分子的方法,包括:i)获得可操纵地连接于启动子的核酸分子,所述核酸分子编码多肽,所述多肽可能是脂肪酸去饱和酶,ii)将所述核酸分子引入细胞,其中所述启动子在所述细胞中具有活性,iii)在所述细胞中表达所述核酸分子;iv)分析所述细胞中的脂肪酸组成;和v)基于以下事实选择参与脂肪酸去饱和作用的核酸分子:所述多肽具有δ6去饱和酶活性并具有至少两种、优选全部三种以下特性:a)对作为脂肪酸底物的ala的δ6去饱和酶活性高于对la的δ6去饱和酶活性,优选地在植物细胞中是这样,b)对作为脂肪酸底物的ala-coa的δ6去饱和酶活性高于对作为脂肪酸底物的连接于pc的sn-2位置的ala的δ6去饱和酶活性,优选地在植物细胞中是这样,和c)对ala具有δ8去饱和酶活性,优选地在植物细胞中是这样。在所述方法的一个实施方式中,所述多肽的氨基酸序列与seqidno:10具有至少77%的相同性和/或与seqidno:8具有至少67%的相同性。本发明还提供选择参与脂肪酸去饱和作用的核酸分子的方法,包括:i)获得可操纵地连接于启动子的核酸分子,所述核酸分子编码多肽,所述多肽可能是脂肪酸去饱和酶,ii)将所述核酸分子引入细胞,其中所述启动子在所述细胞中具有活性,iii)在所述细胞中表达所述核酸分子;iv)分析所述细胞中的脂肪酸组成;和v)基于以下事实选择参与脂肪酸去饱和作用的核酸分子:所述多肽具有δ6去饱和酶活性和δ8去饱和酶活性。在一个实施方式中,所述多肽的氨基酸序列与seqidno:8具有至少67%的相同性。在另一个实施方式中,本发明的方法的步骤(v)包括选择编码对酰基辅酶a底物具有活性的去饱和酶的核酸分子或编码前端去饱和酶的核酸分子。本发明还提供本文中所定义的外源性多核苷酸的组合,当它们用于产生重组细胞时,它们在重组细胞中表达至少两种脂肪酸去饱和酶和两种脂肪酸延长酶的组合、和/或在重组细胞中产生lc-pufa。本发明还提供基本上纯化的和/或重组脂肪酸δ5延长酶,其中当由外源性多核苷酸在细胞中表达时,所述延长酶对epa具有活性,以至少60%、至少65%、至少70%或至少75%的效率产生dpa。在一个实施方式中,所述δ5延长酶的特征在于具有本文所定义的任何一或多种特性。本发明还提供基本上纯化的和/或重组脂肪酸ω3去饱和酶,当由外源性多核苷酸在细胞中表达时,其能够进行以下至少一种去饱和作用:将ara去饱和化为epa、将dgla去饱和化为eta、将gla去饱和化为sda、将ara去饱和化为epa并将dgla去饱和化为eta、将ara去饱和化为epa并将gla去饱和化为sda、或所有这三种去饱和作用。在一个实施方式中,本发明的ω3去饱和酶的特征在于具有本文所定义的任何一或多种特性。本发明还提供基本上纯化的和/或重组脂肪酸δ6去饱和酶,其中所述去饱和酶进一步具有至少两种、优选全部三种以下特性:i)对作为脂肪酸底物的ala的δ6去饱和酶活性高于对la的δ6去饱和酶活性,优选地在植物细胞中是这样,ii)对作为脂肪酸底物的ala-coa的δ6去饱和酶活性高于对作为脂肪酸底物的连接于pc的sn-2位置的ala的δ6去饱和酶活性,优选地在植物细胞中是这样,和iii)对etra具有δ8去饱和酶活性,优选地在植物细胞中是这样。本发明还提供基本上纯化的和/或重组脂肪酸δ6去饱和酶,其中所述去饱和酶对ω3底物的活性高于对相应的ω6底物的活性,且其中所述去饱和酶对ala具有活性,当所述去饱和酶由外源性多核苷酸在细胞中表达时以至少5%、至少7.5%、或至少10%的效率产生sda,或当其在酵母细胞中表达时以至少35%的效率产生sda。在一个实施方式中,本发明的δ6去饱和酶的特征在于具有本文所定义的任何一或多种特性。本发明还提供基本上纯化的和/或重组脂肪酸δ6延长酶和δ9延长酶,其中所述延长酶的δ6延长酶活性高于δ9延长酶活性。在一个实施方式中,本发明的δ6延长酶和δ9延长酶的特征在于具有本文所定义的任何一或多种特性。本发明还提供基本上纯化的和/或重组脂肪酸δ5去饱和酶,其包含seqidno:13所示的氨基酸序列、其生物学活性片段、或与seqidno:13具有至少53%相同性的氨基酸序列。本发明还提供基本上纯化的和/或重组脂肪酸δ9延长酶,其包含seqidno:28、94和96任一所示的氨基酸序列、其生物学活性片段、与seqidno:28具有至少81%相同性的氨基酸序列,或与seqidno:94和/或seqidno:96具有至少50%相同性的氨基酸序列。在一个实施方式中,所述δ9延长酶包含seqidno:94或seqidno:96所示的氨基酸序列、其生物学活性片段、或与seqidno:94和/或seqidno:96具有至少50%相同性的氨基酸序列,且其中所述延长酶对ω6底物的活性高于对相应的ω3底物的活性。在另一个方面,本发明提供基本上纯化的和/或重组二酰基甘油酰基转移酶,其包含seqidno:108所示的氨基酸序列、其生物学活性片段、或与seqidno:108具有至少54%相同性的氨基酸序列。在一个实施方式中,本发明的去饱和酶或延长酶可纯化自微藻类。优选的微藻类是巴夫藻、塔胞藻和微胞藻。本发明还提供分离的和/或外源性多核苷酸,其包含:i)选自seqidno:3、5、7、9、11、12、14、16、18、19、27、29、93、95、107或125至129中任一序列的核苷酸序列,ii)编码本发明的去饱和酶或延长酶的核苷酸序列,iii)与seqidno:3、5、7、9、11、12、14、16、18、19、27、29、93、95、107或125至129所示的一或多种序列具有至少50%相同性的核苷酸序列,和/或iv)在严格性条件下与i)至iii)中任一序列杂交的序列。在一个实施方式中,所述分离的和/或外源性多核苷酸包含与seqidno:3和/或seqidno:126具有至少57%相同性的核苷酸序列,并编码δ6延长酶。在另一个实施方式中,所述分离的和/或外源性多核苷酸包含与seqidno:14、seqidno:16、seqidno:18和/或seqidno:19具有至少50%相同性的核苷酸序列,并编码ω3去饱和酶。在另一个实施方式中,所述分离的和/或外源性多核苷酸包含与seqidno:5和/或seqidno:128具有至少50%相同性的核苷酸序列,并编码δ5延长酶。在一个实施方式中,所述分离的和/或外源性多核苷酸包含与seqidno:7、seqidno:9、seqidno:11和/或seqidno:125具有至少75%相同性的核苷酸序列,并编码δ6去饱和酶。在另一个实施方式中,所述分离的和/或外源性多核苷酸包含与seqidno:12具有至少60%相同性的核苷酸序列,并编码δ5去饱和酶。在另一个实施方式中,所述分离的和/或外源性多核苷酸包含与seqidno:27、seqidno:29、seqidno:93和/或seqidno:96具有至少50%相同性的核苷酸序列,并编码δ9延长酶。在另一个实施方式中,所述分离的和/或外源性多核苷酸包含与seqidno:107具有至少60%相同性的核苷酸序列,并编码二酰基甘油酰基转移酶。在一个具体的实施方式中,所述分离的和/或外源性多核苷酸与seqidno:3、5、7、9、11、12、14、16、18、19、27、29、93、95、107或125至129所示序列之一具有至少80%、或至少90%、或至少95%、或至少99%的相同性。本发明还提供用于整合到和/或已整合到植物细胞基因组中的dna构建体,所述构建体包含一簇至少三个编码调控植物细胞内脂肪酸合成的蛋白质的可读框,优选地每一蛋白质是脂肪酸去饱和酶或脂肪酸延长酶,其中具有相同转录方向的各可读框间隔至少750bp、至少1,000bp或至少1,250bp,且至少有两个可读框具有不同的转录方向,其中每一可读框可操纵地连接于在植物细胞中具有活性的启动子,且每一启动子可独立地是相同的或不同的。优选地,所述dna构建体中至少有两个启动子是不同的。一或多个或每一所述可读框优选地可操纵地连接于异源性5’前导序列,每一前导序列可独立地是相同的或不同的,其中与特定可读框的天然存在的5’前导序列相比,每一异源性5’前导序列使翻译效率提高。在本发明的dna构建体中,所述异源性5’前导序列优选地是烟草花叶病毒(tmv)5’前导序列。在本发明的dna构建体中,所述蛋白质优选地是延长酶和/或去饱和酶,更优选地是本申请所述的组合。在另一个实施方式中,本发明的dna构建体仅具有三个或四个被翻译成蛋白质的可读框。本发明还提供载体,其包含本发明的多核苷酸和/或本发明的dna构建体。优选地,所述多核苷酸可操纵地连接于启动子。本发明还提供产生本发明的去饱和酶或延长酶的方法,所述方法包括在细胞或无细胞表达系统中表达本发明的多核苷酸、本发明的dna构建体和/或本发明的载体。本发明人还令人惊异地发现,可将至少三种独立的包含不同外源性多核苷酸的染色体外转移核酸瞬时转染至真核细胞内并可组合检测细胞内每一外源性多核苷酸的活性。因此,在另一方面,本发明还提供以至少三种不同的外源性多核苷酸瞬时转染真核细胞的方法,所述方法包括:i)获得至少a)第一细菌,其包含包含第一外源性多核苷酸的染色体外转移核酸,b)第二细菌,其包含包含第二外源性多核苷酸的染色体外转移核酸,和c)第三细菌,其包含包含第三外源性多核苷酸的染色体外转移核酸,和ii)将所述细胞与步骤i)的细菌接触,其中每一染色体外转移核酸从细菌转移至细胞中以产生瞬时转染的细胞,其中每一外源性多核苷酸包含在所述细胞中具有活性的启动子,其中每一启动子可以独立地是相同的或不同的,且其中至少有一种外源性多核苷酸编码沉默阻抑物。可以一或多种所述细菌依次或同时进行步骤ii)。例如,细胞可以接触第一细菌,然后接触第二细菌,等等。在另一实例中,细菌同时接触各种细菌,优选地为所述细菌的混合物。不同细菌的浓度可彼此不同或可以是相同的或相近的。所述细菌可以是混合的分离物(pooledisolates),例如包含来自菌株文库的多种分离物。在一个实施方式中,所述方法还包括获得所述细胞然后使所述细胞接触一或多种额外的细菌,每一细菌包含包含不同外源性多核苷酸的染色体外转移核酸。例如,在一个实施方式中,所述方法包括获得所述细胞然后使所述细胞接触第四细菌,所述第四细菌包含包含第四外源性多核苷酸的染色体外转移核酸。在另外一实施方式中,所述方法包括获得所述细胞然后使所述细胞接触第五细菌,所述第五细菌包含包含第五外源性多核苷酸的染色体外转移核酸。在另外一实施方式中,所述方法包括获得所述细胞然后使所述细胞接触第六细菌,所述第六细菌包含包含第六外源性多核苷酸的染色体外转移核酸。在另外一实施方式中,所述方法包括获得所述细胞然后使所述细胞接触第七细菌,所述第七细菌包含包含第七外源性多核苷酸的染色体外转移核酸。在又一实施方式中,所述方法包括获得所述细胞然后使所述细胞接触第八细菌,所述第八细菌包含包含第八外源性多核苷酸的染色体外转移核酸。优选地,所述的不同外源性多核苷酸编码不同rna分子和/或多肽。在一个实施方式中,每一外源性多核苷酸编码形成酶途径的一部分的酶或是此种酶的候选物。上述方面特别可用于研究形成大的和/或复杂的生物学途径的多核苷酸和/或多肽。因此,在优选的实施方式中,每一外源性多核苷酸编码酶或是此种酶的候选物,所述酶参与脂肪酸合成、脂肪酸修饰、二酰基甘油组装、三酰基甘油组装、或其中两种或更多种的组合。在一个实施方式中,一或多种所述细菌的形式是原生质体。可用于本发明的细菌的实例包括,但不限于,土壤杆菌、根瘤菌、苜蓿中华根瘤菌(sinorhizobiummeliloti)、百脉根中慢生根瘤菌(mezorhizobiumloti)、弗氏志贺菌(shigellaflexneri)、鼠伤寒沙门氏菌(salmonellatyphimurium)、猪霍乱沙门氏菌(salmonellacholeraesuis)、单核细胞增多性李司忒菌(listeriamonocytogenes)、大肠杆菌(escherichiacoli)、假结核病耶尔森菌(yersiniapseudotuberculosis)和小肠结肠炎耶尔森菌(yersiniaenterocolitica)。可用于本发明的染色体外转移核酸的实例包括,但不限于,p-dna、土壤杆菌、t-dna、或它们的组合。优选地,上述方面的细胞是植物细胞或哺乳动物细胞。在一个实施方式中,所述细胞是组织或器官的一部分。在另一个实施方式中,所述细胞是植物细胞,所述组织或器官是叶、茎、根、分生组织、愈伤组织、或胚珠。本发明人还确定了,如果启动子是种子特异性启动子,外源性多核苷酸在叶细胞中的表达可共表达种子特异性转录因子(例如多叶子叶2(leafycotyledon2)、视褐质3(fusca3)或脱落酸敏感3(abscisicacid-senstive3))而增强。多叶子叶2蛋白的实例包括,但不限于,wo01/70777中所述的那些。因此,在优选的实施方式中,所胡植物细胞是植物的叶细胞,至少一种所述启动子是种子特异性启动子,且至少一种所述外源性多核苷酸编码种子特异性转录因子例如多叶子叶2。在一个实施方式中,所述外源性多核苷酸无一是病毒基因。在一个实施方式中,一或多种所述外源性多核苷酸在所述染色体外转移核酸中仅以所述核酸序列的单拷贝形式存在,而不是所述核酸序列的多聚体或部分多聚体。在另一个实施方式中,至少一种所述染色体外转移核酸不包括在所述细胞中具有功能的复制起始点,优选地不是病毒复制起始点,更优选地不是fbnyv复制起始点。在另一个实施方式中,所述外源性多核苷酸无一编码病毒复制酶或病毒移动蛋白,例如wo2007/137788和marillonnet等(2005)所述的那些。本发明还提供筛选瞬时转染的细胞的所需活性的方法,所述方法包括以至少三种本发明的外源性多核苷酸瞬时转染真核细胞,并测试所述细胞的所需活性。本发明人还发现,通过以不同的染色体外转移核酸来提供6种以上不同基因,可增强所述6种以上不同基因对细胞、特别是植物细胞的转染。因此,在另一方面,本发明提供以至少六种不同的外源性多核苷酸转化真核细胞的方法,所述方法包括i)获得至少a)第一细菌,其包含第一染色体外转移核酸,所述第一染色体外转移核酸包含三种、四种、五种或六种不同的外源性多核苷酸,和b)第二细菌,其包含不同于所述第一染色体外转移核酸的第二染色体外转移核酸,所述第二染色体外转移核酸包含三种、四种、五种或六种不同的外源性多核苷酸,ii)将所述细胞与步骤i)的细菌接触,和iii)任选地选择被所述第一和第二染色体外转移核酸的外源性多核苷酸稳定转化的细胞,其中每一所述第一和第二染色体外转移核酸的外源性多核苷酸由所述细菌转移至所述细胞中以产生转化的细胞,其中每一所述外源性多核苷酸包含启动子,所述启动子在所述细胞中或可由其衍生的细胞中具有活性,且其中每一启动子可以独立地是相同的或不同的。可以两种细菌依次或同时进行步骤i)a)和i)b)。例如,所述细胞可先接触第一细菌,然后接触第二细菌。与第二细菌接触的细胞可以是与第一细菌接触后的细胞的子代细胞或衍生自与第一细菌接触后的细胞。在另一实例中,所述细胞同时与这两种细菌接触。在一个实施方式中,所述i)第一染色体外转移核酸只有3-6种、3-5种、3-4种、4-6种、4-5种或5-6种不同的外源性多核苷酸,和ii)第二染色体外转移核酸只有3-6种、3-5种、3-4种、4-6种、4-5种或5-6种不同的外源性多核苷酸。优选地,每一外源性多核苷酸编码多肽,且其中各多肽是不同的。在另一个实施方式中,i)第一染色体外转移核酸包含两种外源性多核苷酸,它们独立地编码选自如下一组的多肽:δ6去饱和酶、δ12去饱和酶和δ15去饱和酶,和ii)第二染色体外转移核酸包含编码多肽的外源性多核苷酸,所述多肽是该组中的第三种酶。优选地,所述细胞是植物细胞,且所述方法还包括从所述稳定转化的细胞再生转化植物的步骤。本发明还提供细胞,所述细胞是通过所述以至少三种本发明的外源性多核苷酸瞬时转染真核细胞的方法而产生的,或是通过所述以至少6种不同的本发明外源性多核苷酸转化真核细胞的方法而产生的。在又一方面中,本发明提供产生以至少六种不同的外源性多核苷酸稳定转化的植物的方法,所述方法包括i)获得包含第一外源性基因组区的第一种稳定转化植物,所述第一外源性基因组区包含3、4、5、或6种不同的外源性多核苷酸,ii)获得与所述第一种稳定转化植物具有性亲和性并包含第二外源性基因组区的第二种稳定转化植物,所述第二外源性基因组区不同于所述第一外源性基因组区并包含3、4、5、或6种不同的外源性多核苷酸,iii)使所述第一种稳定转化植物与所述第二种稳定转化植物杂交,和iv)选择由步骤iii)产生的包含所述第一和第二基因组区的植物或其子代,由此产生稳定转化植物,其中每一所述外源性多核苷酸包含启动子,所述启动子在植物中具有活性,且其中每一启动子可以独立地是相同的或不同的。在优选的实施方式中,按照前述本发明的dna构建体中所述来定向和间隔所述第一和/或第二外源性基因组区的外源性多核苷酸。在所述第一和第二外源性基因组区中,任一启动子序列可出现多次或仅出现一次,或者,在所述第一和第二外源性基因组区中,一或多种启动子可出现多次而一或多种其他启动子仅出现一次。每一植物启动子可独立地优先在所述植物的组织或器官中具有活性,例如,相对于其他组织或器官而言,在叶或种子中具有活性。这可使得所有引入的蛋白质编码区在所述植物器官或组织中同时表达或重叠表达。在并列的实施方式中,一或多种启动子在所述植物中组成型表达,而一或多种其他启动子优先在植物器官或组织中表达。在一个实施方式中,步骤i)包括通过以下步骤产生所述第一种稳定转化植物a)将植物细胞与包含第一染色体外转移核酸的第一细菌接触,所述第一染色体外转移核酸包含三种、四种、五种或六种不同的外源性多核苷酸,b)从步骤a)的植物细胞产生稳定转化植物,和任选地c)产生来自步骤b)的稳定转化植物的子代植物;和/或步骤ii)包括通过以下步骤产生所述第二种稳定转化植物,d)将植物细胞与包含第二染色体外转移核酸的第二细菌接触,所述第二染色体外转移核酸包含三种、四种、五种或六种不同的外源性多核苷酸,e)从步骤d)的植物细胞产生稳定转化植物,和任选地f)产生来自步骤e)的稳定转化植物的子代植物。在另一个方面,本发明提供产生以至少六种不同的外源性多核苷酸稳定转化的植物的方法,所述方法包括i)获得包含第一外源性基因组区的第一种稳定转化植物或植物部分,所述第一外源性基因组区包含3、4、5、或6种不同的外源性多核苷酸,ii)使所述第一种稳定转化植物或植物部分的细胞接触包含染色体外转移核酸的细菌,所述染色体外转移核酸包含三种、四种、五种或六种不同的外源性多核苷酸,iii)从所述细胞产生植物,和iv)任选地,选择由步骤iii)产生的包含所述至少六种不同的外源性多核苷酸的植物。对于上述各方面的“将细胞与步骤i)的细菌接触”这一步骤,正如本领域人员能够理解的那样,这一步骤要在合适的条件下进行合适的时间,以便染色体外转移核酸从细菌转移至细胞。在另一方面,本发明提供真核细胞至少包含a)包含第一外源性多核苷酸的第一染色体外转移核酸,b)包含第二外源性多核苷酸的第二染色体外转移核酸,和c)包含第三外源性多核苷酸的第三染色体外转移核酸。在一个实施方式中,所述细菌进一步一或多种额外的细菌,每一细菌包含包含外源性多核苷酸的染色体外转移核酸。本发明还提供植物或其子代或种子,其包含包含3、4、5、或6种不同的外源性多核苷酸的第一外源性基因组区,和包含3、4、5、或6种不同的外源性多核苷酸的第二外源性基因组区。所述外源性基因组区的外源性多核苷酸优选地按照前述dna构建体中所述来定向和间隔。本发明还提供包含本发明的细胞的转基因非人类生物体。在一个实施方式中,所述生物体的每一细胞均是本发明的细胞。优选地,所述转基因非人类生物体是转基因植物,更优选地是转基因油料种子植物,其产生下文所列的的油。在进一步的实施方式中,所述转基因植物包含至少一种额外的编码沉默阻抑物的外源性多核苷酸,其可操纵地连接于植物贮藏器官特异性启动子,其中所述植物具有正常的表型。本发明还提供种子,其包含本发明的细胞或得自本发明的转基因植物。本发明还提供油,其由本发明的细胞、本发明的转基因非人类生物体、或本发明的种子产生,或得自本发明的细胞、本发明的转基因非人类生物体、或本发明的种子。在一个实施方式中,所述油是通过对来自油料种子的油进行提取而获得的。在一个实施方式中,所述油介花油(欧洲油菜(brassicanapus)、芜菁(brassicarapa))、芥子油(芥菜(brassicajuncea))、其他芸苔油、葵花子油(向日葵(helianthusannus))、亚麻籽油(栽培亚麻(linumusitatissimum))、大豆油(大豆(glycinemax))、红花油(红花(carthamustinctorius))、玉米油(玉蜀黍(zeamays))、烟草油(烟草(nicotianatabacum))、花生油(花生(arachishypogaea))、棕榈油、棉籽油(陆地棉(gossypiumhirsutum))、椰子油(椰子(cocosnucifera))、鳄梨油(鳄梨(perseaamericana))、橄榄油(橄榄(oleaeuropaea))、腰果油(腰果(anacardiumoccidentale))、夏威夷果油(夏威夷果(macadamiaintergrifolia))、扁桃仁油(扁桃(prunusamygdalus))或拟南芥籽油(拟南芥(arabidopsisthaliana))。本发明还提供脂肪酸,其由本发明的细胞、本发明的转基因非人类生物体、或本发明的种子产生,或得自本发明的细胞、本发明的转基因非人类生物体、或本发明的种子。本发明还提供产生含不饱和脂肪酸的油的方法,所述方法包括从本发明的细胞、本发明的转基因非人类生物体、或本发明的种子提取油。本发明还提供组合物,其包含本发明的细胞、本发明的去饱和酶或延长酶、本发明的多核苷酸、本发明的dna构建体、本发明的载体、本发明的油或本发明的脂肪酸。本发明还提供饲料、化妆品或化学制品,其包含本发明的细胞、本发明的转基因非人类生物体、本发明的种子、本发明的油和/或本发明的脂肪酸。本发明还提供进行去饱和酶反应的方法,所述方法包括使与coa酯化的多不饱和脂肪酸接触本发明的去饱和酶。本发明还提供基本上纯化的抗体或其片段,其特异性结合本发明的去饱和酶或延长酶.本发明还提供治疗或预防可能从pufa获益的病症的方法,所述方法包括给对象施用本发明的细胞、本发明的去饱和酶或延长酶、本发明的多核苷酸、本发明的dna构建体、本发明的载体、本发明的转基因非人类生物体、本发明的种子、本发明的油或本发明的脂肪酸和/或本发明的饲料。在一个实施方式中,所述病症是心律失常、血管成形术、炎症、哮喘、银屑病、骨质疏松症、肾脏结石、aids、多发性硬化、类风湿关节炎、克隆病、精神分裂症、癌症、胎儿酒精综合征、注意力缺陷多动症、囊性纤维化、苯丙酮尿症、单相抑郁、攻击敌意、肾上腺脑白质营养不良、冠心病、高血压、糖尿病、肥胖、阿尔茨海默病、慢性阻塞性肺疾病、溃疡性结肠炎、血管成形术后再狭窄、湿疹、高血压、血小板聚集、胃肠道出血、子宫内膜异位症、经前期综合征、肌痛脑脊髓炎、病毒感染后慢性疲劳或眼部疾病。本发明还提供本发明的细胞、本发明的去饱和酶或延长酶、本发明的多核苷酸、本发明的dna构建体、本发明的载体、本发明的转基因非人类生物体、本发明的种子、本发明的油或本发明的脂肪酸和/或本发明的饲料在制备用于治疗或预防可能从pufa获益的病症的药物中的用途。本发明人已经令人惊异地发现,沉默阻抑物可优先地表达于植物贮藏器官以增强转基因在植物中的表达水平,而不明显影响植物的发育。因此,本发明提供植物细胞,其包含:i)编码沉默阻抑物的第一外源性多核苷酸,其可操纵地连接于植物贮藏器官特异性启动子,和ii)编码rna分子的第二外源性多核苷酸,其可操纵地连接于在植物贮藏器官中指导基因转录的启动子。优选地,所述植物贮藏器官特异性启动子是种子特异性启动子,例如子叶特异性启动子或胚乳特异性启动子。在一个实施方式中,所述沉默阻抑物是病毒阻抑蛋白,例如,但不限于,p1、p19、v2、p38、p15、pe-po和rpv-p0。典型地,如果植物组成型地表达病毒阻抑蛋白,则植物具有异常的表型,但如果沉默阻抑物特异性表达于贮藏器官,则植物具有正常的表型。此类病毒阻抑蛋白的实例包括,但不限于,p1、p19和p15。在进一步的实施方式中,所述病毒阻抑蛋白降低microrna的积累和/或降低microrna的导向型裂解。rna分子本身可以是功能性的,例如,但不限于,反义多核苷酸、催化性多核苷酸、dsrna和/或microrna。或者,所述rna分子可编码具有所需功能的多肽,例如,但不限于,参与脂肪酸合成或修饰的酶、种子贮藏蛋白(例如,谷类麦谷蛋白或麦醇溶蛋白)、参与碳水化合物的合成或修饰、次级代谢的酶或药物。药用蛋白质的实例包括,但不限于,抗体以及抗体相关分子及其片段、抗原性多肽,它们能够例如产生针对癌症、感染因子的免疫保护,细胞因子例如,粒细胞巨噬细胞集落刺激因子、干扰素-α、人血清白蛋白和促红细胞生成素。在进一步的实施方式中,所述细胞包含至少一种、至少两种、至少三种、至少四种或至少五种或更多额外的不同的外源性多核苷酸,每一外源性多核苷酸编码rna分子并可操纵地连接于在贮藏器官中指导基因转录的启动子。每一外源性多核苷酸可以可操纵地连接于同一启动子、不同的启动子或它们的组合。在一个实施方式中,所述外源性多核苷酸是dna。在进一步的实施方式中,所述细胞存在于植物贮藏器官例如种子中。在优选的实施方式中,与缺乏所述第一外源性多核苷酸的同源细胞相比,所述rna分子以升高的水平存在。优选地,所述水平增加至少10%、至少20%、更优选地至少30%。在另一实施方式中,与缺乏所述第一外源性多核苷酸的同源细胞相比,由至少所述额外的外源性多核苷酸编码的至少一种rna分子以升高的水平存在。本发明还提供转基因植物,其包含以上方面的细胞。在一个实施方式中,所述植物的每一细胞均是以上方面所定义的细胞。在特别优选的实施方式中,与缺乏所述细胞的植物相比,所述植物具有正常的表型。在另一方面,本发明提供植物贮藏器官,其包含以上方面的细胞和/或其得自前面定义的转基因植物。在一个实施方式中,所述植物贮藏器官是种子。在另一方面,本发明提供获得在贮藏器官中具有升高水平的rna分子的表型正常植物的方法,包括a)向植物细胞中引入i)编码沉默阻抑物的第一外源性多核苷酸,其可操纵地连接于植物贮藏器官特异性启动子,和ii)编码rna分子的第二外源性多核苷酸,其可操纵地连接于在植物贮藏器官中指导基因转录的启动子,b)从步骤a)的细胞再生转化植物,c)生长所述转化植物直至其产生贮藏器官,d)确定所述贮藏器官中的rna分子水平,和e)选择表型正常的植物,但其中所述贮藏器官内的rna分子的水平与缺乏所述第一外源性多核苷酸的相应的贮藏器官相比是升高的。在又一方面,本发明提供获得在贮藏器官中rna分子稳定表达的表型正常植物的方法,包括a)向植物细胞中引入i)编码沉默阻抑物的第一外源性多核苷酸,其可操纵地连接于植物贮藏器官特异性启动子,和ii)编码rna分子的第二外源性多核苷酸,其可操纵地连接于在植物贮藏器官中指导基因转录的启动子,b)从步骤a)的细胞再生转化植物,c)从步骤b)的植物产生包含贮藏器官的第三代子代植物,和d)选择第三代子代植物,其中所述贮藏器官中的所述rna分子的水平是前代植物的贮藏器官中水平的至少90%。优选地,以上方面的外源性多核苷酸稳定整合到所述细胞的基因组中。在又一方面,本发明提供在转基因植物的贮藏器官中稳定表达rna分子的方法,包括i)表达编码沉默阻抑物的、可操纵地连接于植物贮藏器官特异性启动子的第一外源性多核苷酸,和ii)表达编码rna分子的、可操纵地连接于在植物贮藏器官中指导基因转录的启动子的第二外源性多核苷酸,其中所述转基因植物是从以所述外源性多核苷酸转化的母本植物获得的至少第三代子代植物,且其中所述植物的贮藏器官中的所述rna分子的水平是前代植物的贮藏器官中水平的至少90%。在一个实施方式中,所述植物生长在田间。显然,本发明一个方面的优选的特征也适用于本发明的许多其他方面。在本说明书中,“包含”一词或其变化形式例如“包括”应理解为旨在纳入所提及的元件、整数或步骤或是元件、整数或步骤的组,但不排除任何其他的元件、整数或步骤或是元件、整数或步骤的组。下面通过以下非限制性实施例并结合附图进一步阐述本发明。附图说明图1.需氧dha生物合成途径。图2.在pc、coa库和tag库之间转移脂肪酸的各种酰基交换酶(acylexchangeenzymes)。改编自singh等(2005)。图3.微胞藻cs-0170δ6-延长酶和相关基因之间的多重比对。aav33630,c20-多不饱和脂肪酸延长酶[巴夫藻ccmp459];aay15135,延长酶1[盐生巴夫藻];abr67690,c20延长酶[绿色巴夫藻(pavlovaviridis)];aav67797,多不饱和脂肪酸延长酶1[ostreococcustauri];cal55414,多不饱和脂肪酸延长酶2(iss)[ostreococcustauri];xp_001419791,推定的蛋白质[ostreococcuslucimarinuscce9901];miccs0170-d6e,微胞藻cs-0170δ6-延长酶(本发明);aav67800,多不饱和脂肪酸延长酶2[假微型海链藻(thalassiosirapseudonana)];xp_001416454,推定的蛋白质[ostreococcuslucimarinuscce9901];abc18313,多不饱和脂肪酸延长酶1[破囊壶菌(thraustochytriumsp.)fjn-10];aav67799,多不饱和脂肪酸延长酶1[假微型海链藻];aaw70157,δ-6-延长酶[三角褐指藻];abc18314,多不饱和脂肪酸延长酶2[破囊壶菌fjn-10];cad58540,未命名蛋白质产物[球等鞭金藻]。图4.塔胞藻cs-0140δ6-延长酶和相关基因之间的多重比对。aal84174,多不饱和脂肪酸特异性延长酶1[小立碗藓];aat85662,多不饱和脂肪酸延长酶[地钱(marchantiapolymorpha)];aav67797,多不饱和脂肪酸延长酶1[ostreococcustauri];abo94747,推定的蛋白质[ostreococcuslucimarinuscce9901];pyrco-d6e,塔胞藻cs-0140δ6-延长酶(本发明);abc18313,多不饱和脂肪酸延长酶1[破囊壶菌fjn-10];baf97073,多不饱和脂肪酸延长酶[高山被孢霉];xp_001567488,长链多不饱和脂肪酸延长酶样蛋白[巴西利什曼原虫(leishmaniabraziliensis)mhom/br/75/m2904];bae71129,δ5-延长酶[地钱];xp_001779105;推定的蛋白质[小立碗藓]。图5.塔胞藻cs-0140δ5-延长酶和相关基因之间的多重比对。aai52204,elovl4蛋白[鮐];cag01780,未命名蛋白质产物[墨綠凹鼻魨(tetraodonnigroviridis)];aav67800,多不饱和脂肪酸延长酶2[假微型海链藻];aav33630,c20-多不饱和脂肪酸延长酶[巴夫藻ccmp459];abr67690,c20延长酶[绿色巴夫藻];aay15135,延长酶1[盐生巴夫藻];aav67798,多不饱和脂肪酸延长酶2[ostreococcustauri];abo98084,推定的蛋白质[ostreococcuslucimarinuscce9901];pyrco-d5e,塔胞藻cs-0140δ5-延长酶(本发明)。图6.微胞藻ccmp1545δ6-去饱和酶和相关基因之间的多重比对。aam09687,δ5-脂肪酸去饱和酶[破囊壶菌atcc21685];aav33631,δ4-去饱和酶[球等鞭金藻];aaw70159,δ6-去饱和酶[ostreococcustauri];abo99366,推定的蛋白质[ostreococcuslucimarinuscce9901];mic-d6d,微胞藻ccmp1545δ6-去饱和酶(本发明);abf58685,δ5-去饱和酶[perkinsusmarinus];abl96295,δ5-去饱和酶[盐生巴夫藻]。图7.ostreococcuslucimarinusδ6-去饱和酶和相关基因之间的多重比对。aam09687,δ5-脂肪酸去饱和酶[破囊壶菌atcc21685];aar27297,δ6-去饱和酶[淀粉丝菌(amylomycesrouxii)];aas93682,δ6-脂肪酸去饱和酶[稻根霉菌(rhizopusoryzae)];aav33631,δ4-去饱和酶[球等鞭金藻];aaw70159,δ6-去饱和酶[ostreococcustauri];ostlu-d6d,ostreococcuslucimarinusδ6-去饱和酶(本发明);abf58685,δ5-去饱和酶[perkinsusmarinus];abl96295,δ5-去饱和酶[盐生巴夫藻];edq92231,推定的蛋白质[领鞭毛虫(monosigabrevicollis)mx1];cam41728,脂肪酸去饱和酶,推定的[巴西利什曼原虫];cam65683,脂肪酸去饱和酶,推定的[婴儿利什曼原虫(leishmaniainfantum)]。图8.塔胞藻cs-0140δ5-去饱和酶和相关基因之间的多重比对。aam09687,δ5-脂肪酸去饱和酶[破囊壶菌atcc21685];pyrco-d5d,塔胞藻cs-0140δ5-去饱和酶(本发明);aat85661,δ6-脂肪酸去饱和酶[地钱];aax14505,δ6-脂肪酸去饱和酶[假微型海链藻];abp49078,δ6-脂肪酸去饱和酶[三角褐指藻];aaw70159,δ6-去饱和酶[ostreococcustauri];xp_001421073,推定的蛋白质[ostreococcuslucimarinuscce9901];abf58685,δ5-去饱和酶[perkinsusmarinus];caj07076,脂肪酸去饱和酶,推定的[硕大利什曼原虫(leishmaniamajor)];abl96295,δ5-去饱和酶[盐生巴夫藻];edq92231,推定的蛋白质[领鞭毛虫mx1]。图9.ostreococcustauri(ot)(seqidno:30)、ostreococcuslucimarinus(ol)(seqidno:10)和微胞藻(m)ccmp1545(seqidno:8)的δ6-去饱和酶蛋白序列的多重比对。图10.显示各种去饱和酶之间的关系的系统发育树。pavsa-d5d=盐生巴夫藻δ5-去饱和酶(abl96295);thr21685-d5d=破囊壶菌atcc21685δ5-去饱和酶(aam09687);mic1545-d6d=微胞藻ccmp1545δ6-去饱和酶(本发明);ostta-d6d=ostreococcustauriδ6-去饱和酶(aaw70159);ostlu-d6d=ostreococcuslucimarinusδ6-去饱和酶(本发明);galga-d6d=原鸡(gallusgallus)δ6-去饱和酶(xp_421053);homsa-d6d=智人δ6-去饱和酶(aag23121);musmu-d6d=小家鼠δ6-去饱和酶(np_062673);danre-d5/6d=鮐δ5-/δ6-去饱和酶(aag25710);spaau-d6d=金黄色石斑鱼(sparusaurata)推定的δ6-去饱和酶(aal17639);scoma-d6d=大菱鲆(scophthalmusmaximus)δ6-去饱和酶(aas49163);oncmy-d6d=虹鱒δ6-去饱和酶(aak26745);salsa-d5d=salmosalarδ5-去饱和酶(aal82631);prifa-d6d=primulafarinosaδ6-去饱和酶(aap23034);euggr-d6d=小眼虫δ6-去饱和酶;borof-d6d=琉璃苣(boragoofficianalis)δ6-去饱和酶(aac49700);caeel-d6d=秀丽隐杆线虫δ6-去饱和酶(aac15586);rhior-d6d=稻根霉菌δ6-去饱和酶(aas93682);moral-d6d=高山被孢霉δ6-去饱和酶(aaf08685);moris-d6d=深黄被孢霉(mortierellaisabellina)δ6-去饱和酶(aal73948);marpo-d6d=地钱δ6-去饱和酶(aat85661);cerpu-d6d=角齿藓(ceratodonpurpureus)δ6-去饱和酶(cab94993);phatr-d6d=三角褐指藻δ6-去饱和酶(aal92563);thaps-d6d=假微型海链藻δ6-去饱和酶(aax14505);pavsa-d8d=盐生巴夫藻δ8-去饱和酶(abl96296);phatr-d5d=三角褐指藻δ5-去饱和酶(aal92562);marpo-d5d=地钱δ5-去饱和酶(aat85663);moral-d5d=高山被孢霉δ5-去饱和酶(aar28035);dicdi-d5d=盘基网柄菌(dictyosteliumdiscoideum)δ5-去饱和酶(baa37090)。图11.来自以linp-mic1545-d6d-lint构建体转化的t2拟南芥种子的gc结果。显示了单个事件1-19的sda和gla水平,各柱状图上方显示了ω3到ω6的转换效率的比。细小微胞藻δ6-去饱和酶对ω3底物显示出明显的偏向性。图12.构成渗入到本氏烟草(nicotianabenthamiana)中的epa途径的酶的转换效率。epa途径含有δ6-去饱和酶(车前叶蓝蓟(echiumplantagineum)δ6-去饱和酶、ostreococcustauriδ6-去饱和酶或细小微胞藻(micromonaspusilla)δ6-去饱和酶)、pyramimonascordataδ6-延长酶和盐生巴夫藻δ5-去饱和酶。a显示各途径的ω3库转换效率;b.含有车前叶蓝蓟途径、细小微胞藻途径和含有这两种去饱和酶的途径之间的直接比较;c.含有o.tauri途径、细小微胞藻途径和含有这两种酰基-coa去饱和酶的途径之间的直接比较。图13.gc和gc-ms确认通过瞬时表达的微胞藻rcc299ω3去饱和酶在本氏烟草中产生epa。图14.二元载体pjp101acq的图显示了该二元载体的关键特征,包括启动子方向、tmv前导序列定位、间隔区域定位和基因插入的独特克隆位点。nost=nos终止子,fp1=截短的napin终止子,linint=linin终止子。图15.二元载体pjp107的图。图16.确定在基于叶的分析法中实现近最大基因活性所需的土壤杆菌的浓度。以不同培养密度的含有二元表达构建体igδ9elo的土壤杆菌agl1进行渗入后的本氏烟草中的球等鞭金藻δ9-延长酶(igδ9elo)活性。将共渗入的p19设定在od600nm为0.4的浓度。y-轴显示的是产生eda和etra的igδ9elo活性的总和。图17.采用在叶子中瞬时表达或稳定表达的方法比较lc-pufa途径的转基因表达。转换效率基于总脂肪酸特征谱。a结果来自(qi等,2004);b结果来自(robert等,2005)。图18.本氏烟草中的代谢适应。a.是对金枪鱼油中产生的脂肪酸甲基酯(fame)的气相色谱(gc)示踪,其仅含有少量的epa,但含有大量的dha。b.和c.是对来自以单基因camv35s二元构建体(含有细小微胞藻δ6-去饱和酶、p.cordataδ6-延长酶、盐生巴夫藻δ5-去饱和酶、p.cordataδ5-延长酶(b.)或盐生巴夫藻δ5-延长酶(c.)和盐生巴夫藻δ4-去饱和酶)瞬时转化的本氏烟草叶组织中的tag成分的fame的gc示踪。epa在使用盐生巴夫藻δ5-延长酶的样品中的累积证实了如何可以通过仔细选择途径中的单个基因而使得代谢途径发生适应。图19.载体pjp3075中包含转基因的区域的图。图20.载体pjp3059中包含转基因的区域的图。图21.载体pjp3060中包含转基因的区域的图。图22.载体pjp3115中包含转基因的区域的图。图23.载体pjp3116中包含转基因的区域的图。序列表要点seqidno:1-编码微胞藻cs-0170δ6-延长酶的可读框。seqidno:2-微胞藻cs-0170δ6-延长酶。seqidno:3-编码塔胞藻cs-0140δ6-延长酶/δ9-延长酶的可读框。seqidno:4-塔胞藻cs-0140δ6-延长酶/δ9-延长酶。seqidno:5-编码塔胞藻cs-0140δ5-延长酶的可读框。seqidno:6-塔胞藻cs-0140δ5-延长酶。seqidno:7-编码微胞藻ccmp1545δ6-去饱和酶/δ8-去饱和酶的可读框。seqidno:8-微胞藻ccmp1545δ6-去饱和酶/δ8-去饱和酶。seqidno:9-编码ostreococcuslucimarinusδ6-去饱和酶的可读框。seqidno:10-ostreococcuslucimarinusδ6-去饱和酶。seqidno:11-用于在植物中表达ostreococcuslucimarinusδ6-去饱和酶的经密码子优化的可读框。seqidno:12-编码塔胞藻cs-0140δ5-去饱和酶的可读框。seqidno:13-塔胞藻cs-0140δ5-去饱和酶。seqidno:14-编码微胞藻cs-0170ω3-去饱和酶的部分可读框。seqidno:15–部分微胞藻cs-0170ω3-去饱和酶。seqidno:16-编码微胞藻rcc299ω3-去饱和酶的可读框。seqidno:17-微胞藻rcc299ω3-去饱和酶。seqidno:18-用于在植物中表达微胞藻rcc299ω3-去饱和酶的经密码子优化的可读框。seqidno:19-编码微胞藻ccmp1545ω3-去饱和酶的可读框。seqidno:20-微胞藻ccmp1545ω3-去饱和酶。seqidno:21-编码球等鞭金藻δ9-延长酶的可读框。seqidno:22-球等鞭金藻δ9-延长酶。seqidno:23-编码盐生巴夫藻δ8-去饱和酶的可读框。seqidno:24-盐生巴夫藻δ8-去饱和酶。seqidno:25-编码盐生巴夫藻δ5-去饱和酶的可读框。seqidno:26-盐生巴夫藻δ5-去饱和酶。seqidno:27-编码emilianiahuxleyiccmp1516δ9延长酶的可读框。seqidno:28-emilianiahuxleyiccmp1516δ9延长酶。seqidno:29-用于在植物中表达emilianiahuxleyiδ9延长酶的经密码子优化的可读框。seqidno:30-ostreococcustauriδ6-去饱和酶。seqidno:31-延长酶共有结构域1。seqidno:32-延长酶共有结构域2。seqidno:33-延长酶共有结构域3。seqidno:34-延长酶共有结构域4。seqidno:35-延长酶共有结构域5。seqidno:36-延长酶共有结构域6。seqidno:37-去饱和酶共有结构域1。seqidno:38-去饱和酶共有结构域2。seqidno:39-去饱和酶共有结构域3。seqidno:40-去饱和酶共有结构域4。seqidno:41-71和78-92-寡核苷酸引物。seqidno:72-编码盐生巴夫藻δ4-去饱和酶的可读框。seqidno:73-盐生巴夫藻δ4-去饱和酶。seqidno:74-编码拟南芥二酰基甘油酰基转移酶1的可读框。seqidno:75-拟南芥二酰基甘油酰基转移酶1。seqidno:76-延长酶共有结构域7。seqidno:77-延长酶共有结构域8。seqidno:93-编码pavlovapinguisδ9-延长酶的可读框。seqidno:94-pavlovapinguisδ9-延长酶。seqidno:95-编码盐生巴夫藻δ9-延长酶的可读框。seqidno:96-盐生巴夫藻δ9-延长酶。seqidno:97–p19病毒阻抑物。seqidno:98–v2病毒阻抑物。seqidno:99–p38病毒阻抑物。seqidno:100–pe-p0病毒阻抑物。seqidno:101–rpv-p0病毒阻抑物。seqidno:102–编码p19病毒阻抑物的可读框。seqidno:103–编码v2病毒阻抑物的可读框。seqidno:104–编码p38病毒阻抑物的可读框。seqidno:105–编码pe-p0病毒阻抑物的可读框。seqidno:106–编码rpv-p0病毒阻抑物的可读框。seqidno:107–密码子优化的编码微胞藻ccmp1545二酰基甘油酰基转移酶2的可读框。seqidno:108-微胞藻ccmp1545二酰基甘油酰基转移酶2。seqidno:109-124–转移核酸边界序列。seqidno:125-用于在植物中表达微胞藻ccmp1545δ6去饱和酶/δ8去饱和酶的经密码子优化的可读框。seqidno:126-用于在植物中表达塔胞藻cs-0140δ6延长酶/δ9延长酶的经密码子优化的可读框(在3’端被截短并编码功能性延长酶)。seqidno:127-用于在植物中表达盐生巴夫藻δ5去饱和酶的经密码子优化的可读框。seqidno:128-用于在植物中表达塔胞藻cs-0140δ5延长酶的经密码子优化的可读框。seqidno:129-用于在植物中表达盐生巴夫藻δ4去饱和酶的经密码子优化的可读框。发明详述通用技术和定义除非另有说明,否则本说明书中所使用的科技术语均具有与本领域(例如细胞培养、分子遗传学、脂肪酸合成、转基因植物、蛋白质化学和生物化学等领域)普通技术人员通常所理解的含义相同的含义。除非另有说明,本发明所采用的重组蛋白质、细胞培养和免疫学技术均为本领域人员所熟知的标准技术。此类技术在文献中有充分的描述和解释,例如参见j.perbal,apracticalguidetomolecularcloning,johnwileyandsons(1984);j.sambrook等,molecularcloning:alaboratorymanual,coldspringharbourlaboratorypress(1989);t.a.brown(编辑),essentialmolecularbiology:apracticalapproach,volumes1and2,irlpress(1991);d.m.gloverandb.d.hames(编辑),dnacloning:apracticalapproach,volumes1-4,irlpress(1995and1996);和f.m.ausubel等(编辑),currentprotocolsinmolecularbiology,greenepub.associatesandwiley-interscience(1988,包括迄今所有的更新版);edharlowanddavidlane(编辑)antibodies:alaboratorymanual,coldspringharbourlaboratory,(1988);和j.e.coligan等(编辑)currentprotocolsinimmunology,johnwiley&sons(包括迄今所有的更新版),通过引用将上述文献并入本申请。选定的定义在本文中,术语"脂肪酸"指的是羧酸(有机酸),通常具有长的脂肪族尾部,可以是饱和的或不饱和的。典型地,脂肪酸具有碳碳键连接的链,其长度为至少8个碳原子,更优选地长度为至少12个碳。绝大多数天然存在的脂肪酸具有偶数个碳原子,因为它们的生物合成涉及具有2个碳原子的乙酸酯。脂肪酸可以是游离状态的(非酯化的)或者是酯化的形式,例如作为甘油三酯、甘油二酯、单酰基甘油酯的部分、酰基辅酶a(硫酯)结合型或其他结合形式。脂肪酸可酯化为磷脂,例如磷脂酰胆碱、磷脂酰乙醇胺、磷脂酰丝氨酸、磷脂酰甘油、磷脂酰肌醇或二磷脂酰甘油的形式。"饱和脂肪酸"在其链上不含有任何双键或其他官能团。术语"饱和"指的是氢,其中所有的碳(除羧基[-cooh]之外)均含有尽可能多的氢。换言之,ω端含有3个氢(ch3-)而链内的各个碳含有2个氢(-ch2-)。"不饱和脂肪酸"与饱和脂肪酸形式相似,不同之处为沿着链存在一或多个烯烃官能团,每一烯烃将链中的单键的"-ch2-ch2-"部分置换为双键的"-ch=ch-"部分(即,与另一碳双键连接的碳)。链中连接于该双键任一侧的2个相邻碳原子可以顺式或反式构型存在。在本文中,术语“单不饱和脂肪酸”指的是这样的脂肪酸,其碳链中包含至少12个碳原子且链中仅有一个烯烃基(碳碳双键)。在本文中,术语“多不饱和脂肪酸”或"pufa"指的是这样的脂肪酸,其碳链中包含至少12个碳原子和至少2个烯烃基(碳碳双键)。在本文中,术语"长链多不饱和脂肪酸”或"lc-pufa"指的是这样的脂肪酸,其碳链中包含至少20个碳原子和至少2个碳碳双键,因此也包括vlc-pufa。在本文中,术语"极长链多不饱和脂肪酸"和"vlc-pufa"指的是这样的脂肪酸,其碳链中包含至少22个碳原子和至少3个碳碳双键。通常,脂肪酸碳链的碳原子数涉及的是非分支碳链。如果碳链是分支的,则碳原子数不包括侧基碳原子。在一个实施方式中,所述长链多不饱和脂肪酸ω3脂肪酸,即脂肪酸自甲基端开始的第三个碳碳键发生去饱和(碳碳双键)。在另一个实施方式中,所述长链多不饱和脂肪酸ω6脂肪酸,即脂肪酸自甲基端开始的第六个碳碳键发生去饱和(碳碳双键)。在进一步的实施方式中,所述长链多不饱和脂肪酸选自:花生四烯酸(ara,20:4δ5,8,11,14;ω6)、二十碳四烯酸(eta,20:4δ8,11,14,17,ω3)、二十碳五烯酸(epa,20:5δ5,8,11,14,17;ω3)、二十二碳五烯酸(dpa,22:5δ7,10,13,16,19,ω3)、或二十二碳六烯酸(dha,22:6δ4,7,10,13,16,19,ω3)。lc-pufa也可以是双高-γ-亚油酸(dgla)或二十碳三烯酸(etra,20:3δ11,14,17,ω3)。显然,本发明所产生的lc-pufa可以是任何或全部上述各项的混合物,并可包括其他lc-pufa或任何这些lc-pufa的衍生物。在优选的实施方式中,ω3脂肪酸是epa、dpa、和/或dha,优选地,epa和/或dpa,或优选地,dpa和/或dha。此外,在本文中,术语"长链多不饱和脂肪酸"和"极长链多不饱和脂肪酸"指的是游离状态的(非酯化的)或者是酯化的形式的脂肪酸,例如作为甘油三酯、甘油二酯、单酰基甘油酯的部分、酰基辅酶a(硫酯)结合型或其他结合形式。脂肪酸可酯化为磷脂,例如磷脂酰胆碱、磷脂酰乙醇胺、磷脂酰丝氨酸、磷脂酰甘油、磷脂酰肌醇或二磷脂酰甘油的形式。因此,lc-pufa可以混合物的形式存在于细胞内或提取自细胞、组织或生物体的纯化的油或脂质中。在优选的实施方式中,本发明提供的油包含至少75%或85%的三酰基甘油,其余部分以其他形式的脂质(例如本文中所提到的那些)存在,至少所述的三酰基甘油包含lc-pufa。所述油可被进一步纯化或处理,例如以强碱水解去除游离脂肪酸或被分级、蒸馏等等。可用于本发明的去饱和酶、延长酶和酰基转移酶蛋白及其编码基因是本领域已知的或是其同源物或衍生物。表1列出了这些基因的实例及其编码的蛋白质的大小。已被发现参与lc-pufa生物合成的去饱和酶均属于所谓的“前端”去饱和酶。在本文中,术语“前端去饱和酶”指的是许多这样的一类酶,这些酶在脂质的酰基链的羧基与已经存在的不饱和部分之间引入双键,它们在结构上的特征为存在n-端细胞色素b5结构域以及典型的脂肪酸去饱和酶结构域,后者包括三个高度保守性的组氨酸盒(napier等,1997)。可如下测试可用于本发明的任何延长酶或去饱和酶的活性:在细胞例如酵母细胞或植物细胞中表达编码所述酶的基因,并确定所述细胞产生lc-pufa的能力与具有可比性的不表达所述酶的细胞相比是否升高。在一个实施方式中,可用于本发明的去饱和酶和/或延长酶可纯化自微藻类。尽管某些酶在本文中被具体描述为是"双功能性的",但不使用该词并不一定暗示一种具体的酶除具体提及的活性以外便不具有其他活性。去饱和酶类在本文中,术语“去饱和酶”指的是这样的酶,其能够在脂肪酸底物的酰基中引入碳碳双键,所述脂肪酸底物典型地是酯化的形式,例如是脂肪酸辅酶a酯。酰基可与磷脂例如磷脂酰胆碱(pc)发生酯化,或与酰基载体蛋白(acp)发生酯化,或在优选的实施方式中与辅酶a发生酯化。相应地,去饱和酶通常可分为3类。在一个实施方式中,所述去饱和酶是前端去饱和酶。在本申请中,术语"δ4去饱和酶"指的是这样的蛋白质,其所进行的去饱和酶反应导致在脂肪酸底物的自羧基端起的第4位处引入碳碳双键。"δ4去饱和酶"至少能够将dpa转换为dha。在除哺乳动物之外的生物体中,所述从dpa产生dha的去饱和步骤由δ4去饱和酶催化,已经从淡水原生生物小眼虫和海洋生物破囊壶菌中分离了编码此酶的基因(qiu等,2001;meyer等,2003)。在一个实施方式中,所述δ4去饱和酶包含seqidno:73所示的氨基酸序列、其生物学活性片段、或与seqidno:73具有至少80%相同性的氨基酸序列。在本申请中,"δ5去饱和酶"指的是这样的蛋白质,其所进行的去饱和酶反应导致在脂肪酸底物的自羧基端起的第5位处引入碳碳双键。表1列出了δ5去饱和酶的实例。在一个实施方式中,所述δ5去饱和酶包含seqidno:26所示的氨基酸序列、其生物学活性片段、或与seqidno:26具有至少80%相同性的氨基酸序列。在另一个实施方式中,所述δ5去饱和酶包含seqidno:13所示的氨基酸序列、其生物学活性片段、或与seqidno:13具有至少53%相同性的氨基酸序列。在本申请中,"δ6去饱和酶"指的是这样的蛋白质,其所进行的去饱和酶反应导致在脂肪酸底物的自羧基端起的第6位处引入碳碳双键。表1列出了δ6去饱和酶的实例。在一个实施方式中,所述δ6去饱和酶的特征为还具有至少两种、优选地全部三种以下特性,优选地在植物细胞中是这样:i)对α-亚麻酸(ala,18:3δ9,12,15,ω3)脂肪酸底物的δ6去饱和酶活性高于对亚油酸(la,18:2δ9,12,ω6)的活性;ii)对作为脂肪酸底物的ala-coa的δ6去饱和酶活性高于对作为脂肪酸底物的连接至pc的sn-2位置的ala的活性;和iii)对etra具有δ8去饱和酶活性。在另一实施方式,所述δ6去饱和酶对ω3底物的活性高于对相应的ω6底物的活性,并对ala具有活性,当由重组细胞中的外源性多核苷酸表达时以至少5%,更优选地至少7.5%,或最优选地至少10%的效率产生octadecatetraenoicacid(十八碳四烯酸,sda,18:4δ6,9,12,15,ω3),当在酵母细胞中表达时该效率是至少35%。在一个实施方式中,所述δ6去饱和酶对作为脂肪酸底物的ala的活性高于对la的活性,例如,对ala的δ6去饱和酶活性是对la的至少两倍。在另一个实施方式中,所述δ6去饱和酶对作为脂肪酸底物的ala-coa的活性高于对作为脂肪酸底物的连接至pc的sn-2位置的ala的活性,例如,对前者的δ6去饱和酶活性是对后者的至少5倍或至少10倍。在一个实施方式中,所述δ6去饱和酶对eta不具有可检测到的δ5去饱和酶活性。在另一个实施方式中,所述δ6去饱和酶包含seqidno:10所示的氨基酸序列、其生物学活性片段、或与seqidno:10具有至少77%相同性的氨基酸序列。在另一个实施方式中,所述δ6去饱和酶包含seqidno:8所示的氨基酸序列、其生物学活性片段、或与seqidno:8具有至少67%相同性的氨基酸序列。所述δ6去饱和酶还可具有δ8去饱和酶活性。在本申请中,"δ8去饱和酶"指的是这样的蛋白质,其所进行的去饱和酶反应导致在脂肪酸底物的自羧基端起的第8位处引入碳碳双键。所述δ8去饱和酶至少能够将etra转换为eta。表1列出了δ8去饱和酶的实例。在一个实施方式中,所述δ8去饱和酶包含seqidno:24所示的氨基酸序列、其生物学活性片段、或与seqidno:24具有至少80%相同性的氨基酸序列。在本申请中,"ω3去饱和酶"指的是这样的蛋白质,其所进行的去饱和酶反应导致在脂肪酸底物的自甲基端起的第3位处引入碳碳双键。ω3去饱和酶的实例包括pereira等(2004a)、horiguchi等(1998)、berberich等(1998)和spychalla等(1997)所描述的那些。在一个实施方式中,所述ω3去饱和酶至少能够进行将ara转换为epa、将dgla转换为eta、将γ-亚麻酸(gla,18:3δ6,9,12,ω6)转换为sda中的一种转换、将ara转换为epa和将dgla转换为eta这两种转换、将ara转换为epa和将gla转换为sda这两种转换、或全部这三种转换。在一个实施方式中,所述ω3去饱和酶对具有至少三个碳碳双键的c20脂肪酸、优选地ara,具有δ17去饱和酶活性。在另一个实施方式中,所述ω3去饱和酶对具有三个碳碳双键的c18脂肪酸、优选地gla,具有δ15去饱和酶活性。在本申请中,"δ15去饱和酶"指的是这样的蛋白质,其所进行的去饱和酶反应导致在脂肪酸底物的自羧基端起的第15位处引入碳碳双键。在本申请中,"δ17去饱和酶"指的是这样的蛋白质,其所进行的去饱和酶反应导致在脂肪酸底物的自羧基端起的第17位处引入碳碳双键。在另一个实施方式中,所述ω3去饱和酶对酰基辅酶a底物(例如,ara-coa)的活性高于对相应的酰基-pc底物的活性。在本申请中,“相应的酰基-pc底物”指的是在磷酯酰胆碱(pc)的sn-2位置发生酯化的脂肪酸,其中所述脂肪酸与所述酰基辅酶a底物中的脂肪酸相同。在一个实施方式中,所述活性是至少两倍高。在进一步的实施方式中,所述ω3去饱和酶包含seqidno:15、17或20所示的氨基酸序列、其生物学活性片段、或与seqidno:15具有至少35%相同性的氨基酸序列、与seqidno:17具有至少60%相同性的氨基酸序列和/或与seqidno:20具有至少60%相同性的氨基酸序列。在另一实施方式中,用于本发明的去饱和酶对酰基辅酶a底物的活性高于对相应的酰基-pc底物的活性。如上所述,“相应的酰基-pc底物”指的是在磷酯酰胆碱(pc)的sn-2位置发生酯化的脂肪酸,其中所述脂肪酸与所述酰基辅酶a底物中的脂肪酸相同。在一个实施方式中,所述活性是至少两倍高。在一个实施方式中,所述去饱和酶是δ5或δ6去饱和酶,表2给出了它们的非限制性实例。表2.对酰基辅酶a底物的活性高于对相应的酰基-pc底物的活性的去饱和酶延长酶类生化证据提示脂肪酸延长作用包括4个步骤:缩合、还原、脱水以及第二步还原。就本发明而言,"延长酶"指的是这样的多肽,其在合适的生理学条件下,在存在延长作用复合体的其他成员的情况下,催化缩合步骤。已经发现,相应的酰基链的延长仅需要在细胞中异源或同源表达延长作用蛋白复合体的缩合组份("延长酶")。因此,所引入的延长酶能够成功募集转基因宿主的还原和脱水活性,以进行成功的酰基延长作用。就脂肪酸底物的链长及其去饱和程度而言,认为延长反应的特异性在于所述缩合组份。该组份也被认为在延长反应中具有限速作用。在本申请中,"δ5延长酶"至少能够将epa转换为dpa。δ5延长酶的实例包括wo2005/103253中所描述的那些。在一个实施方式中,所述δ5延长酶对epa具有活性并以至少60%,更优选地至少65%,更优选地至少70%或最优选地至少75%的效率产生dpa。在进一步的实施方式中,所述δ5延长酶包含seqidno:6所示的氨基酸序列、其生物学活性片段、或与seqidno:6具有至少47%相同性的氨基酸序列。在本申请中,"δ6延长酶"至少能够将sda转换为eta。δ6延长酶的实例包括表1所列的那些。在一个实施方式中,所述延长酶包含seqidno:4所示的氨基酸序列、其生物学活性片段、或与seqidno:4具有至少55%相同性的氨基酸序列。在本申请中,"δ9延长酶"至少能够将ala转换为etra。δ9延长酶的实例包括表1所列的那些。在一个实施方式中,所述δ9延长酶包含seqidno:22所示的氨基酸序列、其生物学活性片段、或与seqidno:22具有至少80%相同性的氨基酸序列。在另一个实施方式中,所述δ9延长酶包含seqidno:28所示的氨基酸序列、其生物学活性片段、或与seqidno:28具有至少81%相同性的氨基酸序列。在另一个实施方式中,所述δ9延长酶包含seqidno:94所示的氨基酸序列、其生物学活性片段、或与seqidno:94具有至少50%相同性的氨基酸序列。在另一个实施方式中,所述δ9延长酶包含seqidno:96所示的氨基酸序列、其生物学活性片段、或与seqidno:96具有至少50%相同性的氨基酸序列。在进一步的实施方式中,所述δ9延长酶包含seqidno:94或seqidno:96所示的氨基酸序列、其生物学活性片段、或与seqidno:94和/或seqidno:96具有至少50%相同性的氨基酸序列,且其中所述延长酶对ω6底物的活性高于对相应的ω3底物的活性。在本文中,术语“对ω6底物的活性高于对相应的ω3底物的活性”指的是所述酶对底物的相对活性不同于ω3去饱和酶的作用。优选地,ω6底物是la而ω3底物是ala。在本申请中,"具有δ6延长酶和δ9延长酶活性的延长酶"至少能够(i)将sda转换为eta和(ii)将ala转换为etra,且其δ6延长酶活性高于δ9延长酶活性。在一个实施方式中,所述延长酶转换sda以产生eta的效率是至少50%,更优选地至少60%,和/或转换ala以产生etra的效率是至少6%,或更优选地至少9%。在另一个实施方式中,所述延长酶的δ6延长酶活性是δ9延长酶活性的至少大约6.5倍。在另一个实施方式中,所述延长酶不具有可检测到的δ5延长酶活性。在另一实施方式中,所述延长酶包含seqidno:4所示的氨基酸序列、其生物学活性片段、或与seqidno:4具有至少55%相同性的氨基酸序列。其他酶在本文中,术语“二酰基甘油酰基转移酶”(ec2.3.1.20;dgat)指的是这样的蛋白质,其将脂肪酰基从酰基-coa转移至二酰基甘油底物,以产生三酰基甘油。因此,术语“二酰基甘油酰基转移酶活性”指的是将酰基-coa转移至二酰基甘油以产生三酰基甘油。已知有三种类型的dgat,分别称为dgat1、dgat2和dgat3。dgat1多肽典型地具有10个跨膜结构域,dgat2典型地具有2个跨膜结构域,而dgat3典型地是可溶性的。dgat1多肽的实例包括由来自以下物种的dgat1基因编码的多肽:烟曲霉(aspergillusfumigatus)(登录号xp_755172)、拟南芥(cab44774)、蓖麻(ricinuscommunis)(aar11479)、油桐(verniciafordii)(abc94472)、咸虾花(vernoniagalamensis)(abv21945,abv21946)、鬼箭羽(euonymusalatus)(aav31083)、秀丽隐杆线虫(aaf82410)、褐家鼠(rattusnorvegicus)(np_445889)、智人(np_036211),以及它们的变体和/或突变体。dgat2多肽的实例包括由来自以下物种的dgat2基因编码的多肽:拟南芥(登录号np_566952)、蓖麻(aay16324)、油桐(abc94474)、拉曼被孢霉(mortierellaramanniana)(aak84179)、智人(q96pd7,q58ht5)、欧洲牛(bostaurus)(q70vd8)、小家鼠(musmusculus)(aak84175)、微胞藻ccmp1545,以及它们的变体和/或突变体。dgat3多肽的实例包括由来自花生(花生,saha,等,2006)的dgat3基因编码的多肽,及其变体和/或突变体。多肽/肽本发明还提供多肽,它们可以是纯化的或重组的。"基本上纯化的多肽"或"纯化的多肽"指的是这样的多肽,其通常已经与在产生所述多肽的细胞中或在天然状态下与之结合的那些脂质、核酸、其他的肽以及其他污染分子分离。优选地,基本上纯化的多肽至少60%、更优选地至少75%、且更优选地至少90%不含在产生所述多肽的细胞中或在天然状态下与之天然结合的其他成分。术语"重组"当涉及多肽时指的是,当所述多肽由细胞或者无细胞表达系统产生时,与其天然状态相比,所述多肽产生的量或速度发生改变。在一个实施方式中,细胞在天然状态下不产生所述多肽。不过,细胞可包含使得产生的所述多肽的量发生改变的非内源性基因。本发明的重组多肽包括细胞、组织、器官或无细胞表达系统中的多肽,即尚未自产生所述多肽的转基因(重组)细胞中的其他组分分离或纯化的多肽,也包括在此类细胞或无细胞系统中产生并随后自至少某些其他成分中纯化出来的多肽。术语"多肽"和"蛋白质"通常可互换使用。一种多肽或一类多肽可通过其氨基酸序列与参比氨基酸序列的相同性(%相同性)程度来定义,或通过其与一种参比氨基酸序列的%相同性大于其与另一参比氨基酸序列的%相同性来定义。多肽与参比氨基酸序列的%相同性通常通过gap分析(needlemanandwunsch,1970;gcg程序)确定,使用的参数为:缺口创建罚分=5,而缺口延伸罚分=0.3。查询序列长度为至少15个氨基酸,且gap分析在至少15个氨基酸的区域比对两个序列。更优选地,查询序列长度为至少50个氨基酸,且gap分析在至少50个氨基酸的区域比对两个序列。更优选地,查询序列长度为至少100个氨基酸,且gap分析在至少100个氨基酸的区域比对两个序列。甚至更优选地,查询序列长度为至少250个氨基酸,且gap分析在至少250个氨基酸的区域比对两个序列。甚至更优选地,gap分析比对两个序列的全长。所述多肽或一类多肽可与所述参比多肽具有相同的酶活性,或具有不同于所述参比多肽的活性,或缺乏所述参比多肽的活性。优选地,所述多肽的酶活性是所述参比多肽的至少10%。在本申请中,"生物学活性"片段是本发明的多肽的一部分,其保持全长参比多肽的所定义的活性,例如具有去饱和酶和/或延长酶活性或其他酶活性。本申请中所使用的生物学活性片段不包括全长多肽。生物学活性片段可以是任何长度,条件是它们保持所定义的活性。优选地,生物学活性片段保持全长蛋白质活性的至少10%。就所定义的多肽或酶而言,可以理解高于上述数值的百分比相同性是优选的实施方式。因此,在适用的情况下,与最小的百分比相同性数值相比,优选地所述多肽/酶包含这样的氨基酸序列,其与相应的指定seqidno具有至少60%、更优选地至少65%、更优选地至少70%、更优选地至少75%、更优选地至少76%、更优选地至少80%、更优选地至少85%、更优选地至少90%、更优选地至少91%、更优选地至少92%、更优选地至少93%、更优选地至少94%、更优选地至少95%、更优选地至少96%、更优选地至少97%、更优选地至少98%、更优选地至少99%、更优选地至少99.1%、更优选地至少99.2%、更优选地至少99.3%、更优选地至少99.4%、更优选地至少99.5%、更优选地至少99.6%、更优选地至少99.7%、更优选地至少99.8%、且甚至更优选地至少99.9%的相同性。在一个实施方式中,本发明的基本上纯化的和/或重组δ6去饱和酶不包含登录号eeh58637.1或xp_001421073.1提供的序列。在另一个实施方式中,本发明的基本上纯化的和/或重组ω3去饱和酶不包含登录号xp_002505536.1提供的序列。在另一个实施方式中,本发明的基本上纯化的和/或重组dgat不包含登录号eeh54819.1提供的序列。本发明的多肽的氨基酸序列突变体可通过在本发明的核酸内引入合适的核苷酸改变或通过体外合成所需多肽而制备。此类突变包括,例如,氨基酸序列内残基的缺失、插入或取代。可通过缺失、插入和取代的组合而获得最终的构建体,条件是最终的肽产物具有所需的特征。可通过本领域所知的任何技术制备突变的(改变的)肽。例如,可对本发明的多核苷酸进行体外诱变。此类体外诱变技术包括将多核苷酸亚克隆到合适的载体内,将载体转化到"增变株(mutator)"菌株如大肠杆菌(e.coli)xl-1red(stratagene)内并将转化的细菌增殖合适的代数。在另一个实例中,对本发明的多核苷酸进行dna重排,参见harayama(1998)。可采用在此所述的技术容易地筛选源自突变的/改变的dna的产物以确定它们是否具有去饱和酶和/或延长酶活性。在设计氨基酸序列突变体时,突变的位置和突变的性质取决于要修饰的特征。可单个地或系列地修饰突变位点,例如通过(1)先选择保守性氨基酸进行取代,然后根据所得结果进行更加激进的选择,(2)缺失靶残基,或(3)在邻近被定位的位点处插入其他残基。氨基酸序列缺失通常为大约1至15个残基,更优选地大约1至10个残基,且典型地为大约1至5个连续的残基。取代突变是去除多肽分子内的至少一个氨基酸残基并在该处插入一个不同的残基。取代诱变的最感兴趣的位点包括那些被鉴定为是活性位点的位点。感兴趣的其他位点是那些在各种菌株或物种之间均相同的特定残基。这些位置可能对生物学活性十分重要。这些位点,特别是那些具有至少3个其他同等保守的位点的序列内的位点,优选地以相对保守的方式进行取代。表3在"示例性取代"一栏给出了此类保守性取代。在优选的实施方式中,与天然存在的多肽相比,突变体/变体多肽具有1或2或3或4个保守性氨基酸改变。表1给出了保守性氨基酸改变的详情。表1给出了保守性氨基酸改变的详情。本领域人员能够理解,可以合理地预期此类少数的改变不会改变在重组细胞内表达的所述多肽的活性。表3.示例性取代原残基示例性取代ala(a)val;leu;ile;glyarg(r)lysasn(n)gln;hisasp(d)glucys(c)sergln(q)asn;hisglu(e)aspgly(g)pro,alahis(h)asn;glnile(i)leu;val;alaleu(l)ile;val;met;ala;phelys(k)argmet(m)leu;phephe(f)leu;val;alapro(p)glyser(s)thrthr(t)sertrp(w)tyrtyr(y)trp;pheval(v)ile;leu;met;phe;ala本发明也包括在合成过程中或合成之后被差异性修饰的本发明的多肽,例如通过生物素化、苄化作用、糖基化、乙酰化、磷酸化、酰胺化、通过已知的保护/阻断基团进行衍生作用、蛋白裂解、连接于抗体分子或其他细胞配体等等。这些修饰可提高本发明的多肽的稳定性和/或生物活性。可以多种形式产生本发明的多肽,包括产生和回收天然多肽、产生和回收重组多肽以及化学合成所述多肽。在一个实施方式中,如下产生重组多肽,在有效产生所述多肽的条件下培养能够表达所述多肽的细胞。重组多肽可随后从细胞分泌并被回收,或从细胞中提取并回收,并优选地自污染分子中纯化出来。其可以被进一步地以化学或酶学方法修饰或者不进行这样的修饰。用于培养的优选的细胞是本发明的重组细胞。有效的培养条件包括,但不限于,允许多肽产生的有效的培养基、生物反应器、温度、ph和氧条件。有效的培养基指的是可将细胞培养于其中以产生本发明的多肽的任何培养基。该培养基典型地包含具有可同化的碳源、氮源和磷酸源以及合适的盐、矿物质、金属和其他营养物如维生素的水性培养基。本发明的细胞可培养于常规的发酵生物反应器、摇瓶、试管、微滴定板和培养皿中。可在适合于重组细胞的温度、ph和氧含量进行培养。此类培养条件是本领域人员熟知的。产生所述多肽的更优选的细胞是植物中的细胞,特别是植物种子中的细胞。在本发明中,除非另有说明,术语"抗体"包括完整抗体的片段,所述片段仍保留与靶分析物的结合活性,也包括包含所述片段的化合物。此类片段包括fv、f(ab')和f(ab')2片段,以及单链抗体(scfv)。本发明的抗体可以是多克隆或单克隆的,并可采用本领域的标准工艺制备。多核苷酸本发明还提供多核苷酸,其可以是,例如,基因、分离的多核苷酸或嵌合dna。其可以是dna或rna,可以是基因组来源的或合成的,双链的或单链的,并与碳水化合物、脂质、蛋白质或其他材料组合,以进行在此所定义的具体活性。术语"多核苷酸"在此与术语"核酸分子"互换使用。“分离的多核苷酸”指的是这样的多核苷酸,如果所述多核苷酸是从自然来源获得的,其已经与天然状态下与之结合或连接的那些多核苷酸序列分离,或是非天然存在的多核苷酸。优选地,分离的多核苷酸至少60%、更优选地至少75%、且更优选地至少90%不含与之天然结合的其他成分。在本文中,术语"基因"取其最广泛的含义,并包括脱氧核糖核苷酸序列,后者包含转录区和(如果翻译的话)结构基因的蛋白质编码区、并包括邻近所述编码区5’和3’端且位于距所述编码区5’和3’端之任一端至少大约2kb距离处的序列,所述序列参与该基因的表达。就此而言,基因包括调控信号,例如与特定基因天然相关的启动子、增强子、终止信号和/或多聚腺苷酸信号,或异源性调控信号,在后一种情况中,该基因称为"嵌合基因"。位于蛋白质编码区5’端且存在于mrna上的序列称为5’非翻译序列。位于蛋白质编码区3’端或下游且存在于mrna上的序列称为3’非翻译序列。术语“基因”涵盖cdna和基因组形式的基因。基因的基因组形式或克隆含有被非编码序列中断的编码区,这些非编码序列称为"内含子"或"干扰区"或"干扰区"或"干扰序列"。内含子是被转录至核rna(hnrna)内的基因区段。内含子可含有调控元件例如增强子。内含子要自核转录产物或初级转录产物去除或“剪切掉”,因此信使rna(mrna)转录产物内没有内含子。mrna的功能是在翻译过程中确定新生多肽内的氨基酸序列或次序。术语"基因"包括编码全部或部分在此所述的蛋白质的合成分子或融合分子以及上述任一的互补核苷酸序列。在本申请中,"嵌合dna"指的是任何这样的dna分子,其不是天然位置上的天然dna分子,在此也称为"dna构建体"。典型地,嵌合dna或嵌合基因包含在天然情况下并不在一起的调控序列、转录的序列或蛋白编码序列。因此,嵌合dna或嵌合基因可包含不同来源的调控序列和编码序列,或包含同一来源但以不同于天然情况下所见的方式排列的调控序列和编码序列。术语"内源性"在此指的是在未修饰植物与所研究的植物中在相同的发育阶段均正常存在或产生的物质。"内源性基因"指的是生物体基因组中存在于其天然位置上的天然基因。在本申请中,"重组核酸分子"、"重组多核苷酸"或其变化形式指的是通过重组dna技术而构建或修饰的核酸分子。术语"外来多核苷酸"或"外源性多核苷酸"或"异源性多核苷酸"等指的是通过实验操控引入细胞基因组内的任何核酸。外来或外源基因可以是插入至非天然生物体内的基因,引入至天然宿主细胞内的新位置上的天然基因,或嵌合基因。"转基因"是已经通过转化程序而引入至基因组中的基因。术语"遗传学修饰"、"转基因"及其变化形式包括通过转化或转导将基因引入至细胞内,突变细胞内的基因,并改变或调节已进行上述这些步骤的细胞或生物体或它们的子代内的基因调控。“基因组区”在此指的是基因组内的位置,其中已经将转基因或一组转基因(在此也称为簇)插入至细胞内或其祖先内。此类区域仅包括通过人工干预例如通过在此所述的方法而插入的那些核苷酸。术语"外源性"当涉及多核苷酸时指的是,当所述多核苷酸存在于细胞中时,与其天然状态相比,所述多核苷酸的量发生改变。在一个实施方式中,所述细胞天然条件下不包含所述多核苷酸。不过,所述细胞可包含导致所编码的多肽产生的量发生改变的非内源性多核苷酸。本发明的外源多核苷酸包括尚未与它们存在于其中的转基因(重组)细胞或无细胞表达系统中的其他成分分离的多核苷酸,也包括在此类细胞或无细胞系统中产生并随后自至少某些其他成分中纯化出来的多核苷酸。外源多核苷酸(核酸)可以是天然存在的一段连续的核苷酸,或是包含由来自不同来源的(天然存在的和/或合成的)两或多段连续的核苷酸连接形成的单一多核苷酸。典型地,此类嵌合多核苷酸至少包含编码本发明的多肽的可读框,该可读框可操纵地连接于适合在感兴趣的细胞中驱动所述可读框转录的启动子。在本文中,术语“不同的外源性多核苷酸”或其变化形式指的是各种多核苷酸的核苷酸序列具有至少一个、优选地多个核苷酸的不同。多核苷酸编码的rna可以在细胞内翻译成蛋白质或者不翻译。在一个实例中,优选的是每一多核苷酸编码具有不同活性的蛋白质。在另一实例中,每一外源性多核苷酸与另一外源性多核苷酸的相同性少于95%、少于90%、或少于80%。优选地,外源性多核苷酸编码功能性蛋白质/酶。此外,优选的是不同的外源性多核苷酸是不重叠的,即每一多核苷酸是例如染色体外转移核酸的不同区域,其与另一外源性多核苷酸不重叠。至少,每一外源性多核苷酸具有转录起始和终止位点以及所指定的启动子。一个外源性多核苷酸可包含或不包含内含子。就所定义的多核苷酸而言,可以理解高于上述数值的百分比相同性是优选的实施方式。因此,在适用的情况下,就最小的百分比相同性数值而言,优选地所述多核苷酸包含这样的多核苷酸序列,其与相应的指定seqidno具有至少60%、更优选地至少65%、更优选地至少70%、更优选地至少75%、更优选地至少80%、更优选地至少85%、更优选地至少90%、更优选地至少91%、更优选地至少92%、更优选地至少93%、更优选地至少94%、更优选地至少95%、更优选地至少96%、更优选地至少97%、更优选地至少98%、更优选地至少99%、更优选地至少99.1%、更优选地至少99.2%、更优选地至少99.3%、更优选地至少99.4%、更优选地至少99.5%、更优选地至少99.6%、更优选地至少99.7%、更优选地至少99.8%、且甚至更优选地至少99.9%的相同性。在一个实施方式中,本发明的编码δ6去饱和酶的分离的和/或外源性多核苷酸不包含来自微胞藻或ostreococcus基因组的推定为分别编码登录号eeh58637.1或xp_001421073.1中提供的氨基酸序列的序列。在另一个实施方式中,本发明的编码ω3去饱和酶的分离的和/或外源性多核苷酸不包含来自微胞藻基因组的推定为编码登录号xp_002505536.1中提供的氨基酸序列的序列。在另一个实施方式中,本发明的编码dgat的分离的和/或外源性多核苷酸不包含来自微胞藻基因组的推定为编码登录号eeh54819.1中提供的氨基酸序列的序列。本发明的多核苷酸可在严格性条件下与编码本发明的多肽的多核苷酸选择性杂交。在本文中,在严格性条件下指的是(1)杂交过程中采用变性剂例如甲酰胺,例如,50%(v/v)甲酰胺含0.1%(w/v)牛血清白蛋白,0.1%ficoll,0.1%聚乙烯吡咯烷酮,50mm磷酸钠缓冲液(ph6.5)含750mmnacl,75mm柠檬酸钠,42℃;或(2)采用50%甲酰胺,5xssc(0.75mnacl,0.075m柠檬酸钠),50mm磷酸钠(ph6.8),0.1%焦磷酸钠,5x登哈特溶液,超声处理的鲑精dna(50g/ml),0.1%sds和10%硫酸葡聚糖,42℃,在0.2xssc和0.1%sds中;和/或(3)采用低离子强度和高洗涤温度,例如,0.015mnacl/0.0015m柠檬酸钠/0.1%sds,50℃。与天然存在的分子相比,本发明的多核苷酸可具有一或多个突变,其可以是核苷酸残基的缺失、插入、或取代。相对于参比序列,具有突变的多核苷酸可以是天然产生的(即分离自天然来源)或合成的(例如,通过在上述核酸上进行定点诱变或dna重排)。因此,本发明的多核苷酸显然可以是天然存在的或重组的。重组载体本发明的一个实施方式包括重组载体,其包含至少一种本发明的核苷酸分子,所述核苷酸分子插入至能够将所述多核苷酸分子输送至宿主细胞内的任何载体内。重组载体包括表达载体。重组载体含有异源多核苷酸序列,即所述多核苷酸序列天然情况下不与本发明的核苷酸分子邻近,且优选地其所来源的物种不同于所述多核苷酸分子所来源的物种。载体可以是rna或dna,真核细胞的或原核细胞的,且典型地是病毒载体、来源于病毒或质粒。质粒载体典型地包括额外的核酸序列,以便于筛选、扩增和将表达盒转化至原核细胞,例如puc-来源的载体、psk-来源的载体、pgem-来源的载体、psp-来源的载体、pbs-来源的载体、或含有一或多个t-dna区域的二元载体。额外的核酸序列包括复制起点(用于载体自主复制)、选择标记物基因(优选地编码抗生素抗性或除草剂抗性)、独特的多克隆位点(用于提供多个位点以便将编码的核酸序列或基因插入核酸构建体中)、以及增强原核细胞和真核细胞(特别是植物细胞)的转化的序列。重组载体包含的本发明的多核苷酸可多于1种,例如3、4、5或6种本发明的多核苷酸的组合,每一个均可操纵地连接于在感兴趣的细胞中起作用的表达调控序列。优选地,所述多于1种的多核苷酸,例如3、4、5或6种多核苷酸,在单独一个重组载体内共价连接在一起,然后可将它们作为一个单独的分子引入细胞内,形成本发明的重组细胞,且优选地整合至该重组细胞的基因组内,例如转基因植物的重组细胞。由此,连接在一起的这些多核苷酸可作为单独一个基因座而被重组细胞或植物的子代整体继承。重组载体或植物可包含2个或更多个此类重组载体,每一个均含有多种多核苷酸,例如其中每一重组载体包含3、4、5或6种多核苷酸。"可操纵地连接"在本文中指的是两个或多个核酸(例如dna)区段之间的功能性关系。典型地,其指的是转录调控元件(启动子)与被转录序列的功能性关系。例如,启动子可操纵地连接于编码序列,例如在此所述的多核苷酸,条件是其在合适的细胞内刺激或调变编码序列的转录。通常,可操纵地连接于被转录序列的启动子转录调控元件与被转录序列在物理上是连续的,即,它们具有顺式作用。不过,某些转录调控元件,例如增强子,不需要与被增强转录的编码序列在物理上连续或位于与之十分接近的位置。当有多个启动子时,每一启动子可独立地是相同的或不同的。重组分子,例如嵌合dna,还可含有(a)一或多种编码信号肽序列的分泌信号,以使得所表达的本发明的多肽被产生所述多肽的细胞分泌,或为所表达的多肽提供定位,例如将多肽保留在细胞的内质网(er)中或转移至质体内,和/或(b)含有融合序列,这导致核酸分子表达为融合蛋白。合适的信号区段的实例包括任何能够指导本发明的多肽分泌或定位的信号区段。优选的信号区段包括,但不限于,nicotiananectarin信号肽(us5,939,288)、烟草延伸信号或大豆油质蛋白油体(soyoleosinoilbody)结合蛋白信号。重组分子还可包含位于本发明的核酸分子的核酸序列周围和/或内部的干扰和/或非翻译序列。为了有助于鉴定转化体,希望核酸构建体在外来或外源性多核苷酸以外还包含选择标记物基因或筛选标记物基因,或包含选择标记物基因或筛选标记物基因作为外来或外源性多核苷酸。"标记物基因"指的是赋予表达该标记物基因的细胞以不同表型的基因,这使得此类被转化的细胞能够与不含有该标记物基因的细胞区别开。选择标记物基因赋予一种性状,以便基于对选择剂(例如,除草剂、抗生素、照射、热或其他对未转化细胞造成损害的处理)的抗性进行"选择"。筛选标记物基因(或报告基因)赋予一种性状,以便通过观察或测试,例如通过“筛选”(例如,β-葡糖醛酸糖苷酶、萤光素酶、gfp或未转化细胞中不存在的其他酶活性),而鉴定。标记物基因和感兴趣的核苷酸序列不一定要连接。标记物的实际选择并非至关重要,只要其与所选择的细胞(例如植物细胞)相组合具有功能(即,选择性)即可。标记物基因和感兴趣的外来或外源性多核苷酸不一定要连接,因为共转化不连接的基因(例如,见us4,399,216)也是有效的植物转化方法。细菌选择标记物的实例是赋予抗生素抗性的标记物,例如氨苄西林、红霉素、氯霉素或四环素抗性,优选地卡那霉素抗性。用于选择植物转化体的示例性选择标记物包括,但不限于,hyg基因,其编码潮霉素b抗性;新霉素磷酸转移酶(nptii)基因,赋予对卡那霉素、巴龙霉素、g418的抗性;大鼠肝脏的谷胱甘肽-s-转移酶基因,赋予对谷胱甘肽衍生的除草剂的抗性,例如,见ep256223;谷氨酰胺合成酶基因,过表达后赋予对谷氨酰胺合成酶抑制剂例如草胺膦(phosphinothricin)的抗性,例如,见wo87/05327;来自产绿色链霉菌(streptomycesviridochromogenes)的乙酰基转移酶基因,赋予对选择剂草胺膦的抗性,例如,见ep275957;编码5-烯醇莽草酸-3-磷酸酯合酶(epsps)的基因,赋予对的耐受性n-膦酰基甲基苷氨酸,例如,见hinchee等(1988);bar基因,赋予对双丙氨膦的抗性,例如,见wo91/02071;腈水解酶基因,例如来自臭鼻克雷伯菌(klebsiellaozaenae)的bxn,其赋予对溴苯腈的抗性(stalker等,1988);二氢叶酸还原酶(dhfr)基因,赋予对氨甲蝶呤的抗性(thillet等,1988);突变的乙酰乳酸合酶基因(als),其赋予对咪唑啉酮、磺酰脲或其他als-抑制性化合物的抗性(ep154,204);突变的邻氨基苯甲酸合酶基因,其赋予对5-甲基色氨酸的抗性;或茅草枯脱卤素酶基因,其赋予对除草剂的抗性。优选的筛选标记物包括,但不限于,uida基因,其编码β-葡糖醛酸糖苷酶(gus),该酶的各种显色底物是已知的;β-半乳糖苷酶基因,其所编码的酶的显色底物是已知的;水母荧光素基因(prasher等,1985),其可用于钙敏感型生物发光检测;绿色荧光蛋白基因(niedz等,1995)或其衍生物;萤光素酶(luc)基因(ow等,1986),其可用于生物发光检测;和其他本领域已知的那些。"报道分子"在本说明书中指的是这样的分子,依据其化学性质,提供可分析鉴定的信号,所述信号有助于通过参照蛋白质产物而确定启动子活性。优选地,核酸构建体稳定整合至细胞例如植物细胞的基因组内。因此,所述核酸可包含合适的元件,使得分子能够整合至基因组内,或者所述构建体被置于合适的载体内,以整合至细胞的染色体内。表达在本申请中,表达载体是dna或rna载体,其能够转化宿主细胞并实现一或多种特定多核苷酸分子的表达。优选地,表达载体还能够在宿主细胞复制。表达载体可以是原核细胞的或真核细胞的,且典型地是病毒或质粒。本发明的表达载体包括在本发明的宿主细胞内有功能(即,指导基因表达)的任何载体,宿主细胞包括细菌、真菌、内寄生物、节肢动物细胞、动物细胞和植物细胞。本发明的特别优选的表达载体可指导基因在酵母和/或植物细胞内表达。本发明的表达载体含有调控序列,例如转录控制序列、翻译控制序列、复制起点和与重组细胞具有相容性并控制本发明的核苷酸分子表达的其他调控序列。具体而言,本发明的多核苷酸或载体包括转录控制序列。转录控制序列是控制转录起始、延长和终止的序列。特别重要的转录控制序列是那些控制转录起始的序列,例如启动子、增强子、操纵子和阻遏物序列。合适的转录控制序列包括能够在至少一种本发明的重组细胞中起作用的转录控制序列。所使用的调控序列的选择取决于靶生物体,例如感兴趣的植物和/或靶器官或组织。此类调控序列可获自任何真核生物体,例如植物或植物病毒,或可化学合成。各种各样的此类转录控制序列是本领域人员已知的。特别优选的转录控制序列是那些在植物中具有转录指导活性的启动子,根据所使用的植物或其部分,可以是组成型的或具有阶段和/或组织特异性。适用于稳定转染植物细胞或建立转基因植物的多种载体已被公开,例如参见pouwels等,cloningvectors:alaboratorymanual,1985,supp.1987;weissbachandweissbach,methodsforplantmolecularbiology,academicpress,1989;和gelvin等,plantmolecularbiologymanual,kluweracademicpublishers,1990。典型地,植物表达载体包括,例如,受5’和3’调控序列转录调控的一或多个克隆的植物基因和显性选择标记物。此类植物表达载体还可含有启动子调控区(例如,调控区控制诱导型或组成型表达、环境调控型或发育调控型表达、或细胞或组织特异性表达)、转录起始位点、核糖体结合位点、rna加工信号、转录终止位点、和/或多腺苷酸化信号。已经公开了在植物细胞内具有活性的多种组成型启动子。用于在植物中进行组成型表达的合适的启动子包括,但不限于,花椰菜花叶病病毒(camv)35s启动子、玄参花叶病病毒(fmv)35s、甘蔗杆状病毒启动子、鸭跖草黄色斑点病毒启动子、来自核酮糖-1,5-二磷酸羧化酶小亚基的光诱导型启动子、稻的细胞质磷酸丙糖异构酶启动子、拟南芥的腺嘌呤磷酸核糖基转移酶启动子、稻肌动蛋白1基因启动子、甘露氨酸合酶和章鱼碱合酶启动子、adh启动子、蔗糖合酶启动子、r基因复合体启动子和叶绿素α/β结合蛋白基因启动子。这些启动子已经用于构建在植物中表达的dna载体;参见,例如wo84/02913。所有这些启动子均已用于构建可在植物中表达的各类重组dna载体。为了在植物的源组织例如叶、种子、根或茎中进行表达,优选地,本发明所用的启动子在这些特定组织中具有相对高的表达。为此,可为基因选择多种具有组织或细胞特异性或增强型的启动子。文献报导的此类启动子的实例包括豌豆的叶绿体谷氨酰胺合成酶gs2启动子、小麦的叶绿体果糖-1,6-二磷酸酶启动子、马铃薯的核光合st-ls1启动子、拟南芥的丝氨酸/苏氨酸激酶启动子和葡糖淀粉酶(chs)启动子。以下启动子也被报导在具有光合作用活性的组织中具有活性,东方落叶松(larixlaricina)的核酮糖-1,5-二磷酸羧化酶启动子、松cab基因cab6的启动子、小麦cab-1基因启动子、菠菜cab-1基因启动子、稻cab1r基因启动子、玉蜀黍的丙酮酸-正磷酸双激酶(ppdk)启动子、烟草lhcb1*2基因的启动子、拟南芥suc2蔗糖-h30协同转运子启动子、菠菜的类囊体膜蛋白基因启动子(psad、psaf、psae、pc、fnr、atpc、atpd、cab、rbcs)。叶绿素α/β-结合蛋白的其他启动子也可用于本发明,例如白芥菜(sinapisalba)lhcb基因和psbp基因的启动子。应答于环境、激素、化学品和/或发育信号而被调控的各种植物基因启动子也可用于在植物细胞中表达rna-结合蛋白基因,包括受到以下调控的启动子:(1)热;(2)光(例如豌豆rbcs-3a启动子、玉蜀黍rbcs启动子);(3)激素,例如脱落酸;(4)致伤(wounding)(例如wuni);或(5)化学制品,例如茉莉酮酸甲酯、水杨酸、类固醇激素、醇、safeners(wo9706269);或者使用(6)器官特异性启动子也是有利的。在本文中,术语“植物贮藏器官特异性启动子”指的是这样的启动子,与其他植物组织相比,其优先在植物的贮藏器官中指导基因转录。优选地,所述启动子仅在贮藏器官中指导感兴趣的基因的表达,和/或在植物的其他部分例如叶中,通过northern印迹分析和/或rt-pcr检测不到感兴趣的基因的表达。典型地,启动子在贮藏器官的生长和发育过程中驱动基因表达,特别是在贮藏器官中合成并积聚贮藏化合物的阶段中。此类启动子可在整个植物贮藏器官中驱动基因表达,或仅在其部分中,例如双子叶植物种子的种皮、胚或子叶中或单子叶植物种子的胚乳或糊粉层中驱动基因表达。为了在植物的库组织(sinktissues)中表达,例如在马铃薯的块茎、番茄的果实、或大豆、芸苔、棉花、玉蜀黍、小麦、稻和大麦的种子中表达,本发明所用的启动子优选地在这些特异性组织中具有相对高的表达。已知多种具有块茎特异性或增强型表达的基因启动子,包括i类patatin启动子、马铃薯块茎adpgpp基因启动子(大和小亚单位)、蔗糖合酶启动子、包括22kd蛋白质复合物和蛋白酶抑制剂的主要块茎蛋白的启动子、颗粒结合型淀粉合酶基因(gbss)的启动子以及其他i和ii类patatin启动子。其他启动子也可用于在特定组织例如种子或果实中表达蛋白质。可使用β-伴大豆球蛋白启动子或其他种子特异性启动子例如napin、玉米醇溶蛋白、核丝(linin)和菜豆球蛋白启动子。还可使用根特异性启动子。所述启动子的实例是酸性壳多糖酶基因启动子。还可使用已经得到鉴定的camv35s启动子的根特异性亚结构域来实现在根组织中的表达。在一个特别优选的实施方式中,启动子在脂肪酸和油进行生物合成的组织和器官中指导表达。在种子发育过程中,此类启动子在合适的时机起作用,改变种子的油的组成。在进一步特别优选的实施方式中,且在本发明的一些方面,启动子是植物贮藏器官特异性启动子。在一个实施方式中,所述植物贮藏器官特异性启动子是种子特异性启动子。在更加优选的实施方式中,相对于在种子的胚或植物的其他器官例如叶中的表达而言,所述启动子优先在双子叶植物的子叶或单子叶植物的胚乳中指导表达。用于种子特异性表达的优选的启动子包括:i)来自编码参与种子中脂肪酸生物合成和积聚的酶(例如去饱和酶和延长酶)的基因的启动子,ii)来自编码种子贮藏蛋白的基因的启动子,和iii)来自编码参与种子中碳水化合物生物合成和积聚的酶的基因的启动子。合适的种子特异性启动子是油料种子油菜napin基因启动子(us5,608,152)、蚕豆(viciafaba)usp启动子(baumlein等,1991)、拟南芥油质蛋白启动子(wo98/45461)、菜豆(phaseolusvulgaris)的菜豆球蛋白启动子(美国5,504,200)、芸苔属bce4启动子(wo91/13980)或豆球蛋白b4启动子(baumlein等,1992),以及在单子叶植物例如玉蜀黍、大麦、小麦、黑麦、稻等中引起种子特异性的表达启动子。合适的著名启动子是大麦lpt2或lpt1基因启动子(wo95/15389和wo95/23230)或wo99/16890所述的启动子(大麦的大麦醇溶蛋白基因的启动子、稻的谷蛋白基因的启动子、稻的oryzin基因的启动子、稻的醇溶谷蛋白基因的启动子、小麦的麦醇溶蛋白基因的启动子、小麦的谷蛋白基因的启动子、玉蜀黍的玉米醇溶蛋白基因的启动子、燕麦的谷蛋白基因的启动子、蜀黍kasirin基因的启动子、黑麦的黑麦醇溶蛋白基因的启动子)。其他启动子包括broun等(1998)、potenza等(2004)、us20070192902和us20030159173所述的启动子。在一个实施方式中,所述种子特异性启动子优先表达于种子的特定部分,例如子叶或胚乳。子叶特异性启动子的实例包括,但不限于,fp1启动子(ellerstrom等,1996)、豌豆豆球蛋白启动子(perrin等,2000)和豆植物凝血素启动子(perrin等,2000)。胚乳特异性启动子的实例包括,但不限于,玉米的玉米醇溶蛋白-1启动子(chikwamba等,2003)、稻谷蛋白-1启动子(yang等,2003)、大麦d-大麦醇溶蛋白启动子(horvath等,2000)和小麦hmw麦谷蛋白启动子(alvarez等,2000)。在进一步的实施方式中,所述种子特异性启动子在胚中和/或在种子萌发后不表达,或是仅以低水平表达。在另一个实施方式中,所述植物贮藏器官特异性启动子是块茎特异性启动子。实例包括,但不限于,马铃薯patatinb33、pat21和gbss启动子,以及甜马铃薯sporamin启动子(综述见potenza等,2004)。在优选的实施方式中,相对于块茎的外层(皮)或块茎的胚而言,所述启动子优先在块茎的木髓中指导表达。在另一个实施方式中,所述植物贮藏器官特异性启动子是果实特异性启动子。实例包括,但不限于,番茄多聚半乳糖醛酸酶、e8和pds启动子以及苹果acc氧化酶启动子(综述见potenza等,2004)。在优选的实施方式中,相对于果实的皮或果实内的种子而言,所述启动子优先在果实的可食用部分例如果实的木髓中指导表达。可从选定的启动子衍生出5'非翻译前导序列以表达本发明的多核苷酸的异源基因序列,或者,5'非翻译前导序列可以与待产生的酶的编码区是异源的,需要的话还可对其进行特异性修饰以便增加mrna的翻译。如何优化转基因的表达可参见koziel等(1996)的综述。5'非翻译区也可得自植物病毒rna(烟草花叶病病毒、烟草刻蚀病毒、玉蜀黍矮花叶病病毒、紫花苜蓿花叶病病毒等等)、合适的真核细胞基因、植物基因(小麦和玉蜀黍叶绿素a/b结合蛋白基因前导区)、或合成的基因序列。本发明不限于其中所述非翻译区衍生自伴随启动子序列的5'非翻译序列的构建体。前导序列也可衍生自无关的启动子或编码序列。可用于本发明的前导序列包括玉蜀黍hsp70前导区(美国5,362,865和美国5,859,347)和实施例8中例证的tmvω元件。转录的终止是通过嵌合载体内与感兴趣的多核苷酸可操纵地连接的3'非翻译dna序列而实现的。重组dna分子的3'非翻译区含有多腺苷酸化信号,其在植物中的作用是导致在rna的3'端添加腺嘌呤核苷酸。3'非翻译区可得自在植物细胞中表达的各种基因。就此能力而言,常用的是胭脂碱合酶3'非翻译区、豌豆的小亚单位核酮糖二磷酸羟化酶(rubisco)基因的3'非翻译区、大豆的7s种子贮藏蛋白基因的3'非翻译区。含有聚腺苷酸化信号的土壤杆菌(agrobacterium)肿瘤诱导型(ti)质粒基因的3'转录的非翻译区也是合适的。可采用重组dna技术来提高转化的多核苷酸分子的表达,例如提高操控宿主细胞内多核苷酸分子的拷贝数、所述多核苷酸分子的转录效率、所得转录产物的翻译效率、以及转录后修饰的效率。可用于增加本发明的核苷酸分子表达的重组技术包括,但不限于,将多核苷酸分子可操纵地连接于高拷贝数质粒,将多核苷酸分子整合至一或多个宿主细胞染色体中,给质粒添加载体稳定序列,置换或改进转录控制信号(例如启动子、操纵子、增强子),置换或改进翻译控制信号(例如核糖体结合位点、shine-dalgarno序列),改进本发明的核苷酸分子以符合宿主细胞内的密码子使用情况,以及去除使转录产物不稳定的序列。转移核酸本发明的转移核酸至少包含一个、优选地两个边界序列,以及外源性多核苷酸。转移核酸可编码或不编码选择标记物。优选地,转移核酸形成细菌中的二元载体的一部分,其中所述二元载体还包含使得载体在细菌中复制的元件或能够选择或维持含有所述载体的细菌细胞的元件。转移至真核细胞后,二元载体的转移核酸成分能够整合至真核细胞的基因组中。在本文中,术语“染色体外转移核酸”指的是这样的核酸分子,其能够从细菌例如土壤杆菌转移至真核细胞例如植物的叶细胞。染色体外转移核酸是熟知的遗传学元件,能够被转移并随后将其边界内所含的核苷酸序列整合至受者细胞的基因组内。就此而言,转移核酸典型地在其两侧有两个“边界”序列,不过在一些情况中仅在一侧有单个边界,并在转移过程中随机产生所转移的核酸的另一端。合意的外源性多核苷酸典型地位于转移核酸的左侧边界样序列与右侧边界样序列之间。转移核酸内所含的合意的多核苷酸可以可操纵地连于多种不同的启动子和终止子调控元件,以有助于其表达,即多核苷酸的转录和/或翻译。来自土壤杆菌例如根癌土壤杆菌(agrobacteriumtumefaciens)或毛根土壤杆菌(agrobacteriumrhizogenes)的t-dna及其人工变体/突变体可能是转移核酸的表征的最清楚的实例。另一实例是p-dna("植物-dna"),其包含来自植物的t-dna边界样序列。在本申请中,“t-dna”指的是,例如,来自根癌土壤杆菌ti质粒或毛根土壤杆菌ri质粒的t-dna,或是其具有t-dna(转移的-dna)功能的人工变体。t-dna可包含整个t-dna,包括右侧和左侧边界序列,但仅需要包含用于转移的顺式所需的最少序列,即右侧和t-dna边界序列。本发明的t-dna在其右侧和左侧边界序列(如果有的话)之间的任何位置插入了外源性多核苷酸,后者的两侧为位点特异性重组酶的靶位点。编码用于将t-dna转移至植物细胞的反式所需的因子的序列,例如vir基因,可插入至t-dna,或可与t-dna存在于同一复制子中,或优选地在土壤杆菌宿主中的相适应的复制子中式反式的。此类“二元载体系统”是本领域已知的。在本申请中,“p-dna”指的是从植物基因组中分离的转移核酸,或是其人工的变体/突变体,并在每一端或至少一端包含t-dna边界样序列。所述边界样序列优选地与来自土壤杆菌例如根癌土壤杆菌或毛根土壤杆菌的t-dna边界序列具有至少50%、至少60%、至少70%、至少75%、至少80%、至少90%或至少95%,但低于100%的序列相同性。因此,p-dna可替代t-dna,用于将p-dna中所含的核苷酸序列从例如土壤杆菌转移至另一种细胞。在插入待转移的外源性多核苷酸之前,可修饰p-dna以便克隆,p-dna优选地应该不编码任何蛋白质。p-dna的特征在于其含有至少右侧边界序列,优选地也含有左侧边界序列。在本申请中,转移核酸的“边界”序列可分离自选定的生物体例如植物或细菌,或是其人工变体/突变体。边界序列促使并有助于转移与之连接的外源性多核苷酸,并有助于其整合至受者细胞基因组内。在一个实施方式中,边界序列的长度为5-100bp、10-80bp、15-75bp、15-60bp、15-50bp、15-40bp、15-30bp、16-30bp、20-30bp、21-30bp、22-30bp、23-30bp、24-30bp、25-30bp、或26-30bp。来自土壤杆菌的t-dna的边界序列是本领域熟知的,见lacroix等(2008)、tzfira和citovsky(2006)和glevin(2003)。p-dna的边界序列可分离自任何植物,例如马铃薯和小麦。在一个实施方式中,所述p-dna具有核酸序列angatntatn6gt(seqidno:109),其中"n"是任何核苷酸,例如"a,""g,""c,"或"t"。可用于本发明的其他边界序列的实例包括,但不限于,tgacaggatatattggcgggtaaac(seqidno:110);tggcaggatatattgtggtgtaaac(seqidno:111);tggcaggatatataccgttgtaatt(seqidno:112);cggcaggatatattcaattgtaatt(seqidno:113);tggtaggatatataccgttgtaatt(seqidno:114);tggcaggatatatggtactgtaatt(seqidno:115);ygryaggatatatwsnvbkgtaawy(seqidno:116);cggcaggatatatcctgatgtaaat(seqidno:117);tggcaggagttattcgagggtaaac(seqidno:118);tgacaggatatatcgtgatgtcaac(seqidno:119);gggaagtacatattggcgggtaaac(seqidno:120);ttacaggatatattaatatgtatga(seqidno:121);taacatgatatattcccttgtaaat(seqidno:122);tgacaggatatatggtaatgtaaac(seqidno:123);和tggcaggatatataccgatgtaaac(seqidno:124),其中*y=c或t;r=a或g;k=g或t;w=a或t;s=c或g;v=a、c、或g;b=c、g、或t。尽管传统上仅采用土壤杆菌将基因转移至植物细胞,但现在已经鉴定/开发了许多以与土壤杆菌类似的方式起作用的系统。一些非土壤杆菌物种已经被遗传改造为适合进行基因转移(chung等,2006;broothaerts等,2005)。这些包括根瘤菌ngr234、苜蓿中华根瘤菌和百脉根中慢生根瘤菌。通过给细菌提供转化过程所需的机构(即,由土壤杆菌ti-质粒编码的一组致病基因和位于一单独的小二元质粒上的t-dna区段)而使得细菌适合于基因转移。这样的工程化细菌能够转化不同的植物组织(叶盘(leafdisks)、愈伤组织和卵形组织(ovaltissue))、单子叶植物或双子叶植物、以及多种不同植物(例如,烟草、稻)。几十年前,通过将哺乳动物细胞与携带质粒的大肠杆菌的原生质体融合而首次实现了将真核表达质粒从细菌直接转移至真核宿主(schaffner,1980)。其后,能够将基因输送至哺乳动物细胞的细菌的数量稳定增加(weiss,2003),这是由四组人员独立发现的(sizemore等1995;courvalin等,1995;powell等,1996;darji等,1997)。已经发现,弱化的弗氏志贺菌、鼠伤寒沙门氏菌或经由弗氏志贺菌的致病质粒(pwr100)而具有侵袭性的大肠杆菌,在侵入宿主细胞后能够转移表达质粒,并因代谢的弱化而发生细胞内死亡。通过鼻部或口腔粘膜施用此类重组志贺菌或沙门氏菌,诱导了针对该表达质粒所编码的抗原的免疫应答。同时,已发现的能够在体外和体内将表达质粒转移至哺乳动物宿主细胞的细菌的数量已经增加了一倍以上,已经报道的伤寒沙门氏菌(s.typhi)、猪霍乱沙门氏菌、单核细胞增多性李司忒菌、假结核病耶尔森菌和小肠结肠炎耶尔森菌(fennelly等,1999;shiau等,2001;dietrich等,1998,2001;hense等,2001;al-mariri等,2002)。一般而言,可以假设所有能够进入宿主细胞的细胞质并在其中裂解的细菌(如弗氏志贺菌或单核细胞增多性李司忒菌)都应该可以转移dna。这种情况称为“流产”或“自杀”侵入,因为细菌要裂解才能实现dna转移(grillot-courvalin等,1999)。此外,甚至停留在吞噬泡中的许多细菌(如鼠伤寒沙门氏菌)也能做到这一点。因此,已经被工程化为具有侵袭性但不能发生吞噬体逃逸的大肠杆菌重组实验株,仍旧能够将其质粒负载输送至被感染的哺乳动物细胞的核中(grillot-courvalin等,1998)。此外,最近也发现根癌土壤杆菌可将转基因引入哺乳动物细胞(kunik等,2001)。采用染色体外转移元件的转移方法典型地将多个拷贝的该元件转移至受者细胞内。在本文中,术语“瞬时转染”指的是尽管有些外源性多核苷酸可以稳定整合至细胞的基因组内,但并不筛选出稳定整合的细胞。因此,细胞内的许多转移核酸仍然处于染色体外,例如,转移至受者细胞内的外源性多核苷酸中有超过90%的拷贝没有整合至基因组内。在本文中,术语“转染”、“转化”及其变化形式通常可互换使用。“转染的”或“转化的”细胞是经操作引入了外源性多核苷酸的细胞,或是由所述细胞产生的子代细胞。重组细胞本发明还提供重组细胞,优选地重组植物细胞,其是以一或多种重组分子转化的宿主细胞,所述重组分子例如为本发明的多核苷酸、嵌合dnas或重组载体。重组细胞可包含它们的任意组合,例如两种或三种重组载体,或重组载体和一或多种额外的多核苷酸或嵌合dna。本发明的合适的细胞包括任何能够被本发明的多核苷酸、嵌合dna或重组载体转化的细胞,例如,编码本发明的多肽或酶的分子。所述细胞优选地是由此能够用于产生lc-pufa的细胞。重组细胞可以是培养的细胞、体外的细胞、或生物体内的细胞,例如植物或器官例如种子或叶内的细胞。优选地,所述细胞处于植物内,更优选地处于植物的种子内。被引入所述多核苷酸的宿主细胞可以是未转化的细胞,或是已经以一或多种核酸分子转化的细胞。此类核酸分子可与lc-pufa合成有关或无关。本发明的宿主细胞可以内源性地(即,天然地)具有产生本发明的蛋白质的能力,在这种情况中,由其产生的所述重组细胞具有增强的产生所述多肽的能力,或者,所述宿主细胞仅在以一或多种本发明的多核苷酸转化之后才能产生此类蛋白质。在一个实施方式中,本发明的重组细胞具有增强的合成长链多不饱和脂肪酸的能力。在本文中,术语"具有增强的合成长链多不饱和脂肪酸的能力的细胞"是一个将本发明的重组细胞与缺乏本发明的多核苷酸的宿主细胞相比的比较性的术语,其中与天然细胞相比,所述重组细胞产生更多的长链多不饱和脂肪酸,或更高浓度的lc-pufa例如epa、dpa或dha(相对于其他脂肪酸)。具有增强的合成另一种产物(例如另一种脂肪酸、脂质、碳水化合物如淀粉、rna分子、多肽、药物或其他产物)的能力的细胞具有相应的含义。本发明的宿主细胞可以是任何能够产生至少一种在此所述的蛋白质的细胞,包括细菌、真菌(包括酵母)、寄生虫、节肢动物、底物和植物细胞。细胞可以是原核细胞或真核细胞。优选的宿主细胞是酵母和植物细胞。在优选的实施方式中,所述植物细胞是种子细胞,特别是种子的子叶或胚乳内的细胞。在一个实施方式中,所述细胞是动物细胞或海藻细胞。动物细胞可以是任何类型的动物细胞,例如,非人类动物细胞、非人类脊椎动物细胞、非人类哺乳动物细胞,或水生动物如鱼类或甲壳类动物、无脊椎动物、昆虫等的细胞。节肢动物细胞的非限制性实例包括昆虫细胞,例如草地夜蛾(spodopterafrugiperda)(sf)细胞,如sf9、sf21、粉纹夜蛾(trichoplusiani)细胞和果蝇s2细胞。可用作本发明的宿主细胞的细菌细胞的实例是聚球藻(synechococcus)(也称集胞藻(synechocystis)),例如细长聚球藻(synechococcuselongatus)。所述细胞可来自适合发酵方法的生物体。在本文中,术语"发酵方法"指的是任何发酵方法或任何包括发酵步骤的方法。发酵方法包括,但不限于,用于产生醇类(例如,乙醇、甲醇、丁醇);有机酸类(例如,柠檬酸、乙酸、甲叉丁二酸、乳酸、葡糖酸);酮类(例如,丙酮);氨基酸(例如,谷氨酸);气体(例如,h2和co2);抗生素(例如,青霉素和四环素);酶;维生素(例如,核黄素、β-胡罗卜素);和激素的发酵方法。发酵方法还包括消费性酿酒业(例如啤酒和葡萄酒)、乳品业(例如发酵乳产品)、皮革业和烟草业所使用的发酵方法。优选的发酵方法包括醇类发酵方法,这是本领域人员所熟知的。优选的发酵方法是厌氧发酵方法,这是本领域人员所熟知的。合适的发酵细胞,典型地是微生物,能够将糖例如葡萄糖或麦芽糖直接或间接发酵为(即转换为)所需的发酵产物。发酵微生物的实例包括真菌生物体,例如酵母。在本文中,“酵母”包括酵母属(saccharomycesspp.),酿酒酵母(saccharomycescerevisiae)、卡尔酵母(saccharomycescarlbergensis)、假丝酵母、克鲁维酵母、毕赤酵母属物种、汉逊酵母、木霉、斯氏油脂酵母(lipomycesstarkey)和解脂耶氏酵母(yarrowialipolytica)。优选的酵母包括酵母属物种的菌株,特别是酿酒酵母。转基因植物本发明还提供包含本发明的细胞的植物,例如包含一或多种本发明的多核苷酸的转基因植物。术语"植物"在本文中作为名词指的是整个植物,但作为形容词使用时指的是存在于植物中的、自植物获得的、来源于植物的或与植物有关的任何物质,例如,植物器官(例如,叶、茎、根、花)、单个的细胞(例如,花粉)、种子、植物细胞等等。术语"植物部分"指的是所有包括植物dna的植物部分,包括营养性结构(vegetativestructures)例如,叶或茎、根、开花器官或结构、花粉、种子、种子部分例如胚、胚乳、盾片或种皮、植物组织例如维管组织、以及它们的细胞和子代。"转基因植物"、"遗传学修饰的植物"或其变化形式指的是这样的植物,其含有基因构建体(“转基因”),该基因构建体在同样物种、品种或栽培变种的野生型植物中是不存在的。就本发明而言,在此所述的转基因植物包括采用重组技术进行遗传学改造以便在所需植物或植物器官中产生至少一种本文所述的多肽的植物及其子代。转基因植物细胞和转基因植物部分也具有相应的含义。“转基因”在此具有生物
技术领域:
的通常含义,并包括通过重组dna或rna技术而产生或改变并被引入至本发明的细胞、优选地植物细胞内的遗传学序列。转基因包括的遗传序列可来源于与引入该转基因的植物细胞属于同样物种、品种或栽培变种的植物细胞,或者来源于不同的物种、品种或栽培变种的植物细胞,或是来源于非植物细胞。典型地,转基因已经通过人为操作而引入细胞例如植物,例如通过转化,但也可采样本领域人员所知的任何方法。术语"种子"和"籽粒"在此可互换使用。“籽粒”指的是成熟的籽粒例如收获的籽粒或虽然仍在植物上但已适合收获的籽粒,不过,根据上下文也可指吸涨或萌芽后的籽粒。成熟籽粒具有的含水量通常低于大约18-20%。“发育中的种子”在本文中指的是成熟之前的种子,典型地见于授粉后或开花期后的植物繁殖结构,不过也可指与植物分离的成熟之前的此类种子。在本文中,术语“植物贮藏器官”指的是植物专门用于以例如蛋白质、碳水化合物、脂肪酸和/或油的形式储藏能量的部分。植物贮藏器官的实例是种子、果实、块状根和块茎。本发明优选的植物贮藏器官是种子。在本文中,术语“具有正常的表型”指的是,本发明的遗传学改造的植物或植物器官,特别是贮藏器官例如种子、块茎或果实,与未改造的植物或植物器官相比,其生长和繁殖能力没有显著降低。在一个实施方式中,所述具有正常的表型的遗传学改造的植物或植物器官包含可操纵地连接于植物贮藏器官特异性启动子的编码沉默阻抑物的外源性多核苷酸,并与不包含所述多核苷酸的同基因植物或器官具有基本上相同的生长或繁殖能力。优选地,如果生长在相同的条件下,所产生的生物量、生长速度、萌发率、贮藏器官大小、种子大小和/或活种子数量,与缺乏所述外源性多核苷酸的植物相比,不低于后者的90%。该术语不包括植物的如下特征,所述特征可能不同于野生型植物,但并不影响植物的商业目的用途,例如,幼苗叶子的芭蕾舞短裙表型(ballerinaphenotype)。本发明所提供的或者预期可用于本发明的植物包括单子叶植物和双子叶植物。在优选的实施方式中,本发明的植物是农作物(例如,谷类和豆类、玉蜀黍、小麦、马铃薯、木薯(tapioca)、稻、蜀黍、粟、木薯、大麦、或豌豆)或其他豆科植物。可种植植物用于产生可食用的根、块茎、叶、茎、花或果实。植物可以是蔬菜或观赏植物。本发明的植物可以是:玉米(zeamays)、芸苔(甘蓝型油菜(brassicanapus)、芜菁(brassicarapassp.))、亚麻(linumusitatissimum)、紫花苜蓿(medicagosativa)、稻(oryzasativa)、黑麦(secalecerale)、蜀黍(sorghumbicolour、sorghumvulgare)、向日葵(helianthusannus)、小麦(tritiumaestivum)、大豆(glycinemax)、烟草(nicotianatabacum)、马铃薯(solanumtuberosum)、花生(arachishypogaea)、棉花(gossypiumhirsutum)、蕃薯(lopmoeabatatus)、木薯(manihotesculenta)、咖啡(cofeaspp.)、椰子(cocosnucifera)、凤梨(ananacomosus)、柑橘(citrusspp.)、可可(theobromacacao)、茶(camelliasenensis)、香蕉(musaspp.)、鳄梨(perseaamericana)、无花果(ficuscasica)、番石榴(psidiumguajava)、芒果(mangiferindica)、橄榄(oleaeuropaea)、木瓜(caricapapaya)、腰果(anacardiumoccidentale)、夏威夷果(macadamiaintergrifolia)、杏仁(prunusamygdalus)、糖用甜菜(betavulgaris)、燕麦、或大麦。在优选的实施方式中,植物是被子植物。在一个实施方式中,植物是油料种子植物,优选地是油料种子农作物植物。在本文中,"油料种子植物"是用于从植物的种子中商品化生产油的植物物种。油料种子植物可以是油料种子油菜(例如芸苔)、玉蜀黍、向日葵、大豆、蜀黍、亚麻(亚麻子)或糖用甜菜。此外,油料种子植物可以是其他的芸苔属(brassicas)、棉花、花生、罂粟、芥菜、蓖麻子、芝麻、红花、或产生坚果的植物。植物在其果实中可产生高水平的油,例如橄榄、油椰子或椰子。本发明可使用的园艺植物是莴苣、菊苣、或芸苔属蔬菜,包括甘蓝、椰菜、或花椰菜。本发明可用于烟草、葫芦、胡萝卜、草莓、番茄或胡椒。在另一个优选的实施方式中,用于产生本发明的转基因植物的非转基因植物(特别是在其种子中)产生的油i)具有低于20%、低于10%或低于5%的18:2脂肪酸和/或ii)具有低于10%或低于5%的18:3脂肪酸。在优选的实施方式中,转基因植物对于已经被引入的每个基因(转基因)均是纯合子,这样其子代的所需表型不会出现分离。转基因植物对于引入的转基因也可以是杂合子,优选地对于所述转基因均一地是杂合子,例如,在从杂交种子生长而来的f1子代中即是如此。此类植物可具有一些优点,例如本领域熟知的杂种优势。在相关的情况中,转基因植物还可包含额外的转基因,所述额外的转基因编码参与产生lc-pufa的酶,例如,但不限于,δ6去饱和酶、δ9延长酶、δ8去饱和酶、δ6延长酶、δ5去饱和酶、ω3去饱和酶、δ4去饱和酶、δ5延长酶、二酰基甘油酰基转移酶、δ17去饱和酶、δ15去饱和酶和/或δ12去饱和酶。具有一或多种这些活性的此类酶的实例是本领域已知的,包括本文和wo05/103253中所述那些(见,例如,wo05/103253的表1)。在具体的实例中,转基因植物至少包含编码以下各项的外源性多核苷酸:a)δ4去饱和酶、δ5去饱和酶、δ6去饱和酶、δ5延长酶和δ6延长酶,b)δ4去饱和酶、δ5去饱和酶、δ8去饱和酶、δ5延长酶和δ9延长酶,c)δ4去饱和酶、δ5去饱和酶、δ6去饱和酶、δ5延长酶、δ6延长酶和δ15去饱和酶,d)δ4去饱和酶、δ5去饱和酶、δ8去饱和酶、δ5延长酶、δ9延长酶和δ15去饱和酶,e)δ4去饱和酶、δ5去饱和酶、δ6去饱和酶、δ5延长酶、δ6延长酶和δ17去饱和酶,或f)δ4去饱和酶、δ5去饱和酶、δ8去饱和酶、δ5延长酶、δ9延长酶和δ17去饱和酶。植物的转化可通过本领域已知的技术产生转基因植物,这些技术例如可参见a.slater等,plantbiotechnology-thegeneticmanipulationofplants,oxforduniversitypress(2003)和p.christouandh.klee,handbookofplantbiotechnology,johnwileyandsons(2004)。在本文中,术语“稳定转化”、“稳定转化的”及其变化形式指的是外源核酸分子整合至细胞的基因组内,由此在细胞分裂过程中传递给子代,而无需对其进行阳性选择。可通过本领域已知的任何手段选择稳定的转化体或其子代,例如在染色体dna上进行southern印迹或基因组dna的原位杂交。土壤杆菌介导的转移是将基因引入植物细胞的广泛适用的系统,这是因为dna可被引入整个植物组织或植物器官或培养的外植体的细胞内,以便瞬时表达或dna稳定整合至植物细胞基因组内。使用土壤杆菌介导的植物整合载体将dna引入植物细胞是本领域熟知的(参见,例如,us5177010、us5104310、us5004863或us5159135)。待转移的dna区域由边界序列限定,干预dna(t-dna)通常被插入至植物基因组内。此外,t-dna的整合是相对精确的过程,其极少产生重排。鉴于其基因转移的简便和确定性,在有效发生土壤杆菌介导的转化的植物品种中成为首选的方法。优选的土壤杆菌转化载体能够在大肠杆菌以及土壤杆菌中复制,因此操作简便(klee等,in:plantdnainfectiousagents,hohnandschell,eds.,springer-verlag,newyork,pp.179-203(1985)。可使用的加速方法包括,例如,微粒轰击等等。将转化核酸分子输送至植物细胞内的方法的一个实例是微粒轰击。该方法的综述见yang等,particle轰击technologyforgenetransfer,oxfordpress,oxford,england(1994)。非生物学颗粒(微粒)可包被上核酸并通过推进力输送至细胞内。示例性的颗粒包括含钨、金、铂等的颗粒。其除了是可重复性地转化单子叶植物的有效手段之外,微粒轰击的特别优势还在于,既不需要分离原生质体,也不需要对土壤杆菌感染的易感性。通过加速而将dna输送至玉蜀黍细胞内的方法的一个示例性实施方式是生物弹道学α-颗粒输送系统,其能够驱动包被有dna的颗粒通过一个屏,例如不锈钢或nytex屏,到达以悬浮培养的玉黍蜀细胞所覆盖的滤膜表面上。适合用于本发明的颗粒输送系统是氦加速枪pds-1000/he,可自bio-radlaboratories获得。为进行轰击,可将悬浮的细胞浓缩于滤膜上。将含有待轰击细胞的滤膜放置在距微粒停止板下方合适的距离处。需要的话,还可在枪和待轰击细胞之间放置一或多个屏。或者,可将不成熟的胚或其他靶细胞设置在固相培养基上。待轰击细胞放置在距微粒停止板下方适当距离处。如果需要,还可在加速装置和待轰击细胞之间放置一或多个屏。通过在此所述的技术可获得1000或更多个瞬时表达标记物基因的细胞焦点。轰击后48小时焦点内表达外源基因产物的细胞数量在1到10个,平均为1到3个。在轰击转化中,可优化轰击前培养条件和轰击参数,以产生最大数量的稳定转化体。轰击的物理学和生物学参数对于该技术均十分重要。物理因素涉及操控dna/微粒沉淀物或影响大颗粒或微粒的飞行和速率的因素。生物学因素包括涉及在轰击之前和轰击后即刻对细胞的操控步骤、调节靶细胞的渗透压以便减轻与轰击相关的损伤、以及转化dna的性质,例如是线性化dna还是完好的超螺旋质粒。轰击前操控被认为对于成功转化未成熟胚而言是特别重要的。在另一个替代性实施方式中,可稳定转化质体。在高等植物中转化质体的已知方法包括用颗粒枪输送含有选择标记物的dna并通过同源重组将dna导向质体基因组(us5,451,513、us5,545,818、us5,877,402、us5,932479和wo99/05265)。因此,预期可以的是,可通过在小规模的研究中调整轰击参数的各个方面以便充分优化条件。特别希望调整的是物理参数,例如间隙距离、飞行距离、组织距离和氦气压力。还可通过改变影响受者细胞的生理学状态并因此可影响转化和整合效率的条件以使得降低损伤因素最小化。例如,可调节渗透压状态、组织水合和受者细胞的亚培养阶段或细胞周期以达到最佳转化。根据在此公开的内容,本领域人员知晓如何进行其他常规的调整。可采用基于磷酸钙沉淀、聚乙二醇处理、电穿孔以及这些处理的组合的方法实现植物原生质体的转化。如何将这些系统应用于不同植物品种取决于自原生质体再生出特定植株的能力。自原生质体再生谷类的示例性方法已被公开(fujimura等,1985;toriyama等,1986;abdullah等,1986)。还可采用其他的细胞转化方法,包括但不限于通过以下方法将dna引入植物:直接将dna转移至花粉,直接将dna注射至植物的繁殖性器官,或直接将dna注射至未成熟胚的细胞内随后对干燥的胚进行再水化。从单个植物原生质体转化体或各种转化的外植体再生、发育和栽培出植物是本领域熟知的(weissbach等,in:methodsforplantmolecularbiology,academicpress,sandiego,calif.,(1988)。这种再生和生长方法典型地包括以下步骤:选择转化的细胞,培养个体化细胞历经从胚发育至有根的小植株阶段的通常阶段。转基因胚和种子的再生类似。得到的转基因的有根的苗随后栽植在合适的植物生长培养基例如土壤中。含有外来的外源基因的植物的发育或再生是本领域熟知的。优选地,再生的植物自花传粉以产生纯合子转基因植物。或者,将得自再生的植物的花粉与由具有重要农艺价值的株系的种子长成的植物进行杂交。反过来,可使用来自这些重要株系的植物的花粉给再生的植物授粉。可采用本领域人员熟知的方法栽培含有所需外源核酸的本发明的转基因植物。主要采用根癌土壤杆菌转化双子叶植物以及获得转基因植物的方法已经用于棉花(美国5,004,863、美国5,159,135、美国5,518,908);大豆(美国5,569,834、美国5,416,011);芸苔属(美国5,463,174);花生(cheng等,1996);和豌豆(grant等,1995)。转化谷类植物例如小麦和大麦以便通过引入外源核酸而在植物中引入遗传变异以及用于从原生质体或未成熟的植物胚再生植物的方法是本领域熟知的,参见例如,ca2,092,588、au61781/94、au667939、us6,100,447、pct/us97/10621、us5,589,617、us6,541,257,其他方法参见专利说明书wo99/14314。优选地,通过根癌土壤杆菌介导的转化方法产生转基因小麦或大麦植物。携带所需核酸构建体的载体可被引入组织培养的植物或外植体的可再生的小麦细胞内、或合适的植物系统例如原生质体中。可再生的小麦细胞优选地来自未成熟胚、成熟胚、来自它们的愈伤组织、或分生组织的盾片。为了证实转基因细胞和植物内存在转基因,可采用本领域人员已知的方法进行聚合酶链反应(pcr)扩增或southern印迹分析。根据产物的性质,可通过多种途径检测转基因表达产物,包括免疫印迹和酶分析。在不同植物组织中定量蛋白质表达和检测复制的一种特别有用的方法是使用报告基因,例如gus。一旦获得转基因植物,可使之生长产生具有所需表型的植物组织或部分。可收获植物组织或植物部分和/或收集种子。种子可用作来源用于生长出具有所需特性的额外的植物、组织或部分。采用土壤杆菌或其他转化方法形成的转基因植物典型地含有位于一条染色体上的单个基因座。此类转基因植物可被称为所添加的基因的半合子。更优选的转基因植物是所添加的基因的纯合子;即,转基因植物含有两个所添加的基因,一对染色体的每一条的相同基因座上具有一个基因。可通过半合子转基因植物自花授粉,使所得种子发芽,并分析所得植物中的感兴趣的基因,从而获得纯合子转基因植物。还要理解,含有两种独立地分离外源基因或基因座的两种不同的转基因植物可杂交,以产生含有两套基因或基因座的子代。合适f1子代的自花授粉可产生两种外源基因或基因座均为纯合子的植物。也预期了与亲代植物进行回交以及与非转基因植物进行异系杂交,以及营养体繁殖。常规采用的对不同性状和作物的其他育种方法可参见fehr,in:breedingmethodsforcultivardevelopment,wilcoxj.ed.,americansocietyofagronomy,madisonwis.(1987)。转基因非人类动物"转基因非人类动物"指的是动物而非人类,所述动物含有同一物种或品种的野生型动物所不具有的基因构建体(“转基因”)。在此所述的“转基因”具有生物
技术领域:
内的通常含义,且包括这样的遗传学序列,所述序列是经重组dna或rna技术产生或改变的且已经被引入至动物细胞内。转基因可包括来源于动物细胞的遗传学序列,所述动物细胞与待引入转基因的细胞可以是相同的物种或不同的物种。典型地,转基因已经通过人为操作引入至动物内,例如,通过转化,但也可通过本领域人员所知的任何方法。产生转基因动物的技术是本领域熟知的。该方面的通用教科书是houdebine,transgenicanimals–generationanduse(harwoodacademic,1997)。可通过任何方法将多核苷酸分子转化至细胞内,所述方法可将多核苷酸分子插入至细胞内。转化技术包括,但不限于,转染、电穿孔、微注射、脂染、吸附和原生质体融合。重组细胞可保持单细胞状态或可生长为组织、器官或多细胞生物体。转化的多核苷酸分子可保持在染色体外或可整合至被转化(即,重组)细胞的染色体内的一或多个位置,使得其能够被表达。异源dna可被引入至例如哺乳动物的受精卵内。例如,可通过微注射、磷酸钙介导的沉淀、脂质体融合、反转录病毒感染或其他手段转化全能或多能干细胞,然后将转化的细胞引入胚胎,并使胚胎发育为转基因动物。在高度优选的方法中,使用含有所需dna的反转录病毒感染发育的胚胎,并自感染的胚胎产生转基因动物。不过,在最优选的方法中,合适的dna被共注射至前核或胚胎的细胞质内,优选地在单个细胞阶段,并使胚胎发育为成熟的转基因动物。产生转基因动物的另一方法涉及通过标准的方法将核酸微注射至原核期卵内。然后培养被注射的卵,随后转移至假孕受者的输卵管内。还可通过核移植技术产生转基因动物。采用这种方法,用质粒稳定转染来自供者动物的成纤维细胞,该质粒整合有置于调控序列控制下的感兴趣的结合结构域或结合区的编码序列。然后将稳定的转染体与去核的卵母细胞融合,培养并转移至雌性受者内。提高外源rna水平和稳定的表达沉默阻抑物转录后基因沉默(ptgs)是核苷酸序列特异性防卫机制,能够将细胞的和病毒的mrna靶向降解。ptgs出现在以外来(外源性)或内源性dna稳定或瞬时转化植物或真菌,导致与所引入的核酸具有序列相似性的rna分子的积聚被降低。普遍认为共表达沉默阻抑物和感兴趣的转基因可升高细胞内由转基因转录而来的rna的水平。尽管这在体外证明是正确的,但在许多整体植物共表达研究中已经发现其具有显著的副作用。更具体地,如mallory等(2002)、chapman等(2004)、chen等(2004)、dunoyer等(2004)、zhang等(2006)、lewsey等(2007)和meng等(2008)所述,表达沉默阻抑物(通常置于组成型启动子控制下)的植物通常具有异常的表型,其程度导致它们无法用于商业生产。如上所述,本发明人发现,可通过将沉默阻抑物的表达局限于植物的贮藏器官或其部分而提高rna分子水平和/或使得rna分子水平在许多世代内保持稳定。在本申请中,“沉默阻抑物”是任何如下所述的多核苷酸或多肽,其能够在植物细胞中表达并提高来自植物细胞内的不同转基因的表达产物的水平,特别是经过从最初转化的植物反复传代之后。在一个实施方式中,所述沉默阻抑物是病毒沉默阻抑物或其突变体。本领域已知大量病毒沉默阻抑物,包括,但不限于,p19、v2、p38、pe-po和rpv-p0。在一个实施方式中,所述病毒沉默阻抑物包含seqidno97至101任一所示的氨基酸序列、其生物学活性片段、或与seqidno97至101中的任何一或多种序列具有至少50%相同性的氨基酸序列,并具有沉默阻抑物的活性。在本文中,术语“使表达稳定”、“稳定表达的”、“稳定表达”及其变化形式指的是,经反复传代后(例如至少3代、至少5待或至少10代)的子代植物中的rna分子的水平与缺乏编码所述沉默阻抑物的外源性多核苷酸的同基因植物相比,基本上是相同的或更高。但是,该术语不排除经反复传代后rna分子的水平与前一代相比有所下降这一可能性,例如每一代损失不低于10%。阻抑物可选自任何来源,例如植物、病毒、哺乳动物等。阻抑物可以是,例如:●兽棚病毒b2;●绿萝潜隐病毒p14;●绿萝潜隐病毒ac2;●非洲木薯花叶病毒ac4;●秋葵黄脉花叶病c2;●秋葵黄脉花叶病c4;●秋葵黄脉花叶病βc1;●番茄褪绿病毒p22;●番茄褪绿病毒cp;●番茄褪绿病毒cpm;●番茄金色花叶病毒al2;●tomatoleafcurljavavirusβc1●番茄黄曲叶病毒v2;●番茄黄曲叶病毒-中国c2●番茄黄曲叶中国病毒y10分离株βc1;●番茄黄曲叶病毒以色列分离株v2;●绿豆黄花叶病毒-豇豆ac2;●木槿褪绿环斑病毒cp;●芜菁皱病毒p38;●芜菁皱病毒cp;●花椰菜花叶病病毒p6;●甜菜黄化病病毒p21;●柑橘腐根病病毒p20;●柑橘腐根病病毒p23;●柑橘腐根病病毒cp;●豇豆花叶病毒scp;●甘薯褪绿矮缩病毒p22●黄瓜花叶病毒2b;●番茄不孕病毒hc-pro●甜菜卷顶病毒l2;●土传小麦花叶病毒19k;●大麦条斑花叶病毒gammab;●poasemilatentvirusgammab;●花生丛簇病毒p15;●稻米矮小病毒pns10;●curubitaphidborneyellowsvirusp0;●甜菜西部黄化病毒p0;●马铃薯病毒xp25;●黄瓜脉黄化病毒p1b;●李痘病毒hc-pro;●甘蔗花叶病毒hc-pro●马铃薯病毒ystrainhc-pro;●烟草蚀纹病毒p1/hc-pro;●芜菁花叶病毒p1/hc-pro;●鸭茅斑点病毒p1;●鸭茅斑点病毒-挪威分离株p1●水稻黄斑点病毒p1;●水稻黄斑点病毒-尼日利亚分离株p1;●水稻白叶病毒ns3●稻条纹病毒ns3●十字花科感染烟草花叶病毒126k;●十字花科感染烟草花叶病毒p122;●烟草花叶病毒p122;●烟草花叶病毒126●烟草花叶病毒130k;●烟草脆裂病毒16k;●番茄丛矮病毒p19;●蕃茄斑萎病毒nss;●苹果褪绿叶斑病毒p50;●葡萄黄脉病毒ap10;●葡萄卷叶相关病毒-2byvp21的同源物,及其变体/突变体。以上所列的提供了可由其获得阻抑物的病毒以及来自每一具体病毒的阻抑物的蛋白质(例如,b2、p14等)或编码区命名。可以使用多个拷贝的阻抑物。不同阻抑物可以一起使用(例如,串联)。rna分子基本上任何适合在植物贮藏器官中表达的rna分子均可与沉默阻抑物共表达。rna分子可影响农业性状、抗虫性、抗病性、除草剂抗性、不育性、籽粒特征等等。所编码的多肽可参与油、淀粉、碳水化合物、营养物等的代谢,或负责合成蛋白质、肽、脂肪酸、脂质、蜡、油、淀粉、糖、碳水化合物、香料、气味(odor)、毒素、类胡罗卜素、激素、聚合物、类黄酮、贮藏蛋白、酚酸、生物碱、木质素、丹宁酸、纤维素、糖蛋白、糖脂等等。在具体的实例中,所述植物中以下的酶或蛋白质的水平是升高的:在植物(例如为芸苔、例如油料种子油菜或向日葵、红花、亚麻、棉、大豆或玉米)中产生油的酶;参与植物(例如马铃薯、玉米和谷物例如小麦、大麦或稻)中淀粉合成的酶;合成药物的酶或其本身即是天然药物的蛋白质,例如药物或兽医产品。预期以本发明的方法产生的多肽的类型包括:用于包括人在内的哺乳动物类的药用蛋白质,例如胰岛素、前胰岛素原、胰岛素原、胰高血糖素、干扰素例如α-干扰素和γ-干扰素、凝血因子例如因子vii、viii、ix、x、xi和xii、生育激素例如黄体生成素、卵泡刺激素、生长因子例如表皮生长因子、血小板衍生的生长因子、粒细胞集落刺激因子、泌乳刺激素、催产素、促甲状腺激素、促肾上腺皮质激素、降钙素、甲状旁腺激素、生长激素抑制素、促红细胞生成素(epo)、酶例如β-葡糖脑苷脂酶、血红蛋白、血清白蛋白、胶原、生长激素、人血清白蛋白、人分泌的碱性磷酸酶、抑肽酶、α1-抗胰蛋白酶、igg1(膦酸酯)、igm(神经肽半抗原)、siga/g(变异链球菌(streptococcusmutans)外源凝集素)、scfv-bryodin1免疫毒素(cd40)、igg(hsv)、lsc(hsv)等等。此外,本发明的方法也可用于产生特异性抗体,包括抗体相关性分子或其活性片段,它们结合,例如,骨形态生成蛋白受体ib型;el6;steapl;mpf;napi3b;sema5b;psca;b型内皮缩血管肽受体;msg783;steap2;trpm4;cripto;cd21;cd79b;fcrh2;her2;nca;mdp;il20rα;短蛋白聚糖(brevican);ephb2r;aslg659;psca;geda;b细胞活化因子受体;cd22;cd79a;cxcr5;hla-dob;p2x5;cd72;ly64;fcrhl;irta2;tenb2;cd20;vegf包括vegf_a、b、c或d;p53;egfr;孕激素受体;组织蛋白酶d;bcl-2;e钙粘蛋白;cea;lewisx;ki67;pcna;cd3;cd4;cd5;cd7;cd11c;cd11d;c-myc;tau;prpsc;或aβ。此外,本发明的方法也可用于产生抗原,所述抗原可通过或不通过摄入所述贮藏器官而输送,抗原的实例包括乙型肝炎病毒包膜蛋白、狂犬病毒糖蛋白、大肠杆菌热不稳定肠毒素、norwalk病毒衣壳蛋白、糖尿病自身抗原、霍乱毒素b亚单位、霍乱毒素b和a2亚单位、轮状病毒肠毒素和产肠毒素大肠杆菌菌毛抗原融合物、猪传播性胃肠炎病毒糖蛋白s、人类鼻病毒15(hrv-14)和1型人免疫缺陷病毒(hiv-1)表位、貂肠炎病毒表位、口蹄疫病毒vp1结构蛋白、人巨细胞病毒糖蛋白b、龋齿(s.mutans)抗原和呼吸道合胞病毒抗原。产生的lc-pufa的水平重组细胞中产生的lc-pufa或lc-pufa的组合的水平十分重要。所述水平可以表示为特定lc-pufa或一组相关的lc-pufa(例如ω3lc-pufa或ω6lc-pufa)、或vlc-pufa、或其他占总脂肪酸的组成(百分比),这可通过本领域已知的方法确定。所述水平还可表示为lc-pufa的含量,例如lc-pufa占包含所述重组细胞的材料干重的百分比,例如lc-pufa占种子干重的百分比。可以理解,就lc-pufa的含量而言,油料种子中产生的lc-pufa可以显著高于不为产油而生长的蔬菜或籽粒,但两者可具有相似的lc-pufa组成,且两者均可用作人或动物摄入lc-pufa的来源。可通过本领域已知的任何方法确定lc-pufa的水平。在优选的方法中,自细胞、组织或生物体提取总脂质,并将脂肪酸转换为甲基酯,随后通过气相色谱(gc)进行分析。此类技术见实施例1。色谱的峰位置可用来鉴定各特定脂肪酸,而各峰下的面积求积分后应用于确定量。在本申请中,除非另有说明,样品中的特定脂肪酸的百分比是按照该脂肪酸的峰下的面积占色谱中的脂肪酸总面积的百分比确定的。这基本上对应于重量百分比(w/w)。脂肪酸的鉴定可通过gc-ms加以确认。总脂质可以通过本领域已知的纯化组份(例如tag组份)的技术进行分离。例如,可进行分析尺度的薄层色谱(tlc)从而将tag与其他脂质组份例如dag、酰基-coa或磷脂分离,以便确定tag具体的脂肪酸组成。在一个实施方式中,所述细胞内脂肪酸中的ara、epa、dpa和dha总计占所述细胞内的总脂肪酸的至少15%,更优选地至少20%或至少25%。在更加优选的实施方式中,这些脂肪酸的总和占所述细胞内的总脂肪酸的至少29%、至少30%或至少31%。在另一个实施方式中,所述细胞内的总脂肪酸具有低于1%的c20:1。在另一个实施方式中,所述细胞内脂肪酸中的dha的量占所述细胞内的总脂肪酸的至少3%,更优选地至少4%,更优选地至少5%或至少7%,或最优选地至少10%。在优选的实施方式中,细胞内可提取的tag包含本段所提及的水平的脂肪酸。还预期了这些特征的每一种可能的组合。例如,所述细胞内脂肪酸中的ara、epa、dpa和dha总计可占细胞内总脂肪酸的至少15%、至少20%、至少25%、至少29%、至少30%或至少31%,其中至少3%、至少4%、至少5%、至少7%或至少10%的细胞内总脂肪酸是dha,而c20:1的水平可低于1%。在这些实施方式的每一实施方式中,重组细胞可以是适合发酵的生物体的细胞,例如,单细胞微生物,其可以是原核生物或真核生物,例如酵母或植物细胞。在优选的实施方式中,细胞是被子植物(高等植物)细胞。在进一步优选的实施方式中,细胞是种子内的细胞,例如,油料种子或籽粒或谷类。重组细胞内lc-pufa产生的水平还可以转换率表示,即所形成的lc-pufa的量占一或多种底物pufa或lc-pufa的百分比。例如,就epa而言,可以表示为epa的水平(占总脂肪酸的百分比)与底物脂肪酸(ala、sda、eta或etra)水平的比率。在一个实施方式中,ala转换为epa的转换效率是至少80%或更优选地至少90%。在另一个实施方式中,ala转换为epa、dpa或dha(以epa、dpa和dha的百分比的总和/ala和ala经δ6-去饱和的所有脂肪酸产物的百分比的总和计算)的转换效率是至少17.3%、或至少23%。在另一个实施方式中,ala转换为dpa或dha(以dpa和dha的百分比的总和/ala和ala经δ6-去饱和的所有脂肪酸产物的百分比的总和计算)的转换效率是至少15.4%、或至少21%。在另一个实施方式中,ala转换为dha(以dha的百分比/ala和ala经δ6-去饱和的所有脂肪酸产物的百分比的总和计算)的转换效率是至少9.5%、或至少10.8%。在另一个实施方式中,epa转换为dha(以dha的百分比/epa和epa经δ5-延长的所有脂肪酸产物的百分比的总和计算)的转换效率是至少45%、或至少50%。在另一个实施方式中,转换sda产生eta(以eta和eta经δ5-去饱和的所有脂肪酸产物的百分比的总和/sda和sda经δ6-延长的所有脂肪酸产物的百分比的总和计算)的转换效率是至少50%,更优选地至少60%。在另一个实施方式中,ala转换为etra的转换效率是至少6%,更优选地至少9%。在另一个实施方式中,通过δ5延长酶步骤将epa转换为dpa(以dpa和dha的百分比的总和/epa、dpa和dha的百分比的总和计算)的转换效率是至少60%,更优选地至少65%,更优选地至少70%或最优选地至少75%。如果用于引入这些基因的亲代细胞被选择为使得所产生的或外源提供的脂肪酸底物的水平达到最佳,则可以使得重组细胞中的lc-pufa含量最大化。可以通过将细胞在最佳条件下生长或温育而使得lc-pufa水平最大化,例如在略低于该细胞的标准温度的温度下生长,认为这样有利于多不饱和脂肪酸的积聚。不过,目前的证据提示,在酵母或植物中外源表达的某些去饱和酶与某些延长酶组合时具有相对低的活性。可通过提供在lc-pufa合成过程中能够利用酰基-coa形式的脂肪酸作为底物的去饱和酶来改善这一点,这在酵母以外的其他重组细胞例如植物细胞中被认为是有利的。油的产生可采用本领域常规使用的技术来提取、加工和分析由本发明的细胞、植物、种子等产生的油。典型地,植物种子经熟化、压榨和提取以产生粗制油,然后进行脱胶、精制、脱色和除味。通常,种子压碎技术是本领域已知的。例如,给油料种子喷水使之变软,并使之含水量升高至,例如8.5%,使用间隙设置为0.23至0.27mm的平滑滚筒将其制成薄片。根据种子的类型,在压碎之前可以不加水。加热可使酶失活,且有助于细胞进一步破坏,使油滴凝结,并使蛋白质凝聚,所有这些均有助于提取加工。通过螺旋压榨机可使得大部分种子油释放出来。从螺旋压榨机排出的饼随后进行溶剂提取,例如以己烷,使用伴热柱(heattracedcolumn)。或者,压榨工序所产生的粗制油可通过沉降罐,其上部具有槽状金属丝网引流构造,以去除压榨工序过程中随油而挤出的固形物。净化的油再通过板框式滤器,以去除任何残余的微小固形颗粒。如果需要,自提取工序所回收的油可与净化的油混合以产生掺合的粗制油。将溶剂从粗制油中去除后,将压榨部分和提取部分混合饼对其进行常规的油加工过程(即,脱胶、碱法净化、脱色和除味)。可通过向粗制油中添加浓磷酸进行脱胶,将不可水合的磷脂转化为可水合形式,并螯合存在的少量金属。通过离心将胶与油分离。可通过添加足量的氢氧化钠溶液滴定全部脂肪酸并移除所形成脂肪酸盐,以精制油。可通过在真空条件下将油加热至260℃并以0.1ml/分钟/100ml油的速度缓慢向油中引入蒸汽而进行除味。喷射30分钟后,将油在真空中冷却。典型地,油被转移至玻璃容器并充入氩气,然后冷藏。如果油量有限,则可置于真空条件下,例如parr反应器内,并加热至260℃,加热时间与用于除味所需的时间一样。这一处理可改善油的颜色并去除绝大部分的挥发性物质。饲料(feedstuffs)本发明包括可用作饲料的组合物。在本发明中,"饲料"包括供人类或动物摄入(包括用于胃肠道和/或胃肠外摄入)的任何食物或制品,当其进入肌体后,(a)用于营养或强健组织或提供能量;和/或(b)保持、恢复或支持足够的营养状态或代谢功能。本发明的饲料包括婴儿和/或幼儿的营养组合物。本发明的饲料包含,例如,本发明的细胞、本发明的植物、本发明的植物部分、本发明的种子、本发明的提取物、本发明的方法的产物、本发明的发酵方法的产物、或本发明的组合物,以及合适的载剂。术语"载剂"取其最为广泛的含义,涵盖具有或不具有营养价值的任何成分。本领域人员能够理解,载剂必须适合于用于饲料(或以足够低的浓度使用),以便其不会对摄入所述饲料的生物体产生有害作用。本发明的饲料包含使用本发明的方法、细胞或植物直接或间接产生的油、脂肪酸酯或脂肪酸。组合物可以是固体或液体的形式。此外,组合物可包括可食用的常量营养物、维生素、和/或矿物质,它们的量符合特定的需要。这些成分的量可以变化,这取决于组合物是用于正常个体还是用于具有特殊需求的个体,例如患有代谢性疾病和类似情况的个体。具有营养价值的合适的载剂的实例包括,但不限于,常量营养物例如可食用的脂肪、糖类和蛋白质。此类可食用的脂肪的实例包括,但不限于,椰子油、琉璃苣油、真菌油、黑醋栗油、大豆油和甘油一酸酯和甘油二酯。此类糖类的实例包括(但不限于):葡萄糖、食用乳糖、和水解淀粉。此外,可用于本发明的营养组合物中的蛋白质的实例包括(但不限于):大豆蛋白、电渗析乳清、电渗析脱脂乳、乳乳清、或这些蛋白质的水解产物。至于维生素和矿物质,以下物质可添加至本发明的饲料组合物中:钙、磷、钾、钠、氯、镁、锰、铁、铜、锌、硒、碘和维生素a、e、d、c、和复合b。其他的此类维生素和矿物质也可添加。用于本发明的饲料组合物的成分可以是半纯化来源的或纯化来源的。半纯化来源的或纯化来源的指的是通过对天然材料进行纯化而制备的物质或通过从头合成而制备的物质。本发明的饲料组合物也可添加至食物中,即使是在不需要饮食增补的情况下也可以。例如,组合物可添加至任何类型的食物中,包括(但不限于):人造黄油、改性黄油、奶酪、乳、酸奶、巧克力、糖果、点心、色拉油、烹饪用油、烹饪用脂肪、肉、鱼和饮料。酵母属物种用于酿造啤酒和葡萄酒以及烘焙,特别是烘制面包。酵母是植物提取物的主要成分。酵母也用作动物饲料的添加剂。显然,可产生遗传工程化的酵母菌株使之适合于合成在此所述的lc-pufa。这些酵母菌株随后可用于食品以及葡萄酒和啤酒酿造,以便提供具有增加的脂肪酸含量的产品。此外,根据本发明产生的脂肪酸或经转化后含有并表达对象基因的宿主细胞也可用作动物食物增补剂,以改变动物组织或乳的脂肪酸组成,使之更适合人类或动物摄入。此类动物的实例包括羊、牛、马等等。此外,本发明的饲料还可用于水产业以提高供人类或动物摄入的鱼的脂肪酸水平。本发明的优选的饲料是植物、种子、或其他植物部分,例如叶和茎,它们可以直接用作人或其他动物的食物或饲料。例如,动物可直接在生长此类植物的田间放牧或以更加定量的可控饲养方式饲喂。本发明包括使用此类植物和植物部分作为饲料以提高人和其他动物的lc-pufa水平。组合物本发明还包括组合物,特别是药物组合物,所述组合物包含一或多种采用本发明的方法产生的脂肪酸和/或所得的油。药物组合物可包含一或多种脂肪酸和/或油,连同标准的、熟知的、非毒性药用可接受的载剂、佐剂或载体,例如磷酸缓冲盐溶液、水、乙醇、多元醇、植物油、湿润剂或乳状液,例如水/油乳状液。组合物可以是液体或固体形式。例如,组合物的形式可以是片剂、胶囊、可摄入的液体或粉末、注射形式、或局部用药膏或乳剂。可通过例如保持分散体系所需要的粒径和通过使用表面活性物质而保持合适的流动性。还可加入等渗剂,例如,糖、氯化钠等等。除了此类惰性修饰剂之外,组合物还可包括佐剂,例如湿润剂、乳化和混悬剂、甜味剂、调味剂和芳香剂。混悬液除了含有活性化合物之外还包含混悬剂,例如乙氧基异硬脂醇(ethoxylatedisostearylalcohols)、聚氧乙烯山梨醇酯和聚氧乙烯山梨糖酯、微晶纤维素、偏氢氧化铝(aluminummetahydroxide)、班脱土、琼脂、和黄芪胶或这些物质的混合物。可通过采用本领域熟知的技术制备固体剂量形式例如片剂和胶囊。例如,根据本发明产生的脂肪酸可压成片剂,其中使用常规的片基例如乳糖、蔗糖和玉米淀粉,联合使用的有粘合剂例如阿拉伯树胶、玉米淀粉或白明胶,崩解剂例如马铃薯淀粉或褐藻酸,以及润滑剂例如硬脂酸或硬脂酸镁。可通过将这些赋形剂连同抗氧化剂和相关的脂肪酸装入白明胶胶囊而制备胶囊。对于静脉施用,可将根据本发明产生的脂肪酸或其衍生物掺入至商品化的配制品中。特定脂肪酸的典型剂量是0.1mg至20g,每天摄入1至5次(高达每日100g),且优选地在每天大约10mg至大约1、2、5、或10g的范围内(一次或分多次摄入)。本领域已知,每天需要最少大约300mg的脂肪酸,特别是lc-pufa。不过,应该理解任何量的脂肪酸对对象均可能是有益的。本发明的药物组合物的可能的施用途径包括,例如,胃肠道(例如口服和直肠)和胃肠外。例如,可口服或直肠施用液体制备物。此外,可将均质的混合物完全分散至水中,在无菌条件下与生理学可接受的稀释剂、防腐剂、缓冲剂或推进物混合物,形成喷剂或吸入剂。待施用于患者的组合物的剂量可由本领域人员确定,这取决于多种因素,例如患者的体重、患者的年龄、患者的总体健康状况、患者的既往病史、患者的免疫状态等等。此外,本发明的组合物可用于化妆品。其可添加至已有的化妆品组合物中一般形成混合物,或是将根据本发明产生的脂肪酸用作化妆品组合物中的唯一的“活性”成分。实施例实施例1:材料和方法培养微藻类在标准培养条件下培养来自csiro活微藻保藏中心(csirocollectionoflivingmicroalgae(http://www.marine.csiro.au/microalgae))的微胞藻cs-0170和塔胞藻cs-0140分离物。将来自保藏中心的原培养物在1lerlenmeyer瓶中按照1:10稀释进行次代培养并连续传代扩增,然后转移至10l聚碳酸酯大瓶。培养基为f/2,是改良的guillard和ryther’s(1962)f培养基,含有半量的营养物,生长温度为20±1℃。其他培养条件包括,光密度为100μmol.photonspar.m-2.s-1,12:12小时的亮:暗光周期,含1%co2的空气充入气泡,速度为200ml.l-1.min-1。分离微藻基因组dna采用dneasyplantminikit系统分离微胞藻cs-0170和塔胞藻cs-0140的基因组dna,方法见所附说明书(qiagen,目录#69106)。分离微藻总rna采用以下方法从微胞藻cs-0170和塔胞藻cs-0140细胞分离总rna。用研钵和杵将2g(湿重)细胞在液氮中粉碎,缓慢撒进大口杯中,其中含有持续搅拌的22ml提取缓冲液。向其中加入5%不溶性聚乙烯吡咯烷酮,90mm2-巯基乙醇,和10mm二硫苏糖醇,混合物继续搅拌10分钟,然后转移至corextm管。加入18.4ml的3m醋酸铵,充分混合。然后将样品6000×g离心20分钟,4℃。将上清液转移至新的管中并加入0.1体积的3mnaac(ph5.2)和0.5体积的冷异丙醇沉淀核酸。-20℃孵育1小时,之后将样品在6000×g旋转离心30分钟。将沉淀团垂悬于1ml水中,以酚/氯仿抽提。将水相转移至新的管中,再次加入0.1体积的3mnaac(ph5.2)和2.5体积的冰冷乙醇沉淀核酸。将沉淀团垂悬于水中并通过分光光度计确定核酸浓度。载体和菌株质粒pyes2和酵母株invsc1来自invitrogen,质粒载体pgemt-easy来自promega,质粒载体pbluescriptiiks-来自stratagene。lazo等(1991)提到的根癌土壤杆菌菌株agl1,coutu等(2007)提到的pore二元载体系列。pcr条件除非另有说明,否则采用标准条件聚合酶链反应(pcr)扩增dna片段。通过改变扩增循环的数量、引物退火温度、mg2+浓度和其他参数的条件最佳化,这在本领域是常规的。缓冲液如聚合酶供货商所述。典型地,反应条件如下。94℃初始变性2-3分钟,然后将反应混合物进行20-40个变性/退火/延伸循环,其中在94℃变性30-60秒,40-60℃引物退火30秒,30-60秒聚合酶延伸70-72℃,随后70-72℃再延伸3分钟。典型地,采用superscriptiiione-steprt-pcr系统(invitrogen)进行反转录-pcr(rt-pcr)扩增,体积为25μl,使用10pmol的正向引物和30pmol的反向引物,mgso4终浓度为2.5mm,400ng的总rna,按照生产商的说明使用缓冲液和核苷酸。典型的温度方案为:1个循环的45℃30分钟进行反转录;然后1个循环的94℃2分钟,随后40个循环的94℃30秒、52℃30秒、70℃1分钟;然后1个循环的72℃2分钟,然后将反应混合物冷却至5℃。5’和3’-race为了获得相应于部分长度基因片段的全长cdna,通过5′-和3′-race(rapidamplificationofcdnaends)方法获得cdna的5’和/或3’末端。使用实施例中所述的基因特异性正向引物和oligo-dt反向引物5′-atttaggtgacactatagttttttttttttttttttv-3′(seqidno:41)分离cdna的3’末端,其中v代表a、g或c,其在所有3’-race反应中均是相同的。采用superscriptiiione-steprt-pcr系统(invitrogen)进行rt-pcr扩增,体积为25μl,使用10pmol的正向引物和30pmol的反向引物,mgso4终浓度为2.5mm,400ng的总rna作为合成cdna的模板,按照生产商的说明使用缓冲液和核苷酸。循环条件是典型的:1个循环45℃30分钟进行反转录;然后1个循环94℃2分钟;随后是40个循环的94℃30秒,52℃30秒,70℃1分钟和1个循环72℃2分钟,随后冷却至5℃。将反应的扩增子连接到pgem-teasy中,克隆至大肠杆菌中,采用标准方法测序。除非另有说明,否则均采用改良的末端转移酶方法分离cdna的5’末端,其中使用2μg总rna作为合成cdna的模板。将10pmol的基因特异性反向引物加入到总rna和10.8μl水中,然后将混合物在65℃加热5分钟,然后冰上冷却2分钟。然后加入以下成分:4μl的superscriptiii第一链cdna缓冲液(invitrogen),1μl的0.1m二硫苏糖醇,1μlrnaseout(invitrogen)和1μl的superscriptiii反转录酶。混合物然后在55℃温育60分钟,然后通过在70℃进一步温育15分钟终止反应。在冰上短暂冷却后,然后将反应物以2单位的rnaseh37℃处理20分钟。然后使用qiaquickpcrpurificationkit(qiagen,目录#28106)纯化cdna。然后使用25μl的洗脱物进行a-加尾(a-tailed),其中使用:10单位的tdt(neb),5μl的nebbuffer#4,5μl的2.5mmcocl2,0.5μl的10mmdatp,总体积为50μl。反应在37℃进行30分钟,随后在70℃处理10分钟灭活酶。然后使用2.5单位的taqdna聚合酶(neb)进行pcr反应,反应混合物包括5μl的a-加尾cdna,10pmol的基因特异性反向引物,30pmol的改良oligo-dt引物5′-atttaggtgacactatagttttttttttttttttttv-3′(seqidno:41),其中v代表a,g或c,按照生产商的说明使用缓冲液和核苷酸。循环条件是典型的:1个循环94℃2分钟;5个循环的94℃20秒,54℃1分钟,72℃1分钟;30个循环的94℃20秒,60℃30秒,72℃1分钟;1个循环72℃5分钟;4℃放置。凝胶电泳后,如果在预期大小的范围没有看到清晰的产物条带,则切取胶上该区域并从胶中纯化dna产物。取1μl的1:20稀释的洗脱物的样品用作第二轮pcr的模板。将反应的扩增子连接到pgem-teasy中并测序。酵母培养并加料前体脂肪酸热休克方法将质粒引入酵母,并在含2%棉子糖作为单一碳源的酵母基本培养基(ymm)板上选择转化体。在含2%棉子糖作为单一碳源的液体ymm中建立克隆接种物培养物。从中取实验培养物接种在ymm+1%np-40,至初始od600为~0.3。在30℃震荡生长培养物(~60rpm)至od600为大约1.0。此时加入半乳糖至终浓度为2%,加入前体脂肪酸至终浓度为0.5mm。培养物在20℃继续震荡培养48小时,然后离心收集。以1%np-40,0.5%np-40和水洗涤细胞沉淀,以去除细胞表面的任何未掺入的脂肪酸。以瞬时表达系统在植物细胞中表达基因基本上按voinnet等(2003)所述以瞬时表达系统在植物细胞中表达基因。含有待从强组成型启动子例如35s启动子表达的编码区的质粒被引入根癌土壤杆菌菌株agl1。用于表达p19病毒沉默阻抑物的嵌合基因35s:p19被另行引入agl1。重组细胞生长在28℃的补充有50mg/ml卡那霉素和50mg/ml利福平的lb培养液中直至静止生长期。然后室温5000g离心15分钟沉淀细菌,然后重悬于含10mmmesph5.7,10mmmgcl2和100um乙酰丁香酮的渗入缓冲液中,使得od600=1.0。细胞在28℃震荡培养3小时,然后与等体积的含有35s:p19和感兴趣的测试嵌合基因的土壤杆菌培养物混合,随后渗入叶组织。渗入后,植物通常再生长5天,然后取叶盘用于gc分析脂肪酸。如果给叶组织补充外源性脂肪酸,则通过将合适的脂肪酸在2m氢氧化铵溶液中60℃加热20分钟预处理脂肪酸,随后同样在60℃将溶液蒸发。所得的盐重悬于0.1m磷酸盐缓冲液(ph7.2)至终浓度为0.5μg/ml。在土壤杆菌渗入后4天将脂肪酸盐注入叶中,在补料后不同时间点取叶盘用于分析脂肪酸组成,例如加入外源性脂肪酸后2-48小时。所用对照为不加人外源性脂肪酸,或用于渗入的土壤杆菌菌株不含有感兴趣的基因。气相色谱(gc)分析脂肪酸脂肪酸制备对于含有大量水分的样品,包括所有本氏烟草叶样品和其他非种子组织,先采用bligh和dyer(1959)所述的方法提取总脂质,然后进行甲基化。将离心的酵母沉淀、拟南芥种子、本氏烟草的总脂质或其他总脂质样品与meoh-chcl3-hcl(10:1:1,v/v/v)在配有teflon-lined螺旋帽的玻璃试管中在90-100℃加热2小时,进行转酯作用形成脂肪酸甲基酯(fame)。fame被抽提至己烷-二氯甲烷(4:1,v/v)中,通过gc和gc-ms进行分析。毛细管气液相色谱(gc)气相色谱(gc)分析fame,使用了agilenttechnologies6890ngc(paloalto,california,usa),其配备有equitytm-1熔融石英毛细管柱(15mx0.1mmi.d.,0.1μm薄膜厚度)、fid、分流/无分流进样器和agilenttechnologies7683series自动采样进样器。氦用作载体气体。以无分流模式进样,炉温120℃。进样后炉温升至270℃,速度为10℃.min-1,最后以5℃.min-1升至310℃。以agilenttechnologieschemstation软件(revb.03.01(317),\paloalto,california,usa)定量峰。气相色谱-质谱(gc-ms)在装有柱上进样器的finnigangcqplusgc-ms离子收集器上在4℃进行gc-ms。使用as2000自动采样器将样品注入连接于hp-5ultra2键合相柱(50mx0.32mmi.d.x0.17μm薄膜厚度)的保留隘口。初始温度45℃保持1分钟,随后以30℃.min-1程控升温至140℃,然后以3℃.min-1至310℃,并保持12分钟。氦用作载体气体。质谱仪操作条件为:电子轰击能量70ev;发射电流250μamp,转运线310℃;源温度240℃;扫描速率0.8scans.s-1,质量范围40-650dalton。获得质谱并以xcaliburtm软件处理。培养酵母并补料前体脂肪酸热休克方法将质粒引入酵母,并在含2%棉子糖作为单一碳源的酵母基本培养基(ymm)板上选择转化体。在含2%棉子糖作为单一碳源的液体ymm中建立克隆接种物培养物。从中取实验培养物接种在ymm+1%np-40,至初始od600为~0.3。在30℃震荡生长培养物(~60rpm)至od600为大约1.0。此时加入半乳糖至终浓度为2%,加入前体脂肪酸至终浓度为0.5mm。培养物在20℃继续震荡培养48小时,然后离心收集。以1%np-40、0.5%np-40和水洗涤细胞沉淀,以去除细胞表面的任何未掺入的脂肪酸。实施例2:从微藻类分离并表征编码δ6-延长酶的cdna分离微胞藻cs-0170δ6-延长酶基因片段csiro活微藻保藏中心的微胞藻cs-0170株(wo2005/103253)被鉴定为具有高天然水平的δ5-和δ6-延长作用的微藻株(表4)。表4.csiro活微藻保藏中心微胞藻cs-0170和塔胞藻cs-0140的脂肪酸转换为了鉴定保守序列,采用clustalw算法比对来自genbank登录号aav67800、abc18314、cad58540、cal55414、aav67797、xp_001416454、aaw70157、aav67799、abc18313、aay15135的延长酶氨基酸序列。在众多具有不同程度相同性的同源区中选择出共有氨基酸序列区段kxxxxxdt(seqidno:31)和myxyy(seqidno:32)(其中每一x独立地是任意氨基酸),分别相应于aay15135的氨基酸位置144-151和204-208。基于这两个区段的序列合成了简并引物5′-aagwwciksgaryisytcgacac-3′(seqidno:42)和5′-aiimirtartasgtgtacat-3′(seqidno:43),其中i=肌苷,w=a或t,r=a或g,y=c或t,k=g或t,m=a或c,s=c或g。使用superscriptiiione-steprt-pcr系统(invitrogen)进行rt-pcr扩增,体积为50μl,使用了20pmol的各种引物,mgso4至终浓度为2.5mm,200ng微胞藻cs-0170总rna,按照说明使用缓冲液和核苷酸。循环条件为:初始48℃30分钟进行反转录,然后1个循环94℃2分钟,随后5个循环的94℃30秒,40℃30秒,70℃30秒;,然后40个循环的94℃30秒,45℃30秒,70℃30秒,然后72℃2分钟。产生了209bp的扩增子,将其连接至pgem-teasy并测序。分离编码微胞藻cs-0170δ6-延长酶的全长cdna设计引物用于通过5′-和3′-race延伸所述209bp片段。如实施例1所述分离基因的3’末端,使用了基因特异性正向引物5′-gaacaacgactgcatcgacgc-3′(seqidno:44)和200ng微胞藻cs-0170总rna。产生了454bp的扩增子,将其连接至pgem-teasy并测序。使用generacerkit(invitrogen,目录#l1500-01)分离基因的5’末端,其中55℃反转录温育1小时以产生5′-适应的cdna,按照所附说明操作。以generacer5′引物5′-cgactggagcacgaggacactga-3′(seqidno:45)和基因特异性反向引物5′-ttgcgcagcaccataaagacggt-3′(seqidno:46)进行pcr扩增,使用了pfuultraiifusiondna聚合酶,体积为50μl,使用10pmol的各种引物,1μl的generacercdna模板,按照生产商的说明使用缓冲液和核苷酸(stratagene,目录#600670)。循环条件为:1个循环94℃2分钟;35个循环的94℃20秒,55℃30秒,72℃30秒;然后72℃2分钟,然后冷却至4℃。1:10稀释该产物并用1μl作为模板进行第二轮pcr,使用了generacer5′巢式引物5′-ggacactgacatggactgaaggagta-3′(seqidno:47)和基因特异性反向引物5′-ttgcgcagcaccataaagacggt-3′(seqidno:46),采用了与第一轮扩增相同的pcr条件。产生了522bp的扩增子,将其连接至pgem-teasy并测序。将这三种扩增子的核苷酸序列组装成一个序列,其为推定的全长序列。然后从微胞藻cs-0170株基因组dna扩增具有短5′utr区的全长编码区,使用了正向引物5′-caggcgacgcgcgccagagtcc-3′(seqidno:48),反向引物5′-ttattagttacttggcctttaccttc-3′(seqidno:49)和pfuultraiifusiondna聚合酶(stratagene)。产生了860bp的扩增子,将其连接至pgem-teasy并测序。该基因可读框的序列为seqidno:1。该基因编码的全长氨基酸序列为seqidno:2。blast分析蛋白质序列显示该分离的cdna编码δ5-延长酶或δ6-延长酶。这两种延长酶在氨基酸水平具有相似性,但从氨基酸序列本身不确定编码何种活性。将其作为查询序列使用blastp搜索genbank蛋白质序列数据库时,微胞藻cs-0170延长酶与其他延长酶之间的最高相同性程度为与登录号cal55414具有65%相同性,后者是ostreococcustauri多不饱和脂肪酸延长酶2的序列。保守性gns1/sur4家族结构域(ncbi保守结构域pfam01151)位于该序列的氨基酸49至274,其通常表明该蛋白质参与长链脂肪酸延长系统。图3给出了基于对与微胞藻cs-0170延长酶具有相似性的序列(包括那些用于设计原始简并引物的序列)的多重比对的序列关系树。在酵母中功能性表征微胞藻cs-0170δ6-延长酶该克隆的完整蛋白质编码区位于pgem-teasy的sali/sphi片段内,将该编码区插入pyes2的xhoi/sphi位点,产生载体pyes2+micelo1,用于引入酵母进行功能性表征。用pyes2+micelo1转化酵母invsc1株的细胞,在不含尿嘧啶的培养基上选择转化体。含有pyes2+micelo1的酵母细胞在培养物中生长,用半乳糖诱导gal启动子以表达micelo1基因。向培养基中加入ala、sda或epa(0.5mm)并在30℃继续培养48小时后,分析总细胞脂质中的脂肪酸。当在培养基中加入ala时,检测到酵母转化体的细胞内脂质中的etra占总脂肪酸的0.2%,这代表较低但仍可检测到的0.4%的转换效率。类似地,当在培养基中加入sda时,检测到酵母转化体的细胞内脂质中的eta占总脂肪酸的0.2%,这代表0.4%的转换效率,表明存在低水平的δ6-延长酶活性。然而,当在培养基中加入epa时,没有检测到酵母转化体的细胞内脂质中存在dpa,表明酵母细胞缺乏δ5-延长酶活性(表5)。表5.以表达分离自微胞藻cs-0170和塔胞藻cs-0140的延长酶的遗传学构建体转化的酵母细胞中的脂肪酸转换分离和表征塔胞藻cs-0140δ6-延长酶分离塔胞藻cs-0140δ6-延长酶基因片段通过对genbank登录号abo94747、cai58897、caj30869、cal23339和aav67797的延长酶氨基酸序列进行比对,我们鉴定到了共有氨基酸序列区段kiyefvdt(seqidno:33)和vhvcmyt(seqidno:34),它们分别对应于aav67797的氨基酸位置143-150和199-205。基于这两个区段的序列合成了简并引物5′-aaratmtaygagttygtigatac-3′(seqidno:50)和5′-taigtgtacatgcacacrtgwaccc-3′(seqidno:51)(缩写如上)。使用superscriptiiione-steprt-pcr系统以100ng的塔胞藻cs-0140总rna进行rt-pcr扩增。产生了191bp的扩增子,将其连接至pgem-teasy并测序。分离全长塔胞藻cs-0140δ6-延长酶基因设计引物用于通过5′-和3′-race延伸所述191bp片段。如实施例1所述分离基因的3’末端,使用了基因特异性正向引物5′-ttcgtggatacgttcatcatgc-3′(seqidno:52)。产生了945bp的扩增子,将其连接至pgem-teasy并测序。使用generacerkit从1μg塔胞藻cs-0140总rna分离基因的5’末端,其中55℃反转录温育1小时以产生5′-适应的cdna,按照所附说明操作。以generacer5′引物和基因特异性反向引物5′-agttgagcgccgccgagaagtac-3′(seqidno:53)进行pcr扩增,使用了pfuultraiifusiondna聚合酶。1:10稀释该产物并用1μl作为模板进行第二轮pcr,使用了generacer5′巢式引物5′-ggacactgacatggactgaaggagta-3′(seqidno:47)和基因特异性反向引物5′-acctggttgacgttgcccttca-3′(seqidno:54),采用了与第一轮扩增相同的pcr条件。产生了743bp的扩增子,将其连接至pgem-teasy并测序。然后将这三部分序列组装成推定的全长序列。然后通过rt-pcr从总rna扩增出具有短5′utr区的全长编码区。使用了正向引物5′-gctatggagttcgctcagcct-3′(seqidno:55)和反向引物5′-ttactactgcttcttgctggccagct-3′(seqidno:56)以及100ng的塔胞藻cs-0140总rna。产生了900bp的扩增子,将其连接至pgem-teasy并测序。该扩增子的可读框的核苷酸序列为seqidno:3,所编码的蛋白质的氨基酸序列为seqidno:4。blast分析显示该全长氨基酸序列(seqidno:4)与其他δ5-延长酶和δ6-延长酶具有相似性。塔胞藻cs-0140延长酶与其他蛋白质之间的最高相同性程度为与登录号aav67797具有54%相同性(blastx),后者是ostreococcustauri多不饱和脂肪酸延长酶1的序列。图4给出了基于对与塔胞藻cs-0140延长酶具有相似性的序列(包括那些用于设计原始简并引物的序列)的多重比对的序列关系树。保守性gns1/sur4家族结构域(ncbi保守结构域pfam01151)位于该序列的氨基酸52至297,其通常表明该蛋白质参与长链脂肪酸延长系统。在酵母中功能性表征塔胞藻cs-0140δ6-延长酶该克隆的完整蛋白质编码区位于pgem-teasy的ecori片段内,将该编码区插入pyes2的ecori位点,产生载体pyes2+pyrco-elo1,用于引入酵母进行功能性表征。用pyes2+pyrco-elo1转化酵母invsc1株的细胞,在不含尿嘧啶的培养基上选择转化体。含有pyes2+pyrco-elo1的酵母细胞在培养物中生长,用半乳糖诱导gal启动子以表达pyrco-elo1cdna。向培养基中加入脂肪酸至终浓度为0.5mm并在30℃继续培养48小时后,然后分析总细胞脂质中的脂肪酸。当在培养基中加入ala时,检测到酵母转化体的细胞内脂质中的etra占总脂肪酸的5.3%,这代表9.3%的转换效率(δ9-延长酶活性)。当在培养基中加入sda时,检测到酵母转化体的细胞内脂质中的eta占34.1%,这代表65.6%的转换效率,这是高水平的δ6-延长酶活性。然而,当在培养基中加入epa时,没有检测到酵母转化体的细胞内脂质中存在dpa(表5),表明该cdna编码的δ6-延长酶活性在酵母细胞中具有一些δ9-延长酶活性,但没有δ5-延长酶活性。有关这两种δ6-延长酶基因的以上结果表明,来自塔胞藻的基因编码的酶的活性大大高于来自微胞藻的基因的活性。这一结果是出乎预料的。已经排出了从微胞藻基因组dna扩增的编码区含有突变或该编码区是不完整的此类可能性。实施例3:从微藻类分离并表征编码δ5-延长酶的cdna分离塔胞藻cs-0140δ5-延长酶基因片段已经鉴定了csiro活微藻保藏中心的塔胞藻cs-0140株是具有高天然水平的δ5-和δ6-延长作用的微藻株(表4)。对genbank登录号aav67798和abo98084的延长酶氨基酸序列进行了比对。从众多相配的序列中我们选出了共有氨基酸序列区段ylelldt(seqidno:35)和mysyy(seqidno:36),它们分别对应于aav67798的氨基酸位置136-142和198-202。基于这两个区段的序列合成了简并引物5′-artayytsgarytrytggayac-3′(seqidno:57)和5′-catkarrtartasgagtacat-3′(seqidno:58)(缩写如上)。按照实施例1所述使用superscriptiiione-steprt-pcr系统进行rt-pcr扩增。然后将0.5μl的作为模板,使用taqdna聚合酶(neb)和相同的引物进行第二轮pcr。产生了200bp的扩增子,将其连接至pgem-teasy并测序。分离全长塔胞藻cs-0140δ5-延长酶基因设计引物用于通过5′-和3′-race延伸所述200bp片段。如实施例1所述分离基因的3’末端,使用了基因特异性正向引物5′-catcataccctgttgatctggtc-3′(seqidno:59)和oligo-dt反向引物。产生了408bp的扩增子,将其连接至pgem-teasy(promega)并测序。使用generacerkit从1μg塔胞藻cs-0140总rna分离基因的5’末端,其中55℃反转录温育1小时以产生5′-适应的cdna,按照所附说明操作。按照生产商所述,使用pfuultraiifusiondna聚合酶,以基因特异性反向引物5′-ccagatcaacagggtatgatggt-3′(seqidno:60)进行pcr扩增。1:10稀释该产物并用1μl作为模板进行第二轮pcr,使用了generacer5′巢式引物5′-ggacactgacatggactgaaggagta-3′(seqidno:47)和基因特异性反向引物5′-cgaaagctggtcaaacttcttgcgcat-3′(seqidno:61)。产生了514bp的扩增子,将其连接至pgem-teasy(promega)并测序。然后将这三部分序列组装成全长序列。然后通过rt-pcr从总rna扩增出具有短5′utr区的全长编码区。使用正向引物5′-aacatggcgtctattgcgattccggct-3′(seqidno:62)和反向引物5′-ttattactgcttcttggcacccttgct-3′(seqidno:63),按照实施例1所述进行rt-pcr扩增。产生了810bp的扩增子,将其连接至pgem-teasy并测序。该插入物的可读框的核苷酸序列为seqidno:5,该cdna所编码的推定的氨基酸序列为seqidno:6。blast分析显示该全长氨基酸序列与其他δ5-延长酶和δ6-延长酶具有同源性。blastp分析发现,塔胞藻cs-0140延长酶与genbank数据库中其他蛋白质之间的最高相同性程度为与登录号abr67690具有46%相同性,后者对应于绿色巴夫藻c20延长酶。图5给出了基于对与塔胞藻cs-0140延长酶具有相似性的序列(包括那些用于设计原始简并引物的序列)的多重比对的序列关系树。在酵母中功能性表征塔胞藻cs-0140δ5-延长酶该克隆的完整蛋白质编码区位于pgem-teasy中的cdna的ecori片段内,将该编码区插入pyes2的ecori位点,产生载体pyes2+pyrco-elo2,用于引入酵母进行功能性表征。用pyes2+pyrco-elo2转化酵母invsc1株的细胞,在不含尿嘧啶的培养基上选择转化体。含有pyes2+pyrco-elo2的酵母细胞在培养物中生长,用半乳糖诱导表达cdna。向培养基中加入脂肪酸并在30℃继续培养48小时,然后分析总细胞脂质中的脂肪酸。当在培养基中加入ala时,检测到酵母转化体的细胞内脂质中的etra占总脂肪酸的0.3%,这代表0.5%的转换效率(δ9-延长酶活性)。当在培养基中加入sda时,检测到酵母转化体的细胞内脂质中的eta占0.7%,这代表1.3%的转换效率(δ6-延长酶活性)。当在培养基中加入epa时,检测到酵母转化体的细胞内脂质中的dpa占1.8%,这代表出乎预料的高达75%的转换效率,表明酵母细胞中存在很强的δ5-延长酶活性(表6)。本发明人认为,在重组细胞内如此高效地将epa转换为dpa还未曾报道。预期该酶在植物内(inplanta)会有类似的高转换效率。保守性gns1/sur4家族结构域(ncbi保守结构域pfam01151)存在于该序列的氨基酸50至267处,这通常表明该蛋白质参与长链脂肪酸延长系统。表6.以表达分离自微胞藻cs-0170和塔胞藻cs-0140的延长酶的遗传学构建体转化的酵母细胞中脂肪酸的转换实施例4:从微藻类分离并表征编码δ6-去饱和酶的基因合成全长微胞藻ccmp1545δ6-去饱和酶基因采用blastp程序分析美国能源部联合基因组研究所(usdepartmentofenergyjointgenomeinstitute)(http://www.jgi.doe.gov/)所产生的微胞藻ccmp1545过滤蛋白质模型基因组序列,使用了ostreococcustauriδ6-去饱和酶氨基酸序列(genbank登录号aaw70159)作为查询序列。这一分析发现微胞藻ccmp1545中存在与aaw70159具有同源性的推定的蛋白质。使用微胞藻ccmp1545推定的蛋白质序列设计并合成了最适合于在双子叶植物例如欧洲油菜中表达的密码子优化的核苷酸序列。该蛋白质编码区的核苷酸序列见seqidno:7。质粒构建体命名为pga4。其氨基酸序列见seqidno:8。使用微胞藻ccmp1545去饱和酶氨基酸序列seqidno:8作为查询序列对genbank数据库中的其他蛋白质进行blastp分析发现,该蛋白质与δ6-去饱和酶具有同源性。最高相同性程度为与登录号aaw70159的氨基酸序列的全长具有66%相同性,后者对应于ostreococcustauriδ6-去饱和酶的序列。图6给出了基于对与微胞藻ccmp1545去饱和酶具有相似性的序列的多重比对的序列关系树。这种前端去饱和酶在氨基酸54至104含有细胞色素b5结构域(ncbi保守结构域pfam00173)并在氨基酸172至428含有δ6-fads-样保守结构域(ncbi保守结构域cd03506)。代表前端去饱和酶的三个组氨酸盒分别存在于该序列的第190-195、227-232和401-405位。同时含有这两种结构域的蛋白质通常是合成高度不饱和脂肪酸所需的前端去饱和酶。有趣的是,这种去饱和酶与aaw70159紧密成簇,而后者是目前已知的唯一经生化方法确认的植物样酰基-coa去饱和酶。在酵母细胞中功能性表征微胞藻ccmp1545δ6-去饱和酶微胞藻去饱和酶的完整编码区位于质粒pga4的kpni-saci片段内,将该编码区插入酵母载体pyes2的kpni-saci位点,产生pyes2+micd6d,用于引入酵母进行功能性表征。用pyes2+micd6d转化酵母invsc1株的细胞,在不含尿嘧啶的培养基上选择转化体。含有pyes2+micd6d的酵母细胞在培养物中生长,用半乳糖诱导。向培养基中加入0.5mm的la、ala、etra、dgla或eta并在30℃继续培养48小时,然后分析总细胞脂质中的脂肪酸。当在培养基中加入la时,检测到酵母转化体的细胞内脂质中的gla占总脂肪酸的3.9%,这代表11.4%的δ6-去饱和作用转换效率。当在培养基中加入ala时,检测到酵母转化体的细胞内脂质中的sda占总脂肪酸的13.9%,这代表39.0%的δ6-去饱和作用转换效率。也就是说,对ω3脂肪酸底物的转换效率是对相应的ω6脂肪酸底物的转换效率的3.5-倍。当在培养基中加入etra时,检测到酵母转化体的细胞内脂质中的eta占总脂肪酸的0.21%,这代表8.0%的δ8-去饱和作用转换效率。然而,当在培养基中加入dgla或eta时,分别没有检测到ara或epa。这表明缺乏任何δ5-去饱和作用活性(表7)。表7.以表达分离自微胞藻ccmp1545、ostreococcuslucimarinus和塔胞藻cs-0140的去饱和酶的遗传学构建体转化的酵母细胞中的脂肪酸转换在植物细胞中功能性表征微胞藻ccmp1545δ6-去饱和酶使用增强型本氏烟草瞬时表达系统按照实施例1所述在植物内证实了微胞藻ccmp1545δ6-去饱和酶(mic1545-d6d)和车前叶蓝蓟δ6-去饱和酶(echpl-d6d;zhou等,2006)(用作阳性对照样品)的酶活性。通过将含有35s启动子的psti片段插入至载体pore04的sfoi位点(经t4dna聚合酶处理以提供平端)(coutu等,2007)而制备命名为35s-pore04的载体。通过将位于swai片段内的pga4的完整编码区插入至35s-pore04的smai-ecorv位点而制备遗传学构建体35s:mic1545-d6d,产生pjp2064。将这些嵌合载体引入根癌土壤杆菌菌株agl1,来自这些培养物的细胞渗入温室中的本氏烟草植物的叶组织。渗入后,植物再生长5天,然后取叶盘进行gc分析,结果显示两种基因在本氏烟草中均发挥δ6-去饱和酶的功能。以车前叶蓝蓟δ6-去饱和酶转化的叶组织含有gla(0.4%)和sda(1.2%),分别代表3.8%和4.4%的转换效率。以微胞藻ccmp1545δ6-去饱和酶转化的叶组织含有sda(2.2%),代表转换效率为6.9%,但没有可检测到的gla。叶组织缺乏gla可能是由于在植物内极度偏向ω3底物ala而非ω6底物la,或是部分由于存在天然的本氏烟草ω3去饱和酶活性,其可能将δ6-去饱和作用所产生的部分gla转换为sda。以往用具有ω3底物偏向性的酰基-pcδ6-去饱和酶进行的实验已经表明这是可能的(sayanova等,2006),不过该研究中没有对其发生的程度进行定量。微胞藻δ6-去饱和酶的ω-3底物偏向性分离自微胞藻的δ6-去饱和酶在植物内与在酵母内一样对ω3底物具有令人惊讶的强偏向性。在酵母中表达的该酶对ω3-去饱和的脂肪酸底物的活性是对相应的ω6-去饱和的脂肪酸底物的活性的3.5倍。鉴于所报道的o.tauri的酶缺乏偏向性(domergue等,2005),因此所观察到的对ω3底物的偏向性完全是令人惊讶且出乎预料的。对o.tauriδ6-去饱和酶在酵母或植物种子中表达的研究表明其对于la和ala具有相似的活性。因此可以预期,与使用对ω3-去饱和的底物不具有偏向性的去饱和酶相比,在植物内使用该基因或其他对ω3-去饱和的脂肪酸底物具有如此高特异性的基因连同作为重组vlc-pufa途径的一部分的其他脂肪酸去饱和酶和延长酶,能够提高epa、dpa和dha的水平。推测导致这种升高的原因是降低了la转换为gla以及随后的ω6pufadgla和ara,真菌或酵母δ17-去饱和酶在植物内不能高效地将它们转换为其ω3对应物。尽管已经分离到了对ω3脂肪酸底物具有偏向性的δ6去饱和酶(sayanova等,2003),但其对磷脂连接的酰基链具有活性。相反,推测从微胞藻获得的所述去饱和酶对酰基辅酶a底物具有活性。微胞藻ccmp1545δ6-去饱和酶的δ6/δ8双重功能有趣的是注意到微胞藻ccmp1545δ6-去饱和酶表现出显著水平的δ8-去饱和酶活性且因此具有显著的双重活性,这不同于不具有可检测到的δ8-去饱和酶活性的ostreococcuslucimarinus酶(下文)。推测这种双重去饱和酶活性可用于构建植物中的双重δ6/δ8-去饱和酶途径,或用于当构建此类途径所使用的延长酶同时具有δ9-延长酶和δ6-延长酶活性的情况中。使用这种基因可能有助于通过将etra转换为eta,其随后被δ5-去饱和为epa,从而降低etra的积聚。合成全长ostreococcuslucimarinusδ6-去饱和酶基因使用ostreococcustauriδ6-去饱和酶核苷酸序列(登录号ay746357)作为查询序列,通过blastx分析非冗余蛋白序列的genbank数据库。根据这一分析鉴定到了ostreococcuslucimarinus的一种基因,其编码登录号为xp_001421073的氨基酸序列的一种部分长度的蛋白质。然后检查了编码xp_001421073的区域两侧的基因组dna序列,以鉴定推定的翻译起始和终止密码子,以便确定全长蛋白质编码区,其核苷酸序列为seqidno:9。随后将该编码区翻译为蛋白质序列,其为seqidno:10。使用这一氨基酸序列来设计并合成最适合于在欧洲油菜和其他双子叶植物中表达的密码子优化的核苷酸序列,其核苷酸序列为seqidno:11。使用ostreococcuslucimarinus去饱和酶氨基酸序列作为查询序列对genbank数据库中的其他蛋白质进行blastp分析,发现seqidno:10与δ6-去饱和酶具有同源性。最高相同性程度为与登录号aaw70159的氨基酸序列的全长具有76%相同性,后者对应于ostreococcustauriδ6-去饱和酶的序列。图7给出了基于对与ostreococcuslucimarinus去饱和酶具有相似性的序列的多重比对的序列关系树。这种前端去饱和酶在氨基酸55至108含有细胞色素b5结构域(ncbi保守结构域pfam00173)并在氨基酸198至444含有δ6-fads-样保守结构域(ncbi保守结构域cd03506)。代表前端去饱和酶的三个组氨酸盒分别存在于该序列的第207-212、244-249和417-421位。同时含有这两种结构域的蛋白质通常是合成高度不饱和脂肪酸所需的前端去饱和酶。有趣的是,这种去饱和酶与aaw70159紧密成簇,而后者是目前已知的唯一经生化方法确认的植物样酰基-coa去饱和酶。在酵母中功能性表征ostreococcuslucimarinusδ6-去饱和酶ostreococcus基因(seqidno:11)的完整编码区位于pgem-teasy的noti片段内,将该编码区插入pyes2的noti位点,产生嵌合载体pyes2+ostlud6d,用于引入酵母进行功能性表征。用pyes2+ostlud6d转化酵母invsc1株的细胞,在不含尿嘧啶的培养基上选择转化体。含有pyes2+ostlud6d的酵母细胞在培养物中生长,然后用半乳糖诱导。向培养基中加入la、ala、sda或epa至每种的终浓度为0.5mm并在30℃继续培养48小时,然后分析细胞脂质中的脂肪酸。当在培养基中加入la时,检测到酵母转化体的细胞内脂质中的gla占总脂肪酸的2.1%,这代表δ6-去饱和作用转换效率为6.6%。当在培养基中加入ala时,检测到酵母转化体的细胞内脂质中的sda占总脂肪酸的13.8%,这代表δ6-去饱和作用转换效率为38.8%。然而,当在培养基中加入etra、dgla或eta时,分别没有检测到eta、ara或epa,表明没有任何δ5-或δ8-去饱和活性(表7),也表明与具有相同长度和不饱和模式的ω6脂肪酸相比,其对ω3脂肪酸底物具有偏向性。去饱和酶的酰基-coa底物特异性与其他δ6-去饱和酶相比,本实施例中所描述的去饱和酶与先前分离自ostreococcustauri的δ6-去饱和酶具有更加密切的关联(图9)。在将这些基因与所述去饱和酶家族的其他成员放在一起的系统发生树中更可以看出这种相似性(图10)。已经报道了ostreococcustauriδ6-去饱和酶对酰基辅酶a底物具有活性(domergue等,2005)。基于这些发现,推测上述基因编码的δ6-去饱和酶可能对酰基辅酶a底物而非酰基-pc底物具有活性。有趣的是,盐生巴夫藻δ5-去饱和酶也与o.tauriδ6-去饱和酶和所述δ8-去饱和酶成簇,形成一个独立的分支。为了确定细小微胞藻(微胞藻ccmp1545)δ6-去饱和酶是否能够以酰基-coa脂肪酸作为底物且由此产生δ6-去饱和的酰基-coa脂肪酸,仅以编码所述去饱和酶的基因构建体转化酿酒酵母,并将所转化的细胞系一式三份的培养物生长在存在250μm外源性18:3δ9,12,15的条件下。从这些培养物提取总脂质并通过薄层色谱(tlc)分级为中性脂质(nl)、磷酯酰胆碱(pc)、磷脂酰肌醇(pi)、磷脂酰丝氨酸(ps)和磷脂酰乙醇胺(pe)等类型,随后从每一类产生fame,并通过gc进行分析。结果显示于表8。表8.以克隆至pyes2的细小微胞藻δ6-去饱和酶转化的酿酒酵母中总脂质和分级的中性脂质(nl)、磷酯酰胆碱(pc)、磷脂酰肌醇(pi)、磷脂酰丝氨酸(ps)和磷脂酰乙醇胺(pe)的脂肪酸组成(占总脂肪酸的百分比)在总脂质中,71%的18:3δ9,12,15已经被δ6-去饱和为18:4δ6,9,12,15。与总脂质提取物相比,在pc级份中没有检测到产物富集。实际上,pc级份中18:4δ6,9,12,15的百分比显著低于总脂质中的百分比(21.0%vs.29.4%),表明所述去饱和酶以酰基-coa硫酯的形式产生18:4δ6,9,12,15(domergue等,2003)。通过转化将编码细小微胞藻δ6-去饱和酶的基因引入拟南芥植物。通过将位于swai片段内的编码细小微胞藻δ6-去饱和酶的完整编码区插入linin-pwvec8的smai位点产生遗传学构建体linin:micpu-d6d,产生linp-mic1545-d6d-lint。该构建体的启动子是来自亚麻的种子特异性linin启动子。将该构建体转化至拟南芥columbia生态型,通过gc分析转化植物的t2种子内的脂肪酸组成(图11)。在酵母和本氏烟草中的生化研究均提示来自细小微胞藻的所述δ6-去饱和酶是酰基-coa去饱和酶。其他研究采用对确保延长步骤的动力学分析作为间接方法来确定去饱和酶产生酰基-coa产物的能力:所述δ6-去饱和产物(sda)在随后δ6-延长步骤(出现于酰基-coa代谢库)中的可利用度受到所述δ6-去饱和酶的底物特异性的影响(domergue等,2003,2005;hoffmann等,2008)。使用来自o.tauri和细小微胞藻的所述δ6-去饱和酶获得了类似的δ6-延长速度,这不同于使用车前叶蓝蓟酰基-pcδ6-去饱和酶时所观察到的显著低水平的延长作用(图12a)。当分析酵母脂质类型中所述δ6-去饱和酶产物sda的分布时观察到了进一步的证据(表8)。与总脂质成分相比,没有观察到pc成分中的富集,不过,可预期的是如果是使用酰基-pc去饱和酶产生sda,应该出现这种富集(domergue等,2005)。我们的研究中所观察到的这种相对低水平的δ6-去饱和作用(图12)是预料之中的,因为本氏烟草叶中的大部分底物la和ala位于质体中,不能用于去饱和作用。不过,由于在制备fame过程中也分离到这些脂肪酸,它们的存在有效降低了所计算的总体转换效率。因此可以预期这些基因的种子特异性转换效率会高得多。酰基-coaδ6-去饱和酶和酰基-pcδ6-去饱和酶之间的比较在植物细胞中进一步比较了微胞藻ccmp1545δ6-去饱和酶、车前叶蓝蓟δ6-去饱和酶和ostreococcustauriδ6-去饱和酶(domergue等,2005)。将实施例4所述的遗传学构建体35s:mic1545-d6d和35s:echpl-d6d与遗传学构建体35s:ostta-d6d进行对比,后者是通过将位于swai片段内的ostreococcustauriδ6-去饱和酶的完整编码区插入至35s-pore04的smai-ecorv位点而制备的,这产生pjp3065。车前叶蓝蓟与o.tauri和细小微胞藻epa途径直接比较显示,酰基-coa去饱和酶途径产生远远更高水平的epa,这是因为两者均有更高效的δ6-去饱和作用和更高效的随后的δ6-延长作用(图12a)。车前叶蓝蓟δ6-去饱和酶催化转换14%的ω3底物(18:3δ9,12,15转换为18:4δ6,9,12,15)和30%的ω6底物(18:2δ9,12转换为18:3δ6,9,12)。使用o.tauriδ6-去饱和酶导致24%的ω3转换和40%的ω6转换,而使用细小微胞藻δ6-去饱和酶导致27%的ω3转换和15%的ω6转换。这些转换导致车前叶蓝蓟途径产生1.3%的20:4δ5,8,11,14和3.4%的20:5δ5,8,11,14,17,o.tauri途径产生1.2%的20:4δ5,8,11,14和9.6%的20:5δ5,8,11,14,17,而细小微胞藻途径产生0.6%的20:4δ5,8,11,14和10.7%的20:5δ5,8,11,14,17。与使用车前叶蓝蓟去饱和酶相比,当使用o.tauri或细小微胞藻δ6-去饱和酶产生底物18:4δ6,9,12,15时,δ6-延长作用远远更高(图12a)。除了细小微胞藻δ6-去饱和酶所显示出的ω3底物特异性之外,p.cordataδ6-延长酶(见实施例2)被证明具有更高的特异性,并且其转换ω3底物18:4δ6,9,12,15的速度远高于转换18:3δ6,9,12的速度(对于细小微胞藻epa途径分别为89%和21%)。使用双重δ6-去饱和酶途径进行对比来研究通过使用含有两种δ6-去饱和酶的途径来提高δ6-去饱和作用的可能性。首先,车前叶蓝蓟酰基-pc去饱和酶和细小微胞藻酰基-coa去饱和酶的组合没有使得转换效率显著高于仅含有细小微胞藻去饱和酶的途径(图12b)。当车前叶蓝蓟与o.tauriδ6-去饱和酶组合时得到了类似的结果。与使用o.tauri或细小微胞藻途径之一相比,o.tauri与细小微胞藻去饱和酶组合的双重酰基-coaδ6-去饱和酶途径也没有导致ω3转换效率升高(图12c)。也测试了在产生epa的途径中使用双重δ6-去饱和酶的效果。首先测试的是在分开的实验中将来自车前叶蓝蓟的酰基-pc去饱和酶与这两种酰基-coa去饱和酶组合。假设加入脂质连接的去饱和酶可能会提高酰基-pc底物la或ala分别向gla或sda的转换。类似地,我们还测试了使用两种酰基-coa去饱和酶是否有可能提高epa的积聚。在本氏烟草中进行的瞬时测定中这些假设无一得到证实。实施例5:分离和表征编码来自微藻类的δ5-去饱和酶的基因分离塔胞藻cs-0140δ5-去饱和酶基因片段对来自genbank登录号abl96295、abp49078、xp_001421073、aam09687、aat85661、aaw70159和aax14505的去饱和酶氨基酸序列进行比对鉴定了共有氨基酸序列区段wknmhnkhha(seqidno:37)和hhlfpsmp(seqidno:38),分别对应于abl96295的氨基酸位置197-206和368-375。使用codehop程序(rose等,1998)基于这两个区段的序列设计了简并引物5′-ggtggaagaacaagcacaacrdncaycaygc-3′(seqidno:64)和5′-gggcatcgtggggwanarrtgrtg-3′(seqidno:65)。使用taqdna聚合酶(neb)进行降落pcr扩增,体积为20μl,使用了10pmol的各种引物,50ng的塔胞藻cs-0140基因组dna,按照所附手册的说明使用缓冲液和核苷酸。循环条件为:1个循环94℃3分钟;20个循环的94℃1分钟,70℃2分钟(每一循环-1℃),72℃1分钟;20个循环的94℃1分钟,55℃1分钟,72℃1分钟;1个循环72℃5分钟;4℃保持。产生了551bp的扩增子,将其连接至pgem-teasy(promega)并测序。分离全长塔胞藻cs-0140δ5-去饱和酶基因如实施例1所述设计引物用于通过5′-和3′-race延伸所述551bp片段。如实施例1所述分离编码所述δ5-去饱和酶的基因的cdna的3’末端,使用了基因特异性正向引物5′-agcgagtacctgcattgggt-3′(seqidno:66)和修饰的oligo-dt反向引物。产生了477bp的扩增子,将其连接至pgem-teasy并测序。如实施例1所述通过改良的末端转移酶方法分离所述基因的5’末端。基因特异性反向引物为5′-atagtgcttggtgcgcaagctgtgcct-3′(seqidno:67)。两轮pcr扩增之后产生了317bp的扩增子,将其连接至pgem-teasy(promega)并测序。将这三部分序列组装成推定的全长基因序列。然后从基因组dna扩增具有短5′utr区的全长蛋白质编码区,pcr扩增使用了正向引物5′-caccatgggaaagggaggcaatgct-3′(seqidno:68)和反向引物5′-ttactagtgcgccttggagtgagat-3′(seqidno:69)和pfuultraiifusiondna聚合酶(stratagene),体积为20μl,使用了4pmol的各种引物和50ng的塔胞藻cs-0140基因组dna,按照所附pfuultraiifusion手册的说明使用缓冲液成分。产生了代表全长cdna的1336bp的扩增子,将其连接至pgem-teasy并测序。该cdna的可读框的核苷酸序列为seqidno:12。blast分析蛋白质序列显示该基因编码的全长氨基酸序列(seqidno:13),与δ5-去饱和酶或δ6-去饱和酶具有相似性。这两种去饱和酶在氨基酸水平具有相似性,但从氨基酸序列本身不确定编码何种活性。按照下文所述进行了酶活性分析,发现所编码的蛋白质具有δ5-去饱和酶活性。经blastp确定,塔胞藻cs-0140去饱和酶与genbank数据库中其他去饱和酶之间的最高相同性程度为与登录号edq92231具有52%相同性,后者是来自领鞭毛虫mx1的具有未确定酶活性的蛋白质的氨基酸序列。图8给出了基于对与塔胞藻cs-0140去饱和酶具有相似性的序列(包括那些用于设计原始简并引物的序列)的多重比对的序列关系树。这种前端去饱和酶在氨基酸16至67含有细胞色素b5结构域(ncbi保守结构域pfam00173)并在氨基酸159至411含有δ6-fads-样保守结构域(ncbi保守结构域cd03506)。代表前端去饱和酶的三个组氨酸盒分别存在于该序列的第175-180、212-217和384-388位氨基酸。含有这些结构域的蛋白质通常是合成多不饱和脂肪酸所需的前端去饱和酶。在酵母中功能性表征塔胞藻cs-0140δ5-去饱和酶该克隆的完整编码区位于pgem-teasy的noti片段内,将该编码区插入pyes2(invitrogen)的noti位点,产生pyes2+pyrco-des2,用于引入酵母进行功能性表征。用pyes2+pyrco-des2转化酵母invsc1株的细胞,在不含尿嘧啶的培养基上选择转化体。含有pyes2+pyrco-des2的酵母细胞在培养物中生长,然后用半乳糖诱导。向培养基中加入0.5mmdla、ala、dgla或eta并在30℃继续培养48小时,然后分析细胞脂质中的脂肪酸。当在培养基中加入dgla时,检测到酵母转化体的细胞内脂质中的ara占总脂肪酸的0.12%,这代表δ5-去饱和作用转换效率为4.0%。当在培养基中加入eta时,检测到酵母转化体的细胞内脂质中的epa占总脂肪酸的0.26%,这代表δ6-去饱和作用转换效率为3.5%。然而,当在培养基中加入la或ala时,酵母转化体分别没有产生gal或sda。这表明该蛋白质在酵母细胞中没有任何δ6-去饱和活性(表7)。在植物细胞中表达p.cordataδ5-去饱和酶使用如实施例1所述的增强型本氏烟草瞬时表达系统在植物内证明以下的酶的酶活性:微胞藻ccmp1545δ6-去饱和酶(seqidno:8,其编码序列为seqidno:7)、塔胞藻cs-0140δ6-延长酶(seqidno:4,其编码序列为seqidno:3)和塔胞藻cs-0140δ5-去饱和酶(seqidno:13,其编码序列为seqidno:12)以及拟南芥dgat1(seqidno:74,其编码序列为seqidno:75)。将位于ecori片段内的塔胞藻cs-0140δ5-去饱和酶的完整编码区插入35s-pore04(实施例4,见上)的ecori位点,产生35s:pyrco-d5d,由此制备编码置于组成型35s启动子控制下的δ5-去饱和酶的遗传学构建体35s:pyrco-d5d。将嵌合载体35s:mic1545-d6d(实施例10)、35s:pyrco-d6e(实施例10)和35s:pyrco-d5d分别引入根癌土壤杆菌菌株agl1,混合来自这些细胞的培养物的转基因细胞,使得混合物渗入温室中的本氏烟草植物的叶组织。渗入后,植物再生长5天,然后取叶盘进行gc分析,结果显示这些基因在本氏烟草中均发挥产生epa的功能。以这些基因转化的叶组织含有sda(1.0%)、eta(0.1%)、epa(10.0%)。叶组织还含有痕量水平的gla、eta和ara。计算出的所述δ5-去饱和酶转换效率为98.8%。本实验证实微藻δ5-去饱和酶能够在植物细胞中以至少90%或至少95%的效率将eta转换为epa。实施例6:分离和表征编码来自微藻类的ω3-去饱和酶的基因分离微胞藻cs-0170ω3-去饱和酶基因片段为了确定微藻类例如微胞藻是否具有编码ω3去饱和酶的基因,且或许会鉴定到这样的基因,搜索微胞藻rcc299株基因组序列中与fad3显示出同源性的基因。但该搜索未能鉴定到任何候选基因。因此,本发明人考虑微胞藻中是否有其他类型的去饱和酶能够充当ω3去饱和酶。这一假设得到了如下发现(实施例4)的支持,即该株中的δ6去饱和酶是属于前端、酰基-coa依赖型的。然而,经检查,微胞藻rcc299基因组似乎含有至少30个推定的脂肪酸去饱和酶的基因,而且不知道其中有哪些(如果有的话)可能编码ω3去饱和酶。在一个实验中,对来自genbank登录号bad91495、abl63813、bad11952和aar20444的去饱和酶氨基酸序列进行比对鉴定到了共有氨基酸序列区段wcighdcg(seqidno:39)和tflqhhdedm(seqidno:40),分部对应于bad91495的氨基酸位置106-113和296-305。使用codehop程序基于这两个区段的序列设计了简并引物5′-tgtggtgcatcggccayganksngg-3′(seqidno:70)和5′-tgtcctcgtcgttgtgctgnarrwangt-3′(seqidno:71)。使用taqdna聚合酶(neb)进行降落pcr扩增,体积为20μl,使用了10pmol的各种引物,50ng的微胞藻cs-0170基因组dna,按照所附手册的说明使用缓冲液和核苷酸。循环条件为:1个循环94℃3分钟;20个循环的94℃1分钟,70℃2分钟(每一循环-1℃),72℃1分钟;35个循环的94℃1分钟,56℃1分钟,72℃1分钟;1个循环72℃5分钟;4℃保持。产生了528bp的扩增子,将其连接至pgem-teasy(promega)并测序。该扩增子的核苷酸序列为seqidno:14,所编码的部分蛋白质序列为seqidno:15。合成全长微胞藻rcc299ω3-去饱和酶基因将简并pcr所产生的528bp片段与完整的微胞藻rcc299过滤蛋白质模型基因组序列(由美国能源部联合基因组研究所(http://www.jgi.doe.gov/)产生)进行比较。blast分析发现在微胞藻rcc299的13号染色体上的一个区域与seqidno:14之间存在具有高同源性的区域。基于这两个序列近乎相同,因此微胞藻cs-0170和rcc299具有密切的关联(微胞藻rcc299的核苷酸序列为seqidno:16)。使用微胞藻rcc299推定的蛋白质序列(seqidno:17)设计并合成了最适合于在欧洲油菜或其他双子叶植物中表达的密码子优化的核苷酸序列(seqidno:18)。在酵母中测试了该基因从seqidno:18的164位核苷酸开始的一个较短形式,但没有检测到ω3去饱和酶活性。blast分析表明全长氨基酸序列(seqidno:17)与fat-1、fat-2和ω3去饱和酶具有同源性。但不可能基于序列本身推断编码何种活性。经blastp确定,微胞藻cs-0170去饱和酶与genbank数据库中其他蛋白质之间的最高相同性程度为与xp_001899085.1具有35%相同性,后者是脂肪酸去饱和酶家族中的brugiamalayi蛋白。该前端去饱和酶含有δ12-fads-样保守结构域(ncbi保守结构域cd03507)。含有这两种结构域的蛋白质通常是合成脂肪酸所需的前端去饱和酶,包括ω3去饱和酶家族。在植物中功能性表征微胞藻rcc299ω3-去饱和酶使用上述增强型本氏烟草瞬时表达系统在植物内测试分离自微胞藻rcc299的全长基因编码的推定的ω3-去饱和酶(mic299-w3d,见上)和致病疫霉δ17-去饱和酶(phyin-d17d,genbank登录号cam55882)(用作阳性对照样品)的酶功能。将位于ecori片段内的seqidno:18所示的完整蛋白质编码区克隆入载体35s-pore04(实施例4)的ecori位点,产生命名为pjp2073的遗传学构建体,由此制备了35s:mic299-w3d构建体。将位于ecori片段内的致病疫霉δ17去饱和酶的完整编码区克隆入载体35s-pore04的ecori位点,产生pjp2074,由此制备了35s:phyin-d17d构建体。类似地,将位于ecori片段内的拟南芥dgat1(af051849)的完整编码区克隆入载体35s-pore04的ecori位点,产生pjp2078,由此制备了35s:arath-dgat1构建体。根癌土壤杆菌菌株agl1在28℃生长于lb培养液中,其中补充了50mg/ml卡那霉素和50mg/ml利福平,直至达到静止生长期。室温下5000g离心15分钟沉淀细菌,然后以od600=1.0垂悬于渗入缓冲液,该渗入缓冲液含有10mmmesph5.7,10mmmgcl2和100μm乙酰丁香酮。细胞随后在28℃震荡培养3小时,然后将等体积的含有35s:p19、35s:arath-dgat1以及35s:phyin-d17d或35s:mic299-w3d的土壤杆菌细胞培养物混合,随后渗入到叶组织中。制备花生四烯酸盐并如上所述补料给转化的叶组织,在补料底物后5小时和24小时取叶盘进行分析。以35s:phyin-d17d构建体渗入的叶斑点,或以35s:mic299-w3d构建体分开地渗入的叶斑点均证实分别以37%和50%的效率将ara(20:4ω6)转换为epa(20:5ω3)(图13),这表明该蛋白质具有δ17-去饱和酶活性。讨论:表征了第一种具有δ17-去饱和酶活性的微藻ω3-去饱和酶本研究所述的微胞藻rcc299ω3去饱和酶是第一种微藻δ17去饱和酶,即植物样δ17去饱和酶,对c20或更长的脂肪酸底物具有活性。并不知道陆生植物具有前端去饱和酶类型的ω3去饱和酶,而是fad3类型的。因此,发现微藻株(其比真菌更接近植物)具有前端去饱和酶类型的ω3去饱和酶是令人惊讶的。基于与其他去饱和酶的同源性,认为有可能的情况是,在上述实验中用作对照基因的真菌致病疫霉去饱和酶对酰基-pc底物具有活性,而微胞藻rcc299去饱和酶对酰基辅酶a底物具有活性。已知其他真菌去饱和酶对酰基-pc底物具有活性。关于微胞藻基因的这一结论与所观察到的其与来自相同株的δ6-去饱和酶基因具有相似性这一点是一致的(实施例4)。可通过底物补料研究进一步测定这种底物优先性,在底物补料研究中,补料给转化组织的底物例如ara可立即被酰基-coa库利用,但却仅在被天然植物(例如,本氏烟草)酰基转移酶转换后才能被酰基-pc库利用。微胞藻rcc299ω3去饱和酶基因对于构建用于在植物中产生epa和下游脂肪酸dpa和dha以及其他ω3vlc-pufa的重组途径是非常有用的,这特别是因为其能够将ω6底物例如ara转换为ω3产物。当其与同样在酰基-coa库中起作用的延长酶例如δ5-延长酶组合时,对酰基辅酶a底物的活性增强了这种用途。此外,微胞藻株的脂肪酸谱表明微胞藻酶还能够将ω6c18脂肪酸例如gla或la分别转换为它们的ω3对应物例如sda或ala。可在酵母细胞或在植物中通过以上就底物ara所述的底物补料而证实gla转换为sda,而la转换为ala则在酵母细胞中得到更好的证实,这是因为植物中存在内源性δ15去饱和酶。鉴定其他ω3-去饱和酶采用blastp程序分析美国能源部联合基因组研究所(http://www.jgi.doe.gov/)所产生的微胞藻ccmp1545过滤蛋白质模型基因组序列,使用了seqidno:17作为查询序列。这一分析发现微胞藻ccmp1545(eugene.0000150179)中存在与seqidno:17具有同源性的基因。其可读框序列为seqidno:19,蛋白质序列为seqidno:20。blast分析表明全长氨基酸序列seqidno:20与fat-1、fat-2和ω3去饱和酶具有同源性。微胞藻ccmp1545去饱和酶与genbank数据库(blastp)中其他蛋白质之间的最高相同性程度为在全长上与seqidno:17具有59%相同性,后者是来自领鞭毛虫mx1的具有未确定酶活性的蛋白质的氨基酸序列。该前端去饱和酶含有δ12-fads-样保守结构域(ncbi保守结构域cd03507)。含有这两种结构域的蛋白质通常是合成脂肪酸所需的前端去饱和酶,包括ω3去饱和酶家族。我们推测该蛋白质在植物内将同样作为具有δ17-去饱和酶活性的ω3去饱和酶而起作用。实施例7:分离和表征编码来自微藻类的δ9-延长酶的其他基因分离并表征emilianiahuxleyiccmp1516δ9-延长酶使用了genbank登录号af390174的氨基酸序列作为查询序列,采用blastp程序分析美国能源部联合基因组研究所(http://www.jgi.doe.gov/)所产生的emilianiahuxleyiccmp1516过滤蛋白质模型基因组序列。这一分析发现emilianiahuxleyiccmp1516中存在与af390174具有同源性的推定的基因。其蛋白质序列为seqidno:28,编码核苷酸序列为seqidno:27。blast分析表明全长氨基酸序列与pufa延长酶具有同源性。emilianiahuxleyiccmp1516延长酶与其他蛋白质之间的最高相同性程度(blastp)为与af390174具有80%相同性。该序列中存在保守性gns1/sur4家族结构域(ncbi保守结构域pfam01151),这通常表明该蛋白质参与长链脂肪酸延长系统。使用emilianiahuxleyiccmp1516推定的蛋白质序列设计并合成了最适合于在双子叶植物例如欧洲油菜中表达的密码子优化的核苷酸序列(seqidno:29)。该质粒构建体命名为0835668_emihu-d9e_pma。分离并表征p.pinguis和盐生巴夫藻δ9-延长酶为了鉴定p.pinguis和盐生巴夫藻δ9-延长酶中可能存在的保守区域,对来自e.huxleyiδ9-延长酶、pll00000665(使用e.huxleyi延长酶序列作为查询序列,通过blast分析鉴定的来自tbestdb的路氏巴夫藻est序列)和genbank登录号aal37626(球等鞭金藻δ9-延长酶)的推定的延长酶氨基酸序列进行比对。由此发现了共有氨基酸序列区段vdtrkgayr(seqidno:76)和fihtimyty(seqidno:77),它们分别对应于emihu-d9e的氨基酸位置40-48和170-178。基于这两个区段的序列设计了简并引物5’-tggtggacacaaggaagggngcntaymg-3’(seqidno:78)和5’-gtaggtgtacatgatggtrtgdatraa-3’(seqidno:79),并使用来自p.pinguis的rna和来自盐生巴夫藻(zhou等,2007)的cdna文库进行rt-pcr和pcr扩增,其中使用superscriptiiitmone-steprt-pcr系统或taqdna聚合酶(neb,ipswich,ma,usa)。通过rt-pcr从p.pinguis中产生了641bp的扩增子,将其连接至pgem-t并测序。设计引物用于通过5′-和3′-race延伸所述641bp片段。rt-pcr分离基因的3’末端,使用了基因特异性正向引物5’-gtccttgctccagggcttccacca-3’(seqidno:80)和oligo-dt-sp6反向引物5’-atttaggtgacactatagtttttttttttttttttt-3’(seqidno:81)。产物以1:10进行稀释,取1.0μl用作模板,以taqdna聚合酶(neb)进行第二轮pcr,使用了基因特异性正向引物5’-ttccagaacgagggcatctacgt-3’(seqidno:82)和相同的反向引物。产生了1079bp的扩增子,将其连接至pgem-easy并测序。从1.0μg的p.pinguiscdna分离该基因的5’末端,所述cdna是使用基因特异性反向引物5’-ttgggtgatctgcatgagcgtgatg-3’(seqidno:83)和末端转移酶a-加尾产生的。然后将该cdna用作模板,使用oligo-dt-sp6引物和基因特异性引物5’-cgaatacttgaagagcttgttggaga-3’(seqidno:84)进行pcr反应。产物以1:10进行稀释,取1.0μl用作模板,使用oligo-dt-sp6引物和基因特异性引物5’-gggctacgagctggcagatgaagca-3’(seqidno:85)进行第二轮pcr。产生了323bp的扩增子,将其连接至pgem-easy并测序。以这三部分序列组装成全长序列。然后从总rnart-pcr扩增具有短5′utr区的全长编码区,rt-pcr扩增使用了正向引物5’-gaaaaaatggttgcgccacccatca-3’(seqidno:86)和反向引物5’-tcactacttcttcttcttgcccgcggc-3’(seqidno:87)。产生了828bp的扩增子,pavpi-elo1,将其连接至pgem-easy并测序(seqidno:93)。p.pinguisδ9-延长酶的推定的氨基酸序列为seqidno:94。类似地,使用简并引物通过pcr从盐生巴夫藻产生了425bp的扩增子,将其连接至pgem-t并测序。设计引物用于通过5′-和3′-race延伸所述425bp片段,通过rt-pcr分离基因的3’末端,使用了基因特异性正向引物5’-ttccggtactcagcggtggcg-3’(seqidno:88)和oligo-dt-sp6反向引物。产生了776bp的扩增子,将其连接至pgem-easy并测序。通过pcr从盐生巴夫藻cdna文库分离该基因的5’末端,使用了m13r引物5’-caggaaacagctatgac-3’(seqidno:89)和基因特异性反向引物5’-acgtagatgccctcgttctg-3’(seqidno:90),按照生产商的说明使用pfuultrafusiondna聚合酶。产生了710bp的扩增子,将其连接至pgem-easy并测序。从这三部分序列组装出全长序列。通过rt-pcr从总rna扩增出具有具有短5′utr区的全长编码区,使用了正向引物5’-caccgaatggcgactgaagggatgcc-3’(seqidno:91)和反向引物5’-ctactcggttttcatgcggttgctgga-3’(seqidno:92)。产生了846bp的扩增子,pavsa-elo3,将其连接至pgem-easy并测序(seqidno:95)。盐生巴夫藻δ9-延长酶推定的氨基酸序列为seqidno:96。在植物细胞中功能性表征δ9-延长酶将位于ecori片段中的分别来自质粒0835668_emihu-d9e_pma、pgemt+pavpi-d9e和pgemt+pavsa-d9e的emiliania延长酶(emihu-d9e)、pavlovapinguis延长酶(pavpi-d9e)和盐生巴夫藻延长酶(pavsa-d9e)的完整编码区插入35s-pore04的ecori位点,产生了35s:emihu-d9e(命名为pjp3027)、35s:pavpi-d9e(命名为pjp3103)、35s:pavsa-d9e(命名为pjp3081)和35s:isoga-d9e(命名为pjp2062)。如实施例1所述,使用增强型本氏烟草瞬时表达系统在植物内证实了emihu-d9e、pavpi-d9e和pavsa-d9e连同作为阳性对照样品的isoga-d9e(qi等,2002)的酶活性。将这些嵌合载体引入根癌土壤杆菌菌株agl1,使来自这些培养物的细胞渗入温室中的本氏烟草植物的叶组织。渗入后,植物再生长5天,然后取叶盘进行gc分析,结果显示,产物脂肪酸的存在表明这两种基因在例如本氏烟草的植物细胞中均发挥δ9-延长酶的功能。以emilianiahuxleyiccmp1516δ9-延长酶转化的叶组织含有20:2δ11,14(6.6%)和20:3δ11,14,17(6.4%),这代表对la和ala的转换效率分别为39.9%和12.4%。以pavlovapinguisδ9-延长酶转化的叶组织含有20:2δ11,14(10.1%)和20:3δ11,14,17(6.6%),这代表转换效率分别为56.0%和13.3%。以盐生巴夫藻δ9-延长酶转化的叶组织含有20:2δ11,14(7.7%)和20:3δ11,14,17(4.6%),这代表转换效率分别为45.0%和9.2%。以球等鞭金藻δ9-延长酶转化的叶组织含有20:2δ11,14(9.2%)和20:3δ11,14,17(7.5%),这代表转换效率分别为48.9%和15.4%(表9)。表9.以δ9-延长酶瞬时转化的本氏烟草叶组织的脂肪酸组成(占总脂肪酸的百分比)。给出了以一式三份进行的不同渗入之间的标准差。叶组织中这种对ω3底物ala的明显的高偏向性是预料之中的,因为本氏烟草叶中的大部分底物ala位于质体内,因此无法被质体外延长作用所利用,也因为在直接甲基化过程中质体和细胞质的ala从叶中被分离,这人为降低了ω3转换率。e.huxleyi和球等鞭金藻δ9-延长酶在本氏烟草中展现出相同的底物偏向性,ω3至ω6的转换率为0.31。观察到的ω6库中最高效的转换涉及盐生巴夫藻δ9-延长酶,其将56.0%的底物发生了转换。相反,13.3%的ω3底物发生了转换,两者的比率为0.24。p.pinguis的酶对ω6底物展现出最高的偏向性,转换率为0.20,因为45.0%的ω6发生转换,而仅有9.2%的ω3发生转换。实施例8.构建包括δ9延长酶的生物合成途以产生ara构建转基因δ-9延长酶途径从二元载体pjp101acq构建了含有球等鞭金藻δ9-延长酶(氨基酸序列genbank登录号af390174–可读框如seqidno:21所示,氨基酸序列如seqidno:22所示)、盐生巴夫藻δ8-去饱和酶(登录号abl96296–可读框如seqidno:23所示,氨基酸序列如seqidno:24所示)、和盐生巴夫藻δ5-去饱和酶(登录号abl96295–可读框如seqidno:25所示,氨基酸序列如seqidno:26所示)的二元载体。此载体没有基因插入物的设计见图14。首先,将含有球等鞭金藻δ9-延长酶的pbluescript克隆的smai-ecorv片段连接至pjp101acq的smai位点以产生pjp105。将含有盐生巴夫藻δ5-去饱和酶的pbluescript克隆的xhoi片段连接至pjp105的xhoi位点以产生pjp106。将含有盐生巴夫藻δ8-去饱和酶的pbluescript克隆的noti片段连接至pjp106的noti位点以产生pjp107,其在图15中图示。关于该设计有若干要点值得关注。首先,所述3个基因中的2个在t-dna上背驰转录,即彼此远离。这么做是为了防止从受关注的任一基因的转录潜在地干扰另一个基因的表达,由此使两者的表达最大化。其次,在该基因构建体中的第三个基因(在此情况下编码所述δ8-去饱和酶)通过插入间隔区而与导向相同方向的第二个基因(编码所述δ9-延长酶)分隔开。据认为上游基因的终止密码子与下游基因的启动密码子之间至少1.0kb的距离会降低前者的转录干扰后者或潜在地引起基因沉默的风险。第三,该三个基因每个的5’-utr被修饰为包括tmv前导序列(已知其用于提供有效率的翻译)。任何其它已知赋予高翻译效率的5’utr序列也可代替所述tmv序列而被使用。通过电穿孔将pjp107引入土壤杆菌菌株agli内并且经转化的菌株被用于将所述基因构建体引入至拟南芥内,所述拟南芥生态型mc49,其是fad3/fae1突变体具有高水平的la作为所述δ8-去饱和酶的潜在初始脂肪酸底物。使用花浸渍法(floraldippingmethod,参见cloughandbent,1998)来进行植物转化和分析。将来自经处理的植物(t0植物)的种子(t1种子)铺板于潮霉素(20mg/l)选择性培养基上,选择转化的植物并将其转移至土壤以建立24株经确认的t1转基因植物。预期这些t1植物的大部分对于所引入的基因构建体都是杂合的。在成熟时收集来自该24株转基因植物的t2种子并分析其脂肪酸组成。这些t2株系包括所述基因构建体是纯合的株系以及所述基因构建体是杂合的株系。从所述t2种子中含有最高ara水平的6个建立t2植物,在含潮霉素(20mg/ml)的ms培养基上选择,以确定所述转基因的存在。例如,所述t2种子栽培自称作fw-10的t1植物,含有5.8%的ara并且在潮霉素培养基上显示出抗性后代对易感后代3:1的分离比,表明fw-10在单一的基因座上含有所述基因构建体。分析了来自fw-10的t3种子组的脂肪酸谱,数据在表10中给出。表10.转化了所述基因构建体pjp107(含有球等鞭金藻δ9-延长酶基因、盐生巴夫藻δ8-去饱和酶基因、和盐生巴夫藻δ5-去饱和酶基因)的拟南芥种子的脂肪酸组成。如表10中所总结的,未经转化的拟南芥(生态型mc49)的种子含有显著量的前体ω6底物la,但却不含有任何ara或预期在所述δ9延长酶途经中出现的中间体脂肪酸。相反,来自经转化的植物fw10-23(含有pjp107构建体)的种子含有显著量的20:2n-6、20:3n-6和20:4n-6(ara),包括21%的ara,用la开始的三个酶步骤的产物。此外,种子油中低水平的ala(在对照mc49中1.0%)被非常有效地转换成epa,其在经转化的株系fw10-23中以1.3%的水平存在。讨论:转换效率及生化含义可以通过测试fw-10-23中底物脂肪酸转换成产物脂肪酸(包括随后的衍生物)的转换百分比来评估所述pjp107构建体所编码的单个酶步骤的相对效率。在ω6库中,球等鞭金藻δ9延长酶显示出45%的la转换成eda以及随后的不饱和脂肪酸。在相同的种子里,盐生巴夫藻δ8-去饱和酶和δ5-去饱和酶分别显示了ω6脂肪酸至其相关产物的90%和95%的转换效率。作为比较,在ω3库中,球等鞭金藻δ9延长酶显示了基本上100%的ala至延长的产物的转换,而盐生巴夫藻δ8-去饱和酶和δ5-去饱和酶分别显示了88%和90%的转换效率。这些酶步骤导致了1.3%epa的合成,尽管拟南芥mc49的背景仅含有低水平的ala。在最戏剧化的结果中,注意到在种子油中未检测到ala,表明基本上100%的ala由所述δ9延长酶转换成ala的延长的产物。有趣的是,注意到fw-10-23中发现的不寻常中间体脂肪酸的水平很低(在ω3库中<0.4%)并且与各种海鲜的食物链中已发现的那些相当(表11)。尽管未经转化的mc49种子油仅含有低水平的ala,并且这可能造成了例如中间体脂肪酸etra的低观测水平;但是据预测,当以具有较高ala水平的遗传背景来组建相同的途径时,所得的种子油依旧具有相对较低水平的etra(<3%)。存在如此低水平的这些中间体很可能是由于经δ9延长的中间体非常有有效的去饱和作用。表11.转化了所述基因构建体pjp107(含有球等鞭金藻δ9-延长酶基因、盐生巴夫藻δ8-去饱和酶基因、和盐生巴夫藻δ5-去饱和酶基因)的拟南芥种子中的脂肪酸,与在海鲜样品的范围中发现的中间体脂肪酸的比较。值得注意的是盐生巴夫藻δ8-去饱和酶在将etra转换成eta方面比起其它经报道的δ8-去饱和酶要有效地多,特别是在与所述δ9延长酶和δ5去饱和酶共表达时。例如,据报道,当小眼虫δ8-去饱和酶与小眼虫或球等鞭金藻δ9-延长酶在大豆胚中共表达时,ω3和ω6底物的转换效率分别是64%和73%。上述实验中所观测的每一步的效率以及ala转换为epa的总转换效率也比qi等(2004)所报道的在拟南芥叶中的高得多,其中他们仅观测到3.0%的epa以及实质水平的不期望的中间体包括etra(4.6%)。已知延长酶仅接近酰基-coa库中的底物。随后的δ8-去饱和酶和δ5-去饱和酶步骤在经转化的种子中被观测到以极高的效率发挥作用,尽管所述经δ9-延长的产物毫无疑问地在酰基-coa库中产生,这一事实强烈表明两种盐生巴夫藻去饱和酶均能够高效地接近酰基辅酶a底物。用所述δ9-延长酶途径生物合成高的ara和epa水平从单个步骤效率的这些数据和观测,预计到可以用修改的δ9-延长酶途径在转基因植物例如拟南芥、芸苔、大豆、亚麻籽或棉花中产生高水平的ara和epa以及随后的dpa和dha。进一步预计到通过再加入三种酶功能的任一种可达到甚至更高的水平,所述三种酶功能即:酰基-coaδ12-去饱和酶功能以提高酰基-coa库中可用的底物la的数量用于δ9-延长作用,第二是加入δ15-去饱和酶以提高ala的水平用于直接转换成epa,以及第三是δ17-去饱和酶其可将ara转换为epa例如实施例6中所述的。更优选地,加入酰基-coaδ12-去饱和酶和所述δ15-去饱和酶或所述δ17-去饱和酶中的任意一种会提供最高的水平。因此,使用能够接近酰基-coa库中底物的酶预期会导致更有效地转换成epa、dha和dha.考虑到拟南芥mc49北京含有fad3突变,其导致低水平的ala聚集(1-3%),则所观测的1.3%epa的合成是值得注意的并且出乎意料的。我们预计,当此(或类似的)δ9-延长酶途径(δ9-延长酶、δ8-去饱和酶、和δ5-去饱和酶)被转化至含有高水平ala的植物时,会得到高水平的epa。例如,我们预计将此途径转化至过表达紫苏(perillafrutescens)δ15-去饱和酶或其它δ15-去饱和酶基因的拟南芥株系,会导致epa的水平为种子油中总脂肪酸的至少25%。实施例9:pufa途径基因在植物细胞中的表达植物稳定转化的一种替代是在叶中转基因的瞬时表达,例如最早由kapila等(1997)所介绍的。用此技术,允许叶细胞的细胞核(zipfel等,2006)经由偏离轴的空气间隔的渗入而转化了在tdna界限内载有表达构建体的土壤杆菌培养物。转基因在叶中的表达显著地由病毒阻抑蛋白的共引入而增强,所述病毒阻抑蛋白如p19(voinnet等,2003)和hc-pro(johansenandcarrington,2001;kasschau等,2003),其抑制宿主细胞的转基因沉默装置并将转基因的表达扩展至更长的时间段。叶具有复杂的脂质代谢,其由质体半乳糖脂单半乳糖基二酰基甘油(mgdg)和二半乳糖基二酰基甘油(dgdg)的巨大的库所控制。更少见的脂肪酸的库存在于质体的室之外包括经卵磷脂(pc)、辅酶a(coa)以及单-和二-酰基甘油酯(mag、dag;ohlrogge和browse,1995)所酯化的那些。此实施例中所用的lc-pufa合成的酶位于内质网(er;napier,2007)其中他们可接触相对较少见的经pc和coa酯化的叶脂质库。pc-coa-连接的反应的代谢产物,例如在er上有活性的那些,可通过二酰基甘油酯-o-酰基转移酶(dgat;bouvier-nave等,2000)的过表达而在三酰基甘油酯(tag)中聚积。与mag或dag相比,位于tag上的脂肪酸更具代谢惰性并且有比较小的可能会重进入脂质生物合成途径或进入质体。重要的是,使用标准薄膜层析技术(tlc)可轻易地将tag从位于叶质体中的丰度更高的脂质类别中分离出来。因此增强的tag聚积和tag/脂质类别纯化的组合可以有助于更完全地理解叶er上的lc-pufa酶反应。在此实施例中用编码去饱和酶和延长酶的基因来测试此系统的lc-pufa生产。用于瞬时表达的质粒构建体通过将基因的编码区克隆至经修饰版本的coutuetal(2007)所述的pore04二元载体中来制备二元载体,所述pore04二元载体中花椰菜花叶病病毒(camv)35s启动子已被克隆至sfoi位点以产生35s-pore04。将球等鞭金藻δ9-延长酶基因编码区(genbank登记号aal37626)(seqidno:21)从基因组dna扩增并克隆至35s-pore04内的ecori位点。将进过植物表达密码子优化的所述三种盐生巴夫藻去饱和酶(genbank登记号abl96296、abl96295和aay15136–每个都在wo2005/103253中有描述)作为swai插入物而克隆至35s-pore04的ecorv-smai位点内。将未经优化的盐生巴夫藻δ5-延长酶(genbank登记号aay15135)作为anxhoi-xbai片段克隆至35s-pore04内的xhoi-nhei位点。由camv35s-推动的版本的p19病毒阻抑物由drpeterwaterhouse所提供。拟南芥的dgat1基因编码区(genbank登记号aaf19262)(seqidno:74)通过rt-pcr获得并作为bamhi-ecorv片段被克隆至35s-pore04中的相应位点。总rna用rneasyminikit(qiagen)分离自致病疫霉以及用platinumsuperscriptiiione-step(qiagen)进行rt-pcr。将所得的含有致病疫霉δ17-去饱和酶蛋白编码区(wo2005/012316)的扩增子克隆pgemt-easy(promega)内并测序。继而将ecori片段克隆至35s-pore04内。土壤杆菌渗入和本氏烟草生长条件载有每个二元载体的根癌土壤杆菌菌株agl1在28℃于补加了适当抗生素的lb培养液中生长。将培养物离心并小心地将其重悬浮两体积的渗入缓冲液(5mmmes,5mmmgso4,ph5.7,100μm乙酰丁香酮)以及进一步生长3小时。测量每个培养物的光密度并制备了培养物的最终组合,从而使每个土壤杆菌构建体等于od600nm0.2或如图16所另外标注。如voinnet等(2003)所述将细胞渗入一个月大的本氏烟草植物的叶的下面,所述本氏烟草植物已被安置在23℃的植物生长室内,且具有10:14的光:暗循环。用记号笔将渗入的区域画圈。在渗入之后,将该植物置于28℃一小时,之后将他们转移到24℃的植物生长室直至分析。除非另有指定,所有的本氏烟草农杆渗入均在存在分离的含p19病毒阻抑蛋白的二元构建体的情况下进行脂质分析本实施例中在用甲醇/hcl/二氯甲烷(dcm;体积10/1/1)溶液于80℃转甲基化2h以产生脂肪酸甲基酯(fame)之后,通过gc和gc-ms分析叶组织的脂肪谱,或脂质类别样品。所述fame在己烷:dcm(4:1,v/v)中提取并在用gc和gc-ms分析前于dcm中进行重建。对于脂质类别分析,用bligh和dyer(1959)所述的方法,从大约50mg鲜重的经渗入的叶组织中提取两次总脂质。中性脂质通过tlc在预包被了硅胶的平板(silicagel60,merck)上用己烷/乙醚/乙酸(体积70/30/1)纯化,而极性脂质用二维tlc分馏,氯仿/甲醇/水(体积65/25/4)用于第一个方向,而氯仿/甲醇/nh4oh/乙基正丙胺(体积130/70/10/1byvol.)用于第二个方向(khozin等,1997)。用碘蒸汽使得脂质的斑点可见,收集入小管并转甲基以产生fame用于如上所述的用于gc分析。tag被作为所存在的脂肪酸的总量的百分比而进行定量,其如上所述通过gc分析并根据对每个脂肪酸所注射的已知量的外部标准进行评估。用agilenttechnologies6890ngc(paloalto,california,usa)来进行gc,其转配了非极性equitytm-1熔融石英毛细管柱(15mx0.1mmi.d.,0.1μm薄膜厚度)、fid、分流/无分流进样器和agilenttechnologies7683series自动取样器以及注射器并使用氦作为载体气。以无分流模式在120℃的炉温注射样品,在注射后将炉温以10℃.min-1升至201℃并最终升至270℃且维持20分钟。用agilenttechnologieschemstation软件(revb.03.01(317),paloalto,california,usa)对峰进行定量。峰应答类似于可靠的nu-checkglcstandard-411(nu-checkprepinc,mn,usa)的脂肪酸,其含有等比例的31种不同的脂肪酸甲基酯,范围从碘苯腈辛酸酯到dha以及几种其它的lc-pufa。峰之间峰应答的轻微差异通过用峰面积乘以每个峰的正态化因子来进行平衡。每种脂肪酸在总脂肪酸中的比例基于脂肪酸的单个和总的峰面积进行计算。进行gc-ms以确认所有形成的新脂肪酸的身份,并且其在finnigangcqplusgc-ms离子收集器(安装了柱上进样器)上于at45℃进行。用as2000自动取样器将样品注射至附着于非极性hp-5ultra2键合相柱(50mx0.32mmi.d.x0.17μm薄膜厚度)的保留隘口上。45℃的初始温度保持1分钟,随后通过温度程序以30℃.min-1升高至140℃继而以3℃.min-1升至310℃并将其保持12分钟。氦被用作载体气。质谱仪操作条件是:电子轰击能量70ev;发射电流250μamp,转运线310℃;源温度240℃;扫描速率0.8次扫描.s-1以及质量范围40-650道尔顿。质谱由xcaliburtm软件获取并处理.用瞬时表达的脂肪酸延长酶修改本氏烟草脂肪酸谱为了评估所需的土壤杆菌的浓度以产生功能性转基因酶的最大生产,表达编码球等鞭金藻δ9-延长酶的基因(igδ9elo;qi等,2002),其已知作用于coa-连接的亚油酸(la)和已知在本氏烟草叶中丰裕的ala底物。在将此基因转移至花椰菜花叶病病毒(camv)35s启动子下游的二元载体后,将此构建体农杆渗入至本氏烟草叶,其中存在p19病毒阻抑蛋白以阻抑宿主介导的转基因沉默,并评估了δ9-延长作用的水平。所述延长作用的产物la和ala、eda和etra分别被检测为接近最大基因活性(得自具有od600=0.2的土壤杆菌培养物)(图16)。然而有趣的是,注意到以浓度十分稀的所述培养物(低至od600=0.05)进行农杆渗入也导致了可轻易检测水平的酶活性。瞬时dgat表达对三酰基甘油聚积的作用接着研究了是否可将本氏烟草叶中的tag库的大小增加提供更大的接收端(sink)以在其中捕获所引入的在er上有活性的脂肪酸生物合成酶的产物。含有拟南芥dgat1(atdgat1)基因(其催化kennedy途径的tag生物合成中的组后一步)的构建体被作为可能的方法进行测试以增加叶中的tag库,因为叶天然仅产生低水平的tag。将该构建体通过如山所述的农杆渗入而引入到本氏烟草叶中。为了测试tag的存在,将经渗入的叶的大约1cm2大小的区段浸入小的含1%水成nileblue(bdh,poole,uk)的皮氏培养皿中,真空渗入3分钟,简单用水清洗,在1%乙酸中温育3分钟,并装载到水中用于保存。在570-670nm用488nm的激发于leicasp2激光扫描共聚焦显微镜上(leicamicrosystems,sydney,australia)收集荧光发射。相同叶的未转化区域被用作对照。用imagej软件评估了每个测定中tag聚积的相对的量。拟南芥dgat1(atdgat1)的瞬时表达导致了显著更多的脂质体的产生,所述脂质体由nileblue染色并用共聚焦显微镜来观测。tag的增加是通过分馏总脂质至tag内来定量的,其中使用中性相的tlc与其他叶脂质类别分离,之后tag的量被测量为tag级分中总脂肪酸的量。p19或p19与atdgat1一起的瞬时表达分别导致tag从46μg.g-1鲜重增加至206μg.g-1鲜重,现实出dgat1基因的加入提高了叶组织中聚积的tag的水平。因此,除非另外指明,则此基因被包括在随后的实验中。外源性脂肪酸底物对瞬时表达的基因的可用性接着测试了对于本氏烟草非天然的外源性脂肪酸底物是否能提供给叶子并变得可用于转基因介导的转换,从而允许单个的酶步骤在分离中进行测试。为了测试此问题,将编码致病疫霉δ17-去饱和酶(piδ17des)(其作用于非天然存在于本氏烟草中的底物ara)的基因进行农杆渗入以产生epa。用piδ17des渗入4天后,以类似于用土壤杆菌培养物转化叶子的方式通过注射而像叶提供ara铵盐。继而在从叶组织中提取总脂质之前,允许该叶代谢底物4小时。这些总脂质的gc和gc-m分析显示出37%的外源提供的ara由δ17-去饱和作用转换成epa,其效果与基于酵母测定中所报道的相当(wo2005/012316).从分离的二元载体快速组装五步lc-pufa途径已经确立本氏烟草系统在确定单一基因的功能以及增强的tag聚积方面是有用的工具,研究了该系统可用于组装theextenttowhichthesystem整个lc-pufa途径的程度。在此研究中,测试了编码5种lc-pufa代谢酶的基因,其产生了平行的两个线性lc-pufa途径,即ω6-pathway,将la转换为dpaω6,以及ω3-途径,将ala转换成dha(图1)。所使用的生物合成基因是球等鞭金藻δ9-延长酶(igδ9elo)、盐生巴夫藻δ8-去饱和酶(psδ8des)、盐生巴夫藻δ5-去饱和酶(psδ5des)、盐生巴夫藻δ5-延长酶(psδ5elo)和盐生巴夫藻δ4-去饱和酶(psδ4des;qi等,2004;robert等,2009;zhou等,2007)。将每个基因分别克隆至如上所述的camv35s启动子下游的植物二元表达载体内,并将这些构建体的混合物(每个以od600nm=0.2的浓度存在)连同atdgat1和p19,农杆渗入至本氏烟草叶的离轴表面内,得到总共7个单个的构建体以及总od600nm=1.4。渗入后5天对叶盘取样并直接从新鲜组织产生脂肪酸甲基酯(fame)并通过gc/ms来分析和坚定(表12)。很清楚的是所有的途径酶都能够接受ω6或ω3pufa的任一作为底物andth并且他们对la或ala的顺序作用分别导致lc-pufa、ara和dha的合成。鉴定出新产生的lc-pufa的百分比是16.9%,包括9.8%的ω6lc-pufa和7.1%的ω6lc-pufa。在所有这新形成的lc-pufa中,ara、epa和dha被认为在营养上很重要,并分别构成叶组织中总脂肪酸的3.6%、2.6%和1.1%。对ω6和ω3途径的每一步计算酶转换效率并与先前的报道进行比较(图17)。ω6和ω3的5步途径的前三步在效果上与qi等的报道类似(qi等,2004),而途径最后两步的效果与robert等(robert等,2005)所受用的相同。瞬时表达的基因与稳定表达的基因的此种对比表明,两种引入途径的方法均产生类似的流通和效果。这些在总脂肪酸谱上计算的转换效率很可能是一种低估,特别是对于第一步,δ9-延长作用,只是由于质体中巨大的la库和ala库的稀释作用。此议题通如下所述的脂质的分馏而完成。表12.产生ω6-和ω3-lc-pufa二者的本氏烟草叶斑点的脂肪酸谱。每个渗入含有土壤杆菌培养物的混合物,所述土壤杆菌培养物载有分离自番茄丛矮病毒和拟南芥二酰基甘油o-酰基转移酶(atdgat1)的p19病毒阻抑物异位表达构建体。lc-pufa途径的渗入包括额外的5个基因,即,球等鞭金藻δ9延长酶(igδ9elo)、盐生巴夫藻δ8-去饱和酶(psδ8des)、盐生巴夫藻δ5-去饱和酶(psδ5des)、盐生巴夫藻δ5-延长酶(psδ5elo)和盐生巴夫藻δ4-去饱和酶(psδ4des)。为了清楚,表中不包括饱和的和较少见的脂肪酸,但其却用于计算百分比。(-)表示无可检测的量的脂肪酸。数据产生自3次重复并显示出标准差。总fame(%)lc-pufa的脂质类别区分为了评估新合成的lc-pufa在tag磷脂和质体半乳糖脂之间的区分将瞬时表达lc-pufa途径基因的本氏烟草叶的总脂质进行如上所述的脂质类别划并确定了其脂肪酸谱(表13)。本氏烟草叶脂质含有高等植物叶中典型的脂质类别和脂肪酸谱(fraser等,2004;moreau等,1998)。新合成的ω6和ω3lc-pufa均主要限于通常在质体外发现的脂质,而质体脂质基本上没有这些脂肪酸。例如,tag和磷脂(pc、pe和pa)–主要的质体外叶脂质–分别包含了高至20.4%和16.9%的新合成的ω6和ω3lc-pufa。值得注意的是,表达全lc-pufa途径的叶,atdgat1和p19产生tag富集了37%的lc-pufa.具有特别兴趣的是营养学上重要的脂肪酸的聚积ara、epa和dha,在叶tag中分别存在7.2%、5.9%和3%。区分显示主要的质体脂质类别,mgdg、dgdg和pg,只分别包含了1.1%和0.3%的新合成的ω6和ω3lc-pufa。尽管这些质体脂质类别代表了叶中脂肪酸最巨大的库,总而言之,这些类别与tag相比只包含小量的ω6和ω3lc-pufa。有趣的是,sqdg脂质类别完全不存在于新合成的lc-pufa。脂质类别区分也被用于计算lc-pufa途径每一步的酶效果,所述lc-pufa途径与用于tag中的脂肪酸的er关联,其无法接触质体脂质(图17)。从这些计算中去除质体脂质类别对ala转换成etra的δ9-延长作用步骤有最显著的效果,将转换效率从16%提高至55%。此步骤的酶转换效率3-倍的增长是由于质体中ala巨大的库其对于此er结合的酶而言不可用(表13)。讨论这些实验显示出,本氏烟草或其它植物叶中的一系列途径基因的瞬间表达模拟了经稳定转化植物中的表达,因此很适宜于且可预计用于产生lc-pufa油料的种子中途径的表达。所述瞬时表达系统提供了可互换的表达平台,其给出了快速且可靠的整个途径的结果,其中在多步骤的重组途径中单个的组分很容易被更换。lc-pufa生物合成的瞬时组装是有力的且可重复的。一式三份进行测定并给出紧凑的数据点,通常标准擦低于5%。所组装的lc-pufa途径中的第一步,δ9延长作用,显示出对la比对ala更高比率的延长作用,与在稳定转化的植物中先前所观测的一样(fraser等,2004)。这种不同不能用酶的脂肪酸底物偏好来解释,酵母中的表达显示出igδ9延长酶具有对la和ala同样的偏好(qi等,2002)。更可能的是对la所观测到的较高比率的延长作用反映出在叶的质体外酰基-coa库(延长酶的作用位点)中存在的la的量高于ala的量(domergue等,2003;fraser等,2004).脂质分类分析证明了所有新形成的lc-pufa在pc库和tag中发现有大致相同的比率。此比率在途径的倒数第二部(产生dta和dpa的δ5-延长作用)有轻微的差异,其在tag上丰度低于pc。相反,最终的δ4去饱和作用的产物,dpaω6和dha,相较于pc而更倾向于在tag上聚集。这些差异可能会揭示出膜编辑的酶的微妙的偏好或atdgat1对这些产物的微妙的偏好。值得注意的是δ5-延长作用和dgat活性均在coa库上出现,这些酶对底物的竞争可以改变脂肪酸在这些pc和tag库中的存在。从这个研究可以得到几个提示。首先,基于叶的瞬时测定显示出适宜于测定脂肪酸酶(单一的或者组合的)。这对于产生容易地与本氏烟草的内源脂肪酸谱区别的脂肪酸的酶而言特别适合。例如本研究中的lc-pufa。对分离的酶的脂肪供给测定以及lc-pufa快速组装成油的证明表明,瞬时叶测定很适合用于er-先关的去饱和作用、延长作用和tag组装。第二,尽管lc-pufa油是植物转化技术现时的目标,但是叶细胞提供了相对于其它异源表达平台的一定范围的优势。叶细胞提供了其它表达宿主不能提供的广泛的代谢产物,而这些现在可以成为需要修改的重组途径的靶标。此外,植物更忠实地处理真核的转基因,包括rna编辑、翻译后修饰和细胞器定位。最后,所述本氏烟草测定可能适用于cdna文库筛选测定。此建议是基于在叶上单一的渗入区中检测到7个不同基因的接近最大的活性,这表明按照此种设置克隆至二元表达载体内的cdna文库可以全身性的渗透到叶内。计算表明,至少7个不同的克隆,包括od600nm0.2的p19,可在单一的斑点中表达。或者,可将形成不完整的或部分途径的基因加入到每个渗入并由此使得汇集的库的克隆能够常规地测试新的步骤或改善的流通。实施例10:在植物细胞中高效的dha生物合成使用如实施例1中所述的增强的本氏烟草瞬时表达系统在植物体中证明了微胞藻ccmp1545δ6-去饱和酶(seqidno:8由seqidno:7编码)、塔胞藻cs-0140δ6-延长酶(seqidno:4由seqidno:3编码)、盐生巴夫藻δ5-去饱和酶(seqidno:26由seqidno:25编码)、塔胞藻cs-0140δ5-延长酶(seqidno:6由seqidno:5编码)和盐生巴夫藻δ4-去饱和酶(seqidno:73由seqidno:72编码)连同拟南芥dgat1(seqidno:74由seqidno:75编码)的酶活性。通过将包含在swai片段内的pga4a的整个编码区插入到35s-pore04(上述实施例4)内的smai-ecorv位点,制备了在组成性35s启动子控制下的编码δ6-去饱和酶的基因构建体35s:mic1545-d6d,产生pjp2064。通过将包含在swai片段内的0804673_pyrco-elo1_pga18的整个编码区插入到35s-pore04内的smai-ecorv位点,制备了编码δ6-延长酶的基因构建体35s:pyrco-d6e,产生pjp2060。通过将包含在swai片段内的0804674_pavsa-d5d_pga15的整个编码区插入到35s-pore04内的smai-ecorv位点,制备了编码δ5-去饱和酶的基因构建体35s:pavsa-d5d,产生pjp2067。通过将包含在swai片段内的0804675_pyrco-elo2_pga4的整个编码区插入到35s-pore04内的smai-ecorv位点,制备了编码δ5-延长酶的基因构建体35s:pyrco-d5e,产生pjp2061。通过将包含在swai片段内的0804676_pavsa-d4d_pga15的整个编码区插入到35s-pore04内的smai-ecorv位点,制备了编码δ4-去饱和酶的基因构建体35s:pavsa-d4d,产生pjp2068。通过将包含在bamhi-ecorv片段内的pxzp513e的整个编码区插入到35s-pore04内的bamhi-ecorv位点,制备了编码酶dgat1的基因构建体35s:arath-dgat1,产生pjp2078。将这些嵌合载体个别地引入到根癌土壤杆菌菌株agl1内,以及将来自这些的培养物的转基因细胞混合并将混合物在温室内渗入到本氏烟草植物的叶组织中。在渗入后将植物再培养5天,随后取叶盘用于gc分析,其表明这些基因在发挥功能以在本氏烟草中产生dha。用这些基因转化的叶组织含有sda(2.3%)、eta(0.7%)、epa(0.8%)、dpa(2.8%)和dha(4.4%)(表14)。叶组织还含有痕量水平的gla、eta和ara。转换效率如下:细胞中所产生的ala的17.4%被转化成epa(包括epa随后被转换成dpa或dha);15.5%的ala被转换成dpa或dha;细胞中所产生的ala的9.6%被转换成dha;而细胞中所产生的δ6-不饱和的ala的40%随后被转换为dha。由于本实验中转基因的瞬时表达,在稳定转化的细胞中会预期有比上述更高的功效。当由tlc分馏从叶组织提取的总脂质以分开脂质类别时,以及由fame来分析tag和极性脂质级分的脂肪酸组成时,观测到在tag中的dha水平作为总脂肪酸百分比是7%,并且在极性脂质中dha的水平是2.8%。极性脂质类别中较低的水平被认为是由于叶中叶绿体脂质的相关贡献,倾向于极性脂质,以及基因的瞬时表达,而不是转基因稳定的插入宿主细胞基因组。表14.来自经去饱和酶和延长酶的组合转化的叶的脂质的脂肪酸组成。此实验证明了,分离的微胞藻ccmp1545δ6-去饱和酶对ω3底物ala与ω6底物la相比具有实质的偏好。该实验也证明了适宜基因的表达可以导致实质百分比的lc-pufa在叶中的聚积,最值得注意的是epa、dpa和dha。次实验也显示了本氏烟草瞬时测定系统用于快速测试各种脂肪酸生物合成途径以及用于选择最佳的基因组合的用途。此系统可用于快速地对具有同源功能的基因的相对活性进行比较,以及比较整个生物合成途径。讨论:在叶和种子组织中高效的dha合成在此数据的基础上预计到,当种子特异性启动子被用于表达此基因组合或者类似的组时,在种子中汇产生相同水平epa、dpa和dha,或甚至更高的水平。所观测到的从ala到epa直到dpa和dha的脂肪酸的高效转变被认为是由于高效的延长酶与酰基-coa去饱和酶的组合,由此主要在酰基-coa库中对脂肪酸起作用。此外,预计可以通过使用具有适当的组织特异性的启动子或者启动子的组合,来实现在转基因植物的叶和种子中或者在种子和叶之外的其它组织中产生epa、dpa、dha和其它lc-pufa。融合的启动子将能够推动在该两种组织类型中酶的产生。所得的植物将或许可用于油料的提取(特别是从种子中),以及具有最少加工的原料。实施例11:植物细胞中更有效的dha生物合成利用如实施例1和实施例10所述的增强型本氏烟草瞬时表达系统,除了拟南芥dgat1(seqidno:74,由seqidno:75编码)以外,植物内(inplanta)证实了微胞藻ccmp1545δ6-去饱和酶(seqidno:8,由seqidno:7编码)、塔胞藻cs-0140δ6-延长酶(seqidno:4,由seqidno:3编码)、盐生巴夫藻δ5-去饱和酶(seqidno:26,由seqidno:25编码)、塔胞藻cs-0140δ5-延长酶(seqidno:6,由seqidno:5编码)和盐生巴夫藻δ4-去饱和酶(seqidno:73,由seqidno:72编码)的酶活性。该实验利用更年幼和健康的本氏烟草植物进行了优化。将实施例10所述的这些嵌合载体分别引入根癌土壤杆菌菌株agl1,并且在温室中将来自这些培养物的转基因细胞混合,并将混合物渗入本氏烟草植物的叶组织。在渗入后使植物再生长5天,然后取叶盘进行gc分析,这表明这些基因在本氏烟草中发挥功能以产生dha(表15和16)。用这些基因转化的叶组织含有sda(2.0%)、eta(0.4%)、epa(0.7%)、dpa(4.3%)和dha(4.4%)。叶组织还有痕量水平的gla、eta和ara。转换效率为:23.4%的细胞中产生的ala转换为epa(包括随后转换为dpa或dha的epa);21.6%的ala转换为dpa或dha;10.9%的细胞中产生的ala转换为dha;而37.2%的细胞中产生的d6-去饱和的ala随后转换为dha。由于该实验中转基因的瞬时表达,在稳定转化的细胞中预期比以上效率更高的效率。当通过tlc分级从叶组织提取的总脂质以分离类型,并且通过fame分析tag和极性脂质组分的脂肪酸组成时,观察到tag中的dha水平是总脂肪酸的15.9%,并且在极性脂质中dha水平为4.4%。极性脂质类型中较低的水平据认为是由于叶子中叶绿体的相对贡献、有利于极性脂质以及基因的瞬时表达而不是转基因稳定插入宿主细胞基因组。表15.来自用去饱和酶和延长酶的组合转化的叶子的脂质的脂肪酸组成表16.来自用去饱和酶和延长酶的组合转化的叶子的转换效率讨论:叶子和种子组织中更有效的dha合成由于叶子和种子组织之间质体外脂质合成机制的重要转换,该结果可能为种子tag的类似进展做好准备(ohlroggeandbrowse,2004;bates等,2007)。我们推测几种因素可能是产量如此大量增加的原因:1.ω3-特异性酰基-coaδ6-去饱和酶的使用增加了ω3途径的流向(fluxdown)并减少了与平行w6底物对随后的代谢步骤的竞争;2.高效δ5-延长酶明显增加了可用于δ4-去饱和作用而转换为dha的dpa的量;3.通过使用独立转录单元和使用病毒阻抑蛋白(p19)减少了基因沉默。当试图改造积累ω3lc-pufaepa和dha的陆生植物时,所述δ6-去饱和酶所表现出的强ω3优选是明显期望的,因为并不要求额外的δ17-去饱和酶活性来将aa(20:4d5,8,11,14)转换为epa,因而简化了代谢工程和调控挑战。除了上述优化步骤以外,使用高效p.cordataδ5-延长酶导致了非常类似于金枪鱼油的tag(油)组分的脂肪酸谱(profile),所述金枪鱼油是一种高dha和低中间体含量的鱼油(图18)。此外,本氏烟草中p.cordataδ5-延长酶所表现出的活性是迄今为止我们所知的最有效的δ5-延长作用,并且该基因的使用有效地克服了在转基因dha生产的其他尝试中所经历的大δ5-延长作用瓶颈。最后,尽管明显要求使用最优基因,但是我们认为这些转基因引入的方法(即,作为独立表达盒和在基因沉默阻抑物的存在下)在本研究中所实现的高水平dha中起到关键作用。因此,lc-pufa生产的代谢工程迄今为止依赖于随机插入宿主基因组的大多基因构建体,尽管许多组通过该方法获得了良好的结果,但是还有迹象表明难以获得表现出所有转基因相等表达的事件(wo2004/017467)。此外,沉默效果可能降低产生的效率(matzke等,2001)。诸如涉及在独立组装单元和改造的小染色体上从头着丝粒形成的人工染色体的替代性转化方法最后可能是成功的稳定lc-pufa代谢工程所要求的(yu等,2007)。实施例12:利用单个生物的基因的完全ala至dha途径的转基因组装利用编码来自盐生巴夫藻的酶的基因重建完全ala至dha途径并在本氏烟草组装,该途径由所述δ9-延长酶、δ8-去饱和酶、δ5-去饱和酶、δ5-延长酶和δ4-去饱和酶组成。土壤杆菌渗入后5天的总叶组织的gc分析证实产生了0.7%dha(表17)。这是首次报导的由单个生物的基因组成的ala至dha转基因途径。表17.瞬时转化了单基因camv35s二元构建体的本氏烟草叶组织的脂肪酸组成(总脂肪酸的百分比)。dha途径由盐生巴夫藻δ9-延长酶、δ8-去饱和酶、δ5-去饱和酶、δ5-延长酶和δ4-去饱和酶组成。示出了一式三份进行的单独渗入之间的标准偏差。实施例13:植物的发育中的种子中vsp的特异性表达将35s启动子控制之下的5个病毒阻抑蛋白(vsp)即p19、v2、p38、pepo和rpv-p0的蛋白编码区初始插入二元载体part27(gleave,1992)以用于植物组织中的组成型表达。这些蛋白如下表征为vsp。p19是来自番茄丛矮病毒(tbsv)的阻抑蛋白,其结合21个核苷酸的长sirna然后指导argonaute指导的同源rna切割(voinnet等,2003)。v2是来自番茄黄化曲叶病毒(tylrv)的阻抑蛋白,其结合植物蛋白sgs3(glick等,2008),这是一种据认为是从ssrna底物产生双链rna中间所需的蛋白(beclin等,2002)。p38是来自芜菁皱病毒(tcv)的阻抑蛋白,其抑制对产生sirna关键的依赖于rna的聚合酶活性(rdrp)并结合切酶蛋白dcl4(dingandvoinnet,2007)。诸如pepo和rpv-p0的p0蛋白来自马铃薯卷叶病毒,其靶向argonaut以增强降解(baumberger等,2007;bortolamiol等,2007)。为了确立这些蛋白作为沉默阻抑物增加转基因表达的功能,将土壤杆菌中5个35s驱动的vsp构建体与35s驱动的gfp构建体共渗入本氏烟草叶子中。在所有的情况下,vsp的存在增加并延长了gfp的表达,这赋予了gfp基因活性的增加水平,特别是在用土壤杆菌柱接种4天后,从而证实在这种测定模式中所述蛋白作为沉默阻抑物的功能。将5个vsp编码区各自插入基于part27骨架载体的第二二元载体pxrz393,从而vsp的表达在种子特异性fp1启动子的控制之下(ellerstrom等,1996),这提供了vsp在双子叶植物的发育中的种子的子叶中的表达。根据实施例1所述的方法,将构建体用于产生转化的拟南芥植物。对于每个编码vsp的嵌合基因获得至少20个转化的植物。如通过在选择培养基和土壤中生长、正常生长为产生成活的种子的可繁殖成体植物所证实的,所述植物是成活的并且大部分表型上是正常的。唯一在幼苗中看起来被改变的形态学表型是在p19、pepo和rpv-p0萌发后从一些种子出现的子叶。与野生型植株不同,用种子特异性编码p19的构建体转化的植株具有扁平的棒状子叶并且没有向下卷曲。用编码pepo的构建体转化的植株产生了“芭蕾舞短裙”表型,其中子叶以向内或凹面卷曲向上。这些植物上的真叶正常发育。表达rpv-p0的植株是茂密的,并且在整个营养生长中趋向于保留茂密的生长习性。v2和p38的植株没有明显的显著表型。除了子叶发育以外,用p19和pope转化的植株正常生长,并且在随后的生长和发育中与对照植物没有区别。在该实验中,表达vsp的启动子fp1的特征为拟南芥种子发育期间发育的子叶中有限但强的表达(ellerstrom等,1996)。在新生子叶表型的基础上,我们认为发育中的种子中p19、pepo或rpv-p0的fp1-驱动的表达可能与正常子叶发育所需的小rna生物发生重叠。子叶发育中这些vsp相关的变化并不影响转基因植物的总体发育,并且植物正常的随后生长和发育表明除了发育中的种子以外的组织中来自fp1启动子的任何vsp渗漏表达是微量且不明显的。这与植物组织中许多vsp的组成型表达是非常有害的之前研究形成对比(mallory等,2002;chapman等,2004;chen等,2004;dunoyer等,2004;zhang等,2006;lewsey等,2007:meng等,2008)。缺少vspv2和p38的表型表明这些vsp以不影响发育的小rna生物发生或识别的方式靶向小rna代谢。利用本氏烟草中的gfp测定检查了每种vsp的功能。实施例14:发现和评价用vsp构建体转化的t1种子的种子特异性可视标记物的开发实施例13的数据表明种子以及随后的子代植物可以耐受来自种子特异性启动子的vsp表达而没有明显的有害效果。本发明人还考虑了如上文所述产生的转基因植物是否仅表达低水平的vsp,从而允许种子存活,有效地针对潜在地表达致死量的vsp并因此在所用条件下无法恢复的转化种子进行选择。为了更准确地评价t1种子中转基因的表达,开发了用于转基因种子中的转化和表达的可视标记物。对于其他双子叶植物种子,拟南芥种子含有被母本种皮所围绕的父本胚和胚乳。在拟南芥的土壤杆菌介导的转化中,父本组织被t-dna转化,而母本组织保持未转化。直到产生下一代的t2种子才获得转化的母本组织。为了提供有用的筛选系统,将编码绿色荧光蛋白(gfp)的基因进行修饰以促进细胞外蛋白的强分泌。这样的gfp定位据证实允许转化的t1种子的可视恢复(nishizawa等,2003),这是通过检测转化的t1胚和胚乳中产生的gfp通过薄但是未转化的种皮的分泌而实现的(fuji等,2007)。编码分泌gfp的嵌合基因如下构建。所述基因含有两个内含子,一个在5’非翻译区(5’utr),第二个在gfp的蛋白编码区。包含这些内含子以增强嵌合基因的表达(chung等,2006),并保证种子中检测到的任何gfp信号只能来自植物细胞中基因的表达而不是土壤杆菌细胞的渗漏表达。根据nishizawa等(2003)的报导,将编码定位于胞质的gfp蛋白的内含子中断形式的人源化gfp(clontech)(brosnan等,2007)修饰以促进通过内质网(er)向质外体的分泌。用于质外体分泌的修饰的gfp构建体包括在gfp的n端作为框内(in-frame)翻译融合而加入的伴大豆球蛋白(conglycinin)分泌肽和在c端加入的4个甘氨酸以促进从er的分泌。对嵌合gfp基因的这些加入通过5’区和用于c端甘氨酸增加的pcr介导的序列修饰的基因合成来进行。将该分泌gfp的编码区插入fp1启动子的下游,其在5’utr中具有来自过氧化氢酶的内含子。将完全fp1-分泌gfp序列插入pore系列二元载体中的nos3’加a信号的上游。将含有编码分泌gfp的嵌合基因的构建体命名为pcw141。为了证实gfp蛋白的表达和分泌,将编码分泌gfp序列但不具有fp1启动子或pcw141的5’utr的基因区亚克隆至pcamv35s–ocs3’表达盒以产生pcw228,并通过土壤杆菌介导的转化引入本氏烟草叶子。基因的表达和gfp蛋白的分泌利用共焦显微术(leica)来证实。在构建体是正确的基础上,通过实施例1所述的土壤杆菌介导的方法将pcw141引入拟南芥col-0生态型的植物。将拟南芥植物的种子用pcw141,fp1-驱动的分泌gfp构建体浸泡,收集并利用配备了荧光检测的解剖镜(leicamfziii)筛选gfp阳性种子。发出绿色荧光的种子是容易鉴定的,即使在群体中大部分t1种子是未转化的。筛选这些gfp阳性种子,并在选择培养基上生长以证实t-dna上连接至嵌合基因构建体的卡那霉素抗性选择标记物基因的存在。汇集其他20个阳性种子,并通过利用抗gfp抗体的蛋白印迹来证实gfp蛋白的表达。利用分泌的gfp标记物选择t1种子中vsp表达将各自在发育的子叶特异性表达的fp1启动子控制之下的编码vsp:p19、p38、v2和p38的4个嵌合基因的每一个分别插入上文所述的gfp选择载体pcw141以产生pcw161、pcw162、pcw163和pcw164。这些二元载体各自具有用于vsp表达和分泌gfp的连接的嵌合基因,从而允许通过gfp表型来鉴定、选择和分析用构建体转化的转基因种子而无需在选择培养基上生长。预期在大部分转化子中,编码vsp的基因会被整合从而与编码gfp的基因连接。收集获得自拟南芥植物的种子,并如上文所述筛选gfp荧光,所述拟南芥植物根据实施例1中的方法用含有vsp-gfp构建体组合的土壤杆菌接种。通过手工收集发出绿色荧光的种子,并在选择培养基上生长以确定它们是否也转化了选择标记物基因。在所有的情况下,gfp阳性种子在选择培养基上生长,并表现出与上文所述用vsp基因转化但没有gfp基因的植物相同的子叶表型。所观察到的gfp样子种子无论如何在选择培养基上生长。这样的种子可以是预期的,如果某些转化事件导致发育中的种子中的细胞,其中vsp表达水平过高并引起致死。转化的群体中不存在此类种子表明,当从fp1启动子表达时,种子中vsp表达是耐受的。不存在此类有害效果与当许多vsp组成型表达时的有害效果的报导形成对比(mallory等,2002;chapman等,2004;chen等,2004;dunoyer等,2004;zhang等,2006;lewsey等,2007:meng等,2008)。因此,使用诸如gfp的可视检测的标记物证实了鉴定、选择和分析连接的基因的有力且有效的方式,所述连接的基因例如编码vsp、去饱和酶、延长酶或其他脂肪酸修饰酶的基因。胚后表达vsp的种子中gfp表达的定量利用荧光显微术和数字成像分析,在从fp1启动子表达vsp的t1和t2种子中定量gfp表达。这些分析清楚地表明,gfp表达不受发育的子叶-特异性启动子控制之下的编码vsp的基因的共引入的影响。分析了gfp-vsp构建体在后续代和独立转化事件中的性能的扩展研究。预计vsp的存在会导致种子的连续代中更稳定且更高的平均表达水平。实施例15:种子中vsp与lc-pufa合成基因的共表达为了确立vsp能够保护或增强种子中的转基因性能,将一些表达载体设计为考虑对宿主介导的抑制(沉默)比对仅具有单基因的载体易感,以增加vsp的相对效率。利用各个基因的相同配置,构建了各自含有种子中dha合成的5个去饱和酶或延长酶基因的一系列载体。认为使得这些构建体对沉默(降低的表达)更易感的一个因素是使用相同的启动子(fp1)来驱动每个基因。使用fp1启动子,因为其相对较小,并且减小了总载体大小和每个编码区之间的间隔。此外,每个基因盒具有相同的朝向,我们认为这会增强沉默的可能性。lc-pufa途径的三个基因具有编码区,其对于最优植物表达是密码子优化(a-b-c)的;而两个(e-d)是获得自微藻类的天然序列。相同组的5个基因之前在叶子中表达以产生组装完整的lc-pufa生物合成途径(实施例11)。在该系列的第一载体中包含编码vspp19的另一基因,在第二载体中包含编码v2的基因,而该系列的第三载体则不具有vsp。如下构建了这些载体pjp3057(图19)、pjp3059(图20)和(图21)并平行转化。首先在克隆载体中包含的fp1启动子和nos终止子之间克隆去饱和酶或延长酶基因或病毒阻抑物基因。然后顺序并以相同的朝向将启动子-基因-终止子盒克隆至二元载体骨架。pjp3057含有整个dha途径,而pjp3059和pjp3060的差异分别是加入了fp1-p19-nos或fp1-v2-nos盒。构建步骤如下。首先,将含有细小微胞藻δ6-去饱和酶的asci-paci片段克隆至pjp2015的asci-paci位点,然后将来自该载体的含有整个启动子-基因-终止子盒的swai片段克隆至pore02的ecorv位点以产生pjp3050。随后,将含有p.cordataδ6-延长酶的asci-paci片段克隆至pjp2015tmv(pjp2015略微修饰的形式,其中tmv前导序列存在于启动子的下游和基因的上游)的asci-paci位点,然后将来自该载体的含有整个启动子-基因-终止子盒的swai片段克隆至pjp3050的t4dna聚合酶处理的saci位点以产生pjp3051。随后,将含有盐生巴夫藻δ5-去饱和酶的asci-paci片段克隆至pjp2015tmv的asci-paci位点,然后将来自该载体的含有整个启动子-基因-终止子盒的swai片段克隆至pjp3051的smai位点以产生pjp3052。随后,将含有盐生巴夫藻δ5-去饱和酶的asci-paci片段克隆至pjp2015tmv的asci-paci位点,然后将来自该载体的含有整个启动子-基因-终止子盒的swai片段克隆至pore02的smai位点以产生pjp3054。随后,将含有p.cordataδ5-延长酶的asci-paci片段克隆至pjp2015tmv的asci-paci位点,然后将来自该载体的含有整个启动子-基因-终止子盒的swai片段克隆至pjp3054的stui位点以产生pjp3055。随后,将含有盐生巴夫藻δ4-去饱和酶的asci-paci片段克隆至pjp2015tmv的asci-paci位点,然后将来自该载体的含有整个启动子-基因-终止子盒的swai片段克隆至pjp3056的sfoi位点以产生pjp3056。随后将pjp3056的pmei-noti片段克隆至pjp3051的pmei-noti位点以产生pjp3057,其是含有从ala产生dha的5个基因的二元载体。随后将含有编码p19病毒阻抑物的嵌合基因的asci-paci片段克隆至pjp2015tmv的asci-paci位点,然后将来自该载体的含有整个启动子-基因-终止子盒的swai片段克隆至pjp3057的zrai位点以产生pjp3059。类似地,将含有编码v2病毒阻抑物的嵌合基因的asci-paci片段克隆至pjp2015tmv的asci-paci位点,然后将来自该载体的含有整个启动子-基因-终止子盒的swai片段克隆至pjp3057的zrai位点以产生pjp3060。将所有三个构建体在拟南芥中转化(生态型columbia)。用每个构建体转化拟南芥植物(col-0生态型)并用pjp3057转化油菜。收集t1种子,在含有除草剂的培养基上分析,并分析所得t2种子的一般形态学变化和lc-pufa合成。使用三个构建体pjp3057、pjp3059和pjp3060产生的转化的植物(拟南芥,生态型columbia)自受精并收集t1种子。将这些种子播种在含有卡那霉素的培养基上以确定t1植物的杂合性/纯合性,并且分析从每个t1植物所得的t2种子的一般形态学变化和lc-pufa合成(表18)。用pjp3057转化的植物的代表性t2种子在种子油中的总脂肪酸中含有sda(0.4%)、eta(0.6%)、epa(0.2%)、dpa(0.3%)和dha(2.4%)。t2植物的种子油还含有gla(1.4%)以及痕量的etra和ara。种子中的转换效率如下:细胞中产生的18.4%的ala为δ6-去饱和的;细胞中产生的89.7%的sda为δ6-延长的;细胞中82.9%的eta为δ5-去饱和的;细胞中93.1%的epa为δ5-延长的;细胞中88.9%的dpa为δ4-去饱和以产生dha(表18)。用pjp3059转化的植物的代表性t2种子含有sda(0.7%)、eta(0.3%)、epa(0.2%)、dpa(0.9%)和dha(1.3%)。种子油还含有gla(0.8%)以及痕量的etra和ara。转换效率如下:细胞中产生的15.7%的ala为δ6-去饱和的;细胞中产生的79.4%的sda为δ6-延长的;细胞中88.9%的eta为δ5-去饱和的;细胞中91.7%的epa为δ5-延长的;细胞中59.1%的dpa为δ4-去饱和以产生dha(表18)。表18.用pjp3057、pjp3059、pjp3060转化的t2拟南芥种子的代表性脂肪酸分布用pjp3060转化的代表性t2种子含有sda(0.6%)、eta(0.7%)、epa(0.3%)、dpa(0.2%)和dha(1.0%)。种子油还含有痕量的gla、etra和ara。转换效率如下:细胞中产生的13.3%的ala为δ6-去饱和的;细胞中产生的78.6%的sda为δ6-延长的;细胞中68.2%的eta为δ5-去饱和的;细胞中80.0%的epa为δ5-延长的;细胞中83.3%的dpa为δ4-去饱和以产生dha(表18)。结果构建体pjp3057中除δ6-去饱和酶之外的所有基因表现出高活性/高转换效率。这表明δ6-、δ5-和δ4-去饱和酶可能作用于酰基辅酶a底物,因为天然底物ala由酰基-pc去饱和酶产生,导致较低的δ6-去饱和作用,并且转基因去饱和酶底物eta和dpa由已知在酰基-coa库中作用的延长酶产生。此外,δ6-和δ5-延长酶步骤的高效率(>80%效率)表明前面紧邻的那个步骤的去饱和酶(分别为δ6-和δ5-去饱和酶)作用于酰基辅酶a底物。可以合理地预期当达到纯合性时,在转基因植物的后代中这些基因的活性会增加,并且作为结果lc-pufa产物的水平会增加。在构建体pjp3057和pjp3059中沉默阻抑物的存在增加了种子油中新脂肪酸的总水平和途径的终产物dha的水平。讨论对于实施例13-15,引入的外源核酸可以被植物作为外源dna或rna检测,从而由于宿主介导的转基因抑制机制而导致降低的表达。这些抑制机制可以通过小rna群体的生物发生而靶向转基因,并且这些小rna指导抑制装置来限制转基因的表达(matzke等,2001)。转基因可以通过各种方式来限制,包括染色体插入位点处的dna直接修饰,例如通过甲基化,或者通过rna(hamiltonandbaulcombe,1999;voinnet等,2003)或蛋白(brodersen等,2008)水平的转录后沉默。引发这样的抑制机制的外源dna或rna的特征尚未完全了解(lindbo等,1993;lechtenberg等,2003)。但是这样的宿主介导的转基因表达抑制更可能具有这样的特性,其要求高表达、多转基因以及具有互相类似或与宿主基因组类似的区域的转基因(schubert等,2004)。而且,转基因性能可以随着随后的每代而逐渐降低,这很可能是由于转基因的启动子和编码区的甲基化(hagan等,2003)。在本文中,我们证实拟南芥中从胚发生后种子特异性启动子表达的病毒阻抑蛋白(vsp)在发育上是耐受的。各种vsp与可定量特性gfp的共表达表明,重组特性在表达vsp的种子中也是耐受的(实施例14)。由于已知vsp阻断构成转基因抑制装置的小rna代谢,所以我们认为种子中具有重组特性的vsp的共表达会保证,这些特性会以高且不降低的水平在许多代中保持。由于植物耐受胚发生后诸如p19和pepo的vsp表达,所以这表明内源性发育信号,至少那些利用小rna的信号在植物发育的这一晚期阶段是较低的或较不重要的。本研究所选的4种vsp很可能在小rna生物发生的不同部分发挥作用,并因此发挥不同程度的功能。通过使用共引入的vsp来降低多转基因盒中的沉默效果,可以预见转基因表达策略的许多变化。首先,可以重复使用具有较少要求的相同表达盒以避免调控序列或编码区之间的序列重复。因此,这一特性可以允许利用相同的启动子-加a信号来构建大多基因表达载体。可选地,单基因的多个拷贝可以用于增加表达水平,并具有降低的可能性或程度的沉默效果的发生或者在植物的代中增加表达稳定性。实施例16:利用种子特异性启动子在植物的叶细胞中瞬时表达基因如下利用增强的本氏烟草瞬时表达系统,在植物的叶组织中证实了微胞藻ccmp1545δ6-去饱和酶(seqidno:8,由seqidno:7编码)、塔胞藻cs-0140δ6-延长酶(seqidno:4,由seqidno:3编码)、盐生巴夫藻δ5-去饱和酶(seqidno:26,由seqidno:25编码)、塔胞藻cs-0140δ5-延长酶(seqidno:6,由seqidno:5编码)和盐生巴夫藻δ4-去饱和酶(seqidno:73,由seqidno:72编码)基因编码的蛋白的酶活性,每个基因均在种子特异性启动子的控制之下。将实施例15所述含有5个dha生物合成基因的嵌合载体pjp3057引入根癌土壤杆菌菌株agl1,每个基因均在种子特异性截短的napin启动子fp1的控制之下。通过将密码子优化的拟南芥多叶子叶2(arath-lec2)基因克隆至35s:pore04的ecori位点来产生命名为35s:lec2的嵌合载体。将35s:lec2构建体单独引入根癌土壤杆菌菌株agl1。将来自含有pjp3057或35s:lec2的agl1单独培养物的转基因细胞混合并且将混合物渗入本氏烟草植物的叶组织。渗入之后使植物再生长4天,然后取叶盘用于叶子脂质中的总脂肪酸和分离的脂质组分的gc分析。这表明这些基因在本氏烟草中发挥功能以产生dha(表19)。用这些基因转化的叶组织含有sda(1.2%)、eta(2.0%)、epa(0.6%)、dpa(1.7%)和dha(2.5%)。所述叶组织还含有gla(2.4%)和痕量的其他长链ω6脂肪酸。将嵌合载体pjp3115和pjp3116(实施例17)引入根癌土壤杆菌菌株agl1。将来自4个含有pjp3115、pjp3116、35s:p19和35s:lec2中任一个的agl1单独培养物的转基因细胞混合并且将混合物渗入本氏烟草植物的叶组织。渗入之后使植物再生长4天,然后取叶盘用于gc分析,显示这些基因在本氏烟草中发挥功能以产生dha(表19)。用这些基因转化的叶组织含有sda(5.6%)、eta(1.4%)、epa(0.2%)、dpa(1.7%)和dha(2.4%)。所述叶组织还含有痕量的长链ω6脂肪酸。该实验证实双构建体pjp3115和pjp3116组合发挥功能以和含有所有8个基因的单构建体一样有效地产生dha。表19.用各种构建体瞬时转化的本氏烟草叶组织的脂肪酸组成(总脂肪酸的百分比)。误差表示一式三份进行的单独渗入之间的标准偏差。讨论:种子特异性构建体的快速失败和验证利用转录因子,在这种情况下为lec2,联合每个在诸如种子特异性启动子的组织特异性启动子的控制之下的一套基因的实验表明,这样的构建体可以在组织特异性启动子不会正常表达的诸如叶的异源系统中测试,并且预测在种子中表达。在叶细胞中瞬时表达种子特异性启动子的能力允许构建体设计的快速验证。确定种子特异性启动子的效果的实验,尤其是在多基因构建体的背景下,先前依赖于稳定转化至油料种子模式植物或农作物,之后产生后代系,然后表型分析可以确定构建体在植物种子中的效果。在本氏烟草中获得的脂肪酸的水平与用如实施例15所述的该相同构建体稳定转化的拟南芥中所见的水平相似的事实增加了对该测定的适用性的信心。实施例17:用于dha生物合成的双构建体如下构建载体pjp3115(图22)。首先,将载体pjp101acq(图14)的sbfi-apai片段克隆至pore03的psti-apai位点以产生pjp3011。然后,将含有密码子优化的细小微胞藻δ6-去饱和酶(seqidno:125)的swai片段克隆至pjp3011中t4dna聚合酶处理的xhoi位点以产生pjp3108。然后将含有密码子优化的盐生巴夫藻δ5-去饱和酶(seqidno:127)的swai片段克隆至pjp3108中t4dna聚合酶处理的noti位点以产生pjp3109。将含有密码子优化的p.cordataδ6-延长酶(seqidno:126)的swai片段克隆至pjp3109中的smai位点以产生pjp3110。然后通过将pjp3110的bsiwi-asisi片段克隆至pore04的bsiwi-asisi位点来将构建体从抗basta构建体转换为抗卡那霉素构建体,产生pjp3111。将含有截短的napin启动子fp1和crepispalestinaδ12-去饱和酶的ncoi(t4dna聚合酶处理)-sbfi片段克隆至pjp3111中的ecorv-psti位点以产生pjp3115。如下构建载体pjp3116(图23)。首先将含有密码子优化的p.cordataδ5-延长酶(seqidno:128)的swai片段克隆至pjp3011中t4dna聚合酶处理的xhoi位点以产生pjp3112。将含有密码子优化的盐生巴夫藻δ4-去饱和酶(seqidno:129)的swai片段克隆至pjp3112中的smai位点以产生pjp3113。然后将含有perillafrutescensδ15-去饱和酶的noti片段克隆至pjp3113中的noti位点以产生pjp3114。然后通过将含有潮霉素抗性盒(由花椰菜花叶病病毒35s启动子随后从二元载体pwvec8获得的cat-1内含子断裂潮霉素b磷酸转移酶基因和nos终止子组成)的xbai-mlui片段克隆至pjp3114的avrii-mlui位点来将构建体从抗basta构建体转变为抗潮霉素构建体以产生pjp3116。将嵌合载体pjp3115和pjp3116分别引入根癌土壤杆菌菌株agl1,并且将来自这些培养物的转基因细胞与用35s:p19转化的agl1混合,然后在温室中将混合物渗入本氏烟草植物的叶组织。渗入之后使植物再生长5天,然后取叶盘用于gc分析,显示这些基因在本氏烟草中发挥功能以产生dha(表20)。用这些基因转化的叶组织含有sda(5.6%)、eta(1.4%)、epa(0.2%)、dpa(1.7%)和dha(2.4%)。所述叶组织还含有痕量的gla、eta和ara。转换效率如下:细胞中98.9%的油酸为δ12-去饱和的(与对照样品没有显著不同);细胞中95.4%的la为δ15-去饱和的;细胞中产生的18.1%的ala为δ6-去饱和的;细胞中产生的50.4%的sda为δ6-延长的;细胞中75.4%的eta为δ5-去饱和的;细胞中95.4%的epa为δ5-延长的;细胞中58.5%的dpa为δ4-去饱和以产生dha。两个构建体均用于转化油菜。收集t1种子并分析一般形态学变化和lc-pufa合成水平。表20.用各种构建体瞬时转化的本氏烟草叶组织的脂肪酸组成(总脂肪酸百分比)。误差表示一式三份进行的单独渗入之间的标准偏差。讨论pjp3115和pjp3116设计为提供产生dha的所有基因的组合,即两个重组载体互补以组成途径。pjp3115编码的δ12-去饱和酶所产生的脂肪酸被用作pjp3116编码的δ15-去饱和酶的底物,pjp3116还含有随后的δ6-去饱和酶、δ6-延长酶和δ5-去饱和酶的基因。然后pjp3115编码的δ5-延长酶作用于δ5-去饱和酶的产物epa,其产物被也由pjp3115编码的δ4-去饱和酶转换为dha。在两个构建体之间划分转基因的原理,其用于单独稳定转化植物与随后的优良株的杂交以组成整个途径,避免了与在单个构建体中含有众多转基因相关的一些问题,例如因为增加的大小而减少的转化效率和减少的基因表达。这些构建体的稳定转化的组合,通过超级转化或通过两个转基因系的杂交,会导致含有dha合成所需的全套基因的转基因植物。还注意到构建体pjp3115含有4个以颠倒形式表达的基因,即2个基因以一个方向而2个基因以另一个方向,这样基因对以发散方式转录(远离对方)。当与构建体pjp107(实施例8)中用于表达3个基因的颠倒设计比较时,得出的结论是在该形式中第4个基因的加入不会阻碍基因的表达。有趣的是注意到与上述其他实验相比相对较低的δ6-延长作用效率(50.4%),其可能是因为编码用于前三个去饱和作用步骤的酶的基因全部从fp1启动子表达而编码δ6-延长酶的基因由拟南芥fae1启动子驱动这一事实。这被认为引起启动子时机的不同,fae1启动子在fp1启动子之后活化。与δ6-延长酶由fp1启动子驱动的前述实验相比,这导致sda的较高积累,然后在δ6-延长酶可以对sda作用之前从δ6-延长酶到达的代谢库去除sda。实施例18:编码微藻dgat2的基因的分离和表征全长细小微胞藻dgat2基因的合成利用来自ostreococcuslucimarinus的推定的氨基酸序列,genbank登录号xp_00141576,作为查询序列用blastp程序分析美国能源部联合基因组研究所(http://www.jgi.doe.gov/)产生的微胞藻ccmp1545过滤蛋白质模型基因组序列。该分析显示在微胞藻ccmp1545中与xp_00141576具有同源性的推定的蛋白的存在。将微胞藻ccmp1545推定的蛋白序列用于设计和合成最适合在诸如欧洲油菜的双子叶植物中表达的密码子优化的核苷酸序列。蛋白编码区的核苷酸序列在seqidno:107中给出。质粒构建体命名为0928814_mic1545-dgat2_pma。氨基酸序列如seqidno:108所示。利用微胞藻ccmp1545去饱和酶氨基酸序列seqidno:108在genbank数据库中查询其他蛋白的blastp分析显示蛋白与dgat具有同源性。最高程度的同一性为与登录号xp_002503155的氨基酸序列,微胞藻rcc299推定的蛋白的序列的全长具有53%的同一性。该基因在氨基酸74-334含有二酰基甘油酰基转移酶基序(ncbi保守结构域pfam03982)。通过将在ecori片段内含有的0928814_mic1545-dgat2_pma的整个编码区在ecori位点插入35s-pore04(以上实施例4)来产生在组成型35s启动子控制之下的编码dgat2的遗传构建体35s:mic1545-dgat2,产生pjp3128。将该嵌合载体引入根癌土壤杆菌菌株agl1并将来自这些培养物的转基因细胞与35s:p19agl1混合,然后在温室中将混合物渗入本氏烟草植物的叶组织。渗入之后使植物再生长4天,然后取叶盘用于脂质分析,显示编码dgat2的基因发挥功能以增加转化的叶细胞中的总tag,偏好多不饱和脂肪酸(表21)。具体地,在转化的细胞中tag中多不饱和脂肪酸的水平增加了至少3倍。本领域人员理解可以对具体实施方式中所给出的发明进行多种变化和/或改动而不脱离所宽泛描述的本发明的实质或范围。以上实施方式在各方面均是示例性的,而不是限制性的。本申请要求2008年11月18日提交的us61/199,669和2009年7月9日提交的us61/270,710的优先权,在此通过引用将其内容并入本申请。本文中所讨论和/或援引的所有文献均以其全部内容并入本申请。本说明书中对文献、法案、材料、装置、物品等等的任何讨论的目的仅在于提供本发明的背景。不应理解为承认它们的任何或全部如同在本申请各项权利要求的优先权日之前已经存在一样构成了现有技术基础或者是本发明所属
技术领域:
的公知常识。参考文献abbadietal.(2004)plantcell16:2734-2748.abbottetal.(1998)science282:2012-2018.abdullahetal.(1986)biotech.4:1087.agabaetal.(2004)marinebiotechnol.(ny)6:251-261.al-maririetal.(2002)infect.immun.70:1915-1923.alvarezetal.(2000)theorapplgenet100:319-327.batesetal.(2007)j.biol.chem.vol.282:31206-31216.baumbergeretal.(2007)curr.biol.17:1609-1614.baumleinetal.(1991)mol.gen.genet.225:459-467.baumleinetal.(1992)plantj.2:233-239.beaudoinetal.(2000)proc.natl.acad.sci.u.s.a.97:6421-6426.beclinetal.(2002)curr.biol.12:684-688.berberich.etal.(1998)plantmol.biol.36:297-306.blighanddyer(1959)canadianj.biochem.37:911-917.bortolamioletal.(2007)curr.biol.17:1615-1621.bouvier-naveetal.(2000)euro.j.biochm.267:85-96.brodersenetal.(2008)science320:1185-1190.broothaertsetal.(2005)nature433:629-633.brosnanetal.(2007)proc.natl.acad.sciu.s.a.104:14741-14746.brounetal.(1998)plantj.13:201-210.capecchi(1980)cell22:479-488.chapmanetal.(2004)gen.dev.18:1179-1186.chenetal.(2004)theplantcell16:1302-1313.chengetal.(1996)plantcellrep.15:653-657.chikwambaetal.(2003)proc.natl.acad.sci.u.s.a.100:11127-11132.choetal.(1999a)j.biol.chem.274:471-477.choetal.(1999b)j.biol.chem.274:37335-37339.chungetal.(2006)bmcgenomics7:120.clapp(1993)clin.perinatol.20:155-168.cloughandbent(1998)plantj.16:735-43.courvalinetal(1995)lifesci.318:1209-1212.coutuetal.(2007)transgenicres.16:771-781.curieletal.(1992)hum.gen.ther.3:147-154.darjietal.(1997)cell91:765-775.denicandweissman(2007)cell130:663-677.dietrichetal.(1998)naturebiotech.18:181-185.dietrichetal.(2001)vaccine19:2506.dingandvoinnet(2007)cell130:413-426.domergueetal.(2002)eur.j.biochem.269:4105-4113.domergueetal.(2003)j.biol.chem.278:35115-35126.domergueetal.(2005)biochem.j.l389:483-490.dunoyeretal.(2004)theplantcell16:1235-1250.eglitisetal.(1988)biotechniques6:608-614.ellerstrometal.(1996)plantmol.biol.32:1019-1027.fennellyetal.(1999)j.immunol.162:1603-1610.fraseretal.(2004)plantphysiol.135:859-866.fujietal.(2007)plantcell19:597-609.fujimuraetal.(1985)planttissueculturelett.2:74.garcia-marotoetal.(2002)lipids37:417-426.girkeetal.(1998)plantj.15:39-48.gleave(1992)plantmol.biol.20:1203-1207.glevinetal(2003)microbiol.mol.biol.rev.67:16-37.glicketal.(2008)proc.natl.acad.sciu.s.a.105-157-161.grahametal.(1973)virology54:536-539.grantetal.(1995)plantcellrep.15:254-258.guillardandrythers(1962)can.j.microbiol.8:229-239.grillot-courvalinetal.(1998)naturebiotech.16:862-866.grillot-courvalin(1999)curr.opin.biotech.10-477-481.haganetal.(2003)plant.biotech.j.1:479-490.hamiltonandbaulcombe(1999)science286:950-952.harayama(1998).trendsbiotechnol.16:76-82.hastingsetal.(2001)proc.natl.acad.sci.u.s.a.98:14304-14309.henseetal.(2001)cellmicrobiol.3:599-609.hincheeetal.(1988)biotechnology6:915-922.hoffmannetal.(2008)jbiol.chem.283:22352-22362.hongetal.(2002a)lipids37:863-868.horiguchietal.(1998)plantcellphysiol.39:540-544.horvathetal.(2000)proc.natl.acad.sci.u.s.a.97:1914-1919.huangetal.(1999)lipids34:649-659.huangetal.(2004)biochimie86(11):793-8.inagakietal.(2002)biosci.biotechnol.biochem.66:613-621.johansenandcarrington(2001)plantphysiol.126-930-938.kajikawaetal.(2004)plantmol.biol.54:335-52.kajikawaetal.(2006)febslett580:149-154.kapilaetal.(1997)plantsci.122:101-108.kasschauetal.(2003)devel.cell4:205-217.khozinetal.(1997)plantphysiol.114:223-230.knutzonetal.(1998)j.biolchem.273:29360-6.kozieletal.(1996)plantmol.biol.32:393-405.kuniketal.(2001)proc.natl.acad.sci.u.s.a.98:1871-1876.lacroixetal.proc.natl.acad.sci.u.s.a.105:15429-15434.lazoetal.(1991)biotechnol.9:693-697.lechtenbergetal.(2003)plantj.507-517.leonardetal.(2000)biochem.j.347:719-724.leonardetal.(2000b)biochem.j.350:765-770.leonardetal.(2002)lipids37:733-740.lewseyetal.(2007)plantj.50:240-252.lindboetal.(1993)plantcell5:1749-1759.loetal.(2003)genomeres.13:455-466.luetal.(1993)j.exp.med.178:2089-2096.malloryetal(2002)nat.biotech.20:622-625.marillonnetetal.(2005)naturebiotechnology23:718-723.matzkeetal.(2001)science293:1080-1083.mengetal.(2008)j.gen.virol.89:2349-2358.meyeretal.(2003)biochem.42:9779-9788.meyeretal.(2004)lipidres45:1899-1909.michaelsonetal.(1998a)j.biol.chem.273:19055-19059.michaelsonetal.(1998b)febslett.439:215-218.moreaueal.(1998)progresslip.res.37:371-391.napier(2007)ann.rev.plant.biol.58:295-319.napieretal.(1998)biochem.j.330:611-614.needlemanandwunsch(1970)j.mol.biol.48:443-453.niedzetal(1995)plantcellreports14:403.nishizawaetal.(2003)plantj.34:647-659.ohlroggeandbrowse(1995)plantcell7:957-970.owetal.(1986)science234:856-859.parker-barnesetal.(2000)proc.natl.acad.sci.usa97:8284-8289.pereiraetal.(2004a)biochem.j.378:665-671.pereiraetal.(2004b)biochem.j.384:357-366.perrinetal.(2000)molbreed6:345-352.potenzaetal.(2004)invitrocelldevbiol–plant40:1-22.prasheretal(1985)biochem.biophys.res.commun.127:31-36.qietal.(2002)febslett.510:159-165.qietal.(2004)nat.biotech.22:739-745.qiuetal.(2001)j.biol.chem.276:31561-31566.reddyandthomas(1996)nat.biotech.14:639-642.reddyetal.(1993)plantmol.biol.22:293-300.robertetal.(2005)func.plantbiol.32:473-479.robertetal.(2009)marinebiotech11:410-418.roseetal.(1998)nucleicacidsres.26:1628-1635.sahaetal.(2006)plantphysiol.141:1533-1543.saitoetal.(2000)eur.j.biochem.267:1813-1818.sakuradanietal.(1999)gene238:445-453.satoetal.(2004)cropsci.44:646-652.sayanovaetal.(1997)proc.natl.acad.sci.u.s.a.94:4211-4216.sayanovaetal.(2003)febslett.542:100-104.sayanovaetal.(2006)planta224:1269-1277.sayanovaetal.(2007)plantphysiol144:455-467.schaffneretal(1980)proc.natl.acad.sci.u.s.a.77:2163-2167.schubertetal.(2004)plantcell16:2561-2572.singhetal.(2005)curr.opin.inplantbiol.8:197-203.sizemoreetal(1995)science270:299-302.shianuetal(2001)vaccine19:3947-3956.sperlingetal.(2000)eur.j.biochem.267:3801-3811.sperlingetal.(2001)arch.biochm.biophys.388:293-8.sprecheretal.(1995)j.lipidres.36:2471-2477.spychallaetal.(1997)proc.natl.acad.sci.u.s.a.94:1142-1147.stalkeretal(1998)j.biol.chem.263:6310-6314.thilletetal(1988)j.biol.chem263:12500-12508.tononetal.(2003)febslett.553:440-444.toriyamaetal.(1986)theor.appl.genet.205:34.trautwein(2001)europeanj.lipidsci.andtech.103:45-55.tvrdik(2000)j.cellbiol.149:707-718.tzfira&citovsky(2006)curr.opin.biotech.17:147-154.voinnetetal.,(2003)plantj.33:949-956.wagneretal.(1992)proc.natl.acad.sci.usa89:6099-6103.wallisandbrowse(1999)arch.biochem.biophys.365:307-316.wattsandbrowse(1999b)arch.biochem.biophys.362:175-182.weissetal(2003)int.j.med.microbiol.293:95:106.whitneyetal.(2003)planta217:983-992.wuetal.(2005)nat.biotech.23:1013-1017.yangetal.(2003)planta216:597-603.yuetal.(2007)proc.natl.acad.sciu.s.a.104:8924-8929.zanketal.(2002)plantj.31:255-268.zanketal.(2005)wo2005/012316zhangetal.(2004)febslett.556:81-85.zhangetal.(2006)20:3255-3268.zhangetal.(2007)yeast25:21-27.zhouetal.(2006)plantsci.170:665-673.zhouetal.(2007)phytochem.68:785-796.zipfeletal.(2006)cell125:749-760.序列表<110>联邦科学技术研究组织<120>产生omega-3脂肪酸的酶和方法<130>508730<150>us61/199,669<151>2008-11-18<150>us61/270,710<151>2009-07-09<160>129<170>patentinversion3.5<210>1<211>825<212>dna<213>micromonascs-0170<400>1atggctttccccgccgtcgcgaccgctgtcttcggtatctatgcgtacgtgatgtactgg60tactccgtccccggcgcgggcttcctcgccgagggcaagcaggacgtcgtggcgtggatc120ggcgatctgtccgtggcgctccccgccaccgccaccgtcctgtacctctccatgtgctac180ttcggcccgaagatcatggccaagagggaggcgttcgagccgaaggggttcatgctggtg240tacaacgcgtaccagaccgtgttcaacatcatcaccgtcgccatcttcatcgctgagctc300aaccggctgggcgtgaaggcgtggggcggccgcctgtcgtggtccgatcccaacagcttc360tacatctgcctcgccatctggctccactacaacaacaagtacctggagctcctcgacacc420gtcttcatggtgctgcgcaagaagaacaaccagctcagcttcctccacatctaccaccac480tgcctcctcatctgggcgtggtggatggtgtgcttcgtcatcaagaacaacgactgcatc540gacgcgtactttggcgcctgcatgaacgctggaattcacgtcatcatgtactcctattac600ctcatggccgcgctgaagatcaagtgcccgtggaagcagtacatcaccatggcgcagatg660ctccagttcgccatcgtctttgcgcacagctgctacgtcatctacgacgggcactgcccg720gcgattctgccgtggtcgcagatgttcgtgatgaccaacatgctggtgctgttcgggcag780ttctacgtgcagacgtacacgaagaagaaggtaaaggccaagtaa825<210>2<211>274<212>prt<213>micromonascs-0170<400>2metalapheproalavalalathralavalpheglyiletyralatyr151015valmettyrtrptyrservalproglyalaglypheleualaglugly202530lysglnaspvalvalalatrpileglyaspleuservalalaleupro354045alathralathrvalleutyrleusermetcystyrpheglyprolys505560ilemetalalysargglualaphegluprolysglyphemetleuval65707580tyrasnalatyrglnthrvalpheasnileilethrvalalailephe859095ilealagluleuasnargleuglyvallysalatrpglyglyargleu100105110sertrpseraspproasnserphetyrilecysleualailetrpleu115120125histyrasnasnlystyrleugluleuleuaspthrvalphemetval130135140leuarglyslysasnasnglnleuserpheleuhisiletyrhishis145150155160cysleuleuiletrpalatrptrpmetvalcysphevalilelysasn165170175asnaspcysileaspalatyrpheglyalacysmetasnalaglyile180185190hisvalilemettyrsertyrtyrleumetalaalaleulysilelys195200205cysprotrplysglntyrilethrmetalaglnmetleuglnpheala210215220ilevalphealahissercystyrvaliletyraspglyhiscyspro225230235240alaileleuprotrpserglnmetphevalmetthrasnmetleuval245250255leupheglyglnphetyrvalglnthrtyrthrlyslyslysvallys260265270alalys<210>3<211>894<212>dna<213>pyramimonascs-0140<400>3atggagttcgctcagcctcttgtggctatggcacaggagcagtatgccgcaattgacgcg60gtggtagcccctgcaattttctcagctaccgacagcatcggttggggtcttaagcccatt120agcagcgcgacaaaggatcttcctctcgttgagagtccgacgccgctcatactgagcctg180ttggcctattttgcgatcgtcggctctgggctggtgtaccgcaaagtattccctcgcaca240gtaaaggggcaagaccccttcctgctgaaggcgctcatgcttgcgcacaacgtgttcctc300attggcctcagtctatacatgtgcttgaagcttgtctacgaggcttacgtcaacaagtac360tccttctggggaaacgcctacaaccccgcacagaccgagatggcgaaggtcatctggatt420ttctacgtctccaagatctatgagttcatggacacgttcatcatgctcttgaagggcaac480gtcaaccaggtctctttcctgcatgtgtaccatcatggctccatctctggtatctggtgg540atgatcacctacgctgcccctggcggtgacgcgtacttctcggcggcgctcaactcgtgg600gtgcacgtgtgcatgtacacgtactacttcatggcggcggtgctgcccaaggacgagaag660accaagcgcaagtacctctggtggggccgctacctgacccagatgcagatgttccagttc720ttcatgaacctgctccaggcggtctacctcctctactcctctagcccctaccccaagttc780atcgcccagctgctggtggtgtacatggtcacgctgctgatgctcttcggcaacttctac840tacatgaagcaccacgcgagcaagaagcagaagctggccagcaagaagcagtag894<210>4<211>297<212>prt<213>pyramimonascs-0140<400>4metgluphealaglnproleuvalalametalaglngluglntyrala151015alaileaspalavalvalalaproalailepheseralathraspser202530ileglytrpglyleulysproileserseralathrlysaspleupro354045leuvalgluserprothrproleuileleuserleuleualatyrphe505560alailevalglyserglyleuvaltyrarglysvalpheproargthr65707580vallysglyglnasppropheleuleulysalaleumetleualahis859095asnvalpheleuileglyleuserleutyrmetcysleulysleuval100105110tyrglualatyrvalasnlystyrserphetrpglyasnalatyrasn115120125proalaglnthrglumetalalysvaliletrpilephetyrvalser130135140lysiletyrgluphemetaspthrpheilemetleuleulysglyasn145150155160valasnglnvalserpheleuhisvaltyrhishisglyserileser165170175glyiletrptrpmetilethrtyralaalaproglyglyaspalatyr180185190pheseralaalaleuasnsertrpvalhisvalcysmettyrthrtyr195200205tyrphemetalaalavalleuprolysaspglulysthrlysarglys210215220tyrleutrptrpglyargtyrleuthrglnmetglnmetpheglnphe225230235240phemetasnleuleuglnalavaltyrleuleutyrserserserpro245250255tyrprolyspheilealaglnleuleuvalvaltyrmetvalthrleu260265270leumetleupheglyasnphetyrtyrmetlyshishisalaserlys275280285lysglnlysleualaserlyslysgln290295<210>5<211>804<212>dna<213>pyramimonascs-0140<400>5atggcgtctattgcgattccggctgcgctggcagggactcttggttatgtgacgtacaat60gtcgcaaacccagatattcctgcatccgagaaggtgcctgcttactttatgcaggtcgag120tattgggggccaacgattgggaccatcggttatcttctgttcatctactttggtaaacgg180attatgcaaaacaggagccagccgtttggcctgaagaacgctatgctggtgtacaacttc240tatcagactttcttcaactcgtactgcatatacctttttgtcacgtcgcaccgcgctcag300gggctgaaagtttggggaaacatccccgatatgactgccaacagctgggggatctcacag360gtgatctggctgcactacaacaacaagtacgttgagctgctggacacgttcttcatggtc420atgcgcaagaagtttgaccagctttcgttcctgcacatttaccatcataccctgttgatc480tggtcttggttcgtggtgatgaaattggagcccgttggggactgctactttggctctagc540gtcaacacgtttgtgcacgtcattatgtactcgtactatggccttgccgcgctcggggtg600aattgcttctggaagaagtacattacgcagattcagatgctgcagttctgtatctgcgct660tcgcactcgatttataccgcctatgtgcagaacaccgcgttctggttgccttacttgcag720ctgtgggtgatggtgaacatgttcgtgttgttcgccaacttctatcgcaagcgctacaag780agcaagggtgccaagaagcagtaa804<210>6<211>267<212>prt<213>pyramimonascs-0140<400>6metalaserilealaileproalaalaleualaglythrleuglytyr151015valthrtyrasnvalalaasnproaspileproalaserglulysval202530proalatyrphemetglnvalglutyrtrpglyprothrileglythr354045ileglytyrleuleupheiletyrpheglylysargilemetglnasn505560argserglnpropheglyleulysasnalametleuvaltyrasnphe65707580tyrglnthrphepheasnsertyrcysiletyrleuphevalthrser859095hisargalaglnglyleulysvaltrpglyasnileproaspmetthr100105110alaasnsertrpglyileserglnvaliletrpleuhistyrasnasn115120125lystyrvalgluleuleuaspthrphephemetvalmetarglyslys130135140pheaspglnleuserpheleuhisiletyrhishisthrleuleuile145150155160trpsertrpphevalvalmetlysleugluprovalglyaspcystyr165170175pheglyserservalasnthrphevalhisvalilemettyrsertyr180185190tyrglyleualaalaleuglyvalasncysphetrplyslystyrile195200205thrglnileglnmetleuglnphecysilecysalaserhisserile210215220tyrthralatyrvalglnasnthralaphetrpleuprotyrleugln225230235240leutrpvalmetvalasnmetphevalleuphealaasnphetyrarg245250255lysargtyrlysserlysglyalalyslysgln260265<210>7<211>1392<212>dna<213>micromonasccmp1545<400>7atgtgtcctcctaagaccgatggaagatcttctcctagatctcctctcaccaggtctaag60tcatctgctgaggctcttgatgctaaggatgcttctaccgctcctgttgatcttaagacc120cttgagcctcatgaacttgctgctaccttcgagactagatgggttagggttgaggatgtt180gagtacgacgtgaccaacttcaaacatcctggtggaagcgtgatcttctacatgcttgct240aacactggtgctgatgctactgaggctttcaaagaatttcacatgcgtagcctcaaggct300tggaagatgcttagagctttgccttctagacctgctgagatcaagagatctgagtctgag360gatgctcctatgcttgaggatttcgctaggtggagagctgaacttgagagggacggattc420ttcaagccttctatcacccatgttgcttaccgtcttttggagcttcttgctactttcgct480cttggaaccgctcttatgtacgctggataccctatcattgctagcgttgtgtacggtgct540ttcttcggagctagatgtggatgggttcaacatgagggtggacacaactctcttaccgga600tctgtgtacgtggataagagacttcaggctatgacttgcggattcggactttctaccagc660ggagagatgtggaaccagatgcataacaagcaccatgctacccctcagaaagttagacac720gacatggatcttgataccactcctgctgtggctttcttcaacaccgctgtggaggataat780agacctaggggattctctagagcttgggctagacttcaagcttggaccttcgttcctgtt840acttctggacttctcgttcaggctttctggatctacgttctccatcctagacaggtgctc900aggaagaagaactacgaggaagcttcttggatgctcgtttctcacgttgttagaaccgct960gttatcaagcttgctaccggatactcttggcctgttgcttactggtggttcactttcgga1020aactggatcgcttacatgtacctcttcgctcacttctctacttctcacactcacctccct1080gttgttccatctgacaagcaccttagctgggttaactacgctgttgatcacaccgttgac1140atcgatccttctcgtggatacgttaactggcttatgggataccttaactgccaggttatc1200caccatctcttccctgatatgcctcaattcagacagcctgaggtgtcaagaagattcgtc1260cctttcgctaagaagtggggactcaactacaaggtgctctcttactacggtgcttggaag1320gctactttcagcaacctcgacaaagttggacagcactactacgttaacggaaaggctgag1380aaggctcactga1392<210>8<211>463<212>prt<213>micromonasccmp1545<400>8metcysproprolysthraspglyargserserproargserproleu151015thrargserlysserseralaglualaleuaspalalysaspalaser202530thralaprovalaspleulysthrleugluprohisgluleualaala354045thrphegluthrargtrpvalargvalgluaspvalglutyraspval505560thrasnphelyshisproglyglyservalilephetyrmetleuala65707580asnthrglyalaaspalathrglualaphelysgluphehismetarg859095serleulysalatrplysmetleuargalaleuproserargproala100105110gluilelysargserglusergluaspalaprometleugluaspphe115120125alaargtrpargalagluleugluargaspglyphephelysproser130135140ilethrhisvalalatyrargleuleugluleuleualathrpheala145150155160leuglythralaleumettyralaglytyrproileilealaserval165170175valtyrglyalaphepheglyalaargcysglytrpvalglnhisglu180185190glyglyhisasnserleuthrglyservaltyrvalasplysargleu195200205glnalametthrcysglypheglyleuserthrserglyglumettrp210215220asnglnmethisasnlyshishisalathrproglnlysvalarghis225230235240aspmetaspleuaspthrthrproalavalalaphepheasnthrala245250255valgluaspasnargproargglypheserargalatrpalaargleu260265270glnalatrpthrphevalprovalthrserglyleuleuvalglnala275280285phetrpiletyrvalleuhisproargglnvalleuarglyslysasn290295300tyrgluglualasertrpmetleuvalserhisvalvalargthrala305310315320valilelysleualathrglytyrsertrpprovalalatyrtrptrp325330335phethrpheglyasntrpilealatyrmettyrleuphealahisphe340345350serthrserhisthrhisleuprovalvalproserasplyshisleu355360365sertrpvalasntyralavalasphisthrvalaspileaspproser370375380argglytyrvalasntrpleumetglytyrleuasncysglnvalile385390395400hishisleupheproaspmetproglnpheargglnprogluvalser405410415argargphevalprophealalyslystrpglyleuasntyrlysval420425430leusertyrtyrglyalatrplysalathrpheserasnleuasplys435440445valglyglnhistyrtyrvalasnglylysalaglulysalahis450455460<210>9<211>1449<212>dna<213>ostreococcuslucimarinus<400>9atgtgcgtcgaaacgaccgaaggcacatcgcgaacgatggcgaacgaacgcacgagctcg60tcgtcgtcgctgagcgaaggcggaacgccgacggtgacggtcgggatgggaagcgaagac120gcggggaagaagactcgaaacgcgagcgtcacggcgtggacgaaagagttggagccgcac180gcgatcgcgaagacgttcgaacggcggtacgtgacgatcgaaggcgtggaatacgatgtg240acggattttaagcatcccggaggatcggttatttattacatgctgtcgaacacgggagcg300gacgcgacggaggcttttaaagagtttcattatcggtcgaaaaaggcgcgcaaggcgttg360gcggcgttgccgcataagccagtggacgcggcgacgcgggaaccgatcgaagatgaggcg420atgctgaaggatttcgcgcagtggcgcaaggaattggagcgtgagggattttttaagccc480tcgccggcgcacgtggcgtatcgattcgccgagctcgcggcgatgttcgcgctcggcacg540gcgttgatgcacgcgcgttggcacgtcgcttccgtgatcgtgtactcgtgtttcttcggc600gcgcgatgcggttgggtgcagcacgagggtgggcacaattcgttgactggaaacatttgg660tgggacaagcgaatccaagccttcgccgcggggttcggcttggcgtcgagtggcgacatg720tggaacaacatgcacaacaagcatcacgcgacgccccaaaaggtgcgacacgatatggat780ctcgacaccactcccacggtggcgttcttcaactccgcggttgaagaaaatcgcccgcgg840ggattcagtaagttgtggttgcgccttcaagcgtggaccttcgtgcccgtgacgtccggt900atggttttgttcttctggatgttcgtcttgcacccgcgtaacgcgctgcgacgcaaaagc960ttcgaagaagcggcttggatgttttccgcgcacgtcattcgcacggcggttatcaaagcc1020gtcaccggctactcctggatcgcctcgtacggcttgttcgcggcgacgatgtgggcgagc1080ggatgttacttgttcgcgcacttttccacgtctcacacgcacttggatgtcgtgccgagc1140gataaacacctctcgtgggtgcgatacgccgtcgatcacacgatcgacatcaatccgaac1200aacagcgtcgtcaactggttgatgggctacttgaactgccaagtcatccatcacctgttc1260ccggatatgcctcagttccgccaacccgaagtctcccgccgattcgtcccgtttgcgaag1320aagtggaacttaaactacaaggtcttgacgtattatggggcctggaaggcgacgttcggc1380aacttgaacgacgtcgggaagcactattacgtgcacggatctcagcgcgtcaaatcaaag1440tcggcgtga1449<210>10<211>482<212>prt<213>ostreococcuslucimarinus<400>10metcysvalgluthrthrgluglythrserargthrmetalaasnglu151015argthrserserserserserleusergluglyglythrprothrval202530thrvalglymetglysergluaspalaglylyslysthrargasnala354045servalthralatrpthrlysgluleugluprohisalailealalys505560thrphegluargargtyrvalthrilegluglyvalglutyraspval65707580thraspphelyshisproglyglyservaliletyrtyrmetleuser859095asnthrglyalaaspalathrglualaphelysgluphehistyrarg100105110serlyslysalaarglysalaleualaalaleuprohislysproval115120125aspalaalathrarggluproilegluaspglualametleulysasp130135140phealaglntrparglysgluleugluarggluglyphephelyspro145150155160serproalahisvalalatyrargphealagluleualaalametphe165170175alaleuglythralaleumethisalaargtrphisvalalaserval180185190ilevaltyrsercysphepheglyalaargcysglytrpvalglnhis195200205gluglyglyhisasnserleuthrglyasniletrptrpasplysarg210215220ileglnalaphealaalaglypheglyleualaserserglyaspmet225230235240trpasnasnmethisasnlyshishisalathrproglnlysvalarg245250255hisaspmetaspleuaspthrthrprothrvalalaphepheasnser260265270alavalglugluasnargproargglypheserlysleutrpleuarg275280285leuglnalatrpthrphevalprovalthrserglymetvalleuphe290295300phetrpmetphevalleuhisproargasnalaleuargarglysser305310315320phegluglualaalatrpmetpheseralahisvalileargthrala325330335valilelysalavalthrglytyrsertrpilealasertyrglyleu340345350phealaalathrmettrpalaserglycystyrleuphealahisphe355360365serthrserhisthrhisleuaspvalvalproserasplyshisleu370375380sertrpvalargtyralavalasphisthrileaspileasnproasn385390395400asnservalvalasntrpleumetglytyrleuasncysglnvalile405410415hishisleupheproaspmetproglnpheargglnprogluvalser420425430argargphevalprophealalyslystrpasnleuasntyrlysval435440445leuthrtyrtyrglyalatrplysalathrpheglyasnleuasnasp450455460valglylyshistyrtyrvalhisglyserglnargvallysserlys465470475480serala<210>11<211>1449<212>dna<213>artificialsequence<220><223>codon-optimizednucleotidesequenceforproductioninplantsofseqidno:10<400>11atgtgtgttgagactactgagggaacctctagaactatggctaacgagaggacctcttct60tcttcttcactctctgagggtggaactcctactgttactgtgggaatgggatctgaggat120gctggaaagaaaaccagaaacgcttctgttactgcttggaccaaagagcttgagcctcac180gctatcgctaagaccttcgagagaagatacgttaccatcgagggtgttgagtacgatgtg240accgatttcaaacaccctggtggatctgtgatctactacatgctctctaacactggtgct300gatgctactgaggctttcaaagagttccactaccgttctaagaaggctagaaaggctctt360gctgctcttcctcacaagcctgttgatgctgctactagagagcctattgaggacgaggct420atgcttaaggatttcgctcagtggagaaaagagttggagagagagggattcttcaagcct480tctcctgctcatgttgcttaccgtttcgctgaactcgctgctatgttcgctcttggaacc540gctcttatgcatgctagatggcacgttgctagcgttatcgtgtactcctgtttcttcgga600gctagatgtggatgggttcaacatgagggtggacacaactctcttaccggaaacatctgg660tgggataagagaatccaagctttcgctgctggattcggacttgcttcttctggtgacatg720tggaacaacatgcacaacaagcaccatgctactcctcagaaagtgagacacgatatggat780cttgataccacccctaccgttgctttcttcaactctgctgtggaggaaaacagacctagg840ggattctctaagctttggctcagacttcaagcttggaccttcgttcctgttacctctgga900atggtgctcttcttctggatgttcgttctccatcctagaaacgctctccgtcgtaagtct960ttcgaagaggctgcttggatgttctctgctcacgttatcagaaccgctgttatcaaggct1020gttaccggatactcttggatcgctagctacggacttttcgctgctactatgtgggcttct1080ggatgctaccttttcgctcacttctctacttctcacacccacctcgatgttgttccatct1140gataagcaccttagctgggttaggtacgctgttgatcacaccatcgacatcaaccctaac1200aactctgttgtgaactggcttatgggataccttaactgccaggttatccaccatctcttc1260cctgatatgcctcaattcagacagcctgaggtgtcaagaagattcgtccctttcgctaag1320aagtggaacctcaactacaaggtgctcacttactacggtgcttggaaggctactttcgga1380aacctcaacgatgttggaaagcactactacgttcacggatctcagagagtgaagagcaag1440agcgcttga1449<210>12<211>1329<212>dna<213>pyramimonascs-0140<400>12atgggaaagggaggcaatgctagcgctcctactgcgaagaaggaggtgttgatcgagggg60aagttttacgatgtcaccgacttcaggcaccccggtggttcgatcatcaagtttctctcg120ggttctggtgctgacgccaccgcttcctaccgcgagttccacgttaggtcagcgaaggca180gacaagttcttgaagacgctgccctcccgcgaagccactccccaggagctgaagcaggcg240gttgagttctccaagctcaacccgccctccgcggagagtgcctctgctcccctgaccgac300cttgccaaggtggaagcgctgaacaaggacttcgaggctttccgtgagcagctcattcag360gagggcttctttaagcccaatatcccgcatgtggtcaagcgcatcacggaagtcgtggcg420atgatggccgtagcctcctggatgatggtgcagaccaacgctcttgttgtgaccctcgga480gttctgatccgcggcattgcacagggccggtgcggttggcttatgcacgagggcggccac540tatagtcttactgggaagatctccattgataggcgtctgcaggagtcaatttacggattc600ggctgtggaatgtccggcgcctggtggcgcaaccagcacaacaagcaccacgcaacccca660cagaagctgcagcatgacgtcgacctggagacccttcctctgatggctttcaacaacgct720gttaccgatagacgcaaggtgaagcctggtagtctccaggctctgtggctcaagtaccag780gccttcctcttcttccccgtgacctcccttctggtcggcctcggttggaccaccgtcctc840caccccaggcacagcttgcgcaccaagcactatttcgagctgctctgcatggctgctcgt900tacgcgagtttcgctgctcttttcgctcccaagtacggacttgcaggagctgccgggctc960tacctcgccaccttcgctgtcgggtgcaactatattttcatcaacttctcggtctctcac1020actcacctgcccgtgagcggtgcgagcgagtacctgcattgggtcgtgtattcggccatc1080cacaccactaacatcaaatccagcatgctgtgcgattggtggatgtcattcctcaacttc1140cagatcgagcatcacctgttcccttcaatgccccagttccgccacaagattatctccccg1200cgtgtaaaggccttgtttgagaagcacggtcttgtgtatgatgtgcgcccctattggggg1260gccatggctgacaccttcaagaacttgaatgacgttggcactcacgcatctcactccaag1320gcgcactag1329<210>13<211>442<212>prt<213>pyramimonascs-0140<400>13metglylysglyglyasnalaseralaprothralalyslysgluval151015leuilegluglylysphetyraspvalthraspphearghisprogly202530glyserileilelyspheleuserglyserglyalaaspalathrala354045sertyrarggluphehisvalargseralalysalaasplyspheleu505560lysthrleuproserargglualathrproglngluleulysglnala65707580valglupheserlysleuasnproproseralagluseralaserala859095proleuthraspleualalysvalglualaleuasnlysasppheglu100105110alaphearggluglnleuileglngluglyphephelysproasnile115120125prohisvalvallysargilethrgluvalvalalametmetalaval130135140alasertrpmetmetvalglnthrasnalaleuvalvalthrleugly145150155160valleuileargglyilealaglnglyargcysglytrpleumethis165170175gluglyglyhistyrserleuthrglylysileserileaspargarg180185190leuglngluseriletyrglypheglycysglymetserglyalatrp195200205trpargasnglnhisasnlyshishisalathrproglnlysleugln210215220hisaspvalaspleugluthrleuproleumetalapheasnasnala225230235240valthraspargarglysvallysproglyserleuglnalaleutrp245250255leulystyrglnalapheleuphepheprovalthrserleuleuval260265270glyleuglytrpthrthrvalleuhisproarghisserleuargthr275280285lyshistyrphegluleuleucysmetalaalaargtyralaserphe290295300alaalaleuphealaprolystyrglyleualaglyalaalaglyleu305310315320tyrleualathrphealavalglycysasntyrilepheileasnphe325330335servalserhisthrhisleuprovalserglyalaserglutyrleu340345350histrpvalvaltyrseralailehisthrthrasnilelysserser355360365metleucysasptrptrpmetserpheleuasnpheglnilegluhis370375380hisleupheprosermetproglnphearghislysileileserpro385390395400argvallysalaleupheglulyshisglyleuvaltyraspvalarg405410415protyrtrpglyalametalaaspthrphelysasnleuasnaspval420425430glythrhisalaserhisserlysalahis435440<210>14<211>528<212>dna<213>micromonascs-0170<400>14tgtggtgcatcggccatgattgggggcacggatccttctccaaaaacgccctcgtcaacg60gcgtcgtcggacacctcacgcactcgccgctgctcgtgcccttttacccgtgggcgtact120cgcacaagcagcaccacaggtttcacaaccacgaggtacgggacatgtcgcacccgtgga180tgtccaaggaggagtacgcggacgtgaaccccgtcgtgagggcgctggcgctggacggtt240ggtggggaaccttcctcggctttcccgggtacctcctcctggaaccgcagtgggccggga300cggacgggtgccacttcaacccgaactccaggctcttcgacagggcgcccaaggatgagc360gggtgaagtgcgcggtttccaccgtggcgtgcgcggcgtttctcggcgcgtccttcgtcg420cgtgcgactcgaaccccgttcactggttcgctcagtacttggcgccgtacttgtgcttct480cgtggtggctcttcaccgtgacatacttacagcacaacgacgaggaca528<210>15<211>175<212>prt<213>micromonascs-0170<400>15trpcysileglyhisasptrpglyhisglyserpheserlysasnala151015leuvalasnglyvalvalglyhisleuthrhisserproleuleuval202530prophetyrprotrpalatyrserhislysglnhishisargphehis354045asnhisgluvalargaspmetserhisprotrpmetserlysgluglu505560tyralaaspvalasnprovalvalargalaleualaleuaspglytrp65707580trpglythrpheleuglypheproglytyrleuleuleugluprogln859095trpalaglythraspglycyshispheasnproasnserargleuphe100105110aspargalaprolysaspgluargvallyscysalavalserthrval115120125alacysalaalapheleuglyalaserphevalalacysaspserasn130135140provalhistrpphealaglntyrleualaprotyrleucyspheser145150155160trptrpleuphethrvalthrtyrleuglnhisasnaspgluasp165170175<210>16<211>1461<212>dna<213>micromonasrcc299<400>16atgaccgccgtcggcggcgggcgtcgacactcccgcgccgcgcgtcccgagcggactcga60gaccgtcggaagcgctcgtcggattccacagagtcggattccgtcgtctcccggacaccc120ggcgacgtacctcccgccgcgctcgttcacgcgatgcccgcgatgatgacctcatccacc180tgtctgcgggccacgtcgccccggatggaatcgacacgccgcggctccctccgcgtcgtc240gacgccgcggcgcccgcacgcaccggcgcatccaagacgggtcggtccgtcccgcgtcaa300ctcccccgcgccatttccgcgccgacgacgaacctcggaaccgcggacgcgccaactccc360gccgccggtcccgtgccgtccctccgagagctgcgcgcatccatcccgaaggagtgcttc420gagcccgacctgggggagtcgctcaagtacgccgcgtacgatctcgccgcgctcgccgcg480tgcttcggcgtcatctccccgcacgtggtggaccacccgtggctgttaccgctgtacgcg540cccctcacgggcaccgtcatgtggatgaacttcgtcgtcggccacgactgcgggcacggc600tccttctccaaatcctccgtcgtcaacggcgtcgtcggccacctcacgcactcgccgctg660ctcgtgcccttctacccgtgggcgtactcgcacaagcagcaccacaggtttcacaaccac720caggtaaaggacatgtcgcacccgtggatgactgcagaggagtacgccgaggtgaacccc780atcgtccgggctctggcgcttgacgggtggtggggaaccttcctcggctttcccgggtac840ctcctcctggaaccgcggtgggccgggacggacgggtgccacttcaacccacaatccagg900ctcttcgacagggcgcccaaggacgagcgggtgaagtgcgcggtttccaccgtggcgtgc960gcggcgtttctcggcgcgtccttcgtcgcgtgcgactcgaacccggcgcactggttcgct1020cagtacttggcgccgtacttgtgcttctcgtggtggctcttcaccgtgacctacctccag1080caccacgactacgacacaacgacgtacgaagagggccagtgggagtacgtgctggggggt1140ttggagacgatcgaccgcgagtttgggcacggcgtggacgagctgacgcatcacatcacc1200gactgccacgtcgcgcaccacatgttctcggacatgccccactaccgcctgcccgcggcg1260accgccggggtgcgatcggtgctggagccccgtgggttgtacaagcgacgggacacgcgg1320gatttcgtgacgaaggtgttcgagttgcacggggacgtcgggcactgcgtggagagcgag1380gcgggggtgaggccgagggcgacgcgcgacgagtgcgcggcggcgttcaacggcgaggag1440tggcgcgaggttcgcaagtag1461<210>17<211>486<212>prt<213>micromonasrcc299<400>17metthralavalglyglyglyargarghisserargalaalaargpro151015gluargthrargaspargarglysargserseraspserthrgluser202530aspservalvalserargthrproglyaspvalproproalaalaleu354045valhisalametproalametmetthrserserthrcysleuargala505560thrserproargmetgluserthrargargglyserleuargvalval65707580aspalaalaalaproalaargthrglyalaserlysthrglyargser859095valproargglnleuproargalaileseralaprothrthrasnleu100105110glythralaaspalaprothrproalaalaglyprovalproserleu115120125arggluleuargalaserileprolysglucysphegluproaspleu130135140glygluserleulystyralaalatyraspleualaalaleualaala145150155160cyspheglyvalileserprohisvalvalasphisprotrpleuleu165170175proleutyralaproleuthrglythrvalmettrpmetasnpheval180185190valglyhisaspcysglyhisglyserpheserlysserservalval195200205asnglyvalvalglyhisleuthrhisserproleuleuvalprophe210215220tyrprotrpalatyrserhislysglnhishisargphehisasnhis225230235240glnvallysaspmetserhisprotrpmetthralagluglutyrala245250255gluvalasnproilevalargalaleualaleuaspglytrptrpgly260265270thrpheleuglypheproglytyrleuleuleugluproargtrpala275280285glythraspglycyshispheasnproglnserargleupheasparg290295300alaprolysaspgluargvallyscysalavalserthrvalalacys305310315320alaalapheleuglyalaserphevalalacysaspserasnproala325330335histrpphealaglntyrleualaprotyrleucysphesertrptrp340345350leuphethrvalthrtyrleuglnhishisasptyraspthrthrthr355360365tyrglugluglyglntrpglutyrvalleuglyglyleugluthrile370375380aspargglupheglyhisglyvalaspgluleuthrhishisilethr385390395400aspcyshisvalalahishismetpheseraspmetprohistyrarg405410415leuproalaalathralaglyvalargservalleugluproarggly420425430leutyrlysargargaspthrargaspphevalthrlysvalpheglu435440445leuhisglyaspvalglyhiscysvalgluserglualaglyvalarg450455460proargalathrargaspglucysalaalaalapheasnglygluglu465470475480trparggluvalarglys485<210>18<211>1461<212>dna<213>artificialsequence<220><223>codon-optimizednucleotidesequenceforproductioninplantsofseqidno:17<400>18atgactgctgttggaggtggaagaaggcattctagagctgctagacctgagagaaccagg60gatcgtagaaagagatcttctgactctaccgagtctgattctgtggtttctaggacccct120ggtgatgttcctcctgctgctcttgttcatgctatgcctgctatgatgacctcttctacc180tgccttagagctacttctcctaggatggaatctaccagaaggggatctctcagagttgtt240gacgctgctgctccagctagaactggtgcttctaagaccggaagatctgttcctagacag300ctccctagagctatctctgctcctactactaaccttggaaccgctgatgctcctactcct360gctgctggacctgttccttctcttagagagcttagggctagcatccctaaagagtgcttc420gagcctgatcttggagagtctctcaagtacgctgcttacgatcttgctgctctcgctgct480tgtttcggagttatctctcctcacgttgttgatcatccttggcttctcccactttacgct540cctcttaccggaactgtgatgtggatgaacttcgttgttggacatgattgcggacacgga600tctttctctaagtctagcgtggttaacggtgttgttggacaccttacccactctcctctt660ctcgttcctttctacccttgggcttactctcataagcagcaccacagattccataaccac720caggtgaaggatatgtctcacccttggatgactgctgaggaatacgctgaggttaaccct780attgtgagggctcttgctcttgatggatggtggggaactttccttggattccctggatac840cttcttcttgagcctagatgggctggaactgatggatgtcacttcaaccctcagtctaga900cttttcgacagggctcctaaggatgaacgtgttaagtgcgctgtttctactgttgcttgc960gctgctttccttggagcttctttcgtggcttgcgattctaaccctgctcattggttcgct1020cagtaccttgctccttacctttgcttctcttggtggctcttcactgttacttacctccag1080caccacgattacgataccactacctacgaggaaggacagtgggaatacgttcttggagga1140ctcgagactatcgatagagaatttggacacggtgttgatgagcttactcaccacatcact1200gattgtcacgtggctcaccacatgttctctgacatgcctcattacagacttccagctgct1260actgctggtgttagatctgttctcgagcctagaggactctacaagagaagagacaccagg1320gatttcgttaccaaggtgttcgagcttcatggtgatgttggacactgcgttgagtctgaa1380gctggtgttaggcctagagctactagagatgagtgcgctgctgcttttaatggtgaagag1440tggagggaagttaggaagtga1461<210>19<211>1389<212>dna<213>micromonasccmp1545<400>19atgacggcggcgacgacgctctcgcgcgcgtcctccgtcgtcggcgtcggcgcaggcgcc60acgtcgaggcgtccggtcgatcgcgccgtctccgccgtcgcgacgtcctcctcgtattcg120cgccgcgcgcgccgcgtcgcgaccgtccgcgcggcgtccagccgcacgtcgtcctccgcg180acgacgacgacgacgacgacgacgcgctccgctctggccatggagaccccgtccacgacg240acgacggacgcggacgacgacgacgacaagaccggcgtcgtcccgtcgctccgagagctc300cgagccgcgatccccgcggagtgcttcgtcccgtgcctgagggagtcgatgaagtacgcc360gcgatcgacctcggtctcctcgccgcctgcttcggtctgtggtctccgctcgtggtggat420aacccgtggatgctcccgctgtacgcccccgtcaccgggacgatcatgtggatgtgcttc480gtcgtcgggcacgactgcgggcacggcagtttcaggtgcgtcagttctacaaacgtcgag540aacaagtggatcaacggcgtcgtcggtcacgcgacgcactcgccgctgctcgtgccgttc600tacccgtgggcgtactcgcacaagcagcaccaccggtttcataaccacgaggagaaggac660atgtcgcacccgtggatgagcgcggagcggtacaaggacacgaacgcgatcgttcggtgc720gcggcgggcggctcggtttctggtttttggttcctcgcgctcgatcacccatggggcgcg780tttctcggcttccccgggtatctcctcctcgagccaaagtggagcagcacggacggctcg840cacttcaacccggccggtcgtctgttcgaccgcgcccccaaggacgaacgtctcaagtgc900gcggtctcgacggtcgcgtgcgcggcgttcctcctcgcgacgttcgccgcgtgcgacggc960cccgcgcagtgggcgacgcagtacctcgcgccgtatctgtgcttctcgtggtggctcttc1020acggtgacctacctccagcaccacgaccacgacacgaagacgtacaaggaaggcgagtgg1080gagtacgtcttggggggattagagacgatcgatcgcgagttcgggtacggcgtggacgag1140gcgacgcatcacatcacggactgccacgtcgcgcaccacatgttttcggacatgccgcac1200tacaacttggagaaggccaccgcgggggtgcggggcgtgctcgagcccaggggactgtat1260aagaagagggacacgagggatttcgcgacgaagatttttgggttgcacaaagacgtcggg1320cactgcgtcgagccggaccggccgcgggcgacgaaggaggagctcgtgaaggcgttgggg1380cgggagtga1389<210>20<211>462<212>prt<213>micromonasccmp1545<400>20metthralaalathrthrleuserargalaserservalvalglyval151015glyalaglyalathrserargargprovalaspargalavalserala202530valalathrsersersertyrserargargalaargargvalalathr354045valargalaalaserserargthrserserseralathrthrthrthr505560thrthrthrthrargseralaleualametgluthrproserthrthr65707580thrthraspalaaspaspaspaspasplysthrglyvalvalproser859095leuarggluleuargalaalaileproalaglucysphevalprocys100105110leuargglusermetlystyralaalaileaspleuglyleuleuala115120125alacyspheglyleutrpserproleuvalvalaspasnprotrpmet130135140leuproleutyralaprovalthrglythrilemettrpmetcysphe145150155160valvalglyhisaspcysglyhisglyserpheargcysvalserser165170175thrasnvalgluasnlystrpileasnglyvalvalglyhisalathr180185190hisserproleuleuvalprophetyrprotrpalatyrserhislys195200205glnhishisargphehisasnhisgluglulysaspmetserhispro210215220trpmetseralagluargtyrlysaspthrasnalailevalargcys225230235240alaalaglyglyservalserglyphetrppheleualaleuasphis245250255protrpglyalapheleuglypheproglytyrleuleuleuglupro260265270lystrpserserthraspglyserhispheasnproalaglyargleu275280285pheaspargalaprolysaspgluargleulyscysalavalserthr290295300valalacysalaalapheleuleualathrphealaalacysaspgly305310315320proalaglntrpalathrglntyrleualaprotyrleucyspheser325330335trptrpleuphethrvalthrtyrleuglnhishisasphisaspthr340345350lysthrtyrlysgluglyglutrpglutyrvalleuglyglyleuglu355360365thrileaspargglupheglytyrglyvalaspglualathrhishis370375380ilethraspcyshisvalalahishismetpheseraspmetprohis385390395400tyrasnleuglulysalathralaglyvalargglyvalleuglupro405410415argglyleutyrlyslysargaspthrargaspphealathrlysile420425430pheglyleuhislysaspvalglyhiscysvalgluproaspargpro435440445argalathrlysglugluleuvallysalaleuglyargglu450455460<210>21<211>792<212>dna<213>isochrysisgalbana<400>21atggccctcgcaaacgacgcgggagagcgcatctgggcggctgtgaccgacccggaaatc60ctcattggcaccttctcgtacttgctactcaaaccgctgctccgcaattccgggctggtg120gatgagaagaagggcgcatacaggacgtccatgatctggtacaacgttctgctggcgctc180ttctctgcgctgagcttctacgtgacggcgaccgccctcggctgggactatggtacgggc240gcgtggctgcgcaggcaaaccggcgacacaccgcagccgctcttccagtgcccgtccccg300gtttgggactcgaagctcttcacatggaccgccaaggcattctattactccaagtacgtg360gagtacctcgacacggcctggctggtgctcaagggcaagagggtctcctttctccaggcc420ttccaccactttggcgcgccgtgggatgtgtacctcggcattcggctgcacaacgagggc480gtatggatcttcatgtttttcaactcgttcattcacaccatcatgtacacctactacggc540ctcaccgccgccgggtataagttcaaggccaagccgctcatcaccgcgatgcagatctgc600cagttcgtgggcggcttcctgttggtctgggactacatcaacgtcccctgcttcaactcg660gacaaagggaagttgttcagctgggctttcaactatgcatacgtcggctcggtcttcttg720ctcttctgccactttttctaccaggacaacttggcaacgaagaaatcggccaaggcgggc780aagcagctctag792<210>22<211>263<212>prt<213>isochrysisgalbana<400>22metalaleualaasnaspalaglygluargiletrpalaalavalthr151015aspprogluileleuileglythrphesertyrleuleuleulyspro202530leuleuargasnserglyleuvalaspglulyslysglyalatyrarg354045thrsermetiletrptyrasnvalleuleualaleupheseralaleu505560serphetyrvalthralathralaleuglytrpasptyrglythrgly65707580alatrpleuargargglnthrglyaspthrproglnproleuphegln859095cysproserprovaltrpaspserlysleuphethrtrpthralalys100105110alaphetyrtyrserlystyrvalglutyrleuaspthralatrpleu115120125valleulysglylysargvalserpheleuglnalaphehishisphe130135140glyalaprotrpaspvaltyrleuglyileargleuhisasnglugly145150155160valtrpilephemetphepheasnserpheilehisthrilemettyr165170175thrtyrtyrglyleuthralaalaglytyrlysphelysalalyspro180185190leuilethralametglnilecysglnphevalglyglypheleuleu195200205valtrpasptyrileasnvalprocyspheasnserasplysglylys210215220leuphesertrpalapheasntyralatyrvalglyservalpheleu225230235240leuphecyshisphephetyrglnaspasnleualathrlyslysser245250255alalysalaglylysglnleu260<210>23<211>1284<212>dna<213>pavlovasalina<400>23atgggacgcggcggagacagcagtgggcaggcgcatccggcggcggagctggcggtcccg60agcgaccgcgcggaggtgagcaacgctgacagcaaagcgctgcacatcgtgctgtatggc120aagcgcgtggatgtgaccaagttccaacgcacgcacccgggtggtagcaaggtcttccgg180atcttccaggaccgcgatgcgacggagcagttcgagtcctaccactcgaagcgcgcgatc240aagatgatggagggcatgctcaagaagtctgaggatgctcccgccgacacgcccttgccc300tcccagtcaccgatggggaaggacttcaaggcgatgatcgagcggcacgttgcagcgggt360tactacgatccatgcccgctcgatgagctgttcaagctcagcctcgtgctcctcccgacc420tttgcgggcatgtacatgctcaaggcgggcgtcggctccccgctctgcggcgccctcatg480gtgagctttggctggtacctcgatggctggctcgcgcacgactatctgcaccactccgtc540ttcaaggggtccgtcgcacgcaccgtcgggtggaacaacgcggcgggctacttcctcggc600ttcgtgcaggggtatgcggtcgagtggtggcgcgcgcggcataacacgcaccacgtgtgc660accaatgaggacggctcggaccccgacatcaaaacggcgccgctgctcatatacgtgcgc720aacaagccgagcatcgccaagcgcctgaacgccttccagcgctaccagcagtactactat780gtgccggtgatggcaatcctcgacctgtactggcggctcgagtcgatcgcctacgtcgcg840atgcgcctgccgaagatgctgccgcaggccctcgcactcgtcgcgcactacgccatcgtc900gcgtgggtctttgcgggcaactaccacctgctcccgctcgtgacggttctgcgcgggttt960ggcactgggatcaccgttttcgcgacgcactacggtgaggacattctcgacgcggaccag1020gtgcgtcacatgacgctcgtcgagcagacggcactcacctcgcgcaacatctcgggcggc1080tggctcgtgaacgtgctcaccggcttcatctcactgcagacggagcaccacctgttcccg1140atgatgccaaccggcaacctcatgactatccagcccgaggtgcgcgccttcttcaagaag1200cacggacttgagtaccgcgagggcaacctcattgagtgcgtgcggcagaacatccgtgcg1260cttgcattcgagcacctgctttga1284<210>24<211>427<212>prt<213>pavlovasalina<400>24metglyargglyglyaspserserglyglnalahisproalaalaglu151015leualavalproseraspargalagluvalserasnalaaspserlys202530alaleuhisilevalleutyrglylysargvalaspvalthrlysphe354045glnargthrhisproglyglyserlysvalpheargilepheglnasp505560argaspalathrgluglnpheglusertyrhisserlysargalaile65707580lysmetmetgluglymetleulyslyssergluaspalaproalaasp859095thrproleuproserglnserprometglylysaspphelysalamet100105110ilegluarghisvalalaalaglytyrtyraspprocysproleuasp115120125gluleuphelysleuserleuvalleuleuprothrphealaglymet130135140tyrmetleulysalaglyvalglyserproleucysglyalaleumet145150155160valserpheglytrptyrleuaspglytrpleualahisasptyrleu165170175hishisservalphelysglyservalalaargthrvalglytrpasn180185190asnalaalaglytyrpheleuglyphevalglnglytyralavalglu195200205trptrpargalaarghisasnthrhishisvalcysthrasngluasp210215220glyseraspproaspilelysthralaproleuleuiletyrvalarg225230235240asnlysproserilealalysargleuasnalapheglnargtyrgln245250255glntyrtyrtyrvalprovalmetalaileleuaspleutyrtrparg260265270leugluserilealatyrvalalametargleuprolysmetleupro275280285glnalaleualaleuvalalahistyralailevalalatrpvalphe290295300alaglyasntyrhisleuleuproleuvalthrvalleuargglyphe305310315320glythrglyilethrvalphealathrhistyrglygluaspileleu325330335aspalaaspglnvalarghismetthrleuvalgluglnthralaleu340345350thrserargasnileserglyglytrpleuvalasnvalleuthrgly355360365pheileserleuglnthrgluhishisleupheprometmetprothr370375380glyasnleumetthrileglnprogluvalargalaphephelyslys385390395400hisglyleuglutyrarggluglyasnleuileglucysvalarggln405410415asnileargalaleualaphegluhisleuleu420425<210>25<211>1278<212>dna<213>pavlovasalina<400>25atgccgccgcgcgatagctactcgtacgccgccccgccgtcggcccagctgcacgaggtc60gataccccgcaggagcatgataagaaggagctcgtcatcggtgaccgcgcgtacgacgtg120accaactttgtgaagcgccacccgggtggcaagatcatcgcataccaggttggcacagat180gcgacggacgcgtacaagcagttccatgtgcggtctgccaaggcggacaagatgctcaag240tcgctgccttcgcgcccggtgcacaagggctactcgccccgccgcgctgacctcattgcc300gacttccaggagttcaccaagcagctggaggcggagggcatgtttgagccgtcgctgccg360cacgtggcataccgcctggcggaggtgatcgcgatgcacgtggccggcgccgcgctcatc420tggcacgggtacaccttcgcgggcattgccatgctcggcgttgtgcagggccgctgcggc480tggctcatgcacgagggcggccactactcgctcacgggcaacattgcttttgaccgtgcc540atccaagtcgcgtgctacggccttggctgcggcatgtcgggcgcgtggtggcgcaaccag600cacaacaagcaccacgcgacgccgcagaagttgcagcacgacgtcgacctcgacaccctc660ccgctcgtcgccttccacgagcggatagccgccaaggtgaagagccccgcgatgaaggcg720tggcttagtatgcaggcgaagctcttcgcgccagtgaccacgctgctggtcgcgctgggc780tggcagctgtacctgcacccgcgccatatgctgcgcaccaagcactacgacgagctcgcg840atgctcggcattcgctacggccttgtcggctacctcgcggcgaactacggcgcggggtac900gtgctcgcgtgctacctgctgtacgtgcagctcggcgccatgtacatcttctgcaacttt960gccgtgtcgcacacacacctgccggttgtcgagcctaacgagcacgcaacgtgggtggag1020tacgccgcgaaccacacgaccaactgctcgccctcgtggtggtgcgactggtggatgtcg1080tacctcaactaccagatcgagcaccacctctacccgtccatgccgcagttccgccacccg1140aagattgcgccgcgggtgaagcagctcttcgagaagcacggcctgcactacgacgtgcgt1200ggctacttcgaggccatggcggacacgtttgccaaccttgacaacgtcgcgcacgcgccg1260gagaagaagatgcagtga1278<210>26<211>425<212>prt<213>pavlovasalina<400>26metproproargaspsertyrsertyralaalaproproseralagln151015leuhisgluvalaspthrproglngluhisasplyslysgluleuval202530ileglyaspargalatyraspvalthrasnphevallysarghispro354045glyglylysileilealatyrglnvalglythraspalathraspala505560tyrlysglnphehisvalargseralalysalaasplysmetleulys65707580serleuproserargprovalhislysglytyrserproargargala859095aspleuilealaasppheglngluphethrlysglnleuglualaglu100105110glymetphegluproserleuprohisvalalatyrargleualaglu115120125valilealamethisvalalaglyalaalaleuiletrphisglytyr130135140thrphealaglyilealametleuglyvalvalglnglyargcysgly145150155160trpleumethisgluglyglyhistyrserleuthrglyasnileala165170175pheaspargalaileglnvalalacystyrglyleuglycysglymet180185190serglyalatrptrpargasnglnhisasnlyshishisalathrpro195200205glnlysleuglnhisaspvalaspleuaspthrleuproleuvalala210215220phehisgluargilealaalalysvallysserproalametlysala225230235240trpleusermetglnalalysleuphealaprovalthrthrleuleu245250255valalaleuglytrpglnleutyrleuhisproarghismetleuarg260265270thrlyshistyraspgluleualametleuglyileargtyrglyleu275280285valglytyrleualaalaasntyrglyalaglytyrvalleualacys290295300tyrleuleutyrvalglnleuglyalamettyrilephecysasnphe305310315320alavalserhisthrhisleuprovalvalgluproasngluhisala325330335thrtrpvalglutyralaalaasnhisthrthrasncysserproser340345350trptrpcysasptrptrpmetsertyrleuasntyrglnilegluhis355360365hisleutyrprosermetproglnphearghisprolysilealapro370375380argvallysglnleupheglulyshisglyleuhistyraspvalarg385390395400glytyrpheglualametalaaspthrphealaasnleuaspasnval405410415alahisalaproglulyslysmetgln420425<210>27<211>801<212>dna<213>emilianiahuxleyiccmp1516<400>27atgctcgatcgcgcctcgtccgacgcggccatctggtctgcggtgtccgatccggaaatc60ctgatcggcactttctcctacctgctgctcaagccgctgctacgcaactcagggctcgtg120gacgagcggaaaggcgcctaccggacctcgatgatctggtacaacgtggtgctcgcgctc180ttctccgcgacgagcttctacgtgactgcgaccgcgctcgggtgggacaagggcaccggc240gagtggctccgcagtctcacgggcgacagcccgcagcagctgtggcaatgcccgtcgagg300gtatgggactccaagctgttcctgtggacggccaaggccttctactactcaaagtacgtg360gagtacctcgacacggcgtggctcgtcctcaaggggaagaaggtctccttcctgcagggc420ttccaccactttggcgcgccgtgggacgtgtacctgggcattcggctgaagaacgagggc480gtgtggatcttcatgttcttcaactcgttcatccacacggtcatgtacacgtactacggc540ctcaccgccgcgggctacaagatccgcggcaagccgatcatcaccgcgatgcaaataagc600cagttcgtcggcggctttgtcctagtgtgggactacatcaacgtgccgtgcttccacgcc660gacgccgggcaggtcttcagctgggtctttaactatgcttacgtcggctccgtctttctg720ctgttctgccacttcttctacatggacaacatcgcgaaggccaaggccaagaaggccgtc780gctacccgcaaggcgctgtga801<210>28<211>266<212>prt<213>emilianiahuxleyiccmp1516<400>28metleuaspargalaserseraspalaalailetrpseralavalser151015aspprogluileleuileglythrphesertyrleuleuleulyspro202530leuleuargasnserglyleuvalaspgluarglysglyalatyrarg354045thrsermetiletrptyrasnvalvalleualaleupheseralathr505560serphetyrvalthralathralaleuglytrpasplysglythrgly65707580glutrpleuargserleuthrglyaspserproglnglnleutrpgln859095cysproserargvaltrpaspserlysleupheleutrpthralalys100105110alaphetyrtyrserlystyrvalglutyrleuaspthralatrpleu115120125valleulysglylyslysvalserpheleuglnglyphehishisphe130135140glyalaprotrpaspvaltyrleuglyileargleulysasnglugly145150155160valtrpilephemetphepheasnserpheilehisthrvalmettyr165170175thrtyrtyrglyleuthralaalaglytyrlysileargglylyspro180185190ileilethralametglnileserglnphevalglyglyphevalleu195200205valtrpasptyrileasnvalprocysphehisalaaspalaglygln210215220valphesertrpvalpheasntyralatyrvalglyservalpheleu225230235240leuphecyshisphephetyrmetaspasnilealalysalalysala245250255lyslysalavalalathrarglysalaleu260265<210>29<211>801<212>dna<213>artificialsequence<220><223>codon-optimizednucleotidesequenceforproductioninplantsofseqidno:28<400>29atgcttgatagagcttcatctgatgctgctatttggagcgctgtttctgatcctgagatc60cttatcggaaccttctcttaccttttgcttaagcctctcctcagaaactctggacttgtg120gatgagagaaagggagcttaccgtacttctatgatctggtacaacgttgttcttgctctt180ttctctgctacctctttctacgttactgctactgctcttggatgggataagggaactggt240gagtggcttagatctcttactggtgattctcctcaacaactttggcagtgcccttctaga300gtttgggacagcaaactcttcttgtggactgctaaagccttctactactccaagtacgtt360gagtaccttgatactgcttggcttgttctcaagggaaagaaggtttcattcctccaggga420ttccatcatttcggtgctccatgggatgtttaccttggaatcaggcttaagaacgaggga480gtttggatcttcatgttcttcaacagcttcatccacactgttatgtacacttactacgga540cttactgctgctggatacaagatcagaggaaagcctatcatcaccgctatgcaaatctct600caattcgttggtggattcgttcttgtgtgggactacatcaacgttccttgtttccatgct660gatgctggacaagttttctcttgggtgttcaactacgcttatgtgggatctgttttcctt720cttttctgccacttcttctacatggacaacattgctaaggctaaggctaaaaaggctgtt780gctaccagaaaggctctttga801<210>30<211>456<212>prt<213>ostreococcustauri<400>30metcysvalgluthrgluasnasnaspglyileprothrvalgluile151015alapheaspglygluarggluargalaglualaasnvallysleuser202530alaglulysmetgluproalaalaleualalysthrphealaargarg354045tyrvalvalilegluglyvalglutyraspvalthraspphelyshis505560proglyglythrvalilephetyralaleuserasnthrglyalaasp65707580alathrglualaphelysgluphehishisargserarglysalaarg859095lysalaleualaalaleuproserargproalalysthralalysval100105110aspaspalaglumetleuglnaspphealalystrparglysgluleu115120125gluargaspglyphephelysproserproalahisvalalatyrarg130135140phealagluleualaalamettyralaleuglythrtyrleumettyr145150155160alaargtyrvalvalserservalleuvaltyralacysphephegly165170175alaargcysglytrpvalglnhisgluglyglyhisserserleuthr180185190glyasniletrptrpasplysargileglnalaphethralaglyphe195200205glyleualaglyserglyaspmettrpasnsermethisasnlyshis210215220hisalathrproglnlysvalarghisaspmetaspleuaspthrthr225230235240proalavalalaphepheasnthralavalgluaspasnargproarg245250255glypheserlystyrtrpleuargleuglnalatrpthrpheilepro260265270valthrserglyleuvalleuleuphetrpmetphepheleuhispro275280285serlysalaleulysglyglylystyrglugluleuvaltrpmetleu290295300alaalahisvalileargthrtrpthrilelysalavalthrglyphe305310315320thralametglnsertyrglyleupheleualathrsertrpvalser325330335glycystyrleuphealahispheserthrserhisthrhisleuasp340345350valvalproalaaspgluhisleusertrpvalargtyralavalasp355360365histhrileaspileaspproserglnglytrpvalasntrpleumet370375380glytyrleuasncysglnvalilehishisleupheprosermetpro385390395400glnpheargglnprogluvalserargargphevalalaphealalys405410415lystrpasnleuasntyrlysvalmetthrtyralaglyalatrplys420425430alathrleuglyasnleuaspasnvalglylyshistyrtyrvalhis435440445glyglnhisserglylysthrala450455<210>31<211>8<212>prt<213>artificialsequence<220><223>elongaseconsensusdomain1<220><221>misc_feature<222>(2)..(6)<223>x=anyaminoacid<400>31lysxaaxaaxaaxaaxaaaspthr15<210>32<211>5<212>prt<213>artificialsequence<220><223>elongaseconsensusdomain2<220><221>misc_feature<222>(3)..(3)<223>x=anyaminoacid<400>32mettyrxaatyrtyr15<210>33<211>8<212>prt<213>artificialsequence<220><223>elongaseconsensusdomain3<400>33lysiletyrgluphevalaspthr15<210>34<211>7<212>prt<213>artificialsequence<220><223>elongaseconsensusdomain4<400>34valhisvalcysmettyrthr15<210>35<211>7<212>prt<213>artificialsequence<220><223>elongaseconsensusdomain5<400>35tyrleugluleuleuaspthr15<210>36<211>5<212>prt<213>artificialsequence<220><223>elongaseconsensusdomain6<400>36mettyrsertyrtyr15<210>37<211>10<212>prt<213>artificialsequence<220><223>desaturaseconsensusdomain1<400>37trplysasnmethisasnlyshishisala1510<210>38<211>8<212>prt<213>artificialsequence<220><223>desaturaseconsensusdomain2<400>38hishisleupheprosermetpro15<210>39<211>8<212>prt<213>artificialsequence<220><223>desaturaseconsensusdomain3<400>39trpcysileglyhisaspcysgly15<210>40<211>10<212>prt<213>artificialsequence<220><223>desaturaseconsensusdomain4<400>40thrpheleuglnhishisaspgluaspmet1510<210>41<211>37<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>41atttaggtgacactatagttttttttttttttttttv37<210>42<211>23<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<220><221>misc_feature<222>(7)..(7)<223>n=inosine<220><221>misc_feature<222>(14)..(14)<223>n=inosine<400>42aagwwcnksgarynsytcgacac23<210>43<211>20<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<220><221>misc_feature<223>n=inosine<220><221>misc_feature<222>(2)..(3)<223>n=inosine<220><221>misc_feature<222>(5)..(5)<223>n=inosine<400>43annmnrtartasgtgtacat20<210>44<211>21<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>44gaacaacgactgcatcgacgc21<210>45<211>23<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>45cgactggagcacgaggacactga23<210>46<211>23<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>46ttgcgcagcaccataaagacggt23<210>47<211>26<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>47ggacactgacatggactgaaggagta26<210>48<211>22<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>48caggcgacgcgcgccagagtcc22<210>49<211>26<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>49ttattagttacttggcctttaccttc26<210>50<211>23<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<220><221>misc_feature<222>(18)..(18)<223>n=inosine<400>50aaratmtaygagttygtngatac23<210>51<211>25<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<220><221>misc_feature<222>(3)..(3)<223>n=inosine<400>51tangtgtacatgcacacrtgwaccc25<210>52<211>22<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>52ttcgtggatacgttcatcatgc22<210>53<211>23<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>53agttgagcgccgccgagaagtac23<210>54<211>22<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>54acctggttgacgttgcccttca22<210>55<211>21<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>55gctatggagttcgctcagcct21<210>56<211>26<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>56ttactactgcttcttgctggccagct26<210>57<211>22<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>57artayytsgarytrytggayac22<210>58<211>21<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>58catkarrtartasgagtacat21<210>59<211>22<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>59catcatccctgttgatctggtc22<210>60<211>23<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>60ccagatcaacagggtatgatggt23<210>61<211>27<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>61cgaaagctggtcaaacttcttgcgcat27<210>62<211>27<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>62aacatggcgtctattgcgattccggct27<210>63<211>27<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>63ttattactgcttcttggcacccttgct27<210>64<211>31<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<220><221>misc_feature<222>(23)..(23)<223>n=inosine<400>64ggtggaagaacaagcacaacrdncaycaygc31<210>65<211>24<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<220><221>misc_feature<222>(16)..(16)<223>n=inosine<400>65gggcatcgtggggwanarrtgrtg24<210>66<211>20<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>66agcgagtacctgcattgggt20<210>67<211>27<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>67atagtgcttggtgcgcaagctgtgcct27<210>68<211>25<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>68caccatgggaaagggaggcaatgct25<210>69<211>25<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>69ttactagtgcgccttggagtgagat25<210>70<211>25<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<220><221>misc_feature<222>(20)..(20)<223>n=inosine<220><221>misc_feature<222>(23)..(23)<223>n=inosine<400>70tgtggtgcatcggccayganksngg25<210>71<211>28<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<220><221>misc_feature<222>(20)..(20)<223>n=inosine<220><221>misc_feature<222>(26)..(26)<223>n=inosine<400>71tgtcctcgtcgttgtgctgnarrwangt28<210>72<211>1344<212>dna<213>pavlovasalina<400>72atgcctccgagcgcggcgaagcagatgggcgcgagcacgggcgtgcatgcgggcgtcaca60gattcgtcggccttcacgcgcaaggatgtcgccgacaggccggacctcacgatcgtgggt120gacagcgtgtacgatgcgaaggcgttccgctccgagcatccgggtggcgcgcactttgtg180tcgctgttcggcgggcgcgatgccacggaggcgttcatggagtaccaccggcgcgcctgg240cccaagtcgcgcatgtcgcgcttccacgtcggctctctggcatcgaccgaggagcccgtc300gccgccgatgagggctacctccagctgtgcgctcgcatcgccaagatggtgccgtcggtc360agcagcgggttcgcgccggcgtcgtactgggtgaaggccgggctgatcctcggctccgcg420atcgcgctcgaggcgtacatgctgtacgcgggcaagcgcctgctcccgtcgatcgtgctc480gggtggctgtttgcgctgattggcctgaacatccagcacgatgccaaccacggcgcgctc540tccaagtcggcctcggtcaacctggcgctcgggttgtgccaggactggatcggcgggagc600atgatcctctggctgcaggagcacgttgtcatgcaccacttgcacaccaacgacgttgac660aaggacccggaccagaaggcgcacggcgccctgcggctcaagccgaccgacgcgtggagc720ccgatgcactggctgcagcacctctacctgctgcctggggagacgatgtacgccttcaag780ctgctgtttctcgacatcagcgagctggtgatgtggcggtgggagggcgagcccatcagc840aagctggccgggtacctcttcatgccctcgctgctcctcaagctcaccttctgggcgcgc900tttgtcgcgctgccgctgtacctcgcgcccagcgtgcacacggcggtgtgcatcgcggcg960acggtaatgacggggagcttctacctcgccttcttcttcttcatctcgcacaacttcgag1020ggcgtggcgagcgtcggaccggacggcagcatcaccagcatgacgcgcggcgcatccttc1080ctcaagcggcaggccgagacctcgtccaacgtgggcggcccgctgctcgccacgctcaac1140ggcggcctcaactaccaaatcgagcaccacctcttccccagggtgcaccacggcttctac1200cctcgcctcgcgccgttggtcaaggcggagctcgaggcgcgcggcattgagtacaagcac1260taccccaccatatggagcaacctggcatccacgctgaggcacatgtacgcgctcggccgc1320aggccgcgcagcaaggcggagtga1344<210>73<211>447<212>prt<213>pavlovasalina<400>73metproproseralaalalysglnmetglyalaserthrglyvalhis151015alaglyvalthraspserseralaphethrarglysaspvalalaasp202530argproaspleuthrilevalglyaspservaltyraspalalysala354045pheargsergluhisproglyglyalahisphevalserleuphegly505560glyargaspalathrglualaphemetglutyrhisargargalatrp65707580prolysserargmetserargphehisvalglyserleualaserthr859095glugluprovalalaalaaspgluglytyrleuglnleucysalaarg100105110ilealalysmetvalproservalserserglyphealaproalaser115120125tyrtrpvallysalaglyleuileleuglyseralailealaleuglu130135140alatyrmetleutyralaglylysargleuleuproserilevalleu145150155160glytrpleuphealaleuileglyleuasnileglnhisaspalaasn165170175hisglyalaleuserlysseralaservalasnleualaleuglyleu180185190cysglnasptrpileglyglysermetileleutrpleuglngluhis195200205valvalmethishisleuhisthrasnaspvalasplysaspproasp210215220glnlysalahisglyalaleuargleulysprothraspalatrpser225230235240promethistrpleuglnhisleutyrleuleuproglygluthrmet245250255tyralaphelysleuleupheleuaspilesergluleuvalmettrp260265270argtrpgluglygluproileserlysleualaglytyrleuphemet275280285proserleuleuleulysleuthrphetrpalaargphevalalaleu290295300proleutyrleualaproservalhisthralavalcysilealaala305310315320thrvalmetthrglyserphetyrleualaphephephepheileser325330335hisasnphegluglyvalalaservalglyproaspglyserilethr340345350sermetthrargglyalaserpheleulysargglnalagluthrser355360365serasnvalglyglyproleuleualathrleuasnglyglyleuasn370375380tyrglnilegluhishisleupheproargvalhishisglyphetyr385390395400proargleualaproleuvallysalagluleuglualaargglyile405410415glutyrlyshistyrprothriletrpserasnleualaserthrleu420425430arghismettyralaleuglyargargproargserlysalaglu435440445<210>74<211>1563<212>dna<213>arabidopsisthaliana<400>74atggcgattttggattctgctggcgttactacggtgacggagaacggtggcggagagttc60gtcgatcttgataggcttcgtcgacggaaatcgagatcggattcttctaacggacttctt120ctctctggttccgataataattctccttcggatgatgttggagctcccgccgacgttagg180gatcggattgattccgttgttaacgatgacgctcagggaacagccaatttggccggagat240aataacggtggtggcgataataacggtggtggaagaggcggcggagaaggaagaggaaac300gccgatgctacgtttacgtatcgaccgtcggttccagctcatcggagggcgagagagagt360ccacttagctccgacgcaatcttcaaacagagccatgccggattattcaacctctgtgta420gtagttcttattgctgtaaacagtagactcatcatcgaaaatcttatgaagtatggttgg480ttgatcagaacggatttctggtttagttcaagatcgctgcgagattggccgcttttcatg540tgttgtatatccctttcgatctttcctttggctgcctttacggttgagaaattggtactt600cagaaatacatatcagaacctgttgtcatctttcttcatattattatcaccatgacagag660gttttgtatccagtttacgtcaccctaaggtgtgattctgcttttttatcaggtgtcact720ttgatgctcctcacttgcattgtgtggctaaagttggtttcttatgctcatactagctat780gacataagatccctagccaatgcagctgataaggccaatcctgaagtctcctactacgtt840agcttgaagagcttggcatatttcatggtcgctcccacattgtgttatcagccaagttat900ccacgttctgcatgtatacggaagggttgggtggctcgtcaatttgcaaaactggtcata960ttcaccggattcatgggatttataatagaacaatatataaatcctattgtcaggaactca1020aagcatcctttgaaaggcgatcttctatatgctattgaaagagtgttgaagctttcagtt1080ccaaatttatatgtgtggctctgcatgttctactgcttcttccacctttggttaaacata1140ttggcagagcttctctgcttcggggatcgtgaattctacaaagattggtggaatgcaaaa1200agtgtgggagattactggagaatgtggaatatgcctgttcataaatggatggttcgacat1260atatacttcccgtgcttgcgcagcaagataccaaagacactcgccattatcattgctttc1320ctagtctctgcagtctttcatgagctatgcatcgcagttccttgtcgtctcttcaagcta1380tgggcttttcttgggattatgtttcaggtgcctttggtcttcatcacaaactatctacag1440gaaaggtttggctcaacggtggggaacatgatcttctggttcatcttctgcattttcgga1500caaccgatgtgtgtgcttctttattaccacgacctgatgaaccgaaaaggatcgatgtca1560tga1563<210>75<211>520<212>prt<213>arabidopsisthaliana<400>75metalaileleuaspseralaglyvalthrthrvalthrgluasngly151015glyglygluphevalaspleuaspargleuargargarglysserarg202530seraspserserasnglyleuleuleuserglyseraspasnasnser354045proseraspaspvalglyalaproalaaspvalargaspargileasp505560servalvalasnaspaspalaglnglythralaasnleualaglyasp65707580asnasnglyglyglyaspasnasnglyglyglyargglyglyglyglu859095glyargglyasnalaaspalathrphethrtyrargproservalpro100105110alahisargargalaarggluserproleuserseraspalailephe115120125lysglnserhisalaglyleupheasnleucysvalvalvalleuile130135140alavalasnserargleuileilegluasnleumetlystyrglytrp145150155160leuileargthraspphetrppheserserargserleuargasptrp165170175proleuphemetcyscysileserleuserilepheproleualaala180185190phethrvalglulysleuvalleuglnlystyrilesergluproval195200205valilepheleuhisileileilethrmetthrgluvalleutyrpro210215220valtyrvalthrleuargcysaspseralapheleuserglyvalthr225230235240leumetleuleuthrcysilevaltrpleulysleuvalsertyrala245250255histhrsertyraspileargserleualaasnalaalaasplysala260265270asnprogluvalsertyrtyrvalserleulysserleualatyrphe275280285metvalalaprothrleucystyrglnprosertyrproargserala290295300cysilearglysglytrpvalalaargglnphealalysleuvalile305310315320phethrglyphemetglypheileilegluglntyrileasnproile325330335valargasnserlyshisproleulysglyaspleuleutyralaile340345350gluargvalleulysleuservalproasnleutyrvaltrpleucys355360365metphetyrcysphephehisleutrpleuasnileleualagluleu370375380leucyspheglyasparggluphetyrlysasptrptrpasnalalys385390395400servalglyasptyrtrpargmettrpasnmetprovalhislystrp405410415metvalarghisiletyrpheprocysleuargserlysileprolys420425430thrleualaileileilealapheleuvalseralavalphehisglu435440445leucysilealavalprocysargleuphelysleutrpalapheleu450455460glyilemetpheglnvalproleuvalpheilethrasntyrleugln465470475480gluargpheglyserthrvalglyasnmetilephetrppheilephe485490495cysilepheglyglnprometcysvalleuleutyrtyrhisaspleu500505510metasnarglysglysermetser515520<210>76<211>9<212>prt<213>artificialsequence<220><223>elongaseconsenusdomain7<400>76valaspthrarglysglyalatyrarg15<210>77<211>9<212>prt<213>artificialsequence<220><223>elongaseconsenusdomain8<400>77pheilehisthrilemettyrthrtyr15<210>78<211>28<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<220><221>misc_feature<222>(20)..(20)<223>nisa,c,g,ort<220><221>misc_feature<222>(23)..(23)<223>nisa,c,g,ort<400>78tggtggacacaaggaagggngcntaymg28<210>79<211>27<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>79gtaggtgtacatgatggtrtgdatraa27<210>80<211>24<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>80gtccttgctccagggcttccacca24<210>81<211>36<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>81atttaggtgacactatagtttttttttttttttttt36<210>82<211>23<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>82ttccagaacgagggcatctacgt23<210>83<211>25<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>83ttgggtgatctgcatgagcgtgatg25<210>84<211>26<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>84cgaatacttgaagagcttgttggaga26<210>85<211>25<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>85gggctacgagctggcagatgaagca25<210>86<211>25<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>86gaaaaaatggttgcgccacccatca25<210>87<211>27<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>87tcactacttcttcttcttgcccgcggc27<210>88<211>21<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>88ttccggtactcagcggtggcg21<210>89<211>17<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>89caggaaacagctatgac17<210>90<211>20<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>90acgtagatgccctcgttctg20<210>91<211>26<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>91caccgaatggcgactgaagggatgcc26<210>92<211>27<212>dna<213>artificialsequence<220><223>oligonucleotideprimer<400>92ctactcggttttcatgcggttgctgga27<210>93<211>819<212>dna<213>pavlovapinguis<400>93atggttgcgccacccatcacgctcgagtggctgctttcgccgaagctcaaggatgcagtg60ttcggtggggaggtgctctacttctccattgcctacctgtttcttgcgcccattttgaag120cgcaccccgttggtggacacgcggaagggcgcgtataagagtggtatgatcgcgtacaac180gtgatcatgtgcgtgttctcgctggtgtgcttcatctgccagctcgcagccctgggctat240gacatgggctacttgcagtgggtgcgtgacctcacaggggacgagattgtccccctctac300caggacgtgtccccgtcccccgccttctccaacaagctcttcaagtattcgtctattgcc360ttccactactccaagtatgttgagtacatggacaccgcatggctggtgatgaagggcaag420cccgtgtccttgctccagggcttccaccactttggcgccgcctgggacacctactttggc480atcaccttccagaacgagggcatctacgtgttcgtggtgctcaacgccttcatccacacg540atcatgtacgcatactacgcggccactgcggcgggtctcaagttctcactgaagttcgtc600atcacgctcatgcagatcacccaattcaacgtgggcttcgtaatggtgtatcactacatc660accctggagtacttccgcaactcaccggagctcgtcttctcctaccttttcaactatgcg720tacgtctgcacggttctcctcctcttcatgcagttcttctacatggacaactttggcaag780aagaaggccgctgccgccgcgggcaagaagaagaagtag819<210>94<211>272<212>prt<213>pavlovapinguis<400>94metvalalaproproilethrleuglutrpleuleuserprolysleu151015lysaspalavalpheglyglygluvalleutyrpheserilealatyr202530leupheleualaproileleulysargthrproleuvalaspthrarg354045lysglyalatyrlysserglymetilealatyrasnvalilemetcys505560valpheserleuvalcyspheilecysglnleualaalaleuglytyr65707580aspmetglytyrleuglntrpvalargaspleuthrglyaspgluile859095valproleutyrglnaspvalserproserproalapheserasnlys100105110leuphelystyrserserilealaphehistyrserlystyrvalglu115120125tyrmetaspthralatrpleuvalmetlysglylysprovalserleu130135140leuglnglyphehishispheglyalaalatrpaspthrtyrphegly145150155160ilethrpheglnasngluglyiletyrvalphevalvalleuasnala165170175pheilehisthrilemettyralatyrtyralaalathralaalagly180185190leulyspheserleulysphevalilethrleumetglnilethrgln195200205pheasnvalglyphevalmetvaltyrhistyrilethrleuglutyr210215220pheargasnserprogluleuvalphesertyrleupheasntyrala225230235240tyrvalcysthrvalleuleuleuphemetglnphephetyrmetasp245250255asnpheglylyslyslysalaalaalaalaalaglylyslyslyslys260265270<210>95<211>840<212>dna<213>pavlovasalina<400>95atggcgactgaagggatgccggcgataacgctggactggctgctctcgcccgggctgaag60gatgccgtaattggcggggaggtgctctacttttcgcttgggtatctgctgctcgagccc120atcctcaagcgctcaccgtttgtggacaagcgcaagggcgcataccgcaacggcatgatc180gcgtacaacatcctcatgtgcggtttctcgctggtatgcttcgtgtgccagatggcggcg240ctcggccttgatcgcggccacctgcagtttgtccgcgacctcacgggcgacagcgtggtg300cagctctaccaggacgtgagcccatcccctgcattcgcgaacaagctcttccggtactca360gcggtggcgttccactactcaaagtacgtggagtacatggacacagcgtggcttgtgctg420aagggcaagcccgtctcgttcctgcagggcttccaccacttcggcgccgcgtgggacacc480tactttggcatcacgtttcagaacgagggcacctacgtctttgtgctgctcaacgcattc540atccacacaatcatgtacacctactacggcgcgacggcagcgggcatcaaaatctcgatg600aagccgctgatcaccctcatgcagatcacgcagttcctgctgggcttcgcgctcgtctac660ccgtacattgacctcggctacttccgtgcgtcgcccgagctcgtgtggagctacctgttc720aactatgcgtacgtactcatggtgctcttcctcttcatgcgcttcttctaccacgacaac780tttagcaagcacaagccaatctcgcgcatcgactccagcaaccgcatgaaaaccgagtag840<210>96<211>279<212>prt<213>pavlovasalina<400>96metalathrgluglymetproalailethrleuasptrpleuleuser151015proglyleulysaspalavalileglyglygluvalleutyrpheser202530leuglytyrleuleuleugluproileleulysargserpropheval354045asplysarglysglyalatyrargasnglymetilealatyrasnile505560leumetcysglypheserleuvalcysphevalcysglnmetalaala65707580leuglyleuaspargglyhisleuglnphevalargaspleuthrgly859095aspservalvalglnleutyrglnaspvalserproserproalaphe100105110alaasnlysleupheargtyrseralavalalaphehistyrserlys115120125tyrvalglutyrmetaspthralatrpleuvalleulysglylyspro130135140valserpheleuglnglyphehishispheglyalaalatrpaspthr145150155160tyrpheglyilethrpheglnasngluglythrtyrvalphevalleu165170175leuasnalapheilehisthrilemettyrthrtyrtyrglyalathr180185190alaalaglyilelysilesermetlysproleuilethrleumetgln195200205ilethrglnpheleuleuglyphealaleuvaltyrprotyrileasp210215220leuglytyrpheargalaserprogluleuvaltrpsertyrleuphe225230235240asntyralatyrvalleumetvalleupheleuphemetargphephe245250255tyrhisaspasnpheserlyshislysproileserargileaspser260265270serasnargmetlysthrglu275<210>97<211>172<212>prt<213>tomatobushystuntvirus<400>97metgluargalaileglnglyasnaspalaarggluglnalaasnser151015gluargtrpaspglyglyserglyglythrthrserprophelysleu202530proaspgluserprosertrpthrglutrpargleuhisasnaspglu354045thrasnserasnglnaspasnproleuglyphelysglusertrpgly505560pheglylysvalvalphelysargtyrleuargtyraspargthrglu65707580alaserleuhisargvalleuglysertrpthrglyaspservalasn859095tyralaalaserargphepheglypheaspglnileglycysthrtyr100105110serileargpheargglyvalserilethrvalserglyglyserarg115120125thrleuglnhisleucysglumetalaileargserlysglngluleu130135140leuglnleualaproilegluvalgluserasnvalserargglycys145150155160progluglythrgluthrpheglulysgluserglu165170<210>98<211>116<212>prt<213>tomatoyellowleafcurlvirus<400>98mettrpaspproleuleuasnglupheprogluservalhisglyphe151015argcysmetleualailelystyrleuglnservalglugluthrtyr202530gluproasnthrleuglyhisaspleuileargaspleuileserval354045valargalaargasptyrvalglualathrargargtyrasnhisphe505560hisalaargleugluglyserprolysalagluleuargglnproile65707580glnglnprocyscyscysprohiscysproarghislysglnalathr859095ilemetaspvalglnalahisvalproglualaglnasnileglnasn100105110valserlyspro115<210>99<211>351<212>prt<213>turnipcrinklevirus<400>99metgluasnaspproargvalarglysphealasergluglyalagln151015trpalailelystrpglnlyslysglytrpserserleuthrserarg202530glnlysglnthralaargalaalametglyilelysleuserproval354045alaglnprovalglnlysvalthrargleuseralaprovalalaleu505560alatyrarggluvalserthrglnproargvalserthralaargasp65707580glyilethrargserglysergluleuilethrthrleulyslysasn859095thraspthrgluprolystyrthrthralavalleuasnproserglu100105110proglythrpheasnglnleuilelysglualaalaglntyrglulys115120125tyrargphethrserleuargpheargtyrserprometserproser130135140thrthrglyglylysvalalaleualapheaspargaspalaalalys145150155160proproproasnaspleualaserleutyrasnilegluglycysval165170175serservalprotrpthrglypheileleuthrvalprothraspser180185190thraspargphevalalaaspglyileseraspprolysleuvalasn195200205pheglylysleuilemetalathrtyrglyglnglyalaasnaspala210215220alaglnleuglygluvalargvalglutyrthrvalglnleulysasn225230235240argthrglyserthrseraspalaglnileglyaspphealaglyval245250255lysaspglyproargleuvalsertrpserlysthrlysglythrala260265270glytrpgluhisaspcyshispheleuglythrglyasnpheserleu275280285thrleuphetyrglulysalaprovalserglyleugluasnalaasp290295300alaserasppheservalleuglyglualaalaalaglyservalgln305310315320trpalaglyvallysvalalagluargglyglnservallysmetval325330335thrthrglugluglnproargglylystrpglnalaleuargile340345350<210>100<211>303<212>prt<213>peamosaicvirus<400>100methisglyilegluglnproglnleuproleuasptyrvalhisarg151015cysalaserthrserpheleuleualaserleuaspglyleuleuser202530glualaarggluleuserglyproleualaleuilethrsersertyr354045tyrleuleuvalserilealaleucystrpalaileproglyserphe505560trptyrargproglycystrpleuglnprovalserglyargasnleu65707580ilephecysglyprothrglualaleuglnargpheargleutyrala859095alaargleuglyleuvalleusergluasncysproarghisglygln100105110seralaalailethrleuglnsertyrtrpalaleuproasnasnile115120125trpmetaspmetalaglnleuaspleuleuthrphesermetproile130135140alaasnthrphealatyrleualaaspcysglualaargphepropro145150155160ilevalgluglyvalglyseralatyrtyrvalprothrleuleugly165170175leuthrhisglnaspproargleutyrleualaleuargargargasn180185190leuaspleuserglygluprohisargvalargproglyvalleuglu195200205sermetalaleuleucysserservalargserthrserargserarg210215220glnileproproleutyrglyservalleuhishisvalleuglyleu225230235240alagluargaspcysileleupheaspthraspserasntyrserser245250255tyrthrhisargvalleugluglnaspargasnargalaaspglnser260265270leupheserileaspleuglutyrvalhisaspleugluleuileala275280285leuglytyrseraspgluaspaspgluaspleuaspasnphephe290295300<210>101<211>256<212>prt<213>cerealyellowdwarfvirus<400>101metpheilealaglnprocysglyargvalleuvalpheaspvalala151015serargthrproserphephethrargtyrservalgluleuserleu202530argvalleuaspprophephethrargalavalthrasppheargtyr354045thrglnasngluileaspleuphecysvalserleuglypheleuleu505560proileleuleuthrglyglusertyrsertrpargglyhisleuasn65707580leuproleusertyrthrgluleuleuvalargtrpglyleualaval859095glytyrpheprothrpheserthraspglyaspileargglnasnpro100105110gluleuargileaspleuserthrmetserthrargserphetyrglu115120125glnpheleuleuargtyrasnthrserglyleualalysalaileval130135140glyglnglnglucyspheglnserglymetgluserphelysargphe145150155160leuhistyrargleuthrcyspheglusercysleuproargproarg165170175trpgluserproleualaproglyprotyrleuaspargalapheglu180185190alathrleuleuglyargmetvalglyhisasnglnleuleuphethr195200205glyleuserseraspilethrargtyrtyrasngluleuvalvalglu210215220glyvalprovalalaphetrpaspalaalaglyilethrleuhishis225230235240alaglygluglutyrpheserasnsertyrileglnlysileleugln245250255<210>102<211>519<212>dna<213>tomatobushystuntvirus<400>102atggaacgagctatacaaggaaacgacgctagggaacaagctaacagtgaacgttgggat60ggaggatcaggaggtaccacttctcccttcaaacttcctgacgaaagtccgagttggact120gagtggcggctacataacgatgagacgaattcgaatcaagataatccccttggtttcaag180gaaagctggggtttcgggaaagttgtatttaagagatatctcagatacgacaggacggaa240gcttcactgcacagagtccttggatcttggacgggagattcggttaactatgcagcatct300cgatttttcggtttcgaccagatcggatgtacctatagtattcggtttcgaggagttagt360atcaccgtttctggagggtcgcgaactcttcagcatctctgtgagatggcaattcggtct420aagcaagaactgctacagcttgccccaatcgaagtggaaagtaatgtatcaagaggatgc480cctgaaggtactgagaccttcgaaaaagaaagcgagtaa519<210>103<211>351<212>dna<213>tomatoyellowleafcurlvirus<400>103atgtgggatccacttctaaatgaatttcctgaatctgttcacggatttcgttgtatgtta60gctattaaatatttgcagtccgttgaggaaacttacgagcccaatacattgggccacgat120ttaattagggatcttatatctgttgtaagggcccgtgactatgtcgaagcgaccaggcga180tataatcatttccacgcccgcctcgaaggttcgccgaaggctgaacttcgacagcccata240cagcagccgtgctgctgtccccattgtccaaggcacaaacaagcgacgatcatggacgta300caggcccatgtaccggaagcccagaatatacagaatgtatcgaagccctga351<210>104<211>1056<212>dna<213>turnipcrinklevirus<400>104atggaaaatgatcctagagtccgaaagttcgcatccgagggcgcccaatgggcgataaag60tggcagaagaagggctggtcatccctaaccagcagacagaaacagaccgcccgcgcagcg120atggggatcaagctctcccctgtggcgcaacctgtgcagaaagtgactcgactgagtgct180ccggtggctctcgcctaccgcgaggtttccacccagcctcgggtttctactgccagggac240ggcataaccagaagcggttctgaactgatcacaaccctgaagaagaacactgacactgaa300cctaagtacaccacagctgtgcttaacccaagcgaacccggaacattcaaccaactcatc360aaggaggcggcccagtatgaaaaataccgattcacgtcactcagatttaggtactctccc420atgagcccttcaaccaccgggggcaaggtggctctggcattcgaccgagacgctgccaaa480cctccgcccaacgacctcgcttccctctacaacatagagggttgtgtatctagcgtgccc540tggacagggtttattttgaccgtcccaacagattctactgaccgctttgtggcggatggt600atcagcgatccaaagcttgtcaatttcggcaagctcatcatggccacctatggccaagga660gccaatgatgccgcccaactcggtgaagtgcgagtcgagtacaccgtgcagctcaagaac720agaactggctcaaccagcgacgcccagattggggacttcgcgggtgttaaggacggaccc780aggttggtctcgtggtccaagaccaagggaacagctgggtgggagcacgattgtcatttt840ctcggaaccggaaacttctcgttgacactgttctacgagaaggcgcccgtttcggggcta900gaaaacgcagacgcctctgacttctcggtcctgggagaagccgcagcaggtagtgtccaa960tgggctggagtgaaggtagcagaaaggggacaaagcgtgaaaatggtcacaactgaggag1020cagccaaggggaaaatggcaagcactcaggatttag1056<210>105<211>912<212>dna<213>peamosaicvirus<400>105atgcacggaattgagcagcctcaactaccgctagattacgttcaccgttgcgcatcaacc60tccttcttgctcgcatcactagatggcctcctttctgaagcccgtgaactctcagggcct120ctggctctcattacttctagctattacttacttgtttctattgccctctgctgggcaatc180cctggatccttctggtataggcctggctgctggttgcagccagtctcagggcggaatctc240atcttttgcggccctaccgaggccttgcaacgattccgtctgtacgctgccagacttggg300ttggtcctgtcagagaactgcccaagacacggccaatcagcagcaatcacccttcaatca360tactgggcacttcctaacaacatctggatggacatggcccaattggacttgctcaccttc420tcaatgccaattgctaatacatttgcctacttggcagattgtgaagcaagatttcctcct480attgttgaaggagtgggatctgcttactatgtgccaacgctgctcggacttactcaccaa540gaccccaggctttatcttgcgcttcgcaggagaaaccttgatcttagtggcgaacctcat600agagttcgtcctggtgtcctggagtctatggctttgctctgttctagtgtacgtagcaca660agccgttccaggcaaattcctcctttatatggcagcgttttgcaccacgttttgggcctg720gccgagagagactgcatcctctttgatacggatagtaactactcctcttacactcatcgg780gttcttgaacaagaccggaatcgggctgatcagtcattgtttagcattgacttggaatat840gttcatgacctggagcttattgccctgggttactctgatgaagatgatgaagatcttgat900aacttcttctag912<210>106<211>771<212>dna<213>cerealyellowdwarfvirus<400>106atgttcatcgcccaaccttgcgggcgagttcttgtgttcgacgtcgcctcccgcacgcca60tcgttcttcactcgttatagtgttgaactctcgctccgtgttctagacccattcttcacg120cgagcagtaacagatttccgatacacccaaaatgaaatcgatttattttgtgtgtctctt180ggctttctgttgccaattctcctcacaggagaatcttactcttggcgcggtcacctcaac240ctccccctttcttacaccgaattacttgttcgatgggggctcgcagtggggtacttccct300accttctccactgatggtgacattcgacagaacccagaactccgcatcgacctgtccacc360atgtcaacccgctctttctacgagcagttcctactcagatataacacgagtgggttggca420aaagctatcgtcggacagcaagagtgctttcaaagcggcatggagtcttttaaaagattc480ctacactaccgcctcacgtgctttgaaagctgccttccacgacctcgttgggaaagtcct540ttggctcctggtccttatctggacagggcttttgaggcaactcttctcggccgtatggtc600ggtcataaccaactactctttaccggtttgtcttctgatatcactaggtattataacgag660ttggttgtggaaggcgtgccggtggctttttgggacgctgccggcattactttgcatcac720gctggtgaagaatatttttcgaattcttacattcaaaagattcttcaatga771<210>107<211>1005<212>dna<213>micromonasccmp1545<400>107atgaccgctgctccagcttctgctagagatcctgctcttgctactggatctggatctctt60tacaaccacctcatgatgttcgcttggcttggagttgttcatgctgttgttgctcttgtt120gctgttgcttgctacttccttcctcatcctgttgctacttgtgctatcgctctcgttgct180cttgctgctcttactcctgttactactcctcatcctgcttggggacttgctatcgctaga240gctatcactaaggctgctgtgagatacttccctcttactatggaatgggaggatgagaga300gcttaccttgacgctgctgctaagggtgttcctgctgttatcggacttgagcctcattct360gttctccctttgtctatcgttgctttcggaaactacttcttcttcaccgaatctacacct420gagtgcgtgagaaactctagagctttggctactggaaccatcttcgttatccctgttctt480aagcacctttggtcttggcttggaatggatgctatctctaggcgtgctatgaagactctt540ctcgatgatggaagatctgttctcatcatccctggtggagttgctgagtgccttcaaatg600aggcctggtgttgagactatctacctcaagaagaggttcggattcgttaagcttgctatc660caaaccggtgcttctcttgttcctgctttcactttcggacagaccagatcttactcttac720tggcgtcttggacctcctcttgttcctcatgttgttgctgaggttttcgctagagcttgt780agagttgctcctatggttttctggggaaagtggggatctcctatccctaacatggttcct840atgcacactgttgttggaaagcctatccctgttaagaagcagtctgagccttctaacgag900tacgttcaagagaagctcaacgagttcgttgctgctatggaatctctttacgctagacac960aagggaaagcacggatacgctgagtctactctcgttgttctttga1005<210>108<211>334<212>prt<213>micromonasccmp1545<400>108metthralaalaproalaseralaargaspproalaleualathrgly151015serglyserleutyrasnhisleumetmetphealatrpleuglyval202530valhisalavalvalalaleuvalalavalalacystyrpheleupro354045hisprovalalathrcysalailealaleuvalalaleualaalaleu505560thrprovalthrthrprohisproalatrpglyleualailealaarg65707580alailethrlysalaalavalargtyrpheproleuthrmetglutrp859095gluaspgluargalatyrleuaspalaalaalalysglyvalproala100105110valileglyleugluprohisservalleuproleuserilevalala115120125pheglyasntyrphephephethrgluserthrproglucysvalarg130135140asnserargalaleualathrglythrilephevalileprovalleu145150155160lyshisleutrpsertrpleuglymetaspalaileserargargala165170175metlysthrleuleuaspaspglyargservalleuileileprogly180185190glyvalalaglucysleuglnmetargproglyvalgluthriletyr195200205leulyslysargpheglyphevallysleualaileglnthrglyala210215220serleuvalproalaphethrpheglyglnthrargsertyrsertyr225230235240trpargleuglyproproleuvalprohisvalvalalagluvalphe245250255alaargalacysargvalalaprometvalphetrpglylystrpgly260265270serproileproasnmetvalpromethisthrvalvalglylyspro275280285ileprovallyslysglnsergluproserasnglutyrvalglnglu290295300lysleuasngluphevalalaalametgluserleutyralaarghis305310315320lysglylyshisglytyralagluserthrleuvalvalleu325330<210>109<211>17<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<220><221>misc_feature<222>(2)..(2)<223>nisa,c,g,ort<220><221>misc_feature<222>(6)..(6)<223>nisa,c,g,ort<220><221>misc_feature<222>(10)..(15)<223>nisa,c,g,ort<400>109angatntatnnnnnngt17<210>110<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>110tgacaggatatattggcgggtaaac25<210>111<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>111tggcaggatatattgtggtgtaaac25<210>112<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>112tggcaggatatataccgttgtaatt25<210>113<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>113cggcaggatatattcaattgtaatt25<210>114<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>114tggtaggatatataccgttgtaatt25<210>115<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>115tggcaggatatatggtactgtaatt25<210>116<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<220><221>misc_feature<222>(1)..(1)<223>y=cort<220><221>misc_feature<222>(3)..(3)<223>r=aorg<220><221>misc_feature<222>(4)..(4)<223>y=cort<220><221>misc_feature<222>(14)..(14)<223>w=aort<220><221>misc_feature<222>(15)..(15)<223>s=corg<220><221>misc_feature<222>(16)..(16)<223>nisa,c,g,ort<220><221>misc_feature<222>(17)..(17)<223>v=a,corg<220><221>misc_feature<222>(18)..(18)<223>b=c,gort<220><221>misc_feature<222>(19)..(19)<223>k=gort<220><221>misc_feature<222>(24)..(24)<223>w=aort<220><221>misc_feature<222>(25)..(25)<223>y=cort<400>116ygryaggatatatwsnvbkgtaawy25<210>117<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>117cggcaggatatatcctgatgtaaat25<210>118<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>118tggcaggagttattcgagggtaaac25<210>119<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>119tgacaggatatatcgtgatgtcaac25<210>120<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>120gggaagtacatattggcgggtaaac25<210>121<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>121ttacaggatatattaatatgtatga25<210>122<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>122taacatgatatattcccttgtaaat25<210>123<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>123tgacaggatatatggtaatgtaaac25<210>124<211>25<212>dna<213>artificialsequence<220><223>transfernucleicacidbordersequence<400>124tggcaggatatataccgatgtaaac25<210>125<211>1395<212>dna<213>artificialsequence<220><223>codon-optimizednucleotidesequenceforproductioninplantsofseqidno:7<400>125atgtgtcctcctaagaccgatggaagatcttctcctagatctcctctcaccaggtctaag60tcatctgctgaggctcttgatgctaaggatgcttctaccgctcctgttgatcttaagacc120cttgagcctcatgaacttgctgctaccttcgagactagatgggttagggttgaggatgtt180gagtacgacgtgaccaacttcaaacatcctggtggaagcgtgatcttctacatgcttgct240aacactggtgctgatgctactgaggctttcaaagaatttcacatgcgtagcctcaaggct300tggaagatgcttagagctttgccttctagacctgctgagatcaagagatctgagtctgag360gatgctcctatgcttgaggatttcgctaggtggagagctgaacttgagagggacggattc420ttcaagccttctatcacccatgttgcttaccgtcttttggagcttcttgctactttcgct480cttggaaccgctcttatgtacgctggataccctatcattgctagcgttgtgtacggtgct540ttcttcggagctagatgtggatgggttcaacatgagggtggacacaactctcttaccgga600tctgtgtacgtggataagagacttcaggctatgacttgcggattcggactttctaccagc660ggagagatgtggaaccagatgcataacaagcaccatgctacccctcagaaagttagacac720gacatggatcttgataccactcctgctgtggctttcttcaacaccgctgtggaggataat780agacctaggggattctctagagcttgggctagacttcaagcttggaccttcgttcctgtt840acttctggacttctcgttcaggctttctggatctacgttctccatcctagacaggtgctc900aggaagaagaactacgaggaagcttcttggatgctcgtttctcacgttgttagaaccgct960gttatcaagcttgctaccggatactcttggcctgttgcttactggtggttcactttcgga1020aactggatcgcttacatgtacctcttcgctcacttctctacttctcacactcacctccct1080gttgttccatctgacaagcaccttagctgggttaactacgctgttgatcacaccgttgac1140atcgatccttctcgtggatacgttaactggcttatgggataccttaactgccaggttatc1200caccatctcttccctgatatgcctcaattcagacagcctgaggtgtcaagaagattcgtc1260cctttcgctaagaagtggggactcaactacaaggtgctctcttactacggtgcttggaag1320gctactttcagcaacctcgacaaagttggacagcactactacgttaacggaaaggctgag1380aaggctcactgatga1395<210>126<211>870<212>dna<213>artificialsequence<220><223>codon-optimizednucleotidesequenceforproductioninplantsofseqidno:3<400>126atggaatttgctcaacctctcgttgctatggctcaagagcagtacgctgctatcgatgct60gttgttgctcctgctatcttctctgctaccgactctattggatggggactcaagcctatc120tcttctgctactaaggatctccctctcgttgaatctcctacccctcttatcctttctctc180ctcgcttacttcgctatcgttggttctggactcgtttaccgtaaagtgttccctagaacc240gttaagggacaggatcctttccttctcaaggctcttatgctcgctcacaacgttttcctt300atcggactcagcctttacatgtgcctcaagctcgtttacgaggcttacgtgaacaagtac360tccttctggggaaacgcttacaaccctgctcaaaccgagatggctaaggtgatctggatc420ttctacgtgtccaagatctacgagttcatggacaccttcatcatgcttctcaagggaaac480gttaaccaggtttccttcctccatgtttaccaccacggatctatctctggaatctggtgg540atgatcacttatgctgctccaggtggagatgcttacttctctgctgctctcaactcttgg600gttcatgtgtgcatgtacacctactacttcatggctgctgttcttcctaaggacgaaaag660accaagagaaagtacctttggtggggaagataccttacccagatgcaaatgttccagttc720ttcatgaaccttctccaggctgtttacctcctctactcttcttctccttaccctaagttc780attgctcaactcctcgttgtttacatggttaccctcctcatgcttttcggaaacttctac840tacatgaagcaccacgcttctaagtgataa870<210>127<211>1281<212>dna<213>artificialsequence<220><223>codon-optimizednucleotidesequenceforproductioninplantsofseqidno:25<400>127atgcctcctagggactcttactcttacgctgctcctccttctgctcaacttcacgaggtt60gacactcctcaagagcacgacaagaaagagcttgttatcggagatagggcttacgatgtg120accaacttcgttaagagacaccctggtggaaagatcattgcttaccaagtgggaactgat180gctaccgatgcttacaagcagttccatgtgagatctgctaaggctgacaagatgctcaag240tctctcccttctagacctgttcacaagggatactctcctagaagagctgatcttatcgct300gacttccaagagttcactaagcaacttgaggctgagggaatgttcgaaccttctctccct360catgttgcttaccgtcttgctgaggttatcgctatgcatgttgctggtgctgctcttatc420tggcacggatacactttcgctggaatcgctatgcttggagttgttcagggaagatgcgga480tggcttatgcatgagggtggacactactctcttaccggaaacattgctttcgatagggct540atccaagttgcttgttacggacttggatgcggaatgtctggtgcttggtggagaaaccag600cataacaagcaccatgctactcctcaaaagctccagcacgatgttgatcttgataccctc660cctctcgttgctttccatgagagaatcgctgctaaggttaagtctcctgctatgaaggct720tggctctccatgcaagctaaactcttcgctcctgttaccactcttcttgttgctcttgga780tggcagctttaccttcaccctagacacatgctcagaactaagcactacgacgagcttgct840atgcttggtatcagatacggacttgtgggataccttgctgctaactacggtgctggatac900gttcttgcttgctaccttctctacgttcagcttggagctatgtacatcttctgcaacttc960gctgtttctcacactcatctccctgttgttgagcctaacgagcatgctacttgggttgag1020tacgctgctaaccacactactaactgctctccatcttggtggtgtgattggtggatgagc1080tacctcaactaccagatcgagcatcacctttacccttctatgcctcagttcaggcatcct1140aagatcgctcctagagtgaagcaactcttcgagaagcacggacttcactacgatgtgcgt1200ggatacttcgaggctatggctgatactttcgctaacctcgataacgttgctcatgctcct1260gagaagaaaatgcaatgatga1281<210>128<211>807<212>dna<213>artificialsequence<220><223>codon-optimizednucleotidesequenceforproductioninplantsofseqidno:5<400>128atggcttctatcgctatccctgctgctcttgctggaactcttggatacgtgacctacaac60gtggctaaccctgatattcctgcttctgagaaggttccagcttacttcatgcaagtggag120tactggggacctactatcggaactatcggttacctcctcttcatctacttcggaaagcgt180atcatgcaaaacagaagccagcctttcggacttaagaacgctatgctcgtgtacaacttc240taccagaccttcttcaacagctactgcatctacctcttcgttacctctcatagggctcag300ggacttaaagtttggggaaacatccctgatatgaccgctaactcttggggaatctctcag360gttatctggctccactacaacaacaagtacgtggagcttctcgataccttcttcatggtg420atgaggaagaagttcgaccagctttctttccttcacatctaccaccacactcttctcatc480tggtcatggttcgtggttatgaagctcgagcctgttggagattgctacttcggatctagc540gttaacaccttcgtgcacgtgatcatgtactcttactacggacttgctgctcttggagtt600aactgcttctggaagaagtacatcacccagatccagatgcttcagttctgtatctgcgct660tctcactctatctacaccgcttacgttcagaacactgctttctggcttccttaccttcag720ctctgggtgatggttaacatgttcgtgctcttcgctaacttctaccgtaaaaggtacaag780agcaagggtgctaagaagcagtgataa807<210>129<211>1347<212>dna<213>artificialsequence<220><223>codon-optimizednucleotidesequenceforproductioninplantsofseqidno:72<400>129atgcctccatctgctgctaaacagatgggagcttctactggtgttcacgctggtgttacc60gattcttctgctttcaccagaaaggatgtggctgatagacctgatcttaccatcgttggt120gactctgtgtacgatgctaaggctttcagatctgagcatcctggtggtgctcatttcgtt180tcactcttcggaggaagagatgctactgaggctttcatggaataccacagaagagcttgg240cctaagtctaggatgtctaggttccatgttggatctcttgcttctaccgaggaacctgtt300gctgctgatgagggataccttcagctttgtgctaggatcgctaagatggtgccttctgtg360tcatctggattcgctccagcttcttactgggttaaggctggacttatcctcggatctgct420atcgctcttgaggcttacatgctctacgctggaaagagacttctcccttctatcgttctt480ggatggctcttcgctcttatcggacttaacatccagcatgacgctaaccatggtgctttg540tctaagtctgctagcgttaaccttgctcttggactttgtcaggattggatcggaggatct600atgatcctttggctccaagagcatgttgttatgcaccacctccacaccaacgatgttgat660aaggaccctgatcaaaaggctcatggtgctcttagactcaagcctaccgatgcttggtca720cctatgcattggcttcagcacctttaccttctccctggtgaaactatgtacgctttcaag780ctcctcttcctcgatatctctgagcttgtgatgtggagatgggagggtgaacctatctct840aagctcgctggatacctcttcatgccttctcttctcctcaagcttaccttctgggctaga900ttcgttgctcttcctctttacctcgctccttctgttcatactgctgtgtgtatcgctgct960actgttatgaccggaagcttctaccttgctttcttcttcttcatcagccacaacttcgag1020ggtgttgcttctgttggacctgatggatctatcacctctatgaccaggggagcttctttc1080cttaagaggcaggctgagacttcttctaatgtgggaggacctcttcttgctactcttaac1140ggtggactcaactaccaaatcgagcaccaccttttccctagagttcaccacggattctac1200cctagacttgctcctcttgtgaaggctgaacttgaggctagaggaatcgagtacaagcac1260taccctaccatctggtctaacctcgcttctaccctcagacatatgtacgctcttggaaga1320aggcctagatctaaggctgagtgatga1347当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1