靶向骨髓瘤BCMA抗原的转基因T细胞及其制备方法与应用与流程
本发明涉及靶向骨髓瘤bcma抗原的转基因t细胞及其制备方法与应用。
背景技术:
多发性骨髓瘤是一种发生于骨髓的血癌,通常是由骨髓中的抗感染浆细胞癌变后增殖失控引起。多发性骨髓瘤可能导致免疫力下降并引起包括骨和肾脏等其它诸多问题。全球多发性骨髓瘤每年大约有114,200新增病例,并且每年有超过79,000人因此死亡。多发性骨髓瘤在我国的发病率约为十万分之一到十万分之二,欧美国家发病率为十万分之四。
多发性骨髓瘤的治疗经历了传统的化疗时期、造血干细胞移植时期、一直到目前的沙利度胺、来那度胺和硼替佐米等为代表的新药时期。即使如此,多发性骨髓瘤在很大程度上依然是一种难以治愈的疾病,只有大约45%的患者在确诊后能活过5年。许多患者暂停治疗之后病情还会反复复发。复发之后患者的5年内存活率不足20%。特别是那些对蛋白酶体抑制剂(硼替佐米和卡非佐米)及免疫调节剂(来那度胺,沙利度胺和泊马度胺)耐药的患者预后极差。在没有卡非佐米和泊马度胺的时代,这些患者的预期总生存只有9个月。fda在2015年12月批准了一种单抗治疗mm,靶向cd38。另外一种单抗靶向cs1完成随机、开放标签、多中心2期临床试验。anti-cd38daratumumab治疗响应率82.9%,对照药物组是63.2%;中位无疾病生存期是9.3月,对照药物组是6.5月。anti-cs1elotuzumab治疗响应率79%,对照药物组是66%;中位无疾病生存期是19.4个月,对照药物组是14.9个月。
2015年12月,美国jamesn.kochenderfer,md报道,靶向bcma的cart临床试验12患者,2cr,1vgpr,1pr,5sd。考虑到剂量使用,该疗法在9x10e6perkg时,疗效可能会有80%以上的cr。
2016年12月,bluebird公司报道,靶向bcma的cart临床试验11患者,低剂量出现1患者pr,高剂量出现2患者cr,1患者vgpr,3患者pr。
cart技术的基本原理嵌合抗原受体t细胞(car-t细胞)是将能识别某种肿瘤抗原的抗体的抗原结合部与cd3-ζ链或fcεriγ的胞内部分在体外偶联为一个嵌合蛋白,通过基因转导的方法转染患者的t细胞,使其表达嵌合抗原受体(car)。患者的t细胞被“重编码”后,生成大量肿瘤特异性的car-t细胞。
第一代car由识别肿瘤表面抗原的单链抗体(singlechainfragmentvariable,scfv)和免疫受体酪氨酸活化基序(immunoreceptortyrosine-basedactivationmotifs,itam,通常为cd3-ζ和fcεriγ)组成。早期的实验证明了car-t的可行性,然而第一代car只能引起短暂的t细胞增值和较低的细胞因子分泌,不能提供长时间的t细胞扩增信号和持续的体内抗肿瘤效应。
依照t细胞活化的双信号学说,t细胞的激活和增殖需要共刺激信号;第二代、第三代car引入了共刺激分子信号序列(costimulatorymolecule,cm),旨在提高t细胞的细胞毒活性、增殖性与存活时间,促进细胞因子的释放。现有的国内外第2代cart细胞培养技术,大致遵循类似的技术路线,即在分离患者外周血单个核细胞后,起始培养1~2天后,转染逆转录病毒(retroviral),或慢病毒(lentiviral),或转座子(transponson)为载体的基因表达嵌合抗原受体(chimericantigenreceptor,car)分子,加共刺激因素(il2等生长因子,辅助以cd3cd28抗体,或者抗原表达的细胞株等)培养7~30天后,回输患者。
欧美少量临床试验的数据表明,现有的bcmachimericantigenreceptort(cart)技术,治疗多发性骨髓瘤客观治愈率(objectiveresponse,obb)达50%左右。还存在的临床不足如下:
1、患者在治疗过程中需要密切监护,有中度的crs,主导原因之一是数十倍于正常水平的血清il6。欧美临床对此有比较好的处理方案,主要是il6r单抗或大剂量类固醇激素;
2、治疗有效率低于典型的cd19cart70-90%完全治愈率,有效率有待提高,有待于克服肿瘤微环境的抑制;
3、治疗成功后,患者体内长期缺乏生成抗体的浆细胞,因而必须每2~3周注射一次免疫球蛋白以保持基本免疫需求;
4、治疗过程中,部分患者丢失cart细胞。
技术实现要素:
针对上述现有技术,本发明提供了一种靶向骨髓瘤bcma抗原的转基因t细胞,及其制备方法。本发明在cart构建中,引入了egfr的识别序列,必要时可用egfr单抗cetuximab消除cart细胞,并且敲除了pd1、ctla4基因,解除了其对cart细胞的抑制,增强了cart细胞克服肿瘤微环境抑制免疫细胞功能。
本发明是通过以下技术方案实现的:
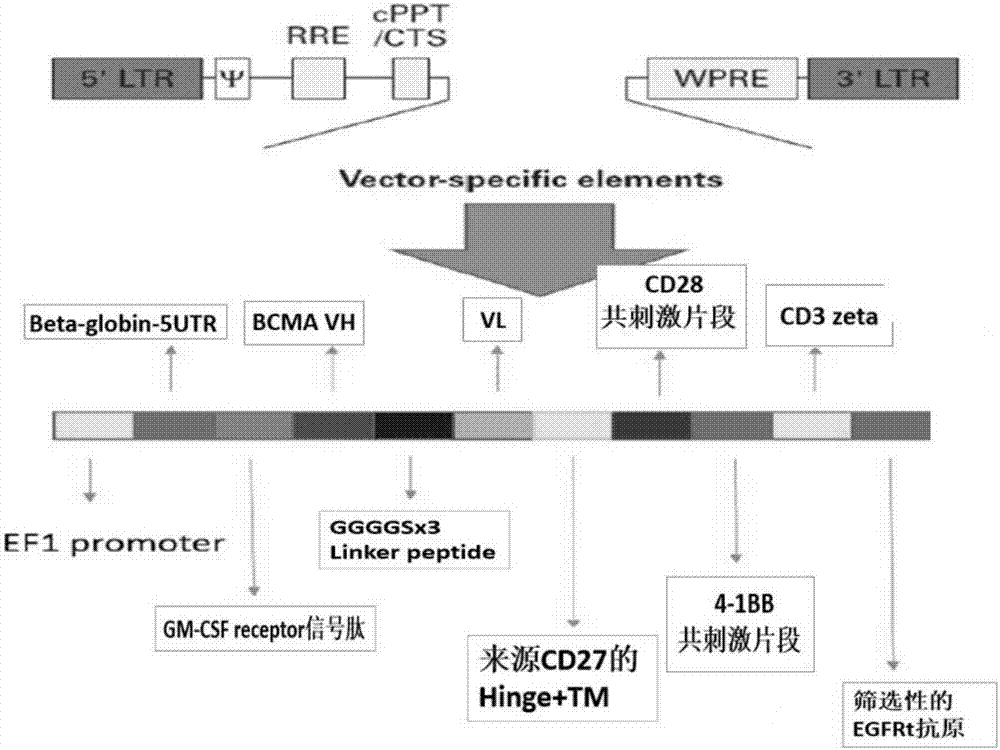
一种抗bcma嵌合抗原受体,其氨基酸序列如seqidno:1所示,包括以下顺式串联的结构域:抗bcma抗体的scfv片段bcmascfv、cd27的绞链区和跨膜区、cd28的胞内信号结构域、4-1bb的胞内信号结构域、cd3ζ的胞内信号结构域、t2a和egfrt抗原。
一种编码上述抗bcma嵌合抗原受体的基因,其核苷酸序列如seqidno:2所示;其结构为:anti-bcmascfv-cd27hinge-tm-cd28-41bb-cd3zeta,包括依次串联的scfc序列、cd27跨膜序列和膜外序列、cd28受体细胞内共刺激序列、是4-1bb受体细胞内共刺激序列、cd3zeta序列、t2a序列、egfrt的识别序列(这些序列的cdna序列拼接后,以软件作密码子优化,以适合于人类细胞表达,去除了某些限制性内切酶序列,去除了潜在的强二级结构,去除了gc或at密集区,得到seqidno:2所示的序列)。
一种重组表达载体,为慢病毒表达载体、逆转录病毒表达载体、腺病毒表达载体、腺相关病毒表达载体或质粒,其包含有上述编码抗bcma嵌合抗原受体的基因。该重组表达载体的构建方法,为常规方法。
一种靶向骨髓瘤bcma抗原的转基因t细胞,为包含有上述重组表达载体的、且敲除了pd1基因或/和ctla4基因的原始细胞,或染色体中整合有上述抗bcma嵌合抗原受体的基因的、且敲除了pd1基因或/和ctla4基因的原始细胞,所述原始细胞为cd4+t细胞或cd8+t细胞。
一种靶向骨髓瘤bcma抗原的转基因t细胞的制备方法:grna、crispr-cas9mrna、hdr混合,电转重组t细胞(400v,0.5ms),即得;所述grna为靶向pd1基因的grna或靶向ctla4基因的grna,所述hdr为pd1基因的hdr或ctla4基因的hdr;
所述重组t细胞,为包含有上述重组表达载体的原始细胞,或染色体中整合有上述抗bcma嵌合抗原受体的基因的原始细胞(该重组t细胞,可通过常规的方法获得:构建组表达载体后,慢病毒转染即可得到),所述原始细胞为cd4+t细胞或cd8+t细胞。
所述crispr-cas9mrna,是通过体外转录spcas9-2.0得到。spcas9(bb)-2a-puro(px459)v2.0(简称spcas9-2.0)为现有技术中已有的序列,为张峰(zhangfeng)所发布,其核苷酸序列如seqidno:3所示(序列所示为相对应的dna序列);克隆出spcas9-2.0的cas9蛋白的mrna,克隆到puc19质粒中,经过invitro转录和5’加帽反应,形成成熟的mrna,供细胞转染。
所述靶向pd1基因的grna,其核苷酸序列如seqidno:4所示(序列所示为相对应的dna序列)。
所述靶向ctla4基因的grna,其核苷酸序列如seqidno:5所示(序列所示为相对应的dna序列)。
所述pd1基因的hdr,其核苷酸序列如seqidno:6所示。
所述ctla4基因的hdr,其核苷酸序列如seqidno:7所示。
所述靶向骨髓瘤bcma抗原的转基因t细胞在制备治疗多发性骨髓瘤的药物中的应用。
本发明的技术方案,针对背景技术中的问题1和3,本发明提出必要时用egfr单抗cetuximab消除cart细胞,因此,在cart构建中,引入了egfr的识别序列。针对背景技术中的问题2,本专发明提出用基因编辑的方法,敲除pd1、ctla4基因(两种基因分别敲除,不宜同时敲除,理由是:1、基因敲除的效率低,两种同时敲除时效率更低;2、pd1、ctla4基因均位于2号染色体,同时敲除有可能会导致染色体大片段缺失),解除对cart细胞的抑制,增强cart细胞克服肿瘤微环境抑制免疫细胞功能。针对背景技术中的问题3,本发明提出用egfr单抗cetuximab消除cart细胞。针对背景技术中的问题4,本发明改进了bcmacart的培养工艺。
附图说明
图1:慢病毒载体构建简图。
图2:car分子及附属筛选标记的设计图。
图3:各种构建的bcmacart在k562-bcma细胞株培养后的短期增殖曲线。
图4:各种构建的bcmacart对k562-bcma细胞株的杀伤活性。
图5:ap1903分子诱导各种构建的bcmacart程序死亡图。
图6:cetuximab诱导各种构建的bcmacart细胞毒性(cytotoxicity)图。
具体实施方式
下面结合实施例对本发明作进一步的说明。
下述实施例中所涉及的仪器、试剂、材料等,若无特别说明,均为现有技术中已有的常规仪器、试剂、材料等,可通过正规商业途径获得。下述实施例中所涉及的实验方法,检测方法等,若无特别说明,均为现有技术中已有的常规实验方法,检测方法等。
实施例1构建慢病毒载体
慢病毒载体构建简图如图1所示。
car分子及附属筛选标记的设计如图2所示。
所构建的car分子,其核苷酸序列如seqidno:2所示。
慢病毒包被:为常规方法。简单而言,用rpmi1640+10%fbs培养293t细胞,待细胞90%密度后,用lipo2000混合转染质粒,转染293t细胞,收获慢病毒后,离心浓缩。质粒转染293t细胞的方法按照invitrogen厂商的lipofactamine2000的试剂指南。质粒有:慢病毒表达质粒,辅助质粒3种(pmdlg/prre,prsv-rev,pmd2.g),混合质粒的摩尔比例为2:1:1:0.5。转染24~48小时后,收获含有慢病毒的上清;加clontech的lenti-xconcentrator,混合后1500g,4c离心45min,去除上清,获取慢病毒沉淀,用于后续实验。
实施例2培养和慢病毒转染t细胞
健康人或肿瘤患者的外周血抽取50~100ml,或者用cobraspectra血细胞分离机获取单个核细胞,经过ficoll分离后,用cd4+或cd8+磁珠分选(磁珠购自stemcelltechnologies公司)。t细胞用培养1天后(培养基配方:lonzaxvivo15,成人血清10%,il2300-500iu/ml,penicillin(100units/ml)andstreptomycin(100μg/ml),用抗cd3cd28的磁珠(与t细胞1:3混合)培养24小时后,感染慢病毒。
慢病毒感染人cd4+t或cd8+t细胞:制备和浓缩后的慢病毒感染参考takararetronectin说明书,简单描述如下:
制备retronectin浓度20~100μg/ml,铺板使用密度是4~20μg/cm2,室温2小时后,吸去上清备用;用慢病毒加到上述板125~250μl/cm2,37c温浴4~6小时;待感染t细胞以密度0.5~2.5×104cells/cm2铺板。t细胞感染24小时后换液。
刺激bcmacart细胞生长8~12天,保持细胞浓度在1~2×106cells/ml。然后分选bcmacart的性能。
实施例3敲除pd1基因、ctla4基因
电转染crispr-cas9进行t细胞的基因编辑,分别进行敲除pd1基因、ctla4基因的实验,具体方式如下:
体外合成的crispr-cas9mrna、grna混合hdr,电转t细胞(400v,0.5ms)。细胞在lonzaxvivo15,成人血清10%,il2300-500iu/ml培养3天后,收获基因组dna。然后做genomicpcr(靶向pd1的exon2附近区域的引物,forward:ttcctcacctctctccatctc;reverse:ctctctttgatctgcgcctt,如seqidno:8、seqidno:9所示。靶向ctla4的exon2附近区域的引物:forward:tgagttcactgagttccctttg;reverse:gaaatggctttgctcaccaatta,如seqidno:10、seqidno:11所示),agarose胶纯化后,进行ta克隆,纯化单个克隆后测序获取indel突变信息,计算突变和非突变序列的信息,基因编辑效率pd1:13.5%,ctla4:15.1%。
所述crispr-cas9mrna核苷酸序列如seqidno:3所示;
敲除pd1基因时,靶向pd1基因的grna,核苷酸序列如seqidno:4所示;pd1基因的hdr核苷酸序列如seqidno:6所示;
敲除ctla4基因时,靶向ctla4基因的grna,核苷酸序列如seqidno:5所示;ctla4基因的hdr核苷酸序列如seqidno:7所示。
实施例4bcmacart的细胞性能鉴定
图3:各种构建的bcmacart在k562-bcma细胞株培养后的短期增殖曲线。完全培养基为lonzaxvivo15,成人血清10%,il2300-500iu/ml。各个组别为:bcmacart(car分子构建不含cd28-41bb共刺激序列);bcmacart(car分子构建不含cd28共刺激序列);bcmacart(car分子构建不含41bb共刺激序列);bcmacart(car分子构建含cd28-41bb共刺激序列)。通过图3可见,本发明构建的bcmacart(含cd28-41bb共刺激序列),效果明显优于其它,有显著差异。
图4:各种构建的bcmacart对k562-bcma细胞株的杀伤活性。完全培养基为lonzaxvivo15,成人血清10%,il2300-500iu/ml。3各个组别为:bcmacart(car受体含cd28共刺激序列);bcmacart(car受体构建含41bb共刺激序列);bcmacart(car受体含cd28-41bb共刺激序列)。
图5:ap1903分子诱导各种构建的bcmacart程序死亡图。完全培养基为lonzaxvivo15,成人血清10%,il2300-500iu/ml。体外培养皿中,在培养基中添加5nmap1903分子后,bcmacart细胞短时间内进入程序死亡,活性细胞数量下降。各个组别为:载体gfp阴性对照;bcmacart(car受体胞外区含cd27序列);bcmacart(car受体胞外区含cd8序列)。
图6:cetuximab诱导各种构建的bcmacart细胞毒性(cytotoxicity)图。完全培养基为lonzaxvivo15,非热灭活的成人血清10%,il2300-500iu/ml。体外培养皿中,在培养基中添加1000ng/mlcetuximab后,bcmacart细胞短时间内进入程序死亡,活性细胞数量下降。各个组别为:载体gfp阴性对照;bcmacart(car受体含cd28序列);bcmacart(car受体含cd28-41bb序列)。
序列表
<110>银丰生物工程基团有限公司
湖北省银丰鼎诚生物工程有限公司
<120>靶向骨髓瘤bcma抗原的转基因t细胞及其制备方法与应用
<141>2017-10-18
<160>11
<170>siposequencelisting1.0
<210>1
<211>932
<212>prt
<213>artificialsequence
<400>1
metleuleuleuvalthrserleuleuleucysgluleuprohispro
151015
alapheleuleuileprogluglnileglnleuvalglnserglypro
202530
gluleulyslysproglygluthrvallysilesercyslysalaser
354045
glytyrthrphethrasptyrserileasntrpvallysargalapro
505560
glylysglyleulystrpmetglytrpileasnthrgluthrargglu
65707580
proalatyralatyrasppheargglyargphealapheserleuglu
859095
thrseralaserthralatyrleuglnileasnasnleulystyrglu
100105110
aspthralathrtyrphecysalaleuasptyrsertyralametasp
115120125
tyrtrpglyglnglythrservalthrvalserserglyglyglygly
130135140
serglyglyglyglyserglyglyglyglyseraspilevalleuthr
145150155160
glnserproproserleualametserleuglylysargalathrile
165170175
sercysargalasergluservalthrileleuglyserhisleuile
180185190
histrptyrglnglnlysproglyglnproprothrleuleuilegln
195200205
leualaserasnvalglnthrglyvalproalaargpheserglyser
210215220
glyserargthraspphethrleuthrileaspprovalglugluasp
225230235240
aspvalalavaltyrtyrcysleuglnserargthrileproargthr
245250255
pheglyglyglythrlysleugluilelysglyglyglyglyserpro
260265270
thrhisleuprotyrvalserglumetleuglualaargthralagly
275280285
hismetglnthrleualaasppheargglnleuproalaargthrleu
290295300
serthrhistrpproproglnargserleuglyserserasppheile
305310315320
argileleuvalilepheserglymetpheleuvalphethrleuala
325330335
glyalaleupheleuhisglnargserlysargserargglyglyhis
340345350
serasptyrmetasnmetthrproargargproglyprothrarglys
355360365
histyrglnprotyralaproproargaspphealaalatyrargser
370375380
servalvallysargglyarglyslysleuleutyrilephelysgln
385390395400
prophemetargprovalglnthrthrglnglugluaspglycysser
405410415
cysargpheproglugluglugluglyglycysgluleuargvallys
420425430
pheserargseralaaspalaproalatyrglnglnglyglnasngln
435440445
leutyrasngluleuasnleuglyargarggluglutyraspvalleu
450455460
asplysargargglyargaspproglumetglyglylysproargarg
465470475480
lysasnproglngluglyleutyrasngluleuglnlysasplysmet
485490495
alaglualatyrsergluileglymetlysglygluargargarggly
500505510
lysglyhisaspglyleutyrglnglyleuserthralathrlysasp
515520525
thrtyraspalaleuhismetglnalaleuproproarggluglyarg
530535540
glyserleuleuthrcysglyaspvalglugluasnproglyproala
545550555560
cysglyalaaspsertyrglumetglugluaspglyvalarglyscys
565570575
lyslyscysgluglyprocysarglysvalcysasnglyileglyile
580585590
glygluphelysaspserleuserileasnalathrasnilelyshis
595600605
phelysasncysthrserileserglyaspleuhisileleuproval
610615620
alapheargglyaspserphethrhisthrproproleuaspprogln
625630635640
gluleuaspileleulysthrvallysgluilethrglypheleuleu
645650655
ileglnalatrpprogluasnargthraspleuhisalaphegluasn
660665670
leugluileileargglyargthrlysglnhisglyglnpheserleu
675680685
alavalvalserleuasnilethrserleuglyleuargserleulys
690695700
gluileseraspglyaspvalileileserglyasnlysasnleucys
705710715720
tyralaasnthrileasntrplyslysleupheglythrserglygln
725730735
lysthrlysileileserasnargglygluasnsercyslysalathr
740745750
glyglnvalcyshisalaleucysserprogluglycystrpglypro
755760765
gluproargaspcysvalsercysargasnvalserargglyargglu
770775780
cysvalasplyscysasnleuleugluglygluproargglupheval
785790795800
gluasnserglucysileglncyshisproglucysleuproglnala
805810815
metasnilethrcysthrglyargglyproaspasncysileglncys
820825830
alahistyrileaspglyprohiscysvallysthrcysproalagly
835840845
valmetglygluasnasnthrleuvaltrplystyralaaspalagly
850855860
hisvalcyshisleucyshisproasncysthrtyrglycysthrgly
865870875880
proglyleugluglycysprothrasnglyprolysileproserile
885890895
alathrglymetvalglyalaleuleuleuleuleuvalvalalaleu
900905910
glyileglyleuphemetargargarghisilevalarglysargthr
915920925
leuargargleu
930
<210>2
<211>2781
<212>dna
<213>artificialsequence
<400>2
atgctgctgttggtcacatctttgttgctctgtgaacttccgcatccggcattccttctc60
attcctgagcaaattcagcttgttcaaagtggcccagaactgaagaaacccggggaaacc120
gtgaagattagttgcaaagcgagtggttatacgttcacggattattccataaactgggtg180
aagcgagcgccgggcaagggtcttaaatggatgggctggattaacacagaaacccgggaa240
cctgcctatgcgtatgattttagagggcgatttgcgttttctcttgagacatcagcaagc300
acggcctacttgcagataaacaacctcaagtacgaagataccgcaacatacttttgtgct360
ttggattactcatatgcgatggattattggggtcagggaacaagtgtcacggtgagtagc420
ggcggagggggatctggtggcggaggttccggcggagggggatctgacatagtccttacc480
cagagtccaccttccctggctatgtccctcgggaaaagagcgaccatcagctgtagagcg540
agtgagtcagtcaccattctcggcagtcatttgatccactggtatcagcaaaagcccgga600
cagccaccgactttgctgatccaactggcctccaatgtccaaacaggtgtgcccgcccgg660
tttagcggatcagggtcccggacagactttacattgacgatagaccctgtcgaggaagat720
gacgtagcggtatactattgtctccagtcaagaacgatcccacgcacattcggcggtgga780
actaaactcgagataaaaggcgggggaggttcaccgacacacctgccgtacgtttctgaa840
atgctggaggctcgcactgctggccacatgcagaccctcgctgactttaggcaactgcca900
gcacggaccctcagcacccactggcctccacagcggagtcttggctcaagtgactttatc960
aggatactcgtgatattttcaggcatgtttttggtttttactctcgcgggtgcactcttt1020
ctgcatcaacggagcaaaaggtctaggggagggcacagcgattacatgaacatgactcct1080
cggcggccagggccgacacgcaagcactaccaaccctatgcgccgccgagagattttgcg1140
gcttaccgctcaagtgtcgttaaaaggggtcgaaaaaagctgctgtatatattcaaacaa1200
cccttcatgagaccagtccaaaccacgcaagaggaggatgggtgttcctgtcgatttcct1260
gaagaggaagaaggagggtgcgaacttagggtaaaattcagccgatccgctgatgcccct1320
gcgtaccaacaaggacagaaccagctgtataacgagctcaacctgggccggagggaagaa1380
tacgacgtcttggataaaagacgcgggcgggaccccgaaatggggggaaagccgaggcga1440
aaaaatcctcaggagggtttgtacaacgaacttcaaaaagacaagatggccgaggcctat1500
tccgagatagggatgaaaggagagcggcgccgagggaaaggtcacgacgggctgtaccaa1560
ggtctgtcaactgcaaccaaggatacctatgacgcccttcatatgcaggccttgcctcct1620
agagagggcagaggcagcctgctgacctgcggcgacgtggaggagaaccccggccccatg1680
gcctgtggggccgacagctatgagatggaggaagacggcgtccgcaagtgtaagaagtgc1740
gaagggccttgccgcaaagtgtgtaacggaataggtattggtgaatttaaagactcactc1800
tccataaatgctacgaatattaaacacttcaaaaactgcacctccatcagtggcgatctc1860
cacatcctgccggtggcatttaggggtgactccttcacacatactcctcctctggatcca1920
caggaactggatattctgaaaaccgtaaaggaaatcacagggtttttgctgattcaggct1980
tggcctgaaaacaggacggacctccatgcctttgagaacctagaaatcatacgcggcagg2040
accaagcaacatggtcagttttctcttgcagtcgtcagcctgaacataacatccttggga2100
ttacgctccctcaaggagataagtgatggagatgtgataatttcaggaaacaaaaatttg2160
tgctatgcaaatacaataaactggaaaaaactgtttgggacctccggtcagaaaaccaaa2220
attataagcaacagaggtgaaaacagctgcaaggccacaggccaggtctgccatgccttg2280
tgctcccccgagggctgctggggcccggagcccagggactgcgtctcttgccggaatgtc2340
agccgaggcagggaatgcgtggacaagtgcaaccttctggagggtgagccaagggagttt2400
gtggagaactctgagtgcatacagtgccacccagagtgcctgcctcaggccatgaacatc2460
acctgcacaggacggggaccagacaactgtatccagtgtgcccactacattgacggcccc2520
cactgcgtcaagacctgcccggcaggagtcatgggagaaaacaacaccctggtctggaag2580
tacgcagacgccggccatgtgtgccacctgtgccatccaaactgcacctacggatgcact2640
gggccaggtcttgaaggctgtccaacgaatgggcctaagatcccgtccatcgccactggg2700
atggtgggggccctcctcttgctgctggtggtggccctggggatcggcctcttcatgcga2760
aggcgccacatcgtttgataa2781
<210>3
<211>4869
<212>dna
<213>artificialsequence
<400>3
atggccccaaagaagaagcggaaggtcggtatccacggagtcccagcagccgacaagaag60
tacagcatcggcctggacatcggcaccaactctgtgggctgggccgtgatcaccgacgag120
tacaaggtgcccagcaagaaattcaaggtgctgggcaacaccgaccggcacagcatcaag180
aagaacctgatcggagccctgctgttcgacagcggcgaaacagccgaggccacccggctg240
aagagaaccgccagaagaagatacaccagacggaagaaccggatctgctatctgcaagag300
atcttcagcaacgagatggccaaggtggacgacagcttcttccacagactggaagagtcc360
ttcctggtggaagaggataagaagcacgagcggcaccccatcttcggcaacatcgtggac420
gaggtggcctaccacgagaagtaccccaccatctaccacctgagaaagaaactggtggac480
agcaccgacaaggccgacctgcggctgatctatctggccctggcccacatgatcaagttc540
cggggccacttcctgatcgagggcgacctgaaccccgacaacagcgacgtggacaagctg600
ttcatccagctggtgcagacctacaaccagctgttcgaggaaaaccccatcaacgccagc660
ggcgtggacgccaaggccatcctgtctgccagactgagcaagagcagacggctggaaaat720
ctgatcgcccagctgcccggcgagaagaagaatggcctgttcggaaacctgattgccctg780
agcctgggcctgacccccaacttcaagagcaacttcgacctggccgaggatgccaaactg840
cagctgagcaaggacacctacgacgacgacctggacaacctgctggcccagatcggcgac900
cagtacgccgacctgtttctggccgccaagaacctgtccgacgccatcctgctgagcgac960
atcctgagagtgaacaccgagatcaccaaggcccccctgagcgcctctatgatcaagaga1020
tacgacgagcaccaccaggacctgaccctgctgaaagctctcgtgcggcagcagctgcct1080
gagaagtacaaagagattttcttcgaccagagcaagaacggctacgccggctacattgac1140
ggcggagccagccaggaagagttctacaagttcatcaagcccatcctggaaaagatggac1200
ggcaccgaggaactgctcgtgaagctgaacagagaggacctgctgcggaagcagcggacc1260
ttcgacaacggcagcatcccccaccagatccacctgggagagctgcacgccattctgcgg1320
cggcaggaagatttttacccattcctgaaggacaaccgggaaaagatcgagaagatcctg1380
accttccgcatcccctactacgtgggccctctggccaggggaaacagcagattcgcctgg1440
atgaccagaaagagcgaggaaaccatcaccccctggaacttcgaggaagtggtggacaag1500
ggcgcttccgcccagagcttcatcgagcggatgaccaacttcgataagaacctgcccaac1560
gagaaggtgctgcccaagcacagcctgctgtacgagtacttcaccgtgtataacgagctg1620
accaaagtgaaatacgtgaccgagggaatgagaaagcccgccttcctgagcggcgagcag1680
aaaaaggccatcgtggacctgctgttcaagaccaaccggaaagtgaccgtgaagcagctg1740
aaagaggactacttcaagaaaatcgagtgcttcgactccgtggaaatctccggcgtggaa1800
gatcggttcaacgcctccctgggcacataccacgatctgctgaaaattatcaaggacaag1860
gacttcctggacaatgaggaaaacgaggacattctggaagatatcgtgctgaccctgaca1920
ctgtttgaggacagagagatgatcgaggaacggctgaaaacctatgcccacctgttcgac1980
gacaaagtgatgaagcagctgaagcggcggagatacaccggctggggcaggctgagccgg2040
aagctgatcaacggcatccgggacaagcagtccggcaagacaatcctggatttcctgaag2100
tccgacggcttcgccaacagaaacttcatgcagctgatccacgacgacagcctgaccttt2160
aaagaggacatccagaaagcccaggtgtccggccagggcgatagcctgcacgagcacatt2220
gccaatctggccggcagccccgccattaagaagggcatcctgcagacagtgaaggtggtg2280
gacgagctcgtgaaagtgatgggccggcacaagcccgagaacatcgtgatcgaaatggcc2340
agagagaaccagaccacccagaagggacagaagaacagccgcgagagaatgaagcggatc2400
gaagagggcatcaaagagctgggcagccagatcctgaaagaacaccccgtggaaaacacc2460
cagctgcagaacgagaagctgtacctgtactacctgcagaatgggcgggatatgtacgtg2520
gaccaggaactggacatcaaccggctgtccgactacgatgtggaccatatcgtgcctcag2580
agctttctgaaggacgactccatcgacaacaaggtgctgaccagaagcgacaagaaccgg2640
ggcaagagcgacaacgtgccctccgaagaggtcgtgaagaagatgaagaactactggcgg2700
cagctgctgaacgccaagctgattacccagagaaagttcgacaatctgaccaaggccgag2760
agaggcggcctgagcgaactggataaggccggcttcatcaagagacagctggtggaaacc2820
cggcagatcacaaagcacgtggcacagatcctggactcccggatgaacactaagtacgac2880
gagaatgacaagctgatccgggaagtgaaagtgatcaccctgaagtccaagctggtgtcc2940
gatttccggaaggatttccagttttacaaagtgcgcgagatcaacaactaccaccacgcc3000
cacgacgcctacctgaacgccgtcgtgggaaccgccctgatcaaaaagtaccctaagctg3060
gaaagcgagttcgtgtacggcgactacaaggtgtacgacgtgcggaagatgatcgccaag3120
agcgagcaggaaatcggcaaggctaccgccaagtacttcttctacagcaacatcatgaac3180
tttttcaagaccgagattaccctggccaacggcgagatccggaagcggcctctgatcgag3240
acaaacggcgaaaccggggagatcgtgtgggataagggccgggattttgccaccgtgcgg3300
aaagtgctgagcatgccccaagtgaatatcgtgaaaaagaccgaggtgcagacaggcggc3360
ttcagcaaagagtctatcctgcccaagaggaacagcgataagctgatcgccagaaagaag3420
gactgggaccctaagaagtacggcggcttcgacagccccaccgtggcctattctgtgctg3480
gtggtggccaaagtggaaaagggcaagtccaagaaactgaagagtgtgaaagagctgctg3540
gggatcaccatcatggaaagaagcagcttcgagaagaatcccatcgactttctggaagcc3600
aagggctacaaagaagtgaaaaaggacctgatcatcaagctgcctaagtactccctgttc3660
gagctggaaaacggccggaagagaatgctggcctctgccggcgaactgcagaagggaaac3720
gaactggccctgccctccaaatatgtgaacttcctgtacctggccagccactatgagaag3780
ctgaagggctcccccgaggataatgagcagaaacagctgtttgtggaacagcacaagcac3840
tacctggacgagatcatcgagcagatcagcgagttctccaagagagtgatcctggccgac3900
gctaatctggacaaagtgctgtccgcctacaacaagcaccgggataagcccatcagagag3960
caggccgagaatatcatccacctgtttaccctgaccaatctgggagcccctgccgccttc4020
aagtactttgacaccaccatcgaccggaagaggtacaccagcaccaaagaggtgctggac4080
gccaccctgatccaccagagcatcaccggcctgtacgagacacggatcgacctgtctcag4140
ctgggaggcgacaaaaggccggcggccacgaaaaaggccggccaggcaaaaaagaaaaag4200
gaattcggcagtggagagggcagaggaagtctgctaacatgcggtgacgtcgaggagaat4260
cctggcccaatgaccgagtacaagcccacggtgcgcctcgccacccgcgacgacgtcccc4320
agggccgtacgcaccctcgccgccgcgttcgccgactaccccgccacgcgccacaccgtc4380
gatccggaccgccacatcgagcgggtcaccgagctgcaagaactcttcctcacgcgcgtc4440
gggctcgacatcggcaaggtgtgggtcgcggacgacggcgccgcggtggcggtctggacc4500
acgccggagagcgtcgaagcgggggcggtgttcgccgagatcggcccgcgcatggccgag4560
ttgagcggttcccggctggccgcgcagcaacagatggaaggcctcctggcgccgcaccgg4620
cccaaggagcccgcgtggttcctggccaccgtcggagtctcgcccgaccaccagggcaag4680
ggtctgggcagcgccgtcgtgctccccggagtggaggcggccgagcgcgccggggtgccc4740
gccttcctggagacctccgcgccccgcaacctccccttctacgagcggctcggcttcacc4800
gtcaccgccgacgtcgaggtgcccgaaggaccgcgcacctggtgcatgacccgcaagccc4860
ggtgcctga4869
<210>4
<211>98
<212>dna
<213>artificialsequence
<400>4
gcggagagcttcgtgctaaacgttttagagctagaaatagcaagttaaaataaggctagt60
ccgttatcaacttgaaaaagtggcaccgagtcggtgct98
<210>5
<211>98
<212>dna
<213>artificialsequence
<400>5
ggtgcggcaacctacatgatggttttagagctagaaatagcaagttaaaataaggctagt60
ccgttatcaacttgaaaaagtggcaccgagtcggtgct98
<210>6
<211>100
<212>dna
<213>artificialsequence
<400>6
aacgccaccttcacctgcagcttctccaacacatcggagagcttcgtgtgataatggtac60
cgcatgagccccagcaaccagacggacaagctggccgctt100
<210>7
<211>99
<212>dna
<213>artificialsequence
<400>7
caggctgacagccaggtgactgaagtctgtgcggcaacctacatgtgataaaatgagttg60
accttcctagatgattccatctgcacgggcacctccagt99
<210>8
<211>21
<212>dna
<213>artificialsequence
<400>8
ttcctcacctctctccatctc21
<210>9
<211>20
<212>dna
<213>artificialsequence
<400>9
ctctctttgatctgcgcctt20
<210>10
<211>22
<212>dna
<213>artificialsequence
<400>10
tgagttcactgagttccctttg22
<210>11
<211>23
<212>dna
<213>artificialsequence
<400>11
gaaatggctttgctcaccaatta23
- 还没有人留言评论。精彩留言会获得点赞!
- 阻断UBE2M介导Neddylation修饰在肾细胞癌治疗中的应用的制造方法与工艺
- 无载体共组装肿瘤靶向抗癌纳米药物及其制备方法与应用与流程
- 一种用于判断靶向单克隆抗体治疗肿瘤疗效的预测分子的制造方法与工艺
- 一种靶向CD47的免疫检查点抑制剂药物组合物及其制备方法与流程
- 具有淋巴靶向功能的生物自组装纳米晶注射剂及制备方法与流程
- 用于检测肝癌的基因标志物及其用途的制造方法与工艺
- 特异性shRNA筛选及其靶向Ang‑2基因抑制肺癌细胞的验证方法与流程
- 一种多功能自动化生物细胞提取分离控制系统及采用该系统实现的细胞提取分离方法与流程
- 一种融合蛋白及光敏剂复合物及其制备方法和应用与流程
- 具有可视化和显示光斑边界功能的光动力治疗系统的制造方法与工艺
- 一种用于判断靶向单克隆抗体治疗肿瘤疗效的预测分子的制造方法与工艺
- 一种检测肺癌相关基因热点突变的试剂盒及其应用的制造方法与工艺
- 一种为肺癌提供离体个体化药物测试的方法及培养基与流程
- 一种谷胱甘肽/超声双重刺激响应纳米聚合物囊泡、其制备方法及应用与流程
- 一种多重刺激响应交联型聚合物纳米水凝胶、其制备方法及应用与流程
- 一种靶向CD47的免疫检查点抑制剂药物组合物及其制备方法与流程
- 具有淋巴靶向功能的生物自组装纳米晶注射剂及制备方法与流程
- 用于检测肝癌的基因标志物及其用途的制造方法与工艺
- 特异性shRNA筛选及其靶向Ang‑2基因抑制肺癌细胞的验证方法与流程
- 一种具有靶向功能仿病毒结构高分子囊泡及其制备与应用的制造方法与工艺
- 肿瘤靶向核磁共振/荧光双模态成像造影剂及其制备和应用的制造方法与工艺
- pH氧化还原双敏感的PAMAM靶向纳米给药载体及其制备方法与制造工艺
- 具有卵巢肿瘤靶向D构型多肽及其基因递释系统的制造方法与工艺
- 靶向survivin纳米抗体及其制备方法和应用与制造工艺
- 用于PSMA靶向的成像和放射疗法的金属/放射性金属标记的PSMA抑制物的制造方法与工艺
- 一种线粒体靶向的粘度荧光探针及其制备方法和应用与制造工艺
- 一种兼具荧光可视、磁靶向和pH敏多功能的复合空心微球药物载体及其制备方法与制造工艺
- 一种用于修饰微泡的多肽以及靶向GBM的药物制剂的制造方法与工艺
- 白果内酯作为脑靶向增效剂在制备防治脑肿瘤药物的应用的制造方法与工艺
- 机器人辅助精确靶向穿刺软组织目标柔性针夹紧固定装置的制造方法