多发性骨髓瘤预后相关基因突变检测试剂盒及检测方法与流程
本发明属于医学及分子生物学技术领域,特别涉及一种多发性骨髓瘤预后相关基因突变检测试剂盒。本发明还提供了一种使用该试剂盒检测多发性骨髓瘤预后相关基因突变的方法。
背景技术:
多发性骨髓瘤(Multiple Myeloma,MM)是一种恶性浆细胞病,其肿瘤细胞起源于骨髓中的浆细胞,而浆细胞是B淋巴细胞发育到最终功能阶段的细胞,因此多发性骨髓瘤可以归到B淋巴细胞淋巴瘤的范围。
传统的MM常规检测内容包括筛查试验、诊断试验、评估肿瘤负荷和预后试验,主要通过血液中异常蛋白类型及浓度等的检测进行诊断,如评估肿瘤负荷和预后试验通常包括:1、骨髓涂片荧光原位杂交分析;2、血清β2微球蛋白浓度;3、血清白蛋白浓度;4、血清和尿中的单克隆蛋白定量。因MM临床表现多种多样,易与其它疾病混淆,传统的诊断方法所依据的检测手段误检漏检率较高,且检测过程繁琐。
随着基因研究的不断进展,对MM的发病机制及诊断治疗有了基因层面的深入了解。MM发病是由于B淋巴细胞分化为浆细胞过程中遗传基因发生突变,约半数病例发生染色体易位,即癌基因易位到14号染色体的免疫球蛋白重链基因(IgH基因易位),导致癌基因过表达和细胞增殖失控;其它病理特征是细胞具有部分奇数三倍体,即染色体3、5、7、9、11、15、19和21。这些众多三体的表现称为超二倍性。目前WHO将其归为B细胞淋巴瘤的一种,称为浆细胞骨髓瘤/浆细胞瘤,其特征为骨髓浆细胞异常增生伴有单克隆免疫球蛋白或轻链(M蛋白)过度生成,极少数患者可以是不产生M蛋白的未分泌型MM,MM占所有癌症比例1%,在血液系肿瘤中占10%。
研究发现MM患者最常见的基因突变发生在RAS基因家族,其中一种是在美国癌症研究所(Institute of Cancer Research,ICR)中发现的,而且在很多种癌症中可检测。发表在Journal of Clinicaloncolog杂志上的一项研究表明,九种基因特征需要被检测来鉴定能从强化治疗中获益的高危患者。这项研究由伦敦肿瘤研究协会研究者领导,第一次把骨髓瘤细胞基因突变与疾病生存期联系起来。研究者发现,43%的患者发生NRAS和KRAS基因突变,表明骨髓瘤RAS基因突变能促使细胞癌变。这项发现可能会为治疗开辟新大道,研究中超过50%的基因突变能成为研究其它类型癌症新药或现存药的靶点。因为骨髓瘤细胞增长和存活要依赖骨髓中其它的细胞,比如成纤维细胞、成骨细胞、破骨细胞、基质细胞和树突状细胞,故以骨髓微环境为靶标的治疗方法有所进展,既往MM的生存期平均3-5年,近些年由于新药的应用,其平均生存期已经延长为5-7年,不少患者可生存10年以上。
鉴于上述类型基因在MM患者及正常组织中的表达特点,其突变情况可以作为重要的MM辅助诊断以及预后评估的标志,通过检测获得的预后相关基因突变数据可以为进一步完成MM预后评估提供分析基础,因此,如何快速全面的获得检测对象MM预后相关基因的突变数据,成为需要解决的技术问题。
技术实现要素:
本发明针对当前MM预后评估相关检测误检漏检率高且过程繁琐的问题,提供了新的MM预后相关基因突变检测技术,综合应用PCR扩增和基因测序技术,从MM高突变基因中选择二十二个基因,开发出了针对MM特异性的一组预后评估的相关基因突变检测试剂盒及检测方法,可快速全面的获得检测标本MM预后相关基因突变的数据。
为解决上述技术问题,本发明的实施例提供一种多发性骨髓瘤预后相关基因突变检测试剂盒,包含分别与各检测基因对应的多对特异性引物,每对所述特异性引物均包括一条上游引物和一条下游引物,用于对各所述检测基因的热点突变区段分别进行PCR扩增;
所述检测基因包括九个核心检测基因:KRAS、NRAS、TP53、BRAF、DIS3、FAM46C、RB1、FAT4和ATM。
优选的,所述特异性引物在PCR扩增时对应的各所述核心检测基因的所述热点突变区段分别为:KRAS基因的第2、3和4号外显子;NRAS基因的第2和3号外显子;TP53基因的第5、6、7和8号外显子;BRAF基因的第11和15号外显子;DIS3基因的第5、8、9、17、18、20和21号外显子;FAM46C基因的第2号外显子;RB1基因的第8和19号外显子;FAT4基因的第1和16号外显子;ATM基因的第10、41和62号外显子。
作为所述试剂盒的优选,所述检测基因还包括十三个辅助检测基因:PIK3CA、ZFHX4、VCAN、PRDM1、CCND1、TRAF3、SPEN、ANK2、EGR1、FGFR3、NFKB2、NEB和SP140。
进一步优选的,所述特异性引物在PCR扩增时对应的各所述辅助检测基因的所述热点突变区段分别为:PIK3CA基因的第9号外显子;ZFHX4基因的第11号外显子;VCAN基因的第7号外显子;PRDM1基因的第6号外显子;CCND1基因的第1号外显子;TRAF3基因的第11号外显子;SPEN基因的第11号外显子;ANK2基因的第38号外显子;EGR1基因的第1号外显子;FGFR3基因的第14号外显子;NFKB2基因的第17号外显子;NEB基因的第78号外显子;SP140基因的第2、19和27号外显子。
本发明实施例还提供了一种多发性骨髓瘤预后相关基因突变检测方法,使用了前述技术方案中的所述试剂盒进行检测,包括步骤为:
S1.提取待测样本基因组DNA:采集血液,筛选出阳性淋巴瘤细胞,提取基因组DNA;
S2.进行PCR扩增:使用所述试剂盒中的特异性引物对其对应的所述检测基因的区段分别进行PCR扩增进行PCR扩增,回收扩增产物;
S3.扩增产物测序:对S2中获得的所述扩增产物进行测序,得到测序结果;
S4.基因突变比对:将所述测序结果与数据库中公开的对应基因序列进行比对,获取基因突变检测结果。
优选的,步骤S1中,使用试剂盒柱提法采集浓度为50~200ng/μL的DNA溶液50μL。
优选的,步骤S1中,使用所述试剂盒中的特异性引物配制引物浓度为5pmol/μL的引物使用液用于PCR扩增。
优选的,执行所述步骤S3前对所述扩增产物进行电泳检测。
优选的,所述步骤S3中使用3500XL测序仪对所述扩增产物进行上机测序。
优选的,所述步骤S4中使用Mutation Surveyor软件和VECTOR NTI软件将所述测序结果与NCBI数据库中序列进行比对分析,寻找发生突变的基因位点,获得基因突变检测结果。
本发明技术方案综合应用PCR和测序两项技术,对MM预后相关基因的突变情况实现快速检测,选择包含热点突变位点的KRAS、NRAS、TP53、BRAF、DIS3、FAM46C、RB1、FAT4、ATM九个基因作为核心检测基因,再选取包含热点突变位点的PIK3CA、ZFHX4、VCAN、PRDM1、CCND1、TRAF3、SPEN、ANK2、EGR1、FGFR3、NFKB2、NEB、SP140十三个基因作为辅助检测基因,通过相应的特异性引物扩增前述二十二个检测基因中包含热点突变的特定外显子区段,通过测序反应对PCR扩增所得到的目的片段进行测序,再与NCBI(美国国家生物技术信息中心)上所公布的基因序列进行比对获得突变结果。本发明的上述技术方案的有益效果如下:
1.依据MM的发病机制以及相关的信号通路,全面的选择相关基因,适用性强,适用于多种核型异常的MM检测;
2.检测特异性强,检测过程简单,准确性高,且更加快速灵敏,检测结果明确清晰,目的性强;
3.检测标本为检测对象的骨髓血且用量少,利于检测对象自身,使用的实验器材较少,节约成本;
4.检测试剂使用低毒的荧光染料,无需放射性标记,安全性好。
附图说明
图1为本发明MM预后基因突变检测方法的基本流程图;
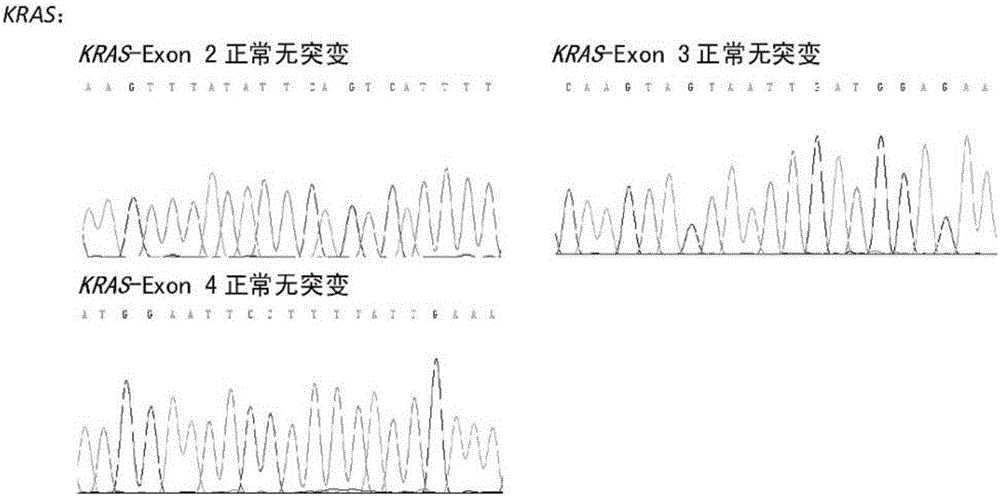
图2为本发明实施例中核心检测基因KRAS的突变分析结果;
图3为本发明实施例中核心检测基因NRAS的突变分析结果;
图4为本发明实施例中核心检测基因TP53的突变分析结果;
图5为本发明实施例中核心检测基因BRAF的突变分析结果;
图6为本发明实施例中核心检测基因DIS3的突变分析结果;
图7为本发明实施例中核心检测基因FAM46C的突变分析结果;
图8为本发明实施例中核心检测基因RB1的突变分析结果;
图9为本发明实施例中核心检测基因FAT4的突变分析结果;
图10为本发明实施例中核心检测基因ATM的突变分析结果;
图11为本发明实施例中辅助检测基因PIK3CA的突变分析结果;
图12为本发明实施例中辅助检测基因ZFHX4的突变分析结果;
图13为本发明实施例中辅助检测基因VCAN的突变分析结果;
图14为本发明实施例中辅助检测基因PRDM1的突变分析结果;
图15为本发明实施例中辅助检测基因CCND1的突变分析结果;
图16为本发明实施例中辅助检测基因TRAF3的突变分析结果;
图17为本发明实施例中辅助检测基因SPEN的突变分析结果;
图18为本发明实施例中辅助检测基因ANK2的突变分析结果;
图19为本发明实施例中辅助检测基因EGR1的突变分析结果;
图20本发明实施例中辅助检测基因FGFR3的突变分析结果;
图21为本发明实施例中辅助检测基因NFKB2的突变分析结果;
图22为本发明实施例中辅助检测基因NEB的突变分析结果;
图23为本发明实施例中辅助检测基因SP140的突变分析结果。
具体实施方式
为使本发明要解决的技术问题、技术方案和优点更加清楚,下面将结合附图及具体实施例进行详细描述。
本发明针对当前MM预后评估相关检测误检漏检率高且过程繁琐的问题,提供了新的MM预后相关基因突变检测技术,可快速全面的获得检测对象MM预后相关基因突变的数据。
本实施例提供的一种多发性骨髓瘤预后相关基因突变检测试剂盒,包含分别与各检测基因对应的多对特异性引物,每对所述特异性引物均包括一条上游引物和一条下游引物,用于对各检测基因的热点突变区段分别进行PCR扩增;
本实施例所述检测基因包括九个核心检测基因:KRAS、NRAS、TP53、BRAF、DIS3、FAM46C、RB1、FAT4和ATM,其基因序列来源为NCBI基因库,具体标识见下表1:
上表中,各基因功能如下:
MM患者最常见的基因突变在是RAS基因家族,其中一种是在ICR中发现的,而且在很多种癌症中可检测。研究者发现43%的患者发生NRAS和KRAS突变——表明骨髓瘤RAS突变能促使细胞癌变。NRAS、BRAF以及KRAS基因属于致癌基因,这些基因突变通过激活MAPK/ERK激酶信号通路发挥功能。NRAS,KRAS基因更加倾向于在复发的病人中发生突变;
TP53基因突变与染色体的结构异常、差的预后相关;
BRAF基因突变的患者比BRAF野生型对于BRAF抑制剂PLX4720更加敏感,在BRAF突变的MM患者中,BRAF抑制下调MAP激酶信号通路的活性;
DIS3基因编码的蛋白是一个肿瘤抑制因子,高度保守的核糖核酸外切酶,它的主要功能是在于细胞循环以及微管组装。基因的功能是肿瘤抑制基因,基因突变限制了该蛋白的功能,导致细胞增殖率变低,预后效果不好。该基因的突变与FAM46C基因突变想排斥,而伴随着该基因的突变发生的往往存在RB1基因的缺失;
FAM46C基因被认为是肿瘤抑制因子,与预后相关,主要是通过点突变以及纯合缺失来实现。该基因的异常表达与预后生存期相关,可以被认为疾病低风险值的评估标准;
RB1基因在MM中属于致病基因;
FAT4基因隶属于FAT家族,是一个肿瘤抑制因子,与HIPPO信号通路相关,该基因突变导致组织的过度生长;
ATM基因突变与差的预后相关。
作为一种优选的实施方式,所述检测基因还可包括十三个辅助检测基因:PIK3CA、ZFHX4、VCAN、PRDM1、CCND1、TRAF3、SPEN、ANK2、EGR1、FGFR3、NFKB2、NEB和SP140,其基因序列来源同样为NCBI基因库,具体标识见下表2:
上表中,各基因功能如下:
PIK3CA基因突变往往伴随着KRAS突变,特别是G12C位点突变,PI3K信号通路至关重要,因为它可以调节下游的信号通路,从而促进细胞的增值、生长以及代谢;
ZFHX4基因属于DNA损伤修复信号通路,其突变与差的预后相关;
VCAN基因蛋白属于ggrecan/versican蛋白多糖家族,与组织形态发生和维护相关;
PRDM1基因是浆细胞分化过程中的转录因子;
CCND1基因属于细胞循环因子,在MM中是染色体易位的靶点,该基因突变与差的预后相关;
TRAF3基因蛋白属于NF-kB信号通路的一员,NF-kB信号通路在20%的MM病人中出现,与MM的发病原因相关,该信号通路的激活是通过是个基因的突变激活的,其中TRAF3是其中发生率最高的基因;
SPEN基因与B淋巴细胞发育相关;
ANK2基因参与细胞活性、活化、增殖、细胞与细胞间的相互作用和维护特殊的结构域;
EGR1基因通过JUN信号通路诱导骨髓瘤细胞的生长,该基因的突变与好的预后相关;
FGFR3基因属于致病基因,在MM中大量表达;
NFKB2基因蛋白参与了NF-kB信号通路的组成型激活;
NEB基因蛋白与细胞骨架构建相关;
SP140基因编码的蛋白是一个肿瘤抑制因子。
上述实施方式中共计二十二个检测基因中的热点突变区段见下表3:
作为一种实施方式,本发明试剂盒中所包含的特异性引物的序列具体见下表4:
在满足PCR引物的设计要求的前提下,特异性引物可根据引物设计原理,设计为不同的序列。
本发明另一实施例提供了多发性骨髓瘤预后相关基因突变检测方法,所述方法使用了前述实施例中的多发性骨髓瘤预后相关基因突变检测试剂盒,如图1所示,包括以下步骤:
S1.提取待测样本基因组DNA:采集血液,筛选出阳性淋巴瘤细胞,提取基因组DNA;
S2.进行PCR扩增:使用所述试剂盒中的特异性引物对其对应的所述检测基因的区段分别进行PCR扩增进行PCR扩增,回收扩增产物;所述检测基因根据试剂盒中的特异性引物作出不同选择,可包括前述九个核心检测基因,也可进一步包括前述十三个辅助检测基因;
S3.扩增产物测序:对S2中获得的所述扩增产物进行测序,得到测序结果;
S4.基因突变比对:将所述测序结果与数据库中公开的对应基因序列进行比对,获取基因突变检测结果。
具体的实施方式如下:
步骤S1中,采用临床标本确诊的MM患者的骨髓样品,提取样品的基因组DNA;提取基因组DNA可使用常规的试剂盒柱提法,按照所用试剂盒的说明书进行操作,收集到50μL浓度为50~200ng/μL的DNA溶液,可直接用于检测或在-20℃下保存;
步骤S2中,使用所述试剂盒中的特异性引物配制溶液,其中特异性引物浓度为5pmol/μL;
步骤S3中,进行PCR扩增的扩增反应体系见下表5:
使用的特异性引物序列参见上表4,PCR扩增可使用普通PCR仪,扩增反应程序见下表6:
为保证PCR扩增结果,可在测序前先对PCR扩增产物进行电泳检测,空白对照为无扩增条带,如样本条带大小正确(参照上表4中记载的扩增产物大小)且条带单一,即可将PCR扩增产物送检测序;
步骤S3中,使用3500XL测序仪,依常规操作方法,对PCR扩增产物进行上机测序;
步骤S4中,使用Mutation Surveyor软件和VECTOR NTI软件对PCR扩增产物测序所得待测序列与NCBI数据库中序列进行比对分析,寻找基因突变位点,获得检测结果。
如图2至图23所示为采用本实施例检测方法对一病例进行检测得到的前述二十二个MM预后相关基因突变的分析谱图,根据图示结果,检测到MM预后相关的FAT4基因中存在一个突变,ATM基因存在三个突变,其它基因未见突变,其中:FAT4基因第1号外显子错义突变是单核苷酸多态性位点;ATM基因第41号外显子周围内含子缺失突变病理意义不明,第62号外显子周围两个内含子的突变也属于单核苷酸多态性位点。
通过对上述基因突变检测结果的进一步分析,可以辅助进行MM患者的诊断,并对其预后进行评估。
应理解,上述实施例只为说明本发明的技术构思及特点,其目的在于供本领域技术人员了解本发明的内容并据以实施,并非具体实施方式的穷举,并不能以此限制本发明的保护范围。凡根据本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的宗旨和范围,其均应涵盖在本发明的权利要求范围当中。
SEQUENCE LISTING
<110>北京海思特临床检验所有限公司
<120>多发性骨髓瘤预后相关基因突变检测试剂盒及检测方法
<160>82
<170> PatentIn version 3.5
<210>1
<211>20
<212> DNA
<213>人工序列
<400> 1
ggcctgctga aaatgactga 20
<210>2
<211>20
<212> DNA
<213>人工序列
<400>2
gtcctgcacc agtaatatgc 20
<210>3
<211> 19
<212> DNA
<213>人工序列
<400>3
tcatctttgg agcaggaac 19
<210>4
<211>20
<212> DNA
<213>人工序列
<400>4
Tggcattagc aaagactcaa 20
<210>5
<211>21
<212> DNA
<213>人工序列
<400>5
Tgaagatgta cctatggtcc t 21
<210>6
<211> 19
<212> DNA
<213>人工序列
<400>6
accaaagcca aaagcagta 19
<210>7
<211>20
<212> DNA
<213>人工序列
<400>7
ggccgatatt aatccggtgt 20
<210>8
<211>20
<212> DNA
<213>人工序列
<400>8
tccgacaagt gagagacagg 20
<210>9
<211>18
<212> DNA
<213>人工序列
<400>9
ccccttaccc tccacacc 18
<210> 10
<211>21
<212> DNA
<213>人工序列
<400> 10
tggtaacctc atttccccata 21
<210> 11
<211>20
<212> DNA
<213>人工序列
<400> 11
gactttcaac tctgtctcct 20
<210> 12
<211>20
<212> DNA
<213>人工序列
<400> 12
atcagtgagg aatcagaggc 20
<210> 13
<211>20
<212> DNA
<213>人工序列
<400> 13
gcctctgatt cctcactgat 20
<210> 14
<211>20
<212> DNA
<213>人工序列
<400> 14
cactgacaac cacccttaac 20
<210> 15
<211>20
<212> DNA
<213>人工序列
<400> 15
cctcatcttg ggcctgtgtt 20
<210> 16
<211>20
<212> DNA
<213>人工序列
<400> 16
cagtgtgcag ggtggcaagt 20
<210> 17
<211>20
<212> DNA
<213>人工序列
<400> 17
ggacctgatt tccttactgc 20
<210> 18
<211> 20
<212> DNA
<213>人工序列
<400> 18
gcttcttgtc ctgcttgctt 20
<210> 19
<211>23
<212> DNA
<213>人工序列
<400> 19
ggatctcttc ctgtatccct ctc 23
<210>20
<211> 19
<212> DNA
<213>人工序列
<400>20
atcagccagc ctcagcttc 19
<210>21
<211>24
<212> DNA
<213>人工序列
<400>21
gttttccttt acttactaca cctc 24
<210>22
<211>22
<212> DNA
<213>人工序列
<400>22
aaatagcctc aattcttacc at 22
<210>23
<211>18
<212> DNA
<213>人工序列
<400>23
gcggcatctt ttggtttg 18
<210>24
<211>19
<212> DNA
<213>人工序列
<400>24
ttcattgtcg ccatgaatc 19
<210>25
<211>18
<212> DNA
<213>人工序列
<400>25
cctttctttt gcctccat 18
<210>26
<211>21
<212> DNA
<213>人工序列
<400>26
tgctacttac caggtcactt t 21
<210>27
<211>22
<212> DNA
<213>人工序列
<400>27
tttagaatgt aagagctgga cc 22
<210>28
<211>21
<212> DNA
<213>人工序列
<400>28
tttggcagaa aactaagaac a 21
<210>29
<211>21
<212> DNA
<213>人工序列
<400>29
cgtttctaat aatgcctctt g 21
<210>30
<211>21
<212> DNA
<213>人工序列
<400>30
ttaccttttc agtaatgctc c 21
<210>31
<211>20
<212> DNA
<213>人工序列
<400>31
aatatggtgc tcttccctcc 20
<210>32
<211>19
<212> DNA
<213>人工序列
<400>32
tgggtatgaa aagccactg 19
<210>33
<211>21
<212> DNA
<213>人工序列
<400>33
cctcaaatgt aaggaagcaa t 21
<210>34
<211>21
<212> DNA
<213>人工序列
<400>34
gagaaaacag gatttgctat g 21
<210>35
<211> 18
<212> DNA
<213>人工序列
<400>35
tatggtgctc ttccctcc 18
<210>36
<211> 18
<212> DNA
<213>人工序列
<400>36
attgagccat tttgtgcc 18
<210>37
<211>19
<212> DNA
<213>人工序列
<400>37
gggctgcaaa gacctggac 19
<210>38
<211>22
<212> DNA
<213>人工序列
<400>38
agcgaagtgg ttttgaaggt aa 22
<210>39
<211> 18
<212> DNA
<213>人工序列
<400>39
ggttcacctc gaacaccc 18
<210>40
<211>19
<212> DNA
<213>人工序列
<400>40
gattccagag tgagggagc 19
<210>41
<211>21
<212> DNA
<213>人工序列
<400>41
tctaataagg cagtaatccc c 21
<210>42
<211>20
<212> DNA
<213>人工序列
<400>42
ctcgcaacat tatcatttcc 20
<210>43
<211> 19
<212> DNA
<213>人工序列
<400>43
ggggacaggt ctcggtttc 19
<210>44
<211>22
<212> DNA
<213>人工序列
<400>44
gatagcaaac atacctttct ga 22
<210>45
<211>20
<212> DNA
<213>人工序列
<400>45
ttctatctgc tgtggactgg 20
<210>46
<211> 19
<212> DNA
<213>人工序列
<400>46
tttacctgtt tgggcatct 19
<210>47
<211>22
<212> DNA
<213>人工序列
<400>47
tcctgccaat ttaggaagta gg 22
<210>48
<211>22
<212> DNA
<213>人工序列
<400>48
gcaggctgac ccagtaaata ac 22
<210>49
<211>20
<212> DNA
<213>人工序列
<400>49
gtttgccacc ttcattagtt 20
<210>50
<211>20
<212> DNA
<213>人工序列
<400>50
gaaatgaaca gggtgctatg 20
<210>51
<211>21
<212> DNA
<213>人工序列
<400>51
cgtctaatga aagcccactct 21
<210>52
<211>21
<212> DNA
<213>人工序列
<400>52
gggctgaagg catttctaaa c 21
<210>53
<211>20
<212> DNA
<213>人工序列
<400>53
tggcaagggt aaacagatcc 20
<210>54
<211>20
<212> DNA
<213>人工序列
<400>54
ctcaggcact cctctcaacc 20
<210>55
<211>20
<212> DNA
<213>人工序列
<400>55
agttcaggca aagacatcca 20
<210>56
<211>22
<212> DNA
<213>人工序列
<400>56
cggcgtcttc atcagtaaac at 22
<210>57
<211>20
<212> DNA
<213>人工序列
<400>57
atagtagcac ccaagagcct 20
<210>58
<211>18
<212> DNA
<213>人工序列
<400>58
atccatcgcc ctcttgtt 18
<210>59
<211>21
<212> DNA
<213>人工序列
<400>59
tcttggagca gaaatgttag g 21
<210>60
<211> 18
<212> DNA
<213>人工序列
<400>60
aggatgggac aacgcaga 18
<210>61
<211> 19
<212> DNA
<213>人工序列
<400>61
acaggggagt tttgttgaa 19
<210>62
<211> 18
<212> DNA
<213>人工序列
<400>62
agaaacacca cggcaaac 18
<210>63
<211>20
<212> DNA
<213>人工序列
<400>63
ttacagccag cctttctaca 20
<210>64
<211> 18
<212> DNA
<213>人工序列
<400>64
tctccagtgg gcttcttg 18
<210>65
<211> 18
<212> DNA
<213>人工序列
<400>65
gccccaggca cagtcaat 18
<210>66
<211> 19
<212> DNA
<213>人工序列
<400>66
tgacctggga cttggacac 19
<210>67
<211>20
<212> DNA
<213>人工序列
<400>67
agcagaatct ccacaagcag 20
<210>68
<211> 19
<212> DNA
<213>人工序列
<400>68
aaagattggg aagggacat 19
<210>69
<211> 19
<212> DNA
<213>人工序列
<400>69
gcacgcttct cagtgttcc 19
<210>70
<211> 19
<212> DNA
<213>人工序列
<400>70
tccttggcat ccctctaac 19
<210>71
<211> 18
<212> DNA
<213>人工序列
<400>71
ccgcaatgtg ctggtgac 18
<210>72
<211>18
<212> DNA
<213>人工序列
<400>72
ggaaggtgag gtcaggct 18
<210>73
<211>20
<212> DNA
<213>人工序列
<400>73
tccccatctc acctgactaa 20
<210>74
<211>20
<212> DNA
<213>人工序列
<400>74
ctgagggtga gactgaccag 20
<210>75
<211>19
<212> DNA
<213>人工序列
<400>75
agaggcatag gatgggttc 19
<210>76
<211>21
<212> DNA
<213>人工序列
<400>76
ggaaggtatg attctggcaa c 21
<210>77
<211>19
<212> DNA
<213>人工序列
<400>77
gtttcttagg atggtcgca 19
<210>78
<211>21
<212> DNA
<213>人工序列
<400>78
ctcctgtcac ctcctaacct a 21
<210>79
<211>22
<212> DNA
<213>人工序列
<400>79
attctcagtt gaaaggtgtc tc 22
<210>80
<211>20
<212> DNA
<213>人工序列
<400>80
tgatgagata cttggggttg 20
<210>81
<211>20
<212> DNA
<213>人工序列
<400>81
tttcctaagt atccctcgtg 20
<210>82
<211>22
<212> DNA
<213>人工序列
<400>82
tacttgtgat gccttcagat ag 22
- 还没有人留言评论。精彩留言会获得点赞!