一种大豆矮杆性状紧密连锁的SNP标记及其应用的制作方法
本发明属于基因工程技术领域,具体涉及一种大豆矮杆性状紧密连锁的SNP标记及其应用。
背景技术:
作物矮杆性状的应用实现了第一次绿色革命,是高产育种的第一个里程碑。自1967年,大豆矮秆性状应用于育种后,世界范围内不断创造大豆高产纪录(Cooper,1981,Cooper,1991),因此矮杆性状已成为大豆育种的一个重要研究内容。然而在大豆育种中,虽然植株高矮很容易肉眼鉴别,但由于水、肥、种植密度和生育期等因素影响比较严重,尤其在育种早世代单株选择时,矮杆性状的鉴定不够方便和准确。
随着生物技术的发展,分子标记技术应用于作物育种,加快了作物育种的效率。分子标记技术能够对目标性状的基因型进行鉴定,不受环境,生育时期等很多限制。大豆矮杆性状属于简单质量遗传性状,是由一对或两对隐形主效基因控制的(kilen,1975;Boerma),有的还伴若干修饰基因控制(何平,陈恒鹤,W.R.Feh)。因此,利用矮杆性状连锁的分子标记进行矮杆性状的鉴定是可行的。
目前,关于矮杆基因的遗传定位研究,目前利用矮杆或半矮秆材料进行了较多株高性状QTL定位。统计大豆数据库发现,在A1、B1、C1、C2、D1a、D1b、E、F、G、H、I、J、L、M、N和O等16条染色体上大约有31个QTL,其中在B1(47Mb-72Mb),C2(106Mb-131Mb),F(1Mb-22.5Mb,60Mb-85Mb),J(11Mb-28Mb),L(8Mb-15Mb),65Mb-93Mb),M(17Mb-41Mb)位点,在不同遗传群体里,发现了共性的QTL。但由于目前针对矮杆质量性状进行的基因定位研究还未见报道,因此,我们对大豆矮杆的分子遗传和机制还不了解。
现有大豆矮杆性状的鉴定技术或方法是在大豆开花后期,株高稳定了,通过肉眼去鉴定是否为矮杆材料;鉴定受时间和生育时期的限制。另外,由于株高受栽培密度、机械损伤,田间降水等环境条件的影响,仅仅调查一株不能准确判断是高还是矮,必须群体调查准确;在矮杆育种中,由于矮杆是隐性基因控制的,仅占分离群体的1/2[1-(1/2)r],如在育种早世代进行选育,育种群体中表现为矮杆材料较少,因此,育种规模要比较大,才能够筛选出优良的矮杆育种材料,育种成本高,育种效率低。
技术实现要素:
为了解决上述现有技术中存在的问题,本发明提供了一种大豆矮杆性状紧密连锁的SNP标记,及其该标记用于筛选矮杆性状大豆材料的应用和方法。
本发明主要通过以下技术方案实现:
一种大豆矮杆性状紧密连锁的SNP标记,根据本发明的实施例,所述的SNP标记包含:第一SNP标记GM19_45000827和第二SNP标记GM19_45361938,所述的第一SNP标记GM19_45000827为大豆基因组v1.0的第19号染色体45000827位置的碱基为C或A;所述的第二SNP标记GM19_45361938为大豆基因组v1.0的第19号染色体44902890位置的碱基为G或A。
本发明所述的GM19_45000827处碱基是C的大豆为矮杆,碱基是A的大豆为高杆;所述的GM19_45361938处碱基是G的大豆为矮杆,碱基是A的大豆为高杆。
本发明的另一个目的,本发明还提供了一种用于检测前面所述的SNP标记的KASP引物对,具体的所述的引物对包括:
GM19_45000827的正向引物1:ACATGCCTACCAACAAAAG,正向引物2:CGGTGTAGTTCGGGAAACAA,反向引物:CGGTGTAGTTCGGGAAACAC;
GM19_45361938的正向引物1:AGATTCCCAGGCGCAACTTG,正向引物2:AGATTCCCAGGCGCAACTTA,反向引物:TTTGTAGCTTCTGTTTATAG。
利用本发明的引物对能够有效地对待测大豆的上述与大豆矮杆性状相关的SNP标记所在的片段进行PCR扩增,进而通过检测PCR产物的荧光信号,确定待测大豆该种SNP标记位点的基因型,进而能够有效预测待测大豆矮杆性状。
本发明的另一个目的,本发明还提供了用于检测前面所述的SNP标记的试剂盒,该试剂盒包含前面所述的用于检测本发明的SNP标记的引物对,即本发明试剂盒包含具有SEQ ID NO:1-6所示的核苷酸序列的引物对。
本发明的另一个目的,本发明还提供了前面所述的本发明的SN P标记、引物对或试剂盒在大豆矮杆性状分子标记辅助育种中的应用。
本发明的另一个目的,本发明还提供了一种筛选具有矮杆性状的大豆材料的方法,其步骤如下:
1)提取待检测大豆基因组DNA;
2)检测:以待检测大豆基因组DNA为模板,利用前面所述的引物进行PCR扩增反应;
3)结果判断:利用LightCycler 480软件(v1.5)的Endpoint分析模块检测PCR扩增产物,根据信号表现区分基因型。其中,绿信号与公交9112基因型一致,是高杆基因型。蓝信号与吉密豆1号基因型一致,是矮杆基因型;红信号为杂合基因型,下代高矮杆分离;粉色和其它为基因型不确定或样品缺失。
其中,第一标记GM19_45000827的检测矮杆准确率为96.77%,第二标记GM19_45361938检测矮杆准确率为93.48%。
本发明的有益效果:
1)简化基因组法遗传定位到2个与大豆矮杆性状连锁最为紧密的SNP标记,利用这两个SNP标记结合KASP PCR技术,能够从基因型上鉴定矮秆性状,可以在大豆不同生长时期,不受环境,群体大小和生育时期等条件的限制,能够快熟、准确、规模化地鉴定大豆植株矮杆性状。
2)在大豆矮秆育种中,解决了矮杆性状仅仅在开花后期才能调查和单一植株受环境影响性状调查不准确必须群体调查的问题;而且在矮杆育种后代材料选育中,在种子期就能够鉴定出隐性基因控制的矮杆基因的材料。
3)在达到相同育种效果下,大大减少了田间育种面积,进而减少田间试验材料和劳务成本。
附图说明
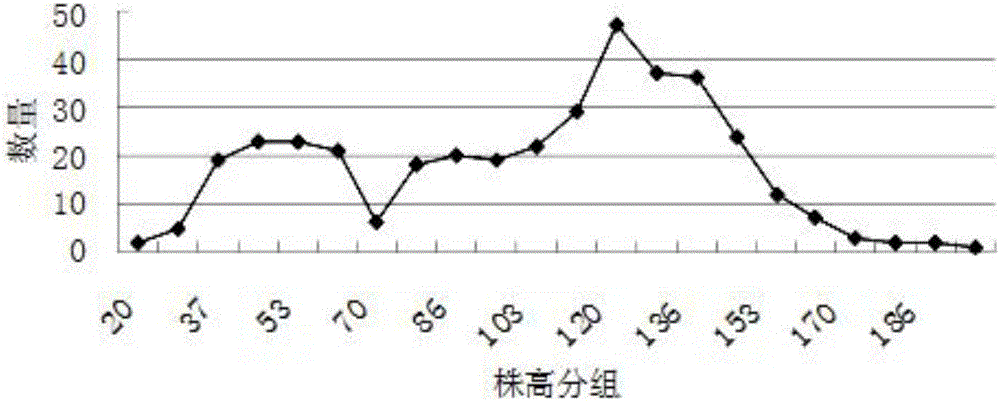
图1是本申请密豆1号×公交9112组合F2群体内377个个体株高分布频率曲线;
图2是本申请大豆20个染色体上个SNP的Δ(SNP_index)拟合曲线;
图3是本申请11个SNP在密豆1号和公交9112间的多态性检测结果;其中,a为公交9112,b为吉密豆1号,1-12分别GM19_44412186,GM19_44546232,GM19_44595030,GM19_44622364,GM19_44779426,GM19_44815533,GM19_44902890,GM19_45000827,GM19_45361938,GM19_45423153,GM19_4549471611个SNP引物;
图4是8个稳定多态关联SNP标记所构建的遗传连锁图谱;Dwarf为矮杆基因;
图5是本发明实施例紧密连锁标记GM19_45000827对吉密豆1号和公交9112杂交后代KASP SNP PCR产物的检测;
图6是本发明实施例紧密连锁标记GM19_45361938对吉密豆1号和公交9112杂交后代KASP SNP PCR产物的检测。
具体实施方式
以下将配合附图及实施例来详细说明本申请的实施方式,藉此对本申请如何应用技术手段来解决技术问题并达成技术功效的实现过程能充分理解并据以实施。
实施例1与大豆矮杆性状紧密连锁的SNP标记的获得
1、遗传定位矮杆性状
(1)利用“吉密豆1号”和“公交9112”杂交获得的377个F2单株DNA及其F2:3株高表现进行大豆矮杆性状的遗传定位。
(2)调查F2:3遗传群体各个株系的植株高度,55垄距,15厘米株距,2两粒播种的栽培密度条件下,大于70的定义为非矮杆,小于等于70的定义为矮杆。利用X2测验分析F2:3株系株高分离是否符合3:1(图1),如果符合3:1,则矮杆性状为单基因控制的,视为质量性状。
(3)CTAB法提取其F2单株DNA,无菌水溶解DNA,浓度要大于50ng/ul,OD260/280=1.8-2.1;利用0.1%琼脂糖凝胶检测DNA,条带完整。
(4)选择其F2:3株高表现极端高的40F2单株和株高极端矮的40F2单株。等量混合40个F2单株DNA,分别构建矮杆基因池和非矮杆基因池。
(5)简化基因组BSA法构建吉密豆1号、公交9112、矮杆基因池和非矮杆基因池文库(Sun et al,2013)并测序。
(6)Δ(SNP_index)拟合曲线关联分析法定位矮杆性状(Hill,2013),并确定Δ(SNP_index)拟合回归阈值为0.8704。在基因组区间内,有3个或三个以上大于阈值且连续分布的SNP,这样的基因组区间,即为与矮杆性状关联的区域。研究发现这样的区间仅有一个(图2),位于williams 82大豆基因组(1.0版本)19号染色体44372629bp-45495014bp位置区间,区间物理距离为1.12Mb。这个区域有18个连续大于阈值的SNP标签(表1),区域平均拟合回归值为0.8796。
表1矮杆性状关联区域18个连续大于阈值的SNP拟合值
实施例2矮杆性状连锁的SNP标记开发
(1)针对其中均匀分布的12个标签(Marker1098514,Marker1168024,Marker1143721,Marker1110430,Marker1120990,Marker1086045,Marker1118571,Marker11080703,Marker1093726,Marker1102167,Marker1094495,Marker1061721)中的12个SNP位点(44412186,44546232,44595030,44622364,44779426,44815533,44902890,4,5000827,45361938,45423153,45494716),利用KASP技术(LGC生物公司)设计并合成11个SNP引物,具体引物对序列如下:
GM19_44412186引物对序列:
F1:TACTTTATTTCGTTAGGTCT
F2:TACTTTATTTCGTTAGGTCC
R:TTACCATAACATACAAAGAC
GM19_44546232引物对序列:
F1:ATGGAAACAATTTAAATGA
F2:ATGGAAACAATTTAAATGT
R:ATTGTGACTAATTAATCGTA
GM19_44595030引物对序列:
F1:TTTAACTCATTAAGGTTTAT
F2:TTTAACTCATTAAGGTTTAG
R:CATTAGCAAACAAAAAAGGA
GM19_44622364引物对序列:
F1:TTCGCTAACTGCCTTGCCCG
F2:TTCGCTAACTGCCTTGCCCA
R:CTTTTCGGACACCCTCTTAG
GM19_44779426引物对序列:
F1:TCTAGATTATTAGTATAAGT
F2:TCTAGATTATTAGTATAAGC
R:AGTGAGACCTTTAATTTACT
GM19_44815533引物对序列:
F1:TCATGTGCGGTATACTTACT
F2:TCATGTGCGGTATACTTACC
R:ACCATTATC TATTCTAGGGA
GM19_44902890引物对序列:
F1:TGGCAATCACTTGTTTTAAA
F2:TGGCAATCACTTGTTTTAAT
R:CGTTTGACACTTCAGATGTT
GM19_45000827引物对序列:
F1:CGGTGTAGTTCGGGAAACAC
F2:CGGTGTAGTTCGGGAAACAA
R:ACATGCCTACCAACAAAAGA
GM19_45361938引物对序列:
F1:AGATTCCCAGGCGCAACTTG
F2:AGATTCCCAGGCGCAACTTA
R:TTTGTAGCTTCTGTTTATAG
GM19_45422153引物对序列:
F1:GTCTTGGTCCGCCTTCGTCC
F2:GTCTTGGTCCGCCTTCGTCT
R:CACAGGTAGCAGCTTAGCAA
GM19_45494716引物对序列:
F1:ATTAGCCCTGGCTGAAGAAG
F2:ATTAGCCCTGGCTGAAGAAA
R:GCTCATGTTGGATGAGCTTT
(2)KASP SNP PCR扩增父母本,即吉密豆1号和公交9112,设置三个重复,通过检测SNP是否在吉密豆1号和公交9112存在多态性,来验证是否可作为SNP标记。LightCycler 480软件(v1.5)的Endpoint分析模块检测发现,8个SNP引物,即GM19_44412186,GM19_44546232,GM19_44595030,GM19_44779426,GM19_44902890,GM19_45000827、GM19_45361938和GM19_45494716在吉密豆1号和公交9112间,三个重复荧光显色皆为为蓝绿差异,即存在稳定的多态性(图3,其中d表示绿色,e表示蓝色,c表示粉色)。其中,PCR反应体系为3ul:DNA 1.5ul(5-30ng/ul),2×KASP Master MIX缓冲液1.5ul,KASP assay 0.05ul;PCR反应条件为:95℃预变性10分钟;95℃变性20秒,65℃退火延伸1.00(每循环降0.8度),10个循环;95℃变性20秒,65℃退火延伸1分钟,36个循环;最后降温到10℃。
(3)在吉密豆1号和公交9112杂交的377个F2遗传个体中,对8个多态SNP引物(GM1944412186,GM1944546232,GM1944595030,GM1944779426,GM1944902890,GM19_45000827、GM19_45361938和GM19_45494716)进行KASP PCR扩增,得到377个F2个体在8个SNP位点的基因型数据。
(4)结合377个F2:3株系株高表型数据,用JoinMap 3.0软件进行连锁分析,计算重组率;并利用Kosambi函数将重组率转化成遗传图距,确定标记和基因之间的排列顺序,利用MapChart 2.0进行连锁图谱的绘制(图4)。将矮杆基因定位到SNP标记GM19_45000827和GM19_45361938标记间,矮杆基因与两个紧密连锁标记的遗传距离分别为,2个SNP标记间的物理距离约为361kb。
施例3利用KASP SNP PCR技术对大豆矮杆性状进行基因型鉴定
(1)采用CTAB法提取用于鉴定的大豆材料的DNA,无菌水溶解并稀释到5-30ng/ul,用于KASP PCR反应鉴定基因型。
(2)KASP SNP PCR反应体系、反应条件和基因型分析方法按照实施例2中的条件进行。
(3)利用2个标记(GM19_45000827和GM19_45361938)的引物,对“吉密豆1号”ד公交9112”的272个杂交后代F4:5的株系进行鉴定,LightCycler 480软件(v1.5)的Endpoint分析模块检测发现(图5、图6),
对于标记GM19_45000827而言,共115绿色信号,93个蓝色信号,40红色信号,24个粉色信号或其它;对于GM19_45361938而言,共111绿色信号,92个蓝色信号,48红色信号,21个粉色信号或其它。其中,绿信号,与公交9112高杆亲本基因型一致,为高杆基因型(DWDW)。蓝信号,与吉密豆号基因型一致,为矮杆基因型(dwd w);红信号,杂合基因型(DWdw);粉色信号或其它,无法确定基因型或无信号(unknown或negtive)。
(4)统计矮杆鉴定符合率发现,对于GM19_45000827而言,93个表现蓝色信号的矮杆基因型(dwdw)的个体中90个是矮杆品系,矮杆鉴定准确率为96.77%。对于GM19_45361938而言,92个表现蓝色信号的矮杆基因型(dwdw)的个体中86个是矮杆品系,矮杆鉴定准确率93.48%。因此,通过对株高性状的表现型测定结果和KASP SNP基因型分型结果的统计分析,证明这2个SNP标记能够高度准确地对大豆矮杆和高杆进行良好分型。
上述说明示出并描述了发明的若干优选实施例,但如前所述,应当理解发明并非局限于本文所披露的形式,不应看作是对其他实施例的排除,而可用于各种其他组合、修改和环境,并能够在本文所述发明构想范围内,通过上述教导或相关领域的技术或知识进行改动。而本领域人员所进行的改动和变化不脱离发明的精神和范围,则都应在发明所附权利要求的保护范围内。
- 还没有人留言评论。精彩留言会获得点赞!