一种引物组的设计方法及其应用与流程
本发明属于生物
技术领域:
,涉及一种引物组的设计方法及其应用,尤其涉及一种免疫组库的引物组的设计方法及其应用。
背景技术:
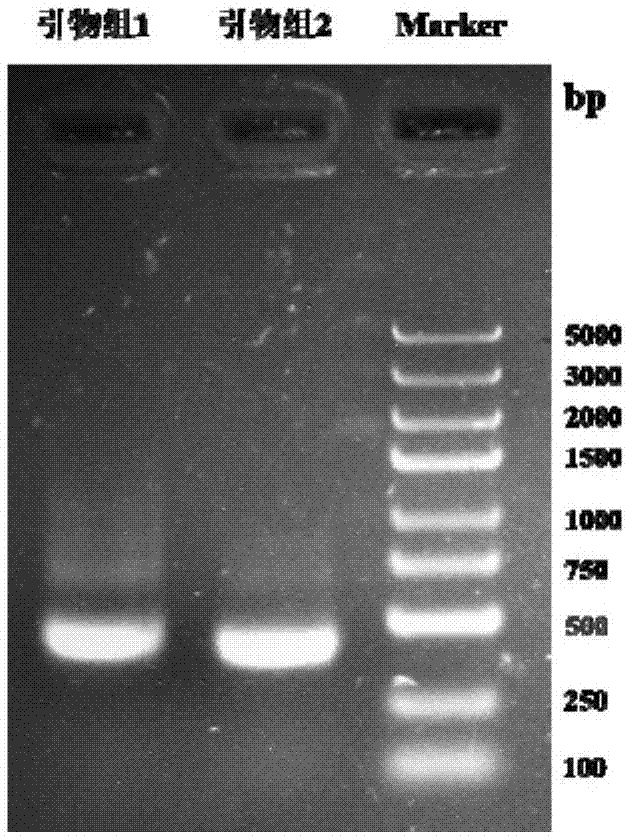
:免疫组库(immunerepertoire,ir)是指在任何指定时间内,某个个体的循环系统中所有功能多样性b细胞和t细胞的多态性总和。人体负责保卫机体的免疫细胞主要有t细胞、b细胞、巨噬细胞、树突细胞等。t、b细胞是人体主要的淋巴细胞,分别负责细胞免疫和体液免疫,其中,b细胞约占外周淋巴细胞总数的20%。bcr是由两条重链和两条轻链连接而成的,其中重链分为可变区(v区)、恒定区(c区)、跨膜区及胞质区;轻链则只有v区和c区。其中,v区由vh和vl两个结构域组成,各由三个互补决定区(cdr)组成,分别为cdr1、cdr2和cdr3,此三个cdr区共同参与抗体对抗原的识别,共同决定bcr、tcr的抗原特异性。个体中cdr区的氨基酸组成和排列顺序呈现高度多样性,在同一个体内,cdr区多样性可达109-1012,构成容量巨大的bcr库,赋予个体识别各种抗原、产生特异性抗体的巨大潜能。目前新兴的免疫组库研究重点就集中在研究cdr基因的多样性上。因此,需要进行免疫组库的扩增。目前,免疫组库扩增方法主要有:5’-race法、多重pcr方法、唯一分子标识符(uniquemolecularidentifiers,简称uid)法等。5’-race法即5'末端的快速扩增法,该法用特异性引物进行逆转录后,在cdna的一链5'末端加入接头,进行二次无偏移的pcr扩增,并通过亲和素磁珠富集,获得含目标区域的序列。该方法扩增引物只需要基因特异性引物如bcr/tcr的c区保守区引物,可以减少多重pcr偏差,但是,该法只能用于扩增rna并且要分选出所要研究的特定类型的细胞;实验较普通多重pcr复杂,且有基因转录本长度和gc含量的偏好性。该方法使用的引物仅仅设计在c区一端,由于是快速扩增,所以产物长度范围较大。5'race法,可实现不同克隆等效扩增,单一克隆数高达105,可从低丰度的转录本中快速扩增cdna的5’末端,扩增区域cdr。可以最大程度的避免pcr扩增的偏向性。该方法具有操作繁琐,实验样本打断会丢失部分序列,重复性较差等缺陷。多重pcr法:在同一pcr反应体系里加入两对以上引物,同时扩增出多个核酸片段的pcr反应,其反应原理、反应试剂、操作过程与一般pcr相同。多重pcr与普通pcr相同,不用将样本打断,数据完整,但是扩增具有偏向性。免疫基因多样性的进化通过基因复制、基因突变完成,可通过设计多重pcr扩增引物扩增得到目的bcr/tcr基因。该方法通常在v区和j/c区的保守区域设计引物实现多重pcr扩增,但由于引物不同pcr扩增的效率必然不同造成扩增偏差(amplificationbias),有些引物被大量扩增有些却几乎没有扩出来,只有通过几次优化找到最优的引物浓度组合来消除pcr扩增偏差,但这种方法对新的引物不具有通用性,因而增加了寻找最佳引物浓度的复杂性。通常可变区基因的克隆,是参考kabat数据库中的抗体序列,针对抗体可变区的保守区域设计若干套通用引物,采用rt-pcr法从人的淋巴b细胞的cdna文库中扩增可变区基因,该方法简单实用,通常5’端通用引物设计在第一骨架区或前导肽区;3’端通用引物设计在恒定区或j链区,但是该方法得到的抗体序列比较短,fr1前的序列无法测到。uid法,即在大规模pcr扩增目标分子前为每个分子加入唯一的uid,这种uid是随机合成的一般12~16个寡聚核苷酸(randombarcode),他们的随机组合会产生庞大的数量来为样品中特定的每个分子加上不同的标签,这样即使pcr扩增不均匀也可以通过计算的方法消除这种偏差,同时还能矫正pcr和测序错误。但这种方法需要很长的引物,会导致扩增效率下降和目标基因片段缩短,并且需要极高的通量才能覆盖所有的uid,该方法目前主要应用于igh/tcr的rna测序。针对免疫组库的引物设计,目前技术很难设计一个上游引物用于识别bcr基因的5’端序列,根据fr1的区域进行引物设计会导致抗体的上游信息无法覆盖到,使得测序基因信息不完整;对于文献2(wangx,stollarbd.humanimmunoglobulinvariableregiongeneanalysisbysinglecellrt-pcr.jimmunolmethods2000;244:217–25)中的引物,虽然引物的数量较少,但是覆盖度较低。多重引物pcr技术的免疫组库测序方法,仅仅对bcr测序的话,就需要几十对引物,这样,庞大的引物数量会让整个pcr的扩增效率和特异性变的很差。鉴于现有的抗体免疫组库扩增存在上述各种缺陷,因此,研发一种简洁高效、覆盖率高、错配率低的引物组设计方法,具有广阔的应用前景和巨大的市场价值。技术实现要素:针对现有技术的不足及实际的需求,本发明提供一种引物组的设计方法及其应用,巧妙地提取抗体fr1的起始位点前的序列并设计候选引物库,通过生物信息学设计分析,筛选得到引物组,该方法适于所有具有免疫系统的物种免疫组库的引物组的设计,具有方法简单、覆盖度高、错配率低、效率高等优势,并可大大节省免疫组库扩增的成本。为达此目的,本发明采用以下技术方案:第一方面,本发明提供一种引物组的设计方法,包括如下步骤:(1)获取物种的抗体重链和/或轻链的种系基因数据以及物种的基因组数据;(2)引物选择区域定位:将抗体重链和/或轻链的种系基因比对到物种的基因组数据上,找到抗体fr1的起始位点,提取起始位点前的序列;(3)候选引物库设计:对步骤(2)提取的起始位点前的序列进行切片,从5’端的第一个碱基开始,依次移位切取固定长度的引物序列,形成候选引物库;(4)进行cluster聚类分析,筛选得到初始引物组;(5)将初始引物组与抗体数据库比对,筛选得到最终引物组。本发明中,发明人在长期的科研实践中,针对现有技术中免疫组库引物组设计存在的缺陷和弊端,为解决引物数量过多导致的效率降低、引物数量较少产生的覆盖度较低以及无法覆盖上游信息等问题,根据扩增长度的需要,巧妙地在抗体fr1起始位点的上游,提取fr1起始位点前的序列,并从5’端的第一个碱基开始,依次移位切取固定长度的引物序列,形成候选引物库,聚类分析后筛选得到引物组,经过实验验证,采用该方法得到的引物组能够显著提高覆盖度和实验效率,降低错配率,方法简单,节省成本。优选地,步骤(1)所述物种包括人、小鼠、大鼠、兔、恒河猴、绵羊、猪、硬骨鱼类、软骨鱼类、大西洋鳕鱼、海峡鲶鱼、彩虹鳟鱼、斑马鱼、鸭嘴兽、羊驼、食蟹猕猴、牛、狗、鸡或三文鱼中的任意一种或至少两种的组合。优选地,步骤(2)所述起始位点前的序列的长度范围为1-300bp,例如可以是1bp、5bp、10bp、20bp、30bp、40bp、50bp、60bp、70bp、80bp、90bp、100bp、110bp、120bp、130bp、140bp、150bp、160bp、170bp、180bp、190bp、200bp、210bp、220bp、230bp、240bp、250bp、260bp、270bp、280bp、290bp或300bp。优选地,步骤(3)所述引物序列的固定长度范围为16-26bp,例如可以是16bp、17bp、18bp、19bp、20bp、21bp或22bp,优选为18-22bp。优选地,步骤(4)所述聚类分析的方法为:通过cd-hit软件,设置-c参数,将候选引物库序列按照-c参数设定进行聚类。优选地,所述-c参数设置为0.8-1.0,例如可以是0.8、0.9或1.0,优选为0.9。优选地,步骤(4)所述筛选的方法为:将cluster根据大小排序,选择前10-90的聚类,提取每个cluster的代表序列,根据引物设计原则,筛选得到初始引物组。所述前10-90的聚类例如可以是前10、前20、前30、前40、前50、前60、前70、前80或前90。优选地,步骤(5)所述比对的方法为:将初始引物组与所对应物种的抗体数据库比对,得到每条序列在抗体库的覆盖度,将该值按照大小排序,选取排名前10-40的引物作为最终引物组。所述前10-40的引物例如可以是前10、前16、前20、前25、前30、前35或前40。作为优选技术方案,一种引物组的设计方法,具体包括如下步骤:(1)获取物种的抗体轻链和/或重链的种系基因数据以及物种的基因组数据;其中,所述物种包括人、小鼠、大鼠、兔、恒河猴、绵羊、猪、硬骨鱼类、软骨鱼类、大西洋鳕鱼、海峡鲶鱼、彩虹鳟鱼、斑马鱼、鸭嘴兽、羊驼、食蟹猕猴、牛、狗、鸡或三文鱼中的任意一种或至少两种的组合;(2)引物选择区域定位:将抗体重链和/或轻链的种系基因比对到物种的抗体所在染色体的基因组数据上,找到抗体fr1的起始位点,提取起始位点前1-300bp的序列;(3)候选引物库设计:对步骤(2)提取的起始位点前的序列进行切片,从5’端的第一个碱基开始,依次移位切取固定长度的引物序列,引物的长度范围为18-22bp,形成候选引物库;(4)进行cluster聚类分析,筛选得到初始引物组;所述聚类分析的方法为:通过cd-hit软件,设置-c参数,将候选引物库序列按照-c参数设定进行聚类;所述-c参数的大小设置为0.9;所述筛选的方法为:将cluster根据大小排序,选择前10-90的聚类,提取每个cluster的代表序列,根据引物设计原则,筛选得到初始引物组;(5)将初始引物组与对应物种的抗体数据库比对,得到每条引物在抗体库的覆盖度,将该值按照大小排序,选取排名前10-40的引物作为最终引物组。第二方面,本发明提供一种采用第一方面所述方法设计得到的人b细胞免疫组库重链引物组,所述引物组的序列包含如seqno.1-seqno.16所示序列;详细序列如下:seqno.1:atggacatactttgttccaseqno.2:ccatggagtttgggctgagcseqno.3:ggctgagctgggttttccttseqno.4:ctgagctgggttttccttgtseqno.5:ctcctggtggcagctcccagseqno.6:cctcctcctggtggcagctcseqno.7:cagctcccagatgtgagtgtseqno.8:gctgggttttccttgttgctseqno.9:atgaaacacctgtggttcttseqno.10:cctggaggatcctcttcttgseqno.11:ggttttccttgttgctatttseqno.12:tggtggcagctcccagatgtseqno.13:ggacgtgagtgagagaaacaseqno.14:tcctcaccatggactggaccseqno.15:cttgttggtattttaaaaggseqno.16:gaggatcctcttcttggtgg.优选地,所述引物组的序列包含如seqidno.1-seqidno.16所示序列中的任意十五条引物序列。所述任意十五条引物序列为随机从seqidno.1-seqidno.16所示序列中选取得到。进一步优选地,所述引物组的序列包含如seqidno.1-seqidno.16所示序列中的任意十条引物序列。所述任意十条引物序列为随机从seqidno.1-seqidno.16所示序列中选取得到。第三方面,本发明提供一种如第一方面所述方法或第二方面所述引物组在免疫组库扩增中的应用。第四方面,本发明提供一种如第一方面所述方法或第二方面所述引物组用于制备扩增免疫组库的试剂盒的应用。与现有技术相比,本发明具有如下有益效果:本发明提供的方法简洁高效,能显著提高多重pcr的反应效率和覆盖度,覆盖度高达95%-100%,降低错配率,抗体的完整性更高,节省成本。附图说明图1为本发明的人b细胞免疫组库pcr扩增电泳图;图2为本发明的引物组扩增出的cdr3的长度分布图;图3为本发明的igvh的cdr3的种系基因(germline)分布图。具体实施方式为更进一步阐述本发明所采取的技术手段及其效果,以下结合附图并通过具体实施方式来进一步说明本发明的技术方案,但本发明并非局限在实施例范围内。实施例1人b细胞免疫组库引物组的设计(1)提取人的germline基因从imgt数据库(http://imgt.org/vquest/refseqh.html)下载人的重链germline基因:提取出361条基因,其中1条fasta序列信息如下:>m99641|ighv1-18*01|homosapiens|f|v-region|188..483|296nt|1|||||296+24=320|||(2)引物选择区域定位将361条序列比对到14号染色体“hs_ref_grch38.p12_chr14.fa”上,提取序列起始位点前150bp的碱基序列组;(3)候选引物库设计按照每20bp形成69430条引物起始数据集,形成候选引物库。(4)cluster聚类分析将候选引物库用cd-hit软件分类,设置-c参数为0.9,进行聚类分析;将cluster根据大小排序,选择前90的聚类,提取每个cluster的代表序列,根据引物设计原则,筛选得到初始引物组;引物原始数据组cluster聚类分析,”>”后面的数字为候选引物库里面的引物序列编号,%表示候选引物序列与代表序列的相似度,聚类的部分结果如下表1:表1由表1可知,cluster1中的序列和代表序列的相似度,即通过cd-hit筛选后,cluster中的95%以上的集合中前35个序列。(5)将初始引物组与人的抗体数据库比对,得到每条序列在抗体库的覆盖度,将该值按照大小排序,选取排名前20,分析gc含量、退火温度等,最终选择16条作为最终重链引物组进行下一步实验验证。选择的16条的引物序列(seqno.1-seqno.16)即筛选出的人b细胞免疫组库引物组,详细序列如下(5’-3’):seqno.1:atggacatactttgttccaseqno.2:ccatggagtttgggctgagcseqno.3:ggctgagctgggttttccttseqno.4:ctgagctgggttttccttgtseqno.5:ctcctggtggcagctcccagseqno.6:cctcctcctggtggcagctcseqno.7:cagctcccagatgtgagtgtseqno.8:gctgggttttccttgttgctseqno.9:atgaaacacctgtggttcttseqno.10:cctggaggatcctcttcttgseqno.11:ggttttccttgttgctatttseqno.12:tggtggcagctcccagatgtseqno.13:ggacgtgagtgagagaaacaseqno.14:tcctcaccatggactggaccseqno.15:cttgttggtattttaaaaggseqno.16:gaggatcctcttcttggtgg后续引物组性能评估时以seqno.1-seqno.16作为上游引物扩增免疫组库;以seqno.17作为下游引物扩增免疫组库,下游引物序列seqno.17为(5’-3’):seqno.17:ggggaagaccgatgggcccttggtgg实施例2引物组性能评估(1)样本制备使用人淋巴细胞分离液分离外周血b淋巴细胞,提取rna,逆转录得cdna,步骤如下:1)收集的新鲜外周血样(5个)各10毫升(ml),按lymphoprep试剂盒说明书操作,获得相对较纯的外周血单核细胞(pbmc);2)提取rna,并用nanodrop2000测定rna的浓度及纯度,使用tianscriptm-mlv逆转录酶试剂盒(货号er104),逆转录后得cdna(即样本:st-1、st-2、st-3、st-4、st-5)为扩增模板,备用;逆转录反应步骤如下:a)体系配置:按照表2配置逆转录反应体系。表2组分用量oligo(dt)12-182μl总rna1μgdntp2μlddh2o用ddh2o将体系补足至15μlb)65℃加热5min后迅速在冰上冷却2min,简短离心收集反应液后加入4μl5×first-strandbuffer及rnasin(40u/μl);c)加入1μl(200u)tianscriptm-mlv并轻轻用移液器混匀;d)42℃温浴1h10min;e)85℃加热5min终止反应置于冰上用于后续实验。(2)多重pcr扩增1)体系配置加入上游引物以及下游引物,配置多重pcr体系,其中,该体系中的上游引物组(seqidno:1-seqidno:16)的等摩尔混合物,引物总浓度是20μμ;下游引物(seqno.17)总浓度是20μμ;cdna(即样本:st-1、st-2、st-3、st-4、st-5)为扩增模板,多重pcr反应体系按照表3配置;表3组分体积10×pcr缓冲液5μl上游引物组2μl下游引物2μldntp1μltaq酶0.5μlcdna5μlddh2o34.5μl总计50μl2)pcr反应按多重pcr的条件设置pcr仪器程序,进行多重pcr:pcr结束后,4℃保存pcr产物并电泳检测,挑选片段长度约为500bp的片段,割胶回收,得到纯化后的抗体dna片段,胶回收步骤采用qiagen公司qiaquick胶纯化试剂盒,按常规实验操作进行;nanodrop2000测试dna浓度,并进行高通量测序。多重pcr条件如下表4:表4pcr扩增后经凝胶电泳得到人b细胞免疫组库pcr扩增电泳图见图(1),由图(1)可知,在500bp附近有明显的条带,且本发明引物组1(即seqidno:1-seqidno:16)与引物组2(文献1(1.high-throughputisolationofimmunoglobulingenesfromsinglehumanbcellsandexpressionasmonoclonalantibodies.liaohx1,levesquemc,nagela,etal.jvirolmethods.2009;158(1-2):171-9.)公开引物)扩增出的条带范围基本一致。可以初步判断,本发明设计的引物组(seqidno:1-seqidno:16)可以扩增出人b细胞免疫组库的重链序列。(3)引物利用率分析采用生物信息学bowtie软件,分析引物(seqidno:1-seqidno:16)与5个样本(st-1、st-2、st-3、st-4、st-5)的序列匹配情况,以5’race方法为对照(即该方法扩增出的抗体序列为基准),分析得到的引物的利用率如下表5所示;表5上表中:0错配表示不允许引物序列和抗体序列发生错配;1错配表示允许引物序列的1个碱基和抗体序列发生错配;2错配表示允许引物序列的2个碱基和抗体序列发生错配。上表结果显示:设计的引物组(seqidno:1-seqidno:16)与样本具有很好的匹配。(4)覆盖度分析以样本st-1、st-2、st-3、st-4、st-5为扩增模板,然后分别用本发明设计的引物(seqno.1-seqno.16,即表中引物组1)和文献1中的引物(表中引物组2)进行pcr扩增,以5’race方法为对照(即该方法扩增出的抗体序列为基准),分析本发明引物扩增出的抗体序列的覆盖度,覆盖度分析结果如下表6:表6覆盖度等于“pcr法”除以“5’race法”再乘以百分之百。其中:“pcr法”指多重pcr法扩增出的与5’race法扩增出的抗体序列相一致的条数;“5’race法”指采用5’race法扩增出的抗体序列的总条数。上表中,对测试的5个样本,引物组1扩增出的的抗体序列的平均覆盖度在95.01%以上,比文献中引物组2的覆盖平均为89.35%,本发明设计引物组1比文献中公开的引物的覆盖度平均高出5.66%,本发明设计的引物组具有很高的覆盖度。实施例3免疫组库生物信息分析用设计的引物组(seqidno:1-seqidno:16)为上游引物,seqidno:17为下游引物,扩增出的人b细胞免疫组库,进行后续生物信息学分析,分析结果如下:(1)cdr3区域长度分析cdr3氨基酸序列中,1个氨基酸(aa)的变化可能导致受体构象的改变,因此,cdr3氨基酸序列长度的变化可以反映出cdr3基因连接区的多样性,通过igblast软件分析免疫组库的序列信息后,得到免疫组库的cdr3长度分布情况(见图(2)),和cdr3区域中种系基因(germline)使用情况(见图(3))。由图2可以发现,cdr3的氨基酸长度主要集中在为9-12个氨基酸之间。(2)cdr3区域中种系基因(germline)使用情况图3为igvh的cdr3的germline分布和使用分布;横坐标为不同的germline,纵坐标为某种germline的数量,结果见图(3);由图(3)可知,不同germline的丰度非常不一致,有些片段丰度显著高于其他片段,该样本的vh的germline主要集中在igvh3-7*01。(3)cdr3区域中氨基酸的种类,以及氨基酸序列出现的频率和长度情况见表7所示;表7由表7可知,cdr3的序列变化很大,有些片段丰度如“avdsnyqli”等高于其他片段,可以根据cdr3的丰度,判断免疫组库中的特征抗体,为后续研究和治疗等提供思路。综上所述,本发明提供一种引物组的设计方法,根据扩增长度的需要,巧妙地在抗体fr1起始位点的上游,提取fr1起始位点前的序列,并从5’端的第一个碱基开始,依次移位切取固定长度的引物序列,形成候选引物库,聚类分析后筛选得到引物组,采用该方法得到的引物组能够显著提高覆盖度和实验效率,降低错配率,方法简单,节省成本。申请人声明,本发明通过上述实施例来说明本发明的详细方法,但本发明并不局限于上述详细方法,即不意味着本发明必须依赖上述详细方法才能实施。所属
技术领域:
的技术人员应该明了,对本发明的任何改进,对本发明产品各原料的等效替换及辅助成分的添加、具体方式的选择等,均落在本发明的保护范围和公开范围之内。序列表<110>苏州泓迅生物科技股份有限公司<120>一种引物组的设计方法及其应用<130>2018年<141>2018-11-06<160>17<170>siposequencelisting1.0<210>1<211>19<212>dna<213>人工合成()<400>1atggacatactttgttcca19<210>2<211>20<212>dna<213>人工合成()<400>2ccatggagtttgggctgagc20<210>3<211>20<212>dna<213>人工合成()<400>3ggctgagctgggttttcctt20<210>4<211>20<212>dna<213>人工合成()<400>4ctgagctgggttttccttgt20<210>5<211>20<212>dna<213>人工合成()<400>5ctcctggtggcagctcccag20<210>6<211>20<212>dna<213>人工合成()<400>6cctcctcctggtggcagctc20<210>7<211>20<212>dna<213>人工合成()<400>7cagctcccagatgtgagtgt20<210>8<211>20<212>dna<213>人工合成()<400>8gctgggttttccttgttgct20<210>9<211>20<212>dna<213>人工合成()<400>9atgaaacacctgtggttctt20<210>10<211>20<212>dna<213>人工合成()<400>10cctggaggatcctcttcttg20<210>11<211>20<212>dna<213>人工合成()<400>11ggttttccttgttgctattt20<210>12<211>20<212>dna<213>人工合成()<400>12tggtggcagctcccagatgt20<210>13<211>20<212>dna<213>人工合成()<400>13ggacgtgagtgagagaaaca20<210>14<211>20<212>dna<213>人工合成()<400>14tcctcaccatggactggacc20<210>15<211>20<212>dna<213>人工合成()<400>15cttgttggtattttaaaagg20<210>16<211>20<212>dna<213>人工合成()<400>16gaggatcctcttcttggtgg20<210>17<211>26<212>dna<213>人工合成()<400>17ggggaagaccgatgggcccttggtgg26当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1