Wnt5a基因作为猪产仔数性状相关检测的分子标记的制作方法
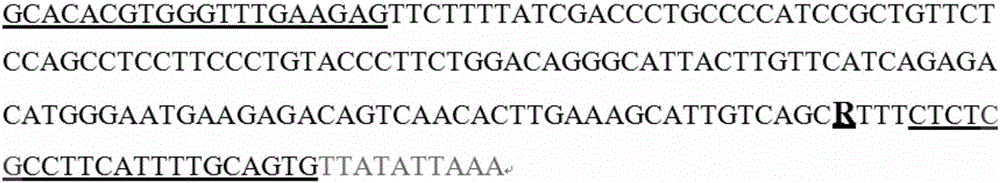
本发明属于猪分子标记筛选技术领域,具体涉及Wnt5a基因作为猪产仔数性状相关检测的分子标记。所述的分子标记从猪WNT5A基因中克隆得到,本发明还包括猪WNT5A基因3′UTR序列突变位点的检测方法与应用。
背景技术:
母猪产仔数是猪繁殖性状的一个重要指标,是影响养猪业经济效益的重要因素。猪妊娠早期胚胎附植是一个关键步骤,因为在这个过程中胚胎死亡率高(Park et al.,2000),是导致产仔数下降的一个重要原因(Geisert et al.,2002)因此研究与猪早期胚胎附植相关的基因对于猪产仔数的遗传改良有重要的意义。
子宫的容受性和囊胚的植入之间的相互作用在胚胎附植时起关键性作用,在妊娠时子宫要经历形态上、细胞上和分子水平上的变化。子宫的重塑需要转录因子、蛋白同源转化、生长因子、成形素和信号转导分子(Cha et al.,2012)。遗传干扰对这些信号通路的影响可以导致附植失败或者延迟附植,这样对整个妊娠期都有影响,最终导致妊娠失败(Song et al.,2002;Ye et al.,2005;Sun et al.,2012)。Wnt信号通路在器官形成和决定细胞命运中发挥作用(Angers and Moon,2009;MacDonald et al.,2009;van Amerongen and Nusse,2009)。在经典的信号通路中,Wnt配体结合Fzd和LRP5/LRP6受体来稳定β-catenin发生核异位,然后β-catenin结合到T细胞因子/淋巴增强子结合因子来激活基因的转录。在非经典的Wnt信号通路中,Wnt不依赖β-catenin作为中介物,直接调控细胞移动、细胞形状和细胞极性(Kikuchi et al.,2011;van Amerongen and Nusse,2009;Veeman et al.,2003)。
Wnt5a基因是一种分泌型糖蛋白,属于高度保守蛋白家族一员,调控许多重要的生物学过程包括决定细胞命运、胚胎发育、细胞增殖、迁移和分化。Wnt5a被认为是非经典Wnt配体,在蟾蜍、斑马鱼和小鼠中敲除Wnt5a后直接影响了细胞运动和极性(Gao et al.,2011;Miyoshi et al.,2012;Schambony and Wedlich,2007;Yamaguchi et al.,1999)。目前,研究发现Wnt5a基因在小鼠胚胎附植过程中具有重要作用。用免疫荧光的方法检测到Wnt5a在小鼠妊娠第3天和4天时在子宫内膜上皮细胞和基质中表达,随着附植的发生,在妊娠第5天时基质细胞M侧(附植侧)检测到Wnt5a高表达,第8天时表达量降低(Cha et al.,2014)。这些结果表明Wnt5a在小鼠妊娠第3天和4天时的表达模式与子宫的容受性有关,从而有利于胚胎附植。在Wnt5a敲除小鼠和Wnt5a超表达小鼠的子宫内可以看到没有附植的囊泡,这两种小鼠都存在附植失败和严重的生育问题。鉴于以上研究我们认为Wnt5a基因可能在猪胚胎附植中也发挥着关键性的作用,而对基因突变的多态性位点在群体中的进行关联分析是研究基因功能的一个有力的方法,所以申请人对Wnt5a基因进行了多态性和关联分析,以期为猪的繁殖性状(主要是猪产仔数性状)提供遗传标记资源。
技术实现要素:
发明的目的在于筛选一种与猪产仔数性状相关的分子标记。本发明通过克隆Wnt5a基因3′UTR序列,寻找突变位点以及基因多态性的检测方法,为猪产仔数性状检测提供一种分子标记和方法。
本发明通过以下技术方案实现:
一种与猪产仔数性状相关的分子标记,通过如下步骤筛选得到:
(1)根据NCBI数据库里猪Wnt5a基因的cDNA序列(GeneBank Accession:XM_005669658.2)克隆得到Wnt5a基因的部分DNA片段(它的DNA序列如序列表SEQ ID NO:1和图2所述。)在序列表SEQ ID NO:1的第213位碱基处存在一个A213-G213的碱基突变(等位基因突变),该突变导致BcgI-RFLP多态性(该突变位于Wnt5a基因3′UTR区域内)。
(2)利用双荧光素酶报告实验发现这个突变位点显著影响miR-628-5p与Wnt5a的结合效率,验证这个突变位点是功能性突变位点。
一种筛选与猪产仔数性状相关分子标记方法,包括下列步骤:
(1)用猪Wnt5a基因的3′UTR序列为模板设计引物;提取猪基因组DNA,设计引物(该引物的核苷酸序列如下所示:正向5′-ATCACACCCTGAGTGTCACG-3′,反向5′-TGTGGAAAGAGGTGTCCTGG-3′),PCR扩增、PCR产物纯化和测序,获得如序列表SEQ ID NO:1所示的核苷酸序列。
(2)通过分析A/G位点的突变,设计一对用于酶切分析的突变引物(酶切突变引物如下所示:正向5’-GCACACGTGGGTTTGAAGAG-3’,反向5’-TTTAATATAA CACTGCAAAATGAAGGCGAGAG-3’),采用BcgI进行酶切检测多态性,所述的扩增的酶切分型得序列如序列表SEQ ID NO:6所示,
(3)应用PCR-RFLP的方法对序列表SEQ ID NO:1所示的第213位碱基突变进行检测,并初步进行其基因型和猪产仔数性状之间的关联分析。
本发明为猪的分子标记辅助选择提供了一个新的标记。
更详细的技术方案请参见说明书的《附图说明》及《具体实施方式》中的实施例。
附图说明
序列表SEQ ID NO:1是扩增的Wnt5a基因3′UTR的部分序列(即本发明的分子标记),序列长度为366bp。
序列表SEQ ID NO:2和3是扩增的Wnt5a基因用于酶切分型的引物对的序列。
序列表SEQ ID NO:4和5是验证序列表SEQ ID NO:1的BcgI-RFLP多态性的突变引物对的序列。
序列表SEQ ID NO:6是扩增的Wnt5a基因用于酶切分型的核苷酸序列,序列长度为185bp,在该序列的150位碱基处存在一个等位基因突变。
图1:本发明技术流程图。
图2:本发明中猪Wnt5a基因用于PCR-RFLP检测的DNA片段,即本发明制备的分子标记(下划线部分为引物,英文字母R代表突变位点,等位基因的突变以(A/G)表示,红色字体为突变引物中突变位点,蓝色字体为保护碱基)。
图3:双荧光素酶报告实验结果,SNP显著影响miR-628-5p与Wnt5a的结合效率。
图4:本发明中Wnt5a基因BcgI-RFLP的三种基因型(AA AG GG)电泳结果。附图标记说明;图中M:DNA分子量标准(marker I,购自宝生物工程大连有限公司)。
图5:是本发明的分子标记的核苷酸序列图。
具体实施方式
实施例1 Wnt5a基因的克隆
(1)引物设计
用NCBI里提供的猪Wnt5a基因的3′UTR序列作为参考序列设计引物进行PCR扩增,该引物对的核苷酸序列如下所述:
Wnt5a:正向引物:5′-ATCACACCCTGAGTGTCACG-3′
反向引物:5′-TGTGGAAAGAGGTGTCCTGG-3′
(2)PCR产物的克隆和测序
将纯化后的PCR产物与pMD-18T载体(购自宝生物工程大连有限公司),在4℃水浴过夜连接;无菌状态下取100-120μl感受态细胞于1.5ml Ependorff管中,将5μl的连接产物加入混匀,在冰上放置30min,42℃热激90s,冰浴3-4min,加入400μl无抗生素的LB液体培养基,于37℃振荡培养45min。取100μl上述LB涂布于异丙基硫代-β-D-半乳糖苷(IPTG)X-gal的琼脂平板上,于37℃平放1h后倒置培养。挑取平板上的单菌落,接种于2-3ml LB中,于37℃,300r/min培养过夜。用1.5ml EP管12000r/min离心30秒收集菌体制备少量质粒。测序后得到一条长度为如SEQ NO:1所示的3′UTR序列(长度为366bp),包含了Wnt5a基因的3′UTR部分序列。
实施例2 Wnt5a基因3′UTR的SNP显著影响miR-628-5p与靶基因的结合效率
1、构建Wnt5a基因3′UTR-psiCHECK-2双荧光素酶报告载体
(1)扩增目的片段回收纯化。
(2)取保存的100μL含有psi-CHECKTM-2载体的菌液加入到5mL含有氨苄青霉素的LB培养基中,扩大培养12个小时,用天根生物(北京)技术公司生产的质粒小提试剂盒从扩大培养的菌液中提取质粒DNA,按说明书操作,用NanoDrop分光光度计(Thermo)测定回收质粒的浓度。
(3)用限制性内切酶Xho I(Fermenters,ER0691)和Not I(Fermenters,ER0691)同时对目的片段和载体进行双酶切。用T4DNA连接酶(Fermentas,EL0011)连接载体和目的片段。向10μL连接产物中加入30uL大肠杆菌感受态并轻轻吹打混匀,冰上放置30min;然后42℃热激90s,取出后冰浴2min;加入600μL无抗生素的LB液体培养基,37℃,250r/m振荡培养60min;取80-100μL浑浊菌液涂布于琼脂平板上,37℃平放1-3h后倒置培养过夜。用10uL灭菌枪头挑取细菌培养皿上的单菌落,分别接种于800μL含100mg/L氨苄青霉素的LB液体培养基中,37℃,2500r/m振荡培养6-8h;通过菌液PCR初步筛选阳性克隆,然后将阳性克隆送测序公司进行测序以最终确定阳性克隆。
(4)用去内毒素质粒小量提取试剂盒(Endo-free Plasmid mimi KitⅡ,OMEGA)提取筛选出的阳性克隆AA基因型和GG基因型质粒DNA,用于下一步的细胞转染试验。
2、细胞培养和转染
(1)将储存于液氮的PK15细胞于37-40℃水浴中快速复苏,培养于二氧化碳培养箱(37℃,5%CO2)。PK15细胞培养基为含10%胎牛血清(购自杭州四季青生物工程材料有限公司)的DMEM(Hyclone)培养基(含1%的青霉素与链霉素复合物)。用于转染的培养基为Opti-MEM(Gbico)培养基。转染后更换的培养基为含10%胎牛血清的DMEM(Hyclone)培养基(不含1%的青霉素与链霉素复合物)。
(2)转染前一天将PK15细胞均匀接种于24孔板,每孔细胞数量以转染时达到75%-85%的汇合度为准。每孔转染500ng重组质粒DNA和50pmol miRNA mimics或者Duplex N.C,转染试剂(Lipofectamine 2000,invitrogen)2μL。转染步骤按照转染试剂说明书进行。转染4-6h后为细胞更换含10%FBS的DMEM培养基(无双抗)。
3、双荧光素酶报告载体的荧光检测与分析
在转染后16-18h,用裂解液处理转染的细胞并进行荧光素酶活性检测分析。用双荧光素酶检测试剂盒(Reporter Assay System,Promega)分别测定细胞中萤火虫与海肾荧光素酶的活性。操作步骤按照试剂盒说明书进行。得到原始结果后,用海肾荧光素酶的荧光值除去萤火虫荧光素酶的荧光值,从而得到每个样品中校正后的海肾荧光素酶的荧光值,最后对不同实验组的数据进行显著性分析检验。每组实验重复三次,每次有四孔重复。结果表明,Wnt5a(A)和miR-628-5p共转染后荧光值显著低于Wnt5a(A)和NC共转染后的荧光值,说明miR-628-5p的靶基因Wnt5a的互补序列预测准确,SNP位于miR-628-5p的靶基因Wnt5a的互补序列中;而Wnt5a(G)和miR-628-5p共转染后荧光值升高,而且与Wnt5a(G)和NC共转染后荧光值没有差异,说明该SNP显著影响miR-628-5p与Wnt5a的结合效率。
实施例3 PCR-RFLP诊断方法的建立
通过分析上述克隆得到的366bp序列(实施例1)的SNP位点,发现没有合适内切酶对该SNP位点进行酶切分型验证,故重新设计突变引物进行扩增,突变引物扩增得到的序列可以用BcgI内切酶进行酶切分型验证,具体步骤如下:
(1)引物序列:
正向引物:5’-GCACACGTGGGTTTGAAGAG-3’
反向引物:5’-TTTAATATAA CACTGCAAAATGAAGGCGAGAG
运用上述步骤(1)中引物扩增得到长度为185bp的序列,对应于序列表SEQ ID NO:6,这段序列就是申请人下面用于BcgI酶进行酶切分型的序列。完整序列信息如下:
GCACACGTGGGTTTGAAGAGTTCTTTTATCGACCCTGCCCCATCCGCTGTTCTCCAGCCTCCTTCCCTGTACCCTTCTGGACAGGGCATTACTTGTTCATCAGAGACATGGGAATGAAGAGACAGTCAACACTTGAAAGCATTGTCAGCRTTTCTCTCGCCTTCATTTTGCAGTGTTATATTAAA
上述序列中的下划线R是申请人需要酶切分型验证的突变位点。
(2)PCR扩增条件
PCR反应总体积为10μl,其中将上述实施例1中扩增得到的366bp产物,稀释100倍作为模板,加入1μl,5μlPCR Mix,正向引物、反向引物各0.2μl。PCR扩增程序为:94℃5min,循环32次94℃30s,60℃30s,然后72℃15s,最后72℃延伸5min。PCR反应产物用2%琼脂糖凝胶电泳检测,得到185bp特异扩增片段。测序结果发现该185bp片段存在一个BcgI酶切位点,其中第150bp处为多态性位点。
(3)PCR-RFLP检测条件
PCR产物酶切反应体积是10μl,其中1×buffer 1μl,PCR产物3μl,限制性内切酶BcgI为0.5μl(1U),用H2O补足10μl,将样品混匀后离心,37℃水浴1h,用3%琼脂糖凝胶电泳检测酶切结果,记录基因型,在紫外灯下拍照。对该位点两个纯合子的测序结果显示,当第150bp位置是G时,则该BcgI酶切位点不存在,BcgI酶切后检测结果只有1个片段,长度是185bp(定为等位基因G);但存在G150→A150替换时,其结果导致第136bp处一个BcgI酶切位点的产生,得到2个片段,长度分别为136bp和49bp(定为等位基因A),三种基因型AA、AG、GG(如图4所述)。
实施例4 本发明的分子标记在猪产仔数标记性状关联分析中的应用
本实施例关联分析所用的实验猪群来自中国福建省某猪场的大白猪,包括332头母猪,记录第一胎总产仔数数据。繁殖性状取自猪场2013-2015年的繁殖记录资料,记录繁殖性状包括总产仔数(TNB)、产活仔数(NBA)、出生窝重和断奶重(WW)等,并对全部332头母猪进行基因型检测,确定其基因型。
根据采集样本的繁殖记录信息和群体结构,申请人运用混合线性模型来统计分析Wnt5a基因SNP位点的基因型效应及其与产仔数性状的关系。
采用SAS软件对该突变位点的多态性进行性状分析,具体模型如下:
Yijkl=μ+genei+sirej+seasonk+eijkl
Y为性状值,μ为总体均数,其中固定效应包括:基因型效应genei;随机效应包括:公猪效应sirej,季度效应seasonk;eijkl为随机误差。
对大白猪Wnt5a基因BcgI-RFLP多态性位点进行性状关联分析,该突变位点在332个个体中,有207个AA基因型的个体,有117个AG基因型的个体,有8个GG基因型的个体。采用SAS软件对该突变位点的多态性进行性状关联分析,发现该突变位点的多态性与第一胎总产仔数有显著影响(P<0.05),其中AA基因型个体和AG基因型个体的第一胎总产仔数均显著高于GG基因型个体。结果见表1。
表1猪WNT5A基因SNP位点与第一胎总产仔数的关联分析结果
注:表中性状值为平均数±标准误,P<0.05代表有显著性差异。
由表1可知,Wnt5a基因SNP位点对第一胎总产仔数有显著影响((P<0.05),其中AA基因型个体和AG基因型个体的第一胎总产仔数均显著高于GG基因型个体。
主要参考文献
1.Park K,Inoue T,Ueda M et al.CD9is expressed on human endometrial epithelial cells in association with integrinsα6,α3,andβ1[J].Molecular Human Reproduction.2000,(6):252-257;
2.Geisert R,Schmitt R.Early embryonic survival in the pig:Can it be improved[J].Journal of Animal Science.2002,80:54-65;
3.Cha,J.,Sun,X.,and Dey,S.K.Mechanisms of implantation:strategies for successful pregnancy[J].Nat Med.2012,18:1754–176;
4.Song,H.,Lim,H.,Paria,B.C.,Matsumoto,H.,Swift,L.L.,Morrow,J.,Bonventre,J.V.,and Dey,S.K.Cytosolic phospholipase A2alpha is crucial[correction of A2alpha deficiency is crucial]for‘on-time’embryo implantation that directs subsequent development[J].Development.2002,129:2879–2889;
5.Ye,X.,Hama,K.,Contos,J.J.,Anliker,B.,Inoue,A.,Skinner,M.K.,Suzuki,H.,Amano,T.,Kennedy,G.,Arai,H.,et al.LPA3-mediated lysophosphatidic acid signalling in embryo implantation and spacing[J].Nature.2005,435:104–108;
6.Sun,X.,Zhang,L.,Xie,H.,Wan,H.,Magella,B.,Whitsett,J.A.,and Dey,S.K.Kruppel-like factor 5(KLF5)is critical for conferring uterine receptivity to implantation[J].Proc.Natl.Acad.Sci.USA.2012,109:1145–1150;
7.Angers,S.,and Moon,R.T.Proximal events in Wnt signal transduction[J].Nat.Rev.Mol.Cell Biol.2009,10:468–477;
8.MacDonald,B.T.,Tamai,K.,and He,X.Wnt/beta-catenin signaling:components,mechanisms,and diseases[J].Dev Cell.2009,17:9–26;
9.van Amerongen,R.,and Nusse,R.Towards an integrated view of Wnt signaling in development[J].Development.2009,136:3205–3214;
10.Kikuchi,A.,Yamamoto,H.,Sato,A.,and Matsumoto,S.New insights into the mechanism of Wnt signaling pathway activation[J].Int.Rev.Cell Mol.Biol.2011,291:21–71;
11.Veeman,M.T.,Axelrod,J.D.,and Moon,R.T.A second canon.Functions and mechanisms of beta-catenin-independent Wnt signaling[J].Dev.Cell.2003,5:367–377;
12.Gao,B.,Song,H.,Bishop,K.,Elliot,G.,Garrett,L.,English,M.A.,Andre,P.,Robinson,J.,Sood,R.,Minami,Y.,et al.Wnt signaling gradients establish planar cell polarity by inducing Vangl2 phosphorylation through Ror2[J].Dev.Cell.2011,20:163–176;
13.Miyoshi,H.,Ajima,R.,Luo,C.T.,Yamaguchi,T.P.,and Stappenbeck,T.S.Wnt5a potentiates TGF-b signaling to promote colonic crypt regeneration after tissue injury[J].Science.2012,338:108–113;
14.Schambony,A.,and Wedlich,D.Wnt-5A/Ror2 regulate expression of XPAPC through an alternative noncanonical signaling pathway[J].Dev.Cell.2007,12:779–792;
15.Yamaguchi,T.P.,Bradley,A.,McMahon,A.P.,and Jones,S.A Wnt5a pathway underlies outgrowth of multiple structures in the vertebrate embryo[J].Development.1999,126:1211–1223;16.Jeeyeon Cha,Amanda Bartos,Craig Park,Xiaofei Sun,Yingju Li,Sang-Wook Cha,2 Rieko Ajima,Hsin-Yi Henry Ho,Terry P.Yamaguchi,and Sudhansu K.Dey Appropriate Crypt Formation in the Uterus for Embryo Homing and Implantation Requires Wnt5a-ROR Signaling[J].Cell Reports.2014,8:382–392。
SEQUENCE LISTING
<110> 华中农业大学
<120> Wnt5a基因作为猪产仔数性状相关检测的分子标记
<130>
<141> 2016-10-20
<160> 6
<170> PatentIn version 3.1
<210> 1
<211> 366
<212> DNA
<213> 猪(Sus scrofa)
<220>
<221> gene
<222> (1)..(366)
<223>
<220>
<221> mutation
<222> (213)..(213)
<223>
<400> 1
atcacaccct gagtgtcacg aagtagcctt gtttctgagg aatctggaag gtcttactga 60
tatgcacacg tgggtttgaa gagttctttt atcgaccctg ccccatccgc tgttctccag 120
cctccttccc tgtacccttc tggacagggc attacttgtt catcagagac atgggaatga 180
agagacagtc aacacttgaa agcattgtca gcttttctct tcccttcatt ttgcagtgtg 240
gaaactggtg cccattagat ctgatagtac ttatttgagt ccccagggaa aatcctgctc 300
acgacatatc tagctagttt tgtaattttt tttttttttt tttaaaccag gacacctctt 360
tccaca 366
<210> 2
<211> 20
<212> DNA
<213> 人工序列
<220>
<221> primer_bind
<222> (1)..(20)
<223>
<400> 2
atcacaccct gagtgtcacg 20
<210> 3
<211> 20
<212> DNA
<213> 人工序列
<220>
<221> primer_bind
<222> (1)..(20)
<223>
<400> 3
tgtggaaaga ggtgtcctgg 20
<210> 4
<211> 20
<212> DNA
<213> 人工序列
<220>
<221> primer_bind
<222> (1)..(20)
<223>
<400> 4
gcacacgtgg gtttgaagag 20
<210> 5
<211> 32
<212> DNA
<213> 人工序列
<220>
<221> primer_bind
<222> (1)..(32)
<223>
<400> 5
tttaatataa cactgcaaaa tgaaggcgag ag 32
<210> 6
<211> 185
<212> DNA
<213> 猪(Sus scrofa)
<220>
<221> gene
<222> (1)..(185)
<223>
<220>
<221> mutation
<222> (150)..(150)
<223>
<400> 6
gcacacgtgg gtttgaagag ttcttttatc gaccctgccc catccgctgt tctccagcct 60
ccttccctgt acccttctgg acagggcatt acttgttcat cagagacatg ggaatgaaga 120
gacagtcaac acttgaaagc attgtcagca tttctctcgc cttcattttg cagtgttata 180
ttaaa 185
- 还没有人留言评论。精彩留言会获得点赞!