一种提高枯草芽孢杆菌普鲁兰酶分泌的方法与流程
本发明涉及一种提高枯草芽孢杆菌普鲁兰酶分泌的方法,属于发酵工程领域。
背景技术:
普鲁兰酶(EC 3.2.1.41)是一种水解普鲁兰糖、支链淀粉等分支多糖类物质中α-1,6糖苷键的脱支淀粉酶。在工业生产中普鲁兰酶通常与α-淀粉酶、β-淀粉酶、葡糖淀粉酶、环糊精葡糖苷转移酶协同作用来获得葡萄糖、果糖、麦芽糖浆、环糊精、直链淀粉等产品。由于普鲁兰酶的加入可以明显提高这些产品的产量同时缩短生产周期,因此在低分子糖浆,啤酒,酒精燃料,抗性淀粉等物质的生产企业中应用广泛。
然而普鲁兰酶在枯草芽孢杆菌中的分泌量却相对较低,例如,重组菌株Bacillus subtilisWB800-pMA0911-PUL表达了来源于Bacillus naganoensis的普鲁兰酶,其胞外普鲁兰酶酶活仅为3.9U·ml-1,将载体pMA0911中的HpaII启动子用强启动子P43替换掉,胞外酶活提高到8.7U·ml-1,但分泌量仍然较低[Wan Song,Yao Nie,Xiao Qing Mu,Yan Xu,Enhancement of extracellular expression of Bacillus naganoensispullulanase from recombinant Bacillus subtilis:Effects of promoter and host.Protein Expression and Purification,2016,124:23-31]。再如,有研究者将枯草芽孢杆菌中用于表达普鲁兰酶的启动子和信号肽都进行优化,但胞外普鲁兰酶酶活仅为2.82U·ml-1,分泌量十分有限[Y.P.Wang,Y.H.Liu,Z.X.Wang,F.P.Lu,Influence of promoter and signal peptide on the expression of pullulanase in Bacillus subtilis.Biotechnology Letter,2014,36:1783-1789],这就很大程度上限制了普鲁兰酶在工业生产中的应用。
由于非离子型表面活性剂通常具有亲水基团和疏水集团,当这些集团与枯草芽孢杆菌的细胞壁和细胞膜接近时会破坏其肽聚糖,磷脂等物质的排列方式和结构,从而使细胞的透性增大。据我们所知,尚未有通过非离子型表面活性剂的添加来提高枯草芽孢杆菌中普鲁兰酶分泌效率的报道。本研究通过一些非离子型表面活性剂的添加和浓度的优化提高了枯草芽孢杆菌中普鲁兰酶的分泌效率,降低了工业上普鲁兰酶的胞外生产纯化成本。
技术实现要素:
本发明首先提供了一种重组表达普鲁兰酶的重组枯草芽孢杆菌,是以枯草芽孢杆菌WB800为宿主,以枯草芽孢杆菌168来源的启动子ylb(F4)替换掉pP43NMK上的启动子P43得到的载体作为表达载体,表达Anoxybacillus sp.WB42来源的普鲁兰酶基因。
在本发明的一种实施方式中,所述重组表达普鲁兰酶的重组枯草芽孢杆菌的构建方法包括以下步骤:
(1)将Anoxybacillus sp.WB42来源的普鲁兰酶基因pulA和质粒pP43NMK同时用限制性内切酶KpnI和HindIII双酶切,纯化回收后的载体pP43NMK与目的基因pulA,进行连接得到pP43NMK-pulA;
(2)以枯草芽孢杆菌168来源的启动子ylb(F4)替换掉pP43NMK-pulA上的启动子P43,得到pPylbNMK-pulA;
(3)将重组质粒pPylbNMK-pulA转入枯草芽孢杆菌WB800菌株,得到重组表达普鲁兰酶的重组枯草芽孢杆菌。
本发明还提供应用所述重组枯草芽孢杆菌分泌表达普鲁兰酶的方法,是将重组枯草芽孢杆菌的种子液接种至LB液体培养基中,在35~37℃、180~200rpm培养,当菌体将要进入生长稳定期时,加入终浓度为0.4~0.6%的甜菜碱或0.2~0.4%的吐温-80或0.2~0.5%的司盘80。
在本发明的一种实施方式中,将重组枯草芽孢杆菌的种子液按接种量1%接种至LB液体培养基中,在37℃,200rpm培养,当菌体将要进入生长稳定期时,加入终浓度为0.4%的甜菜碱或0.2%的吐温-80或0.4%的司盘80。
本发明通过构建重组表达普鲁兰酶的枯草芽孢杆菌,并在以此重组菌发酵生产普鲁兰酶的过程中,添加终浓度为0.4%的甜菜碱或0.2%的吐温-80或0.4%的司盘80,普鲁兰酶的分泌效率可以达到76.13%、68.72%和72.84%,胞外pulA酶活分别达到22.95IU/ml、19.13IU/ml、20.11IU/ml。
附图说明
图1基因片段pulA PCR产物的核酸电泳图
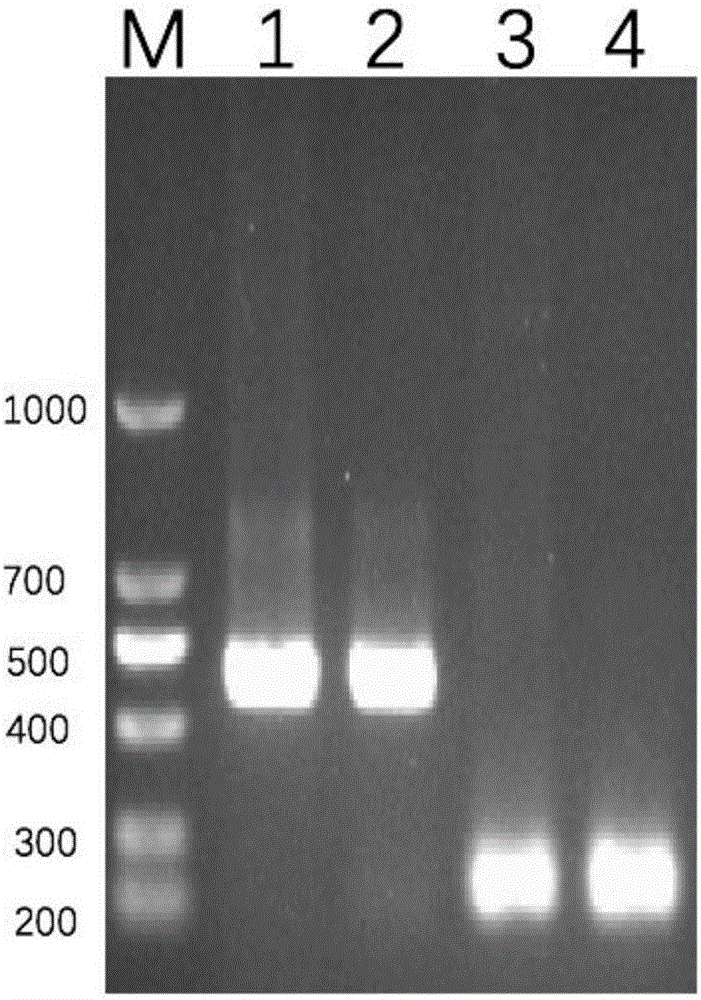
图2启动子ylb(F4)PCR产物的核酸电泳图(3,4泳道)
图3全质粒PCR产物pPylbNMK-pulA的核酸电泳图
图4添加表面活性剂后Bacillus subtilis WB800-pPylbNMK-pulA的生长曲线
图5 Bacillus subtilisWB800-pPylbNMK-pulA发酵上清液SDS-PAGE(箭头所指为目的蛋白pulA,大小约84Kd);1.对照,2.甜菜碱,3.甘氨酸,4.P-40,5.曲拉通X-100,6.吐温-80,7.十二烷基硫酸钠,8.月桂酰肌氨酸钠,9.司盘-80,10.S-185
具体实施方式
实施例1重组菌株Bacillus subtilis WB800-pPylbNMK-pulA的构建
(1)普鲁兰酶基因的克隆
根据Anoxybacillus sp.WB42来源的普鲁兰酶基因序列设计引物,引物如下:
上游引物P1:
GGGGTACCATTATAGGTAAGAGAGGAATGTACACATGCTAACGGTTCATCGGACGTTTG,酶切位点为KpnI;
下游引物P2:CCCAAGCTTTTAAGTCAGTCCTTTCACAAGCACGACTG,酶切位点为HindIII;
扩增体系:
模板1μL(Anoxybacillus sp.WB42基因组DNA)
上游引物P1 1μL
下游引物P2 1μL
dNTP(2.5mM each)4μL
5×PrimeSTAR Buffer(Mg2+Plus)10μL
PrimeSTAR HS DNA Polymerase(2.5U/μL)0.5μL
H2O 32.5μL
用于扩增目的基因的PCR条件:
将PCR扩增产物进行琼脂糖凝胶电泳检测,结果附图1,其大小与目的基因大小相似,目的基因pulA序列见序列SEQ ID NO.1。
(2)E.coli JM109-pP43NMK-pulA重组菌株的构建
用质粒小提试剂盒(TIANGEN公司)从E.coli JM109-pP43NMK中提取质粒pP43NMK(张晓舟.枯草杆菌新型表达系统和遗传操作体系的建立和应用[D].南京:南京农业大学,2006).
将上述提取的目的基因pulA和质粒pP43NMK同时用限制性内切酶KpnI和HindIII双酶切,体系如下:
KpnI 1μL
HindIII 1μL
10×QC Buffer2μL
pulA/pP43NMK 10μL人工序列
H2O 6μL
酶切于37℃,反应4-6h即可,用DNA纯化试剂盒(TOYOBO公司)对DNA进行纯化回收。
pP43NMK与目的基因pulA的连接:将上述纯化回收后的载体pP43NMK与目的基因pulA连接,体系如下:
连接酶solutionI 5μL
pP43NMK 1μL
目的基因pulA 4μL
16℃,过夜连接,转化E.coli JM109菌株,并挑取转化子进行DNA测序(上海睿迪公司)。(3)N-乙酰转移酶基因启动子ylb(F4)的克隆
参考文献中报道的启动子ylb(F4)序列(Xiaoxia Yu,Jiangtao Xu,Xiaoqing Liu,Xiaoyu Chu,Ping Wang,Jian Tian,NingfengWu&YunliuFan.Identification of a highly effici entstationary phase promoter in Bacillus subtilis.Scientific Reports.|5:18405|DOI:10.1038/srep18405(2015))设计引物,引物如下:
上游引物P3:(与载体pP43NMK的P43启动子上游同源)GACCGAGATTTTTTTGAGCAACTGGATCCACTTCTCAAAGATCCCATGTGC
下游引物P4:(与基因pulA序列的5’端反向互补)GCTTCAAACGTCCGATGAACCGTTAGCATACAAATCTCCCCCTTTGTTGTT
扩增体系:
模板1μL(Bacillus subtilis 168基因组DNA)
上游引物P3 1μL
下游引物P4 1μL
dNTP(2.5mM each)4μL
5×PrimeSTAR Buffer(Mg2+Plus)10μL
PrimeSTAR HS DNA Polymerase(2.5U/μL)0.5μL
H2O 32.5μL
用于扩增启动子ylb(F4)序列的PCR条件:
将PCR扩增产物进行琼脂糖凝胶电泳检测,结果如图2,其大小与目标序列大小相似,启动子ylb(F4)序列见SEQ ID NO.6。
(4)重组质粒pPylbNMK-pulA的构建
以上述扩增的启动子ylb(F4)片段为引物,测序正确的质粒pP43NMK-pulA为模板进行全质粒PCR,用启动子ylb(F4)替换掉pP43NMK-pulA上的启动子P43。
扩增体系:
模板1μL(质粒pP43NMK-pulA)
上下游引物(扩增得到的启动子ylb(F4)片段)2μL
dNTP(2.5mM each)4μL
5×PrimeSTAR Buffer(Mg2+Plus)10μL
PrimeSTAR HS DNA Polymerase(2.5U/μL)0.5μL
H2O 32.5μL
用于得到线性重组子pPylbNMK-pulA的PCR条件:
将PCR扩增产物进行琼脂糖凝胶电泳检测,结果如图3。
将上述得到的线性重组子pPylbNMK-pulA用限制性内切酶DpnI消化,体系如下:
DpnI 1μL
pPylbNMK-pulA 5μL
10×QC Buffer 1μL
H2O 1μL
37℃,反应10h即可。
将上述经过DpnI消化的线性重组子pPylbNMK-pulA转化E.coli JM109,并挑取转化子进行DNA测序(上海睿迪公司)。
(5)将测序正确的重组质粒pPylbNMK-pulA转入Bacillus subtilis WB800菌株。
实施例2
(1)用接种环沾取少量Bacillus subtilisWB800-pPylbNMK-pulA菌液,在含有50μg/ml卡那霉素的固体LB平板上划线,之后放到37℃培养箱中培养过夜。第二天挑取单菌落至装有10mlLB液体培养基的试管中,加入终浓度为50μg/ml的卡那霉素,在37℃,200rpm摇床上培养约10h作为种子液。
(2)将种子液以1%的接种量分别接种至50mlLB液体培养基中,加入终浓度为50μg/ml的卡那霉素,在37℃,200rpm摇床上培养并定时取样在600nm吸光值下测量菌体OD。当菌体将要进入生长稳定期时(摇瓶发酵的第21.5h)分别加入终浓度为0.2%的非离子型表面活性剂甜菜碱,甘氨酸,P-40,曲拉通X-100,吐温80,十二烷基硫酸钠,月桂酰肌氨酸钠,司盘80,S-185,菌体生长情况如图1。
(3)在摇瓶发酵的第48h分别从上述10个发酵摇瓶中取500μl菌液,在4℃,12000rpm条件下,离心10min,分离出上清液作为细胞外蛋白样品液。菌体用1M pH5.8的HAc-NaAc缓冲液洗涤3次后加入500μl pH8.0的含有200μg/ml溶菌酶的TE缓冲液重悬,并于37℃金属浴中放置2h,使枯草芽孢杆菌细胞壁充分分解,用超声破碎仪在42%功率下破碎5min,之后在4℃,12000rpm条件下,离心10min,收集上清作为细胞内蛋白样品液。
(4)用DNS显色法分别测定第48h细胞外蛋白样品液和细胞内蛋白样品液的普鲁兰酶活力,并计算出第48h相对胞外pulA酶活、相对总pulA酶活、分泌效率及相对细胞密度,结果如表1。
DNS显色法测普鲁兰酶酶活具体步骤:在1.5mlEP管中加入40μl浓度为6%的普鲁兰糖底物,再加入10μl1MpH5.8的HAc-NaAc缓冲液,放入65℃的金属浴中保温5min,快速加入20μl酶液并于旋涡振荡器混匀,65℃反应15min后加入70μlDNS试剂终止反应。将温度升高至100℃保温6min,并将最终反应液稀释72倍,用于测量其在476nm下的吸光值。普鲁兰酶活力定义为在上述特定条件下,单位体积反应液中1min内普鲁兰酶催化生成1μmol还原力的还原糖所需要的酶量。
表1 不同种表面活性剂对Bacillus subtilis WB800-pPylbNMK-pulA中pulA分泌的影响
注:相对胞外pulA酶活=胞外pulA酶活(不同种表面活性剂)/胞外pulA酶活(对照)×100%;
分泌效率=胞外pulA酶活/总pulA酶活×100%
相对总pulA酶活=总pulA酶活(不同种表面活性剂)/总pulA酶活(对照)×100%;
相对细胞密度=OD600(不同种表面活性剂)/OD600(对照)×100%
从表1中可以看出加入表面活性剂甜菜碱、吐温-80、司盘-80后,总pulA酶活较对照没有明显下降,且这三种表面活性剂对细胞的裂解作用不如甘氨酸、NP-40、TritonX-100、S-185那样强烈,对pulA的分泌效率分别达到54.21%,59.07%,62.58%,胞外pulA酶活分别达到15.17IU/ml,17.55IU/ml,16.02IU/ml。
在相同的实验条件下,我们对甜菜碱、吐温-80、司盘-80这三种表面活性剂进行浓度的调整,结果分别如下表2,3,4。
表2 不同浓度甜菜碱对B.subtilis WB800-pPylbNMK-pulA中pulA分泌的影响
注:相对胞外pulA酶活=胞外pulA酶活(不同浓度表面活性剂)/胞外pulA酶活(0.2%甜菜碱)×100%;
分泌效率=胞外pulA酶活/总pulA酶活×100%
相对总pulA酶活=总pulA酶活(不同浓度表面活性剂)/总pulA酶活(0.2%甜菜碱)×100%;
相对细胞密度=OD600(不同浓度表面活性剂)/OD600(0.2%甜菜碱)×100%
表3 不同浓度吐温-80对Bacillus subtilis WB800-pPylbNMK-pulA中pulA分泌的影响
注:相对胞外pulA酶活=胞外pulA酶活(不同浓度表面活性剂)/胞外pulA酶活(0.1%吐温-80)×100%;
分泌效率=胞外pulA酶活/总pulA酶活×100%
相对总pulA酶活=总pulA酶活(不同浓度表面活性剂)/总pulA酶活(0.1%吐温-80)×100%;
相对细胞密度=OD600(不同浓度表面活性剂)/OD600(0.1%吐温-80)×100%
表4不同浓度司盘-80对Bacillus subtilis WB800-pPylbNMK-pulA中pulA分泌的影响
注:相对胞外pulA酶活=胞外pulA酶活(不同浓度表面活性剂)/胞外pulA酶活(0.1%司盘-80)×100%;
分泌效率=胞外pulA酶活/总pulA酶活×100%
相对总pulA酶活=总pulA酶活(不同浓度表面活性剂)/总pulA酶活(0.1%司盘-80)×100%;
相对细胞密度=OD600(不同浓度表面活性剂)/OD600(0.1%司盘-80)×100%
从上述表中可以看出表面活性剂甜菜碱、吐温-80、司盘-80的添加浓度分别为0.4%、0.2%、0.4%时对pulA的分泌效果最优,其中0.4%甜菜碱,0.2%吐温-80,0.4%司盘-80对pulA的分泌效率可以达到76.13%,68.72%和72.84%,胞外pulA酶活分别达到22.95IU/ml,19.13IU/ml,20.11IU/ml。
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
SEQUENCE LISTING
<110> 江南大学
<120> 一种提高枯草芽孢杆菌普鲁兰酶分泌的方法
<160> 6
<170> PatentIn version 3.3
<210> 1
<211> 2136
<212> DNA
<213> Anoxybacillus sp.WB42
<400> 1
atgctaacgg ttcatcggac gtttgaagca tatttagata caatggaaac gattacgatt 60
ttagtaccga aatcgtattg ccaagggata gtgcgctcat ttaccatcca aacgccaaac 120
ggagaacgac gcgcactaca aattacgaaa cgcgaagatt tatggacgag cattaaatat 180
gagtgcatcc ccgatgttcc ggtggaaatc ggaaaaagtt atttcattta tgaagaacat 240
ggggcgttta ccgatttgca aatgggagcg gttattcgca ccgaagcatt tgatgagcag 300
ttttattatg acggtgacct tggcattacg tacagcaacg atgccacaac gtttaaactt 360
tgggcaccga cagcaacaga ggtaaaacta aagctcatta acaaagcagg aaaagaagaa 420
caaatctcga tgcagcgcgg agaaaaaggg gtatggtgcg cgacagtact cggtaattta 480
gatgggatat actatacgta tttagcgtgt attaaccttg tttggcgaga agcggtcgac 540
ccgtatgctg tagcggtatc ggtgaatgga gaatacgggg tcgtcgtcga tctcgccaaa 600
acgcgagtgc ccaaaccagc gctacctccg cttactgccc cagtcgatgc cattatttat 660
gaaatgcacg ttcgtgattt tacaattgat cctcatagcg gcgttgtcca taaaggaaag 720
tatctaggac tgactgaatt tccaacaaca gaaccgtctg gaacagtgac ggggcttgct 780
tatttgaaac aacttggggt tacacacgta gagctgcttc cggtgaacga ttttgccggg 840
gtcgatgaac gtgaaccgga aaaagagtac aactgggggt acaatccgct tcactacaac 900
gcgccggaag gaagttatgc gaccgatcct tacgatccgt atgcgcgtat tcatgaattg 960
aaacgagcaa ttcgcgcttt gcagcaagaa ggtattcgtg tcattctcga tgtcgtttat 1020
aaccacgttt acattcgcga acagtcgtca ctagaaaaac tcgtgcctgg gtattatttt 1080
cgccatgata tttacggtat gccatcgaac gggacagggg tcggaaatga tcttgccccc 1140
gagcgcaaaa tggttcgcaa gctcattgtc gattccgttc gtttctggct tacagagtat 1200
ggcattgatg ggtttcgttt tgatttaatg ggcattctcg atattgacac gatgaaagaa 1260
gtcgaggcag ttgttcgtgc gctccatcca tccgctctat tgctcggcga aggatgggat 1320
ttgccgacac cattgccgtc ggaaaaaaaa gcaacgatgc aaaatgccca ccttctgccg 1380
acgattgcct tttttaacga tcggtttcgc gattatgtca aagggagtac gtttcattta 1440
ggggaacaag gatttgttct aggaaacagc gcacatcgcg aacaagtgaa acgagtaatc 1500
gaaggaagcc atcatttgtt ttctcaacca acgcaaacgg ttaactacgt cgaatcacac 1560
gacaaccata cgctttggga caaaatgagc atcgccaact attacgagcg agaaaccatt 1620
cgtaaaaaac gacaaaaatt agcgacagcg atgaccttat tagcacaagg cattccattt 1680
ttgcatagcg gccaagaatt ttaccgtaca aaacaaggag tagaaaatag ttataacgct 1740
ccagatgaca ttaaccgcat cgattggacg agaaaaagca tgcacgaaca agacgtccgt 1800
tacgtgcaag gattgattcg gctgcggaaa tggcacggtg cttttcgttt tcaaacagtg 1860
gaagaaataa gaaaccatct tgtatggctt gaaccgatgc cgtcgacagt gctcgctttt 1920
catctttacg acgtatcagc gtatgggccg tggcgtgata ttattgtcat tcaccataat 1980
gaagaaacac ggctagcagt tgcgctccct gatgaagaaa gatggtatgt cgtatgtgat 2040
gaaacaagga gtggaatcga tcctctttac gcggcgacaa aaaaaatcga gctgcaagga 2100
attggaacag tcgtgcttgt gaaaggactg acttaa 2136
<210> 2
<211> 59
<212> DNA
<213> 人工序列
<400> 2
ggggtaccat tataggtaag agaggaatgt acacatgcta acggttcatc ggacgtttg 59
<210> 3
<211> 38
<212> DNA
<213> 人工序列
<400> 3
cccaagcttt taagtcagtc ctttcacaag cacgactg 38
<210> 4
<211> 51
<212> DNA
<213> 人工序列
<400> 4
gaccgagatt tttttgagca actggatcca cttctcaaag atcccatgtg c 51
<210> 5
<211> 51
<212> DNA
<213> 人工序列
<400> 5
gcttcaaacg tccgatgaac cgttagcata caaatctccc cctttgttgt t 51
<210> 6
<211> 103
<212> DNA
<213> Bacillus subtilis 168
<400> 6
acttctcaaa gatcccatgt gcttaaaatt aaagtttaaa tatttggatt ttttaaataa 60
agcgtttaca atatatgtag aaacaacaaa gggggagatt tgt 103
- 还没有人留言评论。精彩留言会获得点赞!