一种凡纳滨对虾血蓝蛋白抗菌肽及其应用的制作方法
本发明涉及水产养殖的技术领域,尤其涉及一种凡纳滨对虾血蓝蛋白抗菌肽及其应用。
背景技术:
近年来,随着经济的发展,人们对水产品的需求日益增加,在捕捞量不能满足市场需求的情况下,水产养殖业得到了迅猛的发展。对虾是水产养殖中增长最快的产品之一,我国是养殖对虾年产量最高的国家。然而,与国际对虾养殖业相比,我国、我省对虾养殖业相对效益较低、发展后劲不足,可持续发展前景不容乐观。究其原因,主要与目前我国对虾养殖病害频发和生物安全所致对虾出口屡遭贸易壁垒等有关。
血蓝蛋白又称血蓝素,是一种多功能蛋白,过去被称为呼吸蛋白,它是位于节肢动物和软体动物血淋巴中的含铜呼吸蛋白,是节肢动物和软体动物血淋巴中发现的含铜的蓝绿色高分子量蛋白质,是氧的传递体,并且是已知的能与氧气可逆结合的唯一铜蛋白。
抗菌肽(antibac-terialpeptide)又叫抗微生物肽(antimicrobialpep-tide)、抗生素肽(antibioticspeptide),是由多种生物细胞特定基因编码经外界条件诱导产生的一类具有广谱抗细菌、真菌、病毒、原虫、抑杀肿瘤细胞等活性作用的多肽。迄今为止,国内外报道大约有2000多种抗菌肽被鉴定、分离出来,以天然抗菌肽作模板进行人工合成的模拟肽已达数千种。在生物界自然存在的抗菌肽根据其结构的不同可以分为四大类:β折叠型抗菌肽、α螺旋型抗菌肽、延伸结构型多肽和loop结构型多肽。
目前,我国对虾养殖业急需解决的共性关键问题包括:对虾传染性疫病的防控、品种的选育、营养研究、养殖环境控制以及对虾加工技术升级等。而要成功解决这些问题,阐明对虾免疫防御机制是前提和基础。有鉴于此,本发明人研究和设计了一种凡纳滨对虾血蓝蛋白抗菌肽及其应用,本案由此产生。
技术实现要素:
本发明的目的在于提供一种凡纳滨对虾血蓝蛋白抗菌肽及其应用,通过凡纳滨对虾血蓝蛋白抗菌肽s10构效关系的研究,为寻找新的对虾病害防控、良种选育的策略提供思路,促进我国对虾养殖业的健康、持续发展。
为了解决上述目的,本发明采用的技术方案如下:
一种凡纳滨对虾血蓝蛋白抗菌肽,其氨基酸序列如序列表seqidno:1所示。
作为实施例的优选方式,所述抗菌肽的分子量为1736da。
一种凡纳滨对虾血蓝蛋白抗菌肽的应用,在制备治疗或预防细菌的药物中的应用。
作为实施例的优选方式,所述细菌包括溶藻酸弧菌、副溶血弧菌、大肠杆菌、嗜水气单胞菌、金黄色葡萄球菌、河弧菌及乙型链球菌。
本发明以凡纳滨对虾血蓝蛋白为研究对象,凡纳滨对虾的血蓝蛋白能产生功能性的抗菌肽,并且通过生物信息学在线预测肽段发现血蓝蛋白亚基中的12种肽段具有抗菌活性的可能性最大。在此基础之上选择一种肽段s10,用圆二色谱法得出最大吸收波长下的吸光度,再用cdpro软件计算出α-螺旋和β-折叠等结构的含量,因此初步预测s10的二级结构。然后研究s10对于七种水产致病菌的抗菌活性,最后先用pymol软件对该肽进行3d模型预测,再用核磁共振法验证其二级结构。实验结果表明,该肽对七种致病菌都具有抑制作用。同时核磁共振法得到结构与软件预测基本一致。本发明为进一步研究凡纳滨对虾的免疫防御机制提供了基础。
附图说明
图1抗菌肽s10的3d模型;
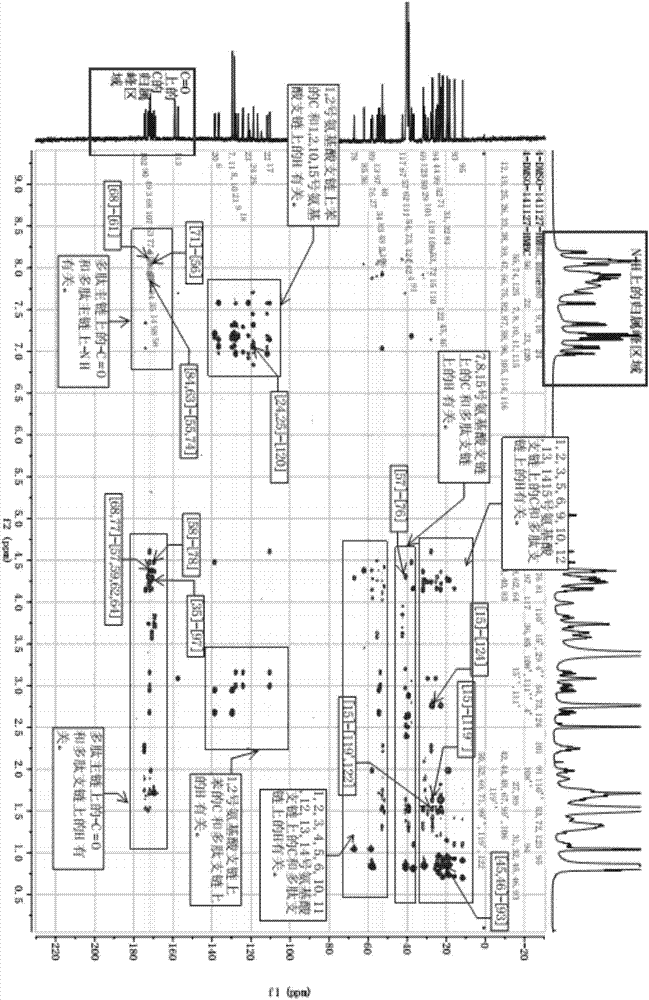
图2抗菌肽s10的hmbc谱;其中,横坐标为h谱,纵坐标为c谱;
图3抗菌肽s10的noesy谱;其中,横纵坐标皆为h谱;
图4为本发明抗菌肽s10可能的空间结构。
具体实施方式
实施例1凡纳滨对虾血蓝蛋白抗菌肽的预测
首先运用antibpserver、camp、apd2三个在线预测软件,预测凡纳滨对虾血蓝蛋白大、小亚基抗菌肽段,如表1、表2所示,总共预测得到34条可能具有抗菌活性的小分子片段,其中11条多肽序列(黑体加粗表示)可能会形成α螺旋结构,且能与病原菌膜相互作用而发挥抑菌作用,这些预测多肽分子量大小范围为1.5~1.9kda。另外,预测结果显示,在血蓝蛋白n端的α螺旋域、c端的ig-like域以及中间部分的保守铜离子结合域均有活性片段,其中铜离子结合域和ig-like域占主要区域。
表1凡纳滨对虾血蓝蛋白大亚基抗菌肽段的预测
注:1、加粗多肽序列:根据其理化性质推测可能形成α螺旋结构,且能与病原菌膜相互作用发挥抑菌作用;2、ⅰdomain:α螺旋域;ⅱdomain:铜离子结合域;ⅲdomain:ig-like域。
表2凡纳滨对虾血蓝蛋白小亚基抗菌肽段预测
注:1、加粗多肽序列:根据其理化性质推测可能形成α螺旋结构,且能与病原菌膜相互作用发挥抑菌作用;2、ⅰdomain:α螺旋域;ⅱdomain:铜离子结合域;ⅲdomain:ig-like域。
实施例2采用圆二色谱法分析凡纳滨对虾血蓝蛋白抗菌肽二级结构含量
将实施1所述的11种(表1和表2中的加粗肽段)可能会形成α螺旋结构,且能与病原菌膜相互作用而发挥抑菌作用的肽段进行圆二色谱扫描,分析其结构含量。
2.1实验器材
2.1.1实验材料
实验用抗菌肽由北京中科亚光生物科技有限公司(scilightbiotechnology,llc)合成,多肽n端乙酰化、c端酰胺化修饰,纯度为﹥95%。
2.1.2实验仪器
紫外分光光度计(uv5200)厂家:上海元析仪器有限公司
圆二色谱仪(jascoj-810)厂家:日本jasco
比色皿:1mm石英比色皿
2.2样品制备
利用紫外分光光度计在扫描范围是190nm-400nm中扫描11个样品的最长吸收波长,往样品中逐步加水直至吸光度为1,记录数据为圆二色谱作前期准备。
2.3样品测试步骤
2.3.1、开启仪器
开启电脑,完全开启氮气瓶总阀,打开减压阀,使表头保持在0.1pa。开启仪器侧面的氮气流量计,是气流保持在3-5l/min。打开仪器电源,仪器开始点灯(约需0.5min),待仪器面板上“open”灯亮后,双击“spectramanager”图标,在spectramanager对话框中选择所需的测量法“spectrummeasurement”,仪器开始自我检查,约2min后,自检后,出现spectrummeasurement对话框,根据测试要求设置参数。
2.3.2、参数设置
parameters设置参数:
波长范围:190-250nm
数据采集点:0.5nm
scanningspeed:100nm/min
scanningmode:continuous
狭峰宽度:1nm
datamode设置:cd或fdcd(荧光cd)测试方法
option:选择注解有关参数
2.3.3、样品测试
将配制好的样品装进1mm石英比色皿中,放入样品架中,溶液样品架放在靠光圈侧,盖上盖板,点击start开始测试,当波长扫描至400nm一下,开启氮气保护,扫描结束自动生成spectraanalysis对话框。
2.3.4、谱图处理
点击生成的对话框中file菜单,选择saveas将cd谱图保存生成.jws文件。
2.3.5、关机
退出cd程序后关闭仪器主机电源,然后关去氮气瓶总阀,排出残留氮气,最后关闭仪器上的氮气流量计。
2.4数据处理
1、利用cdpro软件,把cd数据降序导出到记事本,另存为.inp格式,将其复制到cdpro文件中,先运行crdata.exe,在运行sellon3.exe执行计算,结果打开sellon3.out查看。
2、计算方法:
α螺旋含量=h(r)+h(d);
β折叠=s(r)+s(d);
转角=trn;
无规则=unrd。
2.5实验数据
表3对11个肽的cdpro软件处理数据
2.6抗菌肽段选取
本发明从表3的11种肽段选取其中的抗菌肽s10进行进一步的构效关系研究。从表3得知,抗菌肽s10的α螺旋含量为20.6%、β折叠含量为15.1%、β转角含量为26.1%,说明肽s10的15个氨基酸残基中α螺旋结构的含量只有3个氨基酸残基,不能构成α螺旋结构,β转角含量有4个氨基酸残基,可以形成β转角结构。
实施例3采用平板计数法验证抗菌肽s10的抑菌活性
3.1操作步骤
实验用抗菌肽由北京中科亚光生物科技有限公司(scilightbiotechnology,llc)合成,多肽n端乙酰化、c端酰胺化修饰,纯度为﹥95%。
对虾血蓝蛋白化学合成抗菌肽段抑菌活性分析采用以下方法进行,具体流程如下:
(1)先将置于-80℃保存的七种水产致病菌(溶藻酸弧菌、副溶血弧菌、大肠杆菌、嗜水气单胞菌、金黄色葡萄球菌、河弧菌、乙型链球菌)分别进行小量活化(50μl菌种+1ml肉汤液体培养基),接着在37℃恒温条件下用摇床培养6-12h直至对数生长期(或者也可以在28℃下培养大约24h);
(2)分别取活化后的菌液100μl于离心小管中,再加入900μl无菌水进行稀释,即为10-1倍,依次稀释至10-6—10-7倍。
(3)将稀释后的菌液10μl与1.0mg/ml、0.5mg/ml、0.25mg/ml、0.125mg/ml不同组分的多肽样液10μl混匀,放于30℃的恒温水浴锅当中进行2h的孵育,之后吸取10μl的混合液涂于平板,每组设置2个平行,在37℃的培养条件下倒置,培养12—24h后对菌落进行计数(以无菌水作为阴性对照)。
(4)计算抑菌率(%)=(阴性对照菌落数-实验组菌落数)/阴性对照菌落数×100。
3.2结果分析
表4肽s10对七种致病菌的抑菌率(%)
由表4可知,肽s10对七种致病菌均具有抑制作用,其中对副溶血弧菌、乙型链球菌、河弧菌、大肠杆菌、嗜水气单胞菌有较强的抑制作用,对金黄色葡萄球菌和溶藻酸弧菌的抑制作用相对较弱。
实施例4采用核磁共振法研究抗菌肽s10二级结构
4.1抗菌肽s10的3d模型预测
通过网站http://swissmodel.expasy.org对肽s10的氨基酸序列进行三维结构预测,选择相似度较高的三维结构图,下载.pdb文件,在pymol软件中打开进行查看与编辑,得到肽s10的3d模型图如图1所示。图中最右侧部分预测肽s10可能含有转角结构,图中中间部分预测可能为平行。进一步的结构预测需要通过核磁共振法进行验证。
4.2核磁共振法预测抗菌肽s10的二级结构
4.2.1
将样品送到中科新生命公司,获得抗菌肽s10的c谱、h谱、hmbc谱(代表c与h的远程关系)、noesy谱(代表h与h的远程关系)。
4.2.2
根据肽s10的氨基酸序列用chemoffice软件画出其化学结构式,并应用mestrenova软件将化学结构式自动归属于c谱、h谱上,从而得出各个c、h所对应的峰的位置。
4.2.3
在mestrenova软件中打开抗菌肽s10的c谱、h谱、hmbc谱和noesy谱,将h谱作为hmbc谱的横坐标,c谱作为hmbc谱的纵坐标,h谱作为noesy谱的横纵坐标,导入化学式进行归属,在hmbc谱上分析h与远程c的可能相关关系,在noesy谱上分析h与远程h的可能相关关系。
4.3核磁共振法预测抗菌肽s10二级结构的结果分析
4.3.1抗菌肽s10的化学式
抗菌肽s10的肽段序列:fwvslkggktsierk,翻译成三字符号为:
phe-trp-val-ser-leu-lys-gly-gly-lys-thr-ser-ile-glu-arg-lys。
4.3.2抗菌肽s10的nmr谱图
抗菌肽s10的二级结构解析主要分析nmr谱图中的hmbc谱和noesy谱。
核磁共振法的图谱分析和数据处理结果如图2、图3所示。
从图2hmbc谱图可以得出以下信息:
1、第1、2、3、5、6、9、10、12、13、14、15号氨基酸支链上的c与多肽支链上的h可能有联系;
2、第1、2、3、4、5、6、10、11、12、13、14号氨基酸支链上的c与多肽支链上的h可能有关系;
3、第1、2号氨基酸支链上苯的c与1、2、10、15号氨基酸支链上的h可能有联系;
4、第1、2号氨基酸支链上苯的c与多肽支链上的h可能有关系;
5、第7、8、15号氨基酸支链上的c与多肽支链上的h可能有联系;
6、第55、74号氨基酸上的c与第63、84号氨基酸上的h可能有关系;
7、第56号氨基酸上的c与第71号氨基酸上的h可能有关系;
8、第57、59、62、64号氨基酸上的c与第68、77号氨基酸上的h可能有关系;
9、第61号氨基酸上的c与第68号氨基酸上的h可能有关系;
10、第76号氨基酸上的c与第57号氨基酸上的h可能有关系;
11、第78号氨基酸上的c与第58号氨基酸上的h可能有关系;
12、第93号氨基酸上的c与第45、46号氨基酸上的h可能有关系;
13、第97号氨基酸上的c与第35号氨基酸上的h可能有关系;
14、第119、122、124号氨基酸上的c与第15号氨基酸上的h可能有关系;
15、第120号氨基酸上的c与第24、25号氨基酸上的h可能有关系;
16、多肽主链上的-c=o与多肽支链上的h可能有关系。
从图3noesy谱图可以得出以下信息:
1、第116号氨基酸上的h与第15号氨基酸上的h可能有关系;
2、第110号氨基酸上的h与第31、32号氨基酸上的h可能有关系;
3、第108、111号氨基酸上的h与第27号氨基酸上的h可能有关系;
4、第97号氨基酸上的h与第36号氨基酸上的h可能有关系;
5、第91号氨基酸上的h与第45、46号氨基酸上的h可能有关系;
6、第76号氨基酸上的h与第110号氨基酸上的h可能有关系;
7、第62、64号氨基酸上的h与第69、71、72号氨基酸上的h可能有关系;
8、第61、103号氨基酸上的h与第36、62号氨基酸上的h可能有关系;
9、第34、40、83号氨基酸上的h与第48、50、52、93、89、99号氨基酸上的h可能有关系。
4.3.3讨论
4.3.3.1α螺旋、β折叠、β转角结构的特征:
α螺旋:规则排布的氢键使得螺旋结构稳定,氢键排布的方式是:每一个氨基酸残基的n—h和其氨基侧相隔三个氨基酸残基的c=o之间形成氢键。
β折叠:肽键的平面折叠呈锯齿状,相邻肽链主链的n-h和c=o间具有规则的氢键,在β-折叠当中,所有的肽键均与链间氢键的形成有关,β-折叠的长轴和氢键之间是垂直关系。
β转角:转角i的特点是:第4个残基的酰胺氮和第1个氨基酸残基羰基氧形成氢键;转角ⅱ的特点是:第3个残基一般是甘氨酸。
4.3.3.2
从肽s10的hmbc谱图可以得出:在s10肽段序列的第6个至第9个氨基酸中,74号h与63号c之间以及61号h与68号c之间、56号h与71号c之间存在关系点,而且s10肽段序列的第8个氨基酸为甘氨酸,满足β转角特征依据,可以推断出s10肽段序列的第6个至第9个氨基酸之间形成β转角。其他点表示这各个c与h之间远程的关系,影响着s10肽段的空间结构。例如,1、2、3、5、6、9、10、12、13、14、15号氨基酸支链上的c和多肽支链上的h有关,说明存在相互作用力,导致肽链的两端相互靠近,所以可以推出肽链在空间结构上不是一条笔直的直线,而是存在弧度。
从肽s10的noesy谱图可以得出:第5个氨基酸和第6个氨基酸上的h与h之间存在关系作用点,第8个氨基酸与第9个氨基酸上的h与h之间存在关系作用点,因此进一步证明了β转角可能存在;第1至5个氨基酸上的h与第10至15个氨基酸上的h之间存在关系作用点,进一步证明了s10肽段的肽链在空间结构上不是一条直线。结果如图4所示。
总之,本发明采用圆二色谱法测定出肽s10的α螺旋含量为20.6%、β折叠含量为15.1%、β转角含量为26.1%,说明肽s10的15个氨基酸残基中α螺旋结构的含量只有3个氨基酸残基不能构成α螺旋结构,β转角含量有4个氨基酸残基,可以形成β转角结构;抑菌实验验证抗菌肽s10对七种致病菌均具有抑制作用,其中对副溶血弧菌、乙型链球菌、河弧菌、大肠杆菌、嗜水气单胞菌有较强的抑制作用,对金黄色葡萄球菌和溶藻酸弧菌的抑制作用相对较弱;通过3d模型和核磁共振方法,预测肽s10可能含有β转角结构,两端可能为平行,类似u型;通过预测推出s10较强的抑菌活性可能与形成β转角的空间结构有关,后续可以通过替换或者破坏肽s10中的β转角结构,对新合成肽的抗菌活性进行实验,从而进一步验证这一结论。
以上所述,仅为本发明较佳实施例而已,故不能依此限定本发明实施的范围,即依本发明专利范围及说明书内容所作的等效变化与修饰,皆应仍属本发明涵盖的范围内。
- 还没有人留言评论。精彩留言会获得点赞!
- 凡纳滨对虾功能基因EST‑SSR标记及其特异性引物和检测方法与流程
- 一种用于凡纳滨对虾遗传性别鉴定的DNA探针序列及获取方法与流程
- 一种凡纳滨对虾发酵饲料及其生产方法与流程
- 凡纳滨对虾Na,K‑ATPase α亚基基因及SNP标记筛查的扩增引物和方法与流程
- 高密度养殖凡纳滨对虾专用复合微量元素营养剂及其制备工艺的制造方法与工艺
- 一种凡纳滨对虾亲虾投喂方法与流程
- 一种利用海藻下脚料制作的凡纳滨对虾环保配合饲料及其制备方法与流程
- 一种与凡纳滨对虾耐低盐性状相关的SNP标记、扩增引物及其应用的制造方法与工艺
- 高密度养殖凡纳滨对虾专用复合维生素营养剂及其制备方法与流程
- 一种来源于凡纳滨对虾血蓝蛋白的抗肿瘤肽及其应用的制造方法与工艺