一种RNA甲基化酶TRDMT1基因敲除的HEK293细胞系及其构建方法与流程
本发明涉及基因工程领域,具体涉及一个利用CRISPR/Cas9系统构建RNA甲基化酶TRDMT1基因敲除的HEK293细胞系的方法。
背景技术:
哺乳动物中TRDMT1(tRNA aspartic acid methyltransferase 1)是RNA介导的表观遗传的一个必须成分。TRDMT1之前被认为是DNA甲基转移酶,最近的研究表明其功能主要为RNA甲基转移酶。RNA甲基化作为普遍的表观遗传标记,在基因调控中发挥关键作用的。至今为止,已经发现RNA存在100多种修饰,其中甲基化是主要的修饰形式,6-甲基腺嘌呤(m6A)和5-甲基胞嘧啶(m5C)是其中最具有代表性的两种修饰。有研究指出,这种修饰可以稳定tRNA的二级结构,影响氨酰化的形成及密码子的识别。此外,m5C修饰还存在于rRNA结合tRNA发挥翻译活性的区域。由于检测技术的限制,真核生物mRNA一开始被认为是缺乏m5C修饰的,直到第一次全转录组m5C定位发现,m5C在人类mRNA和ncRNA中是一种重要的修饰。近年来越来越多的证据表明,m5C修饰对mRNA结构的稳定及功能的调节起重要的作用。
CRISPR/Cas9系统是第三代人工核酸内切酶,与第一代锌指核酸内切酶(ZFNs)以及第二代类转录激活因子效应物核酸酶(TALEN)相比,更简便,成本低,更容易在实验室使用。CRISP/Cas9系统原理是成熟的crRNA和traRNA结合,形成gRNA。gRNA定位到靶序列,介导Cas9蛋白识别PAM序列,在目的片段进行切割,形成DNA双链(Double-strand break,DBS)缺口,启动基因组自我修复功能,进行非同源末端链接以及同源重组,来实现对基因组基因定点敲除、特异性突变以及定点修饰。本研究所用HEK293细胞系,即人胚肾细胞系,是293细胞的一种衍生株,来源于人胚肾,正常的细胞形态为上皮样,贴壁生长。该细胞系极少表达细胞外配体所需的内生受体,且比较容易转染,是一个很常用的表达外源基因的细胞株。本发明利用CRISPR/Cas9系统的精确靶向性,构建RNA甲基化酶TRDMT1基因敲除的HEK293细胞系,为RNA甲基化研究提供一种有效的工具。
技术实现要素:
本发明目的是提供一种RNA甲基化酶TRDMT1基因敲除的HEK293细胞系及其构建方法。其主要原理是:利用CRISPR/Cas9技术在基因组通过gRNA定点结合,Cas9蛋白精确切割形成双链缺口,通过同源和非同源重组,来实现移码突变,从而达到基因组层面敲除。而在细胞中通过同源重组的方式定向敲入转录终止信号,终止基因的表达,达到敲除的效果。同时通过抗性基因进行快速筛选,从而建立稳定敲除的细胞系。所获得细胞系,性状稳定,为进一步为研究TRDMT1基因功能提供可靠的细胞模型,也为RNA甲基化功能研究提供一种有效的方法。
为了实现上述目的,本发明的技术方案如下:
一种RNA甲基化酶TRDMT1基因敲除的HEK293细胞系的构建方法,通过两个分别包含抗生素筛选基因的同源重组载体、gRNA载体TRDMT1-gRNA,以及表达Cas9的载体混合转染HEK293细胞,将两个抗生素筛选基因和转录终止信号片段整合到TRDMT1基因的第一外显子前,以终止其转录,通过抗性筛选,获得RNA甲基化酶TRDMT1基因敲除的HEK293细胞系。
本发明方法中,抗生素筛选基因同源重组载体包括插入片段,所述的插入片段包括剪切受体、自我剪切2A肽、转录终止信号片段、抗生素筛选基因;所述转录终止信号片段位于抗生素筛选基因下游。
所述的gRNA载体中,gRNA序列为:GCTGGAGCTATACAGCGGCG(SEQ ID NO.15)。
优选地,抗生素筛选基因同源重组载体,是两个载体TRDMT1-LOXN-Puro和TRDMT1-LOXP-Neo,所述的载体TRDMT1-LOXN-Puro中,抗生素筛选基因是序列如SEQ ID NO.35所示的嘌呤霉素抗性基因PuroR;所述的载体TRDMT1-LOXP-Neo中,抗生素筛选基因是序列如SEQ ID NO.34所示的新霉素抗性基因NeoR。
所述自我剪切2A肽为T2A,其核苷酸序列为SEQ ID NO.36。
所述转录终止信号为BGH polyA,其核苷酸序列为SEQ ID NO.37。
所述的Cas9载体为MLM3613。
所述的抗生素筛选同源重组载体是利用BamBHⅠ可以允许不同的位点以不同的黏性末端连接的特性,采用一步法构建。
具体地,本发明方法包括发下步骤:
1)构建分别含有两种不同抗生素基因的同源重组载体:将靶点左端同源臂TRDMT1-LR、筛选序列Puromycin、转录终止序列BGH poly(A)和靶点右同源臂TRDMT1-RR构进载体骨架T-Easy-Vector-multi-5-BamHⅠ中,形成TRDMT1-LOXN-Puro同源重组载体。以相同的方法构建TRDMT1-LOXP-Neo同源重组载体;
2)设计gRNA,构建含有靶向基因的TRDMT1-gRNA载体:利用重叠PCR方法合成gRNA序列,根据gRNA骨架载体T-Vector-U6-gRNA-is-C连接末端序列的特点,进行退火磷酸化得到含有粘性末端的片段gRNA,将含有粘性末端的片段gRNA连接到gRNA骨架载体上,得到TRDMT1-gRNA载体;
3)将步骤1)中所获得的含有抗生素基因的一对同源重组载体TRDMT1-LOXN-Puro和TRDMT1-LOXP-Neo、步骤2)中所获得的含有靶向基因的TRDMT1-gRNA载体,以及Cas9表达载体MLM3613,4个载体共转染HEK293细胞;
4)抗性筛选:将步骤3)转染的细胞用Puromycin和Neomycin抗生素同时进行筛选,最终获得稳转细胞株,从而建立TRDMT1双等位基因敲除的HEK293稳转细胞系。
本发明还提供了由上述方法构建获得的细胞系。
本发明的有益效果在于:
1)传统的特定基因表达的沉默,通过干扰途径实现:如RNA干扰、MO干扰,也有通过ZFN、TALEN等实现敲除,本研究使用的CRISPR/Cas9技术,可以精确的进行定点编辑;
2)获得了一种RNA甲基化酶TRDMT1基因敲除的HEK293细胞系,细胞特性稳定,有利于进行TRDMT1蛋白功能的研究;
3)构建了一种通过在基因序列第一个外显子前插入转录终止信号,达到敲除细胞系的方法。
附图说明
图1为LOXN-Puro结构示意图以及PCR扩增引物位置示意图。
图2为靶位点左端同源臂TRDMT1-LR结构示意图以及PCR扩增引物位置示意图。
图3为靶位点右端同源臂TRDMT1-RR结构示意图以及PCR扩增引物位置示意图。
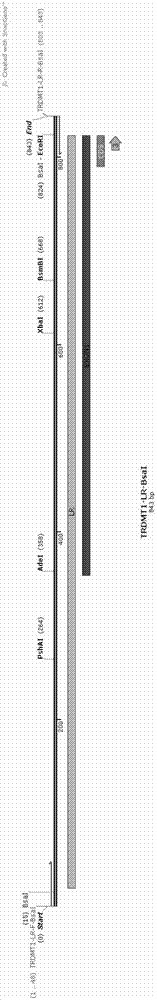
图4为TRDMT1-LOXN-Puro载体图谱,包括启动子T7、SP6、lac,靶点左端同源臂TRDMT1-LR,右端同源臂TRDMT1-RR、抗性基因Puro以及转录终止序列BGH poly(A)。
图5为LOXP-Neo结构示意图以及PCR扩增引物位置示意图。
图6为TRDMT1-LOXP-Neo载体图谱。包括启动子T7、SP6、lac,靶点左端同源臂TRDMT1-LR,右端同源臂TRDMT1-RR、抗性基因Neo以及转录终止序列BGH poly(A)。
图7为gRNA和检测引物位置图谱。
图8为载体TRDMT1-gRNA载体图谱。包括启动子T7、SP6以及gRNA。
图9为不同gRNA引导定位效果示意图,图中第一种野生型条带:477bp,突变型条带:178bp+299bp。第二种野生型条带:734bp;突变型条带:181bp+553bp。
图10为不同细胞株TRDMT1mRNA相对表达量。
图11为不同时期同一细胞株TRDMT1mRNA相对表达。
具体实施方式
为了阐明本发明的技术方案及技术目的,下面结合附图及具体实施方式对本发明做进一步的介绍。下述实施例中的方法,如无特别说明,均为常规方法。
实施例1
构建一种RNA甲基化酶TRDMT1基因敲除的HEK293细胞系的方法。构建分别含有两种不同抗生素基因的同源重组载体TRDMT1-LOXN-Puro和TRDMT1-LOXP-Neo。利用重叠PCR方法合成gRNA序列,构建含有靶向基因的TRDMT1-gRNA载体。将所获得的含有抗生素筛选基因的一对同源重组载体TRDMT1-LOXN-Puro和TRDMT1-LOXP-Neo,含有靶向基因的TRDMT1-gRNA载体,以及Cas9载体MLM3613,四个载体共转染HEK293细胞。将转染的细胞添加Puromycin和Neomycin抗生素进行抗性筛选,最终获得稳转HEK293-TRDMT1Knockout细胞株,从而建立TRDMT1基因敲除的HEK293细胞系,命名为HEK293-TKO细胞系。具体步骤如下:
1.构建TRDMT1-LOXN-Puro同源重组载体
1.1构建同源重组片段LOXN-Puro
利用PCR的方法在Puro-BGH poly(A)片段5’端添加T2A(SEQ ID No.36)和LOXN序列(剪切受体),构建同源重组片段LOXN-Puro(图1)。引物序列如表1:
表1
以pSMPUW-IRES-Puro(CELL BIOLABS,INC.VPK-215)为模板,使用高保真酶(Super-Fidelity DNA Polymerase,Vazyme)扩增Puro-BGH poly(A)片段,并与T2A、LOXN拼接。其反应体系包括:30-50ng pSMPUW-IRES-Puro模板,1μL(10μM)上游引物(Puro-T2A、HELPER-T2A-1、HELPER-T2A-2、T2A-loxN-F)与1μL(10μM)下游引物BGH-Xba1-R别作为扩增引物,0.5μLSuper-Fidelity DNA Polymerase,5μL 5×SF Buffer(with 10Mm MgSO4),0.5μL(10Mm each)dNTP Mix和ddH2O补足25μL。其反应条件为:95℃3min;95℃15s,60℃30s,72℃30s,35循环;72℃5min;4℃维持。
1.2扩增左端同源臂TRDMT1-LR
利用引物扩增靶位点左端同源臂TRDMT1-LR(图2)。引物序列如表2:
表2
以HEK293细胞(ATCC,CRL-1573)基因组为模板,使用高保真酶(Super-Fidelity DNA Polymerase,诺维赞生物公司)扩增TRDMT1-LR片段。其反应体系包括:50-250ng HEK293基因组模板,1μL(10μM)上游引物TRDMT1-LR-F-BsaI与1μL(10μM)下游引物TRDMT1-LR-R-BsaI分别作为扩增引物,0.5μLSuper-Fidelity DNA Polymerase,5μL 5×SF Buffer(with 10Mm MgSO4),0.5μL(10Mm each)dNTP Mix和ddH2O补足25μL。其反应条件为:95℃3min;95℃15s,60℃30s,72℃30s,35循环;72℃5min;4℃维持。
1.3扩增右端同源臂TRDMT1-RR
利用引物扩增靶位点右端同源臂TRDMT1-RR(图3)。引物序列如表3:
表3
以HEK293基因组为模板,使用高保真酶(Super-Fidelity DNA Polymerase,诺维赞生物公司)扩增TRDMT1-RR片段。其反应体系包括:50-250ngHEK293基因组模板,1μL(10μM)上游引物TRDMT1-LR-F-BsaI与1μL(10μM)下游引物TRDMT1-RR-R-BsaI分别作为扩增引物,0.5μLSuper-Fidelity DNA Polymerase,5μL 5×SF Buffer(with10Mm MgSO4),0.5μL(10Mm each)dNTP Mix和ddH2O补足25μL。其反应条件为:95℃3min;95℃15s,60℃30s,72℃30s,35循环;72℃5min;4℃维持。
1.4一步法构建同源重组载体TRDMT1-LOXN-Puro
T-Easy-Vector-multi-5-BamHⅠ载体制备:IRES2从pIRES2-EGFP载体(Clontech,6029-1)扩增,引物Multi5-F:TGGCTCTAGAGACGGATAAGGCCGGTGTGCGTTTGT(SEQ ID No.10),Multi5-R:CGGATCCGCACATGTAAAGCATGTGCACC(SEQ ID No.11)。TA克隆连接T载体(Promega,A1360)得T-Easy-Vector-multi-5-BamHⅠ。
T-Easy-Vector-multi-5-BamHⅠ载体用BsmBⅠ和BamHⅠ进行双酶切,TRDMT1-LR用BsaⅠ进行酶切,LOXN-PURO用EcoRⅠ和XbaⅠ进行酶切,TRDMT1-RR用XbaⅠ和BamHⅠ切。通过简单的一步法构建同源重组载体,用T4连接酶连接,目的片段及载体骨架之间摩尔比为1:1:1:1,16℃过夜链接。
对连接产物进行转化,用M13F、M13R(金唯智公司)对单克隆菌进行PCR检验,将获得的阳性克隆送公司测序,获得TRDMT1-LOXN-Puro载体(图4)。
2.构建TRDMT1-LOXP-Neo同源重组载体
2.1构建同源重组片段LOXP-Neo
利用PCR的方法在Neo-BGH poly(A)片段5’端添加T2A(SEQ ID No.36)和LOXP序列(剪切受体),构建同源重组片段LOXP-Neo(图5)。引物序列如表4:
表4
以pcDNA3.1(+)(Invitrogen,V790-20)为模板,使用高保真酶(Super-Fidelity DNA Polymerase,诺维赞生物公司)扩增Neo-BGH poly(A)片段,并与T2A、LOXP拼接。其反应体系包括:30-50ng pcDNA3.1(+)模板,1μL(10μM)上游引物(Neo-T2A、HELPER-T2A-1、HELPER-T2A-2、T2A-loxP-F)与1μL(10μM)下游引物BGH-Xba1-R别作为扩增引物,0.5μLSuper-Fidelity DNA Polymerase,5μL 5×SF Buffer(with 10Mm MgSO4),0.5μL(10Mm each)dNTP Mix和ddH2O补足25μL。其反应条件为:95℃3min;95℃15s,60℃30s,72℃30s,35循环;72℃5min;4℃维持。
2.2扩增左端同源臂TRDMT1-LR
利用引物扩增靶位点左端同源臂TRDMT1-LR(图2)。引物序列如表5:
表5
以HEK293基因组为模板,使用高保真酶(Super-Fidelity DNA Polymerase,诺维赞生物公司)扩增TRDMT1-LR片段。其反应体系包括:50-250ng HEK293基因组模板,1μL(10μM)上游引物TRDMT1-LR-F-BsaI与1μL(10μM)下游引物TRDMT1-LR-R-BsaI分别作为扩增引物,0.5μLSuper-Fidelity DNA Polymerase,5μL 5×SF Buffer(with10Mm MgSO4),0.5μL(10Mm each)dNTP Mix和ddH2O补足25μL。其反应条件为:95℃3min;95℃15s,60℃30s,72℃30s,35循环;72℃5min;4℃维持。
2.3扩增右端同源臂TRDMT1-RR
利用引物扩增靶位点右端同源臂TRDMT1-RR(图3)。引物序列如表6:
表6
以HEK293基因组为模板,使用高保真酶(Super-Fidelity DNA Polymerase,诺维赞生物公司)扩增TRDMT1-RR片段。其反应体系包括:50-250ngHEK293基因组模板,1μL(10μM)上游引物TRDMT1-LR-F-BsaI与1μL(10μM)下游引物TRDMT1-RR-R-BsaI分别作为扩增引物,0.5μLSuper-Fidelity DNA Polymerase,5μL 5×SF Buffer(with10Mm MgSO4),0.5μL(10Mm each)dNTP Mix和ddH2O补足25μL。其反应条件为:95℃3min;95℃15s,60℃30s,72℃30s,35循环;72℃5min;4℃维持。
2.4一步法构建同源重组载体TRDMT1-LOXP-Neo
T-Easy-Vector-multi-5-BamHⅠ载体用BsmBⅠ和BamHⅠ进行双酶切,TRDMT1-LR用BsaⅠ进行酶切,LOXN-PURO用EcoRⅠ和XbaⅠ进行酶切,TRDMT1-RR用XbaⅠ和BamHⅠ切。通过简单的一步法构建同源重组载体。
2.5用T4连接酶连接,目的片段及载体骨架之间摩尔比为1:1:1:1,16℃过夜链接。
2.6对连接产物进行转化,用M13F、M13R对单克隆菌进行PCR检验,将获得的阳性克隆送公司测序,获得TRDMT1-LOXP-Neo载体(图6)。
3.构建TRDMT1-gRNA载体
3.1设计针对TRDMT1的gRNA
根据人TRDMT1(ENSG00000107614)第一段外显子基因序列分析得到gRNA(图7),选取评分较高的4个,序列分别如表7:
表7
3.2合成gRNA的模版
制备gRNA骨架载体T-Vector-U6-gRNA-is-C。U6-gRNA从pX330-U6-Chimeric_BB-CBh-hSpCas9载体(addgene,42230)扩增,引物U6-F:TGTACAAAAAAGCAGGCTTTA(SEQ ID No.18),U6-R:CACTAGTGATTTAATGCCAACT(SEQ ID No.19)。TA克隆连接T载体(Promega,A1360)得到T-Vector-U6-gRNA-is-C。
利用重叠PCR方法合成4个gRNA序列,50μL反应体系:gRNA上下游引物(序列如表8(10umol/μL)各2μL、退火Buffer 46μL。反应程序:95℃金属水浴锅热激5min后,自然降至室温。根据gRNA骨架载体T-Vector-U6-gRNA-is-C链接末端的序列特点,进行退火磷酸化得到含有黏性末端的片段gRNA,将含有粘性末端的片段gRNA连接到gRNA骨架载体上,得到TRDMT1-gRNA载体(图8),对应4个gRNA,将获得的4个载体分别命名为TRDMT1-gRNA1、TRDMT1-gRNA2、TRDMT1-gRNA3和TRDMT1-gRNA4。
表8
3.3 gRNA定位效率验证
分别将4个TRDMT1-gRNA载体与Cas9表达载体MLM3613(购买于addgene公司)混合共转染
HEK293细胞中,每组2个重复,转染72小时提取基因组,以其为模板,以表9中的TRDMT1-Test1-F/R和TRDMT1-Test2-F/R为引物分别进行PCR扩增并对产物进行PCR退火处理。用T7核酸内切酶对1μgPCR产物进行酶切,用1%琼脂糖跑电泳检测4个gRNA的定位效果。结果(图9)显示gRNA2效率最高,故将其用于后续实验。检测引物如图7。表9
4.转染HEK293细胞
将HEK293细胞铺至6孔板中,将质粒Cas9表达载体MLM3613、gRNA载体、TRDMT1-LOXN-Puro和TRDMT1-LOXP-Neo按照1:1:1:1,每种质粒各按1ug通过Lip2000Transfection Reagent转染试剂共转染HEK293细胞,共4个重复。同时转染Egfp观察转染效率,未转染的作为对照组,转染6小时换成含10%血清的MEM。
5.筛选获得TRDMT1基因去表达的稳定细胞株
5.1筛选HEK293细胞对抗生素Puromycin和Neomycin的敏感浓度
Puromycin和Neomycin抗生素在高浓度情况下对细胞具有杀伤作用。转染后,其携带Puromycin和Neomycin的抗性基因,能抵抗Puromycin和Neomycin对细胞的杀伤作用。筛选出HEK293敏感浓度,可以使阴性细胞死亡,保留阳性细胞。将HEK293铺至12孔板中,加入不同浓度的Puromycin,连续每天观察细胞状态,获取能够在7天有效时间内将细胞完全杀死的抗生素浓度。并将此浓度定位后期HEK293细胞的敏感浓度,实验中检测到的HEK293敏感浓度为0.5-1mg/ml。用上述同样的方法检测到HEK293对Neomycin的敏感浓度为400mg/ml。
5.2筛选稳转细胞株
细胞转染72小时后,1:3对细胞进行传代。使用Puromycin和Neomycin两个抗性基因筛选便于得到双等位基因敲出的细胞系。由于Neomycin药筛作用较慢,Puromycin对细胞毒性大,药筛作用快,因此先用Puromycin进行抗性筛选,易筛除未转的正常HEK293细胞。待细胞贴壁,加0.5-1mg/ml的Puromycin进行抗性筛选。筛选两天,换成0.5-1mg/ml的Puromycin和400mg/ml的Neomycin进行药筛,每隔两天换一次培养基,药筛至对照组细胞全部死亡后,实验组培养基换成一半浓度Puromycin和一半浓度Neomycin维持敲入基因的稳定。待实验组存活的单克隆细胞株长到一定程度,用克隆环转移至新的培养皿中。当单克隆扩大培养足够量时,消化一部分提取RNA,进行反转录,通过RT-qPCR方法检测TRDMT1的转录水平。最终获得了RNA甲基化酶TRDMT1双等位基因敲除的HEK293细胞株,即HEK293-TRDMT1Knockout细胞株,命名为HEK293-TKO细胞系。
实施例2:TRDMT1去表达细胞系筛选
在加入Puromycin和Neomycin抗生素筛选1个月后,用克隆环挑取单克隆细胞。用记号笔在培养皿底部圈出单克隆细胞,去除培养基,将克隆环置于单克隆细胞上,滴入1滴胰酶,在显微镜下观察(室温大概1min)。保证不移动克隆环,用培养基吹打细胞,转移至24孔板中。单克隆细胞扩大培养过程中,加入0.25-0.5mg/ml的Puromycin和200mg/ml的Neomycin。
实施例3:TRDMT1去表达细胞系鉴定
RT-qPCR检测各筛选克隆株中TRDMT1mRNA表达量,从mRNA水平对细胞基因组进行鉴定1目标细胞RNA提取
用酚氯仿抽提法提取疑似正确细胞系的RNA,提取方法如下:
a.消化细胞,离心去除培养基,加入适量TRIzol充分裂解细胞。
b.加入TRIzol试剂后,室温静置5min。再加入1/5体积TRIzol的三氯甲烷,振荡混匀,室温静置5min。
c.用4℃离心机以12000g离心15min,吸取上清,加入等体积的异丙醇,混匀。
d.用4℃离心机以12000g离心10min,弃上清,加入1ml 75%乙醇洗涤两次。
e.用4℃离心机以12000g瞬离,吸除剩余75%乙醇,在超净台中通风干燥,根据RNA白色沉淀的量加入RNase-Free水,在DNA/RNA定量仪测浓度后保存于-80℃。
2TaKaRa的反转录试剂盒合成cDNA
2.1将获得RNA反转录为cDNA
按照表10去除基因组DNA体系配置混合液,混匀,42℃孵育2min。
表10去除基因组DNA反应体系
按照表11反转录反应体系配置混合液,PCR反应程序为:37℃,15min;85℃,5s;4℃,∞。
表11反转录反应体系
2.2TRDMT1基因的表达水平检测
用qPCR的Master Mix试剂进行RT-qPCR检测TRDMT1基因的表达水平。根据RNA甲基化酶TRDMT1的基因结构,用primer5.0设计qPCR引物,引物序列如表12:
表12
qPCR反应体系配制如表13:
表13 qPCR反应体系配制
qPCR反应程序如表14:
表14 qPCR反应程序
RT-qPCR结果(图10)显示:单克隆细胞株TRDMT1基因发生了转录终止,去表达效率高达99.94%,最终获得了RNA甲基化酶TRDMT基因敲除的HEK293细胞系。后期对不同时间同一TRDMT1基因敲除的HEK293细胞系进行了TRDMT1表达水平检测,检测结果表明TRDMT1基因敲除稳定,进一步证明获得了稳定的HEK293细胞系(图11)。
以上描述了本发明的基本原理、主要特征和本发明的优点。本行业的技术人员应该了解,本发明不受上述实施例的限制,上述实施例和说明书中描述的只是说明本发明的原理,在不脱离本发明精神和范围的前提下,本发明还会有各种变化和改进,本发明要求保护范围由所附的权利要求书、说明书及其等效物界定。
SEQUENCE LISTING
<110> 扬州大学
<120> 一种RNA甲基化酶TRDMT1基因敲除的HEK293细胞系及其构建方法
<130>
<160> 37
<170> PatentIn version 3.3
<210> 1
<211> 46
<212> DNA
<213> 人工序列
<400> 1
gtgacgtcga ggagaatcct ggcccaatga ccgagtacaa gcccac 46
<210> 2
<211> 48
<212> DNA
<213> 人工序列
<400> 2
gagggcagag gaagtctgct aacatgcggt gacgtcgagg agaatcct 48
<210> 3
<211> 27
<212> DNA
<213> 人工序列
<400> 3
gttatgggag ggcagaggaa gtctgct 27
<210> 4
<211> 55
<212> DNA
<213> 人工序列
<400> 4
tgcgaattca taacttcgta tagtatacct tatacgaagt tatgggaggg cagag 55
<210> 5
<211> 26
<212> DNA
<213> 人工序列
<400> 5
gctctagacc atagagccca ccgcat 26
<210> 6
<211> 48
<212> DNA
<213> 人工序列
<400> 6
tttgctcagg tctcactcta aacattttac ggagtttggc ttttttcg 48
<210> 7
<211> 41
<212> DNA
<213> 人工序列
<400> 7
atgtaacggg tctcgaattc gctgtatagc tccagcaccc g 41
<210> 8
<211> 40
<212> DNA
<213> 人工序列
<400> 8
tttgctcgaa gacctctaga cgtgggcggc atgcaccacg 40
<210> 9
<211> 40
<212> DNA
<213> 人工序列
<400> 9
gagcaaggat ccgtttgctt gataatatct cgtgatgtcg 40
<210> 10
<211> 36
<212> DNA
<213> 人工序列
<400> 10
tggctctaga gacggataag gccggtgtgc gtttgt 36
<210> 11
<211> 29
<212> DNA
<213> 人工序列
<400> 11
cggatccgca catgtaaagc atgtgcacc 29
<210> 12
<211> 50
<212> DNA
<213> 人工序列
<400> 12
gtgacgtcga ggagaatcct ggcccaatga ttgaacaaga tggattgcac 50
<210> 13
<211> 55
<212> DNA
<213> 人工序列
<400> 13
tgcgaattca taacttcgta tagcatacat tatacgaagt tatgggaggg cagag 55
<210> 14
<211> 20
<212> DNA
<213> 人工序列
<400> 14
tgtatagctc cagcacccgc 20
<210> 15
<211> 20
<212> DNA
<213> 人工序列
<400> 15
gctggagcta tacagcggcg 20
<210> 16
<211> 20
<212> DNA
<213> 人工序列
<400> 16
aagctgtata cctgcacaag 20
<210> 17
<211> 20
<212> DNA
<213> 人工序列
<400> 17
ctgtatacct gcacaagtgg 20
<210> 18
<211> 21
<212> DNA
<213> 人工序列
<400> 18
tgtacaaaaa agcaggcttt a 21
<210> 19
<211> 22
<212> DNA
<213> 人工序列
<400> 19
cactagtgat ttaatgccaa ct 22
<210> 20
<211> 25
<212> DNA
<213> 人工序列
<400> 20
caccgtgtat agctccagca cccgc 25
<210> 21
<211> 25
<212> DNA
<213> 人工序列
<400> 21
aaacgcgggt gctggagcta tacac 25
<210> 22
<211> 24
<212> DNA
<213> 人工序列
<400> 22
caccgctgga gctatacagc ggcg 24
<210> 23
<211> 24
<212> DNA
<213> 人工序列
<400> 23
aaaccgccgc tgtatagctc cagc 24
<210> 24
<211> 25
<212> DNA
<213> 人工序列
<400> 24
caccgaagct gtatacctgc acaag 25
<210> 25
<211> 25
<212> DNA
<213> 人工序列
<400> 25
aaaccttgtg caggtataca gcttc 25
<210> 26
<211> 25
<212> DNA
<213> 人工序列
<400> 26
caccgctgta tacctgcaca agtgg 25
<210> 27
<211> 25
<212> DNA
<213> 人工序列
<400> 27
aaacccactt gtgcaggtat acagg 25
<210> 28
<211> 22
<212> DNA
<213> 人工序列
<400> 28
caaagaagga aatggagcgt aa 22
<210> 29
<211> 20
<212> DNA
<213> 人工序列
<400> 29
ctgtttccag gacaaccgag 20
<210> 30
<211> 23
<212> DNA
<213> 人工序列
<400> 30
ttcaaatatt tcacttcatg cct 23
<210> 31
<211> 22
<212> DNA
<213> 人工序列
<400> 31
tcagtgtttc aatacttgtg ct 22
<210> 32
<211> 25
<212> DNA
<213> 人工序列
<400> 32
aagcctgttt gtcattctgt atccc 25
<210> 33
<211> 25
<212> DNA
<213> 人工序列
<400> 33
aaggccatag cacttaaaga cccat 25
<210> 34
<211> 795
<212> DNA
<213> 人工序列
<400> 34
atgattgaac aagatggatt gcacgcaggt tctccggccg cttgggtgga gaggctattc 60
ggctatgact gggcacaaca gacaatcggc tgctctgatg ccgccgtgtt ccggctgtca 120
gcgcaggggc gcccggttct ttttgtcaag accgacctgt ccggtgccct gaatgaactg 180
caggacgagg cagcgcggct atcgtggctg gccacgacgg gcgttccttg cgcagctgtg 240
ctcgacgttg tcactgaagc gggaagggac tggctgctat tgggcgaagt gccggggcag 300
gatctcctgt catctcacct tgctcctgcc gagaaagtat ccatcatggc tgatgcaatg 360
cggcggctgc atacgcttga tccggctacc tgcccattcg accaccaagc gaaacatcgc 420
atcgagcgag cacgtactcg gatggaagcc ggtcttgtcg atcaggatga tctggacgaa 480
gagcatcagg ggctcgcgcc agccgaactg ttcgccaggc tcaaggcgcg catgcccgac 540
ggcgaggatc tcgtcgtgac ccatggcgat gcctgcttgc cgaatatcat ggtggaaaat 600
ggccgctttt ctggattcat cgactgtggc cggctgggtg tggcggaccg ctatcaggac 660
atagcgttgg ctacccgtga tattgctgaa gagcttggcg gcgaatgggc tgaccgcttc 720
ctcgtgcttt acggtatcgc cgctcccgat tcgcagcgca tcgccttcta tcgccttctt 780
gacgagttct tctga 795
<210> 35
<211> 600
<212> DNA
<213> 人工序列
<400> 35
atgaccgagt acaagcccac ggtgcgcctc gccacccgcg acgacgtccc cagggccgta 60
cgcaccctcg ccgccgcgtt cgccgactac cccgccacgc gccacaccgt cgatccggac 120
cgccacatcg agcgggtcac cgagctgcaa gaactcttcc tcacgcgcgt cgggctcgac 180
atcggcaagg tgtgggtcgc ggacgacggc gccgcggtgg cggtctggac cacgccggag 240
agcgtcgaag cgggggcggt gttcgccgag atcggcccgc gcatggccga gttgagcggt 300
tcccggctgg ccgcgcagca acagatggaa ggcctcctgg cgccgcaccg gcccaaggag 360
cccgcgtggt tcctggccac cgtcggcgtc tcgcccgacc accagggcaa gggtctgggc 420
agcgccgtcg tgctccccgg agtggaggcg gccgagcgcg ccggggtgcc cgccttcctg 480
gagacctccg cgccccgcaa cctccccttc tacgagcggc tcggcttcac cgtcaccgcc 540
gacgtcgagt gcccgaagga ccgcgcgacc tggtgcatga cccgcaagcc cggtgcctga 600
<210> 36
<211> 54
<212> DNA
<213> 人工序列
<400> 36
gagggcagag gaagtctgct aacatgcggt gacgtcgagg agaatcctgg ccca 54
<210> 37
<211> 223
<212> DNA
<213> 人工序列
<400> 37
ctgtgccttc tagttgccag ccatctgttg tttgcccctc ccccgtgcct tccttgaccc 60
tggaaggtgc cactcccact gtcctttcct aataaaatga ggaaattgca tcgcattgtc 120
tgagtaggtg tcattctatt ctggggggtg gggtggggca ggacagcaag ggggaggatt 180
gggaagacaa tagcaggcat gctggggatg cggtgggctc tat 223
- 还没有人留言评论。精彩留言会获得点赞!
- 基于纳米颗粒团聚的dna去甲基化酶的生物传感方法
- 一种多肽——葡萄糖基转移酶蛋白-25.52和编码这种多肽的多核苷酸的制作方法
- 用于抑制法呢基蛋白转移酶的苯并(5
- 作为组蛋白脱甲基酶抑制剂的取代的(e)-n’-(1-苯基亚乙基)苯甲酰肼类似物的制作方法
- 利用雷帕霉素特异性甲基化酶标记雷帕霉素的制作方法
- 转录因子creb在调控jhdm2a基因表达及减少猪脂肪沉积中的应用的制作方法
- 一种新的多肽——dna甲基化酶13和编码这种多肽的多核苷酸的制作方法
- (3r)-去-o-甲基毛狄泼老素在制备预防或治疗抑郁症的药物中的用途的制作方法
- 法呢基蛋白转移酶的新的苯基取代的三环抑制剂的制作方法
- 新的法呢基-蛋白转移酶的三环磺酰胺抑制剂的制作方法
- 一种新的多肽——dna甲基化酶13和编码这种多肽的多核苷酸的制作方法
- 具有治疗活性的环亚甲基-1,2-二羧酸酰胺,制备它们的方法和含它们的药物组合物的制作方法
- 组蛋白去乙酰化酶抑制剂的制作方法
- 基于dna聚合酶链反应的甲基化dna检测方法
- 检测Cre位点特异重组酶活性的转基因报告猪及培养方法
- 一种多肽——人含胞嘧啶特异性dna甲基化酶特征性序列片段的蛋白-9.35和编码这种 ...的制作方法
- 一种多肽——脱氧核糖核酸甲基化酶-9.35和编码这种多肽的多核苷酸的制作方法
- 利用酶活性的竞争性干扰和恢复检测被分析物的制作方法
- 一种基于化学发光反应的甲基化酶检测探针、检测试剂盒及检测方法
- 编码甲基化儿茶素生物合成酶的基因的制作方法