人工优化合成的Pat#基因与重组载体以及改变作物抗性的方法与流程
本公开涉及一种人工优化合成的Pat#基因(草铵膦N-乙酰基转移酶基因),含有该基因的重组载体,以及利用所述含有该基因的重组载体改变作物除草剂抗性的方法。
背景技术:
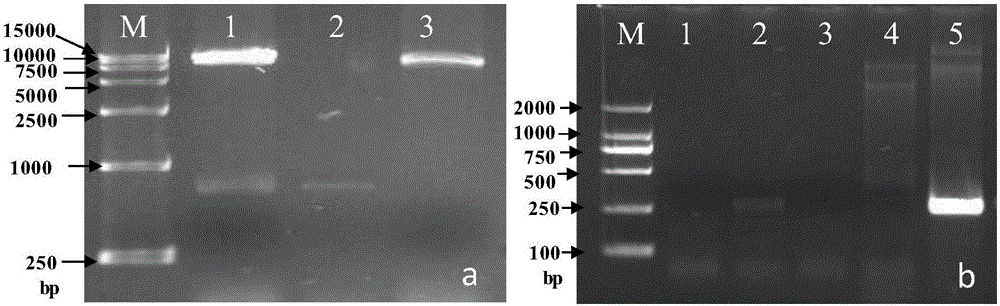
:草铵膦(Glufosinate,Phosphinothricin)是德国赫斯特(Hoechst)公司开发的一种高效、广谱、低毒、低残留的非选择性除草剂,又称为草丁膦(Phosphinothricin,PPT),IUPAC标准名称:2-氨基-4[羟基(甲基)氧膦基]丁酸(2-amino-4-(hydroxy(methyl)phosphonoyl)butanoicacid),易溶于水,不易溶于有机溶剂,对光稳定,在pH5~9的水溶液中易水解。草铵膦可通过植物蒸腾作用在木质部进行传导,生效时间长于百草枯、短于草甘膦,高温、高湿、高光强可以增进草铵膦吸收。在田间条件下,由于草铵膦迅速被土壤微生物降解,故根系吸收不到或吸收很少。草铵膦类除草剂的靶标酶是谷氨酰胺合成酶(Glutaminesynthetase,GS)。谷氨酰胺合成酶存在于所有植物组织中,在植物的氮同化及氮代谢调节中起重要作用,它催化无机氮向有机氮的转化反应,该酶同时也是植物中唯一的铵解毒酶,能够解除由硝酸盐还原、氨基酸降解及光呼吸中释放出的铵的毒性。草铵膦能够特异性地抑制植物的谷氨酰胺合成酶的活性,导致植物体内氮代谢紊乱,过量积累铵,进而使叶绿体解体,光合作用受到抑制,最终导致植物死亡。自从转基因作物商业化以来,抗草铵膦作物面积增长迅速,带动了草铵膦类除草剂销售额的增长。预计到2015年,草铵膦类除草剂的销量将突破10亿美元,在除草剂中仅次于草甘膦(杨益军,2013)。草铵膦乙酰基转移酶(Phosphinothricinacetyltransferase,PAT)基因Pat的序列长度为552bp,序列中G+C碱基含量占总碱基含量的70.1%,密码子第三位G+C含量达93.5%,且起始密码子为GTG,属于典型的产绿色链霉菌(Streptomycesviridochromogenes)的起始密码,与一般真核生物的起始密码子完全不同,编码氨基酸长度为183aa,与抗草铵膦基因Bar的同源性很高,但两者间5’端非编码区域存在调控上的差别(Wohlleben等,1988)。根据一般的生物学原理和研究实践,原始Pat基因起始密码子为GTG,与植物的常用起始密码子ATG不同,且基因中的其它密码子也不符合植物使用密码子的偏好性,用原始Pat基因转化获得的转基因作物对草铵膦的抗性不强,没有生产应用价值,必须经过密码子优化后才能在作物体内高效表达、获得高的除草剂草铵膦抗性。技术实现要素:本公开的目的是获得在高等植物(作物)中能高效表达的草铵膦类除草剂抗性基因以及利用该基因改良转基因植物(作物)对草铵膦类除草剂抗性的方法。为了实现上述目的,本公开提供一种人工优化合成的Pat#基因(草铵膦N-乙酰基转移酶基因),含有该基因的重组载体,以及利用含有该基因的载体改变作物抗性的方法。本公开提供了一种Pat#基因,其为密码子优化的Pat基因,其中,该Pat#基因的序列为SEQIDNo.1所示的序列,且该Pat#基因能编码具有草铵膦N-乙酰基转移酶活性的多肽。本公开还提供了一种Pat#基因,其为密码子优化的Pat基因,其中,该Pat#基因的序列具有信号肽的编码序列和SEQIDNo.1所示的序列,且该Pat#基因能编码N端或/和C端带有信号肽且具有草铵膦N-乙酰基转移酶活性的多肽。本公开还提供了一种Pat#基因,其为密码子优化的Pat基因,其中,该Pat#基因的序列具有提高Pat#基因翻译效率的序列和如上所述的Pat#基因的序列,且该Pat#基因能编码具有草铵膦N-乙酰基转移酶活性的多肽。本公开还提供了一种Pat#基因,其为密码子优化的Pat基因,其中,该Pat#基因的序列为如上所述的Pat#基因的5’端和3’分别连接上相同或不同的内切酶识别序列后得到。本公开还提供了一种Pat#基因表达框,其中,该Pat#基因表达框含有如上所述的Pat#基因、在该Pat#基因的5’端上连接有能够被RNA聚合酶特异识别和高效结合的启动子和在该Pat#基因的3’端上连接有能够给予RNA聚合酶转录终止信号的终止子。本公开还提供了一种Pat#基因重组载体,其中,该Pat#基因重组载体由如上所述的Pat#基因表达框插入骨架载体,得到能够使Pat#基因表达出具有草铵膦N-乙酰基转移酶活性多肽的重组载体。此外,本公开还提供一种改变作物抗性的方法,其中,该方法包括将上述的重组载体转化所述作物,使得到的转基因作物表达Pat#基因、产生具有草铵膦N-乙酰基转移酶活性的多肽并赋予作物除草剂抗性。通过上述技术方案,本公开能够使得转基因水稻至少耐受1000mg/L浓度的草铵膦,因此有效地提高了转基因水稻对草铵膦的抗性。本公开的其他特征和优点将在随后的具体实施方式部分予以详细说明。附图说明附图是用来提供对本公开的进一步理解,并且构成说明书的一部分,与下面的具体实施方式一起用于解释本公开,但并不构成对本公开的限制。在附图中:图1是植物表达载体pC3300-Pat#的构建流程图。图2是表达载体pC3300-Pat#的酶切和PCR鉴定图。a是表达载体pC3300-Pat#的XhoI酶酶切验证图:泳道M为DL15000DNA分子量标记;泳道1为pC3300-Pat#质粒的XhoI酶切片段;泳道2为Pat#基因回收片段;泳道3为pCAMBIA3300质粒经XhoI酶切后回收的大片段。b是表达载体pC3300-Pat#正反向连接PCR鉴定图:泳道M为DL2000DNA分子量标记;泳道1为空白对照;泳道2、3为pCAMBIA3300质粒;泳道4为pMD19T-Pat#质粒;泳道5为pC3300-Pat#质粒。图3a是转基因植株中Pat#基因的PCR检测电泳图:泳道M为DL2000DNA分子量标记;泳道P为质粒pC3300-Pat#阳性对照;泳道B为不加模板的阴性对照;泳道CK为非转基因植株9K阴性对照;泳道1-5为转基因水稻Pat9K的不同单株。图3b是转基因植株中Pat#基因的Southern杂交图:泳道M为DL15000DNA分子量标记;泳道P为质粒pC3300-Pat#阳性对照;泳道CK为非转基因植株9K阴性对照;泳道1-5为转基因水稻Pat9K的不同单株。图4是转基因植株Pat9K和非转基因植株9K中Pat#基因表达产物的相对实时定量PCR检测结果。图5是转基因水稻Pat9K的T2代植株喷洒草铵膦的实验结果。图6是转基因水稻Pat9K的T2代植株的种子在含草铵膦培养基上萌发的实验结果。具体实施方式以下结合附图对本公开的具体实施方式进行详细说明。应当理解的是,此处所描述的具体实施方式仅用于说明和解释本公开,并不用于限制本公开。本公开提供一种Pat#基因,其为密码子优化的Pat基因,其中,该Pat#基因具有SEQIDNo.1所示的序列,且该Pat#基因能编码具有草铵膦N-乙酰基转移酶活性的多肽。所述Pat#基因按照模式植物水稻中粳稻日本晴蛋白质编码基因序列中偏爱密码子的分布原则,首先将原Pat基因序列(NCBI登录号:M22827.1)中的部分稀有密码子置换为水稻中使用频率高的密码子,包括将起始密码子由GTG改为ATG;同时分析发现,原始Pat基因中存在1处潜在的内含子切割识别序列,利用同义密码子替换法,消除内含子切割识别序列;此外原始Pat基因GC含量达到70.1%,具有多个mRNA发夹环结构,起始自由能达到-268.90kcal/mol;在不改变优化基因的氨基酸序列的前提下,利用同义密码子替换法来消除形成的稳定发夹环等mRNA二级结构。与此同时,为了降低DNA序列甲基化的可能性,降低了Pat#基因中密码子GCG、ACG、CCG和TCG的使用频率。优化前和优化后的密码子的相对使用度(RSCU值)如表1中第3列和第5列所示。对优化后的Pat#基因进行分析发现,与水稻和原始Pat基因序列相比,优化后的Pat基因序列的密码子使用频率与水稻模式植物中粳稻日本晴的表达蛋白编码基因序列的密码子使用频率基本一致;与原始Pat基因相比,Pat#基因mRNA二级结构得到优化,起始自由能降到-202.4kcal/mol。对如上所述可能影响Pat基因在模式植物水稻细胞高效表达的密码子进行优化后获得本公开的Pat#基因序列如SEQIDNo.1所示。获得的如SEQIDNo.1所示的Pat#基因序列长度为552bp,与原始Pat基因的DNA序列相比,氨基酸组成和顺序不变,改变碱基101个,占碱基总数的18.3%,更改密码子92个,占基因密码子总数的50.0%,G+C含量从原来的70.1%降为61.41%。根据本公开,在具有SEQIDNo.1所示的序列的前提下,只要该基因能编码具有草铵膦N-乙酰基转移酶活性的多肽,可以在Pat#基因的两端添加任意类型和数目的其他DNA,这些基因也应在本公开的范围内。例如,可在Pat#基因的5’和/或3’端上连接适当的信号肽编码序列,以实现该蛋白质在细胞内或细胞间的转运、定位、分泌、储存等。定位信号肽能够使PAT蛋白产生后定向运输到细胞的特定部位,如:叶绿体、内质网和液泡等,这些特定区域能够为PAT蛋白提供一个相对稳定的环境,有效防止PAT蛋白的降解,可明显提高PAT蛋白的稳定性和积累量,从而提高植物由PAT蛋白所赋予的除草剂抗性。根据本公开的优选的一种实施方式,本公开还提供了一种Pat#基因,其为密码子优化的Pat基因,该Pat#基因的序列具有信号肽的编码序列和SEQIDNo.1所示的序列,且该Pat#基因能编码N端和/或C端带有信号肽且具有草铵膦N-乙酰基转移酶活性的多肽。优选地,所述信号肽编码序列为叶绿体转运肽TP的编码序列。更优选地,所述信号肽编码序列如SEQIDNo.2所示。本公开对于SEQIDNo.2所示的序列与SEQIDNo.1所示的序列的连接方式没有特别的限定,只要二者位于同一读码框,能够表达N端带有TP转运肽的、具有草铵膦N-乙酰基转移酶活性的多肽即可。更进一步优选地,具有信号肽编码序列的Pat#基因的序列为SEQIDNo.3所示,该多肽的氨基酸序列为SEQIDNo.4所示的序列。具体地,所述Pat#基因的5’端有叶绿体转运肽TP的编码序列(如SEQIDNo.2所示),且该Pat#基因能编码N端带有叶绿体转运肽TP、具有草铵膦N-乙酰基转移酶活性的多肽。其中,TP转运肽能够使PAT蛋白靶向性的运输到细胞的叶绿体内。通过以上步骤确定如SEQIDNo.3所示的序列,该优化基因能够编码N端带有TP转运肽的、具有草铵膦N-乙酰基转移酶活性的多肽。在TP转运肽的引导下,PAT蛋白靶向性的运输到细胞的叶绿体内。叶绿体内是草铵膦N-乙酰基转移酶作用的场所,可明显提高PAT蛋白的稳定性和积累量,从而提高植物相对应的抗性水平。但是,本领域技术人员可以确定的是,SEQIDNo.3所示的DNA序列仅是本公开的一种优选实施方式,本公开所述能够编码带有信号肽的、具有草铵膦N-乙酰基转移酶活性的多肽的序列并不限于SEQIDNo.3所示的DNA序列,其中,SEQIDNo.3所示的DNA序列表达出蛋白质的氨基酸序列为SEQIDNo.4所示的氨基酸序列。根据本公开的优选的一种实施方式,本公开还提供了一种Pat#基因,其为密码子优化的Pat基因,该Pat#基因的序列具有提高Pat#基因翻译效率的序列和如上所述的Pat#基因的序列,且该Pat#基因能编码具有草铵膦N-乙酰基转移酶活性的多肽。优选地,提高Pat#基因翻译效率的序列为Kozak序列,所述Kozak序列如SEQIDNo.5所示。更优选地,具有提高Pat#基因翻译效率的序列的Pat#基因的序列如SEQIDNo.6所示。更详细地,为了提高Pat#基因的翻译效率,所述Pat#基因的5’端优选还有SEQIDNo.5所示的DNA序列,SEQIDNo.5所示的DNA序列属于Kozak序列,其中,Kozak序列用来增强真核基因的翻译效率,避免核糖体出现扫描漏洞(Leakyscan)的序列。在本公开中,SEQIDNo.5所示的序列位于SEQIDNo.3所示的序列的5’端,通过以上方法确定如SEQIDNo.6所示的序列。根据本公开的优选的一种实施方式,本公开还提供了一种Pat#基因,其为密码子优化的Pat基因,该Pat#基因的序列为如上所述的Pat#基因的5’端和3’分别连接上相同或不同的内切酶识别序列后得到。优选地,所述内切酶识别序列为XhoⅠ限制性内切酶识别序列,所述XhoⅠ限制性内切酶识别序列如SEQIDNo.7所示。更优选地,具有内切酶识别序列的Pat#基因序列如SEQIDNo.8所示。详细地,为了载体构建的需要,在上述序列的5’与3’端还分别添加了限制性内切酶识别位点。在本公开中,优选地在SEQIDNo.6所示的序列5’与3’端分别添加SEQIDNo.7所示的序列(XhoI限制性内切酶识别序列ctcgag),最终确定如SEQIDNo.8所示的序列。根据本公开的优选的一种实施方式,本公开还提供了一种Pat#基因表达框,该Pat#基因表达框含有如上所述的Pat#基因、在该Pat#基因的5’端上连接有能够被RNA聚合酶特异识别和高效结合的启动子和在该Pat#基因的3’端上连接有能够给予RNA聚合酶转录终止信号的终止子。优选地,所述启动子为35S启动子,所述35S启动子的序列如SEQIDNo.9所示,所述终止子为35SPolyA终止子,所述35SPolyA终止子的序列如SEQIDNo.10所示。更优选地,所述Pat#基因表达框序列如SEQIDNo.11所示。具体地,本领域技术人员均应了解要使目标基因(即Pat#基因)转入生物体内稳定表达,就需要有完整的基因表达框,即目标基因上游(5’端)有启动子序列、下游(3’端)有终止子序列。启动子序列和终止子序列在目标基因表达时起到控制目标基因转录的起始部位、转录强度和转录终止部位的作用,它并不影响目标基因表达出的蛋白质的氨基酸组成和结构,对于目标基因赋予的除草剂抗性特征没有任何影响。因此,本公开中,可以选择任何能够适用于基因表达的启动子、终止子序列与目标基因形成不同组合的基因表达框。根据一般生物学原理,真核和原核生物启动子-基因-终止子组合(表达框)都能实现本公开的Pat#基因的转录;只是本公开的Pat#基因按照模式植物水稻的密码子优化,在真核高等植物中蛋白质翻译效率高。本领域技术人员均应了解不同启动子和终止子对于起始和终止目标基因转录的能力有差异、作用时空有不同。选择合适的启动子和增强启动子的活性可增强外源基因的转录。目前在植物表达载体中应用的启动子有组成表达型启动子(例如:CaMV35S启动子、Ubiquitin启动子、Actinl启动子,等)、诱导表达型启动子(例如:创伤诱导表达启动子、光诱导启动子RbcS、四环素诱导表达启动子,等)、组织特异表达型启动子(例如:花药绒毡层特异表达启动子TA29、胚乳特异表达启动子、维管束特异表达启动子,等)等。根据本公开,优选地,所述Pat#基因的5’端还有SEQIDNo.9所示的序列作为Pat#基因转录的启动子。其中,SEQIDNo.9所示的序列为来源于花椰菜花叶病毒35S蛋白的启动子序列(CaMV35S),作为启动Pat#基因转录的启动子。根据本公开,优选地,所述Pat#基因的3’端还有SEQIDNo.10所示的序列作为Pat#基因转录的终止子。其中,SEQIDNo.10所示的序列为来源于花椰菜花叶病毒的35S蛋白的PolyA终止子序列(35SPolyA),作为终止Pat#基因转录的终止子。最终,形成了如SEQIDNo.11所示的序列,作为Pat#基因表达的完整表达框。另一方面,本公开还提供一种重组载体,该Pat#基因重组载体由上述的Pat#基因表达框插入骨架载体后得到。根据本公开,术语“骨架载体”是指在基因工程重组DNA技术中将所述Pat#基因表达框转移至受体细胞的一种能自我复制的DNA分子;所述骨架载体可以为原核克隆载体(pMD18-T、pMD19-T等)、原核表达载体(pET32a等)或真核表达载体(pCAMBIA3300、pCAMBIA3301等)。其中,原核克隆载体和原核表达载体的主要作用是克隆、验证目标基因和目标蛋白的功能、制作试剂盒的阳性对照物和抗体等,而真核表达载体的作用是为了后续将其转化进作物中并赋予作物目标性状。原核表达载体插入原核启动子和终止子组合成的Pat#基因表达框能实现上述Pat#基因在原核细胞中的表达,真核表达载体插入真核启动子和终止子组合成的Pat#基因表达框能实现上述Pat#基因在真核细胞中的表达。因此,能够满足上述应用的各种T载体、原核载体和真核载体均可用于本公开。优选地,Pat#基因重组载体为真核植物表达载体pC3300-Pat#,它以植物表达载体pCAMBIA3300为骨架载体,插入序列如SEQIDNo.11所示的Pat#基因表达框构建而成。再一方面,本公开还提供一种改变作物抗性的方法,该方法包括将如上所述的重组载体转化所述作物,使得到的转基因作物表达Pat#基因、产生草铵膦N-乙酰基转移酶活性的多肽而具有除草剂抗性。根据本公开,术语“作物”为本领域公知的概念,指大面积栽种或大面积收获、供盈利或口粮用的植物。本公开的基因不仅可用于转入水稻,也可以用于转入玉米、高粱、小麦、棉花、油菜、大豆、土豆、蔬菜及其它具有盈利用途的植物。优选地,所述作物为模式植物水稻,所述水稻包括各种水稻,如各亚种(籼稻、粳稻、爪哇稻)、各生态型(早稻、中稻、晚稻)水稻等。优选地,本公开中优选籼稻品系9K作为转化的受体植物。实现转基因的方法有本领域公知的农杆菌介导转化法、基因枪法和花粉管通道法,等。优选地,本公开所用的转化方法为农杆菌介导转化法。所述农杆菌介导转化法可以为文献(YukohHieietal.TransformationofricemediatedbyAgrobacteriumtumefaciens.PlantMolecularBiology,1997,35:205-218)中报道的方法,也可采用(SeiichiToki,etal.EarlyinfectionofscutellumtissuewithAgrobacteriumallowshigh-speedtransformationofrice.ThePlantJournal,2006,47:969-976)中报道的快速转化方法。本公开采用改进的农杆菌介导快速转化法,具体地,SeiichiToki等人进行农杆菌介导转化时,愈伤组织诱导后不进行继代而直接浸染,而本公开根据所用的愈伤组织的状态,诱导产生的愈伤组织继代后第3天达到最佳状态时再进行浸染,这样改进能够得到更高的转化效率。术语“除草剂抗性”是指作物对某种或某类除草剂具有高于该作物同类一定剂量的耐受能力。本公开中,所述除草剂抗性为对草铵膦类除草剂的抗性;优选地,所述除草剂抗性为至少耐受1000mg/L浓度的草铵膦。下面,通过以下实施例对本公开作更详细的说明。其中,DNA序列SEQIDNo.8委托大连宝生物公司合成;各种PCR引物的合成和基因测序工作委托华大基因公司进行;各种酶均购自大连宝生物公司;pCAMBIA3300载体由澳大利亚农业分子生物学应用中心(CAMBIA,http://www.cambia.org)提供;质粒的提取、纯化和酶切以及其他常规分子生物学操作方法参考《分子克隆》(科学出版社,第3版)进行。本公开实施例中,所用作物为籼稻品系9K,由中国科学院亚热带农业生态研究所培育,已在文献(曾崇华.HD9802S/Kasalath选系中高培养力和抗稻瘟病株系的筛选[D].北京:中国科学院大学,2015)中公开。农杆菌菌株EHA105购自上海北诺生物科技有限公司。本公开中采用文献(SeiichiTokietal.EarlyinfectionofscutellumtissuewithAgrobacteriumallowshigh-speedtransformationofrice.ThePlantJournal,2006,47:969-976)所述的农杆菌介导转化法,但有改良:诱导产生的愈伤组织继代1次,继代后第3d进行浸染;且在共培养和预分化阶段均将固体培养基中的琼脂粉含量提高至12g/L。实施例1本实施例用于说明人工优化的Pat#基因的制备方法。首先,查找原始Pat基因的DNA序列(NCBI登录号:M22827.1),分析原始Pat基因序列的同义密码子的相对使用度(RSCU值)和粳稻日本晴的蛋白质编码基因序列的同义密码子的相对使用度(RSCU值)。其次,分析原始序列中存在的内含子切割识别序列、PolyA加尾识别序列及mRNA发夹环结构。根据分析,在保持PAT蛋白的氨基酸组成、顺序不变的前提下,用粳稻日本晴的偏爱密码子置换原始Pat基因序列中相应的密码子,同时消除影响外源基因在植物细胞内高效表达的元件,最终获得优化后的Pat基因(序列如SEQIDNo.1所示),命名为Pat#基因。分析发现,原始Pat基因中不存在造成植物转录不稳定的AT富集区及ATTTA重复序列、PolyA加尾识别信号序列,但存在1处潜在的内含子切割识别序列,容易甲基化的密码子GCG、ACG、CCG和TCG的使用频率较高,转录产生的mRNA中存在多个稳定的发夹环结构、且起始自由能达到-268.90kcal/mol。利用同义密码子替换法,消除了内含子切割识别序列;在不改变优化基因氨基酸序列的前提下,利用同义密码子替换法来消除形成的稳定发夹环等mRNA二级结构,使优化基因的起始自由能降到-202.40kcal/mol;同时为了防止DNA序列甲基化的可能,适当降低了密码子GCG、ACG、CCG和TCG的使用频率。分析优化后的Pat#基因得到的同义密码子的相对使用度(RSCU值)如表1中第3列所示,与粳稻日本晴和原始Pat基因序列相比,优化后的Pat#基因序列的密码子使用频率与粳稻日本晴的表达蛋白密码子使用频率基本一致。获得的优化Pat#基因序列长度为552bp,与原始Pat基因的DNA序列相比,氨基酸组成和顺序不变,改变碱基101个,占碱基总数的18.3%,更改密码子92个,占基因密码子总数的50.0%,其中起始密码子由GTG改为ATG,G+C含量从原来的70.1%降为61.41%。表1注:括号内数据为在统计的样本中每个密码子使用的次数。RSCU(ij)=x(ij)/〔x(i)/n〕,其中,RSCU(ij)是指第i个氨基酸第j个同义密码子的密码子使用相对概率,x(ij)是指第i个氨基酸第j个同义密码子在一个基因中的出现次数,x(i)是指第i个氨基酸在这个基因中的出现次数,这个数字是该氨基酸对应所有同义密码子的出现次数之和;n是指该氨基酸对应的同义密码子种数。表1展示出了人工优化合成的Pat#基因、原始的Pat基因与粳稻日本晴蛋白编码基因在同义密码子的相对使用度(RSCU值)上的比较。为了使表达的目标蛋白有效地积累和行使功能,在优化好的Pat#基因序列的5’端添加SEQIDNo.2所示的序列,该序列可以把目标蛋白靶向叶绿体。为了提高PAT蛋白的表达效率,可以对起始密码子周边序列进行优化。优选地,在Pat#融合基因的5’端添加了SEQIDNo.5所示的DNA序列,其中SEQIDNo.5所示的DNA序列属于Kozak序列。根据进一步实验的需要,在上述序列的5’与3’端分别添加XhoI内切酶识别序列ctcgag,通过以上步骤最终确定如SEQIDNo.8所示的序列。送至大连宝生物公司合成如SEQIDNo.8所示的序列,并将其连接在pMD19-T载体上,构建成含有Pat#基因的重组载体pMD19T-Pat#,为基因鉴定、基因标准物制备、原核和真核表达载体构建等提供基础。实施例2本实施例用于说明植物表达载体pC3300-Pat#的构建方法,构建流程如图1所示。将质粒载体pCAMBIA3300和pMD19T-Pat#均用限制性内切酶XhoⅠ单酶切,分别回收其大片段和小片段。再将这2个回收片段经去磷酸化后,用T4DNA连接酶连接得到重组质粒,使用重组质粒转化大肠杆菌DH5α感受态细胞,含有重组质粒的阳性菌株扩增后提取重组质粒。限制性内切酶XhoⅠ单酶切和正反向连接PCR验证检测结果(如图2所示)说明重组质粒构建成功,其中验证基因连接方向的上游引物为Pat-F2如SEQIDNO.12所示,验证基因连接方向的下游引物为Pat-R2如SEQIDNO.13所示。把连接方向正确的质粒DNA送样测序,再次验证得到连接方向正确的重组质粒,新重组载体命名为pC3300-Pat#。表2引物名称SEQIDNO.核苷酸序列上游引物Pat-F2125’-gacgcacaatcccactatcc-3’下游引物Pat-R2135’-atctgatcctgccgccattg-3’实施例3本实施例用于说明Pat#基因转化模式植物水稻的方法。1)水稻成熟胚愈伤组织的诱导选用籼稻品系9K作为转化受体,取其成熟种子,剥去颖壳;糙米中加入5%的次氯酸钠溶液消毒40min,倾去上清液,用无菌双蒸水清洗3-5次;将糙米接种于愈伤组织诱导培养基上,32℃黑暗下培养5-7d;挑选从种子盾片部位诱导出的愈伤组织接种于新鲜诱导培养基上,继代3d后用于农杆菌浸染。2)农杆菌介导法转化水稻愈伤组织(a)pC3300-Pat#质粒转化农杆菌感受态细胞。农杆菌感受态细胞的制备及转化方法参考《植物基因工程》(科学出版社,第2版)进行。(b)农杆菌转化水稻成熟胚诱导的愈伤组织。(1)取步骤(a)得到的农杆菌单菌落,均匀涂抹于含50mg/L卡那霉素和50mg/L利福平的LB固体培养基上,28℃倒置培养1-2d至菌落长出;(2)加入相应的液体培养基悬浮菌体成OD值在0.2左右的菌液;(3)将待浸染的愈伤组织转移至灭菌的三角瓶中,加入OD值在0.2左右的农杆菌菌液浸染20min,浸染过程中不断缓慢摇动三角瓶;(4)取出愈伤组织置于灭菌的滤纸上吸干多余菌液后再置于琼脂粉含量为10g/L的固体共培养基上于24℃暗培养3d;(5)取出共培养的愈伤组织,转移到含有6mg/L草铵膦、400mg/L羧苄霉素和500mg/L头孢霉素的筛选培养基上培养2-3周;(6)之后将筛选到的成活愈伤组织转移到含12g/L琼脂粉的预分化培养基上培养2周;(7)将预分化后的愈伤组织转移到分化培养基上,每天12h、1000-1500lux光照下进行分化;(8)7d后愈伤组织出现绿点,14d左右愈伤组织上长出再生小苗,3-4周后将再生小苗转至1/2MS培养基上壮苗培养,待再生植株长到10cm左右时移栽入土。移栽成活的转基因水稻再生植株命名为Pat9K。3)转基因植株的检测(a)转基因植株Pat9K的PCR检测首先,采用CTAB法提取植株总DNA,方法参考《植物基因工程》(科学出版社,第2版)进行。其次,进行PCR检测分析:(1)DNA试验材料:转基因植株Pat9K的总DNA;非转基因阴性对照9K的总DNA;阳性对照pC3300-Pat#质粒。(2)PCR检测Pat#基因:上游引物Pat-F1的序列如SEQIDNo.14所示,下游引物Pat-R1的序列如SEQIDNo.15所示,核酸序列见表3。对Pat#基因的PCR检测结果如图3a所示。其中,泳道M为DL2000DNA分子量标记,泳道B为不加模板的阴性对照,泳道CK为非转基因植株9K的阴性对照,泳道P为质粒pC3300-Pat#的阳性对照,泳道1-5为转基因水稻Pat9K的不同单株。检测结果说明转基因株系Pat9K已成功转入了Pat#基因。表3引物名称SEQIDNO.核苷酸序列上游引物Pat-F1145’-ggattgacgaccttgagcgc-3’下游引物Pat-R1155’-acatcgtgccaaccgccatg-3’(b)转基因植株Pat9K的Southern杂交检测首先,采用CTAB法提取植株总DNA,方法参考《植物基因工程》(科学出版社,第2版)进行。其次,以PCR扩增的方法制备探针,具体方法如下:(1)PCR反应体系:正向引物Pat-F1(10μmol)1μL,反向引物Pat-R1(10μmol)1μL,DNA模板(10-20ng/μL)1μL,10×PCR缓冲液2μL,MgCl22μL,Dig-dUTP标记混合物1μL,H2O13.5μL,TaqDNA聚合酶(5U/μL)0.5μL。(2)PCR反应程序:94℃预变性5min;94℃变性45s,57℃退火45s,72℃延伸1min,34个循环;最后72℃延伸10min。(3)检测扩增产物:制备1.0%的琼脂糖凝胶,取1μLPCR产物稀释5倍后电泳检测。Southern杂交方法参考《植物基因工程》(科学出版社,第2版)进行。Southern杂交检测了5株T1代阳性植株,检测结果见图3b,其中,泳道M为DL15000DNA分子量标记,泳道P为质粒pC3300-Pat#的阳性对照,泳道CK为非转基因植株9K的阴性对照,泳道1-5为转基因水稻Pat9K的不同单株。结果显示,泳道1和3的两株为同一转化体,具有2个拷贝数,另外3株(泳道2、4和5)均有一条不同的特异性目标条带,拷贝数均为1。实施例4本实施例用于说明Pat#基因在籼稻9K中表达。通过测定Pat#基因产物的相对实时表达量,用于说明Pat#基因在籼稻9K中能正常表达。具体操作步骤如下:首先,提取水稻叶片总RNA,提取方法参考《植物基因工程》(科学出版社,第2版)进行。其次,将总RNA2μg利用反转录试剂盒(购自Fermentas公司)合成cDNA20μL,具体步骤参见说明书,-20℃保存备用。然后,进行实时相对定量PCR(qRT-PCR)分析;qRT-PCR分析使用SYBRGreenRT-PCROneStepKit(购自Roche公司)进行扩增,总反应体系10μL,其中加入浓度为100ng/μL的cDNA模板0.5μL,具体步骤参见说明书,采用BIO-RAD荧光定量PCR仪分析仪器。每个测试样品重复3次。(2)检测Pat#基因表达的上游引物Pat-qrt-1F的序列如SEQIDNo.16所示,下游引物Pat-qrt-1R的序列如SEQIDNo.17所示,核苷酸序列见表4。(3)反应程序为:第1步95℃,5min;第2步95℃,10s;第3步60℃,30s;返回第2步,39个循环结束。表4引物名称SEQIDNO.核苷酸序列上游引物Pat-qrt-1F165’-ctgaggtcgaaggggttgt-3’下游引物Pat-qrt-1R175’-gctcttgaggagatgggtgt-3’利用Ct值相对定量法进行数据分析,结果见图4。结果显示,优化后的Pat#基因在转基因水稻Pat9K中可正常表达,转基因水稻Pat9K株系中Pat9K-2和Pat9K-4转基因株系的相对表达量分别为非转基因对照的1.82倍和1.44倍。实施例5本实施例用于检测T2代转基因植株Pat9K喷洒除草剂草铵膦后的抗性表现。用100mL浓度为1000mg/L的草铵膦水溶液喷洒非转基因植株9K和转基因植株Pat9K,10d后观察植株生长情况。结果如图5所示,其中,对照组为非转基因植株9K,Pat9K为转基因株系。由图5可以看出,非转基因对照植株9K已经枯死,而转基因的植株Pat9K则生长正常。说明Pat#基因在转基因水稻中正常表达,能抵抗浓度为1000mg/L草铵膦的处理。实施例6本实施例用于检测转基因水稻T2代植株的种子在含草铵膦培养基上萌发的抗性表现。方法:将灭菌后的非转基因水稻9K种子和转基因水稻Pat9K种子分别接种到含有0、200、400、600、800、1000mg/L草铵膦的1/2MS培养基上,在24℃光照/19℃黑暗交替条件下发芽,10天后观察每个样品的种子发芽情况。种子萌发实验测定转基因水稻对草铵膦抗性的结果见图6,其中,9K为非转基因水稻种子,Pat9K为转Pat#基因水稻Pat9K的种子。结果显示:在除草剂草铵膦存在的情况下,非转基因水稻种子不能正常发芽,而转基因水稻Pat9K种子在试验浓度下均能正常萌发,未受到明显影响。统计转基因水稻种子在草铵膦培养基上萌发的芽长结果表明:当草铵膦浓度为0mg/L时,Pat9K的芽长与对照材料9K间无显著差异;在含草铵膦(200~1000mg/L)的培养基上,转基因水稻Pat9K正常萌发、芽长均与除草剂浓度为0mg/L时没有显著差异,而非转基因水稻种子不能萌发。说明转基因水稻Pat9K具有草铵膦抗性,且抗性浓度不低于1000mg/L。以上结合附图详细描述了本公开的优选实施方式,但是,本公开并不限于上述实施方式中的具体细节,在本公开的技术构思范围内,可以对本公开的技术方案进行多种简单变型,这些简单变型均属于本公开的保护范围。例如,信号肽的改变、增强基因翻译效率序列的改变、启动子的改变等。另外需要说明的是,在上述具体实施方式中所描述的各个具体技术特征,在不矛盾的情况下,可以通过任何合适的方式进行组合。为了避免不必要的重复,本公开对各种可能的组合方式不再另行说明。此外,本公开的各种不同的实施方式之间也可以进行任意组合,只要其不违背本公开的思想,其同样应当视为本公开所公开的内容。SEQUENCELISTING<110>中国科学院亚热带农业生态研究所<120>人工优化合成的Pat#基因与重组载体以及改变作物抗性的方法<130>5849ISA<160>17<170>PatentInversion3.5<210>1<211>552<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>1atgagccccgagaggcgccccgttgagatccgtcccgcaacggccgcagacatggctgca60gtgtgcgacattgtgaatcactacattgagacctccacagtgaacttccggactgagccc120cagactccccaggaatggattgacgaccttgagcgcctgcaggatcgctacccctggttg180gtggctgaggtcgaaggggttgtggcgggcatcgcctacgcgggtccttggaaagcccgg240aacgcctatgactggaccgttgagtccacagtctacgtctctcaccggcaccagcgcttg300ggtttgggatccaccctctacacccatctcctcaagagcatggaggcgcaaggcttcaag360agtgtcgttgccgtcatcggccttcccaacgatccgtctgtccggttgcatgaggccctc420ggatacaccgcgagagggaccctccgagccgcaggctacaagcatggcggttggcacgat480gtcggcttctggcaaagggactttgagctgcctgcaccgcctcgccctgtccgtccggtg540acacagatctga552<210>2<211>147<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>2atggccccaagcgtgatggccagctccgcaaccacggtcgctcccttccaaggcctcaag60tccacggccggcatgccggtggccaggagatccggcaactccagcttcggcaacgtcagc120aatggcggcaggatcagatgcatgcag147<210>3<211>699<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>3atggccccaagcgtgatggccagctccgcaaccacggtcgctcccttccaaggcctcaag60tccacggccggcatgccggtggccaggagatccggcaactccagcttcggcaacgtcagc120aatggcggcaggatcagatgcatgcagatgagccccgagaggcgccccgttgagatccgt180cccgcaacggccgcagacatggctgcagtgtgcgacattgtgaatcactacattgagacc240tccacagtgaacttccggactgagccccagactccccaggaatggattgacgaccttgag300cgcctgcaggatcgctacccctggttggtggctgaggtcgaaggggttgtggcgggcatc360gcctacgcgggtccttggaaagcccggaacgcctatgactggaccgttgagtccacagtc420tacgtctctcaccggcaccagcgcttgggtttgggatccaccctctacacccatctcctc480aagagcatggaggcgcaaggcttcaagagtgtcgttgccgtcatcggccttcccaacgat540ccgtctgtccggttgcatgaggccctcggatacaccgcgagagggaccctccgagccgca600ggctacaagcatggcggttggcacgatgtcggcttctggcaaagggactttgagctgcct660gcaccgcctcgccctgtccgtccggtgacacagatctga699<210>4<211>232<212>PRT<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>4MetAlaProSerValMetAlaSerSerAlaThrThrValAlaProPhe151015GlnGlyLeuLysSerThrAlaGlyMetProValAlaArgArgSerGly202530AsnSerSerPheGlyAsnValSerAsnGlyGlyArgIleArgCysMet354045GlnMetSerProGluArgArgProValGluIleArgProAlaThrAla505560AlaAspMetAlaAlaValCysAspIleValAsnHisTyrIleGluThr65707580SerThrValAsnPheArgThrGluProGlnThrProGlnGluTrpIle859095AspAspLeuGluArgLeuGlnAspArgTyrProTrpLeuValAlaGlu100105110ValGluGlyValValAlaGlyIleAlaTyrAlaGlyProTrpLysAla115120125ArgAsnAlaTyrAspTrpThrValGluSerThrValTyrValSerHis130135140ArgHisGlnArgLeuGlyLeuGlySerThrLeuTyrThrHisLeuLeu145150155160LysSerMetGluAlaGlnGlyPheLysSerValValAlaValIleGly165170175LeuProAsnAspProSerValArgLeuHisGluAlaLeuGlyTyrThr180185190AlaArgGlyThrLeuArgAlaAlaGlyTyrLysHisGlyGlyTrpHis195200205AspValGlyPheTrpGlnArgAspPheGluLeuProAlaProProArg210215220ProValArgProValThrGlnIle225230<210>5<211>6<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>5gccacc6<210>6<211>705<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>6gccaccatggccccaagcgtgatggccagctccgcaaccacggtcgctcccttccaaggc60ctcaagtccacggccggcatgccggtggccaggagatccggcaactccagcttcggcaac120gtcagcaatggcggcaggatcagatgcatgcagatgagccccgagaggcgccccgttgag180atccgtcccgcaacggccgcagacatggctgcagtgtgcgacattgtgaatcactacatt240gagacctccacagtgaacttccggactgagccccagactccccaggaatggattgacgac300cttgagcgcctgcaggatcgctacccctggttggtggctgaggtcgaaggggttgtggcg360ggcatcgcctacgcgggtccttggaaagcccggaacgcctatgactggaccgttgagtcc420acagtctacgtctctcaccggcaccagcgcttgggtttgggatccaccctctacacccat480ctcctcaagagcatggaggcgcaaggcttcaagagtgtcgttgccgtcatcggccttccc540aacgatccgtctgtccggttgcatgaggccctcggatacaccgcgagagggaccctccga600gccgcaggctacaagcatggcggttggcacgatgtcggcttctggcaaagggactttgag660ctgcctgcaccgcctcgccctgtccgtccggtgacacagatctga705<210>7<211>6<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>7ctcgag6<210>8<211>717<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>8ctcgaggccaccatggccccaagcgtgatggccagctccgcaaccacggtcgctcccttc60caaggcctcaagtccacggccggcatgccggtggccaggagatccggcaactccagcttc120ggcaacgtcagcaatggcggcaggatcagatgcatgcagatgagccccgagaggcgcccc180gttgagatccgtcccgcaacggccgcagacatggctgcagtgtgcgacattgtgaatcac240tacattgagacctccacagtgaacttccggactgagccccagactccccaggaatggatt300gacgaccttgagcgcctgcaggatcgctacccctggttggtggctgaggtcgaaggggtt360gtggcgggcatcgcctacgcgggtccttggaaagcccggaacgcctatgactggaccgtt420gagtccacagtctacgtctctcaccggcaccagcgcttgggtttgggatccaccctctac480acccatctcctcaagagcatggaggcgcaaggcttcaagagtgtcgttgccgtcatcggc540cttcccaacgatccgtctgtccggttgcatgaggccctcggatacaccgcgagagggacc600ctccgagccgcaggctacaagcatggcggttggcacgatgtcggcttctggcaaagggac660tttgagctgcctgcaccgcctcgccctgtccgtccggtgacacagatctgactcgag717<210>9<211>770<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>9cgacactctcgtctactccaagaatatcaaagatacagtctcagaagaccaaagggctat60tgagacttttcaacaaagggtaatatcgggaaacctcctcggattccattgcccagctat120ctgtcacttcatcaaaaggacagtagaaaaggaaggtggcacctacaaatgccatcattg180cgataaaggaaaggctatcgttcaagatgcctctgccgacagtggtcccaaagatggacc240cccacccacgaggagcatcgtggaaaaagaagacgttccaaccacgtcttcaaagcaagt300ggattgatgtgataacatggtggagcacgacactctcgtctactccaagaatatcaaaga360tacagtctcagaagaccaaagggctattgagacttttcaacaaagggtaatatcgggaaa420cctcctcggattccattgcccagctatctgtcacttcatcaaaaggacagtagaaaagga480aggtggcacctacaaatgccatcattgcgataaaggaaaggctatcgttcaagatgcctc540tgccgacagtggtcccaaagatggacccccacccacgaggagcatcgtggaaaaagaaga600cgttccaaccacgtcttcaaagcaagtggattgatgtgatatctccactgacgtaaggga660tgacgcacaatcccactatccttcgcaagaccttcctctatataaggaagttcatttcat720ttggagaggacacgctgaaatcaccagtctctctctacaaatctatctct770<210>10<211>203<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>10gtttctccataataatgtgtgagtagttcccagataagggaattagggttcctatagggt60ttcgctcatgtgttgagcatataagaaacccttagtatgtatttgtatttgtaaaatact120tctatcaataaaatttctaattcctaaaaccaaaatccagtactaaaatccagatccccc180gaattaattcggcgttaattcag203<210>11<211>1690<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>11cgacactctcgtctactccaagaatatcaaagatacagtctcagaagaccaaagggctat60tgagacttttcaacaaagggtaatatcgggaaacctcctcggattccattgcccagctat120ctgtcacttcatcaaaaggacagtagaaaaggaaggtggcacctacaaatgccatcattg180cgataaaggaaaggctatcgttcaagatgcctctgccgacagtggtcccaaagatggacc240cccacccacgaggagcatcgtggaaaaagaagacgttccaaccacgtcttcaaagcaagt300ggattgatgtgataacatggtggagcacgacactctcgtctactccaagaatatcaaaga360tacagtctcagaagaccaaagggctattgagacttttcaacaaagggtaatatcgggaaa420cctcctcggattccattgcccagctatctgtcacttcatcaaaaggacagtagaaaagga480aggtggcacctacaaatgccatcattgcgataaaggaaaggctatcgttcaagatgcctc540tgccgacagtggtcccaaagatggacccccacccacgaggagcatcgtggaaaaagaaga600cgttccaaccacgtcttcaaagcaagtggattgatgtgatatctccactgacgtaaggga660tgacgcacaatcccactatccttcgcaagaccttcctctatataaggaagttcatttcat720ttggagaggacacgctgaaatcaccagtctctctctacaaatctatctctctcgaggcca780ccatggccccaagcgtgatggccagctccgcaaccacggtcgctcccttccaaggcctca840agtccacggccggcatgccggtggccaggagatccggcaactccagcttcggcaacgtca900gcaatggcggcaggatcagatgcatgcagatgagccccgagaggcgccccgttgagatcc960gtcccgcaacggccgcagacatggctgcagtgtgcgacattgtgaatcactacattgaga1020cctccacagtgaacttccggactgagccccagactccccaggaatggattgacgaccttg1080agcgcctgcaggatcgctacccctggttggtggctgaggtcgaaggggttgtggcgggca1140tcgcctacgcgggtccttggaaagcccggaacgcctatgactggaccgttgagtccacag1200tctacgtctctcaccggcaccagcgcttgggtttgggatccaccctctacacccatctcc1260tcaagagcatggaggcgcaaggcttcaagagtgtcgttgccgtcatcggccttcccaacg1320atccgtctgtccggttgcatgaggccctcggatacaccgcgagagggaccctccgagccg1380caggctacaagcatggcggttggcacgatgtcggcttctggcaaagggactttgagctgc1440ctgcaccgcctcgccctgtccgtccggtgacacagatctgactcgaggtttctccataat1500aatgtgtgagtagttcccagataagggaattagggttcctatagggtttcgctcatgtgt1560tgagcatataagaaacccttagtatgtatttgtatttgtaaaatacttctatcaataaaa1620tttctaattcctaaaaccaaaatccagtactaaaatccagatcccccgaattaattcggc1680gttaattcag1690<210>12<211>20<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>12gacgcacaatcccactatcc20<210>13<211>20<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>13atctgatcctgccgccattg20<210>14<211>20<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>14ggattgacgaccttgagcgc20<210>15<211>20<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>15acatcgtgccaaccgccatg20<210>16<211>19<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>16ctgaggtcgaaggggttgt19<210>17<211>20<212>DNA<213>ArtificialSequence<220><223>Thissequenceissynthesizedinlab.<400>17gctcttgaggagatgggtgt20当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1
- 一种基因片段、载体pPlasmid‑Clearance及应用的制作方法与工艺
- 一种米曲霉脂肪酶、编码基因、载体、工程菌及其应用的制作方法与工艺
- 一种正电性的基因载体功能的聚合物荧光碳量子点的制备方法及应用与流程
- 人工优化合成的Mat基因与重组载体以及改变作物抗性的方法与流程
- 斜带石斑鱼补体C1INH基因、载体、重组菌株和蛋白及其应用的制造方法与工艺
- 一种几丁质代谢调控基因、编码蛋白、载体、工程菌及其应用的制造方法与工艺
- 用于减弱基因编辑抗病毒转移载体免疫应答的方法和组合物与流程
- 苯丙氨酸变位酶、编码基因、重组载体、宿主细胞、多重PCR引物及它们的应用的制造方法与工艺
- Hg‑snb1基因片段介导的RNAi载体及其在培育抗大豆胞囊线虫转基因植物中的应用的制造方法与工艺
- 纤维素酶及其编码基因、重组载体和应用的制造方法与工艺