一种蔗糖酶基因及其表达与应用的制作方法
本发明涉及生物技术领域,具体地说是一种蔗糖酶编码基因及其表达,以及该蔗糖酶蛋白在工业上的应用。
背景技术:
蔗糖酶(invertase,ec.3.2.1.26)又被称作蔗糖转化酶或β-呋喃果糖苷酶(β-fructofuranosidase),能催化β-d-呋喃果糖苷末端非还原性末端β-d-呋喃果糖苷键水解,不仅能催化蔗糖水解为等摩尔的果糖和葡萄糖,也能催化棉子糖水解生成蜜二糖和果糖。按照氨基酸序列相似性分类,蔗糖酶可被归类为糖苷水解酶中的32家族(gh32)、gh68以及gh100(http://www.cazy.org/)。其中,gh32家族蔗糖酶具有典型的n-和c-端保守结构域,其pfam标志性结构域编号分别为pf0251和pf08244。
蔗糖酶广泛存在于真菌中。近年来,部分真菌来源特别是酵母来源的蔗糖已经应用于工业生产。例如,已经从不同属来源酵母中发现蔗糖酶编码基因,部分蔗糖酶蛋白也已进行系统的酶学性质分析,包括pichiaanomala、arxulaadeninivorans、saccharomycescerevisiae等来源的蔗糖酶等。除此之外,aspergillus属真菌中也发现了蔗糖酶的存在。gongronellasp.属于小克银汉霉科(familycunninghamellaceae)真菌。目前,对该属真菌的研究主要集中于其壳聚糖的编码基因、壳聚糖的合成、以及部分糖苷水解酶如β-葡萄糖苷酶等的研究。然而,目前为止,还没有gongronella属来源蔗糖酶基因的报道。
技术实现要素:
本发明是为避免上述现有技术所存在的不足之处,提供一种蔗糖酶(名称为gspinv)及表达该蔗糖酶基因的工程菌株。
该菌株分类命名为pichiapastoris/ppic9k-gspinv,保藏于中国典型培养物保藏中心,保藏编号为cctccm2017438。。
本发明的蔗糖酶,其原始来源于中土壤来源微生物,具体来源于gongronellasp.,其基因,具有如下核苷酸序列之一:
(1)序列表中的seqidno:1;
(2)序列表中的seqidno:1经取代、缺失或添加一个或几个核苷酸且编码相同功能蛋白质的核苷酸序列。
(3)编码序列表中seqidno:2蛋白质序列的多核苷酸序列;
一种蔗糖酶,具有下列氨基酸序列之一:
(1)序列表中的seqidno:2;
(2)序列表中的seqidno:2经取代、缺失或添加一个或几个氨基残基且编码相同功能蛋白质的氨基酸序列。
本发明的目的还在于提供含有上述蔗糖酶基因的重组表达载体。
本发明的目的还在于提供含有上述蔗糖酶基因的宿主菌。
本发明的目的还在于提供上述蔗糖酶蛋白的制备方法,具体包括以下步骤:
(1)将接种菌株于bmgy液体培养基中,28℃、200rpm/min条件下培养至od600=2~6;离心菌体,用bmmy液体培养基重悬细胞至od600=1.0,每隔24h加入甲醇使其中甲醇浓度为0.5%;将诱导后的发酵液经过过滤分离得到上清液,即是蔗糖酶粗制剂;
(2)将粗制剂放入超滤容器中,4~10℃条件下在磁力搅拌器上进行超滤浓缩,然后将浓缩液溶于磷酸盐缓冲溶液中,离心分离上清液后,用deaesepharoseff离子交换柱纯化,洗脱缓冲液为0.3m(nh4)2so4,以5%,10%,20%,30%,40%,60%,80%,100%梯度洗脱,每次洗脱均冲至基线平稳,流速为1.0ml/min,最终获得纯化的蔗糖酶蛋白。
下面以表达质粒载体ppic9k、宿主菌株pichiapastorisgs115为例,具体介绍含有本发明蔗糖酶基因的重组表达菌株的构建方法,具体包括以下步骤:
(1)由来自gongronellasp.mrna反转录获得的cdna文库序列为模板,以p1和p2为引物进行pcr扩增,得到pcr扩增产物即为本发明蔗糖酶cdna;所述引物为:
p1:5′-cctaggatggtgcttgctgatcct-3′
p2:5′-gcggccgctcaagggcgattgaacg-3′
(2)将步骤(1)所得pcr扩增产物和peasy-t1simple载体连接,获得连接产物;将连接产物转化大肠杆菌trans1-t1感受态细胞,筛选阳性克隆,进行序列分析;挑选序列正确的克隆提取质粒,获得含有蔗糖酶基因(gspinv)的peasy-t1simple重组质粒;
(3)用notⅰ和blnⅰ酶切步骤(2)所得的peasy-t1simple重组质粒和ppic9k质粒;酶切后的载体ppic9k和蔗糖酶基因gspinv片段用t4dna连接酶连接,得到连接产物;连接产物用限制性内切酶sacⅰ对重组质粒gspinv-ppic9k进行单酶切,获得的单酶切产物回收后电击转化pichiapastorisgs115原生质体细胞,筛选阳性克隆,得到含有本发明基因gspinv的工程菌株pichiapastoris/ppic9k-gspinv。
附图说明
图1为本发明pcr扩增产物的电泳图谱;
图1中1和2为pcr扩增产物;m为分子量标准。
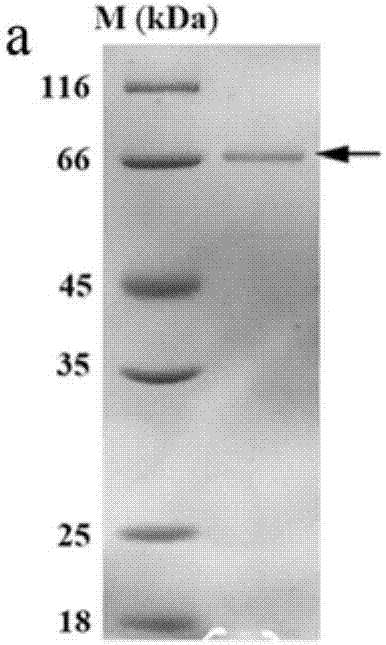
图2为本发明表达并纯化获得目标蛋白的电泳图谱。
图2中a为sds-page图谱,m为标准分子量;b为native-page图谱,电泳后胶块放置于终浓度为200mmol/l蔗糖溶液中45℃孵育30min,孵育结束后用清水冲洗凝胶将剩余的蔗糖溶液去除,将凝胶置于煮沸后的0.2%三苯基氯化四氮唑的naoh溶液中显色。
图3为以蔗糖为底物时测定的gspinv催化的最适ph、ph稳定性、最适温度和温度稳定性。
图3中a为最适ph,b为ph稳定性,c为最适温度,d为温度稳定性
图4为gspinv对不同浓度蔗糖的水解能力。
本发明具体实施方式中的菌株pichiapastoris/ppic9k-gspinv已送往中国典型培养物保藏中心(chinacenterfortypeculturecollection,cctcc)保藏。菌株保藏编号为cctccm2017438。
具体实施方式
下面结合实施例对本发明作进一步说明,但应该理解这些实施例并非限制本发明的范围。同时,下列实施例中的实施方法,如无特别说明,均为本领域常规试剂及常规方法。
(一)、含有本发明蔗糖酶基因的酵母表达菌株的构建
1、gongronellasp.菌株的活化
斜面保藏的gongronellasp.w5挑取至cpda斜面上,28℃静置培养3天以活化菌株。
2、培养基的配制及灭菌
配制发酵培养基:15g蔗糖,1.5gdl-天冬酰胺,0.5gmgso4·7h2o,0.1gna2hpo4·12h2o,0.01gcacl2,0.01gfeso4·7h2o,0.0275g腺嘌呤,50μgvb1,115℃灭菌20min备用。
3、gongronellasp.w5菌株mrna制备
挑取直径活化的4mmgongronellasp.w5菌丝体5块,接种到灭菌的发酵培养基中,28℃,120rpm振荡培养2d,减压抽滤获得菌丝体,保存于-70℃备用。
4、gspinvcdna克隆
获得的菌丝体使用液氮进行研磨,并按照rneasyplantminikit(qiagen)使用说明书抽提总rna。cdna反转录制备使用primescriptrtreagentkit(takara)和下述引物,实验操作按照试剂盒使用说明书进行。
p1:5′-cctaggatggtgcttgctgatcct-3′
p2:5′-gcggccgctcaagggcgattgaacg-3′
获得的cdna按下表进行pcr反应:
pcr扩增条件为94℃预变性5min,94℃变性30s,tm58℃退火30s,72℃延伸2kb/min共30循环,最后在72℃延伸10min,将得到的片段进行1%琼脂糖凝胶检测,结果见图1。得到的蔗糖酶的核苷酸序列如seqidno:1所示,其氨基酸序列如seqidno:2所示。
5、表达载体的构建
将步骤4中得到40ngpcr扩增产物,加入10ngpeasy-t1simple载体(transgen),25℃反应15min后立即置于冰上。连接产物热激转化大肠杆菌trans1-t1感受态细胞,得到的转化子测序验证是否突变;挑选序列正确的克隆提取质粒,获得含有蔗糖酶基因(gspinv)的peasy-t1simple重组质粒;用notⅰ和blnⅰ双酶切所得的peasy-t1simple重组质粒和ppic9k载体,然后用t4dna连接酶将酶切后的gspinv与表达质粒载体连接,建立如下酶切体系:20ngppic9k载体,60nggspinv酶切片段,3ul10×ligationbuffer,1ult4dna连接酶(takara),补水至30ul,16℃连接8h,得到连接产物;连接产物用限制性内切酶sacⅰ对重组质粒gspinv-ppic9k进行单酶切,回收后电转化宿主菌pichiapastorisgs115原生质体细胞,得到的转化子测序验证是否突变,挑选序列正确的转化子得到含有本发明基因gspinv的工程菌株pichiapastoris/ppic9k-gspinv。
(二)、含本发明蔗糖酶基因工程菌的表达与蛋白纯化
将(一)中获得的基因的工程菌株pichiapastoris/ppic9k-gspinv接种于含5mlbmgy液体培养基的试管中,接种量为1%,28℃,200rpm/min,过夜培养;取1ml过夜培养物转接于100mlbmgy的三角瓶中,28℃,200rpm/min,振荡培养至od600=2~6(unicouv2102紫外可见分光光度计,以bmgy培养基为空白);离心菌体,用bmmy液体培养基重悬细胞至od600=1.0,并于28℃,200rpm/min条件下继续培养,每隔24h加入甲醇,使其中甲醇体积浓度为0.5%以诱导蛋白表达。
将诱导8天的发酵液通过离心收集上清液。然后将上清液放入millipore超滤杯中,4℃下在磁力搅拌器上进行超滤浓缩至原体积的1/50,并通过sds-page和native-page检测蛋白纯度,最终获得纯蛋白,结果见图2。
以蔗糖为底物,纯化的蔗糖酶蛋白gspinv的最适ph为5.0,酶在ph5.5-6.0范围内有较高的稳定性,孵育5h后能够维持原酶活力的60%以上。以abts为底物,gspinv在30℃-60℃范围内均能显示催化活性,其最适温度为45℃,45℃时酶的半衰期为90min,结果见图3。
(三)、含本发明蔗糖酶蛋白的deae-sepharoseff离子交换层析纯化
层析过程中所有试剂均用去离子水溶解,经0.22μm滤膜过滤去除杂质,再置于超声清洗仪中超声1h以去除气泡。具体纯化步骤为:
1)启动aktapurifier系统,打开unicorn软件,将a、b泵都放入去离子水中,启动pumpwashbasic程序将系统冲洗一遍。
2)调低流速至0.5ml/min,将deaesepharoseff离子交换柱装入纯化系统。
3)将a、b泵分别放入低盐与高盐缓冲液中,调节流速至1ml/min,先用高盐缓冲液冲洗阴离子交换柱,直到基线平稳,再用低盐缓冲液冲洗至基线平稳。
4)将透析离心好的样品用0.22μm滤膜过滤后采用恒流泵上样,上样体积一般为柱床体积的1-5%,本实验总共上50ml浓缩液。
5)采用梯度洗脱,通过逐渐增加离子强度,从而使与离子交换树脂结合的各个组分被洗脱下来。本实验中用的洗脱缓冲液为0.3m(nh4)2so4,以5%,10%,20%,30%,40%,60%,80%,100%梯度洗脱,每次洗脱均冲至基线平稳,流速为1.0ml/min,自动部分收集器每管收集1.0ml,收集有漆酶活性的组分进行native-page和sds-page检测,合并相应组分,收集纯酶。
(四)、含本发明蔗糖酶蛋白蔗糖水解能力检测
在ph5.0、45℃条件下,在反应体系中分别加入终浓度为0、50、300、600、900、1200、1500、1800和2000mmol/l的蔗糖溶液,测定gspinv酶活力。结果显示,在蔗糖浓度为300mm以下,gspinv的水解活性随着蔗糖浓度的增加而增加;随着蔗糖浓度的继续增加,gspinv的水解活力呈下降趋势,然而,当蔗糖浓度达到1000mmol/l时,活力还保留70%以上,酶蛋白比活力为1500u/mg,说明gspinv具有较好的蔗糖水解活性,且对蔗糖具有高的耐受特性,可用于高浓度果糖糖浆的制备,结果见图4。
以上实施例仅用以说明本发明的技术方案,而非对其限制;尽管参照前述实施例对本发明进行了详细的说明,本领域的普通技术人员应当理解:其依然可以对前述各实施例所记载的技术方案进行修改,或者对其中部分技术特征进行等同替换;而这些修改或者替换,并不使相应技术方案的本质脱离本发明各实施例技术方案的精神和范围。
<110>安徽大学
<120>一种蔗糖酶基因及其表达与应用
<130>...
<160>2
<170>patentinversion3.1
<210>1
<211>1761
<212>dna
<213>gongronella
<400>1
atggtgcttgctgatccttttaccaacatgcaagactcctttggcaagattgagactggt60
tatggcgccaacgacgcggatttcgtcaaatatcgcccaaaattccacgtcattggccct120
gtcaattggatgaacgatccatcggcgccctactttgatggcacgcattaccacatctat180
tatcagcacaatccctatgctcaaatttggggcaacatgacgtggggccatgccacctcg240
acagatctcctccattggcaagatcagccatttgcgctttatcctaattcgacttgggac300
atggagggcgcatttgacggcacatgcatggcggaaaagggatatcaaggcaagacgacg360
ttgatctatacggcggtcagctacagcggcccgacgtacgtcaatggccaagagaagcag420
gtgcttgcggtgacggacgacaatggcaattcgtggacacgcatcaagcctgtgatcaat480
ggcccgcccgacaattttgtggtcaatggcttccgcgacccttatttgttccgctccgcg540
tcgtttgacgcgttcctcaacatgcccaccgcgccagattcgatctacatgacggtgtcg600
tcgggtatcccagataagggcggcagactctggctgtaccactcgtccgattgggaaaat660
tgggatttccatggtccattcctgtttcacccgccaaatcacgtcgcgtcgcccaaccca720
gcattttacggcaatgacggcgtcaattgggaagttgcgtcgtattttgaactccctgat780
ccttctggtggcgattcttggcacattgcaacatggggctcgcaaggtggacgtaatgga840
gaagattttgaccattgggcgctctggactgctggacagcaattggacatgtcgatgccg900
gacaccaacgaggttgcgaagccgacgccacgaattcacgaaaccatgagcggcgtgtat960
gattggggcatcagttacgcgcaaatggccttcaaggatgcaaaggatcccaatggaaga1020
tacctcgccgtcggatgggttcaggatgatgtcattgctcccgccactagcgccaacagg1080
tggaatggtgtccttggtctctaccgcgagctctttatccaagaaatcaatggaattgac1140
gcttctgacccattgctgcaagagggctacgcgagctgggtgtatgacgcgtccacaaac1200
aaagtcaagaccctcggcatgcgtccgttacccgaatacgcgtccatgcgcggcaccggt1260
gtgtggtcactgcccaaaaataccaagaaaaccgacctcagcaatcttgccatccccatt1320
gactcgacccacgtggaaattgacgccgtcatcgccctggacaccaattccgaccccatc1380
agcttcgttgtgcgccaatctgcaaaggaagagaccgtcatcacctatattccttcccaa1440
ggcaatatcttcgtcaatcgcaccgcatccacctcgcgttccgacctgtggcgcacatcc1500
gatgaaacgcacgcgcttccgctcttccgtgtcaacaatggcggcctcaatggcaccggt1560
ggccttgagcccttgcatctgcgcgtctttgtcgacaattcgctgattgaggtctttgca1620
aacgaccgctacgccgtgtccacgcgtatctacccggatgacgccgatgccactagaatg1680
gcattgcgtgcgccagcaaatgtcaagatcacaagcctgaacgtctacccaatcactgac1740
agtgcgttcaatcgcccttga1761
<210>2
<211>586
<212>prt
<213>gongronella
<400>2
metvalleualaaspprophethrasnmetglnaspserpheglylys
151015
ilegluthrglytyrglyalaasnaspalaaspphevallystyrarg
202530
prolysphehisvalileglyprovalasntrpmetasnaspproser
354045
alaprotyrpheaspglythrhistyrhisiletyrtyrglnhisasn
505560
protyralaglniletrpglyasnmetthrtrpglyhisalathrser
65707580
thraspleuleuhistrpglnaspglnprophealaleutyrproasn
859095
serthrtrpaspmetgluglyalapheaspglythrcysmetalaglu
100105110
lysglytyrglnglylysthrthrleuiletyrthralavalsertyr
115120125
serglyprothrtyrvalasnglyglnglulysglnvalleualaval
130135140
thraspaspasnglyasnsertrpthrargilelysprovalileasn
145150155160
glyproproaspasnphevalvalasnglypheargaspprotyrleu
165170175
pheargseralaserpheaspalapheleuasnmetprothralapro
180185190
aspseriletyrmetthrvalserserglyileproasplysglygly
195200205
argleutrpleutyrhisserserasptrpgluasntrpaspphehis
210215220
glypropheleuphehisproproasnhisvalalaserproasnpro
225230235240
alaphetyrglyasnaspglyvalasntrpgluvalalasertyrphe
245250255
gluleuproaspproserglyglyaspsertrphisilealathrtrp
260265270
glyserglnglyglyargasnglygluasppheasphistrpalaleu
275280285
trpthralaglyglnglnleuaspmetsermetproaspthrasnglu
290295300
valalalysprothrproargilehisgluthrmetserglyvaltyr
305310315320
asptrpglyilesertyralaglnmetalaphelysaspalalysasp
325330335
proasnglyargtyrleualavalglytrpvalglnaspaspvalile
340345350
alaproalathrseralaasnargtrpasnglyvalleuglyleutyr
355360365
arggluleupheileglngluileasnglyileaspalaserasppro
370375380
leuleuglngluglytyralasertrpvaltyraspalaserthrasn
385390395400
lysvallysthrleuglymetargproleuproglutyralasermet
405410415
argglythrglyvaltrpserleuprolysasnthrlyslysthrasp
420425430
leuserasnleualaileproileaspserthrhisvalgluileasp
435440445
alavalilealaleuaspthrasnseraspproileserphevalval
450455460
argglnseralalysglugluthrvalilethrtyrileprosergln
465470475480
glyasnilephevalasnargthralaserthrserargseraspleu
485490495
trpargthrseraspgluthrhisalaleuproleupheargvalasn
500505510
asnglyglyleuasnglythrglyglyleugluproleuhisleuarg
515520525
valphevalaspasnserleuilegluvalphealaasnaspargtyr
530535540
alavalserthrargiletyrproaspaspalaaspalathrargmet
545550555560
alaleuargalaproalaasnvallysilethrserleuasnvaltyr
565570575
proilethraspseralapheasnargpro
580585
- 还没有人留言评论。精彩留言会获得点赞!
- 大豆低温诱导人工合成启动子SP5及应用的制造方法与工艺
- 一种棒状杆菌组成型表达载体启动子、含有该启动子及gnd基因的表达载体的制造方法与工艺
- 一种棒状杆菌组成型表达载体启动子、含有该启动子及lrp基因的表达载体的制造方法与工艺
- 一种二值卷积神经网络处理器及其使用方法与流程
- 检测NUP214‑ABL1基因相对表达量的试剂盒和方法与流程
- 环介导的级联放大策略用于高灵敏检测DNA甲基转移酶活性的制造方法与工艺
- 一种基于高压静电场来促进作物优质高效安全生产的嫁接方法与流程
- 一种酶性DNA机器用于miRNA检测的方法与流程
- 转基因玉米MON87403品系特异性实时荧光PCR检测引物、探针、方法和试剂盒与流程
- 葡萄早熟基因VvWRKY13在调控植物中乙烯生物合成中的应用的制造方法与工艺