一种稻瘟病抗性基因Piz‑t功能特异性分子标记及其检测方法和应用与流程
本发明属于农业生物技术领域,特别涉及一种稻瘟病抗性基因Piz-t功能性分子标记Piz-t-InDel及其检测方法与应用。
背景技术:
稻瘟病是危害水稻高产稳产最为严重的病害之一。当前生产中常用的化学防治虽在一定程度上可解决或减轻病害对水稻生产的影响,但农田长期用药不仅对环境有污染作用,而且农药残留物还会影响到稻米的食用品质。长期的生产实践表明,选育与利用抗病品种是防治稻瘟病最为安全有效的方法之一。然而由于稻瘟病菌生理小种致病性有频繁变异的特性,这会导致抗性单一的品种会在种植后3-5年时间里逐渐丧失抗性(殷得所等,2011);因此,挖掘和应用抗性基因是获得持久、广谱抗病品种的重要途径。早期的抗性基因鉴定方法主要是接种鉴定,但常规的接种鉴定方法不能准确地反映出抗病基因型,难以满足抗病分子育种的要求。随着水稻抗病分子遗传学的发展,很多抗性基因被精细定位或克隆(Su et al.2015);随着分子标记的发展及应用,其逐渐适用于抗性基因遗传背景的鉴定以及多抗病基因聚合育种的发展(Hittalmani et al.2000;Jena and Mackill,2008)。
但在已克隆的抗性基因中,一些重要的抗病基因位于同一位点,如Pik和Pi2/Pi9位点上的复等位基因之间、功能型序列与非功能型之间序列高度同源,一般的连锁标记仍然难以准确甄别各种材料的功能抗病基因(Qu et al.2006;Zhou et al.2006;Ashikawa et al.2008;Takahashi et al.2010;Yuan et al.2011;Zhai et al.2011;Yuan et al.2011;Hua et al.2012;Ma et al.2015;Tian et al.2016)。因此,直接分析功能等位基因本身的序列并开发其功能特异性的分子标记来对目标基因进行选择,不仅选择可靠性高,还会大大加快了育种步伐。
到目前为止,至少有六个稻瘟病抗性基因(Piz-t、Pi2、Pi9、Piz、Pigm、Pi50)已经被定位在水稻第6染色体短臂端的Pi2/Pi9基因簇内(Jiang etal.2012),其中Piz-t、Pi2、Pi9和Pi50等已经被成功克隆(Qu et al.2006,Zhou et al.2006)。研究表明,该位点的功能基因多具有广谱高抗的特性,其中Piz-t与水稻叶瘟抗性相关,对国内外多个稻瘟病小种表现出较强的叶瘟抗性。华丽霞等(2015)开发出了Piz-t的显性标记,该标记在抗性育种工作中容易发生错选或漏选的情况。为了能更准确有效地将稻瘟病广谱抗性基因Piz-t应用到水稻抗性育种工作中,有必要开发出该基因的特异性功能标记。鉴于前期我们已开发出Pi2和Pi9功能标记(杨立明等,2014;2015),本研究将着重于分型Piz-t与Pi2/Pi9位点其它功能基因及等位非功能基因的特异性标记。
技术实现要素:
解决的技术问题:为了克服现有技术的不足与缺点,本发明的首要目的在于提供一种稻瘟病抗性基因Piz-t基因功能特异性分子标记Piz-t-InDel。
本发明的另一发明目的在于提供所述的稻瘟病抗性基因Piz-t基因特异性分子标记Piz-t-InDel的检测方法。
本发明的再一发明目的在于提供所述的稻瘟病抗性基因Piz-t基因功能特异性分子标记Piz-t-InDel的应用。
技术方案:一种稻瘟病抗性基因Piz-t功能特异性分子标记,所述分子标记是通过引物对SEQ ID NO.1和SEQ ID NO.2从水稻基因组DNA中扩增出与稻瘟病抗性基因Piz-t呈特异性带型的分子标记,所述引物对序列为:
SEQ ID NO.1Fl:5’-TTACTAGAATCGCTCCAT-3’;
SEQ ID NO.2Rl:5’-ACTGCGTGCGACTTGA-3’。
所述与稻瘟病抗性基因Piz-t呈特异性带型的分子标记的序列如SEQ ID NO.3所示。
检测所述稻瘟病抗性基因Piz-t的功能特异性分子标记的方法,通过比对多个水稻稻瘟病抗性基因Piz-t的等位基因序列,包含以下步骤:(1)从公共数据库中下载得到的Piz-t及其同源的基因组序列,以及测序品种日本晴相对应区域的基因组序列,针对Piz-t位点进行序列比对,筛查Piz-t特异的、能区别于该位点其他稻瘟病等位抗性基因的插入/缺失(insertion-deletion,InDel)位点;(2)利用步骤(1)获得的InDel信息,根据InDel标记的设计原理,在所述InDel位点处上下游处设计基因特异性引物,引物对碱基序列如SEQ ID NO.1和SEQ ID NO.2所示;(3)以携带有稻瘟病抗性基因Piz-t的稻瘟病抗性品种IRBLZT-T的总DNA为模板,进行PCR扩增,所获得的PCR产物即为稻瘟病抗性基因Piz-t基因特异性分子标记Piz-t-InDel。
上述稻瘟病抗性基因Piz-t基因特异性分子标记在鉴别水稻品种稻瘟病抗性基因中的应用。
上述稻瘟病抗性基因Piz-t基因特异性分子标记在鉴别Pi2/Pi9基因簇区域不同的稻瘟病抗性基因中的应用。
一种鉴别水稻品种稻瘟病抗性基因的试剂盒,含有核酸序列如SEQ ID NO.3所示的分子标记,以及如SEQ ID NO.1和SEQ ID NO.2所述的引物对。
本发明的原理:InDel是指在近缘种或同一物种不同个体之间基因组同一位点的序列发生了不同大小核苷酸片段的插入或缺失,即一个序列上某一位点相比同源的另一个序列插入或缺失了一个或多个碱基(Weber et al,2002)。InDel标记基于PCR扩增技术,本质上属于长度多态性标记(Hyten et al,2010)。InDel标记稳定性好、多态性高、分型系统简单(Jander et al,2002);与分型系统复杂的SNP标记相比,InDel检测更加简单便捷,对仪器设备和技术要求较低,在电泳技术平台上即可进行。目前已开始应用于动植物群体遗传分析、分子辅助育种以及人类法医遗传学、医学诊断等领域。本发明通过多序列比对的方法寻找Piz-t基因序列上出现的InDel,由于这个InDel在Piz-t与Pi2/Pi9的其它功能/非功能基因有27bp的差异,因此很容易将Piz-t从中区分出来。本发明据此设计引物对F1/R1,通过常规PCR扩增,在聚丙烯酰胺凝胶电泳检测时,以不同大小的片断得以呈现。
有益效果:(1)本发明提供的分子标记特异性高:Piz-t所在的基因组区域存在着包括Piz-t在内的多个具有抗性基因特征的候选基因,这些候选基因在序列上高度同源;此外,Piz-t与该位点上已经克隆的稻瘟病抗性基因在碱基序列上高度一致。本发明提供的分子标记是本发明发明人经过不断的进行序列多态性搜查并通过实验验证得到的,能明显地将Piz-t与存在于该基因组区域内与Piz-t高度同源的旁系同源基因进行区分。
(2)本发明提供的分子标记在实际应用中,低成本、高通量:目前大多是利用电泳平台进行分型,该分型平台快捷经济,不需要复杂的实验设备,可操作性强。电泳分型平台有琼脂糖凝胶电泳、变性或非变性聚丙烯酰胺凝胶电泳以及毛细管电泳。本发明提供的分子标记只需PCR结合琼脂糖凝胶电泳或非变性聚丙烯酰胺凝胶电泳,成本低、通量高、加上特异性高(即准确度高),特别适用于生产实践中。
(3)本发明是第一个针对Piz-t基因内部序列所开发的Piz-t基因特异性InDel标记。本发明能通过电泳检测的方法成功地将Piz-t与定位在该位点上的其它稻瘟病抗性基因区分开,到目前为止,还没有有关这类标记的报道。本发明所提供的Piz-t特异性分子标记Piz-t-InDel是一个共显性标记,在实际应用过程中其可靠性及准确性优于显性标记。本发明可应用于Piz-t的种质资源筛选、转基因鉴定和基因聚合以及基于MAS技术的水稻抗性育种工作中。该标记存在于Piz-t基因内部,因此对Piz-t的筛选能力理论值可达100%,其综合性能优于已报道的与Piz-t连锁的分子标记和功能标记。
(4)本发明提供的分子标记能简便地应用于遗传背景不同的群体中。现有的与Piz-t连锁的分子标记大部分都是针对同一个群体2个不同亲本的序列多态性所开发的,这些标记在其它群体中的适用性有限。华丽霞等(2015)等开发的功能标记,不仅鉴定过程比较繁琐,而且还是显性标记,不适应于大规模的种质资源鉴定及MAS育种中。本发明适用于任何遗传背景下的转基因育种,基因聚合以及基于MAS技术的抗性育种,无需重复进行亲本多态性的筛选,大大提高了育种效率。
因此,本发明所提供的稻瘟病抗性基因Piz-t基因特异性分子标记具有重要的应用价值,利用此标记可以提高该基因在种质资源筛选、分子标记辅助选择育种、基因聚合育种,以及转基因育种中利用的效率。
附图说明
图1是Piz-t基因特异性分子标记Piz-t-InDel在Pi2/Pi9基因簇区域不同稻瘟病抗性基因及日本晴、9311的验证结果图,其中:泳道DL500为DNA ladder,泳道的DNA模板依次为IRBLzt-T(Piz-t)、IR65482-4-136-2-2、谷梅2号、谷梅4、二八占、Fukunishiki。
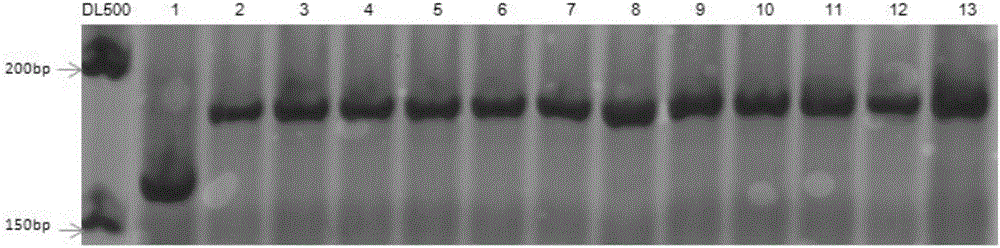
图2是Piz-t基因特异性分子标记Piz-t-InDel在其它水稻品种中的验证结果图,其中:泳道DL500为DNA ladder,泳道1-13的DNA模板依次为IRBLzt-T(Piz-t)、矮洋稻、辽粳5号、TQ155、Masoli、BG1100、TRAT、5011、培矮64、9311、杨恢136-4、中农青、34重庆恢。
具体实施方式
一种稻瘟病抗性基因Piz-t基因功能特异性分子标记Piz-t-InDel,是通过引物对SEQ ID NO.1和SEQ ID NO.2从水稻基因组DNA中扩增出与稻瘟病抗性基因Piz-t呈特异性带型的分子标记;SEQ ID NO.1(5’-3’):TTACTAGAATCGCTCCAT;SEQ ID NO.2(5’-3’):ACTGCGTGCGACTTGA;
所述的水稻品种为IRBLzt-T(Piz-t供体亲本);
所述的稻瘟病抗性基因Piz-t包括编码NBS-LRR类蛋白的基因片段I,序列如下所示
SEQ ID NO.3:
TTACTGGAATCGCTCCATTTTCGTATTTGAAAATATTTGATTATGTTTTTTATGTGGGGTTTCTGATTCCAATTAAAAAAA-TGAAAATAAAAATGGTATGATGGTTTCCGTTCGTTATGCATGCGCGGAACAATGGATCTCACTAATCAAGTAGCACGCAGT
所述的稻瘟病抗性基因Piz-t的功能特异性分子标记Piz-t-InDel的检测方法,通过比对多个水稻稻瘟病抗性基因Piz-t的等位基因序列,包含以下步骤:(1)从公共数据库中下载得到的Piz-t及其同源的基因组序列Pi50以及测序品种日本晴(Nipponbare)相对应区域的基因组序列,针对NBS1~9-Piz-t位点进行序列比对,筛查Piz-t特异的、能区别于该位点其他稻瘟病等位抗性基因的插入/缺失(insertion-deletion,InDel)位点。(2)利用步骤(1)获得的InDel信息,根据InDel标记的设计原理,在所述InDel位点处上下游100-200bp处设计基因特异性引物,利用引物对扩增IRBLz-FU(Piz)、谷梅4(Pigm(t))、二八占(Pi50(t))、谷梅2(P26(t))、IR65482-4-136-2-2(Pi40(t))、日本晴和9311,测序比对,直至在NBS2-Piz-t筛选到能明显区分出Piz-t引物对,碱基序列如下:Fl:5’-TTACTAGAATCGCTCCAT-3’;Rl:5’-ACTGCGTGCGACTTGA-3’;(3)以携带有稻瘟病抗性基因Piz-t的稻瘟病抗性品种IRBLzt-T的总DNA为模板,进行PCR扩增,所获得的PCR产物即为稻瘟病抗性基因Piz-t基因特异性分子标记Piz-t-InDel。
稻瘟病抗性基因Piz-t基因特异性分子标记Piz-t-InDel在鉴别水稻品种稻瘟病抗性基因的应用,特别适合于在鉴别Pi2/Pi9基因簇区域不同的稻瘟病抗性基因的应用;优选包含如下步骤:(1)扩增:利用引物Fl和Rl对待检测的水稻品种的基因组进行PCR;(2)检测:利用聚丙烯酰胺凝胶电泳检测,若检测到分子量大小为163bp的核苷酸片段,则携带有稻瘟病抗性基因Piz-t;若检测到分子量大小为190bp,则待检测的水稻品种不携带Piz-t位点的功能基因。
下面结合实施例及附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。
实施例1
稻瘟病抗性基因Piz-t基因特异性分子标记及其引物设计与检测:
(I)Piz-t插入/缺失(insertion-deletion,InDel)位点的分析:
从公共数据库中下载得到Piz-t等水稻供体品种的部分基因组序列,针对NBS1~9-Piz-t位点进行序列比对,筛查Piz-t特异的、能区别于该位点其他稻瘟病等位抗性基因的特异性插入/缺失(InDel)差异位点设计引物,扩增IRBLz-FU(Piz)、谷梅2(P26(t))、谷梅4(Pigm(t))、二八占(Pi50(t))、IR65482-4-136-2-2(Pi40(t))、日本晴,测序、比对结果下表所示:
其中,----即为鉴定到的Piz-t基因特异性缺失序列;
(2)设计引物:
根据InDel标记的设计原理,在所述InDel位点处上下游100-200bp处设计引物,引物对碱基序列如下所示:
Fl:5′-TTACTAGAATCGCTCCAT-3′;
Rl:5′-ACTGCGTGCGACTTGA-3′。
(3)选择水稻代表品种:选择携带有Piz-t基因以及Pi2/Pi9基因族等位基因的代表品种如下:
IRBLzt-T(Piz-t)、IR65482-4-136-2-2(Pi40(t))、谷梅2(P26(t))、谷梅4(Pigm(t))、二八占(Pi50(t))、Fukunishiki。
(4)PCR扩增,获得含有Piz-t基因特异性InDel的片段
利用上述引物对Fl和R1,以上述水稻品种的总DNA为模板,进行常规PCR扩增后进行聚丙烯酰胺凝胶电泳检测,得到的结果如图1所示。
扩增反应体系如下:
2x Reaction Mix:12.5μL
引物Fl(10μΜ):1μL
引物Rl(10μΜ):1μL
Golden DNA Polymerase:0.2μL
DNA模板(20-50ng/μL):1μL
ddH2O:补足至25μL。
PCR温度循环条件如下:94℃5分钟;94℃30秒、63℃30秒、72℃30秒,35个循环;72℃7分钟;10℃保存。
PCR反应结束后,取适量样品在8%的聚丙烯酰胺凝胶上进行电泳检测,电泳条件为90V,1小时。
其中,图1的泳道I为以Piz-t供体品种水稻品种IRBLzt-T基因组为模板PCR得到的片段,与稻瘟病抗性基因Piz-t呈特异性带型,即泳道1为Piz-t基因特异性分子标记Piz-t-InDel。图1的结果显示,Piz-t基因特异性分子标记既能区分抗感等位基因,又能区分Pik位点上所鉴定到的其它抗性基因。也就是说,携带有Piz-t的水稻品种,其PCR扩增产物的电泳检测条带以163bp呈现,其它Pi2/Pi9基因族等位基因稻瘟病抗性基因及非功能基因以电泳检测条带190bp呈现。
实施例2
抗性基因Piz-t基因特异性分子标记在其它水稻品种中的检测应用:
选择13个代表性水稻品种,依次为:IRBLzt-T(Piz-t)、矮洋稻、辽粳5号、TQ155、Masoli、BG1100、TRAT、5011、培矮64、9311、杨恢136-4、中农青、34重庆恢。
分别提取其基因组DNA,并以此为模板,按照实施例1的方法,进行PCR扩增、酶切及电泳检测。根据酶切产物(条带)的大小,可以把抗性基因Piz-t与其他稻瘟病抗性基因区分开来。如图2所示,在抗谱表型为Piz-t型的个体(携带有稻瘟病抗性基因Piz-t的抗病品种IRBLZT-T,泳道1的样品)中能检测到Piz-t-InDel基因特异性分子标记的存在,而其他抗谱表型的个体则以另外一种带型呈现。可见,试验结果与设计分析的相吻合,说明抗性基因Piz-t基因特异性分子标记可在鉴别Piz-t基因与其他Pi2/Pi9基因簇等位基因等方面得到应用。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
SEQUENCE LISTING
<110> 杨海军
<120> 一种稻瘟病抗性基因Piz-t功能特异性分子标记及其应用
<130>
<160> 10
<170> PatentIn version 3.3
<210> 1
<211> 18
<212> DNA
<213> 人工序列
<400> 1
ttactagaat cgctccat 18
<210> 2
<211> 16
<212> DNA
<213> 人工序列
<400> 2
actgcgtgcg acttga 16
<210> 3
<211> 162
<212> DNA
<213> 人工序列
<400> 3
ttactggaat cgctccattt tcgtatttga aaatatttga ttatgttttt tatgtggggt 60
ttctgattcc aattaaaaaa atgaaaataa aaatggtatg atggtttccg ttcgttatgc 120
atgcgcggaa caatggatct cactaatcaa gtagcacgca gt 162
<210> 4
<211> 35
<212> DNA
<213> 人工序列
<400> 4
taaaaatggt atgatggttt ccgttcgtta tgcat 35
<210> 5
<211> 62
<212> DNA
<213> 人工序列
<400> 5
taaaaatggt atgatggttt ccgttcgttt tcgagccgtt tcatccctaa ttgcacatgc 60
at 62
<210> 6
<211> 62
<212> DNA
<213> 人工序列
<400> 6
taaaaatggt atgatggttt ccgttcgttt tcgagccgtt tcatccctaa ttgcacatgc 60
at 62
<210> 7
<211> 62
<212> DNA
<213> 人工序列
<400> 7
taaaaatggt atgatggttt ccgttcgttt tcgagccgtt tcatccctaa ttgcacatgc 60
at 62
<210> 8
<211> 62
<212> DNA
<213> 人工序列
<400> 8
taaaaatggt atgatggttt ccgttcgttt tcgagccgtt tcatccctaa ttgcacatgc 60
at 62
<210> 9
<211> 62
<212> DNA
<213> 人工序列
<400> 9
taaaaatggt atgatggttt ccgttcgttt tcgagccgtt tcatccctaa ttgcacatgc 60
at 62
<210> 10
<211> 62
<212> DNA
<213> 人工序列
<400> 10
taaaaatggt atgatggttt ccgttcgttt tcgagccgtt tcatccctaa ttgcacatgc 60
at 62
- 还没有人留言评论。精彩留言会获得点赞!
- 水稻稻瘟病抗性基因Pi2‑1的分子标记方法及应用与流程
- 一种稻瘟病抗性基因Piz‑t功能特异性分子标记及其检测方法和应用与流程
- 一种接种架及稻瘟病菌接种系统的制作方法与工艺
- 谷梅4号抗稻瘟病基因Pigm的分子标记及其应用的制作方法与工艺
- 水稻稻瘟病抗性基因Pita、Pib分子标记方法与流程
- 一种基于猪非典型瘟病毒E2基因的荧光定量RT‑PCR检测试剂盒的制作方法与工艺
- 稻瘟病菌LAMP检测引物组及应用的制造方法与工艺
- 一种制备稻瘟病菌促分裂原活化蛋白激酶MoMps1晶体的方法与流程
- 水稻抗稻瘟病Pi9基因的共显性功能分子标记Pi9InDel1及其应用的制造方法与工艺
- 水稻抗稻瘟病Pi9基因的共显性功能分子标记Pi9InDel2及其应用的制造方法与工艺
- 玉米低镁条件下特异性转运镁离子基因ZmMGT10及其应用的制造方法与工艺
- CRISPR‑Cas9特异性敲除猪SLA‑3基因的方法及用于特异性靶向SLA‑3基因的sgRNA与制造工艺
- 毛酸浆的分子特异性标记引物及鉴别方法与制造工艺
- 一种检测CYP2C9基因多态性的ARMS引物的制造方法与工艺
- 构建在海马体区域特异性敲除IKKα基因的小鼠模型的方法及打靶载体和试剂盒与制造工艺
- 使用重叠非等位基因特异性引物和等位基因特异性阻断剂寡核苷酸的组合物进行等位基因特异性扩增的制造方法与工艺
- 一种利用线粒体D-loop区特异区域鉴定鼋的标记引物及方法
- 一种用于检测大豆拟茎点种腐病菌的特异性lamp引物组合物及其应用
- 一种针对膨胀污泥中Eikelboom Type 0092型丝状菌群特异性引物和方法
- 脯氨酸特异性蛋白酶基因及其应用
- 一种泰国库蠓的特异基因及其分子鉴定方法
- 一种美丽库蠓特异基因及其分子鉴定方法
- 一种特异于禽类原始生殖细胞的基因转移系统及方法
- CRISPR-Cas9特异性敲除猪SALL1基因的方法及用于特异性靶向SALL1基因的sgRNA的制作方法
- CRISPR-Cas9特异性敲除猪CMAH基因的方法及用于特异性靶向CMAH基因的sgRNA的制作方法
- CRISPR-Cas9特异性敲除猪SLA-2基因的方法及用于特异性靶向SLA-2基因的sgRNA的制作方法
- 一种安宁铗蠓的特异基因及其分子鉴定方法
- CRISPR-Cas9特异性敲除猪GGTA1基因的方法及用于特异性靶向GGTA1基因的sgRNA的制作方法
- 一种蓝腹库蠓的特异基因及其分子鉴定方法
- 扬子鳄抗病毒潜力检测的ⅰ类mhc基因的特异性引物及分型方法
- 一种检测水稻粒宽等位基因的特异性PCR分子标记和方法与流程
- 丝状真菌中多个基因拷贝的同时位点特异性整合的制造方法与工艺
- 一种特异性敲减人BRCC3基因慢病毒的构建及在肺癌中的应用的制造方法与工艺
- 烟粉虱MED隐种高温耐受相关基因特异性探针及其应用的制造方法与工艺
- CRISPR/SaCas9特异性敲除人CXCR4基因的方法与流程
- 玉米低镁条件下特异性转运镁离子基因ZmMGT10及其应用的制造方法与工艺
- CRISPR‑Cas9特异性敲除猪SLA‑3基因的方法及用于特异性靶向SLA‑3基因的sgRNA与制造工艺
- 构建在海马体区域特异性敲除IKKα基因的小鼠模型的方法及打靶载体和试剂盒与制造工艺
- 使用重叠非等位基因特异性引物和等位基因特异性阻断剂寡核苷酸的组合物进行等位基因特异性扩增的制造方法与工艺
- 用于检测烟草对tmv抗性的n基因特异性引物对、检测方法及试剂盒的制作方法