一种提高4-α-糖基转移酶可溶性表达水平的方法与流程
本发明涉及一种提高4-α-糖基转移酶可溶性表达水平的方法,属于发酵工程技术领域。
背景技术
5-α-糖基转移酶是一种多功能酶,属于淀粉水解酶家族。由于4-α-糖基转移酶具有独特的催化特性,可以催化环化、歧化、水解和偶合四种反应,使其在改性淀粉、大环糊精等生产中具有广泛的用途。
如在淀粉改性方面,chok.h.等人应用来源于栖热水生菌的4-α-糖基转移酶对大米淀粉进行处理,结果表明经过该酶处理后,淀粉的回生现象得到很大程度的抑制,hav.do等人应用来源于栖热水生菌的4-α-糖基转移酶制备淀粉凝胶,分别测定不同加酶量、不同处理时间下玉米淀粉凝胶的质构特性,最终制备出粘弹性较好的理想凝胶,也有研究已经证实,采用微生物来源的4-α-糖基转移酶制备淀粉质凝胶,可以得到与明胶性质相似的淀粉质凝胶;在生产大环糊精方面,4-α-糖基转移酶可以通过其环化作用转化淀粉生成大环糊精,实现一步法制备大环糊精。
因此,如何实现4-α-糖基转移酶的工业生产以及高效生产一直是一个研究热点。
目前,已经有许多研究是基于基因工程技术,通过构建基因工程菌以尝试提高4-α-糖基转移酶的表达量,如许燕等构建产栖热水生菌嗜热4-α-糖基转移酶重组大肠杆菌,并对该没进行了纯化和性质分析,结果显示该酶比活力为156.25u/mg,该酶可以催化淀粉制备大圆环糊精,最高转化率为45.58%;张磊等构建了产thermusthermophilushb84-α-糖基转移酶重组大肠杆菌,采用两步法获得了纯酶,该酶可以催化麦芽糖生成葡萄糖、麦芽三糖、麦芽四糖、麦芽五糖和麦芽六糖。
但是,上述研究或者对4-α-糖基转移酶的表达依处在一个很低的水平,或者由于表达量过低而没有报道酶的实际表达量,因此,如何实现4-α-糖基转移酶的工业生产以及高效生产仍需进一步研究。
技术实现要素:
为解决上述问题,本发明提供了一种提高4-α-糖基转移酶可溶性表达水平的方法。此方法从4-α-糖基转移酶的发酵生产工艺入手,通过在4-α-糖基转移酶生产菌株的发酵培养基中添加一定量的氧化三甲胺和/或脯氨酸,以提高其对4-α-糖基转移酶的表达水平;采用此方法发酵48h,可成功将细胞破碎上清液中的4-α-糖基转移酶酶活提高至1302.6u/ml;且此方法具有工艺简单易行,适合工业化生产的优点。
本发明技术方案如下:
本发明提供了一种提高4-α-糖基转移酶可溶性表达水平的方法,所述方法为使用添加了氧化三甲胺和/或脯氨酸的发酵培养基进行发酵培养;所述发酵培养所用的生产菌株为可产4-α-糖基转移酶的菌株。
在本发明的一种实施方式中,所述生产菌株包含可产4-α-糖基转移酶的基因工程菌。
在本发明的一种实施方式中,所述基因工程菌包含产4-α-糖基转移酶的重组枯草芽孢杆菌以及可产4-α-糖基转移酶的重组大肠杆菌。
在本发明的一种实施方式中,所述基因工程菌为重组大肠杆菌bl21(de3)/petduet-groes-groel-4gt或重组枯草芽孢杆菌bacillussubtilis168/pht01-4gt。
在本发明的一种实施方式中,所述氧化三甲胺在发酵培养基中的添加量为0.5~10g/l。
在本发明的一种实施方式中,所述氧化三甲胺在发酵培养基中的添加量为2.3g/l。
在本发明的一种实施方式中,所述脯氨酸在发酵培养基中的添加量为0.5~5g/l。
在本发明的一种实施方式中,所述所述脯氨酸在发酵培养基中的添加量为1.15g/l。
在本发明的一种实施方式中,所述发酵培养基的成分包含甘油4~10g/l、蛋白胨0.5~3g/l、酵母粉0.5~5g/l、kh2po45~15g/l、(nh4)2hpo41~6g/l、柠檬酸0.5~4g/l、mgso40.2~1.5g/l以及微量元素液5~20ml/l。
在本发明的一种实施方式中,所述所述发酵培养基的成分包含甘油8g/l、蛋白胨1g/l、酵母粉2g/l、kh2po413.5g/l、(nh4)2hpo44g/l、柠檬酸1.7g/l、mgso40.68g/l以及微量元素液10ml/l。
在本发明的一种实施方式中,所述微量元素液的成分包含feso4·7h2o5~20g/l、znso4·7h2o1.5~5g/l、cuso4·5h2o0.5~2g/l、mnso4·4h2o0.2~1.0g/l、na2b4o7·10h2o0.1~0.5g/l、cacl20.5~5g/l、(nh4)6mo7o25~50g/l。
在本发明的一种实施方式中,所述微量元素液的成分包含feso4·7h2o10g/l、znso4·7h2o2.25g/l、cuso4·5h2o1.0g/l、mnso4·4h2o0.5g/l、na2b4o7·10h2o0.23g/l、cacl22.0g/l、(nh4)6mo7o240.1g/l。
本发明提供了上述一种提高4-α-糖基转移酶可溶性表达水平的方法在4-α-糖基转移酶制备、淀粉改性以及大环糊精生产方面的应用。
有益效果:
本发明的方法从4-α-糖基转移酶的发酵生产工艺入手,通过在4-α-糖基转移酶生产菌株的发酵培养基中添加一定量的氧化三甲胺和/或脯氨酸,以提高其对4-α-糖基转移酶的表达水平;采用本发明的方法发酵48h,可成功将发酵上清液中的4-α-糖基转移酶酶活提高至1302.6u/ml;且本发明的方法具有工艺简单易行,适合工业化生产的优点。
附图说明
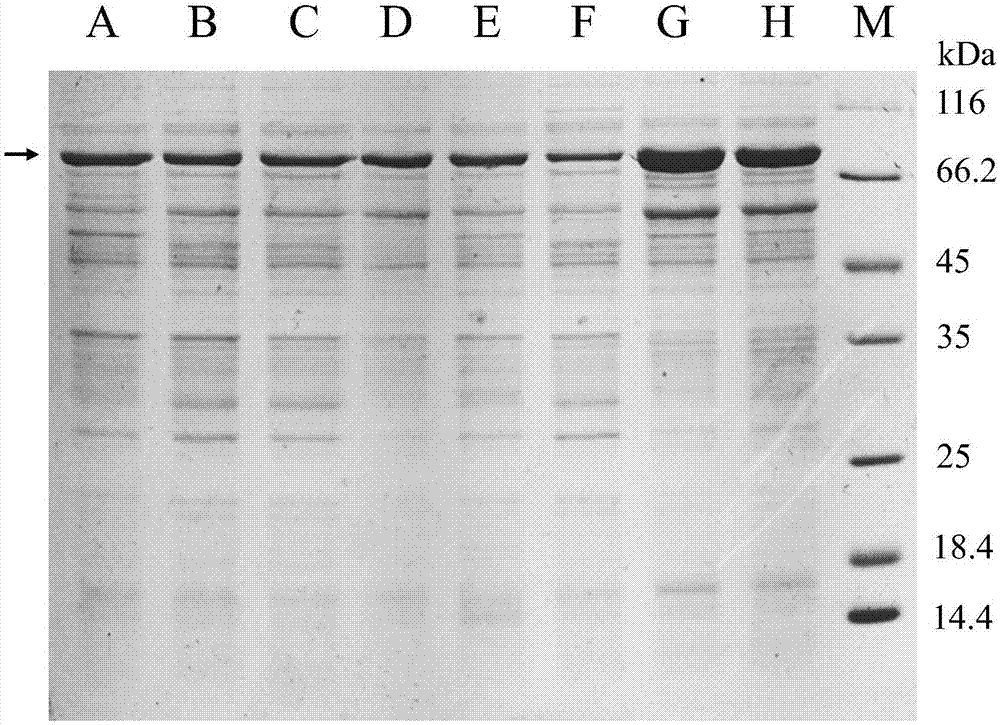
图1:重组菌株在添加不同种类化学添加剂培养基中摇瓶发酵细胞破碎上清样品sds-page电泳图;
图2:重组菌株在3-l发酵罐中菌体生长及产酶曲线。
具体实施方式
通过以下的具体实施方式,对本发明的上述内容再作进一步的详细解释及阐述。
下述实施例中涉及的培养基如下(以g/100ml计):
摇瓶lb液体培养基:酵母粉5,蛋白胨10,nacl10,ph7.0;
摇瓶tb发酵培养基:蛋白胨12,酵母粉24,甘油5,k2hpo4·3h2o16.43,kh2po42.3;
3l发酵罐发酵培养基:甘油8,蛋白胨1,酵母粉2,kh2po413.5,(nh4)2hpo44,柠檬酸1.7,mgso40.68,微量元素液10ml/l;
微量元素液:feso4·7h2o10,znso4·7h2o2.25,cuso4·5h2o1.0,mnso4·4h2o0.5,na2b4o7·10h2o0.23,cacl22.0,(nh4)6mo7o240.1;
3l发酵罐补料培养基:甘油500,mgso4·7h2o20,蛋白胨15,酵母粉30,氧化三甲胺2.3。
下述实施例中涉及的检测方法如下:
4-α-糖基转移酶酶活检测方法:
(1)酶活力测定方法:
配制1%的麦芽糖溶液,即称取0.10g麦芽糖溶解pbs(20mm·l-1,ph7.4)中,然后每个离心管分装300μl,放置75℃水浴锅加热5min。取出冰上加50μl的酶,空白对照管加50μl的去离子水,再放置75℃水浴锅加热30min。加热结束后,取出冰上,生物传感分析仪测定葡萄糖的量(以同样条件下用缓冲液代替酶液的反应体系作为空白对照)。
酶活力(u)定义为:在上述分析测定条件下,1h水解麦芽糖产生1μm的葡萄糖所需的酶量定义为一个酶活力单位。
(2)样品处理及检测
将菌液样品稀释适当倍数,用分光光度计在波长600nm处测定菌液的吸光值,以去离子水作为空白对照(od600=吸光值×稀释倍数)。
再将发酵菌液置于50ml离心管中,于4℃、6000rpm离心20min,弃去上清,收集菌体细胞。将离心后的菌体用等体积的磷酸盐缓冲液重悬,置于冰上进行超声破碎,功率采用130kw,超声3s,间歇5s,共20min。破碎后细胞悬液置于4℃、10000rpm离心5min,收集离心上清(破碎细胞上清部分),然后采用标准的sds-page电泳方法和酶活性检测确定重组蛋白的表达水平和酶活力。
菌体浓度检测方法:
od600的测定:发酵过程中通过测定发酵液的吸光度来监测发酵过程中细胞生长情况。具体方法为发酵液用生理盐水稀释后测定od600的吸光值。
细胞干重测定:准确移取5ml发酵液,10000g离心10min,弃上清后加入生理盐水悬浮菌体沉淀,继续在10000g离心10min,倒掉上清,剩下的菌体在105℃烘干至恒重,冷却后测定细胞干重。
下述实施例中涉及的4-α-糖基转移酶生产菌株如下:
实验室前期构建的重组菌株bl21(de3)/petduet-groes-groel-4gt和bacillussubtilis168/pht01-4gt
重组菌株e.colibl21(de3)/petduet-groes-groel-4gt的构建
1、4-α-糖基转移酶编码基因密码子优化及合成
4-α-糖基转移酶氨基酸序列可由ncbi数据库获得,序列号为wp_004067291.1。
根据大肠杆菌密码子偏好性,通过人工设计并合成经过密码子优化的4-α-糖基转移酶编码核苷酸(dna)序列,经过测序验证,获得目的基因片段(合成的时候在两端分别加上ndei和xhoi酶切位点以便于基因克隆)。
4-α-糖基转移酶的氨基酸序列如seqidno.1所示,其优化后的核苷酸序列如seqidno.2所示。
2、重组质粒pet24-4gt的构建
将经合成的基因片段,采用ndei和xhoi双酶切,胶回收后与以相同的两个酶酶切并胶回收纯化的pet24a(+)载体(购自于novagen公司)进行连接。
连接产物转化大肠杆菌e.colijm109感受态细胞,将转化后感受态细胞涂布含30μg/ml卡那霉素抗性的lb-琼脂平板(含有lb固体培养基的平板培养基),37℃培养过夜;挑取转化子转接到lb培养基中,37℃振荡培养8h,提取质粒;重组质粒经过酶切验证并且基因测序,确认重组载体pet24-4gt正确。
pet24a(+)载体含有t7启动子,可以通过添加iptg或者乳糖来诱导启动目的基因的表达。
3、重组质粒petduet-4gt的构建
将pet24-4gt采用ndei和xhoi双酶切,胶回收后与以相同的两个酶酶切并胶回收纯化的petduet-1载体(购自novagen公司产品)进行连接。
连接产物转化大肠杆菌e.colijm109感受态细胞,将转化后感受态细胞涂布含100μg/ml氨苄青霉素抗性的lb-琼脂平板,37℃培养过夜;挑取转化子转接到lb培养基中,37℃振荡培养8h,提取质粒;重组质粒经过酶切验证并且基因测序,确认重组载体petduet-4gt正确。
petduet-1载体含有两个t7启动子,4gt位于其中一个t7启动子下游,可以通过添加iptg或者乳糖来诱导启动4gt基因的表达。
4、重组质粒petduet-groes-groel-4gt的构建
以重组质粒petduet-4gt为模板,设计引物,扩增得到7313bp的dna片段;
pcr引物如下:
核苷酸序列为seqidno.4的petduet-f:gccaggatccgaattcg
核苷酸序列为seqidno.5的petduet-r:ggtatatctccttcttaaag
pcr反应体系为50μl:ddh2o32.5μl,petduet-4gt质粒dna模板1μl,上游引物petduet-f1μl,下游引物petduet-r1μl,dntpmix4μl,5×primestarbuffer10μl,primestar0.5μl;
pcr反应条件为94℃预变性4min,进行94℃10s,55℃5s,72℃7min30s,共30个循环,最后72℃延伸10min;
反应结束后将pcr产物进行琼脂糖电泳,胶回收备用。
大肠杆菌e.colik12(atcc27325)购于美国菌种保藏中心,以该菌株基因组作为模板,扩增得到分子伴侣蛋白groes-groel编码基因,核苷酸序列如seqidno.3所示;
引物设计如下(引物中用下划线标注序列为与表达载体互补序列,用于in-fusion连接):
核苷酸序列为seqidno.6的groes-groel-for:agaaggagatataccatgaatattcgtccattgcatgatcgc
核苷酸序列为seqidno.7的groes-groel-rev:aattcggatcctggcttacatcatgccgcccatgcca
pcr反应体系为50μl:ddh2o32.5μl,e.colik12基因组dna模板1μl,上游引物petduet-f1μl,下游引物petduet-r1μl,dntpmix4μl,5×primestarbuffer10μl,primestar0.5μl;
pcr反应条件为94℃预变性4min,进行94℃10s,55℃5s,72℃2min10s,共30个循环,最后72℃延伸10min;
反应结束后将pcr产物进行琼脂糖电泳,胶回收备用。
将pcr扩增得到的质粒petduet-4gtdna片段和groes-groel编码基因片段,采用如下连接方式连接:质粒dna片段3μl,groes-groel编码基因dna片段5μl,5×in-fusionhd酶混合液2μl,混合均匀,置于50℃水浴锅中孵育15min,然后置于冰上冷却5min。
将连接产物全部转化大肠杆菌e.colijm109感受态细胞中(大肠杆菌感受态细胞制备及转化参照分子克隆实验手册),将转化后感受态细胞涂布含100μg/ml氨苄青霉素抗性的lb-琼脂平板,37℃培养过夜;挑取转化子转接到lb培养基中,37℃振荡培养8h,提取质粒;重组质粒经过酶切验证并且基因测序,确认正确的重组载体命名为petduet-groes-groel-4gt。
5、重组菌株的构建
将重组表达质粒petduet-groes-groel-4gt转化大肠杆菌e.colibl21(de3)(invitrogen公司)感受态细胞。
重组菌株枯草芽孢杆菌bacillussubtilis168/pht01-4gt的构建
宿主菌株为枯草芽孢杆菌bacillussubtilis168(购自美国菌种保藏中心atcc,保藏编号为23857),表达载体为pht01(购自德国mobitec公司),该质粒启动子为pgrac,可以通过添加iptg诱导目的基因的表达。
枯草芽孢杆菌没有密码子偏好性,因此以含有4-α-糖基转移酶基因petduet-4gt质粒dna为模板,设计引物,扩增得到两端分别带有bamhi和smai酶切位点的4-α-糖基转移酶基因dna片段。
pcr引物如下:
核苷酸序列为seqidno.8的pht01-f:ggatccatgaatattcgtccattgca
核苷酸序列为seqidno.9的pht01-r:cccgggttacatcatgccgcccatgc
pcr反应体系为50μl:ddh2o32.5μl,petduet-4gt质粒dna模板1μl,上游引物pht01-f1μl,下游引物pht01-r1μl,dntpmix4μl,5×primestarbuffer10μl,primestar0.5μl;
pcr反应条件为94℃预变性4min,进行94℃10s,55℃5s,72℃7min30s,共30个循环,最后72℃延伸10min;
反应结束后将pcr产物进行琼脂糖电泳,胶回收后采用bamhi和smai双酶切,胶回收后与以相同的两个酶酶切并胶回收纯化的pht01载体(购自于购自德国mobitec公司)进行连接。
连接产物转化大肠杆菌e.colijm109感受态细胞,将转化后感受态细胞涂布含100μg/ml氨苄青霉素抗性的lb-琼脂平板(含有lb固体培养基的平板培养基),37℃培养过夜;挑取转化子转接到lb培养基中,37℃振荡培养8h,提取质粒;重组质粒经过酶切验证并且基因测序,确认重组载体pht01-4gt正确。将重组表达质粒pht01-4gt转化大肠杆菌e.coli枯草芽孢杆菌bacillussubtilis168感受态细胞,得到重组菌株bacillussubtilis168/pht01-4gt。
实施例1
具体过程如下:
分别以构建的重组大肠杆菌bl21(de3)/petduet-groes-groel-4gt和重组枯草芽孢杆菌bacillussubtilis168/pht01-4gt为发酵菌种。
将构建的重组大肠杆菌bl21(de3)/petduet-groes-groel-4gt和重组枯草芽孢杆菌bacillussubtilis168/pht01-4gt分别接种到到lb液体培养基(分别含有100μg/ml氨苄青霉素和5μg/ml氯霉素)中,置于恒温摇床上200rpm/min,37℃振荡培养8h,作为发酵用种子。
分别取2.5ml种子接种到添加了苯甲醇、肌醇、海藻糖、甜菜碱、甘氨酸、氧化三甲胺和脯氨酸(10mmol/l)的50mltb培养基中(分别含有100μg/ml氨苄青霉素和5μg/ml氯霉素),并且以不添加上述任何一种添加剂的tb培养基(分别含有100μg/ml氨苄青霉素和5μg/ml氯霉素)作为对照。
接种后,置于28℃200r·min-1条件下振荡培养4h,加入0.2mmiptg,诱导培养24h。
将发酵液进行菌体浓度以及4-α-糖基转移酶酶活检测(检测结果见表1和图1)。
结果显示,当使用重组大肠杆菌bl21(de3)/petduet-groes-groel-4gt时,苯甲醇、肌醇、甘氨酸对菌体生长和产酶都有明显的抑制作用,海藻糖、甜菜碱对具体生长有一定的抑制作用,其中,海藻糖不利于重组酶的生产,而甜菜碱对重组酶产量没有明显影响;氧化三甲胺对菌体生长没有影响,但是最有利于产酶水平的提高,酶活力为140.3u/ml;脯氨酸对菌体生长和产酶都有一定的促进作用,酶活力为130.3u/ml。
当使用重组枯草芽孢杆菌bacillussubtilis168/pht01-4gt时,不同添加剂对枯草芽孢杆菌生长没有明显影响,但是对酶活力影响情况与重组大肠杆菌类似。苯甲醇、肌醇、甘氨酸、海藻糖对产酶都有抑制作用,而甜菜碱对重组酶产量没有明显影响;氧化三甲胺有利于产酶水平的提高,酶活力为25.7u/ml;脯氨酸,酶活力为21.9u/ml。
表1菌体浓度和酶活力检测结果
其中,a-h为使用重组大肠杆菌bl21(de3)/petduet-groes-groel-4gt的检测结果;i-p为使用重组枯草芽孢杆菌bacillussubtilis168/pht01-4gt的检测结果。
实施例2
具体过程如下:
分别以构建的重组大肠杆菌bl21(de3)/petduet-groes-groel-4gt和重组枯草芽孢杆菌bacillussubtilis168/pht01-4gt为发酵菌种。
将构建的重组大肠杆菌bl21(de3)/petduet-groes-groel-4gt和重组枯草芽孢杆菌bacillussubtilis168/pht01-4gt分别接种到到lb液体培养基(分别含有100μg/ml氨苄青霉素和5μg/ml氯霉素)中,置于恒温摇床上200rpm/min,37℃振荡培养8h,作为发酵用种子。
分别取2.5ml种子接种到添加了10mmol/l、20mmol/l、40mmol/l、80mmol/l、mmol/l氧化三甲胺和10mmol/l、20mmol/l、40mmol/l、80mmol/l脯氨酸的50mltb培养基中(分别含有100μg/ml氨苄青霉素和5μg/ml氯霉素)。
接种后,置于28℃200r·min-1条件下振荡培养4h,加入0.2mmiptg,诱导培养24h。
将发酵液进行菌体浓度以及4-α-糖基转移酶酶活检测(检测结果见表2)。
结果显示,当使用重组大肠杆菌bl21(de3)/petduet-groes-groel-4gt时,脯氨酸最佳浓度为10mm,此时酶活力为138.5u/ml;氧化三甲胺最佳浓度为20mm,此时酶活力为138.5u/ml。
当使用重组枯草芽孢杆菌bacillussubtilis168/pht01-4gt菌株时,脯氨酸最佳浓度为20mm,此时酶活力为23.1u/ml;氧化三甲胺最佳浓度为30mm,此时酶活力为30.5u/ml。
表2菌体浓度和酶活力检测结果
其中,a-d为使用重组大肠杆菌bl21(de3)/petduet-groes-groel-4gt的检测结果;e-h为使用为使用重组枯草芽孢杆菌bacillussubtilis168/pht01-4gt的检测结果的检测结
实施例3
具体过程如下:
以构建的重组大肠杆菌bl21(de3)/petduet-groes-groel-4gt为发酵菌种。
1、种子培养:将-80℃甘油管保存的重组大肠杆菌e.colibl21(de3)petduet-groes-groel-4gt接种到到lb液体培养基(含有100μg/ml氨苄青霉素)中,置于恒温摇床上200rpm/min,37℃振荡培养8h,作为发酵用种子。
2、发酵产酶:
分批发酵阶段:将种子液以8%接种量接入3l发酵罐发酵培养基(初始体积为1.1l)中,通过控制搅拌转速和通风量使溶氧维持20-30%,控制温度为37℃,流加质量浓度为25%的氨水控制ph7.0,待初始碳源耗尽,分批发酵阶段结束。该阶段培养时间6.5个小时;
补料发酵阶段:并开始以指数流加的方式补加3l发酵罐补料培养基,使菌体以0.15h-1的比生长速率进行生长,溶氧维持在20-30%,流加质量浓度为25%的氨水控制ph7.0;
诱导培养阶段:当菌体浓度达到od600=50时,降低温度至28℃,溶氧继续维持在20-30%,一次补加终浓度为0.2mm的iptg。同时溶氧控制在20-30%,控制ph7.0±0.5,诱导20小时左右,菌体密度及酶活力达到最高,此时发酵可以结束。
发酵培养结束后,离心获得菌体,菌体经过超声破碎后离心,得到超声破碎上清。
发酵培养结束后,离心获得菌体,菌体经过超声破碎后离心,得到超声破碎上清。通过在发酵不同时间取样,分别测定菌体干重和酶活力绘制得到菌体生长及产酶曲线(菌体生长及产酶曲线如图2)。
测定超声破碎上清液中的4-α-糖基转移酶酶活,并且折算成每毫升发酵液酶活力,最高活力为1302.6u/ml。
实施例4
具体过程如下:
以构建的重组枯草芽孢杆菌bacillussubtilis168/pht01-4gt为发酵菌种。
1、种子培养:将-80℃甘油管保存的重组枯草芽孢杆菌bacillussubtilis168/pht01-4gt接种到到lb液体培养基(含有5μg/ml氯霉素)中,置于恒温摇床上200rpm/min,37℃振荡培养8h,作为发酵用种子。
2、发酵产酶:
分批发酵阶段:将种子液以8%接种量接入3l发酵罐发酵培养基(初始体积为1.1l)中,通过控制搅拌转速和通风量使溶氧维持20-30%,控制温度为37℃,流加质量浓度为25%的氨水控制ph7.0,待初始碳源耗尽,分批发酵阶段结束。该阶段培养时间8个小时;
补料发酵阶段:并开始以指数流加的方式补加3l发酵罐补料培养基,使菌体以0.1h-1的比生长速率进行生长,溶氧维持在20-30%,流加质量浓度为25%的氨水控制ph7.0;
诱导培养阶段:当菌体浓度达到od600=30时,降低温度至30℃,溶氧继续维持在20-30%,一次补加终浓度为0.2mm的iptg。同时溶氧控制在20-30%,控制ph7.0±0.5,诱导20小时左右,酶活力达到最高,此时发酵可以结束。测定超声破碎上清液中的4-α-糖基转移酶酶活,并且折算成每毫升发酵液酶活力,最高活力为307.1u/ml。
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
序列表
<110>南京林业大学
<120>一种提高4-α-糖基转移酶可溶性表达水平的方法
<160>9
<170>patentinversion3.3
<210>1
<211>659
<212>prt
<213>人工序列
<400>1
metgluargileasnpheilepheglyilehisasnhisglnproleu
151015
glyasnpheglytrpvalphegluglualatyrasnargsertyrarg
202530
prophemetgluileleugluglupheproglumetlysvalasnval
354045
hispheserglyproleuleuglutrpileglugluasnlysproasp
505560
tyrleuaspleuleuargserleuilelysargglyglnleugluile
65707580
valvalalaglyphetyrgluprovalleualaalaileprolysglu
859095
aspargleuvalglnileglumetleulysasptyralaarglysleu
100105110
glytyraspalalysglyvaltrpleuthrgluargvaltrpglnpro
115120125
gluleuvallysserleuargglualaglyileglutyrvalvalval
130135140
aspasptyrhisphemetseralaglyleuserlysglugluleuphe
145150155160
trpprotyrtyrthrgluaspglyglygluvalilethrvalphepro
165170175
ileaspglulysleuargtyrleuilepropheargprovallyslys
180185190
thrileglutyrleugluserleuthrseraspaspproserlysval
195200205
alavalphehisaspaspglyglulyspheglyvaltrpproglythr
210215220
tyrglutrpvaltyrglulysglytrpleuarggluphepheaspala
225230235240
ilethrserasnglulysileasnleumetthrtyrserglutyrleu
245250255
serlysphethrproargglyleuvaltyrleuproilealasertyr
260265270
pheglumetserglutrpserleuproalalysglnalalysleuphe
275280285
valgluphevalgluglnleulysglugluglylyspheglulystyr
290295300
argvalphevalargglyglyiletrplysasnphephephelystyr
305310315320
progluserasnphemethislysargmetleumetvalserlysala
325330335
valargaspasnproglualaarglystyrileleulysalaglncys
340345350
asnaspalatyrtrphisglyvalpheglyglyiletyrleuprohis
355360365
leuargargthrvaltrpgluasnileilelysalaglnargtyrleu
370375380
lysprogluasnlysileleuaspvalasppheaspglyargalaglu
385390395400
ilemetvalgluasnaspglypheilealathrilelysprohistyr
405410415
glyglyserilephegluleuserserlysarglysalavalasntyr
420425430
asnaspvalleuproargargtrpgluhistyrhisgluvalproglu
435440445
alathrlysproglulysgluserglugluglyilealaserilehis
450455460
gluleuglylysglnileproglugluileargarggluleualatyr
465470475480
asptrpglnleuargalaileleuglnasphispheilelysproglu
485490495
gluthrleuaspasntyrargleuvallystyrhisgluleuglyasp
500505510
phevalasnglnprotyrglutyrglumetilegluasnglyvallys
515520525
leutrparggluglyglyvaltyralagluglulysileproalaarg
530535540
valglulyslysilegluleuthrgluaspglypheilealalystyr
545550555560
argvalleuleuglulysprotyrlysalaleupheglyvalgluile
565570575
asnleualavalhisservalmetglulysproglugluphegluala
580585590
lysgluphegluvalasnaspprotyrglyileglylysvalargile
595600605
gluleuasplysalaalalysvaltrplyspheproilelysthrleu
610615620
serglnserglualaglytrpasppheileglnglnglyvalsertyr
625630635640
thrmetleupheproileglulysgluleugluphethrvalargphe
645650655
arggluleu
<210>2
<211>1986
<212>dna
<213>人工序列
<400>2
catatggaacgtattaattttatttttggtattcataatcatcagccgttaggtaatttt60
ggttgggtgtttgaagaagcctataatcgtagctatcgcccgtttatggaaattctggaa120
gaatttccggagatgaaagttaatgttcattttagcggtccgctgttagagtggattgaa180
gaaaataagccggattatttggatctgctccgtagcctgattaaacgcggtcagttagaa240
attgttgttgcaggcttttatgaaccggtgctggccgccattccgaaagaagatcgctta300
gttcagattgaaatgctgaaagattatgcacgcaaactgggctatgatgccaaaggcgtt360
tggctgaccgagagggtgtggcagccggaactggttaaatcactgcgcgaagcaggcatt420
gaatatgtggttgtggatgattatcattttatgtcagcaggcctgagtaaagaagaactg480
ttttggccgtattataccgaagatggcggcgaagtgattaccgtgtttccgattgatgaa540
aaactgcgctatctgattccgtttcgcccggttaagaaaaccattgaatatctggaatct600
ctgacctcagatgatccgagcaaagttgcagtgtttcatgatgatggcgaaaaatttggt660
gtgtggccgggcacttatgaatgggtgtatgaaaaaggttggttacgcgaatttttcgat720
gccattacctctaacgagaaaattaatctgatgacctatagcgaatatctgtctaagttc780
accccgcgcggcttagtgtatctgccgattgcaagctattttgaaatgtcagagtggagt840
ctgccggcaaaacaggccaaactgtttgtggaatttgtggaacagctgaaagaagaaggt900
aaatttgaaaaatatcgtgtgtttgtgcgcggcggtatttggaaaaatttctttttcaaa960
tatccggaatctaattttatgcataaacggatgctgatggtgagtaaagcagttcgcgat1020
aatccggaagcacgcaaatatattctgaaagcacagtgtaatgatgcctattggcatggt1080
gtgtttggcggcatatacctgccgcatctgcgtcgtaccgtgtgggaaaatattattaaa1140
gcacagcgctatctgaaaccggaaaacaagattctggatgtggattttgatggtcgtgcc1200
gaaattatggtggaaaatgatggctttattgccaccattaaaccgcattatggtggtagt1260
atttttgaactgtctagtaaacgcaaagcagttaattataatgatgtgctgccgcgtcgt1320
tgggaacattatcatgaagtgccggaagccaccaaaccggaaaaagaatcagaagaaggc1380
attgcaagtattcatgaactgggcaaacagattccggaagaaattcgtcgcgaactggcc1440
tatgattggcagctgcgcgccattttgcaggatcattttattaaaccggaagaaacctta1500
gataattatcgcttagttaaatatcatgaactgggcgattttgttaatcagccgtatgaa1560
tatgaaatgattgaaaatggcgttaaactgtggcgcgaaggcggcgtgtatgcagaagaa1620
aaaattccggcccgagtggagaagaagatagaactgaccgaagatggctttattgccaaa1680
tatcgcgtgctgttagaaaaaccgtacaaagccctgtttggcgttgaaattaatctggca1740
gttcatagcgtgatggaaaaaccggaagaatttgaagccaaagaatttgaagttaatgat1800
ccgtatggtattggcaaagtgcgcattgaactggataaagccgccaaagtgtggaaattt1860
ccgattaaaaccctgtcacagagcgaagcaggttgggattttattcagcagggcgtgagc1920
tataccatgctgtttccgattgaaaaagaactggagtttaccgttcgctttcgcgaactg1980
aagctt1986
<210>3
<211>1984
<212>dna
<213>人工序列
<400>3
atgaatattcgtccattgcatgatcgcgtgatcgtcaagcgtaaagaagttgaaactaaa60
tctgctggcggcatcgttctgaccggctctgcagcggctaaatccacccgcggcgaagtg120
ctggctgtcggcaatggccgtatccttgaaaatggcgaagtgaagccgctggatgtgaaa180
gttggcgacatcgttattttcaacgatggctacggtgtgaaatctgagaagatcgacaat240
gaagaagtgttgatcatgtccgaaagcgacattctggcaattgttgaagcgtaatccgcg300
cacgacactgaacatacgaatttaaggaataaagataatggcagctaaagacgtaaaatt360
cggtaacgacgctcgtgtgaaaatgctgcgcggcgtaaacgtactggcagatgcagtgaa420
agttaccctcggtccaaaaggccgtaacgtagttctggataaatctttcggtgcaccgac480
catcaccaaagatggtgtttccgttgctcgtgaaatcgaactggaagacaagttcgaaaa540
tatgggtgcgcagatggtgaaagaagttgcctctaaagcaaacgacgctgcaggcgacgg600
taccaccactgcaaccgtactggctcaggctatcatcactgaaggtctgaaagctgttgc660
tgcgggcatgaacccgatggacctgaaacgtggtatcgacaaagcggttaccgctgcagt720
tgaagaactgaaagcgctgtccgtaccatgctctgactctaaagcgattgctcaggttgg780
taccatctccgctaactccgacgaaaccgtaggtaaactgatcgctgaagcgatggacaa840
agtcggtaaagaaggcgttatcaccgttgaagacggtaccggtctgcaggacgaactgga900
cgtggttgaaggtatgcagttcgaccgtggctacctgtctccttacttcatcaacaagcc960
ggaaactggcgcagtagaactggaaagcccgttcatcctgctggctgacaagaaaatctc1020
caacatccgcgaaatgctgccggttctggaagctgttgccaaagcaggcaaaccgctgct1080
gatcatcgctgaagatgtagaaggcgaagcgctggcaactctggttgttaacaccatgcg1140
tggcatcgtgaaagtcgctgcggttaaagcaccgggcttcggcgatcgtcgtaaagctat1200
gctgcaggatatcgcaaccctgactggcggtaccgtgatctctgaagagatcggtatgga1260
gctggaaaaagcaaccctggaagacctgggtcaggctaaacgtgttgtgatcaacaaaga1320
caccaccactatcatcgatggcgtgggtgaagaagctgcaatccagggccgtgttgctca1380
gatccgtcagcagattgaagaagcaacttctgactacgaccgtgaaaaactgcaggaacg1440
cgtagcgaaactggcaggcggcgttgcagttatcaaagtgggtgctgctaccgaagttga1500
aatgaaagagaaaaaagcacgcgttgaagatgccctgcacgcgacccgtgctgcggtaga1560
agaaggcgtggttgctggtggtggtgttgcgctgatccgcgtagcgtctaaactggctga1620
cctgcgtggtcagaacgaagaccagaacgtgggtatcaaagttgcactgcgtgcaatgga1680
agctccgctgcgtcagatcgtattgaactgcggcgaagaaccgtctgttgttgctaacac1740
cgttaaaggcggcgacggcaactacggttacaacgcagcaaccgaagaatacggcaacat1800
gatcgacatgggtatcctggatccaaccaaagtaactcgttctgctctgcagtacgcagc1860
ttctgtggctggcctgatgatcaccaccgaatgcatggttaccgacctgccgaaaaacga1920
tgcagctgacttaggcgctgctggcggtatgggcggcatgggtggcatgggcggcatgat1980
gtaa1984
<210>4
<211>17
<212>dna
<213>人工序列
<400>4
gccaggatccgaattcg17
<210>5
<211>20
<212>dna
<213>人工序列
<400>5
ggtatatctccttcttaaag20
<210>6
<211>42
<212>dna
<213>人工序列
<400>6
agaaggagatataccatgaatattcgtccattgcatgatcgc42
<210>7
<211>37
<212>dna
<213>人工序列
<400>7
aattcggatcctggcttacatcatgccgcccatgcca37
<210>8
<211>37
<212>dna
<213>人工序列
<400>8
ggatccatgaatattcgtccattgca26
<210>9
<211>37
<212>dna
<213>人工序列
<400>9
cccgggttacatcatgccgcccatgc26
- 还没有人留言评论。精彩留言会获得点赞!