GhPME36基因在调控植物次生壁发育中的应用的制作方法
本发明属于基因工程领域,具体涉及基因GhPME36在调控植物次生壁发育中的应用。
背景技术:
次生壁是细胞停止生长后,在初生壁内侧继续积累的细胞壁层。次生壁出现在初生壁之内,一般较厚且坚硬,常出现在起机械支持和输导作用的植物细胞中,如导管,管胞,厚壁细胞、纤维细胞等。棉纤维的主要结构是细胞壁,他的主要成分是纤维素和果胶类物质,果胶作为细胞壁的重要组成成分,其新陈代谢与纤维品质形成关系密切。而果胶甲酯酶是一种重要的果胶酶,广泛存在于植物和一些具有细胞壁降解作用的微生物中,通过产生可羧基基团或是去甲酯化的反应而改变细胞壁的结构,调控细胞壁的生长发育。近年来,人们从生理、生化、代谢等角度对细胞壁的发育进行了系统地研究,由于细胞壁尤其是次生壁发育的复杂性,传统方法对其研究具有局限性,随着分子生物学的发展,利用基因工程手段对细胞壁大于尤其是次生壁发育期开展研究,对植物的生长调控具有重要意义。
棉花是世界上重要的天然纤维作物之一,是纺织工业重要的原材料。随着纺织工业对棉纤维品质的要求愈来愈高,提高棉纤维品质尤其是纤维比强度的改良变得日益重要,已然成为棉花生物学领域的重要研究热点。由于棉花的纤维细胞是由单个细胞发育而来,细胞壁是整个细胞发育的关键,次生壁是纤维发育的关键时期,棉纤维发育的次生壁加厚期与棉纤维的比强度的形成关系密切,纤维比强度是纤维品质的重要指标,因此,对纤维次生壁加厚期的研究对纤维品质的改良具有重要意义。
技术实现要素:
本发明所要解决的技术问题是如何调控植物的生长发育,依据是次生壁中的果胶类物质在果胶甲酯酶的作用下发生变化,使细胞壁结构改变,从而影响细胞的生长发育,本发明GhPME36基因转入植物中后,使植物根长增加,抽薹开花时间延迟,叶片变长以及变宽,细胞的次生壁增厚,茎秆增粗,具有抗倒伏能力。
为解决上述技术问题,本发明提供了GhPME36基因在调控植物发育中的应用,所述GhPME36基因为SEQ ID No.1所示序列编码的基因。本发明首先提供了该基因家族的功能和结构进化分析编码该类物质的PME基因家族在调控植物生长发育中的应用,所述蛋白结构类型有两种,分别为为类型Ⅰ:包括21个α-螺旋、4个β-折叠或是25个α-螺旋、6个β-折叠,说明PME蛋白家族在三级结构上的高度保守性,推测PME家族在功能上有高度保守性。
为了说明上述问题,本发明还提供了一种测量总酶活的方法,以观察该酶在植物发育中的作用。
本领域的普通技术人员可采用已知方法,对本发明的基因GhPME36的核苷酸序列进行突变,那些经过人工修饰后得到的具有与本发明基因GhPME36的核苷酸序列75%或是更高同一性的核苷酸,只要编码基因GhPME36且具有基因GhPME36的功能,均是衍生于本发明的核苷酸序列且等同于本发明的序列。
上述应用中,所述植物可视为如下的任一种:
双子叶植物,单子叶植物,禾本科植物,十字花科植物,棉花,陆地棉,海岛棉,亚洲棉,拟南芥,野生型拟南芥Columbia。
上述应用中所述的植物发育为细胞壁发育或次生壁发育。
为解决上述问题,本发明还提供了一种培育转基因植物的方法
本发明所提供的培育转基因植物的方法,包括如下步骤:向受体植物中导入编码基因GhPME36,得到生长发育不同于所述受体植物的转基因植物。
上述方法中,所述“向受体植物中导入基因GhPME36”,可通过向受体植物中导入重组质粒实现。
所述重组质粒具体可视为重组质粒pBI121-GhPME36。
本发明具有以下有益效果:
本发明所涉及的PMEs基因,通过甲酯化反应使胞外pH下降,使细胞壁松弛的多种水解酶的活性被激发,如多聚半乳糖醛酸酶和果胶裂解酶等,果胶在这些酶的作用下被降解,导致分子结构被破坏,细胞壁松弛,促进细胞生长,本发明的GhPME36基因转入植物中后,使植物根长增加,抽薹开花时间延迟,叶片变长以及变宽,细胞的次生壁增厚,茎秆增粗,具有抗倒伏能力,通过调控植物次生壁的发育,影响棉花纤维强度的形成,对棉花纤维品质的研究具有重大的意义。
附图说明
图1为 4个挑选出来的优势表达的果胶甲酯酶基因和其直系同源基因对的荧光定量检测图。4个基因的相对定量以histone 3为参考基因,误差棒表示每个样品至少有三个技术重复的标准差;10 DPA 15 DPA 20 DPA 25 DPA 30 DPA: 开花后10、15、20、25和30天的纤维。
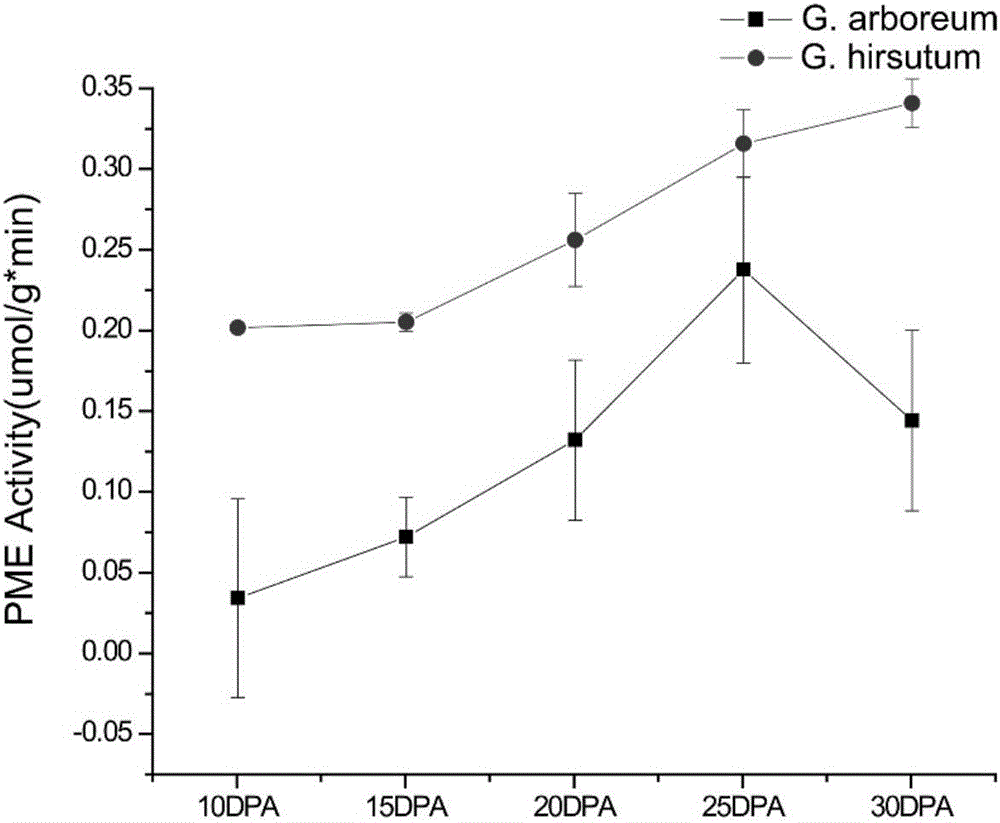
图2为棉纤维果胶甲酯酶酶活性测定图,分别选取开花后10、15、20、25和30天的纤维。
图3为 PCR扩增GhPME36基因的CDS区图,其中1为扩增得到的GhPME36基因CDS全长1368bp,M代表Marker2000。
图4为转基因拟南芥卡那抗性筛选图,图中的苗为所筛选的抗性苗。
图5为GhJAZ在转基因拟南芥中的表达情况图,1-4是转基因拟南芥,5-7是WT阴性对照,8是质粒阳性对照,M表示Marker2000。
图6为转基因株系GhPME36与野生型拟南芥的的表型比较,a表示根长增加,b和c分别表示抽薹开花时间延迟,d代表叶片增宽加长,e代表转基因植株抗倒伏。
图7为转基因株系GhPME36与野生型拟南芥的的细胞壁的电镜扫描图,f和g表示茎秆增粗,Bar=1cm。
具体实施方式
下面结合具体实施方法对本发明进行进一步的详细描述,给出的实例仅为了阐述本发明,而不是为了限制本发明的范围。
下述实例中,试验方法,如无特殊说明,均为常规方法;所用的材料、试剂等,如无特殊说明,均可以从商业途径得到;定量实验,均设置三次重复实验,结果取平均值;材料选择分别为开花后10、15、20、25、30天的棉花纤维组织,在文中简称为10DPA,15DPA等,以此类推。
TransScript All-in-One First-Strand cDNA Synthesis SuperMix for qPCR (One-Step gDNA Removal)、TransStart Tip Green qPCR SuperMix 为全式金公司产品;pMD-18T Vector、PrimeScript RT reagent kit with gDNA Eraser(Perfece Real Time)为Takara公司产品;植物多糖多酚RNA提取试剂盒为天根公司产品。
棉花品种海岛棉S-6、陆地棉69307、亚洲棉石系亚1号,雷蒙德氏棉记载在如下文献中:王中娜,白志川,商海红,袁有禄.亚洲棉NAC转录因子家族的生物信息学分析及相关NAC基因的克隆和功能研究,西南大学,硕士毕业论文,2015。公众可以从中国农业科学院棉花研究所获得该生物材料,该生物材料只为重复本发明的相关实验所用,不可作为其他使用用途。在下文中,陆地棉69307简称为69307、海岛棉S-6简称为s-6、亚洲棉石系亚1号简称为亚洲棉。
野生型拟南芥Columbia记载在如下文献中: A Gigon,AR Matos,D Laffray,Y Zuily-Fodil,AT Pham-Thi.Effect of drought stress on lipid metabolism in the leaves of Arabidopsis thaliana (ecotype Columbia).Annals of Botany,2004,94(3):345-351.
根癌农杆菌GV3101记载在如下文献中:肖卫民,赵名琛,邹敏,苏承刚,杜小兵.拟南芥受伤和接种根癌农杆菌GV3101对转录的影响.《农业生物技术学报》, 2013,21(5):537-545.
质粒pBI121记载在如下文献中:李伟.陆地棉GhAQP及GhNAC家族相关基因克隆、表达及功能初步研究.西南大学硕士论文,2013.
光暗交替培养即光培养和暗培养交替,具体培养周期具体可为:14小时光照培养/10小时黑暗培养。
实施例1、PME家族优势表达基因的实时荧光定量表达分析
1、根据转录组分析,在陆地棉、亚洲棉、雷蒙德氏棉中分别找到了11、5 、3个在纤维发育15 DPA (该时期属于次生壁加厚期) 优势表达的PMEs基因。
2、提取69307和亚洲棉纤维发育不同时期10DPA、15DPA、20DPA、25DPA、30DPA的纤维组织的RNA,反转录成第一条链cDNA,根据优势基因序列设计引物,进行Q-PCR扩增。
3、完成上述实验所使用的程序为95℃预变性15 s ;95℃ 变性5 s,60℃复性34 s,72℃延伸40 s (数据接收);(共40个循环)。
4、完成上述试验所用内参基因为Histon-3,引物序列为Histon-F: GAAGCCTCATCGATACCGTC,Histon-R:CTACCACTACCATCATGGC
5、上述每个反应均设置3次生物重复和3次技术重复,相对定量的差异表达分析采用2–ΔΔCT 法,该方法记载在如下文献中(Analysis of Relative Gene Expression Data Using Real-Time Quantitative PCR and the 2–ΔΔCT Method.Livak and Schmittgen, 2001: 25, 402-408.
6、定量表达的结果分析见图1。
7、结果证明:亚洲棉和陆地棉果胶甲酯酶基因的表达量在纤维发育过程中都有一个峰值,亚洲棉的峰值主要在纤维发育的20 DPA,陆地棉的峰值主要在25 DPA,而且在纤维发育25 DPA均表现为陆地棉高于亚洲棉,说明PME家族确实参与次生壁的发育过程。
实施例2、PME总酶活的测定
1、酶活的测定参照Hagerman的试验方法,该方法记载于如下文献中:Hagerman, A.E.A., P. J. (1986). Continuous spectrophotometric assay for plant pectin methylesterase. J. Agr. Food. Chem. 34, 440-444.
2、上述实验结果如图2。结果表明:棉花纤维发育不同时期果胶甲酯酶的活性存在差异,亚洲棉从纤维发育10 DPA到25 DPA果胶甲酯酶活性随发育时间的增加而逐渐增大,但30 DPA果胶甲酯酶的活性又有所降低。而在陆地棉中果胶甲酯酶的活性从10 DPA到30 DPA持续上升。
实施例3、GhPME36基因的克隆
本发明人从亚洲棉和69307中克隆出基因GhPME36。具体方法如下:
1、模板的获得:用试剂盒从亚洲棉和69307的10DPA、15DPA、20DPA、25DPA、30DPA中分别提取棉纤维的总RNA,然后利用PrimeScript RT reagent kit with gDNA Eraser(Perfece Real Time)反转录出第一链cDNA。
2、人工合成引物GhPME36-F:CGCGGATCCGCGGGCATTGGCACGTCTTCAAAT(下划线为限制内切酶BamHI的识别序列)和GhPME36-R:CGAGCTCGTAAAGCCCTGAAGTGAAAGGC (下划线为限制内切酶SacI的识别序列)。
3、完成步骤1和2后,以步骤1得到的cDNA为模板,以步骤2合成的GhPME36-F和GhPME36-R为引物进行PCR扩增,反应程序:94℃,预变性5min;94℃30s,58℃30s,72℃2min,30个循环;72℃,10min,实验结果见图3,结果得到约1368bp的双链DNA分子,然后进行纯化、回收,得到DNA片段。
4、将步骤3得到的双链DNA分子连接至pMD18TM(simple)T载体,得到重组质粒pMD18-GhPME36。
5、经测序,重组质粒pMD18-GhPME36含有序列表中的序列1所示的DNA分子。
实施例4、转GhPME36基因植株的获得和表型鉴定
一、重组质粒pBI121-GhPME36的构建
1、用限制性内切酶BamHI和SacI双酶切重组质粒pMD18-GhPME36,回收纯化得到DNA片段。
2、用限制性内切酶BamHI和SacI双酶切质粒pBI121,回收约13K的载体骨架。
3、将步骤1和2得到的DNA用T4连接酶16℃过夜连接,得到重组质粒pBI121-GhPME36。
4、根据测序结果,对重组质粒pBI121-GhPME36进行描述如下:向质粒pBI121的限制性内切酶BamHI和SacI酶切位点之间插入核苷酸序列是表中序列1所示的DNA分子。
二、转GhPME36基因植株的获得
1、将重组质粒pBI121-GhPME36导入根癌农杆菌GV3101,得到重组农杆菌。
2、采用拟南芥花序浸花转化法(记载于如下文献中:高建强,梁华,赵军。植物遗传转化农杆菌浸花法研究进展,中国农学通报,2010,26(16):22-25),将步骤1得到的重组农杆菌转至野生型拟南芥中,获得T1代拟转GhPME36基因拟南芥种子。
3、将步骤2获得的T1代拟转基因拟南芥种子播种于含有50mg/L卡那霉素的MS培养基上,能够正常生长的拟南芥(抗性苗)即为T1代拟转GhPME36基因的阳性苗,见图4,T1代拟转GhPME36基因阳性苗收到的种子即为T2代拟转GhPME36基因拟南芥种子。
4、将步骤3筛选得到的不同株系的T2代拟转GhPME36基因的拟南芥种子播种于含有50mg/L卡那霉素的MS培养基上进行生长,如果某株系中能够正常生长的拟南芥(抗性苗)数量与不能够正常生长的拟南芥(非抗性苗)的数目比例为3:1,则该株系为GhPME36基因插入一个拷贝的株系,该株系中的抗性苗收到的种子即为T3代代纯合转GhPME36基因拟南芥。
5、将步骤4筛选到的T3代拟转GhPME36基因的拟南芥种子播种于含有50mg/L卡那霉素的1/2MS培养基上进行筛选,均为抗性苗的即为T3代纯合拟转GhPME36基因的拟南芥。将其中2个T3代纯合拟转GhPME36基因的拟南芥株系分别命名为L1和L2。
三、转空载拟南芥的获得
按照上述步骤二的方法,将重组质粒pBI121-GhPME36替换为pBI121,其他步骤均相同,得到T3代纯合拟空载体拟南芥,简称拟转空载体拟南芥。
四、分子检测
以野生型拟南芥、拟转空载体拟南芥的T3代植株、L1的T3代植株或L2的T3代植株的基因组DNA为模板,以实施例3中人工合成的引物GhPME36-F和GhPME36-R为引物,进行PCR扩增。
PCR扩增的实验结果见图5,结果表明,以L1的T3代植株或L2的T3代植株的基因组DNA为模板,均能扩增得到1368bp的条带;而以野生型拟南芥和拟转空载体拟南芥的基因组DNA为模板,则均不能扩增得到1368bp的条带。因此,L1的T3代植株和L2的T3代植株均鉴定为T3代纯合转GhPME36基因拟南芥,拟转空载体拟南芥鉴定为转空载体拟南芥。
五、转GhPME36基因拟南芥的表型鉴定
1、根长的鉴定
实验重复三次取平均值,每个株系重复30粒种子。
取野生型拟南芥种子、转空载体拟南芥的T3代种子,L1的T3代种子,播种与MS培养基上,20℃光暗交替培养两周,统计根长。
实验结果见图6。根长结果鉴定如下:GhPME36转基因株系与对照野生型比较,根长增加了19.5%。
2、抽薹早晚的鉴定
实验重复三次取平均值,每个株系重复30粒种子。
取野生型拟南芥种子、转空载体拟南芥的T3代种子,L1的T3代种子,播种与MS培养基上,20℃光暗交替培养两周,记录抽薹时间。
实验结果见图6。GhPME36抽薹时间晚于野生型约一周。
3叶片宽度的鉴定
实验重复三次取平均值,每个株系重复30粒种子。
取野生型拟南芥种子、转空载体拟南芥的T3代种子,L1的T3代种子,播种与MS培养基上,20℃光暗交替培养,测量30 天大小幼苗的第1-、3-、5-、7-、9-、11-片莲座叶的叶长和叶宽。
实验结果见图6。GhPME36的叶长比野生型拟南芥的高度分别高了8%、70%、76%、57%、28%和11.5%;GhPME36的第1-、3-、5-、7-的叶宽分别比野生型拟南芥宽了25%、26%、78%和60%。
4、茎秆直径的鉴定
实验重复三次取平均值,每个株系重复30粒种子。
取野生型拟南芥种子、转空载体拟南芥的T3代种子,L1的T3代种子,播种与MS培养基上,20℃光暗交替培养,各取茎秆进行荧光电镜扫描。
实验结果见图7。GhPME36植株的茎秆粗于野生型5%。
<110>中国农业科学院棉花研究所
<120> GhPME36基因在调控植物次生壁发育中的应用
<160>1
<210> 1
<211> 1368
<212> DNA
<400> 1
ATGGTGAAATCCATCCTCGACTCCTCCAAAGGCAACCTCAACCGCTCTAA
CGCCGCCGCCATCTGTCTCAACATCCTTTCTAATTCTGATTACCGCATAA
GATCGACCAATGACGCTCTGGCACGTGGCAAGATCAAGGACGCTCGTGCG
TGGATGAGCGCGGCCCTTTGCTACCAATACGATTGTTGGAGCGCGCTCAA
GTACGTAAATGACACCAAATTGGTCGGTGAAACGATGGCGTTTTTGGACT
CTTTAACGCAACACAGCAGCAACGCGTTGAGCATGATGGTCGCCTACGAT
AATAACGGGGAGGACATAGCCGCGTGGGTCCCTCCCAAGACGGAACGGGA
CGGGTTCTACGAGAACGGGTCGGGTGGGACGGAGCTGGGGTTTAACGGGG
GTTTACCGTCAAACTTGAAGACGGACGTAACGGTGTGTAAAGACGGGAGC
GGAGGGTGTTACAAGACAGTGCAAGAGGCCGTCAACACAGCACCGGCAAA
TGCAGAGAGTACACGCCGTTTCGTGATATATATAAAGGAAGGTGTGTACG
AAGAAACGGTGAGGGTGCCACTGGAGAAGAAAAATGTGGTCTTTTTGGGG
GATGGAATGGGTAAAACCATCATTACGGGTGCTTTAAATGCAGGGATGCC
TGGACTTACCACTTATGAGACTGCTACTGTCGGGGTTCTTGGGGATGGAT
TTATGGCCAGTGGACTCACAATCCGGAACACAGCAGGCCCTGATGCCCAC
CAAGCCGTAGCCTTTAGATCAGATAGTGATCTTTCTGTCATTGAGAACTG
TGAATTCCTAGGCAACCAAGATACTCTCTACGCTCACTCCCTCCGCCAGT
TCTACAGGAAGTGCCGTATTCAGGGGAACGTGGACTTCATCTTCGGGAAT
TCTGCCTCGGTGTTCCAAGATTGTGAAATTTTGGTTGCTCCCCGGCAGGT
AAAACCCGAAAAAGGTGAGAACAATGCTGTGACAGCTCATGGTAGAACTG
ATCCTGCTCAATCGACTGGTCTGGTTTTCCAGAACTGCTTGATCAACGGA
ACTGATGAATACATGAGATACTATTATAGCAAGCCTAAAGTGCACAAGAA
CTTTTTGGGAAGGCCATGGAAGGAATATTCAAGGACAGTTTTTATAAATT
GTGTTATGGAAGCACTTATTAATCCAAATGGATGGATGCCATGGAAGGGT
GACTTTGCATTGAAAACACTCTTCTATGGAGAATTTGGGAATTCTGGTCT
TGGATCCATTCCATTGAACAGAGTTCCATGGAGTACTCAAATTCCACCCC
AGCATGTACATACATTTTCAGTCCAAAATTTCATTCAAGGAGATCAATGG
ATTCCAACATCATCTTGA
- 还没有人留言评论。精彩留言会获得点赞!
- 拟南芥SSCD1基因突变在调控植物体内茉莉酸合成中的应用的制作方法与工艺
- 一种基因调控网络的优化控制方法与流程
- 拟南芥WRKY家族转录因子AtWRKY44基因,蛋白编码序列及其应用的制作方法与工艺
- 针对免疫调控能力相关基因个体化配方食品及其制备方法与流程
- REVOLUTA基因在调控豆科植物小叶片数目和叶茎比中的应用的制作方法与工艺
- 玉米锌铁调控转运体ZmZIP3基因及其应用的制作方法与工艺
- 拟南芥基因SPOC1在调控植株开花期中的应用的制作方法与工艺
- 调控石榴花胚珠发育的PgAG基因及其用途的制作方法与工艺
- 光效调控基因HPE1及其应用的制作方法与工艺
- 菘蓝IiAP2/ERF049基因在调控木脂素类化合物合成中的应用的制作方法与工艺